随着分子生物学及基因工程技术的飞速发展,转基因技术成为研究基因功能和植物改良的重要途径,高效遗传转化体系的建立便成为影响物种间功能基因发掘及研究利用的限制条件。目前,在植物遗传转化中普遍应用的转化方法主要有农杆菌介导法和DNA直接导入法。农杆菌介导法具有操作简单、成本低、转化频率高等优点,但需依赖于物种高效的再生体系,存在再生周期长等缺点[1-2]。DNA 直接导入法主要包括基因枪轰击法以及花粉管通道法。其中基因枪轰击法受体类型广泛,无物种限制,但存在转化频率低、价格昂贵以及后代遗传不稳定等缺点[1-2]。花粉管通道法是由我国科学家周光宇[3]于1979年首先提出和设计的,与其他方法相比,该法操作简便,无需昂贵的仪器和化学药品,注射后直接收获种子,无须经过组织培养再生,且转化周期短,其首先在棉花上获得成功应用[4]。
花生属于比较难于转化的作物,目前花生遗传转化的方法主要有农杆菌介导法和花粉管通道法,使用的外植体有胚小叶、子叶节、胚轴、幼叶等[5-8]。王凤欢等[9]对农杆菌介导的花生胚小叶遗传转化体系进行优化,使抗性丛生芽诱导率可达到15%~93%。贾宇臣等[10]以子叶节为外植体进行研究,获得最佳转化条件。邱金梅等[11]利用农杆菌EHA105菌株侵染花生胚轴外植体,认为外植体在真空中侵染利于转化。钟育海等[12]研究认为在农杆菌菌液中添加烟草提取物,以及超声波处理,可使子叶外植体的分化率达到86%。花粉管通道法最初用于外源DNA的转移,如申馥玉等[13]和梁炫强等[14]提取高抗锈病的花生野生种Arachis glabrata基因组DNA,通过花粉管通道法导入栽培品种白沙1016中,结果获得抗锈病性及其他性状明显转移的株系。王亚等[15]、范乾程等[16]通过花粉管通道法将携带外源基因的重组农杆菌导入花生,PCR检测阳性率分别为34.7%和18.0%。
CRISPR-Cas9 技术是最新发展的基因编辑技术,可以对特定DNA 进行定点切割,并进一步引起靶基因的定点突变。运用 CRISPR/Cas9 技术已经在水稻、小麦、玉米三大作物上实现了高效精确的单碱基定点突变[17],并显著提高了水稻白叶枯病抗性[18]。但基因编辑同样要依赖于成熟高效的遗传转化体系,由于基因编辑是在外源质粒成功转化的基础上发生,所依赖的遗传转化体系需要具有更高的转化效率,方可提高编辑的效率。因此,简单高效遗传转化体系的建立也成为基因编辑有效利用的途径。
花生是重要的油料作物,花生油富含多种脂肪酸,主要包括饱和脂肪酸和不饱和脂肪酸,分别占到总脂肪酸含量的20%和80%。由于饱和脂肪酸被认为对人体有害,故降低饱和脂肪酸含量成为花生育种的目标之一。高等植物中的FatB(acyl-ACP thioesterase, 酰基-ACP 硫脂酶)主要编码生成饱和脂酰碳链的硫酯酶,催化饱和脂肪酸的合成释放。采用RNA干涉技术沉默FatB基因的表达使棉籽油中的C16∶0含量从最初的20%下降到8%;过表达C12∶0-ACP硫酯酶的转基因拟南芥和油菜中月桂酸含量显著增加[19]。在亚麻荠中将FAD2基因突变则获得油酸含量由16%增加到50%的籽粒[20]。因此,通过调控FatB的催化活性,可以对花生籽粒饱和脂肪酸的合成进行调控。
本研究拟构建花生AhFatB基因的CRISPR/Cas9编辑载体,并通过农杆菌介导的花粉管注射法转化花生,在成功转化的基础上获得AhFatB位点被成功编辑的花生籽粒,进一步筛选出饱和脂肪酸含量降低的突变材料,旨在为花生基因编辑的有效展开提供依据和参考。
1 材料和方法
1.1 试验材料
1.1.1 植物材料 花生品种为花育23号,由青岛农业大学花生研究中心保存。
1.1.2 菌株、质粒、载体和试剂药品 大肠杆菌(Escherichia coli)DH5α、根癌农杆菌(Agrobacterium tumefaciens)GV3101购自上海唯地生物技术有限公司(Veidi,中国上海)。CRISPR/Cas9载体构建试剂盒购自杭州百格生物技术有限公司(Biogle,中国杭州)。卡那霉素(Kanamycin)、利福平(Rifampin)、乙酰丁香酮(AS)、MES、MgCl2·6H2O、质粒提取试剂盒均购自生工生物工程(上海)股份有限公司(Sangon,中国上海)。2×Taq Plus Master MixⅡ(Dye Plus)购自南京诺唯赞生物科技有限公司(Vazyme,中国南京)。
1.1.3 引物设计与合成 试验所用引物及其序列列于表1。引物由生工生物工程(上海)股份有限公司合成。CS4-F/CS4-R用于Cas9基因的扩增及测序;A1-F/A1-R用于Aradu.ZG6C0基因的扩增及测序;B1-F/B1-R用于Araip.SBA9Y基因的扩增及测序;U6-F用于靶位点测序;Oligo1、Oligo2用于构建重组载体。
表1 引物及序列
Tab.1 Primers and sequences used in experiments

1.1.4 培养基及侵染液 LB液体培养基+50 mg/L 卡那霉素;YEB液体培养基+50 mg/L卡那霉素+50 mg/L利福平;侵染液:MS液体培养基+100 μmol/L 乙酰丁香酮+10 mmol/L MES+10 mmol/L MgCl2·6H2O。
1.2 试验方法
1.2.1 CRISPR/Cas9编辑载体的构建 设计花生AhFatB编辑位点,根据靶位点合成1对Oligos(Oligo1和 Oligo2,表1),使用杭州百格生物技术有限公司CRISPR/Cas9载体试剂盒,将合成的编辑位点DNA Oligo通过Buffer Anneal连接到PX458载体上(图1),转化大肠杆菌DH5α,挑选单克隆菌落,利用CS4-F和CS4-R引物进行PCR扩增验证,对扩增阳性的菌落利用U6启动子引物U6-F进行测序验证。
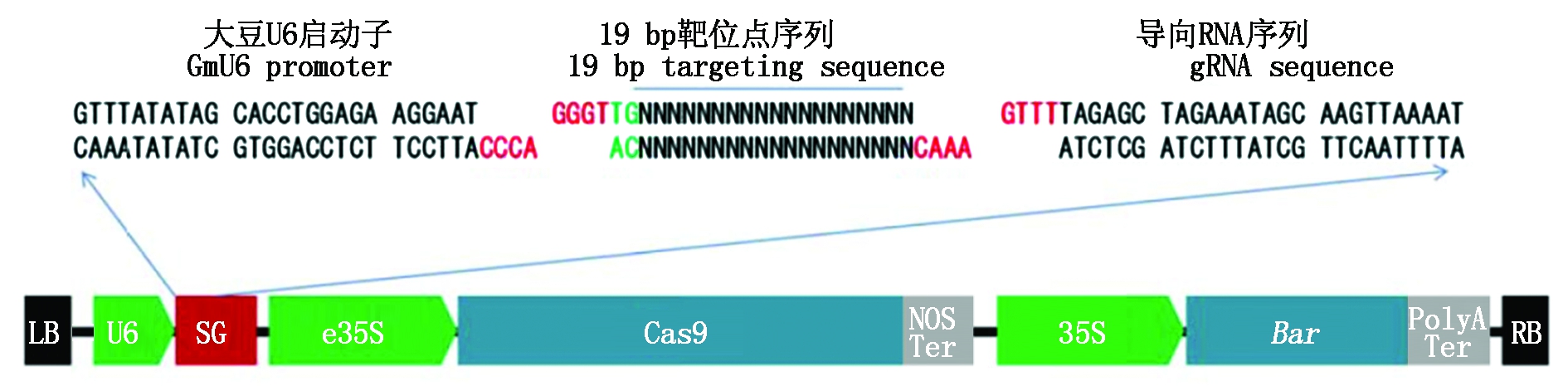
LB.T-DNA左侧;RB.T-DNA右侧;U6.大豆U6启动子;SG.sgRNA(导向RNA);e35S.35S强启动子; Cas9.优化
过的Cas9;NOS Ter.NOS终止子;35S.花椰菜花叶病毒的35S启动子;Bar.Bar选择标记基因;PolyA Ter.PolyA终止子。
LB.Left border of T-DNA; RB. Right border of T-DNA; U6.Soybean U6 promoter; SG.sgRNA; e35S.Enhanced 35S promoter;
Cas9.Optimized Cas9; NOS Ter.NOS terminator; 35S.CaMV 35S promoter; Bar.Bar selection marker gene;PolyA Ter.PolyA terminator.
图1 PX458-Cas9载体
Fig.1 Vector of PX458-Cas9
1.2.2 重组质粒转化农杆菌 取-80 ℃冰箱保存的农杆菌于超净工作台室温融化,取100 μL加入2 μL质粒DNA,用手弹打管底混匀,依次于冰上静置5 min,液氮5 min,37 ℃水浴5 min,冰浴5 min;加入600 μL的无抗生素的YEB液体培养基,于28 ℃摇床200 r/min振荡培养3 h;6 000 r/min离心1 min,弃上清液,加入100 μL的无抗生素的YEB液体培养基,枪头吸打混匀,吸取100 μL的菌液涂布于含有50 mg/L卡那霉素和50 mg/L利福平的YEB平板上,倒置培养3 d;挑单克隆进行菌落PCR扩增验证,保存阳性克隆菌液。
1.2.3 注射浸花法转化花生 将花育23号成熟种子进行催芽处理后播种于花盆中,每盆4粒,出苗后,保留长势良好的2棵植株。于花生始花期开始每天早晨摘去尚未完全开放的花朵,持续7~10 d使其达到盛花期时开始注射。注射前1 d将保存在冰箱中的农杆菌菌液取出,吸取200 μL转入含有50 mg/L卡那霉素和利福平的YEB培养基中,28 ℃,200 r/min,摇菌约10~12 h后,取适量菌液于紫外分光光度计中测量OD600值,待 OD600=0.6~0.8时,先取1 mL菌液保存在4 ℃冰箱待次日摇菌备用,剩余菌液经离心后收集沉淀,加入提前制备好的等体积侵染液,充分混匀后得到菌体注射液。注射于当日8:00之前进行,使用1 mL的医用注射器吸取注射液注入开放花朵的龙骨瓣内(或者花萼管),连续注射10 d,之后再连续摘花7~10 d,对新生果针进行绑绳标记。待荚果发育成熟后收获绑绳标记的花生荚果。
1.2.4 收获籽粒的分子鉴定 取注射后收获的花生荚果籽粒,使用SDS法提取基因组DNA为模板[21],以CS4-F和CS4-R为引物进行PCR扩增检测。扩增体系为:总体积25 μL,包括正反向引物各10 μmol/L,2×Taq Plus Master Mix Ⅱ 12.5 μL,DNA模板50 ng。扩增条件为:95 ℃预变性5 min;然后进行35次循环:95 ℃变性40 s,64 ℃退火40 s,72 ℃延伸1 min;最后72 ℃延伸10 min;4 ℃保存。1%琼脂糖凝胶电泳检测PCR扩增结果,对检测的阳性籽粒DNA,再用A1-F/A1-R及B1-F/B1-R引物进行扩增后测序,验证靶位点编辑情况。
2 结果与分析
2.1 CRISPR/Cas9编辑载体的构建
以转化后的单克隆大肠杆菌为模板,CS4-F、CS4-R为引物进行PCR扩增,扩增出产物为1 249 bp的条带(图2-A),说明重组质粒已成功转入。再取部分阳性克隆送公司测序,测序结果含有靶位点DNA序列的为阳性克隆(图2-B)。对测序成功的重组载体命名为PX458-Cas9-FatB。
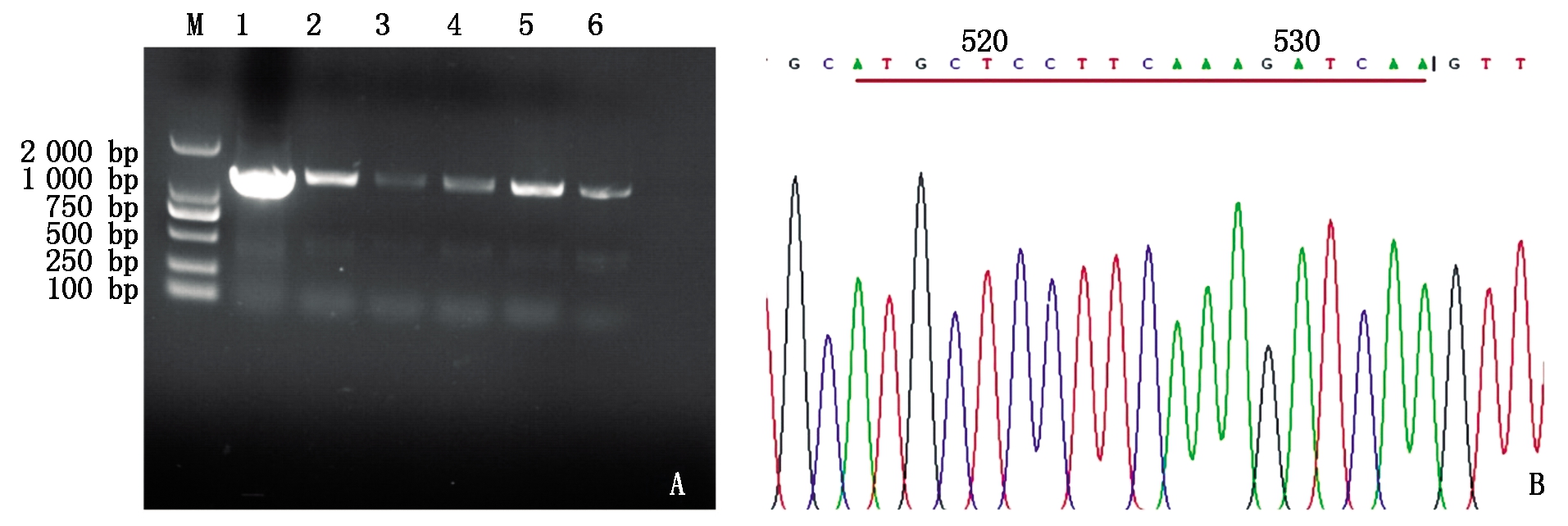
A.重组质粒的PCR扩增验证:M.DL2000;1.阳性对照(1 249 bp);2-6.单克隆。B.重组质粒的测序峰图,横线部分为靶位点序列。
A. PCR amplification verification of recombinant plasmid:M.The molecular weight Marker of DL2000;1. Positive control (1 249 bp);
2-6. Every monoclonal. B. The sequencing peak map of the recombinant plasmid, with the horizontal line as the target sequence.
图2 重组载体PX458-Cas9-FatB的构建
Fig.2 Construction of recombinant vector PX458-Cas9-FatB
2.2 重组质粒转化农杆菌
挑选在含有50 mg/L卡那霉素和50 mg/L利福平的YEB平板上的单克隆菌落,利用CS4-F、CS4-R引物进行菌落PCR扩增验证,能扩增出1 249 bp目的片段的均为阳性菌落,说明重组质粒已成功转入农杆菌(图3)。
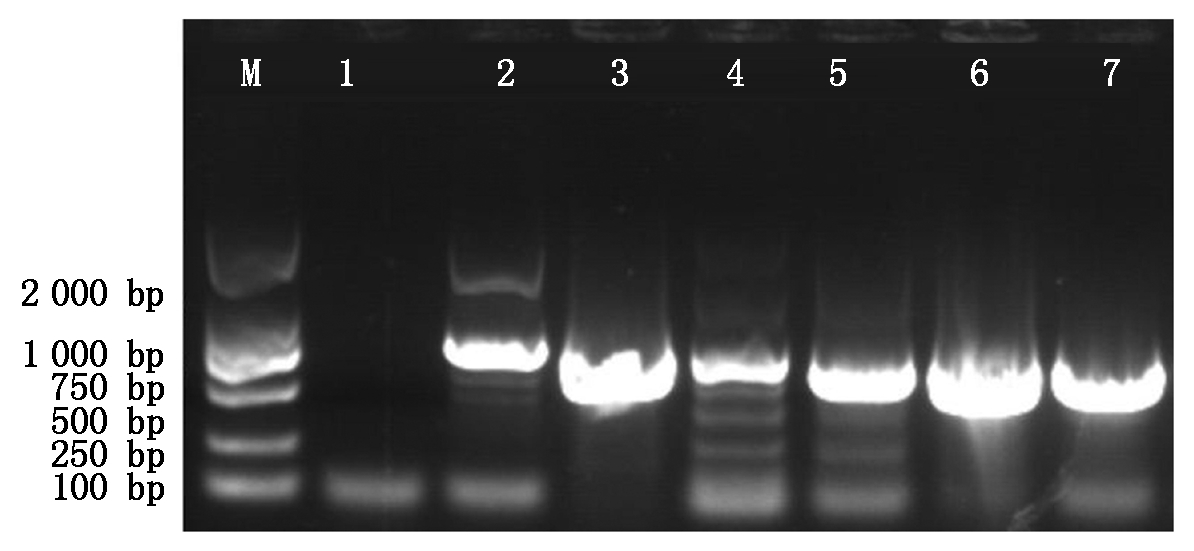
M.DL2000;1.空白对照;2.阳性质粒对照;3-7.农杆菌单克隆。
M.The molecular weight Marker of DL2000; 1.Blank control;
2.Positive plasmid control; 3-7.Agrobacterium
tumefaciens monoclonal.
图3 琼脂糖凝胶电泳检测农杆菌阳性克隆
Fig.3 Detection of Agrobacterium tumefaciens positive clones by agarose gel electrophoresis
2.3 注射浸花法转化花生
使用1 mL的无菌注射器吸取农杆菌注射液,每次吸取之前先将离心管中的注射液摇匀。选择当日盛开的花朵(图4-A),从花生花瓣的龙骨瓣处进行注射,一只手拖住花瓣,另一只手持注射器扎破龙骨瓣进行注射(图4-B),直到花萼管中充满水柱,并且打开翼瓣后看到注射液浸满整个花朵(图4-C)。每朵花大约注射0.1 mL注射液,每天6:00-8:00进行注射,持续7~10 d。随后对新生果针进行绑绳标记(图4-D)。待荚果发育成熟后收获绑绳标记的花生荚果(图4-E、F)。
2.4 注射后收获籽粒的分子鉴定
图5结果显示,注射后收获的籽粒中,能成功扩增出了相应目的条带的籽粒含有外源重组质粒,未能扩增出相应条带的为转基因阴性籽粒。在被检测的274粒籽粒中,共检测获得108粒阳性籽粒,转基因阳性率为39.42%。对扩增阳性的108粒籽粒进行测序验证,发现有1粒在靶位点外发生了编辑,在部分阳性籽粒的自交后代中,发现了靶位点发生编辑的籽粒。

A.待注射的花朵;B.注射龙骨瓣;C.侵染液包满雄蕊且在花萼管有一段水柱;D.尼龙绳捆绑果针;E.收获植株;F.收获的荚果和籽粒。
A.Flowers to be injected; B.Carina injected; C.Infection medium covered with stamens and a column of water in the calyx tube;
D. Pod needles tied with nylon cord; E.Harvested plants; F.Harvested pods and seeds.
图4 注射浸花法转化花生
Fig.4 Peanut transformation by injection soaking flower method
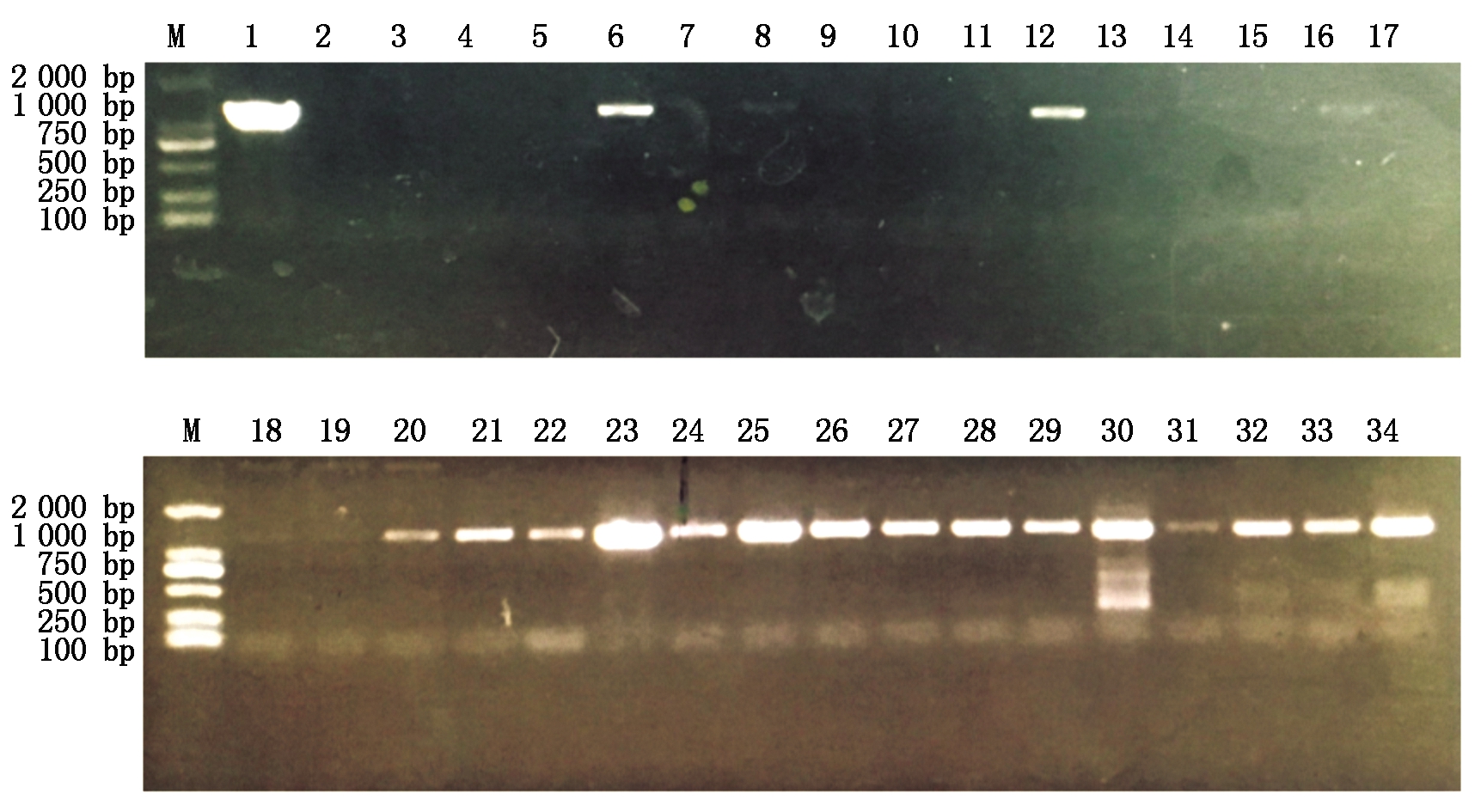
M.DL2000;1.阳性质粒对照;2.空白对照;3.非转基因对照;4-34.转化后收获的籽粒。
M.The molecular weight Marker of DL2000;1.Positive plasmid control;2.Blank control;3.Non-transgenic
control;4-34. Seeds harvested after transformation.
图5 转化后收获籽粒的PCR扩增结果(目的条带为1 249 bp)
Fig.5 Results of PCR amplification of harvested kernels after transformation (target band is 1 249 bp)
3 结论与讨论
利用浸花法进行遗传转化在拟南芥以及油菜等植物上获得了巨大成功[22-23]。花生属于较难转化的作物,青岛农业大学花生研究中心十多年来坚持对该法进行摸索优化,建立了简单高效的注射浸花法遗传转化技术。根据以往经验,影响转化效率最关键的因素是农杆菌的活力,因此,注射液必须要现用现配,具体为注射前1 d晚上摇菌,第2天早上离心后用侵染液重悬菌体,用于当日注射效果最好,第2日注射需要第2日早上再重新配置注射液,如使用第1日的注射液,转化成功率会降低50%~80%。另外在注射过程中,直接注射花萼管虽然可行,但因花萼管较细长,直接用注射器扎破后容易造成花萼管受损折弯,影响受精过程进而降低转化效率。因此,应该尽量选择注射龙骨瓣,注射龙骨瓣直到雄蕊被浸透,农杆菌可随着花粉管通道直接进入胚囊而提高转化效率。有研究者直接利用花粉管注射法转化外源DNA也能达到很好的转移效果,但由于同时导入大片段的序列而引起受体很多基因及性状的改变,而直接注射农杆菌可以保证外源基因的单一进入而增加了转化后代的定向选择效果。因此,使用注射浸花法,将携带外源基因的重组农杆菌通过花粉管通道完成整个转化过程,与其他的遗传转化方法相比较,该法不需要依赖其他设备,操作简单,可以大大降低成本和节约时间。利用此法将花生nsLTPs、Lea-D、WRKY75等基因导入花生,分别获得35%~56%的阳性转化率[24-25]。如果能够更好地控制摘花时间,而且即时标记果针,预计可提高转化率到70%以上。本研究首次使用该转化方法进行CRISPR/Cas9基因编辑,虽然产生较高的阳性籽粒(39.42%),但是经测序结果验证,只有1粒在靶位点外发生编辑,说明应用此法未能实现高效率的基因编辑。有报道说非同源性的启动子会抑制编辑效率[26],本研究使用了大豆的U6启动子对花生进行了编辑,这种非同源性的差异可能是造成编辑效率低的原因之一,目前正在尝试用花生的启动子来替代大豆U6启动子来提高编辑效率。此外,FatB基因在植物的发育过程中起到非常重要的作用[19-20],基因被编辑失活可能会影响到种子的正常发育而被选择淘汰,因而显示出极低的编辑效率。高效的注射浸花法转化技术是否能更有效地用于花生基因编辑,仍需进一步的研究证实。
[1] 苗利娟, 黄冰艳,张新友,董文召,刘娟,秦利,张俊,齐飞艳,石磊. 花生组培再生及农杆菌介导遗传转化研究进展[J]. 中国农学通报, 2017, 33(32): 15-20.doi:10.11924/j.issn.1000-6850.casb16090137.
Miao L J, Huang B Y,Zhang X Y,Dong W Z,Liu J,Qin L,Zhang J,Qi F Y,Shi L.Tissue culture regeneration and Agrobacterium mediated transformation of peanut:research progress[J]. Chinese Agricultural Science Bulletin, 2017, 33(32): 15-20.
[2] 简纯平, 李开绵, 欧文军.花粉管通道法转基因育种研究进展[J].热带作物学报, 2012, 33(5): 956-961. doi:10.3969/j.issn.1000-2561.2012.05.033.
Jian C P, Li K M, Ou W J.Research progress in pollen-tube pathway method in transgenic plants[J].Chinese Journal of Tropical Crops, 2012, 33(5): 956-961.
[3] 周光宇. 从生物化学的角度探讨远缘杂交的理论[J]. 中国农业科学, 1978, 11(2): 16-20.
Zhou G Y.Discussion on the theory of interspecific hybridization from the biochemical perspective[J]. Scientia Agricultura Sinica,1978, 11(2): 16-20.
[4] 周光宇, 陈善葆, 黄骏麒. 农业分子育种研究进展[M]. 北京:中国农业科技出版社, 1993:154-156.
Zhou G Y, Chen S B, Huang J L. Advances in molecular breeding research of agricuiture[M]. Beijing: China Agriculture Science and Technology Press, 1993:154-156.
[5] 王旭达, 于树涛, 张高华, 王鹤, 丰明, 都兴范, 范琦, 于国庆. 农杆菌介导花生转化体系的优化及转化AlDREB2A基因花生的耐旱性研究[J]. 中国农业大学学报, 2018, 23(7): 26-35. doi:10.11841/j.issn.1007-4333.2018.07.04.
Wang X D, Yu S T, Zhang G H, Wang H, Feng M, Du X F, Fan Q, Yu G Q. Optimization of Agrobacterium tumerfaciens mediated peanut transformation system and studies on the drought tolerance of transgenic AlDREB2A peanut[J].Journal of China Agricultural University, 2018, 23(7): 26-35.
[6] 朱军, 韩锁义, 袁美, 贺梁琼, 何国浩, 黄家权. 农杆菌介导的花生遗传转化条件优化[J]. 中国油料作物学报, 2018, 40(2): 191-198. doi:10.7505/j.issn.1007-9084.2018.02.004.
Zhu J, Han S Y, Yuan M, He L Q, He G H, Huang J Q. Optimization of Agrobacterium tumefaciens-mediated transformation in peanut[J]. Chinese Journal of Oil Crop Sciences, 2018, 40(2): 191-198.
[7] Swathi A T,Divya K,Jami S K,Kirti P B.Transgenic tobacco and peanut plants expressing a mustard defensin show resistance to fungal pathogens[J].Plant Cell Reports,2008, 27(11): 1777-1786.doi:10.1007/s00299-008-0596-8.
[8] Tiwari S, Tuli R. Factors promoting efficient in vitro regeneration from de-embryonated cotyledon explants of Arachis hypogaea L.[J]. Plant Cell Tissue and Organ Culture, 2008, 92(1):15-24. doi: 10.1007/s11240-007-9297-1.
[9] 王凤欢,冯滢,马旭策,杨晓欣,张乐. 农杆菌介导花生胚小叶遗传转化体系的优化研究[J]. 河南农业, 2019(17): 23-25. doi:10.15904/j.cnki.hnny.2019.17.012.
Wang F H, Feng Y, Ma X C, Yang X X, Zhang L. Optimization of genetic transformation system of peanut embryo leaflets mediated by Agrobacterium tumefaciens[J]. Agriculture of Henan, 2019(17): 23-25.
[10] 贾宇臣, 王利, 陈琦, 李连国, 李巧玲, 刘德虎.花生子叶节外植体遗传转化体系的建立[J]. 科学技术与工程, 2011, 11(14): 3142-3146. doi:10.3969/j.issn.1671-1815.2011.14.006.
Jia Y C, Wang L, Chen Q, Li L G, Li Q L, Liu D H. Establishment of genetic transformation system of peanut cotyledon node explants[J].Science Technology and Engineering, 2011, 11(14): 3142-3146.
[11] 邱金梅, 温世杰, 刘海燕, 梁炫强.根癌农杆菌介导花生高效遗传转化体系的优化[J]. 中国油料作物学报, 2010,32(2): 208-211.
Qiu J M, Wen S J, Liu H Y, Liang X Q. An effective Agrobacterium tumefaciens-mediated transformation system of peanut (Arachis hypogaea L.)[J].Chinese Journal of Oil Crop Sciences, 2010,32(2): 208-211.
[12] 钟育海, 申艮宝. 植物基因的遗传转化方法[J]. 安徽农学通报, 2010, 16(11): 65-66,206.doi:10.3969/j.issn.1007-7731.2010.11.032.
Zhong Y H,Shen G B.Genetic transformation technique of plant gene[J].Auhui Agricultural Science Bulletin, 2010, 16(11): 65-66,206.
[13] 申馥玉, 朱忠学, 吕祝章, 王传堂. 外源DNA导入技术在花生育种中的应用研究初报[J]. 花生科技, 1990(4): 5-6. doi:10.14001/j.issn.1002-4093.1990.04.002.
Shen F Y, Zhu Z X,Lü Z Z,Wang C T. A preliminary study on the application of exogenous DNA transfer technique in peanut breeding[J].Peanut Science and Technology, 1990(4): 5-6.
[14] 梁炫强, 罗虹. 外源DNA导入技术在花生选育种中的应用研究[J]. 广东农业科学, 1995(6): 15-17.
Luo X Q, Luo H. Study on the application of exogenous DNA transfer technology in peanut selection and breeding[J].Guangdong Agricultural Sciences, 1995(6): 15-17.
[15] 王亚, 郭宝太, 乔利仙, 武秀玲, 王晶珊, 韩立坤, 隋炯明.PyTPS转化花生对其后代耐盐性和品质性状的影响[J]. 华北农学报, 2014, 29(5): 175-179.doi: 10.7668/hbnxb.2014.05.030.
Wang Y, Guo B T, Qiao L X, Wu X L, Wang J S, Han L K,Sui J M.Effect of PyTPS on salt tolerance and quality of transgenic offspring of peanut[J].Acta Agriculturae Boreali-Sinica, 2014, 29(5): 175-179.
[16] 范乾程,谭玲玲,王亚,乔利仙,王晶珊,隋炯明. 花粉管通道法验证花生Oleosin基因启动子的特异性表达[J]. 中国粮油学报, 2013, 28(12): 52-56.doi: 10.3969/j.issn.1003-0174.2013.12.011.
Fan Q C, Tan L L, Wang Y,Qiao L X, Wang J S, Sui J M.Identification of specific expression of promoter of Oleosin gene via pollen-tube pathway method[J].Journal of the Chinese Cereals and Oils Association, 2013, 28(12): 52-56.
[17] Zong Y, Wang Y P, Li C, Zhang R, Chen K L, Ran Y D, Qiu J L, Wang D W, Gao C X. Precise base editing in rice, wheat and maize with a Cas9-cytidine deaminase fusion[J]. Nature Biotechnology, 2017,35(5): 438-440. doi:10.1038/nbt.3811.
[18] Scheben A, Edwards D. Genome editors take on crops[J]. Science, 2017, 355(6330): 1122-1123.doi:10.1126/science.aal4680.
[19] Dong S B, Huang J C, Li Y N, Zhang J, Lin S Z, Zhang Z X. Cloning, characterization, and expression analysis of acyl-acyl carrier protein (ACP)-thioesterase B from seeds of Chinese Spicehush (Lindera communis)[J]. Gene, 2014, 542(1): 16-22. doi:10.1016/j.gene.2014.03.028.
[20] Jiang W Z, Henry I M, Lynagh P G,Comai L, Cahoon E B, Weeks D P. Significant enhancement of fatty acid composition in seeds of the allohexaploid, Camelina sativa, using CRISPR-Cas9 gene editing[J]. Plant Biotechnology Journal,2017, 15(5): 648-657. doi:10.1111/pbi.12663.
[21] 于明洋,孙明明,郭悦,姜平平,雷永,黄冰艳,冯素萍,郭宝珠,隋炯明,王晶珊,乔利仙. 利用回交法快速选育高油酸花生新品系[J].作物学报, 2017, 43(6): 855-861.doi: 10.3724/SP.J.1006.2017.00855.
Yu M Y, Sun M M, Guo Y, Jiang P P, Lei Y, Huang B Y, Feng S P, Guo B Z, Sui J M, Wang J S, Qiao L X.Breeding new peanut line with high oleic acid content using backcross method[J].Acta Agronomica Sinica, 2017,43(6): 855-861.
[22] Clough S J, Bent A F. Floral dip:a simplified method for Agrobacterium-mediated transformation of Arabidopsis thaliana[J]. The Plant Journal,1998, 16(6): 735-743.doi:10.1046/j.1365-313x.1998.00343.x.
[23] Hu D, Bent A F, Hou X L, Li Y. Agrobacterium-mediated vacuum infiltration and floral dip transformation of rapid-cycling Brassica rapa[J]. BMC Plant Biology, 2019, 19(1): 246. doi:10.1186/s12870-019-1843-6.
[24] 郭悦. 花生AhWRKY75基因的克隆、遗传转化及功能分析[D]. 青岛:青岛农业大学, 2018.
Guo Y. Cloning, genetic transformation and functional analysis of AhWRKY75 gene in peanut[D].Qingdao:Qingdao Agricultural University, 2018.
[25] 姜平平. 花生AhLea-D、AhLea-3基因的克隆及其耐盐功能验证[D]. 青岛:青岛农业大学, 2019.
Jiang P P. Cloning and functional analysis of AhLea-D and AhLea-3 genes in peanut[D]. Qingdao: Qingdao Agricultural University, 2019.
[26] Howells R M, Craze M, Bowden S, Wallington E J. Efficient generation of stable, heritable gene edits in wheat using CRISPR/Cas9[J]. BMC Plant Biology, 2018, 18(1): 215. doi:10.1186/s12870-018-1433-z.
