微卫星DNA (Microsatellites) 或称简单重复序列(Simple sequence repeats,SSRs),是一种广泛存在于原核生物和真核生物基因组中的串联重复序列,其重复单元一般由2~6个核苷酸组成[1]。与其他分子标记相比,SSR标记具有分布随机、数量丰富、信息含量高、等位基因变异多、共显性遗传、稳定性好、操作简便优点,目前已广泛应用于遗传图谱构建、分子标记辅助选择育种、遗传多样性分析和品种鉴定等领域[2-4]。SSR标记可以从保守程度较高的编码区获取,也可以从非编码区获取[5]。传统的基因组SSR标记开发相对耗时,价格也比较昂贵。基于表达序列标签(Expressed sequence tags,EST)或cDNA序列开发的SSR标记被称为EST-SSR标记[6],EST-SSR标记是基于已有的EST或转录组等公共数据库资源开发,省去了SSR引物开发过程中的克隆和测序步骤,大幅度降低了开发成本[3]。与传统的基因组SSR标记相比,EST-SSR反映了蛋白质编码区的信息,与基因的表达具有直接或间接的关系,是功能基因的“绝对”的标记。因此,这类标记对于功能基因分离、基因组比较作图和遗传图谱的构建具有更为重要的作用[7-9]。
普通小麦(Triticum aestivum L.,2n = 6x = 42)是世界上重要的粮食作物之一。目前利用SSR标记、SNP标记和SRAP标记已构建了多张小麦的遗传图谱[10-12],这些标记也为小麦的分子标记辅助选择育种奠定了基础。尽管从已公布的小麦的EST数据库中开发了一些EST-SSR标记[13-15],但是与其他物种相比,利用小麦的转录组数据开发EST-SSR标记还相对较少。西华师范大学西南野生动植物资源保护教育部重点实验室前期从小麦雄蕊同源转化为雌蕊突变体(HTS-1)的正常雌蕊(P)、雌蕊化雄蕊(PS)和正常雄蕊(S)中获得了40.88 G的转录组数据,并从这些数据中心挖掘出8 389条EST-SSR序列[16]。本研究从8 389对EST-SSR引物中随机选取了585对得分大于95分的引物进行合成,并在小麦中检测其有效性。以川麦42与川农16杂交并自交后的RIL群体为作图群体,利用筛选出的EST-SSR标记,结合本实验室前期筛选的小麦基因组SSR标记和SRAP标记构建了一张遗传图谱。本研究旨在丰富小麦功能分子标记的数量,为小麦功能基因的定位、比较基因组学、小麦起源和进化等方面的研究奠定基础。
1 材料和方法
1.1 试验材料
川麦42与川农16杂交后再自交构建的重组自交系群体(RILs),F14(2018年),共125个家系,该群体由四川省农业科院的杨武云研究员馈赠。 每一个家系种植1行,行距0.2 m,株距0.2 m,常规大田水肥管理,次年3月采集3~4片新鲜嫩叶,液氮速冻后置于-80 ℃超低温冰箱中保存备用。
1.2 试验方法
1.2.1 DNA提取 利用植物DNA提取试剂盒(兰博生物,中国浙江)提取川麦42、川农16及其RILs家系幼叶的基因组DNA。具体操作参照试剂盒的说明书进行。1%琼脂糖凝胶电泳检测DNA的完整性,Nanodrop 2000微量紫外-可见分光光度计检测DNA的浓度和纯度,选择无降解、纯度高的DNA样品用于后续试验。
1.2.2 EST-SSR位点筛选及引物设计 从本实验室前期利用转录组分析筛选的8 389个SSR位点中选取585个位点用于引物设计[17],选取原则为:二核苷酸、三核苷酸、四核苷酸、五核苷酸、六核苷酸的重复基元的重复次数分别大于或等于10,7,5,4,3,SSR位点大于或等于20 bp,EST序列长度大于150 bp。利用Primer Premier 5.0 软件设计引物,引物得分大于95分,所有EST-SSR引物由生工生物工程(上海)有限公司合成。
1.2.3 PCR扩增及引物筛选 利用川麦42和川农16的DNA对585对EST-SSR引物进行初步筛选。PCR反应体系为10.0 μL:2×Taq Master Mix 5 μL;10 μmol/L的正向引物(F)和反向引物(R)各0.5 μL;30~50 ng/μL的模板DNA 1 μL;ddH2O 3 μL。为减少非特异性扩展,本试验采用Touchdown PCR 程序:95 ℃预变性 3 min; 95 ℃变性 30 s,65 ℃退火60 s (每一循环减1 ℃),72 ℃延伸60 s,共10个循环;95 ℃变性 30 s,55~62 ℃退火60 s (引物的退火温度如表1所示),72 ℃延伸60 s,共30个循环,72 ℃继续延伸5 min。首先,采用1.5%的琼脂糖凝胶电泳检测是否扩增成功;其次,采用8%的聚丙烯酰胺凝胶电泳筛选具有多态性的引物;最后选择多态性引物对125个RIL家系进行PCR扩增和聚丙烯酰胺凝胶电泳。
1.2.4 遗传图谱的构建 对聚丙烯酰胺凝胶电泳结果的带型进行统计,将与母本川麦42带谱一致的标记为“A”,与父本川农16带谱一致的表记为“B”,具有双亲带谱(杂合体)的标记为“H”,整合本实验室之前筛选的SRAP引物和SSR引物[18],利用Jionmap 4.0软件构建遗传图谱,作图函数为Kosambi,设置LOD>3.0,最大遗传距离为50 cM,其他为默认参数。
2 结果与分析
2.1 引物有效性和多态性分析
利用亲本川麦42与川农16对585对EST-SSR引物进行有效性检测,其中555对引物在双亲中能扩增出清晰、稳定的条带,其条带大小在100~300 bp,有效性达94.87%。其余30对被视为无效引物,其中2对引物的扩增条带太大(超过500 bp),6对引物的扩增条带小于50 bp, 22对引物在双亲中均无扩增条带。555对有效引物中有69对引物在川麦42和川农16中表现出多态性,占12.43%。随机选取20个RILs单株进一步验证引物的多态性,结果有44对引物表现出清晰的、稳定的多态性条带,多态性率为7.93%,comp584引物扩增结果如图1所示。这些引物可作为小麦新的EST-SSR标记(部分引物序列如表1所示)。
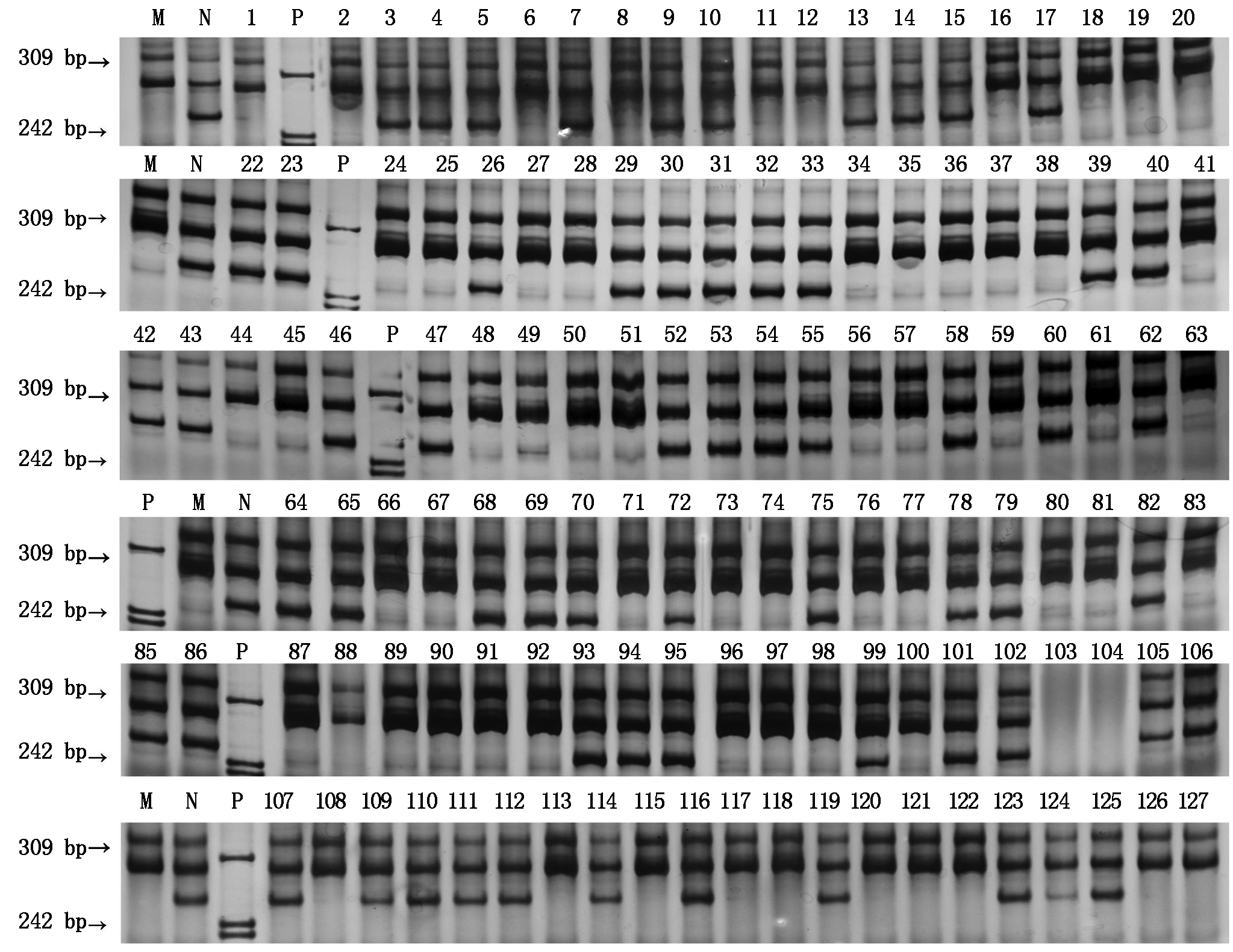
M.川麦42;N.川农16;P.pBR322 Marker;缺少编号为21和84的株系。
M. Chuanmai 42;N. Chuannong 16;P. pBR322 Marker;Missing lines with numbers 21 and 84.
图1 引物comp584扩增川麦42 ×川农16重组自交系的聚丙烯酰胺凝胶电泳图
Fig.1 SDS-PAGE of RILs derived from Chuanmai 42 × Chuannong 16 amplified by comp548 primers
表1 位于遗传图谱上的17对EST-SSR引物特点
Tab.1 The characteristics of 17 pairs of EST-SSR primers on the genetic map

2.2 标记偏分离分析
亲本之间具有多态性的EST-SSR标记的2种基因型在RILs作图群体中的比例应符合1∶1,对44个EST-SSR标记2种基因型在作图群体中的分布进行χ2检验表明,在0.05水平上有13个标记表现为偏分离,占总的EST-SSR标记的29.5%。在13个偏分离标记中有12个标记偏向川麦42,仅1个偏向川农16。
2.3 遗传图谱构建
利用上述44个EST-SSR标记扩增RILs群体的125个系,并整合本实验室前期的SSR标记和SRAP标记数据构建小麦遗传图谱。该图谱由163个标记组成,这些标记分布在12个连锁群上,其中新开发的EST-SSR标记17个,SSR标记和SRAP标记各73个。这些标记覆盖小麦基因组长度1 551.5 cM,相邻标记之间的平均距离为13.11 cM(图2)。其中位于B染色体组上标记最多,共95个,包括SRAP标记43个,SSR标记44个,EST-SSR标记8个,覆盖长度707.4 cM;D染色体组上标记最少,仅11个,包括2个SRAP标记,7个SSR标记和2个EST-SSR标记,覆盖长度为231.5 cM。新开发的17个EST-SSR标记在1B染色体上最多,有4个;其次是1A和4A染色体,各2个EST-SSR标记分布;在2B、3A、3B、3D、4B、5A、5B、6A、6D染色体上各有1个。在具有多态性的EST-SSR标记中能够定位到染色体上的标记为38.6%(表2)。
表2 分子标记在12条染色体上的分布
Tab.2 Distribution of molecular markers on the 12 chromosomes
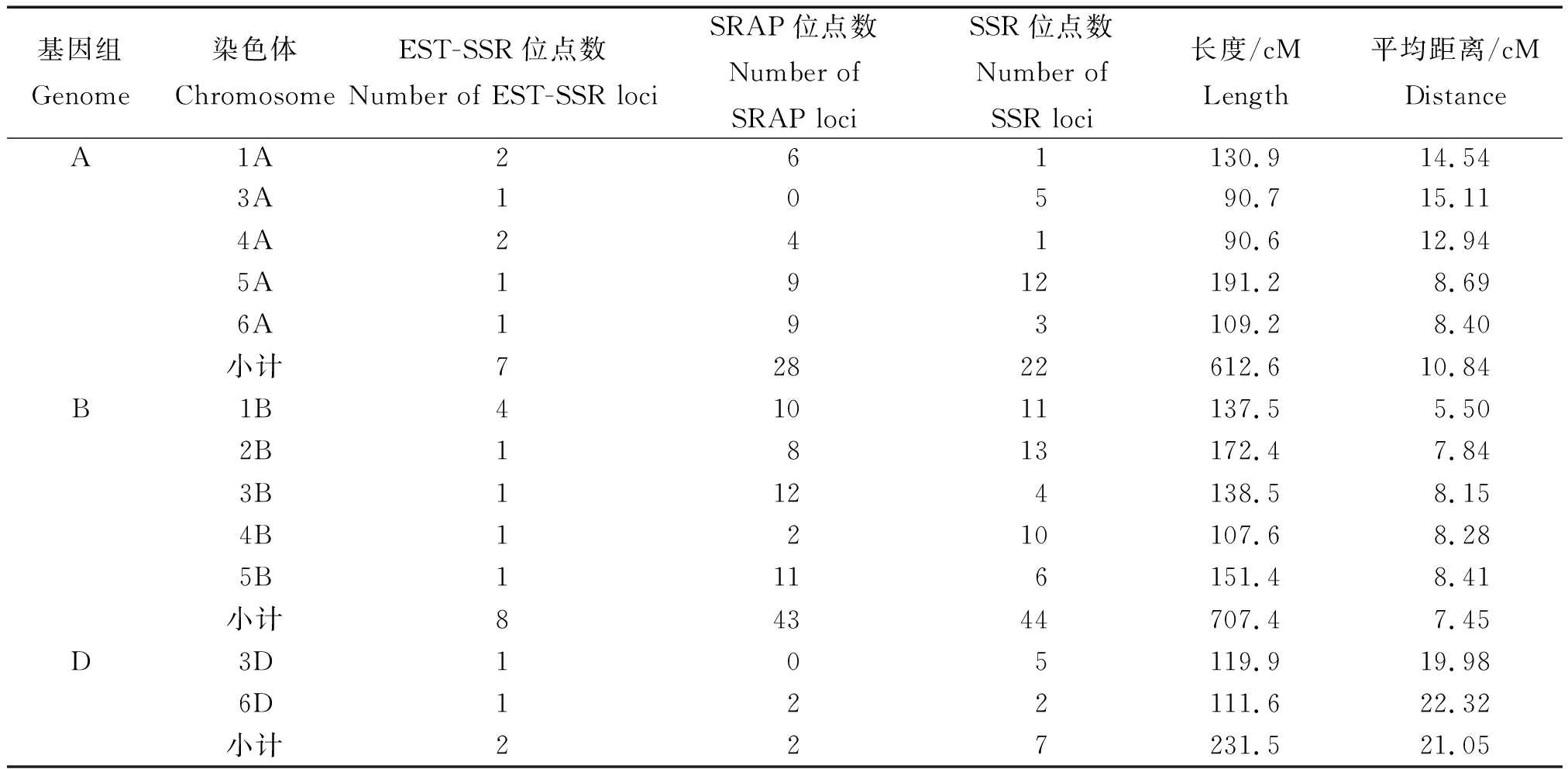
基因组Genome染色体ChromosomeEST-SSR位点数Number of EST-SSR lociSRAP位点数Number of SRAP lociSSR位点数Number of SSR loci长度/cMLength平均距离/cMDistanceA1A261130.914.543A10590.715.114A24190.612.945A1912191.28.696A193109.28.40小计72822612.610.84B1B41011137.55.502B1813172.47.843B1124138.58.154B1210107.68.285B1116151.48.41小计84344707.47.45D3D105119.919.986D122111.622.32小计227231.521.05

图距在染色体的左边,标记名称在染色体的右边;本研究新开发的EST-SSR标记加下划线。
Map distances are indicated on the left side of each chromosome,and the
name of markers on the right side; The newly developed EST-SSR markers in this study were underlined.
图2 EST-SSR标记在遗传图谱上的位置
Fig.2 Location of EST-SSR markers in genetic map
3 讨论与结论
利用诸如基因组文库筛选法、富集法、序列标签微卫星分析法(STMP)和选择性扩增微卫星分析法(SAM)等传统的SSR分子标记开发方法往往是一项耗时长且成本高的工作[19]。随着二代测序技术的迅速崛起,不仅测序错误率大幅度下降,准确率提高,且测序成本也极大降低,这为SSR标记的开发奠定了基础。加之以NCBI(https://www.ncbi.nlm.nih.gov/)为代表的公共数据库资源的开发共享,使得EST序列的查询更为方便,这些都为EST-SSR标记的开发提供了广阔的前景。基于高通量测序平台的转录组测序技术(RNA-Seq),可获取样品在特定时间下特定组织所表达的转录本序列信息,且不受参考基因组有无的影响,因此,RNA-Seq已成为发掘基因功能的重要手段之一。除此之外,RNA-Seq技术也是挖掘EST-SSR标记的有效手段。目前,我国学者在利用EST序列进行小麦EST-SSR标记的开发方面已取得较大的成就,如Gao等[20]利用71 495条EST序列设计出688对EST-SSR引物,其中483个引物对有扩增产物;陈军方等[21]利用ITEC上公布的10 380条EST序列设计出135对EST-SSR引物,其中有效引物82对,并将32对引物定位到除2D、4B和4D外的18条小麦染色体上;潘海涛等[3]利用GenBank/dbEST数据库中的265 362条小麦EST序列设计出596个EST-SSR引物对,选择其中95分以上的194个引物对进行PCR检测,结果表明,165个引物对能扩增出稳定清晰的条带,并将其中的23个EST-SSR位点整合到已有的小麦遗传图谱上。本实验室的Yang等[17]从小麦HTS-1突变体的RNA-Seq数据中鉴定出8 389个潜在的SSR序列,并进行了引物设计;对随机挑选出的300对EST-SSR引物进行鉴定,结果表明有177个引物对能扩增出稳定清晰的条带。上述EST-SSR标记可以作为小麦及其近缘物种的分子标记。本研究是在Yang等[17]的基础上从8 389个SSR引物对中随机选取585对得分大于95分的EST-SSR引物进行合成,其中555对引物在川麦42和川农16中均能扩增出稳定清晰的条带,其大小在50~300 bp,其有效性为94.87%。
在555对有效引物中有44对EST-SSR引物在川麦42和川农16中表现出明显的、稳定的多态性,多态性率为7.93%。其多态率显著低于SRAP标记和SSR标记[18,22]。这主要是由于EST-SSR来源于表达序列,这些序列具有较高的保守性,但也正是由于这个原因使得EST-SSR标记成为一种有功能的分子标记[23]。利用这44个EST-SSR标记,以川麦42与川农16杂交后并自交获得的RILs群体为作图群体,结合了本实验室前期的SRAP和SSR标记数据,构建了一张小麦遗传图谱。含有本次新开发的EST-SSR标记的连锁群共12个,这12个连锁群由163个标记组成,包括EST-SSR标记17个,SSR标记和SRAP标记73个。覆盖小麦基因组长度1 551.5 cM,相邻标记间的平均遗传距离为13.11 cM。除1B、1A和4A染色体外,其余的9条染色体上仅1个EST-SSR标记分布,在具有多态性的EST-SSR标记中能够定位到染色体上的标记为38.6%。普通小麦是异源六倍体,含有A、B、D 3个染色体组,其遗传图谱应包含21个连锁群,但本次利用EST-SSR标记构建的遗传图谱只包含了其中的12个连锁群。在1D、2A、2D、4D、6B、7A、7B和7D 9个连锁群上没有筛选到与之连锁的EST-SSR标记。这可能与EST-SSR标记本身的多态性较低和本试验筛选的标记数量偏少有关。因此,下一步应继续筛选和开发新的EST-SSR标记,不断完善和丰富各连锁群上的分子标记。尝试将本研究获得的图谱与本试验之前构建的遗传图谱进行整合,但由于本次开发的标记数量偏少,且缺乏中间连锁标记,因此,只有极个别EST-SSR标记能整合。但本研究丰富了小麦EST-SSR标记的数目。而这些EST-SSR标记作为基因的一部分其信息含量高、保守性好、在物种间具有良好的通用性等优点[22],因此,在功能基因的定位、比较基因组学和小麦起源和进化研究方面具有重要的价值。
[1] Koelling J,Coles M C,Matthews P D,Schwekendiek A. Development of new microsatellite markers (SSRs) for Humulus lupulus[J]. Molecular Breeding,2012,30:479-484. doi:10.1007/s11032-011-9637-z.
[2] 胡文舜,陈秀萍,郑少泉. 龙眼EST-SSR标记开发及无患子科5个属种质遗传多样性分析[J]. 园艺学报,2019,46(7):1359-1372. doi:10.16420/j.issn.0513-353x.2018-0753.
Hu W S,Chen X P,Zheng S Q. EST-SSR markers developed from Dimocarpus longan and their application in genetic diversity analysis of five genera of Sapindaceae[J]. Acta Horticulturae Sinica,2019,46(7):1359-1372.
[3] 潘海涛,汪俊君,王盈盈,齐照良,李斯深. 小麦EST-SSR标记的开发和遗传作图[J]. 中国农业科学,2010,43(3):452-461. doi:10.3864/j.issn.0578-1752.2010.03.002.
Pan H T,Wang J J,Wang Y Y,Qi Z L,Li S S. Development and mapping of EST-SSR markers in wheat[J]. Scientia Agricultura Sinica,2010,43(3):452-461.
[4] 许云峰,蒋方山,郭营,李瑞军,李斯深. EMS 诱导小麦品种烟农15 突变体的鉴定和EST-SSR分析[J]. 核农学报,2008,22(4):410-414.
Xu Y F,Jiang F S,Guo Y,Li R J,Li S S. Identification and EST-SSR analysis of mutants from wheat variety "Yannong 15" induced by EMS[J]. Journal of Nuclear Agricultural Sciences,2008,22(4):410-414.
[5] Sraphet S, Boonchanawiwat A, Thanyasiriwat T, Boonseng O, Tabata S, Sasamoto S, Shirasawa K, Isobe S, Lightfoot D A, Tangphatsornruang S,Triwitayakorn K.SSR and EST-SSR-based genetic linkage map of cassava (Manihot esculenta Crantz)[J]. Theoretical and Applied Genetics,2011,122(6):1161-1170. doi:10.1007/s00122-010-1520-5.
[6] Chen C X,Zhou P,Choi Y A,Huang S,Gmitter F G. Mining and characterizing microsatellites from citrus ESTs[J]. Theoretical and Applied Genetics,2006,112(7):1248-1257. doi:10.1007/s00122-006-0226-1.
[7] Kalia R K,Rai M K,Kalia S,Singh R,Dhawan A K. Microsatellite markers:An overview of the recent progress in plants[J]. Euphytica,2011,177:309-334.doi:10.1007/s10681-010-0286-9.
[8] Parida S K, Kalia S K, Kaul S, Dalal V, Hemaprabha G, Selvi A, Pandit A, Singh A, Gaikwad K, Sharma T R, Srivastava P S, Singh N K,Mohapatra T. Informative genomic microsatellite markers for efficient genotyping applications in sugarcane[J]. Theoretical and Applied Genetics,2009,118:327-338. doi:10.1007/s00122-008-0902-4.
[9] Vašek J, ![]() ílová D, Melounová M, Svoboda P, Vejl P,
ílová D, Melounová M, Svoboda P, Vejl P, ![]() tikarová R, Vostry′ L, Kuchtová P, Ovesná J.New EST-SSR markers for individual genotyping of opium poppy cultivars (Papaver somniferum L.)[J]. Plants,2019,9(1):10-25. doi:10.3390/plants9010010.
tikarová R, Vostry′ L, Kuchtová P, Ovesná J.New EST-SSR markers for individual genotyping of opium poppy cultivars (Papaver somniferum L.)[J]. Plants,2019,9(1):10-25. doi:10.3390/plants9010010.
[10] Somers D J,Isaac P,Edwards K. A high-density microsatellite consensus map for bread wheat (Triticum aestivum L.)[J]. Theoretical and Applied Genetics,2004,109:1105-1114. doi:10.1007/s00122-004-1740-7.
[11] Yang Z J,Chen Z Y,Peng Z S,Yu Y,Liao M L,Wei S H. Development of a high-density linkage map and mapping of the three-pistil gene (Pis1) in wheat using GBS markers[J]. BMC Gnomics,2017,18:567. doi:10.1186/s12864-017-3960-7.
[12] Yang Q,Yang Z J,Tang H F,Yu Y,Chen Z Y,Wei S H,Sun Q X,Peng Z S. High-density genetic map construction and mapping of the homologous transformation sterility gene (hts) in wheat using GBS markers[J]. BMC Plant Biology,2018,18:301. doi:10.1186/s12870-018-1532-x.
[13] Yu J K,Dake T M,Singh S,Benscher D,Li W L,Gill B,Sorrells M E. Development and mapping of EST-derived simple sequence repeat markers for hexaploid wheat[J]. Genome,2004,47(5):805-818. doi:10.1139/g04-057.
[14] Chen H M,Li L Z,Wei X Y,Li S S,Lei T D,Hu H Z,Wang H G,Zhang X S. Development,chromosome location and genetic mapping of EST-SSR markers in wheat[J]. Chinese Science Bulletin,2005,50(20):2328-2336. doi:10.1007/BF03183744.
[15] Li L Z,Wang J J,Guo Y,Jiang F S,Xu Y F,Wang Y Y,Pan H T,Han G Z,Li R J,Li S S. Development of SSR markers from ESTs of gramineous species and their chromosome location on wheat[J]. Progress in Natural Science,2008,18(12):1485-1490. doi:10.1016/j.pnsc.2008.05.012.
[16] Yang Z J,Peng Z S,Wei S H,Liao M L,Yu Y,Jang Z Y. Pistillody mutant reveals key insights into stamen and pistil development in wheat (Triticum aestivum L.)[J]. BMC Genomics,2015,16:211. doi:10.1186/s12864-015-1453-0.
[17] Yang Z J,Peng Z S,Yang H. Identification of novel and useful EST-SSR markers from de novo transcriptome sequence of wheat (Triticum aestivum L.)[J]. Genetics and Molecular Research,2016,15 (1):15017509.doi:10.4238/gmr.15017509.
[18] Zhao L B,Zhang L,Qu J P,Yu Y,Lu L,Peng Z S,Yang W Y,Wei S H,Yang J,Yang Z J. Novel fluorescent sequence-related amplified polymorphism (FSRAP) markers for the construction of a genetic linkage map of wheat (Triticum aestivum L.)[J]. Genetika,2017,49(3):1081-1093. doi:10.2298/GENSR1703081Z.
[19] 邵阳,范文,黄连冬,高继银,李昕骥,张文驹. 基于RNA-Seq的崇左金花茶EST-SSR标记开发[J]. 复旦学报(自然科学版),2015,54(6):761-767.
Shao Y,Fan W,Huang L D,Gao J Y,Li X J,Zhang W J. Development of EST-SSR markers in Camellia chuongtsoensis based on RNA-Seq[J]. Journal of Fudan University (Natural Science),2015,54(6):761-767.
[20] Gao L F,Jing R J,Huo N X,Li Y,Li X P,Zhou R H,Chang X P,Tang J F,Ma Z Y,Jia J Z. One hundred and one new microsatellite loci derived from ESTs (EST-SSRs) in bread wheat[J]. Theoretical and Applied Genetics,2004,108:1392-1400. doi:10.1007/s00122-003-1554-z.
[21] 陈军方,任正隆,高丽锋,贾继增. 从小麦EST 序列中开发新的SSR 引物[J]. 作物学报,2005,31(2):154-158. doi:10.3321/j.issn:0496-3490.2005.02.004.
Chen J F,Ren Z L,Gao L F,Jia J Z. Developing ne w SSR markers from EST of wheat[J]. Acta Agronomica Sinica,2005,31(2):154-158.
[22] Tang Y L,Li J,Wu Y Q,Wei H T,Li C S,Yang W Y,Chen F. Identification of QTLs for yield-related traits in the recombinant inbred line population derived from the cross between a synthetic hexaploidy wheat-derived variety Chuanmai 42 and a Chinese elite variety Chuannong 16[J]. Agricultural Sciences in China,2011,10(11):1665-1680. doi:10.1016/S1671-2927(11)60165-X.
[23] Sylvia S,Campbel L,Henderson K,Eujay J I,Hanafey M,Powell W,Wolters P.Development of EST-derived microsatellite markers for mapping and germplasm analysis in wheat[C].San Diego,CA,Plant and Animal Genome Ⅷ Conference,2000.
