小麦是世界上重要的粮食作物之一,然而在其生长过程中容易受到真菌、细菌和病毒等多种病原菌的感染,对小麦产量和质量造成严重影响,如较严重的有白粉病、赤霉病等。小麦白粉病是由禾本科布氏白粉菌小麦专化型(Blumeria graminis f.sp.tritici(Bgt))引起的世界性真菌病害,在亚洲、非洲和欧美等主产麦区都有发生,中国的小麦产区也几乎都有发生,并有日趋严重之势[1]。小麦赤霉病是由镰刀菌(Fusarium graminearum)引起的一种世界性病害,近年来,随着全球气候变暖以及耕作制度的变化,小麦赤霉病的发生频率不断增加,发生范围也不断扩大。这些病害的发生都严重影响了小麦的产量和质量[2],因此,解决这些病害问题对小麦生产具有重要意义。由于小麦抗性品种比较少,目前,控制小麦病原菌发生的途径主要是使用杀菌剂,而杀菌剂的大量喷施会造成农药残留和环境污染,因此,培育小麦抗病品种成为解决小麦病害的理想途径。而不管是野生型还是人工选育的小麦抗病品种都比较匮乏,因此,快速、高效培育抗病品种迫在眉睫。传统杂交育种时间长、效率低,而利用现代分子生物学手段,将抗病基因转入栽培品种中使其获得抗病性,是快速、有效获得抗病品种的重要途径,其中分子育种的关键是挖掘更多具有优良抗性的基因[3-4]。
前人的研究中已克隆了一些小麦抗病相关基因[5-6],但抗性基因资源仍非常有限,而且抗病基因抗性单一,有效抗原没有充分利用,加之新的病原菌小种产生速度非常快,许多抗性基因丧失抗性。因此,需要进一步寻找新的抗病基因资源。microRNA(miRNA)是一类生物体内普遍存在的长度为21~25个核苷酸的内源性非编码小分子RNA,主要通过与目标基因互补配对从而在转录后水平调控基因的表达,导致mRNA降解或抑制翻译而参与植物体内多种生物学反应,在整个调控网络中起到关键作用[7-8]。目前,已在许多植物中发现了miRNA的存在,对其功能的研究也越来越广泛和深入。越来越多的研究表明,miRNA在植物抗病中也起着重要作用[9-11] ,其参与的抗病反应主要是通过直接或间接调控抗病相关基因的表达量来实现的。miRNA482/2118超家族中的miRNA能够调控NBS-LRR家族基因(抗病蛋白基因)的沉默,当番茄受到病原菌感染后,NBS-LRR家族基因的表达量迅速下降,植物抗病性发生变化[12]。此外,多主棒孢霉能够引起黄瓜棒孢叶斑病,通过RNA-seq分析,当多主棒孢霉侵染黄瓜时,miR164d、miR396b、Novel-miR1和Novel-miR7表达量发生变化,当过量或抑制表达它们的靶基因时,黄瓜对多主棒孢霉的抗性会发生变化,因此,推测miRNA在黄瓜对多主棒孢霉抗性中起重要作用[13]。研究还发现,棉花中的miRNA168在棉花抗曲叶病毒中起着重要作用,其主要通过在转录水平或直接降解病毒中关键基因的表达量来影响病毒的复制,从而使棉花具有抗病毒性[14]。综上表明,miRNA已成为植物与病原菌互作研究中的重要领域,通过抑制或诱导某种miRNA的产生,可以改变植物的抗病性,并且也从另一个角度应用于植物的抗病研究和育种中。
研究发现,miR395家族成员在受到病原菌侵入的应答反应中起着重要作用[15]。苹果中miR395的靶基因为MdWRKY26,当过量表达miR395时,MdWRKY26表达量下降,而WRKY可以和病程相关蛋白(PR)互作,因此抑制表达miR395时,PR基因表达量升高,苹果的叶斑病抗性增强[16]。棉花中的miR395a和miR395b参与调控棉花与曲叶病毒的互作[15]。通过生物信息学分析发现,miR395a成熟序列在各物种间具有较高的相似性。因此,推测miR395在植物抗病性方面具有一定的保守性。而目前关于其在小麦抗病方面的研究尚未见报道,为此,克隆小麦miR395a的前体序列,构建其过量表达载体并遗传转化小麦,同时分析其组织表达模式,以期为后续研究和解析miR395a在小麦抗病中的功能和作用机制提供材料来源。
1 材料和方法
1.1 试验材料
1.1.1 植物材料 供试材料为小麦栽培品种百农207。
1.1.2 质粒及菌种 大肠杆菌菌种DH5α、农杆菌菌种GV3101、克隆载体质粒pMD19-T、表达载体质粒pCambia1301。
1.1.3 酶和化学试剂 FastPfu酶购自全式金公司;限制性内切酶XbaⅠ和BamHⅠ、Marker DL 2000和T4 DNA连接酶、PrimeScript RT reagent Kit with gDNA Eraser (Perfect Real Time)和Mir-XTM miRNA First-Strand Synthesis Kit均购自TaKaRa公司;TRIzol购自美国Invitrogen公司;琼脂糖凝胶回收试剂盒和质粒小提试剂盒购自天根生物公司。
1.2 试验方法
1.2.1 小麦基因组DNA的提取 采用CTAB法[17]从小麦叶片中提取基因组DNA。
1.2.2 小麦miR395a前体基因的克隆 在NCBI上查找小麦miR395a的前体序列,设计引物,并在上、下游引物上分别加上BamHⅠ和XbaⅠ酶切位点,引物序列如下:上游引物TmiR395-F为5′-GGATCCCTAGAGTTCCCTTGACCGCT-3′,下游引物TmiR395-R为5′-TCTAGAACGATATGTTAACATCGAAT-3′。30 μL PCR反应体系:5×Pfu Buffer 6 μL,FastPfu酶0.6 μL,上、下游引物各1.5 μL,dNTP 3 μL,小麦基因组DNA模板2 μL,ddH2O 15.4 μL。扩增程序:95 ℃ 5 min;95 ℃ 30 s,56 ℃ 30 s,72 ℃ 30 s,33个循环;72 ℃ 10 min。
1.2.3 PCR产物的回收与测序 PCR产物经1%琼脂糖凝胶电泳后,用胶回收试剂盒回收,连接至克隆载体pMD19-T上,转化DH5α感受态细胞,挑取单克隆进行菌落PCR鉴定,之后送至武汉金开瑞公司测序,将测序正确的质粒命名为pMD19-T-miR395a。
1.2.4 小麦miR395a前体的过量表达载体构建 用限制性内切酶BamHⅠ和XbaⅠ对载体pCambia1301和pMD19-T-miR395a分别进行双酶切,完全酶切后,进行琼脂糖凝胶电泳,用PCR产物回收试剂盒(天根琼脂糖凝胶DNA回收试剂盒)进行纯化回收后连接,10 μL连接体系如下:miR395a前体序列片段6 μL,线性化质粒1 μL,10×Buffer 1 μL,T4 DNA连接酶0.2 μL,ddH2O 1.8 μL。16 ℃连接12 h后,转化DH5α感受态细胞,通过菌落PCR和酶切鉴定,筛选出阳性转化子,即过量表达载体,将其命名为pCambia1301-miR395a。
1.2.5 过量表达载体转化农杆菌 将3 μL pCambia1301-miR395a质粒和100 μL农杆菌GV3101感受态细胞混匀,在冰上静置30 min,之后液氮中放置5 min,取出后立即放入37 ℃培养箱5 min,随后加入YEB(不含抗生素)800 μL,在28 ℃恒温摇床中复苏3~5 h,用涂布棒均匀涂在YEB固体培养基(含Rif和Kan抗生素)上。将平板放在28 ℃培养箱中培养2~3 d,通过菌落PCR鉴定,筛选出阳性菌落,备用。
1.2.6 农杆菌介导的小麦遗传转化 小麦的遗传转化方法,参照Supartana等[18]的报道。
1.2.7 转基因小麦的PCR检测 采用CTAB法提取转基因当代小麦叶片的基因组DNA,使用嵌合引物,即在过量表达载体中启动子序列上设计上游引物S1:5′-CTCCTACAAATGCCATCATT-3′,miR395a前体序列上设计下游引物S2:5′-AGAACTTCACAGTGGTCTTA-3′,进行PCR,扩增片段长度约为580 bp,PCR阳性对照模板为质粒pCambia1301-miR395a,阴性对照为水对照和未转基因植株。
1.2.8 组织表达部位分析 总RNA的提取采用TRIzol法,反转录使用TaKaRa的miRNA反转录试剂盒Mir-XTM miRNA First-Strand Synthesis Kit,按照操作说明进行。将反转录产物稀释100倍作为模板,进行荧光定量分析,荧光定量试剂盒使用TaKaRa的PrimeScript RT reagent Kit with gDNA Eraser (Perfect Real Time),按照操作说明进行。每个组织部位3个重复;上游引物为miR395a的特异引物:5′-GTGAAGTGTTTGGGGGAACTC-3′,下游引物为前体序列引物S2:5′-AGAACTTCACAGTGGTCTTA-3′;内参基因为GAPDH。荧光定量的结果采用2-ΔΔCT方法计算。
2 结果与分析
2.1 miR395a成熟序列比对分析
从miRBase(http://www.mirbase.org)中分别获得不同物种的miR395a成熟序列,分析发现,miR395a在不同物种中具有较高的保守性,其中小麦与拟南芥、番茄以及大豆中的miR395a成熟序列具有高度一致性(图1),推测其功能上也具有保守性。

图1 miR395a成熟序列比对分析
Fig.1 Alignment analysis of mature miR395a sequence
2.2 小麦miR395a前体序列的克隆
根据在miRBase和NCBI上获得的miR395a的前体序列,设计引物,拟扩增出384 bp的片段。以小麦叶片的基因组DNA为模板,以TmiR395-F和TmiR395-R为引物,进行PCR扩增,扩增结果如图2所示。对该条带进行回收、连接至克隆载体pMD-19T上,并进行测序。对测序结果在DNAMAN上进行比对分析,发现所获片段与NCBI中获得的miR395a前体序列的碱基完全一致,表明已成功克隆该序列。
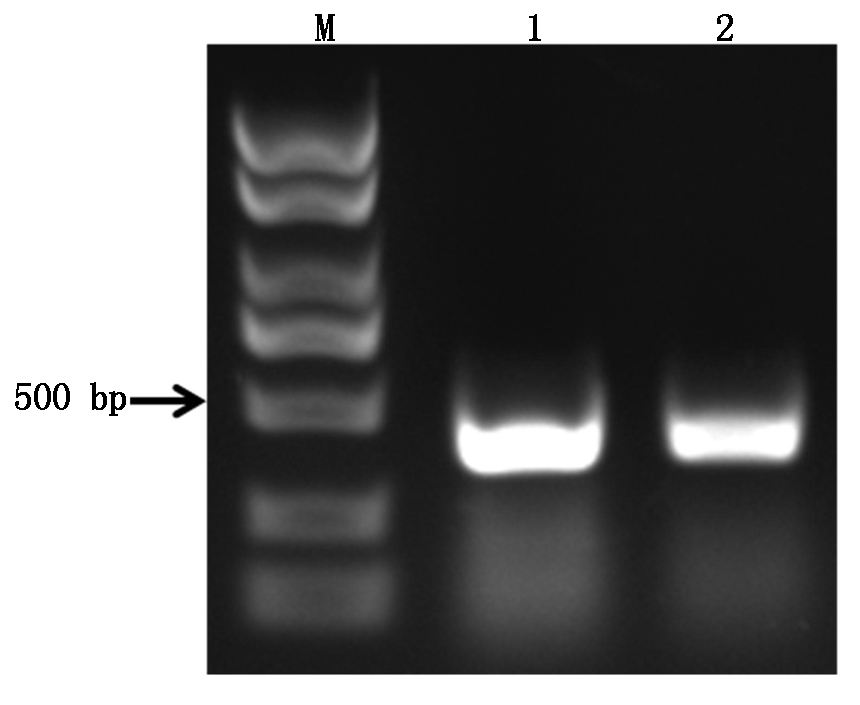
M.DNA标准;1,2.miR395a扩增结果。
M.Marker;1,2.Products of pre-miR395a.
图2 小麦miR395a前体序列的PCR扩增结果
Fig.2 Products of wheat pre-miR395a amplified by PCR
2.3 小麦miR395a前体过量表达载体的构建
将以上测序正确的pMD-19T-miR395a重组质粒和表达载体pCambia1301质粒同时用BamHⅠ和XbaⅠ进行双酶切,将目的基因片段和线性化的pCambia1301切胶回收、连接,菌落PCR初步鉴定阳性转化子pCambia1301-miR395a,进一步通过酶切鉴定,能够切割出预期连入的miR395a前体片段396 bp(图3),证明过量表达载体构建成功。
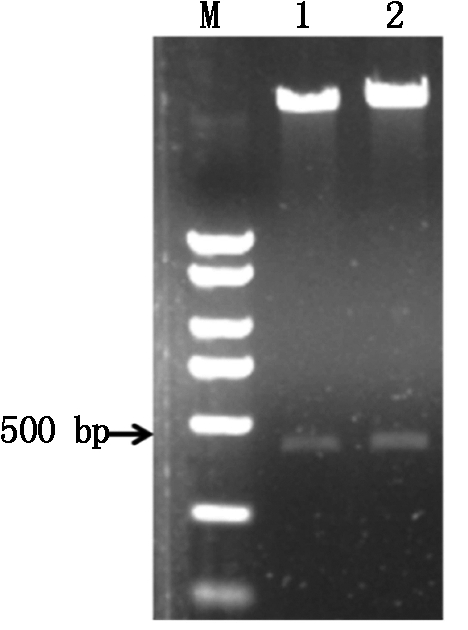
M.DNA标准;1,2.重组载体酶切鉴定结果。
M.Marker;1,2.Products of pCambia1301-miR395a
digested by restriction enzyme.
图3 重组质粒的酶切鉴定
Fig.3 Characterization of recombinant plasmid by restriction enzyme digestion
2.4 农杆菌介导法遗传转化小麦
采用农杆菌介导的方法,将miR395a前体过量表达载体遗传转化小麦,获得56株T0转基因植株(图4),并已收获种子。

图4 小麦转基因T0植株表型
Fig.4 Phenotype of T0 transgenic wheat
2.5 转基因小麦植株的PCR检测
提取56株小麦T0植株的基因组DNA,同时以构建好的过量表达质粒作阳性对照,水和未转基因植株作阴性对照,进行PCR扩增。扩增产物经1%琼脂糖凝胶电泳检测,结果显示,在56株转基因植株中,11株转基因植株扩增出和阳性质粒大小一致的约580 bp片段,且与预期大小一致,而其余45株转基因植株、水对照和未转基因植株均未扩增出目标条带(图5)。同时也进行了GUS染色,与PCR结果一致,说明miR395a前体已成功转入小麦中,阳性率约20%。
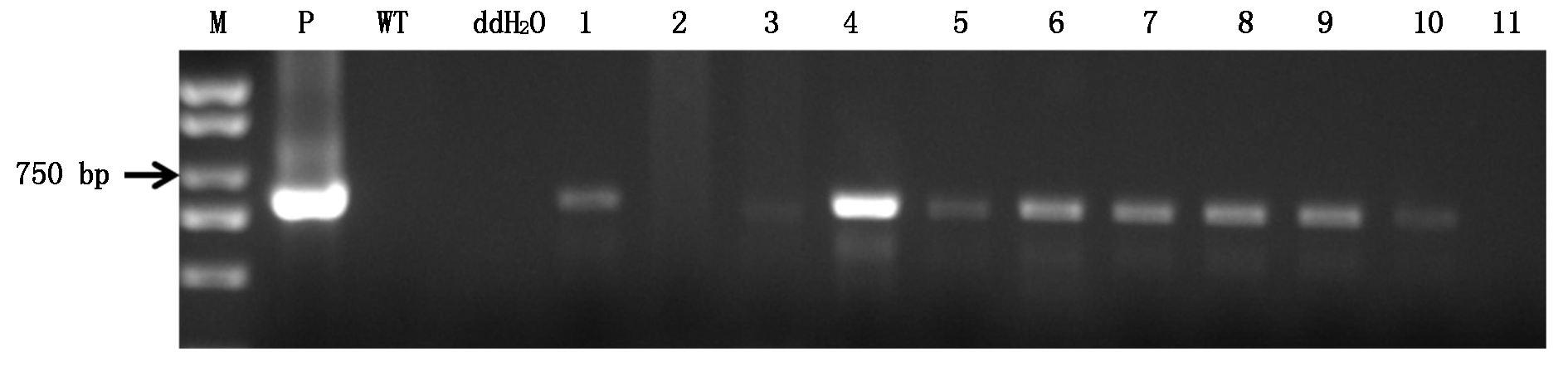
M.DNA标准;P.过量表达载体质粒对照;WT.未转基因植株;ddH2O.水对照;1-11.T0转基因植株。
M.Marker;P.Plasmid;WT.Wild type;ddH2O.Double distilled water;1-11.11 lines of T0 transgenic plants.
图5 转基因小麦的PCR鉴定
Fig.5 RCR identification of transgenic wheat
2.6 小麦miR395a的组织表达模式分析
利用RT-qPCR技术,分析miR395a在小麦不同组织部位中的表达量(图6),结果显示,miR395a为组成型表达,但在不同组织中的表达量有差异,其中在三叶期和抽穗期叶片中的表达量较高,叶鞘中次之,旗叶中表达量最低。
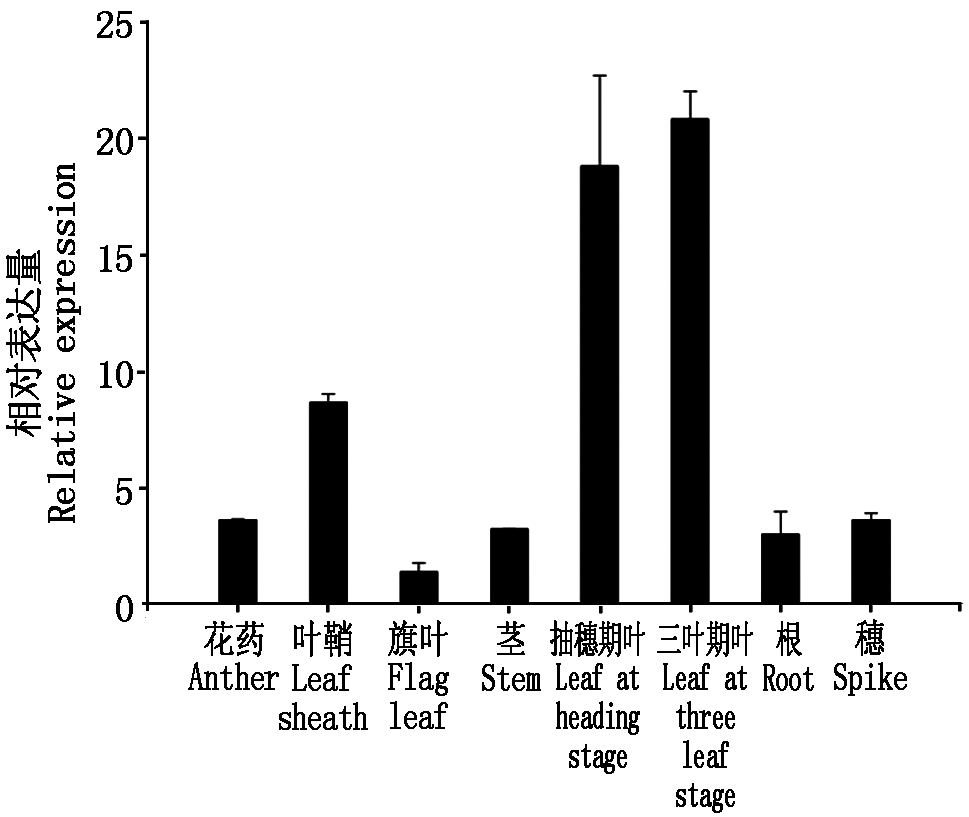
图6 小麦miR395a的组织表达模式分析
Fig.6 Analysis of tissue expression pattern of miB395a in wheat
3 结论与讨论
病害严重影响了小麦的产量和质量,小麦与病原菌互作是目前小麦研究中的热点领域。miRNA在植物抗病中的功能越来越多地被报道,如当番茄受到致病疫霉菌的感染时,其中的一些miRNA表达量发生明显变化,分析它们的靶基因均为一些植物病程相关蛋白,说明miRNA参与番茄对致病疫霉菌的防御反应[19]。前期的试验结果表明,小麦中一些miRNA的表达量也能够响应白粉菌的诱导,其中包括miR395a。而有关miR395家族成员的功能,早期的研究主要集中在其对硫酸盐吸收中的作用[20-21],发现其能够调控拟南芥对SO2的抗性,也即其能参与非生物胁迫的应答反应。最近相关的研究表明,miR395也参与植物抗病反应[14]。
序列分析发现,miR395a的成熟序列在不同物种中具有较高的保守性,推测其功能也具有保守性。而miR395a在小麦抗病中的功能还未见报道。为了进一步研究其功能,本试验克隆了小麦miR395a前体序列,并将其过量表达载体遗传转化至小麦中,在基因组水平设计嵌合引物,通过PCR扩增鉴定出阳性植株,同时通过GUS染色,进一步确定阳性植株。目前这些植株已经成熟并收获种子。同时本研究还对miR395a的组织表达模式进行了分析,结果显示,其为组成型表达,其中在三叶期和抽穗期的叶中表达量最高,表明其在小麦生长各个阶段都可能发挥作用,但主要在叶片中起作用。下一步将筛选阳性T0植株的种子,并对T1叶片接种白粉菌等病原菌,进行小麦抗性鉴定试验,研究miR395a是否具有对白粉病等的抗性反应,并在穗期对转基因小麦进行赤霉病抗性鉴定,进一步深入研究miR395a在小麦广谱抗病中的功能及作用机制。
[1] 高海峰,白微微,刘恩良,赵海燕,李广阔,张航,曾潮武.新疆春小麦品种(系)抗白粉病基因的分子检测[J].河南农业科学,2017,46(10):76-80. doi:10.15933/j.cnki.1004-3268.2017.10.014.
Gao H F,Bai W W,Liu E L,Zhao H Y,Li G K,Zhang H,Zeng C W. Molecular detection of powdery mildew resistant genes of spring wheat varieties(lines) in Xinjiang[J].Journal of Henan Agricultural Sciences,2017,46(10):76-80.
[2] Zou J W, Qiu D, Sun Y L, Zheng C X, Li J T, Wu P P,Wu X F, Wang X M, Zhou Y, Li H J. Pm52:effectiveness of the gene conferring resistance to powdery mildew in wheat cultivar Liangxing 99[J]. Acta Agron Sin,2017,43(3):332-342. doi:10.3724/SP.J.1006.2017.00332.
[3] 夏晴,麦艳娜,董振杰,刘文轩.小麦-野生亲缘种属添加系白粉病新抗源筛选及特异分子标记鉴定[J].河南农业科学,2018,47(6):64-69. doi:10.15933/j.cnki.1004-3268.2018.06.012.
Xia Q,Mai Y N,Dong Z J,Liu W X. Identification of powdery mildew resistance resources from wheat-wild relative disomic addition lines and development of molecular markers of alien chromosome-specialty[J].Journal of Henan Agricultural Sciences,2018,47(6):64-69.
[4] 王掌军,刘妍,王志兰,乔丹,石冰欣,卢春甜,刘凤楼,李清峰,张晓岗,刘生祥.宁春4号与河东乌麦杂交F2产量相关性状和抗病性及其QTL分析[J].河南农业科学,2019,48(8):7-17. doi:10.15933/j.cnki.1004-3268.2019.08.002.
Wang Z J,Liu Y,Wang Z L,Qiao D,Shi B X,Lu C T,Liu F L,Li Q F,Zhang X G,Liu S X. Analysis of yield-related traits,disease resistance and their QTLs of F2 hybrids from Ningchun No.4 and Hedong black wheat[J].Journal of Henan Agricultural Sciences,2019,48(8):7-17.
[5] Xu H X, Yi Y J, Ma P T, Qie Y M, Fu X Y, Xu Y F, Zhang X T, An D G. Molecular tagging of a new broad-spectrum powdery mildew resistance allele Pm2c in Chinese wheat landrace Niaomai[J]. Theor Appl Genet, 2015,128(10):2077-2084. doi:10.1007/s00122-015-2568-z.
[6] Zou B H,Ding Y,Liu H,Hua J. Silencing of copine genes confers common wheat enhanced resistance to powdery mildew[J]. Mol Plant Pathol,2018,19(6):1343-1352. doi:10.1111/mpp.12617.
[7] Voinnet O. Origin,biogenesis,and activity of plant microRNAs[J]. Cell,2009,136(4):669-687. doi:10.1016/j.cell.2009.01.046.
[8] 安凤霞,梁艳,曲彦婷.MicroRNA在调节植物生长发育和逆境胁迫中的作用[J].植物生理学报,2013,49(4):317-323. doi:10.13592/j.cnki.ppj.2013.04.013.
An F X,Liang Y,Qu Y T. Role of microRNA in regulating plant growth and development and stress responses[J]. Plant Physiology Journal,2013,49(4):317-323.
[9] Chen L,Meng J M,Zhai J M,Xu P S,Luan Y S. MicroRNA396a-5p and-3p induce tomato disease susceptibility by suppressing target genes and up-regulation salicylic acid[J]. Plant Sci,2017,265(12):177-187. doi:10.1016/j.plantsci.2017.10.004.
[10] Pradhan B,Naqvi A R,Saraf S,Mukherjee S K,Dey N. Prediction and characterization of tomato leaf curl new delhivirus (TOLCNDV) responsive novel microRNAs in Solanum lycopersicum[J]. Virus Res,2015,195(7):183-195. doi:10.1016/j.virusres.2014.09.001.
[11] Zhu Q H,Fan L J,Liu Y,Xu H,Llewellyn D,Wilson L. miR482 regulation of NBS-LRR defense genes during fungal pathogen infection in cotton[J]. PLoS One,2013,8(12):143-159. doi:10.1371/journal.pone.0084390.
[12] Shivaprasad P V,Chen H M,Patel K,Bond D M,Santos B A C M,Baulcombe D C. A microRNA superfamily regulates nucleotide binding site-leucine-rich repeats and other mRNAs[J]. The Plant Cell,2012,24(3):859-874. doi:10.1105/tpc.111.095380.
[13] Wang X Y,Yu G C,Zhao J Y,Cui N,Yu Y,Fan H Y. Functional identification of Corynespora cassiicola-responsive miRNAs and their targets in cucumber[J]. Front Plant Sci,2019,10:668. doi:10.3389/fpls.2019.00668.
[14] Shweta,Akhter Y,Khan J A. Genome wide identification of cotton (Gossypium hirsutum)-encoded microRNA targets against Cotton leaf curl Burewala virus[J]. Gene,2018,638:60-65. doi:10.1016/j.gene.2017.09.061.
[15] Li L H,Yi H L,Xue M Z,Yi M. miR398 and miR395 are involved in response to SO2 stress in Arabidopsis thaliana[J]. Ecotoxicology,2017,26(9):1181-1187. doi:10.1007/s10646-017-1843-y.
[16] Zhang Q L, Li Y, Zhang Y, Wu C B, Wang S N, Hao L, Wang S Y, Li T T. Md-miR156ab and Md-miR395 target WRKY transcription factors to influence apple resistance to leaf spot disease[J]. Front Plant Sci,2017,8:526. doi:10.3389/fpls.2017.00526.
[17] 张晓祥,王玲,寿路路. 改良CTAB方法快速提取小麦基因组DNA[J]. 农业科学与技术(英文版),2013, 14(7) : 946-949.doi:10.16175/j.cnki.1009-4229.2013.07.015.
Zhang X X,Wang L,Shou L L. Modified CTAB method for extracting genomic DNA from wheat leaf[J]. Agr Sci Tech,2013,14(7):946-949.
[18] Supartana P,Shimizu T,Nogawa M,Shioiri H,Nakajima T,Haramoto N,Nozue M,Kojima M. Development of simple and efficient in planta transformation method for wheat (Triticum aestivum L.) using Agrobacterium tumefaciens[J]. J Biosci Bioeng,2006,102(3):162-170. doi:10.1263/jbb.102.162.
[19] Luan Y S,Cui J M,Zhai J M,Li J,Han L,Meng J. High-throughput sequencing reveals differential expression of miRNAs in tomato inoculated with Phytophthora infestans[J]. Planta,2015,241(6):1405-1416. doi:10.1007/s00425-015-2267-7.
[20] Ai Q,Liang G,Zhang H M,Yu D Q. Control of sulfate concentration by miR395-targeted APS genes in Arabidopsis thaliana[J]. Plant Divers,2016,38(2):92-100. doi:10.1016/j.pld.2015.04.001.
[21] Yuan N,Yuan S R,Li Z G,Li D Y,Hu Q,Luo H. Heterologous expression of a rice miR395 gene in Nicotiana tabacum impairs sulfate homeostasis[J]. Sci Rep,2016,6(1):28791. doi:10.1038/srep28791.
