磷作为维持植物生长发育等生命活动必需的重要矿质元素,对作物的新陈代谢、生长发育、高产及优质具有重要意义。土壤一旦缺磷,将会严重限制作物产量和品质的提升。在我国,土壤有效磷低于10 mg/kg的土壤面积占总耕地面积的50%左右[1]。即使土壤中总磷含量较高,绝大部分有效磷也会很快被土壤胶体吸附或固定,只有20%~30%的磷酸盐被植物吸收和利用[2]。虽然施用磷肥能缓解土壤中磷素供应不足,但是磷矿资源枯竭已成为一个全球普遍关心的问题,而且过度施用磷肥会使水体富营养化。当土壤磷含量较低时,磷高效型品种通过根系吸收、磷素的转运能力和体内对磷同化代谢的能力(代谢效率)获取足够的有效磷来维持植株的正常生长[3]。因此,利用分子生物学手段解析植物自身转换有机磷的分子机制,对提高作物产量和品质具有重要的生态环境意义。
酸性磷酸酶(Acid Phospoatase,ACP)是一种在酸性条件下水解磷酸酯并释放出磷酸的酶,普遍存在于植物组织中,与植物体的有机磷分解再利用有着密切的关系。目前,关于低磷诱导的磷酸酶研究可分为2类:一类是分泌型酸性磷酸酶,能将衰老组织中的有机磷再活化并被转运至幼嫩组织中,如Robinson等[4]研究发现,紫色酸性磷酸酶AtPAP26可促进拟南芥衰老叶片磷的再活化和再利用过程;而另一类是细胞内的酸性磷酸酶,在磷饥饿条件下,细胞内的活化酸性磷酸酶将液泡内的有机磷转化为无机磷,并通过液泡膜上的磷转运蛋白向细胞质转运,从而维持细胞质中磷含量的动态平衡。近10 a来,关于分泌型紫色酸性磷酸酶(PAP)的研究较多,主要集中在根系分泌紫色酸性磷酸酶的功能及其调控机制等领域[5],而对细胞内尤其是液泡内的酸性磷酸酶研究较少。2014年,Zhang等[6]通过全基因组关联分析方法研究发现了酸性磷酸酶基因GmACP1,过表达GmACP1的大豆耐低磷特性显著提高。由此可见,细胞内的酸性磷酸酶在调控植物有机磷代谢过程中同样起着非常重要的作用。
谷子(Setaria italica)作为传统优质杂粮作物之一,具有较强的耐贫瘠特性,其蕴藏着丰富的种质资源和重要的磷高效基因资源,如何快速鉴定耐低磷或磷高效基因的优异等位位点,并应用于作物遗传改良是目前种质资源和基因资源研究的中心任务之一。
鉴于ACP家族基因在植物耐低磷方面的重要性,本研究鉴定了谷子基因组的胞内酸性磷酸酶ACP家族基因,并通过候选基因进行关联分析和单倍型分析,初步检测到一个耐低磷的关键SiACP1基因及其优异单倍型,旨在为谷子SiACP家族基因的功能研究和耐低磷分子育种提供新的基因遗传信息。
1 材料和方法
1.1 谷子SiACP家族基因的鉴定和生物信息学分析
为了筛选较为可靠的谷子SiACP家族基因,首先以功能已知的大豆GmACP1(Glyma08g20820)[6]蛋白序列为模板,在大豆基因组(Wm82.a2.v1)数据库中进行同源序列BlastP比对(同源性P<10e-10),以找到大豆的所有ACP家族基因;再利用hmmbuild软件对检索到的大豆GmACP基因建立HMM模型(Hidden Markov Model,隐马尔科夫模型);然后使用Hmmsearch软件搜索谷子基因组(https://phytozome.jgi.doe.gov/,Setaria italica v2.2)中的ACP家族基因;最后,利用SMART网站(http://smart.embl-heidelberg.de/)检验谷子ACP家族基因的蛋白序列是否包括HAD保守结构域(PF12710),并将蛋白质分子质量大于0.2 ku的谷子SiACP基因作为谷子酸性磷酸酶SiACP家族基因成员。此外,利用ExPASy ProtParam在线软件(https://web.expasy.org/protparam/)分析谷子SiACP家族蛋白的分子量、等电点、疏水性等理化性质。
1.2 谷子SiACP家族基因系统进化分析
为了研究谷子、拟南芥、水稻、玉米、高粱和二穗短柄草ACP家族基因的进化关系,首先通过Clustal X2.1软件[7]比对不同物种的ACP蛋白序列。并使用Mega 7.0软件的Neighbor Joining方法展示不同物种ACP家族蛋白的系统发育树,参数Bootstrap值设为1 000次重复;并用FigTree软件构建不同物种间的ACP家族基因的系统进化树;然后使用Gene Structure Display Server软件[8]展示和绘制不同物种的ACP家族基因结构;最后,使用在线网站MEME(http://meme-suite.org/)[9]预测编码保守结构域的motif,motif的大小设定为5~300个氨基酸。
1.3 谷子SiACP家族基因的表达模式分析
基于水稻粳稻[10]、玉米B73[11]以及Phytozome数据库(https://phytozome.jgi.doe.gov)公布的高粱BT×623和谷子豫谷1号(Yugu 1)转录组(RNA-seq)数据,筛选出ACP家族基因的FPKM值。并利用R语言pheatmaps软件包绘制基因表达的热图,比较谷子和其他禾本科植物ACP家族基因在不同发育时期和不同组织表达模式的差异。
1.4 谷子SiACP基因变异位点与低磷相关性状的关联分析
为了鉴定到与耐低磷相关的谷子SiACP关键基因及其优异变异位点,采用了候选基因关联分析策略,基于中国农业科学院刁现民研究员团队[12]提供的重测序核心谷子种质,用TASSEL 3.0软件提取出SiACP在不同种质中的SNP和InDel,并将最小等位频率(MAF)阈值设为0.05;然后分析多态性位点(SNP和InDel)在SiACP基因上的具体分布(启动子、5′-UTR、3′-UTR、内含子、外显子)和距离起始密码子ATG的距离(变异位点信息);最后结合160份核心谷子种质在正常磷和低磷条件下的苗期株高、根长、地上干质量和根干质量表型数据[13],使用TASSEL 3.0软件的一般线性模型GLM+Q进行候选基因关联分析[14-15]。当表型与基因SiACP多态性位点的关联P值小于0.01时,可被认为基因SiACP中对应的多态性位点与谷子耐低磷相关性状存在显著关联。
1.5 谷子耐低磷SiACP基因的单倍型分析
基于候选基因关联分析结果,对耐低磷相关性状显著关联的基因SiACP进行单倍型分析。从160份种质中挑选出SiACP基因的多态性位点,使用独自开发的Perl脚本CandiHap.pl(https://github.com/xukaili/CandiHap)进行单倍型统计,并对不同单倍型的核心种质进化划分归类,去除掉位点为N(未知)的单倍型;最后用R语言的Ggplot2和Ggsignif软件包绘制对应的表型箱线图,并对不同单体型的耐低磷相关表型进行差异显著性分析。
2 结果与分析
2.1 谷子与其他3种禾本科植物ACP家族基因的鉴定及理化性质分析
以大豆酸性磷酸酶GmACP1基因的蛋白序列为靶序列[4],通过HMM模型预测和同源序列比对,从谷子和其他3种禾本科植物(水稻、高粱、玉米)的全基因组中共鉴定出13个ACP家族基因(表1),按照与大豆GmACP基因的序列同源性顺序进行编号,并根据物种拉丁文名称将4种禾本科ACP基因编号为SiACP1、SiACP2、SiACP3、ZmACP1、ZmACP2、ZmACP3、ZmACP4、SbACP1、SbACP2、SbACP3、OsACP1、OsACP2、OsACP3。通过生物信息学方法对所有禾本科植物ACP基因的染色体分布、编码区和氨基酸长度、分子质量、等电点进行分析。由表1可知,禾本科植物的ACP基因数目和染色体分布具有一定的规律性,谷子(基因组大小为491 Mb)、水稻(466 Mb)和高粱(730 Mb)基因组上均鉴定到3个ACP家族成员,均分布在2条染色体上。其中,不同物种同一条染色体上的2个ACP基因距离为6.40~7.95 Mb;而玉米基因组(2 300 Mb)上共鉴定到4个ACP基因,在3号染色体上也鉴定到2个ACP基因,其相互间距离为21.67 Mb。
表1 谷子和其他3种禾本科植物酸性磷酸酶ACP家族基因成员信息
Tab.1 Information of phosphatase ACP family genes in foxtail millet and other three gramineous plants
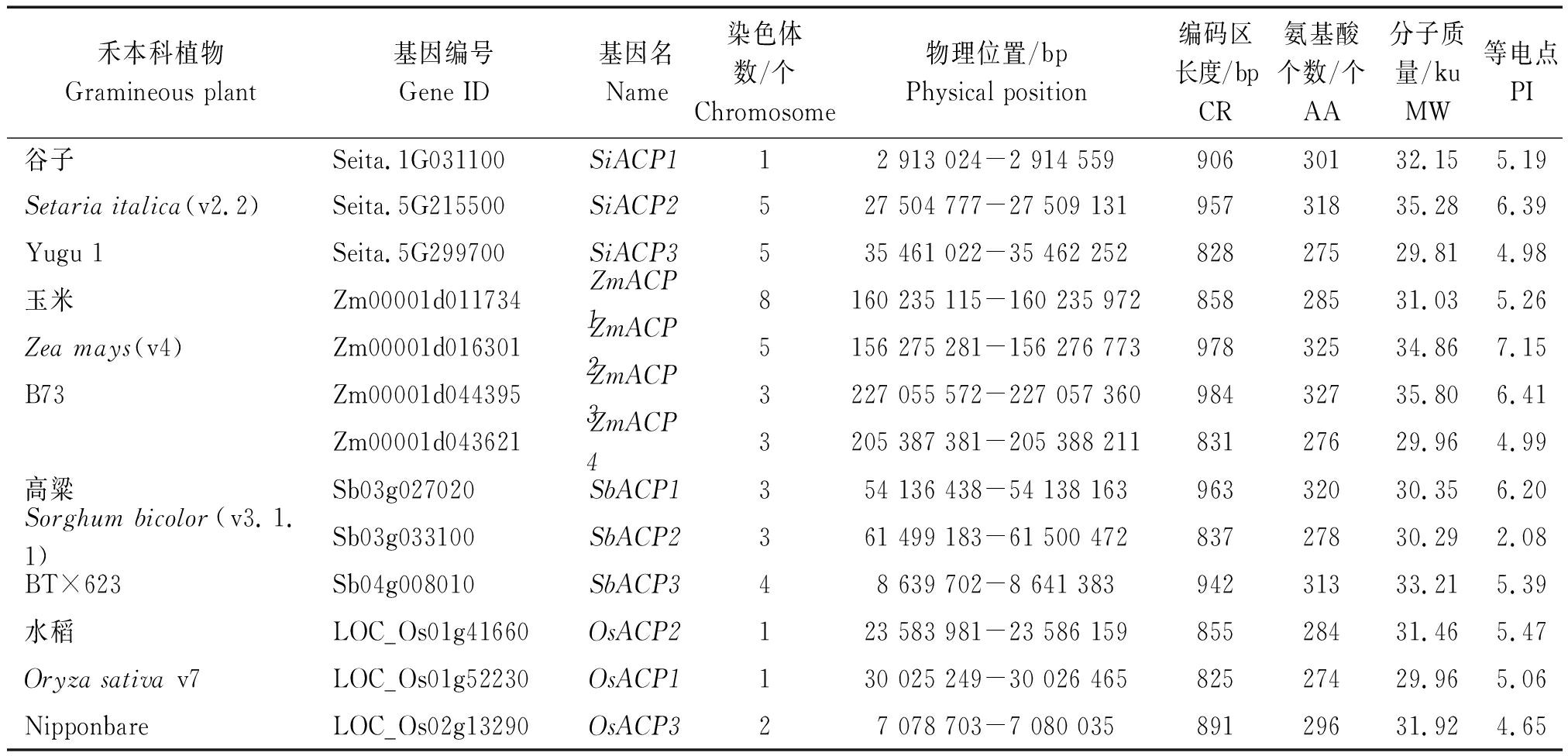
禾本科植物Gramineous plant基因编号Gene ID基因名Name染色体数/个Chromosome物理位置/bpPhysical position编码区长度/bpCR氨基酸个数/个AA分子质量/kuMW等电点PI谷子Seita.1G031100SiACP112 913 024-2 914 55990630132.155.19Setaria italica(v2.2)Seita.5G215500SiACP2527 504 777-27 509 13195731835.286.39 Yugu 1Seita.5G299700SiACP3535 461 022-35 462 25282827529.814.98玉米Zm00001d011734ZmACP18160 235 115-160 235 97285828531.035.26Zea mays(v4)Zm00001d016301ZmACP25156 275 281-156 276 77397832534.867.15B73Zm00001d044395ZmACP33227 055 572-227 057 36098432735.806.41Zm00001d043621ZmACP43205 387 381-205 388 21183127629.964.99高粱Sb03g027020SbACP1354 136 438-54 138 16396332030.356.20Sorghum bicolor(v3.1.1)Sb03g033100SbACP2361 499 183-61 500 47283727830.292.08BT×623Sb04g008010SbACP348 639 702-8 641 38394231333.215.39水稻LOC_Os01g41660OsACP2123 583 981-23 586 15985528431.465.47Oryza sativa v7LOC_Os01g52230OsACP1130 025 249-30 026 46582527429.965.06NipponbareLOC_Os02g13290OsACP327 078 703-7 080 03589129631.924.65
从编码区长度来看,4种禾本科植物的ACP基因编码区长度为825~984 bp,其中,最长编码区的基因和最短编码区的基因均出现在同一条染色体上。例如位于谷子第5号染色体上的SiACP2基因编码区长度为957 bp,SiACP2蛋白质由318个氨基酸组成;而SiACP3基因编码区长度为828 bp,其蛋白质由275个氨基酸组成。除位于5号染色体上的玉米ZmACP2基因等电点大于7外,其他禾本科植物的ACP蛋白质等电点均小于7,推测其编码弱酸性蛋白质,在酸性环境中发挥着一定的作用。鉴定到的所有ACP蛋白质分子质量为29.81~35.80 ku,平均为32.01 ku,且不同ACP蛋白质间的分子质量差异较小。
由此可见,ACP家族基因在禾本科植物的数量和分布具有一定的规律性。玉米的基因组大小(2 300 Mb)明显大于其他禾本科植物基因组大小(466~730 Mb),其含有4个ZmACP基因;而其他的禾本科植物只有3个ACP基因,其中有2个ACP基因位于同一条染色体上,其编码区长度和氨基酸长度均存在明显的差异。所鉴定到的大部分ACP基因可能在酸性环境中发挥着重要的生物学功能。
2.2 谷子和其他物种的ACP基因进化和结构分析
为了研究谷子SiACP家族基因的进化关系,利用谷子SiACP(Yugu 1)、水稻OsACP(粳稻)、玉米ZmACP(B73)、高粱SbACP(BT×623)、拟南芥AtACP(Col)、大豆GmACP(Williams 82)基因的蛋白序列构建了一个单子叶和双子叶物种间的进化关系树。根据系统发育树的结构可将ACP基因家族成员划分为2个亚家族,含6个簇(Ⅰ~Ⅵ)(图1-A)。其中,第1亚族包含了所有双子叶植物的大豆GmACP和拟南芥AtACP蛋白,除了大豆Glyma08g03350蛋白外,其他的大豆GmACP基因不仅蛋白序列的相似性较高,而且均被聚类到第1簇中;而第2亚族中包含了所有单子叶的禾本科植物,包括谷子SiACP、水稻OsACP、高粱SbACP和玉米ZmACP蛋白;第Ⅳ、Ⅴ、Ⅵ簇中至少包含一个SiACP、SbACP、ZmACP和OsACP基因家族成员。由此可见,单子叶植物与双子叶植物的ACP基因在进化过程中有着明显的序列差异,从而形成了单子叶植物ACP基因与双子叶植物ACP基因分支。从单子叶禾本科植物亲缘关系来看,C4植物中ZmACP蛋白与SbACP蛋白亲缘关系最近,与SiACP蛋白亲缘关系次之,而与C3植物水稻ACP蛋白亲缘关系相对较远。由此可见,在禾本科植物间ACP家族基因的亲缘关系划分符合植物分类学特性。
通过绘制不同单子叶植物和双子叶植物的ACP基因结构发现(图1-B),在单子叶植物中,第Ⅴ簇水稻、高粱、玉米和谷子的ACP基因不含内含子,大豆Glyma08g03350和水稻OsACP3基因只含一个内含子。由此推测,这些ACP基因可能产生或分化的时间较晚,其功能相对专一。而其他单双子叶植物的ACP基因至少含有2个内含子,例如SbACP3、ZmACP2和ZmACP3均含2个内含子 ,大豆Glyma15g02670基因含有5个内含子。谷子SiACP1、SiACP3、SiACP2基因分别含有2,0,3个内含子,表明SiACP基因在进化过程中其生物学功能具有明显的差异。
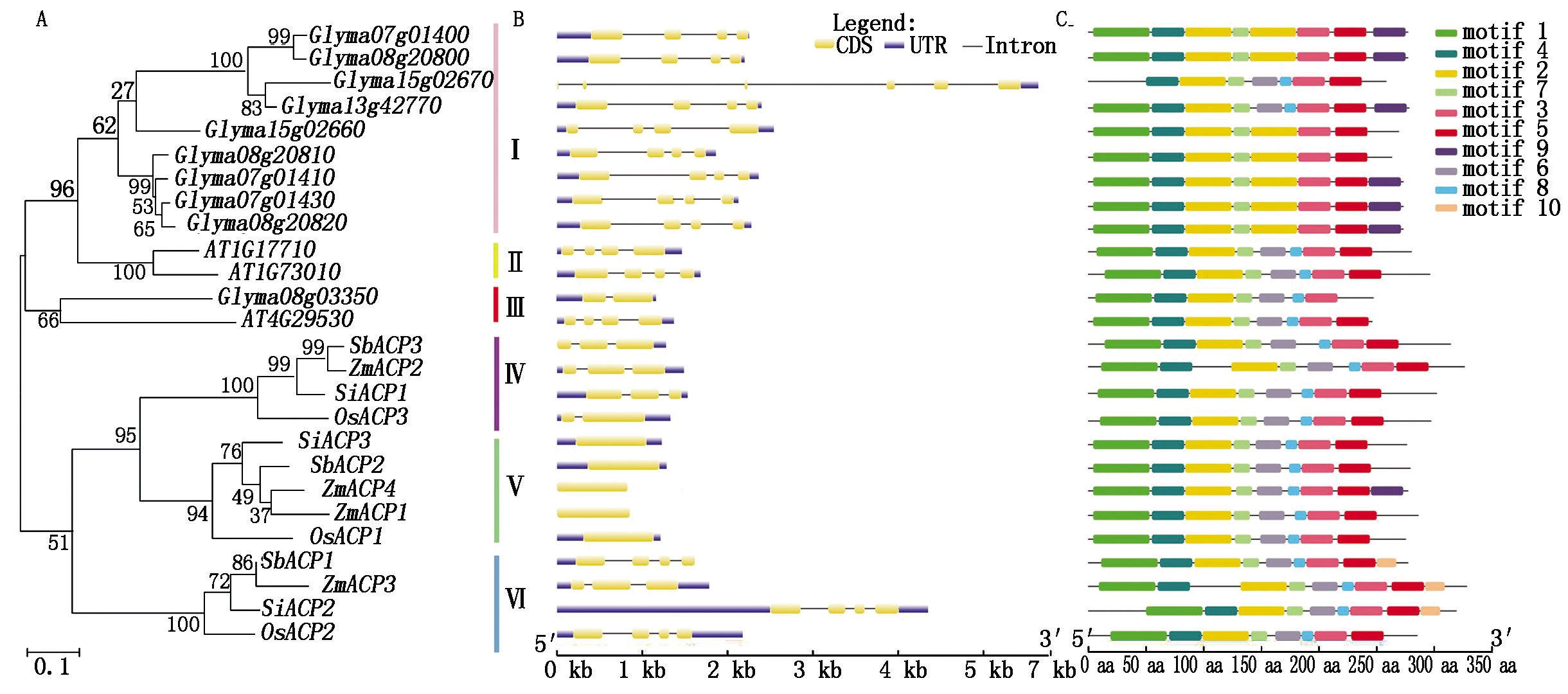
图1 单双子叶植物ACP家族基因进化关系(A)、基因结构分析(B)和蛋白motif组成成分分析(C)
Fig.1 The analysis of phylogenetic relationships (A),gene structure (B) and motif compositions (C) within ACP family genes in monocotyledon and dicotyledonous
从不同单双子叶植物ACP基因的蛋白序列中共检测到7~9个假定的保守基序(motif)(图1-C),包括motif 1、motif 2和motif 3编码的磷酸酶结构域。所有的ACP蛋白均包含3~4个磷酸酶结构域,同一簇的ACP蛋白保守基序类型及排列顺序基本一致。但是每个簇之间motif数目和组成成分存在一定的区别,例如第Ⅰ簇除了Glyma15g02670和Glyma13g42770蛋白序列包含motif 8外,其他的大豆GmACP蛋白序列均不包含motif 8;此外,Ⅱ~Ⅵ簇中除了ZmACP4外其他的ACP蛋白序列均包含motif 9。ACP家族蛋白除了磷酸酶结构域外,其他特异的假定motif是否决定了ACP蛋白在单、双子叶植物的功能差异,还需进一步分析研究。
2.3 谷子SiACP家族基因组织表达特异性分析
为了研究SiACP家族基因在谷子生长发育过程的作用,从不同物种的基因组数据库中筛选出ACP家族基因在水稻、高粱、玉米、谷子的不同组织(根、茎、叶、穗及籽粒)和不同发育时期的表达数据,并对不同物种的ACP基因表达水平绘制热图。由图2可知,不同禾本科植物的ACP基因表达模式具有明显的差异,同一基因在不同组织和不同发育时期表达模式也存在一定的差异。比如,在玉米的生长发育过程中,4个ZmACP基因均在不同组织中表达,且表达量都比较高,而其他3个物种中均有一个表达量相对较低的ACP基因(OsACP3、SbACP1、SiACP1)。

不同颜色表示基因在不同组织的lgFPKM值;DAS为播种后的天数;DAP为开花后的天数。
Different color shows genes at lgFPKM values of different tissues;DAS. Days after sowing; DAP. Days after pollination.
图2 禾本科植物水稻、高粱、玉米和谷子ACP基因在正常条件下不同组织的表达模式分析
Fig.2 Expression analysis of ACP family genes in rice,sorghum,maize and foxtail millet of gramineous plants
不同物种的ACP基因在不同组织与不同发育阶段所表现的时空特异性也存在一定的差异。在水稻中,OsACP1和OsACP2基因在根系和茎秆中的表达量相对较低,主要在叶片中表达,其FKPM值范围为28.70~164.09(图2-A),此外,OsACP1基因在雌蕊和花序中的表达量也相对较高;而OsACP3基因除了在穗部有一定的表达外(FKPM值为9.69),在其他组织中的表达量均很低或没表达。在高粱中,SbACP2和SbACP3基因在整个植株生长发育过程中都有一定的表达(图2-B)。从组织特异性来看,SbACP1基因除了在中部茎秆(FPKM值为26.15)和叶片(FPKM值为30.23)表达量相对较高外,在其他组织的表达量较低(FPKM值为2.55~7.07)。而SbACP2基因主要在茎秆和叶鞘部位表达,其FPKM值分别为26.16和30.23。SbACP3基因主要在根系、茎秆、节点和叶片部位表达,其FPKM值均大于26.06。在玉米中,除了ZmACP3在花药、花丝、胚乳、胚和苞叶组织中没有表达外,其他ZmACP基因均有相对较高的表达量,例如ZmACP1基因在根系和花丝中的FPKM值高达140,ZmACP2和ZmACP4基因在顶部叶片、节点、花药和苞叶的FPKM值均高于123(图2-C)。由此可见,玉米ACP基因主要在生长发育旺盛的组织中表达。在谷子中,所有ACP基因均有一定的表达,其FPKM值为9.21~491.51(图2-D)。从不同组织的基因表达情况看,SiACP1主要在根系中表达,且表达表现出了明显的组织特异性;SiACP2和SiACP3在整个生育期的不同组织中均表达,且表达量都比较高,其FPKM均值为96.70。
由此可见,高粱SbACP和玉米ZmACP基因在不同组织中的表达量明显高于谷子SiACP和水稻OsACP。禾本科作物中,水稻OsACP1基因、高粱SbACP3基因、玉米ZmACP2基因和ZmACP4基因、谷子SiACP2基因表达量明显高于其他ACP基因,这些ACP基因不仅在不同组织发育过程中起着非常重要的作用,而且在植株最高处的叶片ACP基因表达量明显高于基部或中间部位叶片ACP基因。
2.4 谷子SiACP基因的多态性位点与耐低磷相关性状的关联分析
为了分析谷子SiACP1、SiACP2和SiACP3基因在谷子核心种质资源中的多态性,以及挖掘耐低磷相关性状的优异变异位点,本研究基于Jia等[12]利用高通量测序Illumina HiSeq 2000平台重测序的916份谷子种质资源,挑选出具有耐低磷相关表型的160份谷子种质基因型[13],分析了SiACP1、SiACP2、SiACP3基因在试验材料中的变异位点和序列多样性,包括起始密码子ATG上游1 000 bp的启动子。通过过滤掉缺失率和杂合度大于20%、最小等位频率(MAF)小于0.05的多态性位点,最终在SiACP1基因中共检测到87个SNP和2个InDel(图3-A),平均每28 bp存在一个变异位点(SNP或InDel);在SiACP2基因中共检测到11个SNP(图3-B),平均每486 bp有一个SNP;在SiACP3基因中共检测到21个SNP和7个InDel,平均每79 bp有一个变异位点(SNP或InDel)(图3-C)。SiACP基因的核苷酸多态性位点非均匀地分布在启动子、5′-UTR、编码区、非编码区以及3′-UTR。其中,多态性位点最多的SiACP1基因启动子(包含5′-UTR区和3′-UTR区)(π>7.4×10-2)的多态性最高。

图3 谷子SiACP基因的多态性位点分布及其与低磷条件下苗期相关表型的关联分析
Fig.3 Locus distribution of SiACP genes and associate analysis between natural variation in SiACPs and correlated phenotype under low Pi conditions at seedling stage
为了筛选耐低磷相关的关键SiACP基因和优异等位变异位点,本研究基于前期160份谷子核心种质的苗期株高(PH)、根长(RL)、地上部干质量(DW above)和根干质量(RDW)表型数据[13],对候选基因SiACP1、SiACP2和SiACP3进行关联分析。由图3-D、E、F可知,利用GLM+Q模型,在阈值为0.01水平下共鉴定到16个SNP与耐低磷相关性状存在显著关联(表2)。在SiACP1基因的多态性位点中,共检测到11个SNP与低磷条件下株高(PH)、相对株高(RPH)和相对根长(RRL)存在显著关联(P<6.83×10-3),其中位于起始密码子ATG上游第459 bp、第556 bp和第二外显子第1 100 bp的SNP均与LPH(低磷条件的株高)、RPH、RRL表型存在显著关联,但是位于编码区的显著SNP为非同义突变。此外,位于5′UTR区的2个SNP均与LPH、RPH存在显著关联;在SiACP2基因中共检测出5个SNP,仅与低磷条件下相对根长(RRL)存在显著关联(图3-F),并没有鉴定到与表现共性的显著位点,其中,有3个SNP位于基因下游,2个SNP位于SiACP2基因的第2和第3外显子区,其中在起始密码子604 bp处的SNP导致氨基酸从丙氨酸变成甘氨酸;在SiACP3基因的多态性位点中,并没有鉴定到与耐低磷相关性状显著关联的位点。由此可见,SiACP1的启动子区不仅具有较高的多态性,而且存在多个与低磷条件下的株高、相对株高和相对根长表型显著关联的共性SNP,故推断谷子SiACP1基因是导致低磷条件下谷子种质资源表型变异的主要关键细胞内酸性磷酸酶基因。
表2 基因SiACP多态性位点与耐低磷相关性状的关联分析
Tab.2 Associations between the natural variations within SiACPs and phenotype related to low-phosphorous tolerance
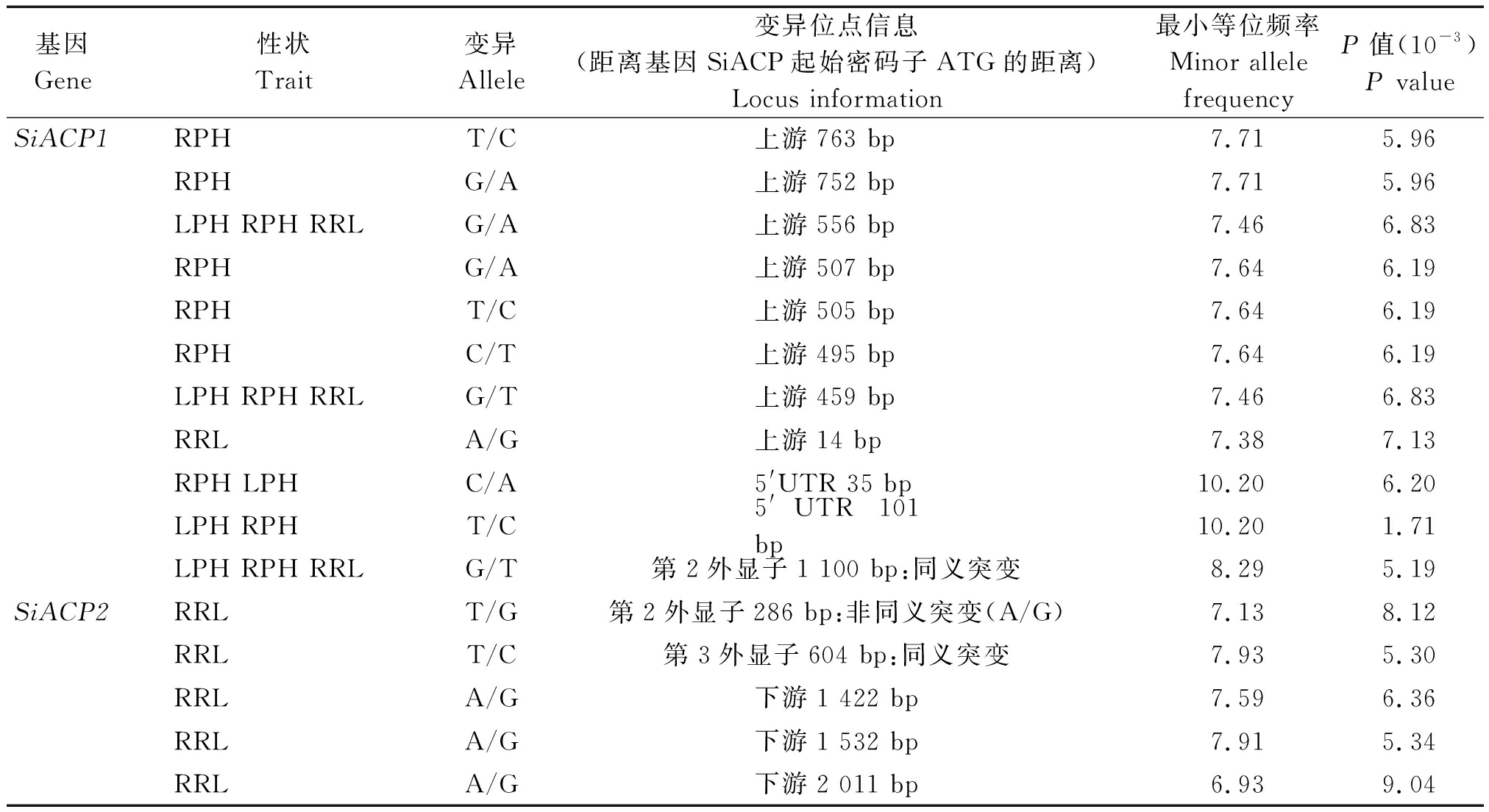
基因Gene性状Trait变异Allele变异位点信息(距离基因SiACP起始密码子ATG的距离)Locus information最小等位频率Minor allelefrequencyP值(10-3)P valueSiACP1RPHT/C上游763 bp7.715.96RPHG/A上游752 bp7.715.96LPH RPH RRLG/A上游556 bp7.466.83RPHG/A上游507 bp7.646.19RPHT/C上游505 bp7.646.19RPHC/T上游495 bp7.646.19LPH RPH RRLG/T上游459 bp7.466.83RRLA/G上游14 bp7.387.13RPH LPHC/A5'UTR 35 bp10.206.20LPH RPHT/C5'UTR 101 bp10.201.71LPH RPH RRLG/T第2外显子1 100 bp:同义突变8.295.19SiACP2RRLT/G第2外显子286 bp:非同义突变(A/G)7.138.12RRLT/C第3外显子604 bp:同义突变7.935.30RRLA/G下游1 422 bp7.596.36RRLA/G下游1 532 bp7.915.34RRLA/G下游2 011 bp6.939.04
2.5 谷子SiACP1基因变异位点的单倍型分析
基于上述候选基因关联分析结果,为了进一步确定SiACP1启动区的SNP是影响ACP基因功能的主要因素,从160份谷子核心种质中筛选到的所有SiACP1的SNP和InDels位点进行单倍型分析。在检测到的89个多态性位点中(SNP/InDel),去除掉SiACP1基因变异位点为N(未知)的种质,最终得到32个SNP和1个InDel。通过对此33个多态性位点的随机组合进行分析,结果共得到7种单倍型,将138份种质不均匀地划分成7种单体型(图4)。其中,有99份种质被划分为ACPHap1(SiACP1基因的第一个单倍型),9份和10份种质(单体型)分别包括单倍型ACPHap2和ACPHap4,而ACPHap3单体型只包含1份种质B078,而其余的ACPHap5、ACPHap6和ACPHap7单体型中均存在杂合变异位点。
通过分析单倍型ACPHap1和ACPHap4的SNP组合时发现,ACPHap1和ACPHap4单倍型存在一个位于基因起始密码子下游1 770 bp的SNP差异,该位点引起非同义突变;而比较单倍型ACPHap1和ACPHap2的SNP组合来看,二者之间存在31个SNP和1个InDel差异。通过整合基因SiACP1多态性位点与耐低磷相关性状的关联分析结果,在与株高、相对株高、相对根长存在显著关联的5个SNP中,有4个SNP是单倍型ACPHap2和ACPHap3所特有的SNP。由此可见,由纯合变异位点组合成的单倍型ACPHap1、ACPHap2和ACPHap3是影响基因SiACP1功能变异的主要单倍型。
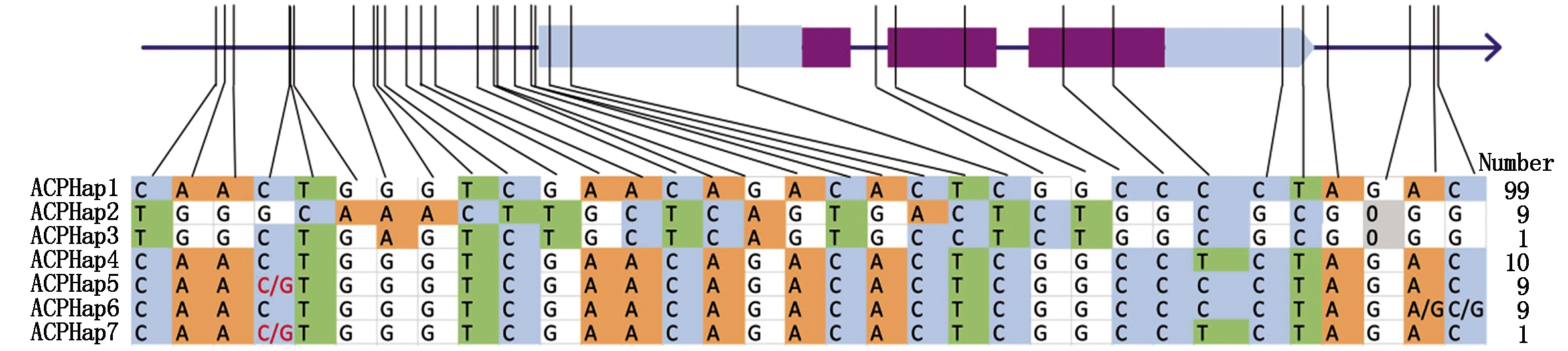
最右侧的数字代表每一个单倍型包含的种质数目。
The right most numbers represents the accessions numbers contenting each haplotype.
图4 基因SiACP1的7种单倍型分布及其对应的谷子单体型数目
Fig.4 Distributions of the 7 haplotypes in SiACP1 gene and its corresponding haplotype number of foxtail millet accessions
鉴于具有ACPHap3单倍型的种质(1份)不能进行表型统计分析外,本研究对ACPHap1、ACPHap2单体型在低磷条件下株高、根长、根干质量、地上部干质量的表型进行T检验差异显著分析(图5)。由图5可知,ACPHap1单体型与ACPHap2单体型在正常磷和低磷条件下株高存在显著差异(P<0.05),而其他表型包括根长、根干质量、地上部干质量均不显著。在同一条件下,对ACPHap1、ACPHap2单体型的相关性状进行方差分析发现,在正常磷条件下ACPHap2单体型株高比ACPHap1单体型株高显著增加了11.92%(3.98 cm,P=0.02)。在低磷条件下,与ACPHap1单体型株高和根长相比,ACPHap2单体型株高显著增加了29.70%(6.60 cm,P=0.001 3),ACPHap2单体型的根长也增加了1.91 cm,但差异未达到显著水平(P=0.074)。

横线上方的数字代表t检验的P值。图6同。
The numbers above the line represent the P value of t test. The same as Fig.6.
图5 不同单体型在正常磷和低磷条件下株高、根长、根和地上部干质量的差异比较分析
Fig.5 The comparative analysis between different haplotype for plant height,root length,
root dry weight and dry weight of aerial part under normal phosphorus and low phosphorus conditions
通过比较分析正常磷和低磷条件下的株高、根长、地上部、根干质量的相对值发现,ACPHap1单体型与ACPHap2单体型的相对株高、相对根长、根干质量存在显著差异(P<0.05)(图6)。例如,ACHap1和ACPHap2单体型的相对株高平均值分别为33.45%和-23.14%;而ACPHap2和ACPHap1单体型的相对根长平均分别为17.38%和-0.02%;此外,ACPHap2单体型的相对根干质量为-2.36%,而ACPHap1单体型的相对根干质量为-22.83%。由此可见,在低磷条件下,ACPHap2单体型的生长发育过程中的根长、株高、地上部和根干质量方面受低磷影响较小,表现出了较强的耐低磷特性。
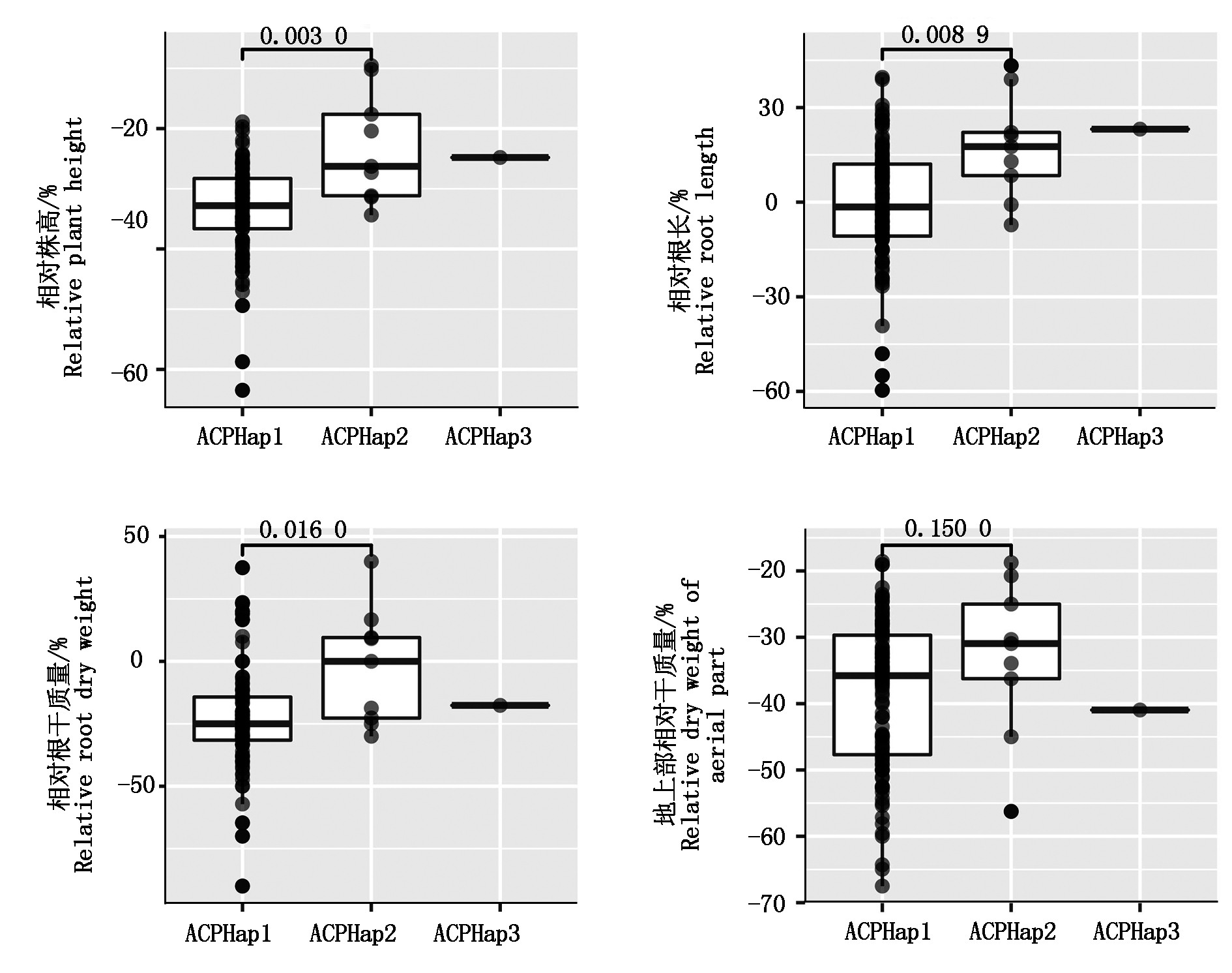
图6 不同单体型在正常磷和低磷条件下的株高、根长、根和地上部干质量相对值比较差异分析
Fig.6 The comparative analysis between different haplotype for relative plant height,root length,root dry weight and dry weight of aerial part under normal phosphorus and low phosphorus conditions
3 结论与讨论
磷是植物生长发育过程中必需的大量营养元素之一,土壤中有效磷含量过低是限制作物高产优质的主要因素。植物在长期进化过程中形成了一系列适应低磷诱导的分子机制,包括重塑根系的形态构型促进根系吸收更多的有效磷、体内有机磷的高效利用[5]。磷酸酶不仅是一种重要的水解酶,参与碳水化合物转化和蛋白质合成;而且是一种受低磷诱导酶,在土壤中与植株体内的有机磷的分解和再利用有着密切的关联。张丽梅等[16]研究缺磷对不同耐低磷玉米基因型酸性磷酸酶活性的影响发现,缺磷使得玉米地上部干质量下降,根干质量、根冠比增加,磷诱导玉米叶片、根组织和根系分泌APase活性升高。当植物受到低磷诱导时,植物体内会合成大量的去磷酸化酶相关基因,从不同的有机磷底物上水解磷酸基团,增加体内的有效磷素,提高植物在缺磷环境对磷的耐性及磷的高效利用。
细胞内的酸性磷酸酶能够将植物液泡内的有机磷转化为无机磷,并通过液泡膜上的磷转运蛋白转向细胞质,维持细胞质中磷含量的动态平衡。在番茄植物中,成功克隆到了一个低磷诱导的酸性磷酸酶基因LePS2,能编码269个氨基酸,该基因只能在低磷条件诱导表达,而不受其他营养物质或非生物胁迫影响[17]。Tian等[18]通过cDNA文库鉴定一个编码271个氨基酸的酸性磷酸酶基因PvPS2发现,该基因在磷饥饿条件下被诱导高表达,且有助于改良菜豆根系形态和结构,耐低磷的植株内磷的转运和酸性磷酸酶活性均增加。Wasaki等[19]在低磷胁迫下对白羽扇豆根分泌酸性磷酸酶基因表达研究发现,植株体内的磷含量降低促进了酸性磷酸酶ACP基因的表达,并加快了丛生根的形成。在大豆中,低磷条件下植物体内的GmACP1基因表达量显著增强,导致酸性磷酸酶活性升高,显著提高植物的耐低磷能力[6]。本研究通过一个功能已知的耐低磷基因GmACP1进行BlastP同源比对,从谷子和其他3种禾本科植物的全基因组水平上共鉴定出13个ACP基因,ACP家族成员的数量和分布具有一定的规律性,均具有高度保守的磷酸酶结构域。其中,在谷子中,本研究共鉴定的3个等电点均小于7的酸性磷酸酶基因SiACP,在酸性环境中可能着一定特殊的生物学功能。家族基因进化树分析发现,单子叶植物ACP基因与双子叶植物ACP基因的蛋白序列、基因结构存在明显的差别,推测在ACP家族基因的进化过程出现在双子叶植物纲和单子叶植物形成之前。在单子叶禾本科植物中,属于C4植物的谷子SiACP基因、高粱SbACP基因和玉米ZmACP基因之间的亲缘关系较近。
谷子作为传统优质杂粮作物,具有耐贫瘠特性,其蕴藏着重要的磷高效基因资源。挖掘谷子优异的磷高效等位变异位点,并通过分子标记加快培育磷高效品种,是解决土壤有效磷缺乏、降低种植成本的主要遗传育种途径之一。近几年,随着测序技术的发展和基因组学研究的不断革新,关联分析成为发掘与目标性状显著有关联的变异位点最有效的数量遗传学手段。Weng等[20]利用175个优良自交系鉴定基因ZmIPT2位点多态性,并与粒质量进行关联分析,结果检测到16个SNP和7个单倍型与粒质量显著关联。Liu等[21]利用候选基因关联策略对玉米ZmDREB基因和耐旱相关性状进行关联分析,结果发现,ZmDREB2.7基因的启动子区InDel位点与根长存在显著关联,该位点是导致ZmDREB2.7基因功能差异的关键位点,此研究不仅解析了玉米耐旱机制,而且为分子设计育种提供了新的选择靶点。此外,Wang等[22]研究发现,ZmVPP1基因启动子区的一个InDel与耐旱性状显著关联,并且发现该InDel是3个MYB反式作用因子,调控着基因ZmVPP1表达,从而提高植株耐旱性。在逆境过程中,基因的启动子在基因表达调控过程中起着重要的作用[23]。
本研究通过候选基因关联分析发现,基因SiACP1的启动子不仅具有多态性,而且存在着多个与低磷条件下的株高、相对株高和相对根长表型显著关联的共性SNP。进一步对SiACP1基因的多态性位点进行单倍型分析,可将138份谷子核心种质划分成7种单体型,结合耐低磷相关表型并成功鉴定到一个与耐低磷相关的有利的单倍型ACPHap2。虽然与Zhang等[6]研究发现的与大豆磷高效相关GmACP1基因的单倍型位置有一定区别(内含子和外显子),但是单子叶植物ACP基因与双子叶植物ACP基因进化过程中出现分歧,推测位于SiACP1基因启动子区的SNP很有可能是影响谷子种质在低磷条件下相关表型变化的主要原因。这些结果均证明,SiACP1基因在进化过程中自然变异位点对于抵御土壤磷缺乏较为重要,将为后期大规模筛选耐低磷种质和遗传育种改良新品种提供一定的基因信息。
[1] 曹宁, 陈新平, 张福锁, 曲东. 从土壤肥力变化预测中国未来磷肥需求[J]. 土壤学报,2007,44(3):536-543. doi:10.11766/trxb200601060322.
Cao N,Chen X P,Zhang F S, Qu D. Prediction of phosphate fertilizer demand in China based on change in soil phosphate fertility[J]. Acta Pedologica Sinica,2007,44(3):536-543.
[2] 邓美菊, 王飞, 毛传澡. 植物磷酸盐转运体及其分子调控机制[J]. 植物生理学报,2017,53(3):377-387. doi:10.13592/j.cnki.ppj.2017.0003.
Deng M J,Wang F,Mao C Z. Plant phosphate transporters and its molecular regulation mechanism[J]. Plant Physiology Journal,2017,53(3):377-387.
[3] 李喜焕, 常文锁, 张彩英. 提高植物磷营养效率(候选)基因研究进展[J]. 植物遗传资源学报,2012,13(1):83-97. doi:10.3969/j.issn.1672-1810.2012.01.014.
Li X H,Chang W S,Zhang C Y. Research progress of candidate genes for improving plant phosphorus-effiency[J]. Journal of Plant Genetic Resources,2012,13 (1):83-97.
[4] Robinson W D,Carson I,Ying S,Ellis K,Plaxton W C. Eliminating the purple acid phosphatase AtPA26 in Arabidopsis thaliana delays leaf senescence and impairs phosphorus remobilization[J]. New Phytologist,2012,196 (4):1024-1029. doi:10.1111/nph.12006.
[5] 田江, 梁翠月, 陆星, 陈倩倩. 根系分泌物调控植物适应低磷胁迫的机制[J]. 华南农业大学学报,2019,40 (5):175-185. doi:10.7671/j.issn.1001-411X.201905068.
Tian J,Liang C Y,Lu X,Chen Q Q. Mechanism of root exudates regulating plant responses to phosphorus deficiency[J]. Journal of South China Agricultural University,2019,40 (5):175-185.
[6] Zhang D,Song H N,Cheng H,Hao D R,Wang H,Kan G Z,Jin H X,Yu D Y. The acid phosphatase-encoding gene GmACP1 contributes to soybean tolerance to low-phosphorus stress[J]. PLoS Genetics,2014,10 (1):e1004061. doi:10.1371/journal.pgen.1004061.
[7] Larkin M A,Blackshields G,Brown N P,Chenna R,McGettigan P A,McWilliam H,Valentin F,Wallace I M,Wilm A,Lopez R,Thompson J D, Gibson T J, Higgin D G. ClustalW and ClustalX version 2.0[J]. Bioinformatics,2007,23(21):2947-2948. doi:10.1093/bioinformatics/btm404.
[8] Hu B,Jin J P,Guo A Y,Zhang H,Luo J C,Gao G. GSDS 2.0:an upgraded gene feature visualization server[J]. Bioinformatics,2015,31(8):1296-1297. doi:10.1093/bioinformatics/btu817.
[9] Bailey T L,Boden M,Buske F A,Frith M,Grant C E,Clementi L,Ren J Y,Li W W,Noble W S. MEME SUITE:tools for motif discovery and searching[J]. Nucleic Acids Research,2009,37 (S2):202-208. doi:10.1093/nar/gkp335.
[10] Xia L,Zou D,Sang J,Xu X J,Yin H Y,Li M W,Wu S Y,Hu S N,Hao L L,Zhang Z. Rice Expression Database (RED):An integrated RNA-Seq-derived gene expression database for rice[J]. Journal of Genetics and Genomics,2017,44 (5):235-241. doi:10.1016/j.jgg.2017.05.003.
[11] Stelpflug S C,Sekhon R S,Vaillancourt B,Hirsch C N,Buell C R,de Leon N,Kaeppler S M. An expanded maize gene expression atlas based on RNA sequencing and its use to explore root development[J]. The Plant Genome,2016,9 (1):1-16. doi:10.3835/plantgenome2015.04.0025.
[12] Jia G Q, Huang X H, Zhi H, Zhao Y, Zhao Q, Li W J, Chai Y, Yang L F, Liu K Y, Lu H Y, Zhu C R, Lu Y Q, Zhou C C, Fan D L, Weng Q J, Guo Y L, Huang T, Zhang L, Lu T T, Feng Q, Hao H F, Liu H K, Lu P, Zhang N, Li Y H, Guo E H, Wang S J, Wang S Y, Liu J R, Zhang W F, Chen G Q, Zhang B J, Li W, Wang Y F, Li H Q, Zhao B H, Li J Y, Diao X M, Han B. A haplotype map of genomic variations and genome-wide association studies of agronomic traits in foxtail millet (Setaria italica)[J]. Nature Genetics,2013,45 (8):957-961. doi:10.1038/ng.2673.
[13] 苑乂川, 陈小雨, 李明明, 李萍, 贾亚涛, 韩渊怀, 邢国芳. 谷子苗期耐低磷种质筛选及其根系保护酶系统对低磷胁迫的响应[J]. 作物学报,2019,45 (4):601-612. doi:10.3724/SP.J.1006.2019.82029.
Yuan Y C,Chen X Y,Li M M,Li P,Jia Y T,Han Y H,Xing G F. Screening of germplasm tolerant to low phosphorus of seedling stage and response of root protective enzymes to low phosphorus in foxtail millet[J]. Acta Agronomica Sinica,2019,45 (4):601-612.
[14] Bradbury P J,Zhang Z W,Kroon D E,Casstevens T M,Ramdoss Y,Buckler E S. TASSEL:software for association mapping of complex traits in diverse samples[J]. Bioinformatics,2007,23 (19):2633-2635. doi:10.1093/bioinformatics/btm308.
[15] Jan H U,Guan M,Yao M,Liu W,Wei D Y,Abbadi A,Zheng M,He X,Chen H,Guan C Y,Nichols R A, Snowdon R J, Hua W, Qian L W. Genome-wide haplotype analysis improves trait predictions in Brassica napus hybrids[J]. Plant Science,2019,283:157-164. doi:10.1016/j.plantsci.2019.02.007.
[16] 张丽梅, 郭再华, 张琳, 贺立源. 缺磷对不同耐低磷玉米基因型酸性磷酸酶活性的影响[J]. 植物营养与肥料学报,2015,21 (4):898-910. doi:10.11674/zwyf.2015.0408.
Zhang L M,Guo Z H,Zhang L,He L Y. Effects of phosphate deficiency on acid phosphatase activities of different maize genotypes tolerant to low-P stress[J]. Joumal of Plant Nutrition and Fertilizers,2015,21 (4):898-910.
[17] Baldwin J C,Karthikeyan A S,Raghothama K G. LEPS2,a phosphorus starvation-induced novel acid phosphatase from tomato[J]. Plant Physiology,2001,125 (2):728-737. doi:10.1104/pp.125.2.728.
[18] Tian J,Venkatachalam P,Liao H,Yan X L,Raghothama K. Molecular cloning and characterization of phosphorus starvation responsive genes in common bean (Phaseolus vulgaris L.)[J]. Planta,2007,227 (1):151-165. doi:10.1007/s00425-007-0603-2.
[19] Wasaki J,Yamamura T,Shinano T,Osaki M. Secreted acid phosphatase is expressed in cluster roots of lupin in response to phosphorus deficiency[J]. Plant and Soil,2003,248(1-2):129-136. doi:10.1023/A:1022332320384.
[20] Weng J F,Li B,Liu C L,Yang X Y,Wang H W,Hao Z F,Li M S,Zhang D G,Ci X K,Li X K,Zhang S H. A non-synonymous SNP within the isopentenyl transferase 2 locus is associated with kernel weight in Chinese maize inbreds (Zea mays L.)[J]. BMC Plant Biology,2013,13 (1):98. doi:10.1186/1471-2229-13-98.
[21] Liu S X, Wang X L, Wang H W, Xin H B, Yang Xi H, Yan J B, Li J S, Tran L S P, Shinozaki K, Yamaguchi-Shinozaki K, Qin F. Genome-wide analysis of ZmDREB genes and their association with natural variation in drought tolerance at seedling stage of Zea mays L.[J]. PLoS Genetics,2013,9 (9):e1003790. doi:10.1371/journal.pgen.1003790.
[22] Wang X L, Wang H W, Liu S X, Ferjani A, Li J S, Yan J B, Yang X H, Qin F. Genetic variation in ZmVPP1 contributes to drought tolerance in maize seedlings[J]. Nature Genetics,2016,48 (10):1233-1241. doi:10.1038/ng.3636.
[23] Zhu L P,Yu Z,Zou C X,Li Q L. Plant stress-inducible promoters and their function[J]. Hereditas,2010,32 (3):229-234. doi:10.3724/sp.j.1005.2010.00229.
