羊传染性脓疱皮炎俗称“羊口疮”,是由羊口疮病毒(Orf virus,ORFV)引起的一种急性、接触性、嗜上皮性的人兽共患传染病[1]。该病毒主要感染绵羊及山羊,特别是羔羊,人类接触也可感染[2]。临床上主要以感染动物的唇、鼻孔、口腔黏膜、乳房、阴部等部位皮肤形成红斑、水疱、脓疱、丘疹和疣状痂皮为特征[3-5]。初生羔羊和幼龄羊感染ORFV后发病率高达100%,死亡率达93%;虽然成年羊感染ORFV后的死亡率较低,但由此引起的采食困难,会导致机体消瘦、生长缓慢,以及继发感染,带来巨大的经济损失。近年来,随着我国养羊数量增加和跨境流通,ORF作为人畜共患的传染病,对人类的健康也产生了威胁,受到社会的广泛关注。然而,由于ORFV特有的免疫机制和免疫逃逸机制,目前的疫苗并不能为羊提供完全的免疫保护。同时,现有的针对羊口疮的治疗药物大多为“对症治疗”的缓解性药物,其治疗效果不甚理想,且费时费力。羊口疮难以根治的特点和反复发作的现状已成为制约养羊产业发展的瓶颈。
ORFV属于痘病毒科副痘病毒属,为双股DNA病毒,共包含130多个基因[6]。由末端基因区(ORFVs001-008和ORFVs112-134)和中间核心基因区(ORFVs009-111)组成,中间核心区包含众多保守基因,多与病毒的复制和形态相关,末端基因区多为非保守基因,与病毒毒力和致病性相关[7-9]。其中末端基因中包括5种编码锚蛋白的基因,分别为ORFV008、ORFV123、ORFV126、ORFV128和ORFV129。ORFV129蛋白与其他ORFV锚蛋白具有相似的结构,包含多个锚蛋白重复基序(ANKs)和1个F-Box样结构域[10-11]。ORFV129蛋白的功能目前不清楚,而蛋白质生物学功能的发挥往往与该蛋白的亚细胞定位密切相关[12]。本研究旨在克隆羊口疮病毒ORFV129基因全长,并对其进行生物信息学分析。进一步构建真核表达载体pEGFP-ORFV129,转染新生山羊睾丸原代细胞研究该蛋白在细胞中的定位特征,为进一步研究ORFV129在诱导机体免疫应答中的作用奠定基础。
1 材料和方法
1.1 毒株、菌株、真核质粒和细胞
羊口疮病毒JYY-ORFV株、新生山羊睾丸原代细胞和pEGFP-N1载体及大肠杆菌DH5α购自宝生物工程(大连)有限公司(TaKaRa)。
1.2 主要试剂及仪器
Premix Taq、DL2000 DNA Marker、T4DNA连接酶均购自宝生物工程(大连)有限公司(TaKaRa);Hind Ⅲ、Pst Ⅰ购自上海玉博生物科技有限公司;胶回收试剂盒、质粒提取试剂盒均购自美国Omega公司;DMEM培养基、胎牛血清和OPTI-MEM 1(1X)均购自美国Gibco公司;RAPI裂解液、广谱蛋白酶抑制剂、广谱磷酸酶抑制剂、组织固定液、一抗鼠源GFP、一抗兔源GADPH均购自武汉博士德生物工程有限公司;HRP标记的山羊抗鼠IgG购自武汉亚科因生物技术有限公司;HRP标记的山羊抗兔IgG购自北京博奥龙免疫技术有限公司;DAPI核酸染料、LipoGeneTM 2000均购自US Everbright® Inc.(简称UE)公司;Triton X-100、DNase Ⅰ均购自广州赛国生物科技有限公司;荧光倒置显微镜购自日本奥林巴斯(Olympus Corporation);激光共聚焦显微镜购自德国卡尔·蔡司公司股份公司(Carl Zeiss AG)。
1.3 ORFV129基因全长的克隆鉴定及生物信息学分析
参照GenBank中登录的羊口疮病毒SY 17株ORFV129(登录号:MG 712417.1),运用Primier Primer 5设计1对特异性引物。上游引物:5′-AACGCCACGCCATGGACGC-3′;下游引物:5′-GATGGGCAGTCACTCGGGCG-3′,引物由上海生工生物工程有限公司合成。选取接种了ORFV且具有明显细胞病变的新生山羊睾丸原代细胞培养物反复冻融3次,12 000 r/min离心2 min,留上清,使用DNA提取试剂盒提取病毒DNA。以提取的病毒DNA为模板,使用上下游引物进行PCR反应,特异性扩增全长ORFV129基因。反应体系:Pre mix Taq 10 μL,上、下游引物各1 μL,模板1 μL,其余用ddH2O补足20 μL。反应条件:94 ℃预变性4 min;94 ℃变性30 s,65 ℃退火30 s,72 ℃延伸1 min,共35个循环;72 ℃再延伸7 min。PCR产物经1.5%琼脂糖凝胶电泳进行检测,切胶回收产物连接pMD19-T载体,并转化DH5α感受态细胞,提取质粒后阳性的重组质粒送上海生工生物工程有限公司测序后保存备用,将鉴定正确的质粒命名为pMD19-ORFV129。对测序鉴定正确的ORFV 129全基因序列进行相关结构和功能的预测:使用SMART(http://smart.emblheidelberg.de/smart/set_mode.cgi?NORMAL=1)软件进行一级结构分析;使用TMHMM Server v. 2.0(http://www.cbs.dtu.dk/services/TMHMM-2.0/)软件进行跨膜区域分析;使用在线软件SignalP 5.0(http://www.cbs.dtu.dk/services/SignalP/)进行信号肽分析以及在线软件WOLF(https://wolfpsort.hgc.jp/)进行蛋白亚细胞定位的预测。
1.4 融合表达ORFV129基因真核重组表达载体的构建及鉴定
参照ORFV SY 17株ORFV129(登录号:MG 712417.1),运用Primier Primer 5设计引物。上游引物5′-AAGCTTATGGACGCCGCCGAGATGGAGGAGCTCGACATC-3′(划线部分为Hind Ⅲ酶切位点);下游引物5′-CTGCAGCTCGGGCGCGCGGCGGTCGCACGAGCGGCGC-3′(划线部分为Pst Ⅰ酶切位点),引物由上海生工生物工程有限公司合成。以1.3中质粒pMD19-ORFV129为模板,PCR扩增带酶切位点的129基因,反应体系及条件同1.3,1.5%琼脂糖凝胶电泳进行检测,切胶回收产物。将胶回收产物和载体pEGFP-N1分别使用Hind Ⅲ、Pst Ⅰ进行双酶切,酶切产物经PCR产物纯化试剂盒纯化后,T4 DNA连接酶16 ℃金属仪连接过夜,转化DH 5α感受态细胞,提取质粒后经PCR、双酶切鉴定为阳性的重组质粒送上海生工生物工程有限公司测序后保存备用,将鉴定正确的质粒命名为pEGFP-ORFV129。
1.5 pEGFP-ORFV129重组质粒转染新生山羊睾丸原代细胞
将生长良好的新生山羊睾丸原代细胞接种于6孔细胞培养板,待细胞汇合度达70%~90%时,按照LipoGeneTM 2000 转染试剂说明书,通过脂质体介导法将重组表达质粒pEGFP-ORFV129转染至生长良好的新生山羊睾丸原代细胞,以空载体pEGFP-N1为转染对照,并以正常生长的细胞为非转染空白对照。
1.6 检测pEGFP-ORFV129重组质粒在睾丸细胞的mRNA表达
在转染12~24 h后,将转染空载pEGFP-N1和融合载体pEGFP-ORFV129的细胞培养板置于荧光倒置显微镜下,观察pEGFP-ORFV129融合蛋白在睾丸细胞中的瞬时表达情况。对于检测转录动力学,在转染48 h后,用TRIzol法提取细胞总RNA,并用DNase Ⅰ消化残留于总RNA中的DNA,总RNA逆转录为cDNA,以cDNA为模板进行PCR扩增,扩增引物和条件同1.4,1.5%琼脂糖凝胶电泳进行检测。通过逆转录PCR(RT-PCR)检测ORFV129基因在细胞中ORFV复制期间的mRNA表达。
1.7 检测ORFV129蛋白在羊睾丸细胞中的蛋白表达
将生长良好的睾丸细胞接种于6孔细胞培养板上,设3组,每组2个重复,分别为空白对照组、pEGFP-N1转染组(4.0 μg)、pEGFP-ORFV129转染组(4.0 μg)。转染36 h后,将贴壁细胞刮取到1.5 mL的离心管,将广谱蛋白酶抑制剂:RAPI细胞裂解液按照1∶100的比例混匀的细胞裂解液加入装有细胞的离心管,在超声波破碎仪作用下破碎,离心,收集蛋白。随后,将提取的细胞总蛋白经SDS-PAGE电泳后,转入聚偏二氟乙烯(PVDF)膜上,经5%(m/V)脱脂奶中,4 ℃封闭过夜。随后分别加入鼠源GFP(1∶400)和兔源GADPH(1∶1 000),室温孵育2 h,10 min/次TBST(3次)的洗膜。利用HRP标记山羊抗鼠IgG(1∶400)和HRP标记的山羊抗兔IgG(1∶400),室温孵育2 h。TBST洗膜3次,随后使用ECL试剂显色,成像。
1.8 融合蛋白ORFV129的亚细胞定位
将睾丸细胞培养于含有盖玻片的6孔细胞培养板,待细胞汇合度达70%~90%时,转染重组表达质粒pEGFP-ORFV129,以空载体pEGFP-N1为对照,重组表达质粒组和空载体组每组设2个平行。转染48 h后吸尽培养液,预冷PBS洗3次,4%的多聚甲醛室温孵育15 min,预冷PBS洗3次;0.1%的Triton X-100室温孵育10 min,预冷PBS洗3次;加入10~50 μmol/L DAPI核酸染料,室温孵育15 min,预冷PBS洗2次;取出盖玻片,将其贴于含有封片剂的载玻片上,于激光共聚焦显微镜下观察ORFV129在细胞内的分布情况,分析其亚细胞定位。
2 结果与分析
2.1 ORFV129基因的PCR扩增、克隆鉴定及生物信息学分析
成功扩增ORFV129基因片段,经琼脂糖凝胶电泳检测可见与预期大小(1 551 bp)相符的目的片段(图1-A)。胶回收克隆转化后,进行菌落PCR,经琼脂糖凝胶电泳检测,与预期ORFV129基因的大小相符(图1-B)。测序结果显示,与NCBI上收录的同一病毒的不同毒株基因序列相似性均在99%以上。利用DNAStar软件分析发现,ORFV129基因共编码516个氨基酸。SMART分析显示,ORFV129基因共含有8个ANK基序,分别位于49-80 aa,85-117 aa,123-157 aa,161-192 aa,196-227 aa,231-262 aa,266-297 aa,302-331 aa(图2-A)。跨膜区域分析显示,ORFV129蛋白不存在跨膜区域,推测其可能作为胞内蛋白而起作用(图2-B)。通过WoLFPSORT在线比对软件预测ORFV129蛋白的亚细胞定位,结果显示蛋白质主要定位于细胞质(图2-C)。
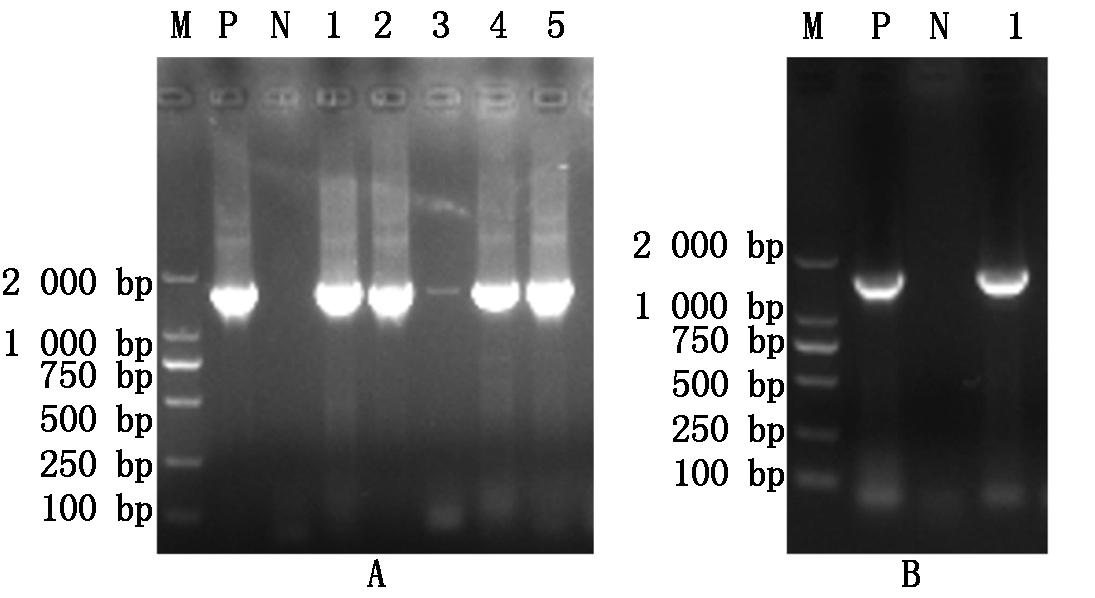
M. 相对分子质量标准(2 000 bp DNA ladder);P. 阳性对照;N. 阴性对照;1-5. ORFV129基因;A. ORFV129基因的PCR扩增;B. 重组质粒pMD-19T-ORFV129 的PCR扩增。
M.Relative molecular mass (2 000 bp DNA ladder);P. Positive product;N. Negative product;1-5. ORFV129 gene;A. ORFV129 gene amplified by PCR;B. Recombinant plasmid pMD-19T-ORFV129 amplified by PCR.
图1 ORFV129基因克隆及重组质粒鉴定
Fig.1 ORFV129 gene cloning and recombinant plasmid identification
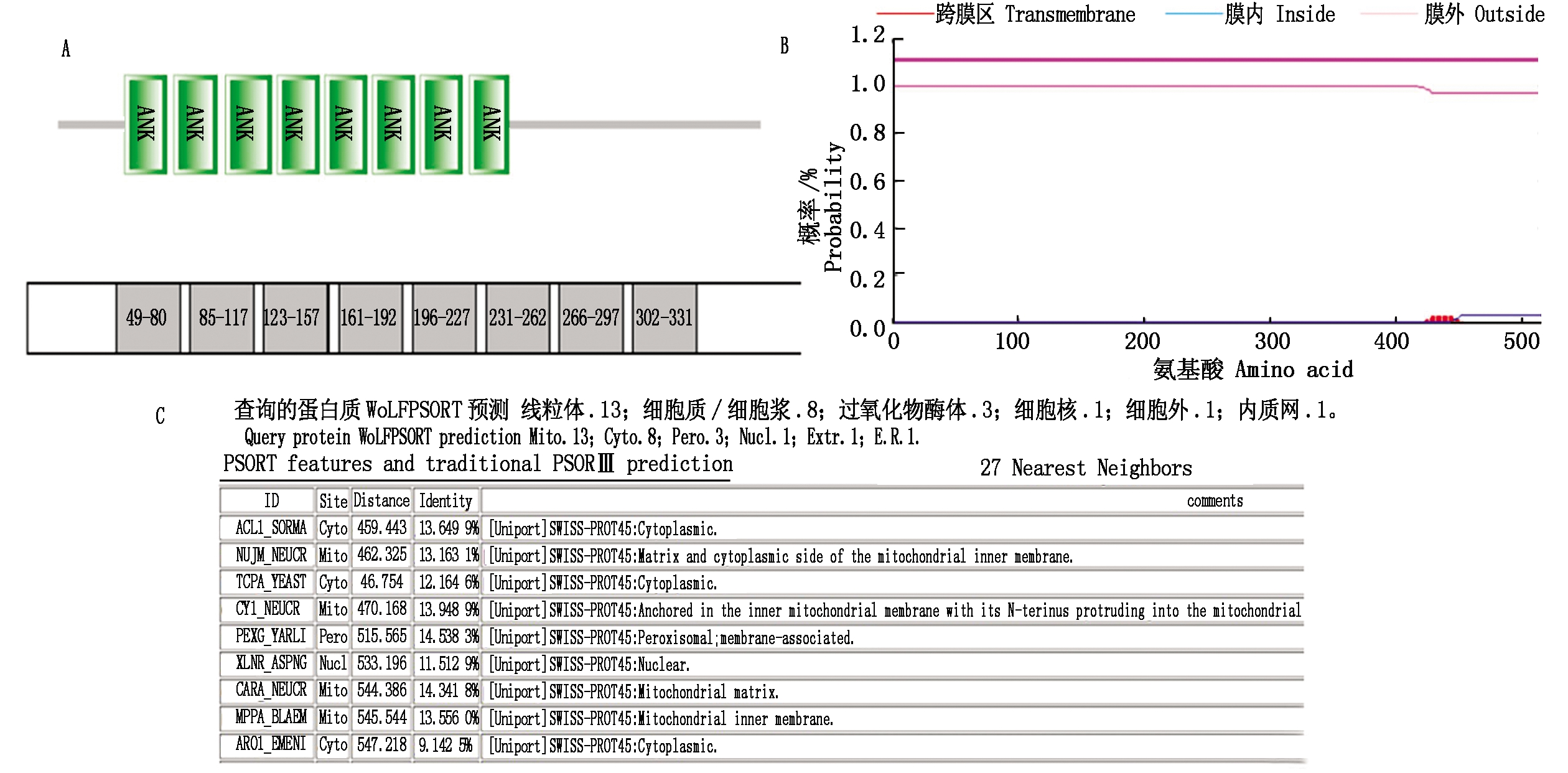
A. ORFV129蛋白的一级结构分析;B. ORFV129蛋白的跨膜区域分析;C. ORFV129蛋白的亚细胞定位分析。
A. Predicted primary-structure of ORFV129 protein;B. Predicted transmembrane region of ORFV129 protein;
C. Predicted subcellular localization of ORFV129 protein.
图2 ORFV129蛋白的生物信息学分析
Fig.2 Bioinformatics analysis of ORFV129 protein
2.2 融合表达ORFV129基因真核重组表达载体的构建及鉴定
使用带Hind Ⅲ、Pst Ⅰ酶切位点的引物进行PCR扩增,结果显示片段大小约为1 600 bp,与预期的片段大小(1 560 bp)相符合。随后,胶回收后定向克隆至pEGFP-N1构建融合重组质粒pEGFP-ORFV129,经双酶切鉴定表明构建的质粒正确(图3)。
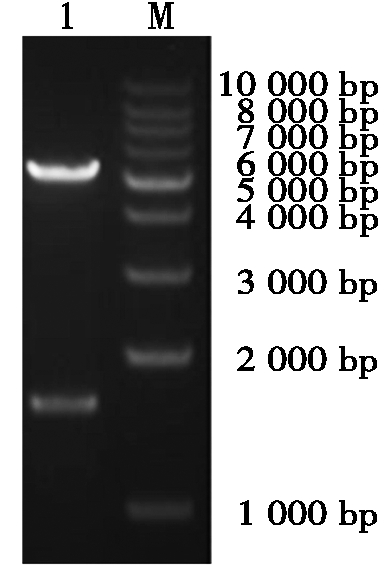
M. 相对分子质量标准(1 kb DNA ladder);
1. pEGFP-ORFV129的Hind Ⅲ、Pst Ⅰ双酶切鉴定。
M. Relative molecular mass (1 kb DNA ladder);
1. pEGFP-ORFV129 digested by Hind Ⅲ and Pst Ⅰ.
图3 pEGFP-ORFV129的酶切鉴定
Fig.3 Enzyme identification of pEGFP-ORFV129
2.3 pEGFP-ORFV129重组质粒在睾丸细胞ORFV复制期间的mRNA表达分析
转染12~24 h后,将融合载体pEGFP-ORFV129和空载体pEGFP-N1置于荧光倒置显微镜下,均能观察到绿色荧光(图4)。mRNA表达检测发现,细胞总RNA经RT-PCR扩增后,可见与预期ORFV129基因大小相似的目的条带(图5),表明ORFV129基因在细胞中ORFV复制期间发生了转录。
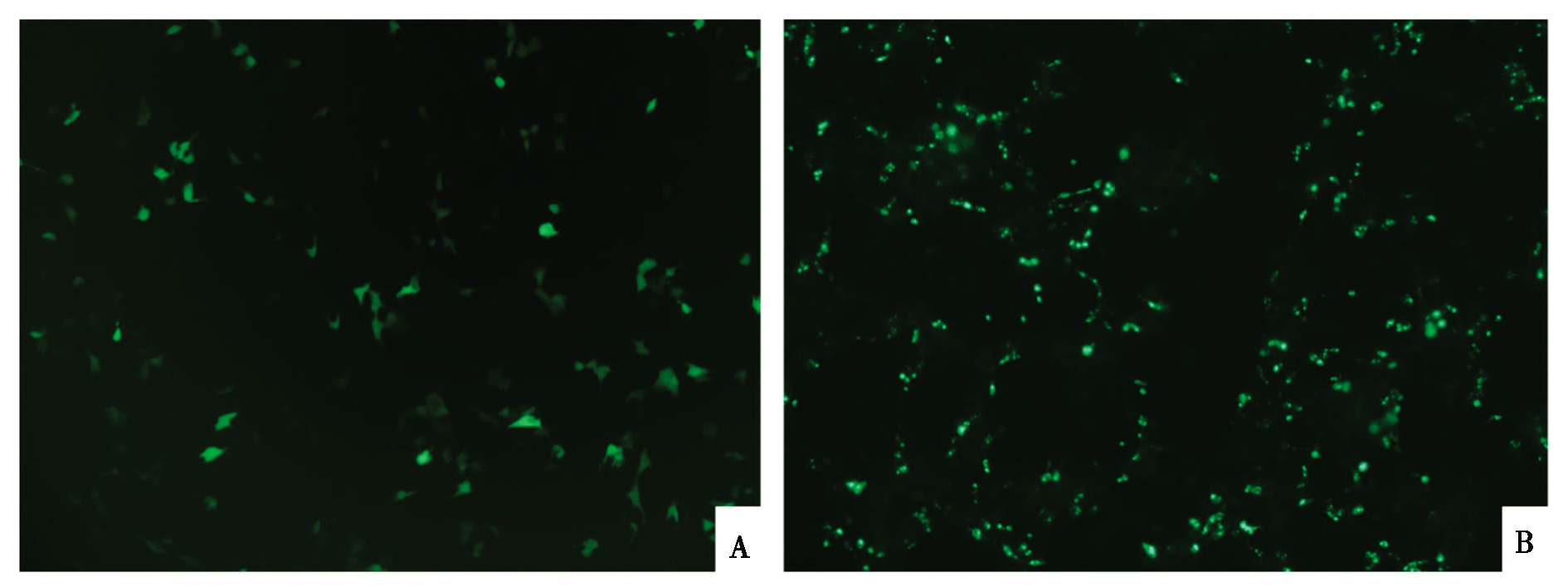
A. 转染pEGFP-N1空载的新生山羊睾丸原代细胞;B. 转染pEGFP-ORFV129的新生山羊睾丸原代细胞。
A. Testis cells of newborn goat transfected with pEGFP-N1;B. Testis cells of newborn goat transfected with pEGFP-ORFV129.
图4 pEGFP-ORFV129重组质粒转染新生山羊睾丸原代细胞
Fig.4 pEGFP-ORFV129 plasmid transfected into testis cells of newborn goat
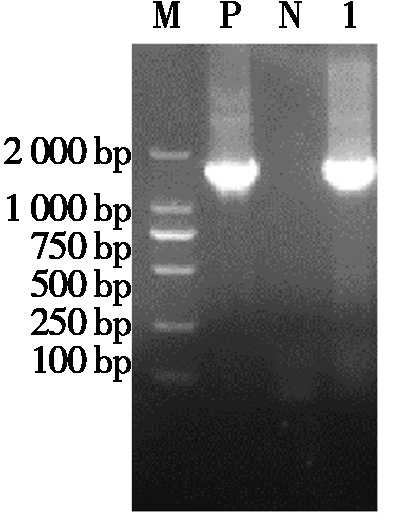
M.相对分子质量标准(2 000 bp DNA ladder);
P.阳性对照;N.无模板对照;1.ORFV129基因。
M.Relative molecular mass (2 000 bp DNA ladder);P.Positive
product;N.No template control;1.ORFV129 gene.
图5 pEGFP-ORFV129重组质粒的mRNA表达分析
Fig.5 mRNA expression analysis of pEGFP-ORFV129
2.4 融合蛋白ORFV129在羊睾丸细胞中的表达
将 pEGFP-N1和pEGFP-ORFV129重组质粒转染羊睾丸原代细胞36 h后,Western Blot 结果显示,空白对照组中没有GFP蛋白的表达,而转染了pEGFP-ORFV129重组质粒的山羊睾丸原代细胞中检测到了ORFV129-GFP蛋白(75 ku
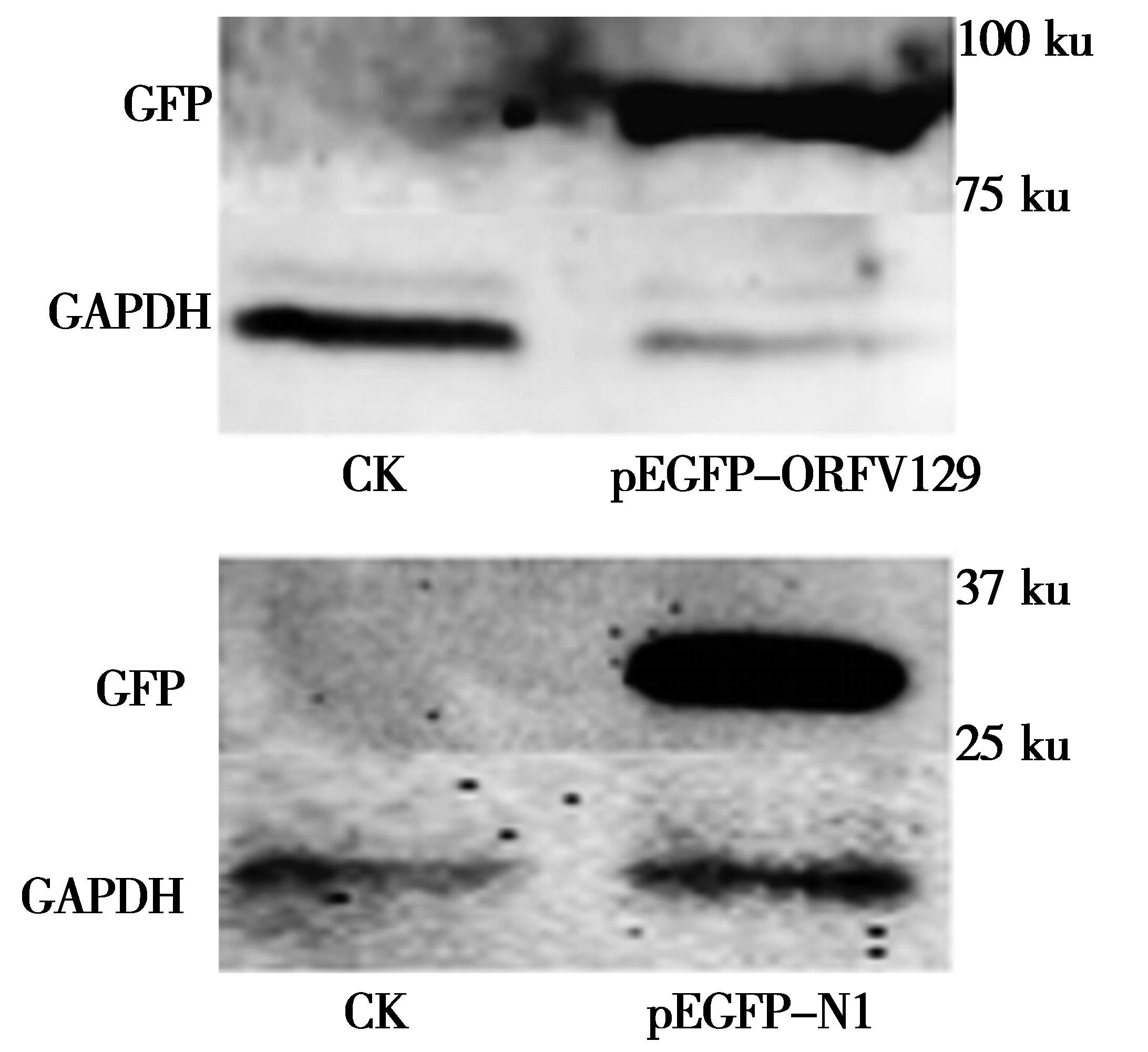
图6 ORF129蛋白表达的Western Blot鉴定
Fig.6 Western Blot identification of ORF129 protein
2.5 融合蛋白ORFV129-GFP的亚细胞定位
将 pEGFP-N1和pEGFP-ORFV129重组质粒转染羊睾丸原代细胞24 h后,经染料DAPI对细胞核进行蓝染,激光共聚焦显微镜下分析ORFV129蛋白在细胞内的定位情况。结果显示,在表达pEGFP-N1的细胞内,绿色荧光弥散性分布于整个细胞;而在表达pEGFP-ORFV129的睾丸细胞中,绿色荧光呈点状地分布于细胞质(图7)。

A. 转染pEGFP-N1的睾丸细胞明场观察;B.转染pEGFP-N1的睾丸细胞绿色荧光观察;C. DAPI 染色后细胞核;D. 图B和C的合并;E. 转染pEGFP-ORFV129的睾丸细胞明场观察;F. 转染pEGFP-ORFV129的睾丸细胞绿色荧光观察;G. DAPI 染色后细胞核;H. 图F和G的合并。
A. The common observation of transfected pEGFP-N1;B. The green fluorescent of transfected pEGFP-N1;C. Nuclei were stained using DAPI;D. The merged image of B and C;E. The common observation of transfected pEGFP-ORFV129;F. The green fluorescent of transfected pEGFP-ORFV129;G. Nuclei were stained using DAPI;H. The merged image of F and G.
图7 共聚焦显微镜下观察pEGFP-ORFV129表达蛋白在睾丸细胞中的定位
Fig.7 Results of pEGFP-ORFV129 expressed protein subcellular localization in testis cells by cofocal
3 讨论与结论
锚蛋白重复序列 (Ankyrinrepeat,ANKs )是普遍存在于生物体中的一种由33个氨基酸残基基序组成的蛋白质序列模体,由2个α螺旋和1个Loop区构成,在细胞信号转导、细胞骨架完整性、细胞周期调控、免疫应答、蛋白转运等过程中均具有重要作用[13-14]。ANKs不仅存在生物体,也存在于痘病毒编码的蛋白,而其他病毒几乎不编码锚蛋白。锚蛋白作为痘病毒编码的蛋白中最大的家族,其特征是具有ANKs的N末端和1个F-Box样的C末端结构域[15-17]。已有研究报道,痘病毒编码的锚蛋白主要功能是可通过多层次的方式来影响NF-κB信号通路。例如,天花病毒(Variola,VARV)编码的蛋白G1R,痘苗病毒(Vaccinia virus,VACV)编码的蛋白CP77,两者均可通过其N端的ANK重复基序,分别与NF-κB1/p105、NF-κB p65蛋白直接或间接结合抑制宿主NF-κB的活化[18-20]。2004年,Shisler等[21-22]发现VACV编码的蛋白K1L可通过竞争与转录因子NF-κB的结合,阻止IκBα的降解,从而抑制NF-κB的活化。同时,该团队也证实K1L蛋白能通过阻碍IKK的磷酸化从而抑制NF-κB的活化。因此,KIL蛋白准确的抑制机制仍然需进一步的研究。VACA A52和K7蛋白均能与肿瘤坏死因子受体相关分子6(TNF receptor-associated factor 6,TRAF6)相互作用,间接抑制NF-κB的激活[23]。羊口疮病毒编码的蛋白ORFV119,能通过与视网膜瘤蛋白相互作用抑制IKK的活化,从而抑制NF-κB信号通路的活化[24]。2014年,Lamb等[25]发现,黏液瘤病毒(Myxoma virus,MYXV)编码的锚蛋白M-T 5、M148R、M149R和M150R均能抑制NF-κB p65蛋白从细胞质迁移到细胞核,减少细胞核内NF-κB p65的聚集,抑制或者降低NF-κB信号通路的活性。同样VACV编码的锚蛋白K1L可通过对NF-κB信号途径不同位点的作用而调控宿主的天然抗病毒免疫应答[26]。
对于ORFV编码的5种锚蛋白,ORFV008蛋白可通过阻止IκBα蛋白磷酸化降解过程而抑制细胞NF-κB p65蛋白核移位,从而减弱细胞NF-κB信号通路的活化[27]。ORFV128蛋白在病毒感染早期表达且定位于宿主的细胞核,通过抑制IκBα蛋白磷酸化降解过程,阻止NF-κB p65蛋白核移位,进而抑制宿主NF-κB信号通路的活化[28]。可见,痘病毒内具有ANKs基序的锚蛋白,能通过对NF-κB信号途径的不同位点的作用而调控宿主的天然抗病毒免疫应答。羊口疮病毒ORFV129基因编码的锚蛋白具有典型的N末端多个ANK重复序列和1个F-Box的C末端结构域。2013年,白刚等[10]利用真核表达系统对ORFV129基因编码的蛋白进行表达并筛选出稳定表达ORFV129基因的细胞株,为研究ORFV129蛋白调控宿主天然免疫应答的作用机制奠定了基础。2016年,葛士坤等[29]对ORFV129基因序列及其编码蛋白进行生物信息学分析,该结果为深入研究ANK在ORFV致病过程中的作用提供了基础数据。对于ORFV编码的129锚蛋白功能目前不清楚,这也正是本研究选择ORFV129蛋白作为研究切入点的重要原因。本研究克隆了羊口疮病毒ORFV129基因全序列,对其进行生物信息学分析;进一步构建真核表达载体pEGFP-ORFV129,并将其转染新生山羊睾丸原代细胞,以空载体pEGFP-N1作为阴性对照,借助激光共聚焦显微镜观察了ORFV129在细胞中的定位;亚细胞定位发现ORFV129分布于细胞质中。ORFV编码的其他锚蛋白,如ORFV126蛋白表达后靶向定位于线粒体内,且ORFV126编码的AR8和AR9对锚蛋白定位于线粒体内起着关键性的作用[30]。ORFV128蛋白表达后靶向定位于细胞核中[17]。ORFV129蛋白是否类似于ORFV126,定位于细胞质内的线粒体,或者其蛋白功能是否与ORFV128的不一致,目前仍不清楚,需要进一步的研究。
本研究成功克隆得到羊口疮病毒ORFV129基因的全长,定位在新生山羊睾丸原代细胞细胞质中,这些结果为进一步研究ORFV129在诱导机体免疫应答中的作用奠定了基础。
[1] 陆承平. 兽医微生物学[M]. 北京:中国农业出版社,2007.
Lu C P. Veterinary microbiology[M]. Beijing:China Agricultural Press,2007.
[2] Bergqvist C,Kurban M,Abbas O. Orf virus infection[J]. Reviews in Medical Virology,2017,27(4):e1932. doi:10.1002/rmv.1932.
[3] Mondal B,Bera A K,Hosamani M,Tembhurne P A,Bandyopadhyay S K. Detection of Orf virus from an outbreak in goats and its genetic relation with other Parapoxviruses[J]. Veterinary Research Communications,2006,30(5):531-539. doi:10.1007/s11259-006-3270-z.
[4] Long M J,Wang Y Y,Chen D X,Wang Y,Wang R X,Gong D Y,He H J,Rock D L,Hao W B,Luo S H. Identification of host cellular proteins LAGE3 and IGFBP6 that interact with Orf virus protein ORFV024[J]. Gene,2018,661:60-67. doi:10.1016/j.gene.2018.03.089.
[5] Angella L C,Gustavo C,Nicole K,Isabel C,Esperanza P P,Antonio T. Erythema multiforme after Orf virus infection[J]. Pediatric Dermatology,2018,35(4):e237-e238. doi:10.1111/pde.13526.
[6] 陈慧芹.羊口疮病毒119蛋白诱导细胞凋亡分子机制的研究[D]. 广州:南方医科大学,2017.
Chen H Q. The molecular mechanisms of cell apoptosis induced by Orf virus (ORFV) 119 protein[D]. Guangzhou:Southern Medical University,2017.
[7] 闫丰超,邵佳,窦永喜. 羊口疮病毒分子生物学的研究进展[J]. 中国兽医科学,2013,43(1):103-109. doi:10.16656/j.issn.1673-4696.2013.01.009.
Yan F C,Shao J,Dou Y X. Progress in molecular biology of Orf virus[J]. Chinese Veterinary Science,2013,43(1):103-109.
[8] Sonnberg S,Seet B T,Pawson T,Fleming S B,Mercer A A. Poxvirus ankyrin repeat proteins are a unique class of F-box proteins that associate with cellular SCF1 ubiquitin ligase complexes[J]. Proceedings of the National Academy of Sciences of the United States of America,2008,105(31):10955-10960. doi:10.1073/pnas.0802042105.
[9] Shen Y W,Feng M,Wu X F. Bombyx mori nucleopolyhedrovirus ORF40 is essential for Budded virus production and Occlusion-derived virus envelopment[J]. Journal of General Virology,2018,99(6):837-850. doi:10.1099/jgv.0.001066.
[10] 白刚,贾怀杰,何小兵,王盈盈,何妍萍,吴润,景志忠. 羊口疮病毒ORF129基因重组质粒的构建及其在BHK-21细胞中的表达[J]. 甘肃农业大学学报,2013,48(5):8-13. doi:10.13432/j.cnki.jgsau.2013.05.002.
Bai G,Jia H J,He X B,Wang Y Y,He Y P,Wu R,Jing Z Z. Construction and expression of the recombinant plasmid of Orf virus ORF129 gene in BHK-21 cells[J]. Journal of Gansu Agricultural University,2013,48(5):8-13.
[11] 杜国玉,刘永杰,吴锦艳,王光祥,尚佑军,张勇.羊口疮病毒ORF129基因的原核表达、纯化及鉴定[J]. 中国畜牧兽医,2018,45(7):1798-1803. doi:10.16431/j.cnki.1671-7236.2018.07.009.
Du G Y,Liu Y J,Wu J Y,Wang G X,Shang Y J,Zhang Y. Prokaryotic expression,purification and identification of ORF129 gene of Orf virus[J]. China Animal Husbandry & Veterinary Medicine,2018,45(7):1798-1803.
[12] Lü Q Z,Guo K K,Xu H,Wang T,Zhang Y M. Identification of putative ORF5 protein of porcine Circovirus type 2 and functional analysis of GFP-fused ORF5 protein[J]. PLoS One,2015,10(7):e0134203. doi:10.1371/journal.pone.0127859.
[13] Lacek K,Bauer B,Bien′kowska-Szewczyk K,Rziha H J. Orf virus (ORFV) ANK-1 protein mitochondrial localization is mediated by ankyrin repeat motifs[J]. Virus Genes,2014,49(1):68-79.doi:10.1007/s11262-014-1069-5.
[14] 王少鑫,靳宝锋,崔立红. 锚蛋白重复序列通过NF-κB信号通路参与肠炎相关肿瘤的调控[J]. 世界华人消化系统,2015,23(2):189-195.
Wang S X,Jin B F,Cui L H. Gankyrin regulates colitis-associated cancer via NF-κB signaling pathway[J]. World Chinese Journal of Digestology,2015,23(2):189-195.
[15] Sonnberg S,Fleming S B,Mercer A A. Phylogenetic analysis of the large family of Poxvirus ankyrin-repeat proteins reveals orthologue groups within and across Chordopoxvirus genera[J]. Journal of General Virology,2011,92(11):2596-2607. doi:10.1099/vir.0.033654-0.
[16] Liu R K,Moss B. Vaccinia virus C9 ankyrin-repeat/F-box protein is a newly identified antagonist of the type Ⅰ interferon-induced antiviral state[J]. Journal of Virology,2018,92(9):e00053-e00018. doi:10.1128/JVI.00053-18.
[17] Odon V,Georgana I,Holley J,Morata J,Maluqyer M C. Novel class of viral ankyrin proteins targeting the host E3 ubiquitin ligase cullin-2[J]. Journal of Virology,2018,92(23):e01374-e01318.doi:10.1128/JVI.01374-18.
[18] Chang S J,Hsiao J C,Sonnberg S,Chiang C T,Yang M H,Tzou D L,Mercer A A,Chang W. Poxvirus host range protein CP77 contains an F-box-like domain that is necessary to suppress NF-κB activation by tumor necrosis factor alpha but is independent of its host range function[J]. Journal of Virology,2009,83(9):4140-4152. doi:10.1128/JVI.01835-08.
[19] Mohamed M R,Rahman M M,Lanchbury J S,Shattuck D,Neff C,Dufford M,van Buuren N,Fagan K,Barry M,Smith S,Damon I,Mcfadden G. Proteomic screening of Variola virus reveals a unique NF-κB inhibitor that is highly conserved among pathogenic orthopoxviruses[J]. Proceedings of the National Academy of Sciences of the United States of America,2009,106(22):9045-9050. doi:10.1073/pnas.0900452106.
[20] Mohamed M R,Rahman M M,Rice A,Moyer R W,Werden S J,Mcfadden G. Cowpox virus expresses a novel ankyrin repeat NF-κB inhibitor that controls inflammatory cell influx into virus-infected tissues and is critical for virus pathogenesis[J]. Journal of Virology,2009,83(18):9223-9236. doi:10.1128/JVI.00861-09.
[21] Shisler J L,Jin X L . The Vaccinia virus K1L gene product inhibits host NF-κB activation by preventing IκBα degradation[J]. Journal of Virology,2004,78(7):3553-3560. doi:10.1128/JVI.78.7.3553-3560.2004.
[22] Aravalli R N,Hu S X,Lokensgard J R. Inhibition of toll-like receptor signaling in primary murine microglia[J]. Journal of Neuroimmune Pharmacology,2008,3(1):5-11. doi:10.1007/s11481-007-9097-8.
[23] Pilato M D,Mejías-Pérez E,Sorzano C O S,Esteban M. Distinct roles of Vaccinia virus NF-κB inhibitor proteins A52,B15 and K7 in the immune response[J]. Journal of Virology,2017,91(13):e00575-e00617. doi:10.1128/JVI.00575-17.
[24] Nagendraprabhu P,Khatiwada S,Chaulagain S,Delhon G,Rock D L. A parapoxviral virion protein targets the retinoblastoma protein to inhibit NF-κB signaling[J]. PLoS Pathogens,2017,13(12):e1006779. doi:10.1371/journal.ppat.1006779.
[25] Lamb S A,Rahman M M,McFadden G. Recombinant Myxoma virus lacking all Poxvirus ankyrin-repeat proteins stimulates multiple cellular anti-viral pathways and exhibits a severe decrease in virulence[J]. Virology,2014,464-465(1):134-145. doi:10.1016/j.virol.2014.06.021.
[26] Meng X Z,Xiang Y. Vaccinia virus K1L protein supports viral replication in human and rabbit cells through a cell-type-specific set of its ankyrin repeat residues that are distinct from its binding site for ACAP2[J]. Virology,2006,353(1):220-233. doi:10.1016/j.virol.2006.05.032.
[27] 冯园,贾怀杰,何小兵,成温玉,陈国华,王春燕,何玉林,景志忠. 口疮病毒B4R锚蛋白抑制细胞NF-κB信号通路的研究[J]. 中国兽医科学,2016,46(11):1412-1417. doi:10.16656/j.issn.1673-4696.2016.11.012.
Feng Y,Jia H J,He X B,Cheng W Y,Chen G H,Wang C Y,He Y L,Jing Z Z. Poxvirus ankyrin protein B4R from Orf virus inhibits cell NF-κB signaling pathway[J]. Chinese Veterinary Science,2016,46(11):1412-1417.
[28] 安雪珂,贾怀杰,冯园,陈国华,何小兵,景志忠,王晓霞. 羊口疮病毒ORF128锚蛋白对HEK 293T 细胞NF-κB信号通路的调节作用及机制[J]. 细胞与分子免疫学杂志,2019,35(3):243-249. doi:10.13423/j.cnki.cjcmi.008759.
An X K,Jia H J,Feng Y,Chen G H,He X B,Jing Z Z,Wang X X. Regulating effect of ORF128 on NF-κB signaling pathway of HEK293T cells and its mechanism[J]. Chinese Journal of Cellular and Molecular Immunology,2019,35(3):243-249.
[29] 葛士坤,张凯照,刘健新,王晴楠,于梦,陶攀,宁章勇. 羊口疮病毒 GDZC 株锚蛋白基因的克隆及序列分析[J]. 中国畜牧兽医,2016,43(12):3085-3093. doi:10.16431/j.cnki.1671-7236.2016.12.001.
Ge S K,Zhang K Z,Liu J X,Wang Q N,Yu M,Tao P,Ning Z Y. Cloning and sequence analysis of ankyrin genes from Orf virus GDZC strain[J]. China Animal Husbandry & Veterinary Medicine,2016,43(12):3085-3093.
[30] Lacek K,Bauer B,Bieńkowska-Szewczyk K,Rziha H J. Orf virus (ORFV) ANK-1 protein mitochondrial localization is mediated by ankyrin repeat motifs[J]. Virus Genes,2014,49(1):68-79. doi:10.1007/s11262-014-1069-5.
