大豆(Glycine max (L.) Merr.)是重要的食用油和植物蛋白的来源,在农业和经济生产中占据举足轻重的地位,提高油分含量和蛋白质含量是大豆育种的主要目标。为了加快育种进程,分子标记技术定位QTL已广泛应用于大豆蛋白质和油分含量的遗传育种研究中,国内外关于大豆蛋白质和油分含量QTL定位的报道已有很多,控制蛋白质和油分性状的 QTL 基本分散在 20条染色体上[1-4]。1992 年,Diers等[5]利用A81356022×PI468916杂交得到F2∶3单株60个,定位了8个蛋白质和9个油分含量 QTL。 1996 年Lee等[6]利用 Young×PI 416937 杂交得到124个株系的F4 分离群体定位了 13 个蛋白质和 6 个油分含量 RFLP 位点,另外,利用 PI 97100×COKER 237 杂交得到111个株系F2的分离群体定位了6个蛋白质和5个油分含量RFLP位点。 2011年,葛振宇等[7]以栽培大豆TK780 与野生大豆 Hidaka4 杂交得到包含96 个株系的重组自交系(RILs),利用该群体定位了 2 个蛋白质含量 QTL和3 个油分含量 QTL。 2013年,Rossi等[3]利用来自加拿大和中国的2个品种OAC Millenium和黑农38杂交得到92个株系的F4∶7 RIL群体,定位了4个油分和4个蛋白含量QTL。2010年,Liang等[8]利用晋豆 23×Huibuzhi得到含有474个株系的F13 RIL群体,利用SSR标记分析共定位到5个蛋白质含量和6个油分含量QTL。2012年,Wang等[2]利用SD02-4-59×A02-381100得到含有87个株系的F5 RIL群体,利用SSR和SNP标记分析共定位到9个油分含量QTL。 2003年,Chung等[9]利用A3733×P1437088A得到包含75个株系的F5 RIL群体,利用SSR标记分析定位到一个蛋白和一个油分含量QTL,位于连锁群I上。2014年,Wang等[10]利用2个群体SD02-4-59 ×A02-381100和 SD02-911×SD00-1501,共定位到16个油分含量和11个蛋白含量QTL。2015年, Han等[11]利用中豆 27×九农 20得到RIL群体,定位到9个蛋白质含量和9个油分含量QTL。截至目前,Soybase 网站(www.soybase.org)统计了241个蛋白含量的 QTL 位点,320 个油分含量的 QTL 位点。
综上所述,国内外定位的蛋白质和油分含量QTL数量很多,但多是运用单一环境进行两亲本衍生的群体定位,同时蛋白质和油分含量表现为数量性状,其遗传基础复杂,这可能使遗传发生较高的偏分离,从而降低遗传作图精度。本研究应用前期构建的关联重组自交系群体,关联重组自交系群体中由于含有共同的亲本(东农L13),突破了以往单一亲本的限制,可以增加后代的多态性以及变异率,使后代在具有更高变异的同时还可以使2个遗传图谱间紧密联系,提高作图精度,拓展图谱丰富度,在研究大豆蛋白质和油分QTL时降低了定位误差,从而提高主效QTL的真实性。
1 材料和方法
1.1 关联重组自交系的构建
2008年用品质性状差异较大的大豆亲本东农L13(蛋白质含量45.50%,油分含量18.74%)、合农60(蛋白质含量38.47%,油分含量22.25%)和黑河36(蛋白质含量39.8%,油分含量19.28%),配制2个杂交组合,分别获得东农L13×合农60的F1和L13×黑河36的F1,将F1自交,自F2世代,在每一单株按单粒传法摘粒,在哈尔滨和海南岛连续自交5代,获得关联重组自交系群体。东农L13×黑河36衍生的重组自交系群体(RIL3613)包含134个家系,东农L13×合农60衍生的重组自交系群体(RIL6013)包含156个家系,用于本研究的蛋白质和油分含量QTL初步定位。
1.2 田间试验与性状调查
分别于2013,2014,2015年分别在黑龙江省克山和哈尔滨进行田间试验。将3个亲本和2个 RIL在田间种植,其中亲本种植40株,田间试验采取随机区组试验设计,3次重复,每小区3行,行长3 m,行距0.7 m,株距0.07 m。田间管理同一般大田栽培。成熟后收获种子应用Perton8620近红外品质分析仪测定蛋白质含量和油分含量[12]。
1.3 数据分析方法
对多年多点条件下蛋白质和油分含量数据进行描述性分析,并使用SAS 9.1软件对不同环境下的表型观测值进行联合方差分析,估算相应的方差分量,通过遗传方差、基因型与环境互作方差和误差方差进行遗传率的估计,遗传率估计的计算方法如下:
其中:h2为遗传率,![]() 为基因型方差,
为基因型方差,![]() 为基因型×环境互作方差, σ2为误差方差,r是重复数, e是环境数。
为基因型×环境互作方差, σ2为误差方差,r是重复数, e是环境数。
1.4 QTL定位分析
基于前期研究构建的连锁图谱[13],对各环境的蛋白质和油分含量分别采用完备复合区间作图法进行QTL定位,其统计分析过程通过IciMapping软件实现。背景标记选择的概率标准为0.001,染色体扫描步长为1 cM,LOD值得全基因组临界值由排列法(Permutation)确定,重复次数为1 000次,第一类错误的临界值为0.05,最小临界值为2.5。
2 结果与分析
2.1 表型数据分析
对3个环境下蛋白质和油分含量的分析(表1)可看出,在RIL3613和RIL6013的蛋白质含量和油分含量存在较大的变异,2个群体的蛋白质含量和油分含量在基因型间或不同环境条件下差异均达极显著水平,且环境和基因型之间存在极显著的互作效应。蛋白质和油分性状在两群体中均表现为连续分布(图1),表明蛋白质含量和油分含量这2个性状是受多基因控制的数量性状,在E1环境下,RIL6013群体油分含量高于RIL3613群体油分含量,RIL3613群体在E2环境下油分含量高于E1环境,两群体在E3环境下油分和蛋白质含量均表现较为集中。因此,可用该群体进行QTL定位研究,并且不同环境下定位结果可能发现不同的QTL。
表1 不同环境下2个群体蛋白质和油分含量的方差分析
Tab.1 Analysis of variance on protein and oil contents across multiple environments in RIL
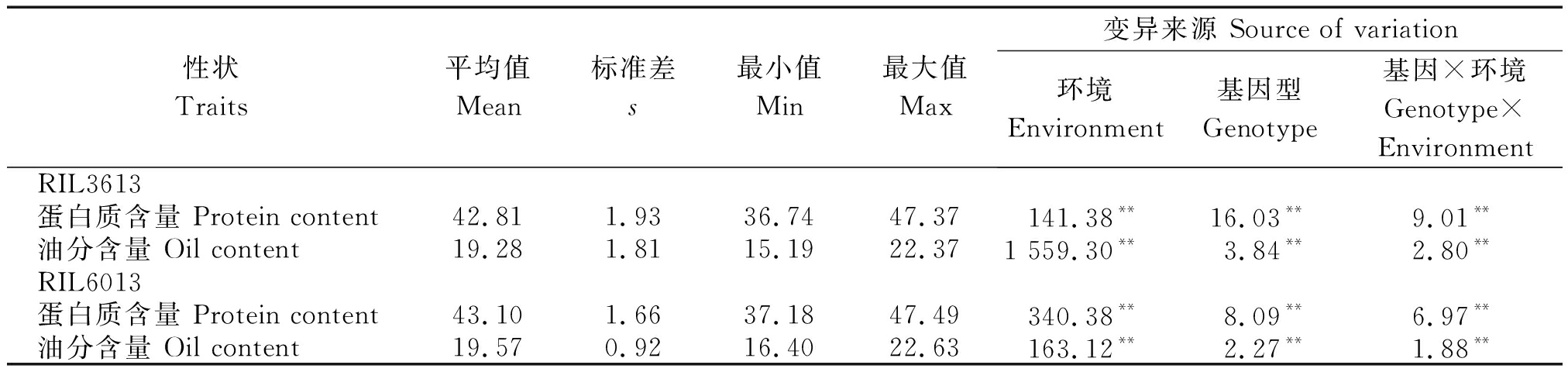
性状Traits平均值Mean标准差s最小值Min最大值Max变异来源 Source of variation环境Environment基因型Genotype基因×环境Genotype×Environment RIL3613蛋白质含量 Protein content42.811.9336.7447.37141.38**16.03**9.01**油分含量 Oil content19.281.8115.1922.371 559.30**3.84**2.80**RIL6013蛋白质含量 Protein content43.101.6637.1847.49340.38**8.09**6.97**油分含量 Oil content19.570.9216.4022.63163.12**2.27**1.88**
注:**.在P<0.01水平差异显著。
Note:**.Significant difference at P<0.01 level.

E1.克山2013;E2.哈尔滨2014;E3.哈尔滨2015。
E1.Keshan in 2013;E2.Harbin in 2014;E3.Harbin in 2015.
图1 不同环境下RIL3613(左)和RIL6013(右)蛋白质和油分含量分布
Fig.1 Distribution of protein and oil content in different environments in RIL3613 (left) and RIL6013 (right)
2.2 蛋白质含量的 QTL 定位
本试验在3个环境中检测到控制大豆蛋白质含量QTL共8个,定位在大豆的7个连锁群上(表2),在RIL3613群体中检测到6个蛋白质含量相关 QTL,分布于连锁群O、B1、F、D2、L、I、G上,LOD值分别为3.11,2.60,4.06,3.04,3.89和4.77,贡献率分别为11.53%,11.76%,7.93%,7.29%,12.11%和7.56%,其中qPro-O-1、qPro-B1-1、qPro-I-1的贡献率高于10%,加性效应分别为1.31,0.38,-0.91,0.82,1.12和0.60,其中,qPro-O-1、qPro-B1-1、qPro-L-1、qPro-I-1和qPro-G-1的正向加性效应来自于亲本东农L13,而qPro-D2-1的正向加性效应来自于亲本黑河36。在RIL6013群体中检测到2个蛋白质含量相关的QTL,分布于连锁群O、F上,LOD值分别为2.65和3.04,贡献率分别为6.53%和9.26%,加性效应分别为0.76和-0.71,其中,qPro-O-2的正向加性效应来自于亲本东农L13,而qPro-F-1的正向加性效应来自于亲本合农60。
表2 不同环境下蛋白质含量QTL定位结果
Tab.2 QTLs underlying protein content in soybean detected in different environments
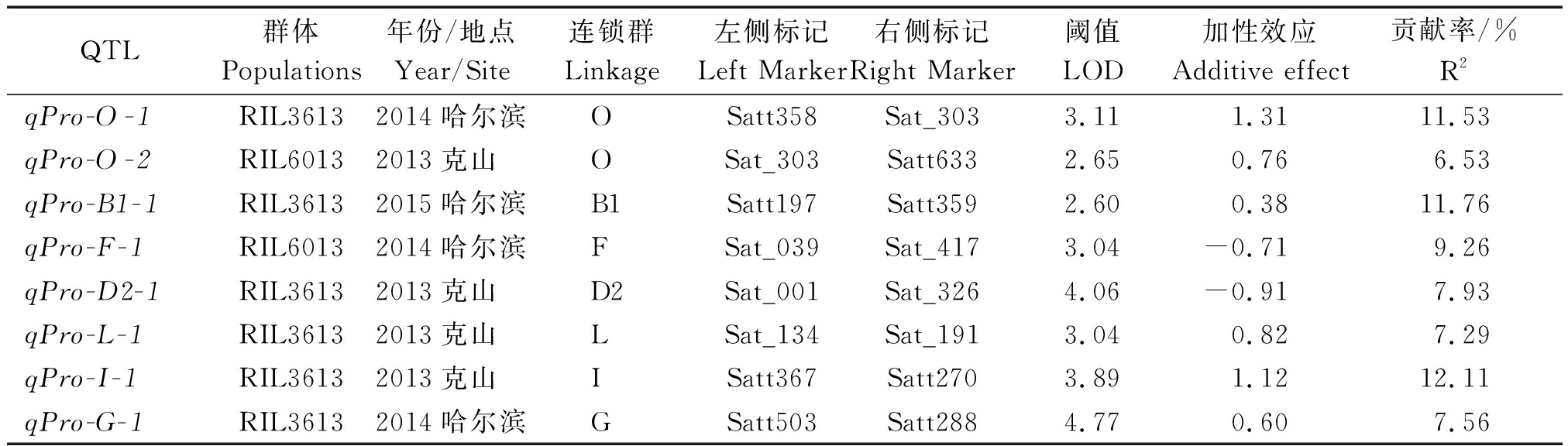
QTL群体Populations年份/地点Year/Site连锁群Linkage左侧标记Left Marker右侧标记Right Marker阈值LOD加性效应Additive effect贡献率/%R2 qPro-O-1RIL36132014哈尔滨OSatt358Sat_3033.111.3111.53qPro-O-2RIL60132013克山 OSat_303Satt6332.650.766.53qPro-B1-1RIL36132015哈尔滨B1Satt197Satt3592.600.3811.76qPro-F-1RIL60132014哈尔滨FSat_039Sat_4173.04-0.719.26qPro-D2-1RIL36132013克山 D2Sat_001Sat_3264.06-0.917.93qPro-L-1RIL36132013克山 LSat_134Sat_1913.040.827.29qPro-I-1RIL36132013克山 ISatt367Satt2703.891.1212.11qPro-G-1RIL36132014哈尔滨GSatt503Satt2884.770.607.56
2.3 油分含量的 QTL 定位
在3个环境中检测控制大豆油分含量QTL 5个(表3),定位在大豆的5个连锁群上,在RIL3613群体中检测到2个油分含量相关QTL,分布于连锁群D1a和A2上,LOD值分别为3.16和5.67,贡献率分别为9.95%和17.73%,加性效应分别为-0.23和-0.38,正向加性效应来自于亲本黑河36,其中,qOil-A2-1可以同时在2013年克山和2014年哈尔滨的2个环境中检测到为多环境通用QTL。在RIL6013群体中检测到3个油分含量相关QTL,分布于连锁群C1、H和D2上,LOD值分别为3.61,2.75和5.33,贡献率分别为11.84%,5.72%和13.62%,加性效应分别为-0.40,-0.27和-0.43,正向加性效应来自于亲本合农60。两群体共检测到贡献率高于10%的QTL有3个,分别为qOil-C1-1、qOil-A2-1、qOil-D2-1。
表3 不同环境下油分含量QTL定位结果
Tab.3 QTLs underlying oil content in soybean detected in different environments
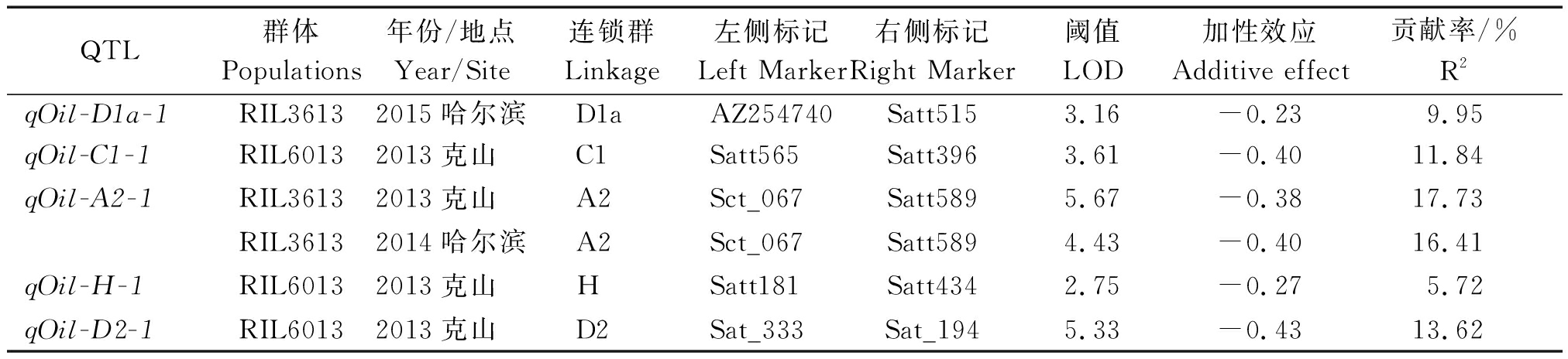
QTL群体Populations年份/地点Year/Site连锁群Linkage左侧标记Left Marker右侧标记Right Marker阈值LOD加性效应Additive effect贡献率/%R2 qOil-D1a-1RIL36132015哈尔滨D1aAZ254740Satt5153.16-0.239.95qOil-C1-1RIL60132013克山 C1Satt565Satt3963.61-0.4011.84qOil-A2-1RIL36132013克山 A2Sct_067Satt5895.67-0.3817.73RIL36132014哈尔滨A2Sct_067Satt5894.43-0.4016.41qOil-H-1RIL60132013克山 HSatt181Satt4342.75-0.275.72qOil-D2-1RIL60132013克山 D2Sat_333Sat_1945.33-0.4313.62
3 讨论
在以往的QTL定位群体中,多是应用单个双亲本衍生的群体,由于分子标记的多态性的原因,使构建的遗传图谱难以覆盖全基因组区域,因此,也不可能发现遗漏基因组区域上的QTL。为覆盖不同基因组区域,可采用同一个亲本与多个亲本杂交衍生的关联重组自交系群体进行图谱构建,通过不同亲本与共同亲本间分子标记的多态性以增加基因组的覆盖范围,进而提高QTL发掘能力。Buckler等[14]构建了包含25个玉米株系的RIL群体的关联重组自交系群体,对玉米开花期性状进行研究,在2个群体中共同发现了大量的微效QTL。李君[15]构建了包含2个小麦RIL的关联重组自交系群体进行QTL检测,其中有13对 QTL 在 2 个 RIL 群体中被同时定位或具有相同的连锁标记区间。本研究采用东农L13作为共同亲本与合农60和黑河36分别杂交,构建了包含2个RIL 群体的关联重组自交系群体进行蛋白质和油分含量的QTL定位。应用RIL3613定位了6个蛋白质含量QTL,而RIL6013定位了2个蛋白质含量QTL。这2个群体定位的QTL在不同的基因组区域,说明应用关联群体可提高QTL的发现能力。
通过比较含有不同遗传背景的QTL的基因组区域,结果发现,O连锁群上的蛋白质QTL(qPro-O-1)基因组所在的区域为5.44(Satt358)~20.93 cM(Sat_303)已经定位1个蛋白质含量QTL和1个油分含量QTL[4,16],O连锁群上的另一个蛋白质QTL(qPro-O-2)基因组所在的区域为20.93(Sat_303)~56.93 cM(Satt633),已经定位1个蛋白质含量QTL和3个油分含量QTL[12,17-19]。B1连锁群上的蛋白质QTL(qPro-B1-1) 基因组所在的区域46.38(Satt197)~102.55 cM(Satt359),已经定位3个蛋白质含量QTL和1个油分含量QTL[4,20-21]。F连锁群上的蛋白质QTL (qPro-F-1)基因组所在的区域为27.87(Sat_039)~135.94 cM(Sat_417),已经定位8个蛋白质含量QTL和5个油分含量QTL[2-3,12,16,21-25]。D2连锁群上的蛋白质QTL (qPro-D2-1)基因组所在的区域为92.12(Sat_001)~112.84 cM(Sat_326),已经定位3个蛋白质含量QTL和4个油分含量QTL[8,11-12,19-20]。L连锁群上的蛋白质QTL (qPro-L-1)基因组所在的区域为28.27(Sat_134)~32.00 cM(Sat_191),在标记Sat_134处与Mao等[12]定位的油分含量QTL有相同的标记,但是否为同一QTL 有待进一步验证。Ⅰ连锁群上的蛋白质QTL (qPro-I-1)基因组所在的区域为27.98(Satt367)~50.11 cM(Satt270),已经定位14个蛋白质含量QTL和12个油分含量QTL[4-5,9-11,22,26-30]。D1a连锁群上的控制油分含量的QTL (qOil-D1a-1)基因组所在的区域为8.23 (AZ254740)~55.68 cM(Satt515),已经定位1个蛋白质含量和1个油分含量QTL[11,22]。A2连锁群上的控制油分含量的QTL (qOil-A2-1)基因组所在的区域为14.99(Sct_067)~33.95 cM(Satt589),与Qi等[4]在A2连锁群上的油分含量QTL在大豆公共图谱上相距很近,可以认为是同一个QTL。D2连锁群上的控制油分含量的QTL (qOil-D2-1)基因组所在的区域为5.38(Sat_333)~86.69 cM(Sat_194),已经定位3个蛋白质含量QTL和11个油分含量QTL[4,6,10-12,15,23]。以上 QTL 与前人的定位结果相一致,是多个遗传背景通用的QTL。但蛋白质QTL(qPro-G-1)和油分含量QTL (qOil-C1-1、qOil-H-1)在公共图谱上并未发现与之重叠区域,是RIL3613和RIL6013的特异性QTL位点,可为以后的分子育种奠定基础。
国内外关于大豆蛋白质含量、油分含量QTL的研究日益增多,研究方法也有所不同,但在以往研究多是利用单环境的数据进行分析,由于蛋白质和油分含量多表现为数量性状,易受环境条件的影响,这将导致一些QTL由于LOD值较高而未被检测到或是出现假阳性。Wang等[10]利用2个重组自交系群体,共定位到12个产量性状,16个油分含量和11个蛋白质含量多环境通用QTL。侯萌等[31]构建了包含147个株系的重组自交系群体,进行油分和蛋白质含量的QTL分析,6个环境下共定位到9个蛋白质含量QTL和11个油分含量QTL,其中有2个油分含量QTL为多环境通用QTL。Ning等[13]利用包含204个株系的大豆4项重组自交系群体,共检测到29个蛋白质含量QTL和39个油分含量QTL,分别有5个蛋白质和10个油分QTL为多环境通用QTL。应用多环境的数据分析,可以同时发现环境特异QTL和多环境通用QTL,较准确估计 QTL 的效应,更有利于搜索稳定的QTL。本研究共检测到1个油分含量QTL为多环境通用QTL,12个QTL为单个环境特异QTL。
在以往进行QTL的研究中,多研究主效QTL,即贡献率值高于10%的QTL。葛振宇等[7]利用包含96个株系的RIL群体,进行油分和蛋白质含量的QTL分析,定位到2个蛋白质含量和3个油分含量QTL,且贡献率均高于10%。侯萌等[31]构建了包含147个株系的重组自交系群体,进行油分和蛋白质含量的QTL分析,共定位到9个蛋白质含量QTL和11个油分含量QTL,其中有6个蛋白质含量和7个油分含量QTL 贡献率高于10%。本研究有3个蛋白质QTL和3个油分含量QTL的贡献率高于10%,这些QTL具有重要的潜在价值,可以对其进行精细定位,为相应基因的图位克隆奠定基础。
本试验定位出了8个蛋白质含量QTL与5个油分含量QTL,有1个蛋白质QTL和2个油分含量QTL为本研究群体特异性QTL;有1个油分含量QTL为多环境通用QTL,其他12个QTL为单个环境特异QTL;在本研究中,有3个蛋白质QTL和3个油分含量QTL为主效QTL,可进行分子设计育种研究。
[1] Palomeque L, Liu L J, Li W B, Hedges B, Cober E R, Rajcan I. QTL in mega-environments: Ⅱ. agronomic trait QTL co-localized with seed yield QTL detected in a population derived from a cross of high-yielding adapted×high-yielding exotic soybean lines[J].Theoretical and Applied Genetics,2009, 119(3): 429-436.doi:10.1007/s00122-009-1048-8.
[2] Wang X Z, Jiang G L, Green M,Scott R A, Hyten D L,Cregan P B. Quantitative trait locus analysis of saturated fatty acids in a population of recombinant inbred lines of soybean[J].Molecular Breeding,2012,30(2):1163-1179.doi:10.1007/s11032-012-9704-0.
[3] Rossi M E, Orf J H, Liu L J, Dong Z M, Rajcan I. Genetic basis of soybean adaptation to North American vs. Asian mega-environments in two independent populations from Canadian×Chinese crosses[J]. Theoretical and Applied Genetics, 2013,126(7):1809-1823.doi:10.1007/s00122-013-2094-9.
[4] Qi Z M, Wu Q, Han X, Sun Y N, Du X Y, Liu C Y, Jiang H W, Hu G H, Chen Q S. Soybean oil content QTL mapping and integrating with meta-analysis method for mining genes[J]. Euphytica, 2011, 179:499-514.doi:10.1007/s10681-011-0386-1.
[5] Diers B W, Keim P, Fehr W R, Shoemaker R C. RFLP analysis of soybean seed protein and oil content[J].Theoretical and Applied Genetics, 1992, 83(5):608-612. doi:10.1007/BF00226905.
[6] Lee S H, Bailey M A R, Mian M A R, Carter T E, Shipe E R, Ashley D A, Parrott W A, Hussey R S, Boerma H R. RFLP loci associated with soybean seed protein and oil content across populations and locations[J]. Theoretical and Applied Genetics, 1996, 93(5-6):649-657.doi:10.1007/bf00224058.
[7] 葛振宇, 刘晓冰, 刘宝辉, 阿部纯, 马凤鸣, 孔凡江. 大豆种子蛋白质和油份性状的QTL定位[J].大豆科学, 2011, 30(6): 901-905.
Ge Z Y, Liu X B, Liu B H, Abe J, Ma F M, Kong F J. QTL mapping of protein and oil content in soybean[J]. Soybean Science, 2011, 30(6): 901-905.
[8] Liang H Z, Yu Y L, Wang S F, Lian Y, Wang T F, Wei Y L, Gong P T, Liu X Y, Fang X J, Zhang M C. QTL mapping of isoflavone,oil and protein contents in soybean (Glycine max L.Merr.)[J]. Agricultural Sciences in China, 2010, 9(8):1108-1116.doi:10.1016/S1671-2927(09)60197-8.
[9] Chung J, Babka H L, Graef G L, Staswick P E, Lee D J, Cregan P B, Shoemarker R C, Specht J E. The seed protein oil and yield QTL on soybean linkage group I[J]. Crop Science, 2003, 43(3):1053-1067.doi:10.2135/cropsci2003.1053.
[10] Wang X Z, Jiang G L, Green M, Scott R A, Song Q J, Hyten D L, Cregan P B. Identification and validation of quantitative trait loci for seed yield, oil and protein contents in two recombinant inbred line populations of soybean[J]. Mol Genet Genomics,2014, 289(5):935-949. doi:10.1007/s00438-014-0865-x.
[11] Han Y P, Teng W L, Wang Y, Zhao X, Wu L, Li D M, Li W B. Unconditional and conditional QTL underlying the genetic interrelationships between soybean seed isoflavone, and protein or oil contents[J]. Plant Breeding, 2015, 134(3):300-309.doi:10.1111/pbr.12259.
[12] Mao T T, Jiang Z F, Han Y P, Teng W L, Zhao X, Li W B. Identification of quantitative trait loci underlying seed protein and oil contents of soybean across multi-genetic backgrounds and environments[J].Plant Breeding, 2013, 132(6):630-641.doi:10.1111/pbr.12091.
[13] Ning H L, Yuan J Q, Dong Q Z, Li W B, Xue H, Wang Y S, Tian Y, Li W B. Identification of QTLs related to the vertical distribution and seed-set of pod number in soybean (Glycine max (L.) Merr.)[J]. PLoS One,2018, 13(4):e0195830.doi:10.1371/journal.pone.0195830.
[14] Buckler E S, Holland J B, Bradbury P J, Acharya C B,Brown P J, Browne C, Ersoz E, Flint-Garcia S, Garcia A, Glaubitz J C,Garcia A, Glaubitz J C, Goodman M M, Harjes C, Guill K, Kroon D E, Larsson S, Lepak N K, Li H H, Mitchell S E, Pressoir G, Peiffer J A, Rosas M O, Rocheford T R, Romay M C, Romero S, Salvo S, Villeda H S, da Silva H S, Sun Q, Tian F, Upadyayula N, Ware D, Yates H, Yu J M, Zhang Z W, Kresovich S, McMullen M D. The genetic architecture of maize flowering time[J].Science, 2009, 325(5941):714-718.doi:10.1126/science.1174276.
[15] 李君. 小麦关联重组自交系群体遗传图谱的构建及主要品质性状的QTL定位[D].泰安:山东农业大学, 2011.doi:10.7666/d.d143624.
Li J. Construction of genetic linkage maps and QTL mapping for quality related traits using two associated RIL populations of wheat[D].Taian:Shandong Agricultural University,2011.
[16] Kabelka E A, Diers B W, Fehr W R, LeRoy A R, Baianu I C, You T, Neece D J, Nelson R L. Putative alleles for increased yield from soybean plant introductions[J]. Crop Science, 2004, 44(3):784-791.doi:10.2135/cropsci2004.0784.
[17] Jun T H, Van K, Kim M Y, Lee S H, Walker D R.Association analysis using SSR markers to find QTL for seed protein content in soybean[J]. Euphytica,2008,162: 179-191.doi:10.1007/s10681-007-9491-6.
[18] Panthee D R, Pantalone V R, West D R, Saxton A M, Sams C E.Quantitative trait loci for seed protein and oil concentration, and seed size in soybean[J].Crop Science,2005,45(5): 2015-2022. doi:10.2135/cropsci2004.0720.
[19] Tajuddin T, Watanabe S, Masuda R, Harada K, Kawano S. Application of near infrared transmittance spectroscopy to the estimation of protein and lipid contents in single seeds of soybean recombinant inbred lines for quantitative trait loci analysis[J]. Journal of Near Infrared Spectroscopy, 2002, 10(1):315.doi:10.1255/jnirs.348.
[20] Gai J Y, Wang Y J, Wu X L, Chen S Y. A comparative study on segregation analysis and QTL mapping of quantitative traits in plants-with a case in soybean[J]. Frontiers of Agriculture in China, 2007, 1(1):1-7.doi:10.1007/s11703-007-0001-3.
[21] Reinprecht Y, Poysa V W, Yu K F, Rajcan I, Ablett G R, Pauls K P. Seed and agronomic QTL in low linolenic acid, lipoxygenase-free soybean (Glycine max (L.) Merrill) germplasm[J]. Genome, 2006, 49(12):1510-1527.doi:10.1139/g06-112.
[22] Brummer E C, Graef G L, Orf J H, Wilcox J R, Shoemaker R C. Mapping QTL for seed protein and oil content in eight soybean populations[J]. Crop Science, 1997, 37(2):370-378.doi:10.2135/cropsci1997.0011183x003700020011x.
[23] Hyten D L, Pantalone V R, Sams C E, Saxton A M, Ellis D L, Stefaniak T R, Schmidt M E. Seed quality QTL in a prominent soybean population[J]. Theoretical and Applied Genetics, 2004, 109(3):552-561.doi:10.1007/s00122-004-1661-5.
[24] Qiu B X, Arelli P R, Sleper D A. RFLP markers associated with soybean cyst nematode resistance and seed composition in a ′Peking′×′Essex′ population[J]. Theoretical and Applied Genetics,1999,98:356-364.doi:10.1007/s001220051080.
[25] Eskandari M, Cober E R, Rajcan I. Genetic control of soybean seed oil: I. QTL and genes associated with seed oil concentration in RIL populations derived from crossing moderately high-oil parents[J]. Theoretical and Applied Genetics, 2013, 126(2):483-495.doi:10.1007/s00122-012-1995-3.
[26] Specht J E, Chase K, Macrander M, Graef G L, Chung J, Markwell J P, Germann M, Orf J H, Lark K G. Soybean response to water: a QTL analysis of drought tolerance[J]. Crop Science,2001, 41(2):493-509.doi:10.2135/cropsci2001.412493x.
[27] Warrington C V, Abdel-Haleem H, Hyten D L, Cregan P B, Orf J H, Killam A S, Bajjalieh N, Li Z, Boerma H R. QTL for seed protein and amino acids in the benning×danbaekkong soybean population[J]. Theoretical and Applied Genetics, 2015,128(5): 839-850.doi:10.1007/s00122-015-2474-4.
[28] Sebolt A M, Shoemaker R C, Diers B W. Analysis of a quantitative trait locus allele from wild soybean that increases seed protein concentration in soybean[J]. Crop Science,2000,40(5):1438-1444.doi:10.2135/cropsci2000.4051438x.
[29] Lu W G, Wen Z X, Li H C, Yuan D H, Li J Y, Zhang H, Huang Z W, Cui S Y, Du W J. Identification of the quantitative trait loci (QTL) underlying water soluble protein content in soybean[J].Theoretical and Applied Genetics,2013,126:425-433.doi:10.1007/s00122-012-1990-8.
[30] Veeranki S,Choubey D. Interferon-inducible p200-family protein IFI16, an innate immune sensor for cytosolic and nuclear double-stranded DNA: regulation of subcellular localization[J]. Molecular Immunology,2012,49(4):567-571.doi:10.1016/j.molimm.2011.11.004.
[31] 侯萌, 齐照明, 韩雪, 辛大伟, 蒋洪蔚, 刘春燕, 吴琼, 隋丽丽, 胡国华, 陈庆山. 大豆蛋白质和油分含量 QTL 定位及互作分析[J]. 中国农业科学, 2014, 47(13): 2680-2689.doi:10.3864/j.issn.0578-1752.2014.13.020.
Hou M, Qi Z M, Han X, Xin D W, Jiang H W, Liu C Y, Wu Q, Sui L L, Hu G H, Chen Q S. QTL Mapping and interaction analysis of seed protein content and oil content in soybean[J]. Scientia Agricultura Sinica, 2014, 47(13): 2680-2689.
