玉米目前是全球第一大作物,也已跃升为我国第一大作物,经济地位和战略地位日益提升,成为政府保证粮食持续增产的最大战略需求。随着我国种业的快速发展,玉米审定品种数量呈逐年上升趋势,目前我国已通过国家或省级审定的品种近1.3万个(截止到2019年),尤其是近几年随着深化种业“放、管、服”,激发育种人员快速释放科技创新积极性,新品种审定进入了快车道,2016-2019年审定玉米品种近5 700个,国家及各省区试、联合体、绿色通道参试品种数量每年接近8 000个。随着玉米品种数量逐年增多,少数骨干亲本的集中应用,品种间的遗传差异越来越小,难以进行有效甄别,如何从源头控制品种多、乱、杂,并有效地管理和甄别数量庞大的玉米品种成为管理的难题[1]。
我国从2002年起开始开展DNA指纹在国家玉米区域试验样品真实性检测中的研究与应用[2-3],2005年辽宁、吉林、河北、河南、山东、四川等重要玉米种植地区开始采用DNA检测参试组合真实性,2012年基本覆盖到全国各省、自治区。2016年随着农作物品种审定制度的改革和参试渠道的扩宽,国家区试和国家联合体、省区试和省联合、绿色通道、自主试验等参试渠道参试的组合数量出现了爆发式的增长,2002年至今已累计检测42 826个玉米参试组合样品的DNA真实性。水稻[4]、小麦[5-6]、大豆[7-8]、油菜[9-10]、棉花[11]、马铃薯[12]、花生[13]、芝麻[14]等作物区域试验也在采用DNA指纹技术对参试组合进行检测,并作为品种审定的依据和参考。
分子标记在区域试验参试组合检测中发挥以下作用:一是构建参试组合DNA指纹数据库,分析参试组合的遗传多样性以及品种选育趋势;二是检测同一参试组合不同年份、不同组别是否存在同名更换;三是对每一份参试组合进行特异性检测,与所有已知品种及同时参试的组合进行比对,从分子水平检测是否具备特异性;四是对参试组合进行一致性、稳定性、纯度测试。DNA指纹在我国品种区域试验中的深入应用,为我国品种管理提供了有效的技术支撑,防止参试组合更换亲本,以及与已知品种相近的问题,从源头解决品种的多、乱、杂的问题。本研究对2014-2019年国家玉米区域试验样品进行DNA指纹的检测和遗传多样性分析,全面了解我国近几年国家玉米参试组合的多样性、选育水平、发展趋势和存在的问题。
1 材料和方法
1.1 试验材料
2014-2019年国家区试参试组合2 949份,其中2014年294份,2015年430份,2016年524份,2017年576份,2018年538份,2019年587份。包括青贮组(QZ)、西南组(XN)、东南组(DN)、爆裂组(BL)、极早熟组(JZS)、东华北组(DHB)、西北组(XB)、黄淮海组(HHH)、鲜食玉米组(包括甜玉米和糯玉米)(XS)、机收组(JS)、热带和亚热带组(RD)。样品信息详见表1,其中鲜食玉米组参试组合包括糯玉米和甜玉米,虽然甜玉米与糯玉米的遗传背景不同,但两者与其他组别遗传背景差异较大,合并为鲜食组进行分析。
表1 样品信息表
Tab.1 Sample information sheet
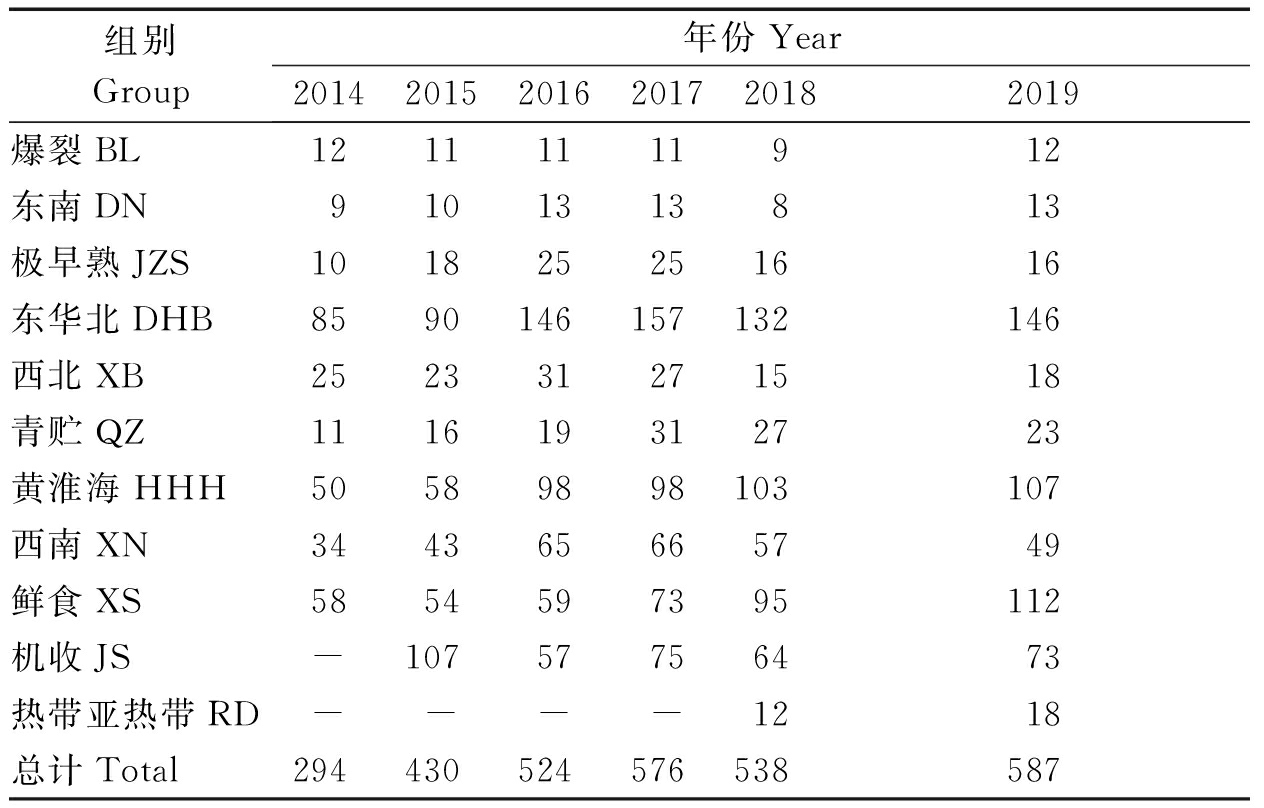
组别Group年份Year201420152016201720182019爆裂BL12111111912东南DN9101313813极早熟JZS101825251616东华北DHB8590146157132146西北XB252331271518青贮QZ111619312723黄淮海HHH50589898103107西南XN344365665749鲜食XS5854597395112机收JS-10757756473热带亚热带RD----1218总计Total294430524576538587
1.2 试验方法
1.2.1 基因组DNA提取 每份供试样品均随机抽取50粒种子形成混合样品,采用CTAB法提取DNA。
1.2.2 PCR扩增 模板DNA 3 μL,0.25 μmol/L引物,1×PCR Buffer,0.15 μmol/L dNTP,2.5 μmol/L MgCl2,1 U Taq酶,反应总体积为20 μL。每对引物中的一条5′端用荧光染料标记,选用PET、NED、VIC、FAM 4种荧光染料(Applied Biosystems, USA公司合成)。PCR反应程序:94 ℃预变性5 min,1个循环;94 ℃变性40 s,60 ℃退火35 s,72 ℃延伸45 s,共35个循环;72 ℃延伸10 min,4 ℃保存。
1.2.3 检测扩增产物 毛细管电泳:PCR产物在毛细管荧光电泳系统ABI 3730XL DNA分析仪 (Applied Biosystems,USA)上进行电泳,在96孔电泳板的单个孔中分别加入1.5 μL PCR产物、9 μL甲酰胺、0.1 μL分子量内标(GeneScanTM-500 LIZ)。95 ℃变性5 min,4 ℃保存10 min,1 000 r/min离心1 min后,于AB 3730XL DNA分析仪上进行电泳。预电泳时间为15 kV,2 min;电泳时间为15 kV,20 min。用Date Collection V1.0软件收集原始数据。
1.2.4 真实性检测 参试组合真实性检测参考NY/T 1432-2014《玉米品种鉴定规程 SSR标记法》。参试组合近似品种筛查对照样品库包括6 039份农业部征集审定品种标准样品、1 861份农业部品种权保护标准样品、同年度及上一年度的参试样品。
1.2.5 遗传分析 采用PowerMarker V3.25软件对等位变异数、基因多样性、杂合率和多样性信息(Polymorphism information content, PIC)进行分析,基于UPGMA(Unweighted pair-group method with arithmetic means)方法的Nei′s (1972)遗传距离进行不同区组的聚类分析。
2 结果与分析
2.1 不同年份和不同区组多样性的比较
不同年份参试组合多样性分析结果表明(表2),2014-2019年国家区试参试组合年际间的PIC、杂合率和遗传多样性基本一致,由于2014和2015年样品数量较少,2014和2015年参试组合的平均基因型数略低于2016-2019年。不同组别参试组合多样性分析结果表明(表3),爆裂组和西北组的PIC和遗传多样性较低,尤其是爆裂组,各指标都低于其他组别;鲜食、青贮和西南3个组别的PIC和遗传多样性较高。
表2 2014-2019年不同年份参试组合多样性比较
Tab.2 Genetic diversity comparison of national regional trail in 2014-2019
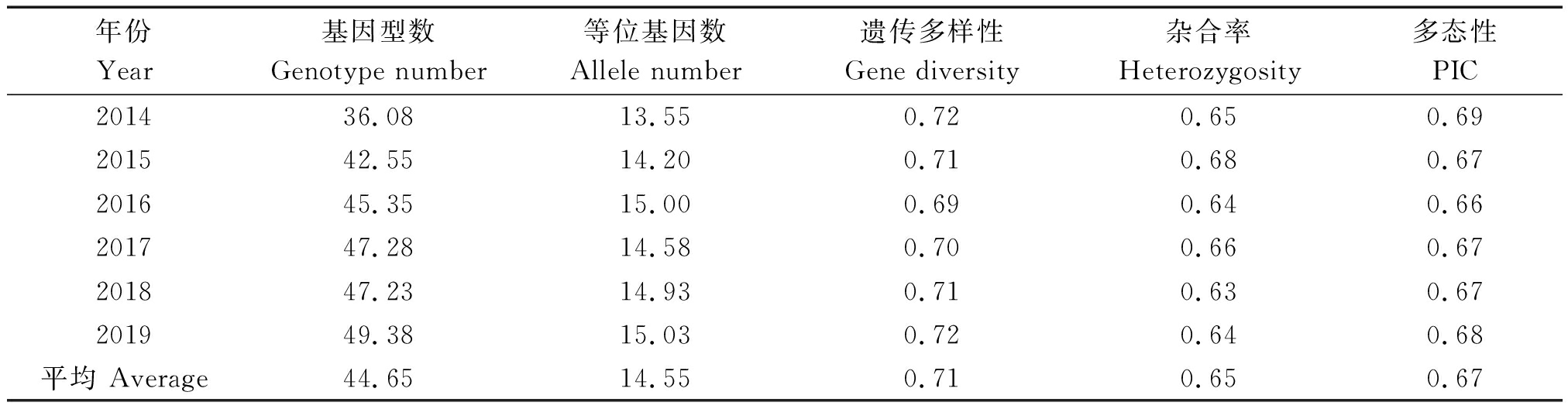
年份Year基因型数Genotype number等位基因数Allele number遗传多样性Gene diversity杂合率Heterozygosity多态性PIC201436.08 13.550.72 0.65 0.69 201542.55 14.20 0.71 0.68 0.67 201645.35 15.00 0.69 0.64 0.66 201747.28 14.58 0.70 0.66 0.67 201847.23 14.93 0.71 0.63 0.67 201949.38 15.03 0.72 0.64 0.68 平均 Average44.65 14.55 0.71 0.65 0.67
表3 不同区组参试组合多样性比较
Tab.3 Genetic diversity comparison among different regional trail group
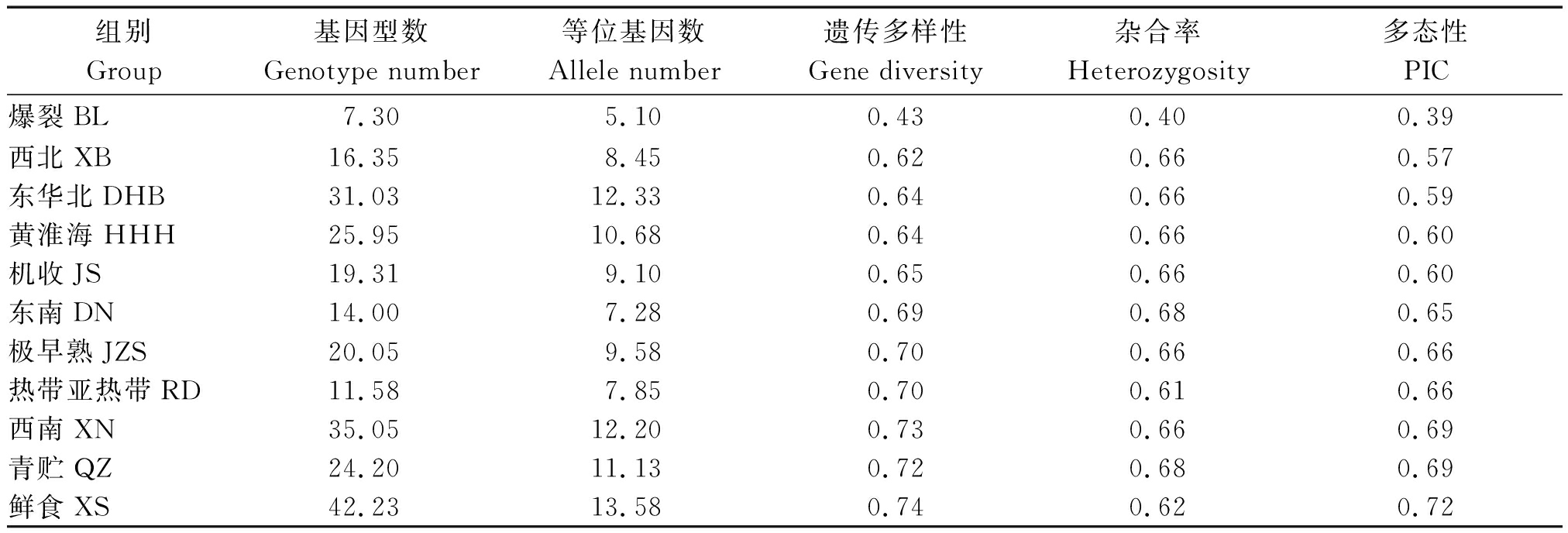
组别Group基因型数Genotype number等位基因数Allele number遗传多样性Gene diversity杂合率Heterozygosity多态性PIC爆裂BL7.305.100.43 0.40 0.39 西北XB16.35 8.45 0.62 0.66 0.57 东华北DHB31.0312.330.640.660.59黄淮海HHH25.9510.680.640.660.60机收JS19.31 9.10 0.65 0.66 0.60 东南DN14.00 7.28 0.69 0.68 0.65 极早熟JZS20.05 9.58 0.70 0.66 0.66 热带亚热带RD11.58 7.85 0.70 0.61 0.66 西南XN35.05 12.20 0.73 0.66 0.69 青贮QZ24.20 11.13 0.72 0.68 0.69 鲜食XS42.23 13.58 0.74 0.62 0.72
2.2 不同区组之间的遗传距离与聚类分析
分析各区组之间的遗传距离和遗传聚类图,结果(表4、图1)表明,爆裂组与其他区组遗传距离最远,遗传聚类时单独成群,其次是热带亚热带、鲜食和极早熟组,东华北和西北之间遗传距离最小。机收组分为机收东北组和机收黄淮海组,2个不同区域机收组之间的遗传差异低于2个区域的常规品种。黄淮海、东华北、西北、机收几个组别之间遗传背景有一定的差异,但遗传背景相似比例明显高于其他组别。
表4 不同组别之间的Nei′s(1972)遗传距离
Tab.4 Nei′s (1972) genetic distance among different regional trail groups
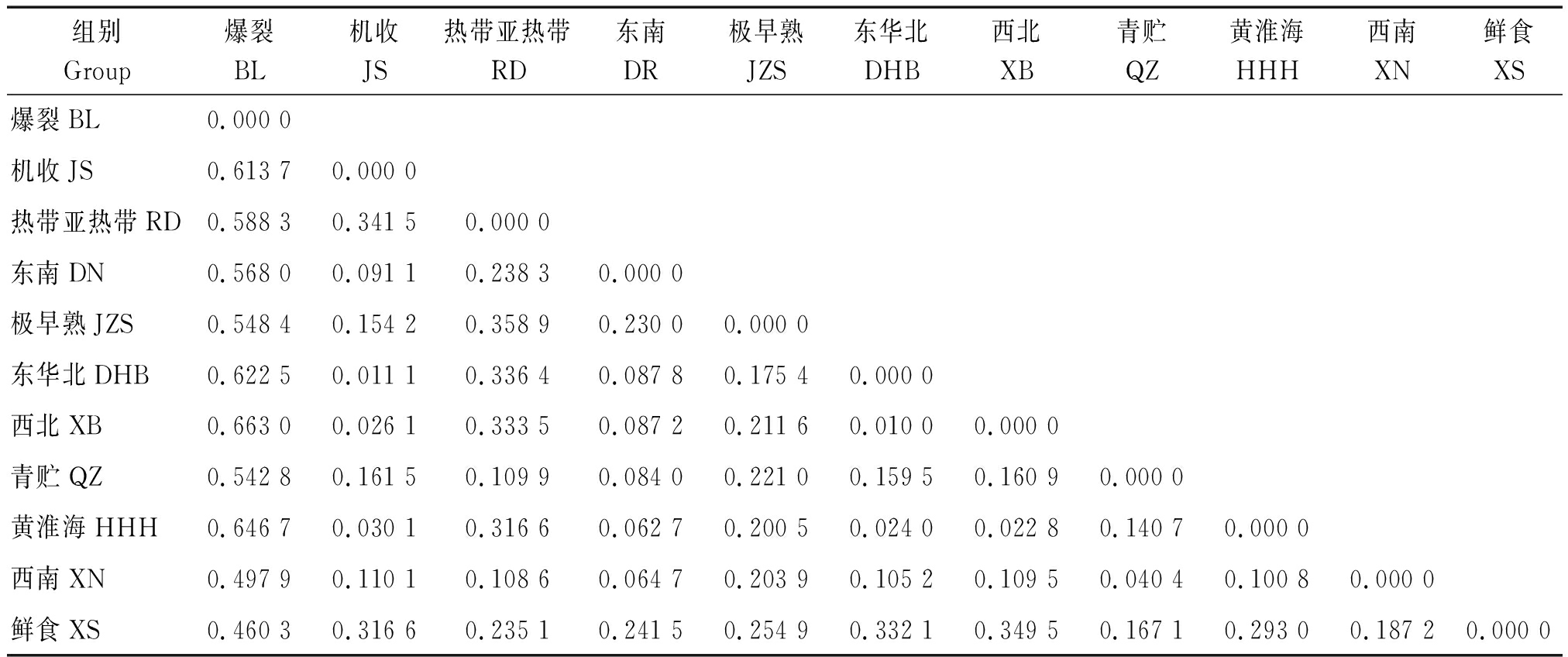
组别Group爆裂BL机收JS热带亚热带RD东南DR极早熟JZS东华北DHB西北XB青贮QZ黄淮海HHH西南XN鲜食XS爆裂BL0.000 0机收JS0.613 70.000 0热带亚热带RD0.588 30.341 50.000 0东南DN0.568 00.091 10.238 30.000 0极早熟JZS0.548 40.154 20.358 90.230 00.000 0东华北DHB0.622 50.011 10.336 40.087 80.175 40.000 0西北XB0.663 00.026 10.333 50.087 20.211 60.010 00.000 0青贮QZ0.542 80.161 50.109 90.084 00.221 00.159 50.160 90.000 0黄淮海HHH0.646 70.030 10.316 60.062 70.200 50.024 00.022 80.140 70.000 0西南XN0.497 90.110 10.108 60.064 70.203 90.105 20.109 50.040 40.100 80.000 0鲜食XS0.460 30.316 60.235 10.241 50.254 90.332 10.349 50.167 10.293 00.187 20.000 0
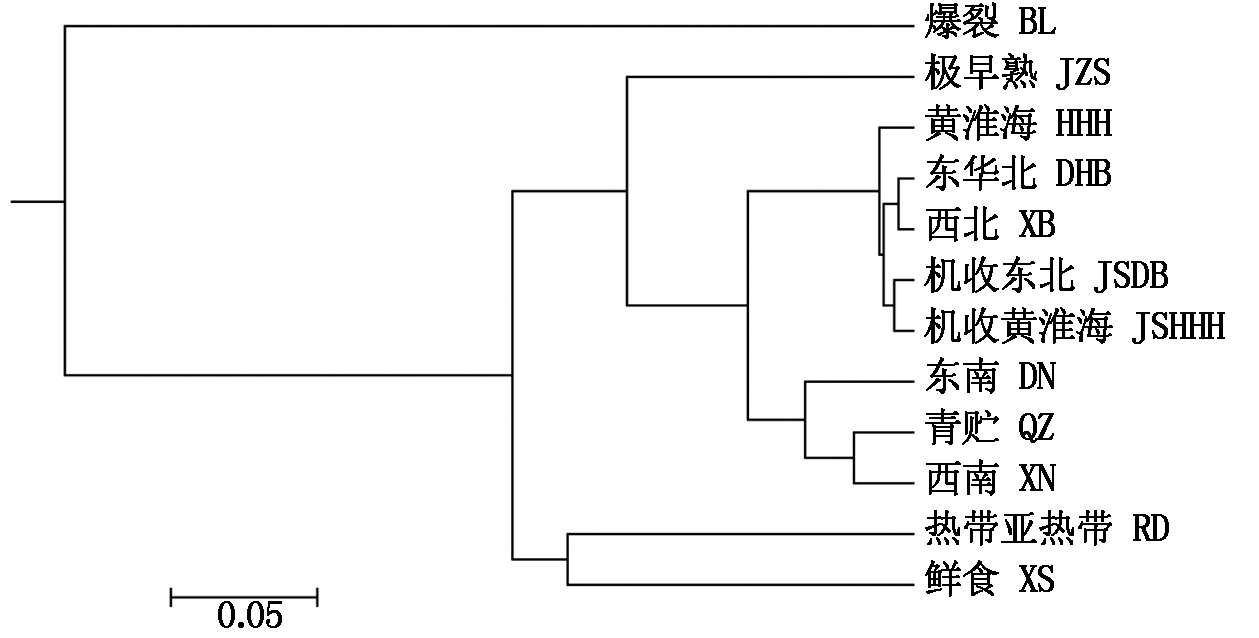
图1 不同区组之间基于UPGMA法的Nei′s遗传聚类图
Fig.1 UPGMA cluster diagram of different trail group (Nei′s)
2.3 参试组合DNA指纹检测与分析
2014-2019年2 949份参试组合共检测出1 152组同名组合,其中1 088组不同来源的同名组合DNA指纹一致,64组不同来源的同名组合DNA指纹差异位点≥2,说明亲本组合发生更换,更换组合的样品占比2.17%。更换组合的样品与对照差异位点大部分在6个以上,表明大部分更换的组合与原组合差异明显。2 949份参试组合与已审定、品种权保护、同一年度及上一年度参试组合比较,共筛查出187套相似品种信息,即6.30%的参试组合与其他品种或组合DNA指纹无明显差异,西北组、东华北组和黄淮海组筛查出的相似品种比例较高,分别为12.90%,10.30%,7.80%,东南组、鲜食组和爆裂组筛查出的相似品种比例较低,分别为1.50%,0.70%,0(图2)。
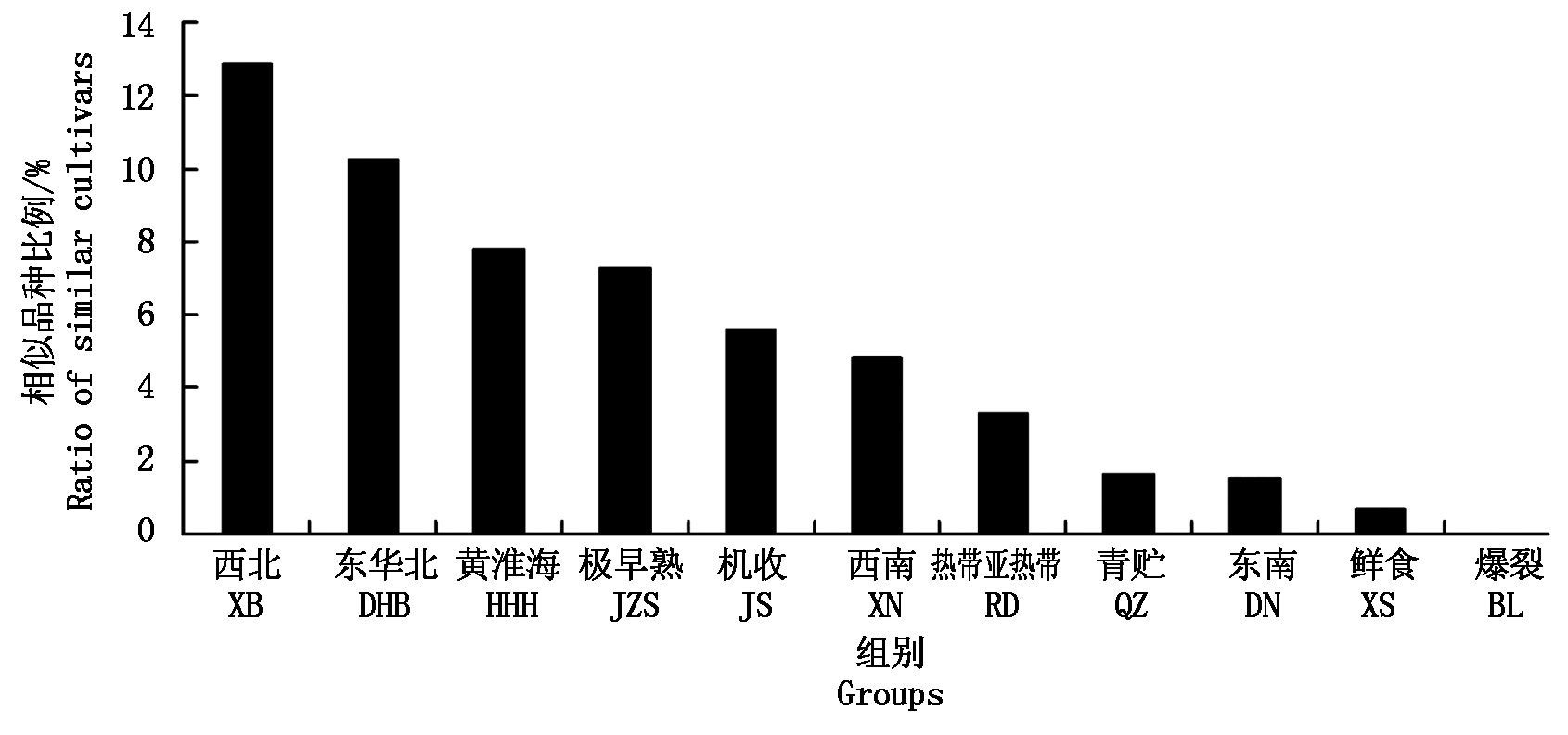
图2 不同区组筛查出的近似品种比例
Fig.2 Proportion of similar varieties detected in different groups
2.4 参试样品与数据库比较差异位点分布
2014-2019年国家区试参试组合与已审定品种、品种权保护品种以及同一年度和上一年度所有参试组合两两比对,年际间随机2个品种的差异位点的分布基本一致,主要分布在28~36个位点之间,表明参试组合整体具有较高的多样性(图3)。2014-2019年的参试组合与数据库比对筛查最近似品种,DNA指纹差异位点在5个以内的品种数呈逐渐下降趋势,表明遗传背景相近的品种比例逐年降低。与数据库比较筛查每个参试组合的最近似品种信息,2014年参试组合与最近似品种比较,频率最高的DNA指纹差异位点为16个,2015-2016年为15个,2017-2019年为13个,表明参试组合之间及与已知品种遗传背景相似性逐年提高(图4)。
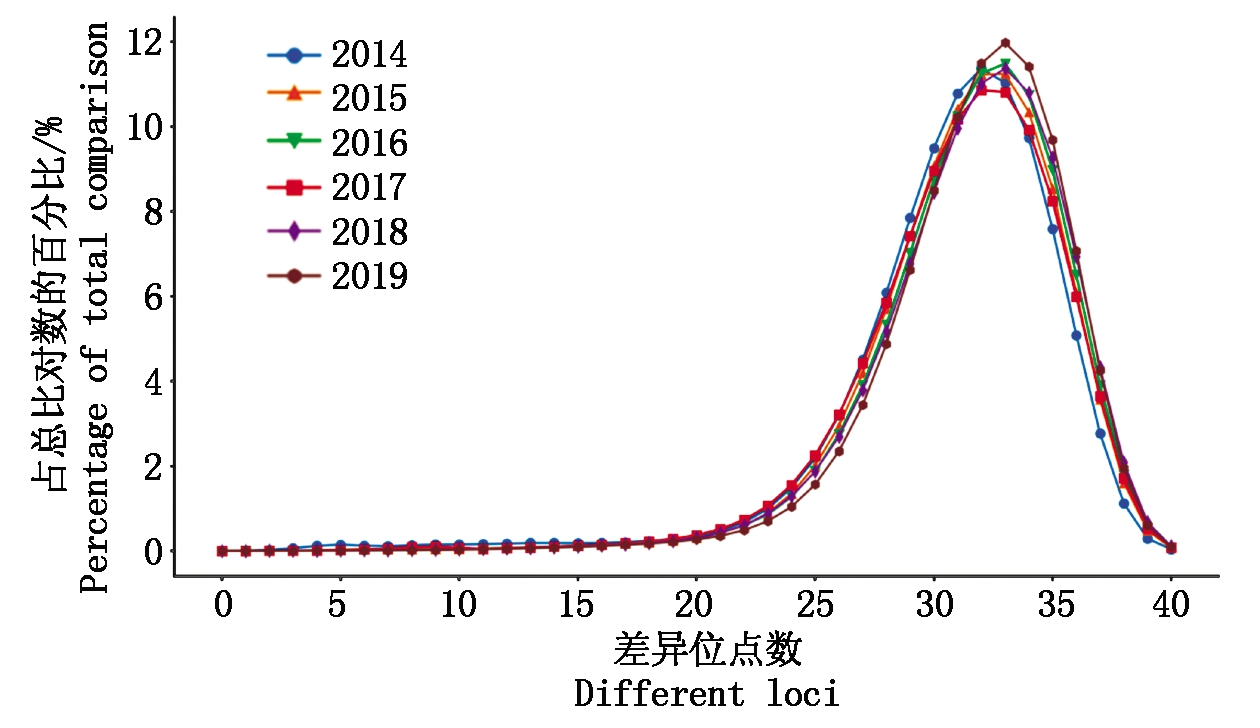
图3 不同年份国家区试参试组合间差异位点数分布
Fig.3 Distribution of the number of differential sites among the national regional trail varieties in different years
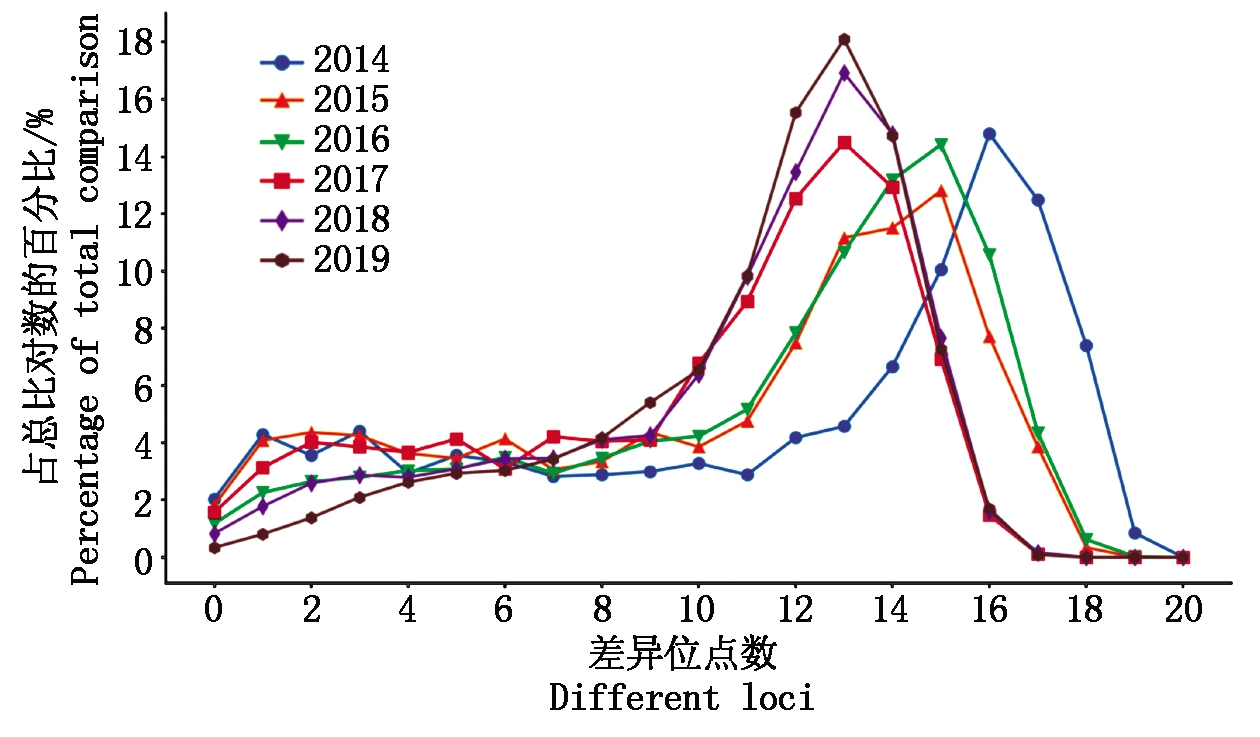
图4 不同年份参试组合与最近似品种比较差异位点分布
Fig.4 Distribution of the most similar variety difference sites in different years
3 结论与讨论
3.1 2014-2019年国家区试参试组合遗传背景的变化趋势
本研究结果表明,随着玉米区域试验持续采用DNA指纹进行检测同名更换和非同名相近的问题,遗传背景相近的参试组合比例在逐年降低,表明直接拿其他品种换名参试或进行高度模仿育种的比例在减少,DNA指纹的持续监控能从源头发挥清理品种多、乱、杂的作用。与王凤格等[15]研究结果比较,本研究分析的2014-2019年国家区试参试组合的基因型数量、等位基因数量、基因多样性、PIC、杂合率均有所提高,表明近10 a来我国玉米选育品种遗传变异和遗传多样性日趋丰富。2014-2019年不同年际间国家区试玉米参试组合之间遗传多样性和差异位点分布基本一致,说明近几年国家区域试验参试组合遗传多样性变化不大,但参试组合与数据库比对最近似品种的差异位点数在减少,侧面反映了近几年来,其他来源参试组合的遗传背景相似度提高了,表明参试组合的激增并未带来种质的创新,参试组合间的遗传背景在逐渐变窄。在不同区组的参试组合中,爆裂组的参试组合多样性水平最低,可能是由于爆裂玉米参试品种参试单位和参试品种较少,关键种质资源相似所致。
3.2 DNA指纹在玉米区域试验中的应用
区域试验是品种审定的重要依据,近几年我国玉米参试组合数量年均达到8 000份,如何准确检测每个品种是否具有特异性,是我国目前品种管理面临的难题,也给DNA指纹技术在区域试验中的应用带来了挑战。虽然2014-2019年检测出的近似品种比率呈现逐年下降的趋势,但由于参试组合数量的大幅增加,每年检测出的DNA指纹相近的品种的绝对数量增加幅度较大。随着分子标记辅助育种技术的不断发展,以及转基因、基因编辑品种的研发,在原有优异品种基础上进行微改良的新品种成为DNA鉴定的难点。随着我国知识产权保护力度的不断增加,为了鼓励原始创新,行业内有人士提出将现行行业标准DNA指纹差异位点判定阈值从“2”调整到“4”。根据近年国家及各省区试、联合体、绿色通道的参试品种信息,SSR差异位点每提高1个将影响约5%的参试组合,根据近几年的参数组合数量计算,1个位点将影响约400个参试组合。DNA指纹差异位点阈值的调整对我国玉米品种选育影响较大,差异位点的提高能抑制大量品种同质化的问题,提高品种原始创新水平。面对新技术、新形式的挑战,需要不断发展和完善DNA指纹技术。
3.3 SNP技术在玉米区域试验中的应用展望
由于SSR只采用了40个位点进行检测,对相似品种的遗传背景扫描标记密度较低,每个位点占比2.50%,不能精确评估相似品种间的遗传关系。SNP作为新一代DNA指纹技术,与其他遗传标记相比,SNP具有以下几个突出的优势:一是在基因组中标记密度更高,分布也更均匀,候选位点多;二是SNP通常为二态位点,数据统计容易,不同来源数据易实现整合和共享,而且试验流程易于自动化;三是单个位点基因分型成本较低;四是具有高通量样品和高通量位点检测的优势。随着技术的不断发展,SNP基因分型成本越来越低,各种SNP基因分型平台已广泛应用于遗传研究和分子标记辅助育种中,包括基于芯片的多位点SNP检测,单SNP基因分型和测序。SNP芯片具有高密度的位点,能更精准地评估相似品种间的遗传背景,适用于区域试验参试组合的特异性筛查。目前多种作物已开发不同通量的SNP芯片[16-21],我国已开发出200K、90K、60K、55K、3 072、384等不同通量的玉米SNP芯片。建议加快SNP芯片鉴定技术在各农作物品种区域试验中的应用研究,为区域试验品种真实性和特异性检测提供更有效的技术方案。
[1] 王凤格, 易红梅, 赵久然, 孙世贤, 杨国航, 任洁, 王璐. DNA指纹技术在玉米区域试验品种真实性及一致性检测中的应用[J]. 分子植物育种, 2016, 14(2): 456-461. doi:10.13271/j.mpb.014.000456.
Wang F G, Yi H M, Zhao J R, Sun S X, Yang G H, Ren J, Wang L. The application of DNA fingerprint technology in maize varieties′ authenticity and consistency identification in maize regional test[J]. Molecular Plant Breeding, 2016, 14(2): 456-461.
[2] 王凤格, 赵久然, 戴景瑞, 王璐, 易红梅, 郭景伦, 孙世贤, 廖琴, 杨国航. 利用SSR标记进行玉米品种一致性检测研究[J]. 分子植物育种, 2007, 5(1): 95-104. doi:10.3969/j.issn.1672-416X.2007.01.018.
Wang F G, Zhao J R, Dai J R, Wang L, Yi H M, Guo J L, Sun S X, Liao Q, Yang G H. Uniformity analysis of maize varieties by a set of SSR markers[J]. Molecular Plant Breeding, 2007, 5(1): 95-104.
[3] 陈发波, 杨克诚, 荣廷昭, 潘光堂. 西南及四川区试玉米组合遗传多样性分析[J]. 作物学报, 2007, 33(6): 991-998. doi:10.3321/j.issn:0496-3490.2007.06.021.
Chen F B, Yang K C, Rong T Z, Pan G T. Analysis of genetic diversity of maize hybrids in the regional tests of Sichuan and Southwest China [J]. Acta Agronomica Sinica, 2007, 33(6): 991-998.
[4] 贺国良, 付高平, 彭从胜, 邓伟, 朱珊, 杨宙, 魏永清, 陈丽妹, 沈林军, 沈显华, 黄仁良. 江西省2018年晚粳区试品种的DNA指纹图谱以及遗传相似性分析[J]. 江西农业大学学报, 2019, 41(5): 843-852. doi:10.13836/j.jjau.2019097.
He G L, Fu G P, Peng C S, Deng W, Zhu S, Yang Z, Wei Y Q, Chen L M, Shen L J, Shen X H, Huang R L. DNA fingerprint map and analysis of genetic diversity of the japonica rice varieties in the regional test in Jiangxi province in 2018[J]. Acta Agriculturae Universitatis Jiangxiensis, 2019, 41(5): 843-852.
[5] 刘丽华, 庞斌双, 刘阳娜, 邱军, 李宏博, 张欣, 王娜, 赵昌平. 2009-2014年国家冬小麦区域试验品系的遗传多样性及群体结构分析[J]. 麦类作物学报, 2016, 36(2): 165-171. doi:10.7606/j.issn.1009-1041.2016.02.06.
Liu L H, Pang B S, Liu Y N, Qiu J, Li H B, Zhang X, Wang N, Zhao C P. Genetic diversity and population structure analysis of winter wheat lines from recent national regional trials in China[J]. Journal of Triticeae Crops, 2016, 36(2): 165-171.
[6] 陈先红, 徐利远, 彭正松, 杜文平, 余桂蓉, 钟昌松, 曲继鹏, 胡凤林. 四川省2005-2006年区试小麦品种(系)的SSR遗传多样性[J]. 分子植物育种, 2007, 5(6): 843-848. doi:10.3969/j.issn.1672-416X.2007.06.015.
Chen X H, Xu L Y, Peng Z S, Du W P, Yu G R, Zhong C S, Qu J P, Hu F L. Genetic diversity of wheat lines from Sichuan field trial in 2005-2006 revealed by SSR markers[J]. Molecular Plant Breeding, 2007, 5(6): 843-848.
[7] 关荣霞, 方宏亮, 何艳琴, 常汝镇, 邱丽娟. 国家大豆区域试验品种(系)SSR位点纯合度分析[J]. 作物学报, 2012, 38(10): 1760-1765. doi:10.3724/SP.J.1006.2012.01760.
Guan R X, Fang H L, He Y Q, Chang R Z, Qiu L J. Molecular homozygosity analysis of soybean varieties (lines) in regional test of China by using SSR markers[J]. Acta Agronomica Sinica, 2012, 38(10): 1760-1765.
[8] 何琳, 何艳琴, 刘业丽, 邱强, 栾怀海, 韩雪, 胡国华. 2012年北方春大豆国家区试大豆品种纯度鉴定、分子ID构建及遗传多样性分析[J]. 中国农学通报, 2014, 30(18): 277-282.
He L, He Y Q, Liu Y L, Qiu Q, Luan H H, Han X, Hu G H. Purity identification, molecular ID establishment and genetic diversity analysis of soybeans attending national regional test of North spring soybean in 2012 [J]. Chinese Agricultural Science Bulletin, 2014, 30(18): 277-282.
[9] 陆光远, 伍晓明, 张冬晓, 刘凤兰, 陈碧云, 高桂珍, 许鲲. SSR标记分析国家油菜区试品种的特异性和一致性[J]. 中国农业科学, 2008, 41(1): 32-42. doi:10.3864/j.issn.0578-1752.2008.01.005.
Lu G Y, Wu X M, Zhang D X, Liu F L, Chen B Y, Gao G Z, Xu K. SSR-based evaluation of distinctness, uniformity and stability (DUS) of rapeseed (Brassica napus L.) varieties being subjected to national official field tests[J]. Scientia Agricultura Sinica, 2008, 41(1): 32-42.
[10] 陈碧云, 伍晓明, 张冬晓, 刘凤兰, 陆光远, 许鲲, 高桂珍. 国家冬油菜区试新品种的SSR指纹图谱分析[J]. 分子植物育种, 2008, 6(4): 709-716.
Chen B Y, Wu X M, Zhang D X, Liu F L, Lu G Y, Xu K, Gao G Z. SSR marker fingerprinting of winter rapeseed varieties in national field trails[J]. Molecular Plant Breeding, 2008, 6(4): 709-716.
[11] 孙宁, 杨付新, 付小琼, 王秀玲, 刘逢举, 李亚丽, 汤磊鹏. 国家棉花区试新品种的SSR指纹图谱分析[J]. 棉花学报, 2011, 23(3): 279-283. doi:10.3969/j.issn.1002-7807.2011.03.015.
Sun N, Yang F X, Fu X Q, Wang X L, Liu F J, Li Y L, Tang L P. SSR marker fingerprinting of cotton varieties in national regional trails[J]. Cotton Science, 2011, 23(3): 279-283.
[12] 李丽, 黄先群, 何天久, 雷尊国, 李其义, 彭义, 黄贵民. 贵州马铃薯审定品种及区试材料的SSR遗传多样性分析[J]. 西南农业学报, 2013, 26(3): 909-913. doi:10.16213/j.cnki.scjas.2013.03.052.
Li L, Huang X Q, He T J, Lei Z G, Li Q Y, Peng Y, Huang G M. Genetic diversity analysis of Guizhou approved and attending regional tests potato by SSR marker[J]. Southwest China Journal of Agricultural Sciences, 2013, 26(3): 909-913.
[13] 陈静, 胡晓辉, 苗华荣, 石运庆, 禹山林. SSR标记分析国家北方花生区试品种的遗传多样性[J]. 植物遗传资源学报, 2009, 10(3): 360-366. doi:10.13430/j.cnki.jpgr.2009.03.004.
Chen J, Hu X H, Miao H R, Shi Y Q, Yu S L. Analysis of genetic diversity of peanut (Arachis hypogaea L.) varieties under the region tests of North China based on SSR marker[J]. Journal of Plant Genetic Resources, 2009, 10(3): 360-366.
[14] 刘红艳, 吴坤, 杨敏敏, 左阳, 赵应忠. 国家芝麻区域试验新品种(系)的DNA指纹分析[J]. 作物学报, 2012, 38(4): 596-605. doi:10.3724/SP.J.1006.2012.00596.
Liu H Y, Wu K, Yang M M, Zuo Y, Zhao Y Z. DNA fingerprinting of sesame (Sesamum indicum L.) varieties (lines) from recent national regional trials in China[J]. Acta Agronomica Sinica, 2012, 38(4): 596-605.
[15] 王凤格, 田红丽, 赵久然, 王璐, 易红梅, 宋伟, 高玉倩, 杨国航. 中国328个玉米品种(组合)SSR标记遗传多样性分析[J]. 中国农业科学, 2014, 47(5): 856-864. doi:10.3864/j.issn.0578-1752.2014.05.003.
Wang F G, Tian H L, Zhao J R, Wang L, Yi H M, Song W, Gao Y Q, Yang G H. Genetic diversity analysis of 328 maize varieties (hybridized combinations) using SSR markers[J]. Scientia Agricultura Sinica, 2014, 47(5): 856-864.
[16] Wang Y P, Cheng X, Shan Q W, Zhang Y, Liu J X, Gao C X, Qiu J L. Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew[J]. Nat Biotechnol, 2014, 32(9):947-951. doi:10.1038/nbt.2969.
[17] Singh N, Jayaswal P K, Panda K, Mandal P, Kumar V, Singh B, Mishra S, Singh Y, Singh R, Rai V, Gupta A, Sharma T R, Singh N K.Single-copy gene based 50K SNP chip for genetic studies and molecular breeding in rice[J]. Sci Rep, 2015, 5: 11600. doi:10.1038/srep11600.
[18] Lee Y G, Jeong N, Kim J H, Lee K, Kim K H, Pirani A, Ha B K, Kang S T, Park B S, Moon J K, Kim N, Jeong S C.Development, validation and genetic analysis of a large soybean SNP genotyping array [J]. Plant J, 2015, 81(4): 625-636. doi:10.1111/tpj.12755.
[19] Hulse-Kemp A M, Lemm J, Plieske J, Ashrafi H, Buyyarapu R, Fang D D, Frelichowski J, Giband M, Hague S, Hinze L L, Kochan K J, Riggs P K, Scheffler J A, Udall J A, Ulloa M, Wang S S, Zhu Q H, Bag S K, Bhardwaj A, Burke J J, Byers R L, Claverie M, Gore M A, Harker D B, Islam M S, Jenkins J N, Jones D C, Lacape J M, Llewellyn D J, Percy R G, Pepper A E, Poland J A, Rai K M, Sawant S V, Singh S K, Spriggs A, Taylor J M, Wang F, Yourstone S M, Zheng X T, Lawley C T, Ganal M W, Van Deynze A, Wilson I W, Stelly D M.Development of a 63K SNP array for cotton and high-density mapping of intraspecific and interspecific populations of Gossypium spp.[J]. G3 (Bethesda), 2015, 5(6): 1187-1209. doi:10.1534/g3.115.018416.
[20] Ganal M W, Durstewitz G, Polley A, Bérard A, Buckler E S, Charcosset A, Clarke J D, Graner E M, Hansen M, Joets J, Paslier M L, McMullen M D, Montalent P, Rose M, Schon C C, Sun Q, Walter H, Martin O C, Falque M. A large maize (Zea mays L.) SNP genotyping array:development and germplasm genotyping, and genetic mapping to compare with the B73 reference genome[J]. PLoS One, 2011, 6(12): e28334. doi:10.1371/journal.pone.0028334.
[21] Xu C, Ren Y H, Jian Y Q, Guo Z F, Zhang Y, Xie C X, Fu J J, Wang H W, Wang G Y, Xu Y B, Li P, Zou C. Development of a maize 55K SNP array with improved genome coverage for molecular breeding [J]. Mol Breed, 2017, 37(31):20. doi:10.1007/s11032-017-0622-z.
