器官的大小作为植物重要的形态特征是其进化与适应环境的结果;同时,器官大小的调控也是一个重要的生物学过程。研究器官大小调控的分子机制对作物生物量的改良具有潜在的应用价值。尽管目前已经发现了许多调控植物器官大小的基因,但是器官大小调控的分子调控网络仍不清楚。进一步分离和鉴定器官大小变异突变体有助于深入了解器官大小调控的分子机制,并且为分子育种改良提供理论基础和基因资源。
植物器官的最终大小是由细胞的数目和细胞的大小所决定[1-3]。植物的器官在发育时分为2个连续的阶段。第一阶段是器官原基中的细胞进行分裂,分裂达到一定的数目后就会停止,细胞体积变化不大。第二阶段是细胞开始进行扩展,细胞的体积开始增加。细胞分裂和细胞扩展的协同调控决定了最终的器官大小[2]。到目前为止,在模式植物拟南芥中已发现许多影响器官大小的因子,包含转录调控、激素信号调控通路及蛋白质合成与修饰等多个水平和途径[4-5]。其中通过促进细胞分裂来影响器官生长的基因有:AUXIN-REGULATED GENE INVOLVED IN ORGAN SIZE(ARGOS)、GROWTH REGULATING FACTOR(GRF)、KLUH(KLU)/CYP78A5和JAGGED(JAG)等[6-10]。有些基因能够通过抑制细胞分裂调控器官大小。例如,过量表达ROTUNDIFOLIA4(ROT4)的突变体rot4-1D,其叶片的细胞分裂受到抑制,表现出变短变圆的表型[11]。拟南芥DA1基因编码一个泛素受体蛋白。da1-1突变体外珠被细胞数目增多,种子花瓣均增大。细胞学实验表明,DA1通过细胞分裂来调控种子大小[12]。eod1(enhancer of da1-1)是da1-1突变体的增强子,EOD1/BIG BROTHER (BB)编码一个E3泛素连接酶。过表达EOD1/BB时,其花瓣和叶片都变小。DA1与EOD1/BB协同抑制细胞分裂来调控器官的大小[13-14]。此外,也发现了一些基因能够通过影响细胞扩展调控器官大小。当过量表达ORGAN SIZE RELATED2(OSR2)时细胞的扩展得到增强,植物器官明显增大。BIGPETAL(BPEp)与AUXIN RESPONSE FACTOR8(ARF8)发生相互作用,在花瓣发育的后期协同限制细胞扩展[15-16]。
李云海课题组前期研究中发现了器官大小的重要调控因子UBIQUITIN SPECIFIC PROTEASE15 (UBP15)/SUPPRESSOR2 OF DA1 (SOD2)。UBP15是一个泛素特异的蛋白酶,通过调控细胞的分裂来调控器官的大小[17]。突变体sod2-1的器官与野生型相比显著变小。UBP15与器官大小的正调控因子DA1能够直接发生互作,并且UBP15作为泛素受体DA1的一个直接下游底物,其蛋白稳定性受DA1的调节。在遗传上,sod2-1能够完全抑制da1-1器官变大的表型。因此,DA1-UBP15是一个重要的器官大小调控途径。
为了进一步鉴定DA1-UBP15途径的新成员或新的器官大小的调控因子,在sod2-1的背景下通过甲基硫酸乙酯(Ethyl-methanesulphonate,EMS)诱变筛选得到sod2-1的抑制子突变体suppressors of sod2(sus40-1D),对sus40-1D sod2-1突变体的花瓣和种子与sod2-1进行对比分析,并通过F2群体构建与重测序分析挖掘sus40-1D的候选基因,为更好了解植物器官大小调控的分子机理奠定基础。
1 材料和方法
1.1 材料和生长条件
以拟南芥突变体sod2-1为原始材料,经EMS诱变获得了器官变大的突变体sus40-1D sod2-1。将sod2-1与sus40-1D sod2-1进行杂交获得F1,并通过F1植株自交得到F2。F2群体用于基因的定位研究。试验所用野生型材料为拟南芥Columbia生态型(Col-0)。生长的温度为22 ℃,光照周期为16 h光照/8 h黑暗。
1.2 分析方法
1.2.1 形态学分析 花瓣、子叶和种子的测量:将花瓣或子叶放在1%的琼脂培养基上铺平,在体式显微镜(LEICA S8APO)下进行拍摄,种子的大小用体式显微镜拍摄。照片利用Image J软件测量器官的面积。统计数据均进行t检验分析。
1.2.2 遗传分析 突变体sod2-1与sus40-1D sod2-1杂交获得F1群体,考察F1植株的表型。F1自交构建F2分离群体,待植株开花后考察F2分离群体中单株的形态,统计器官变小突变体单株和器官变大突变体单株的数目并进行卡方适合性检测。
1.2.3 重测序筛选基因 对sod2-1与sus40-1D sod2-1杂交的F2分离群体,在开花后对器官变大和器官变小的植株各选取45株剪取叶片,然后把各45个样品等量混合进行DNA提取。利用高通量测序与MutMap[18-19]技术分析筛选突变基因。将测序所得的2份混池数据以野生型Col-0基因组做比对获得SNPs数据群,将野生型与混池都含有的SNPs去除,获得混池所特有的SNPs数据。计算每个SNP的突变频次与该位点总深度的比值(SNP-index),SNP-index为1时,其SNP与表型连锁。进一步挑选特异SNPs数据中SNP-index为1的SNP为候选基因。
1.2.4 dCAPS标记 利用dCAPS分子标记网站在线分析,挑选适合的限制性内切酶和上下游引物,PCR扩增后酶切即可作为合适的dCAPS标记。选定2个dCAPS标记位点用来突变位点验证和目的基因的连锁分析(表1)。
表1 用于dCAPS分析的引物及酶
Tab.1 Primers and enzymes for dCAPS analysis

引物Primer引物序列(5'-3')Primer sequence片段长度/bpFragment length酶EnzymedCAPS 1F:GCGTTATCTGTACGTGGCGA188Kpn ⅠR:CAGTTGTCAGAGGCTCATCGAAACAAG-GTACdCAPS 2F:GCAAGCCACTGATCAGGAGAGTTAG191EcoR ⅠR:AGTTGGTCCGGAAGGAGGTT-TAGCGAATT
2 结果与分析
2.1 突变体sus40-1Dsod2-1的分离与鉴定
前期研究表明,器官大小调控因子UBP15/SOD2通过影响细胞的增殖来控制器官大小。为了更好研究UBP15在器官大小中的调控机制,通过对sod2-1突变体种子进行EMS化学诱变处理并进行遗传筛选寻找sod2-1突变体的抑制子。试验结果表明,在M2中成功筛选能抑制sod2-1表型的突变体,得到了多个sod2-1的抑制子突变体,这些抑制子被命名为suppressors of sod2-1(简称sus),其中一个突变体(sus40-1D)显著抑制sod2-1的器官大小的表型(图1-A)。
2.2 sus40-1D抑制了sod2-1的种子和器官大小表型
对主茎上第4~10个花瓣和成熟角果的测量结果显示,sod2-1的种子面积比野生型显著减少了17%,而sus40-1D sod2-1则比野生型显著增加了16%,同时相对sod2-1,种子面积显著变大(图1-B,E);sod2-1花瓣面积比野生型显著减少了22%,而突变体sus40-1D sod2-1比野生型显著增加了21%(图1-C,E);对萌发10 d的子叶测量发现,sus40-1D sod2-1突变体子叶较sod2-1也显著变大(图1-D,E)。这些结果表明,sus40-1D在器官和种子大小方面均能抑制sod2-1的表型。
2.3 突变体sus40-1D的遗传分析
将突变体sus40-1D sod2-1与sod2-1杂交获得F1,考察F1植株叶片的表型,其大小均表现为sus40-1D sod2-1的表型。F1自交构建F2分离群体,于开花后考察F2分离群体并统计168株单株叶片植株的形态,统计结果显示,表现为sod2-1表型的有45株,表现为sus40-1D sod2-1表型的有123株。卡方适合性检测结果显示,F2的分离符合3∶1的分离规律。以上试验结果表明,该突变体器官变大性状受一对显性基因控制(表2)。
2.4 候选基因的筛选与鉴定

A.野生型Col-0、sod2-1和sus40-1D sod2-1的植株形态(标尺:2 cm)。B.野生型Col-0、sod2-1和sus40-1D sod2-1的种子(标尺:1 mm)。C.野生型Col-0、sod2-1和sus40-1D sod2-1的花瓣(标尺:1 mm)。D.野生型Col-0、sod2-1和sus40-1D sod2-1的子叶(标尺:2 mm)。E.野生型Col-0、sod2-1和sus40-1D sod2-1的种子面积、花瓣面积和子叶面积的统计数据(n≥50)。不同字母表示差异显著(P<0.05)。
A. Plants of Col-0, sod2-1 and sus40-1D sod2-1 (Scale bar: 2 cm). B. Seeds of Col-0, sod2-1 and sus40-1D sod2-1(Scale bar: 1 mm) . C. Petals of wild-type Col-0, sod2-1 and sus40-1D sod2-1 (Scale bar: 1 mm). D. Cotyledons of Col-0, sod2-1 and sus40-1D sod2-1 (Scale bar: 2 mm). E. Seed area, petal area and cotyledon area of Col-0, sod2-1 and sus40-1D sod2-1 (n≥50).Different letters indicate significant differences (P<0.05).
图1 突变体表型及相关性状分析
Fig.1 Analysis of mutant phenotypes and related traits
表2 遗传分析
Tab.2 Genetic analyses
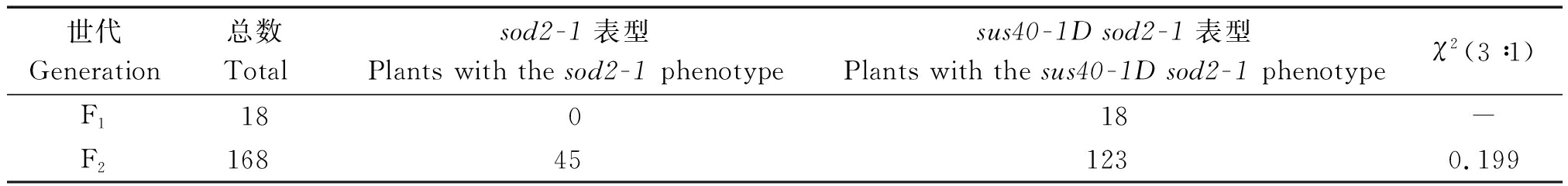
世代Generation总数Totalsod2-1表型Plants with the sod2-1 phenotypesus40-1D sod2-1表型Plants with the sus40-1D sod2-1 phenotypeχ2(3∶1)F118018-F2168451230.199
为了鉴定sus40-1D突变位点,从sus40-1D sod2-1与sod2-1杂交的F2群体中,选取了45株sus40-1D sod2-1表型和45株sod2-1表型的单株分别等量混合提取DNA并进行全基因组重测序。重测序数据分析结果共检测出3 108个SNP,连锁分析发现位于1号染色体上的SNP与sus40-1D sod2-1存在表型连锁。由于sus40-1D是显性突变,因此,在重测序的F2群体中sus40-1D sod2-1表型植株的基因型为纯合突变体或杂合型,重测序的F2群体中sod2-1表型植株的基因型为野生型,说明这些SNPs可能与突变表型连锁。
表3 突变位点分析
Tab.3 Analyses of mutation sites
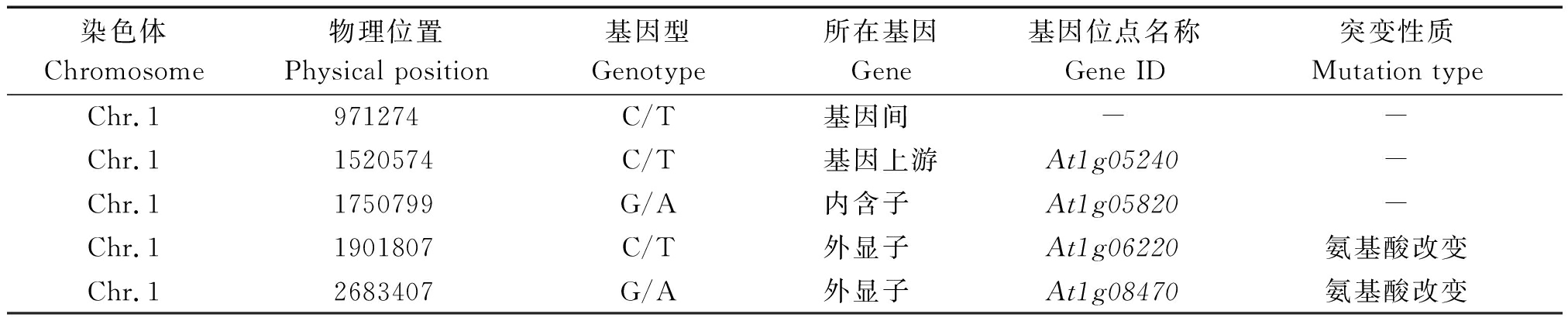
染色体Chromosome物理位置Physical position基因型Genotype所在基因Gene基因位点名称Gene ID突变性质Mutation typeChr.1971274C/T基因间 - -Chr.11520574C/T基因上游At1g05240-Chr.11750799G/A内含子 At1g05820-Chr.11901807C/T外显子 At1g06220氨基酸改变Chr.12683407G/A外显子 At1g08470氨基酸改变
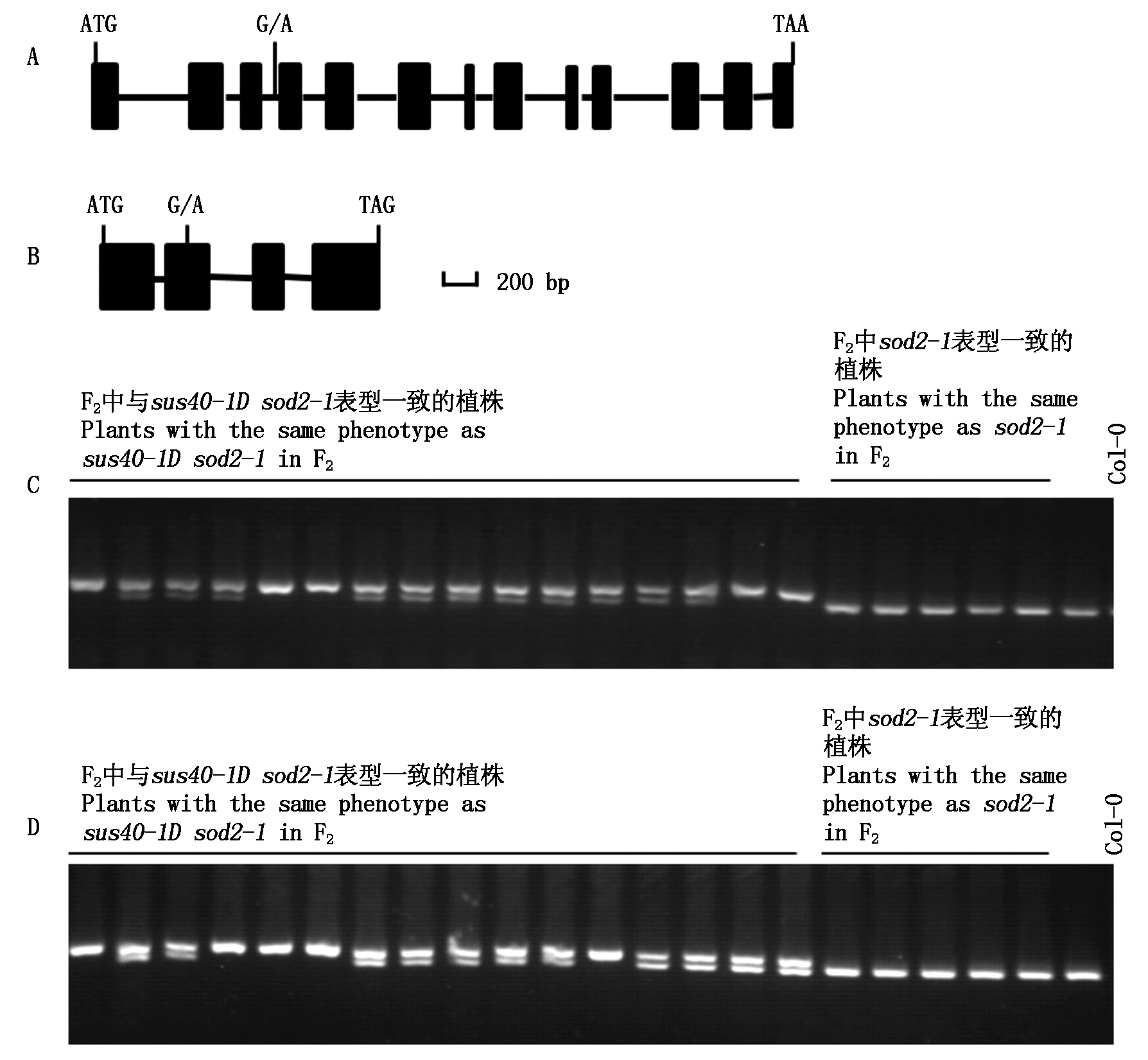
A.候选基因1:At1g05820的基因结构图;B.候选基因2:At1g08470的基因结构图;C.候选基因1的部分F2群体验证;D.候选基因2的部分F2群体验证。
A. The gene structure of candidate gene 1:At1g05820; B. The gene structure of candidate gene 2:At1g08470; C. Partial F2 population verification of candidate gene 1; D. Partial F2 population verification of candidate gene 2.
图2 候选基因鉴定
Fig.2 Identification of candidate genes
对于1号染色体突变位点的分析结果(表3)显示,其中有2个突变位点在外显子区,另外3个突变位点发生在内含子、基因间和基因上游。针对这些SNPs开发dCAPS标记并利用F2分离群体的2种表型植株进行上样检测,发现其中的2个SNP与表型发生共分离,这2个SNP分别发生在At1g05820和At1g08470基因上。其中,SNP1发生在At1g05820的内含子区(图2-A);SNP2发生在At1g08470的外显子区,在2683407处发生由G变为A的碱基突变,导致了非同义突变,由丝氨酸转变成苯丙氨酸,从而导致了蛋白质一级结构的改变(图2-B)。
根据At1g05820、At1g08470基因上的突变位点,分别设计了dCAPS1和dCAPS2 2个分子标记。基于这2个标记对突变体与野生型的分析结果表明,突变体sus40-1D sod2-1中的2个候选基因的确发生了突变。对120个F2分离群体的检测结果显示,该突变位点与表型共分离,基因与表型完全连锁(图2-C,D)。试验结果进一步证明了At1g05820与At1g08470为sus40-1D突变体的候选基因。考虑到SNP2发生在At1g08470的外显子区,并导致了氨基酸的改变,因此,At1g08470是最可能的候选基因。
3 结论与讨论
植物器官大小是十分重要的数量性状,其分子调控机制研究在理解植物生长发育与促进农作物丰产方面具有重要的生物学意义。目前,已经陆续发现了许多调控植物器官大小的基因,但是具体的分子调控机制还未能详细阐述。鉴定发现更多的参与器官大小调控的基因,并阐明器官大小调控因子之间的相互关系,有助于深入了解器官大小调控的内在机理并最终应用到农作物的分子改良研究中。
前期研究表明DA1能够切割UBP15/SOD2从而调控植物器官大小,但是UBP15/SOD2下游成员并不清楚。本研究通过在sod2-1背景下遗传筛选并获得一个抑制子突变体sus40-1D sod2-1,其植株表现出较大的子叶、花瓣与种子。利用基因组重测序技术和连锁分析对突变导致植株变大表型的SNP进行筛查,最终确定位于1号染色体的At1g05820和At1g08470为候选基因。进一步通过分子标记对F2群体分析也进一步证实,这2个突变位点与表型共分离。
基于蛋白结构分析发现,At1g05820编码一个类信号肽酶SPPL5,在拟南芥中SPPL共有AtSPPL1~AtSPPL55种,属于信号肽酶SPP的同系物。序列分析表明,SPPL具有疏水性高的跨膜结构域,现已经在人类和其他脊椎动物中鉴定出具有相似结构的蛋白质[20-21]。在斑马鱼中敲除SPP或SPPL3会引起中枢神经系统功能缺陷并导致细胞死亡[22],在果蝇和秀丽隐杆线虫中SPP敲除则会导致幼虫发育异常[23]。在植物中,SPPL5的功能却鲜有报道。以上这些结果表明,SPP和SPPL在基本生命过程中起着至关重要的作用。At1g08470基因编码异胡豆苷合成酶类似基因(STRICTOSIDINE SYNTHASE-LIKE 3,SSL3),与中华白菜基因存在同源性,但SLL3基因鲜有报道。在拟南芥中SSL基因分为不同的组,串联排列在3号染色体上的4个基因(AtSSL4-AtSSL7)与果蝇和秀丽隐杆线虫的SSL基因相似[24]。SSL5-73个基因在植株正常状态下中低水平表达,在面临各种刺激下能够被诱导表达。而SSL4在植物中具备相对较高的基础水平表达,但诱导表达作用并不明显。本研究中检测到在SSL3基因第2个外显子区发生碱基置换导致非同义突变,由丝氨酸替换成苯丙氨酸。由于sus40-1D突变是一个显性突变,该氨基酸的改变可能增强了该蛋白的功能。
综上所述,本试验通过遗传筛选得到了sod2-1的抑制子突变体sus40-1D,并利用重测序技术、连锁分析和dCAPS分子标记对突变位点进行鉴定,得到了2个候选基因At1g05820和At1g08470。这一发现为DA1-UBP15的下游途径提供了遗传基础。未来需要构建基因组互补载体对sus40-1D sod2-1突变体进行遗传转化与功能验证,最终确定真正SUS40基因。
[1] Orsi C H, Tanksley S D. Natural variation in an ABC transporter gene associated with seed size evolution in tomato species[J]. PLoS Genet, 2009, 5(1):e1000347.doi:10.1371/journal.pgen.1000347.
[2] Anastasiou E, Kenz S, Gerstung M, MacLean D, Timmer J, Fleck C, Lenhard M. Control of plant organ size by KLUH/CYP78A5-dependent intercellular signaling[J]. Developmental Cell, 2007, 13(6):843-856.doi:10.1016/j.devcel.2007.10.001.
[3] Liu C W, Li X H, Thompson D, Wooding K, Chang T L, Tang Z Y, Yu H T, Thomas P J, DeMartino G N. ATP binding and ATP hydrolysis play distinct roles in the function of 26S proteasome[J]. Mol Cell, 2006, 24(1):39-50.doi:10.1016/j.molcel.2006.08.025.
[4] Powell A E, Lenhard M. Control of organ size in plants[J]. Curr Biol, 2012, 22(9):360-367.doi:10.1016/j.cub.2012.02.010.
[5] Krizek B A. Making bigger plants: key regulators of final organ size[J]. Curr Opin Plant Biol, 2009, 12(1):17-22.doi:10.1016/j.pbi.2008.09.006.
[6] Mizukami Y, Fischer R L. Plant organ size control: AINTEGUMENTA regulates growth and cell numbers during organogenesis[J]. Proc Natl Acad Sci, 2000, 97(2):942-947.doi:10.1073/pnas.97.2.942.
[7] Hu Y X, Xie Q, Chua N H. The Arabidopsis auxin-inducible gene ARGOS controls lateral organ size[J]. The Plant Cell, 2003, 15(9):1951-1961.doi:10.1105/tpc.013557.
[8] Horiguchi G, Kim G T, Tsukaya H. The transcription factor AtGRF5 and the transcription coactivator AN3 regulate cell proliferation in leaf primordia of Arabidopsis thaliana[J]. Plant J, 2005, 43(1):68-78.doi:10.1111/j.1365-313X.2005.02429.x.
[9] Kazama T Q, Ichihashi Y, Murata S, Tsukaya H. The mechanism of cell cycle arrest front progression explained by a KLUH/CYP78A5-dependent mobile growth factor in developing leaves of Arabidopsis thaliana[J]. Plant Cell Physiol, 2010, 51(6):1046-1054.doi:10.1093/pcp/pcq051.
[10] Dinneny J R, Yadegari R, Fischer R L, Yanofsky M F, Weigel D. The role of JAGGED in shaping lateral organs[J]. Development, 2004, 131:1101-1110.doi:10.1242/dev.00949.
[11] Wen J Q, Lease K A, Walker J C. DVL, a novel class of small polypeptides: overexpression alters Arabidopsis development[J]. Plant J, 2004, 37(5):668-677.doi:10.1111/j.1365-313x.2003.01994.x.
[12] Li N, Li Y H. Ubiquitin-mediated control of seed size in plants[J]. Front Plant Sci, 2014, 5:332.doi:10.3389/fpls.2014.00332.
[13] Disch S, Anastasiou E, Sharma V K, Laux T, Fletcher J C, Lenhard M. The E3 ubiquitin ligase BIG BROTHER controls Arabidopsis organ size in a dosage-dependent manner[J]. Curr Biol, 2006, 16(3):272-279.doi:10.1016/j.cub.2005.12.026.
[14] Li Y H, Zheng L Y, Corke F, Smith C, Bevan M W. Control of final seed and organ size by the DA1 gene family in Arabidopsis thaliana[J]. Genes Dev, 2008, 22: 1331-1336.doi:10.1101/gad.463608.
[15] Szécsi J, Joly C, Bordji K, Varaud E, Cock J M, Dumas C, Bendahmane M. BIGPETALp, a bHLH transcription factor is involved in the control of Arabidopsis petal size[J]. EMBO J, 2006, 25(16):3912-3920.doi:10.1038/sj.emboj.7601270.
[16] Varaud E, Brioudes F, Szecsi J, Leroux J, Brown S, Perrot-Rechenmann C, Bendahmane M. AUXIN RESPONSE FACTOR8 regulates Arabidopsis petal growth by interacting with the bHLH transcription factor BIGPETALp[J]. Plant Cell, 2011, 23(3):973-983.doi:10.1105/tpc.110.081653.
[17] Du L, Li N, Chen L L, Xu Y X, Li Y, Zhang Y Y, Li C Y, Li Y H. The ubiquitin receptor DA1 regulates seed and organ size by modulating the stability of the ubiquitin-specific protease UBP15/SOD2 in Arabidopsis[J]. The Plant Cell, 2014, 26(2):665-677.doi:10.1105/tpc.114.122663.
[18] Abe A, Kosugi S, Yoshida K, Natsume S, Takagi H, Kanzaki H, Matsumura H, Yoshida K, Mitsuoka C, Tamiru M, Innan H, Cano L, Kamoun S, Terauchi P. Genome sequencing reveals agronomically important loci in rice using MutMap[J]. Nat Biotechnol, 2012, 30(2): 174-178.doi:10.1038/nbt.2095.
[19] 袁金红,李俊华,袁娇娇,贾克利,李书粉,邓传良,高武军.基于全基因组测序的MutMap方法在正向遗传学研究中的应用[J].遗传,2017,39(12):1168-1177.doi:10.16288/j.yczz.17-095.
Yuan J H, Li J H, Yuan J J, Jia K L, Li S F, Deng C L, Gao W J. The application of MutMap in forward genetic studies based on whole-genome sequencing[J]. Hereditas,2017,39(12):1168-1177.
[20] Friedmann E, Lemberg M K, Weihofen A, Dev K K, Dengler U, Rovelli G, Martoglio B. Consensus analysis of signal peptide peptidase and homologous human aspartic proteases reveals opposite topology of catalytic domains compared with presenilins[J]. J Biol Chem, 2004, 279(49):50790-50798.doi:10.1074/jbc.M407898200.
[21] Tamura T, Asakura T, Uemura T, Ueda T, Terauchi K, Misaka T, Abe K. Signal peptide peptidase and its homologs in Arabidopsis thaliana-plant tissue-specific expression and distinct subcellular localization[J]. FEBS J, 2008, 275(1):34-43.doi:10.1111/j.1742-4658.2007.06170.x.
[22] Krawitz P, Haffner C, Fluhrer R, Steiner H, Schmid B, Haass C. Differential localization and identification of a critical aspartate suggest non-redundant proteolytic functions of the presenilin homologues SPPL2b and SPPL3[J]. J Biol Chem, 2005, 280(47):39515-39523.doi:10.1074/jbc.M501645200.
[23] Casso D J, Tanda S, Biehs B, Martoglio B, Kornberg T B. Drosophila signal peptide peptidase is an essential protease for larval development[J]. Genetics, 2005, 170(1):139-148.doi:10.1534/genetics.104.039933.
[24] Fabbri M, Delp G, Schmidt O, Theopold U. Animal and plant members of a gene family with similarity to alkaloid-synthesizing enzymes[J]. Biochemical and Biophysical Research Communications, 2000,271(1):191-196.doi:10.1006/bbrc.2000.2598.
