查尔酮合成酶(Chalcone synthase,CHS)是植物类黄酮生物合成过程中第1个限速酶和关键酶,它催化丙二酰辅酶A与香豆酰辅酶A生成四羟基查尔酮[1-3]。CHS基因最初从欧芹中分离到[4],目前,已克隆得到了许多植物的CHS基因。由于CHS基因在植物黄酮代谢途径中具有重要功能,涉及花色、育性、抵御紫外线和致病因子等诸多植物生理生化过程,该基因得到了广泛、深入的研究[5-8]。此外,黄酮类物质也具有很强的抗氧化性和自由基清除能力,多项证据表明,黄酮类化合物对人体健康有积极的影响,包括抗肿瘤、抗氧化、抗炎症等[9-10]。近年来,植物中黄酮类化合物生物合成代谢关键酶基因的克隆、表达和功能研究已引起了广泛的关注,CHS基因日益成为植物遗传育种、抗逆等领域的研究热点。
作为三大谷物之一,小麦是世界范围的主要粮食作物之一,全球1/3以上的人以小麦为主食。随着气候的变化,小麦的生存环境面临着严峻的挑战[11]。通过改良小麦的遗传背景,提高小麦的品质和抗逆能力是当前研究的重点之一。植物类黄酮不仅参与花色素的合成,还参与抵御逆境的生理活动,同时籽粒中黄酮类物质是重要的抗氧化成分,对人体健康有着重要作用,因此,通过对小麦中类黄酮合成途径的研究并通过育种和分子生物学手段提高小麦籽粒中类黄酮含量对改善小麦品质有着重要意义。为此,对小麦TaCHS基因进行克隆和序列特征分析,并进一步对黄淮麦区119份小麦品种TaCHS基因的多态性进行分析,以期为进一步深入研究TaCHS基因的功能以及不同等位变异与籽粒抗氧化性之间的关系奠定基础。
1 材料和方法
1.1 试验材料
小麦品种周麦98165和扬麦10号于2015-2016年种植于河南农业大学科教园区,小区面积3 m×7 m,在开花期对同日开花的小麦穗进行标记,分别取周麦98165和扬麦10号花后7,14,21,28 d的籽粒样品;小麦品种许科316于2016-2017年种植于河南农业大学科教园区,取花后15 d的根、茎、叶和籽粒样品。所取样品液氮速冻后,保存于-80 ℃冰箱备用。
1.2 DNA与RNA的提取及cDNA第一链的合成
材料DNA用SLS方法在籽粒中提取。小麦根、茎、叶和籽粒中总RNA的提取使用TransZol Plant试剂盒(全式金),并立即用ReverTra Ace qPCR RT Master Mix with gDNA Remover (ToYoBo)进行反转录,合成cDNA第一链。
1.3 TaCHS基因的克隆
参照NCBI中国春TaCHS(AY286095.1)的基因序列设计1对引物TaCHS-F/TaCHS-R(表1),使用Phanta® Max Super-Fidelity DNA Polymerase(南京诺唯赞)在119份小麦品种中克隆TaCHS基因,并在河南尚亚生物技术有限公司进行测序。
表1 本研究所用引物
Tab.1 Primers used in this study

引物名称Primer name引物序列(5'-3')Primer sequence(5'-3')退火温度/℃Annealing temperature用途PurposeTaCHS-FTGTCATTCTTCTCTGCCTCC57.8TaCHS基因克隆TaCHS-RTCTACCAAATCCGTCCTCTC57.8TaCHS-1FCAGATCAGGAAGAGGTACATG-CAC57.6荧光定量PCRTaCHS-1RGACCTCCACGACGACAATG59.7Actin-FCCTCTCTTAGCACTTTTCCAGCA60.0内参Actin-RGTAAGTCCCCTTCACCGACTC60.0
1.4 TaCHS基因编码蛋白的生物信息学分析
用ExPASy-ProtParam(https://web.ex-pasy.org/protparam/)分析蛋白质的分子质量、等电点、稳定性等理化性质;用WOLF PSORT(https://psort.hgc.jp/)和TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)对TaCHS蛋白的信号肽、亚细胞定位及跨膜区进行预测;用InterProScan(http://www.ebi.ac.uk/interpro/)对蛋白质保守结构域进行预测。用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)预测TaCHS蛋白的二级结构,用Phyre2(http://www.sbg.bio.ic.ac.uk/Phyre2/html/page.cgi?id=index)预测蛋白质的三级结构。以TaCHS编码蛋白质序列为探针,在Ensembl Plants(http://plants.ensembl.org/index.html)搜索不同物种的编码序列,利用CDS序列在Mega 5.0中构建基于Neigbor-joining的系统发育树,预测亲缘关系,并在GSDS 2.0(http://gsds.cbi.pku.edu.cn/)中构建不同物种CHS基因的结构图。
1.5 TaCHS基因的表达分析
设计引物TaCHS-1F/TaCHS-1R(表1),并使用TB Green Premix Ex Taq Ⅱ(TaKaRa)分析TaCHS基因在小麦不同组织部位的表达情况以及籽粒灌浆过程中表达量的变化趋势,荧光定量PCR仪为Quantstudio 5 Real-time PCR(Thermo Fisher),以小麦Actin基因为内参。
2 结果与分析
2.1 小麦TaCHS基因的序列分析
以NCBI上中国春TaCHS基因(AY286095.1)为参考序列设计引物TaCHS-F/TaCHS-R,扩增小麦品种许科316,获得其TaCHS基因DNA全长。以TaCHS基因为探针在Ensembl Plants六倍体小麦数据库(http://plants.ensembl.org/Triticum_aestivum/Info/Index)进行比对,得到3条高度相似分别定位于2A、2B、2D短臂上的序列,其对应的序列号分别为TRIAE_CS42_2AS_TGACv1_112825_AA0345780.1、TRIAE_CS42_2BS_TGACv1_147037_AA0476980.1、TRIAE_CS42_2DS_TGACv1_177302_AA0572700.1,其中克隆的TaCHS基因与位于2A染色体的TaCHS拷贝相似度最高,因此,将克隆的TaCHS基因命名为TaCHS-A1a。
对TaCHS-A1a基因进行生物信息学分析,其DNA全长1 283 bp,由2个外显子和1个内含子组成,CDS序列全长1 185 bp,编码394个氨基酸,其蛋白质分子质量为43.15 ku,理论等电点为6.43,分子式为C1907H3048N526O563S25,脂肪族氨基酸指数为87.39,负电荷的氨基酸残基数(Asp+Glu)为47个;正电荷的氨基酸残基数(Arg+Lys)为44个。平均亲水性值(GRAVY)为-0.088,不稳定系数为39.22,其属于疏水稳定蛋白。预测的TaCHS蛋白位于细胞质中,没有分泌通路信号肽,属于非分泌蛋白。TaCHS氨基酸序列不存在跨膜结构域(图1-A),由此推测,TaCHS蛋白是在细胞质基质中合成的,不经过跨膜转运,在细胞质中催化合成类黄酮类物质[12]。TaCHS蛋白在N端和C端都存在1个查尔酮和二苯乙烯合成酶的结构域,属于同源超家族,为Ⅲ型聚酮合成酶。TaCHS蛋白的二级结构(图1-B)是由α-螺旋、延伸链、β-转角和无规则卷曲4种结构模式组成。其中,α-螺旋178处,占45.18%;延伸链60处,占15.23%;β-转角23处,占5.84%;无规则卷曲133处,占33.76%。该蛋白质的3D模型(图1-C)中有388个氨基酸残基,覆盖98%的氨基酸序列,结构预测置信度达到100%。
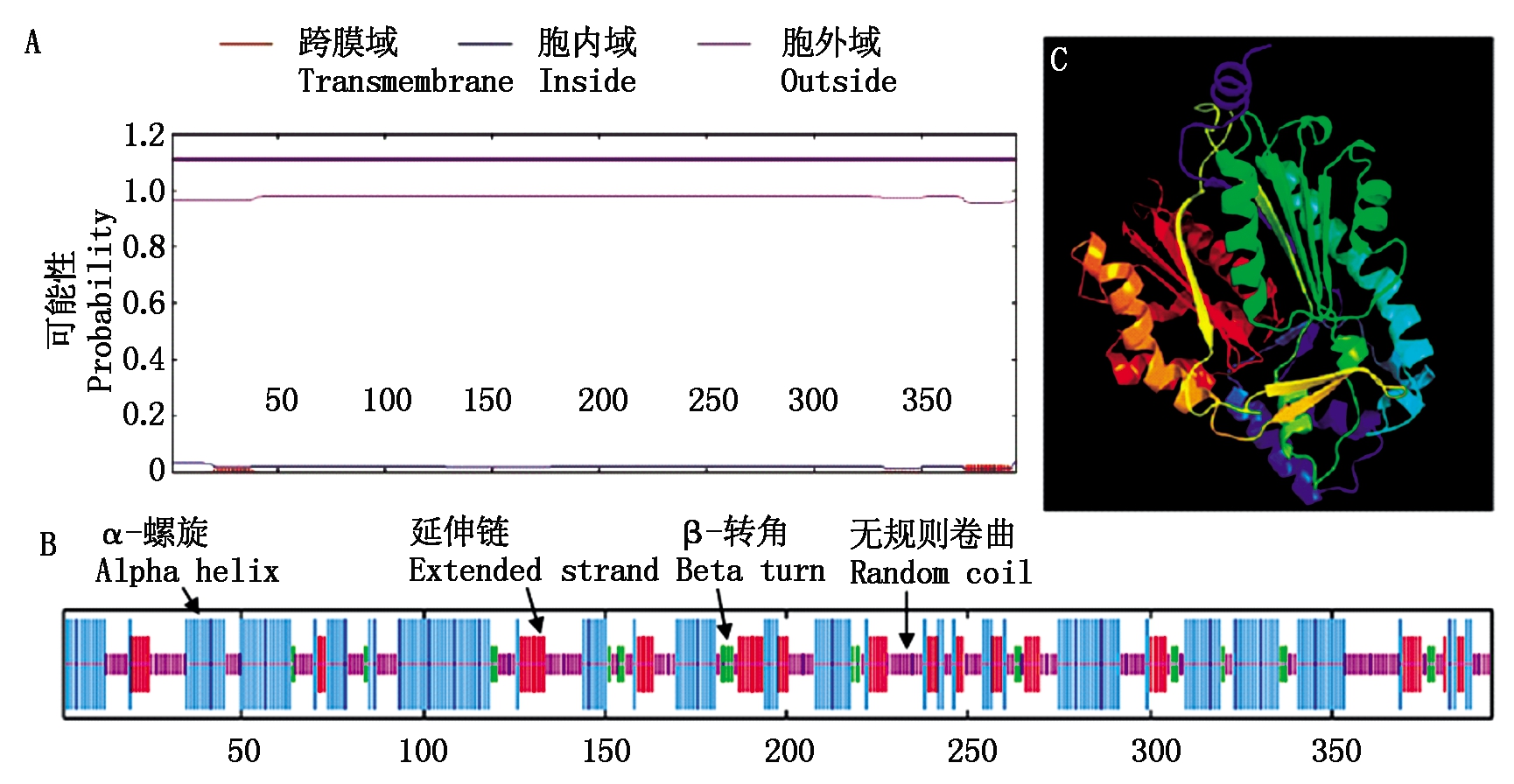
图1 TaCHS蛋白的跨膜结构域(A)、二级(B)和三级(C)结构预测
Fig.1 Transmembrane domain (A), secondary (B) and tertiary (C) structure prediction of the TaCHS protein
2.2 TaCHS基因结构和进化分析
以TaCHS编码蛋白质序列为探针,在Ensembl Plants(http://plants.ensembl.org/index.html)搜索不同物种的编码序列,得到12个物种的CDS序列(表2)。然后利用这些CDS序列在Mega 5.0中构建基于Neigbor-joining的系统发育树,结果显示,12条CDS序列明显聚类为3类,其中小麦的CHS基因与二穗短柄草的亲缘关系最近,属于第一类,与玉米亲缘关系最远,小麦、水稻、玉米等12个物种的CHS基因均由1个内含子和2个外显子组成,CDS序列相似度为78.43%,TaCHS与BdCHS相似度最高,达到81.90%(图2)。
表2 不同物种CHS基因的信息
Tab.2 Information on CHS genes in different species

物种Species 基因ID号Gene ID(Ensembl Plants)琴叶拟南芥Arabidopsis lyratafgenesh2_kg.6_1353_AT5G13930.1甘蓝Brassica oleraceaBo9g166290大豆Glycine maxGLYMA_08G110300水稻Oryza sativaOs11g0530600马铃薯Solanum tuberosumPGSC0003DMG400029620小麦Triticum aestivumTraesCS2A02G025700玉米Zea maysZm00001d032662拟南芥Arabidopsis thalianaAT5G13930高粱Sorghum bicolorSORBI_3005G136800二穗短柄草Brachypodium dis-tachyonBRADI_4g17230v3短花药野生稻Oryza brachyanthaOB11G22140油菜Brassica napusGSBRNA2T00140359001
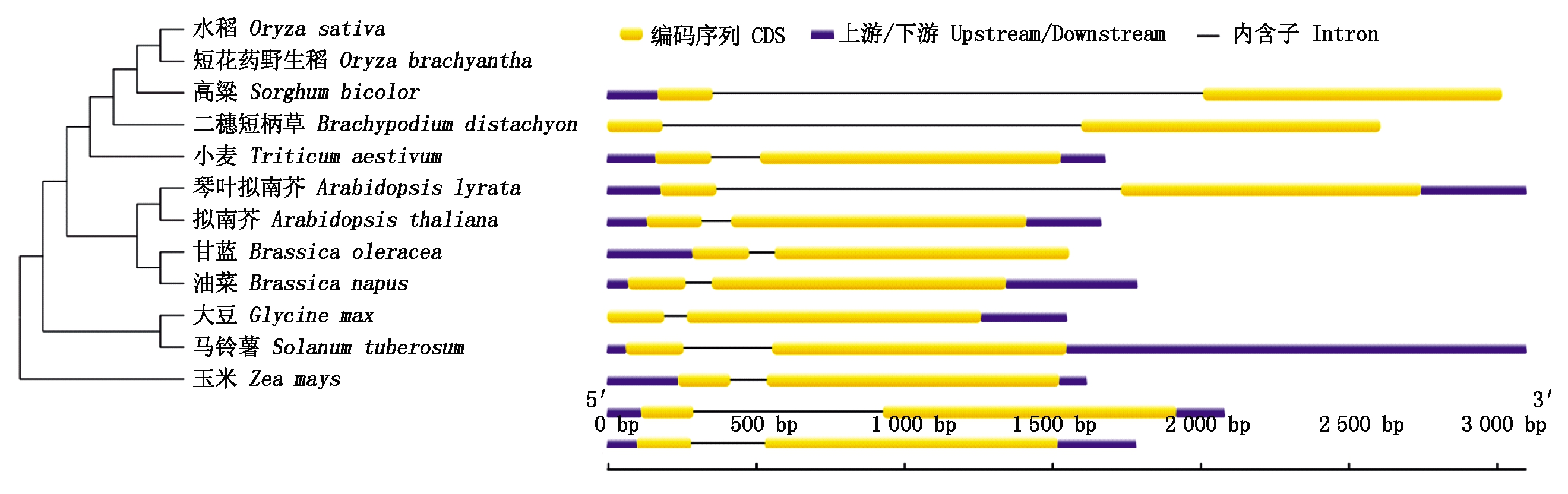
图2 不同来源的CHS基因进化树和基因结构
Fig.2 CHS genetic tree and genetic structure from different sources
2.3 TaCHS基因的表达模式分析
对TaCHS基因的组织表达定量分析结果表明,TaCHS在小麦的根、茎、叶和籽粒中均有所表达,尤其在茎中的表达量最高,显著高于根、叶和籽粒中的表达量,是根中表达量的3倍、叶片中的11倍、籽粒中的16倍(图3-A)。
在灌浆期,TaCHS基因的表达水平在扬麦10号和周麦98165的籽粒中均呈现出先升高后下降再升高的特点,在花后14 d达到第一个峰值;且在整个灌浆期,扬麦10号籽粒中TaCHS的表达量均高于周麦98165,这表明扬麦10号籽粒中可能有较高的类黄酮积累(图3-B)。
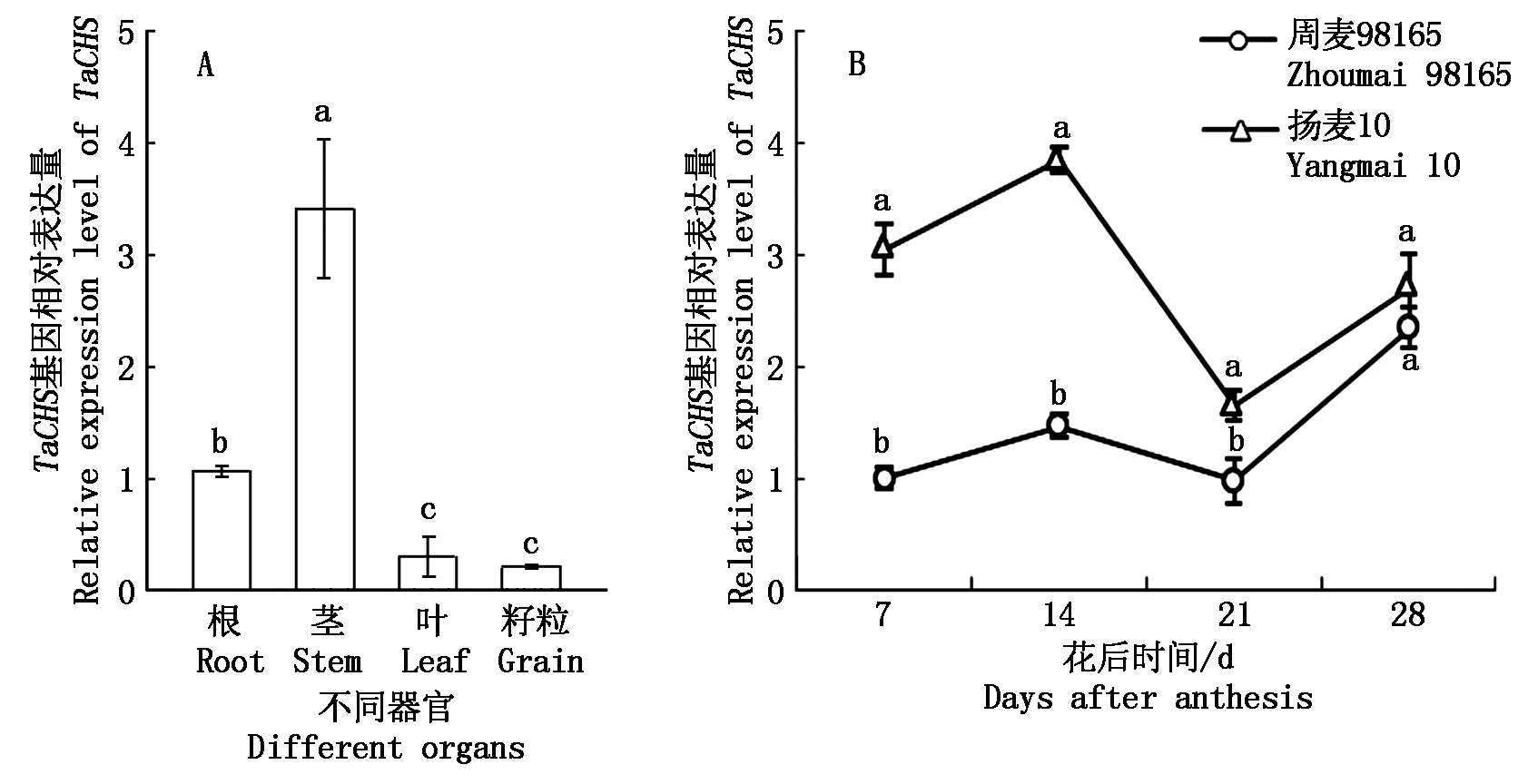
不同字母(a, b, c)表示差异显著(P< 0.05)。
Different letters (a, b, c)indicate significant difference(P< 0.05).
图3 TaCHS基因在小麦中的时空表达模式
Fig.3 Temporal and spatial expression pattern of TaCHS gene in wheat
2.4 TaCHS基因等位变异分析
M.DL2000 DNA Marker;1-6.周麦98165、扬麦10、许科316、郑麦366、大白芒、矮抗58 TaCHS基因的PCR扩增。
M.DL2000 DNA Marker; 1-6.PCR amplification of TaCHS gene in Zhoumai 98165, Yangmai 10, Xuke 316, Zhengmai 366, Dabaimang, Aikang 58.
图4 不同小麦品种TaCHS基因PCR扩增结果
Fig.4 PCR products of TaCHS gene in different wheat varieties
对119份小麦品种的DNA样品进行扩增,经1.5%琼脂糖凝胶电泳检测(图4)后,将有明亮目的条带的样品测序。
通过序列比对分析发现,不同小麦品种TaCHS基因在56个位点处存在单核苷酸突变,并在内含子区域存在小片段的Indel,但其编码的氨基酸序列基本一致,仅在6个位点处存在差异(图5)。在119份品种中共鉴定到4种TaCHS基因类型,分别命名为TaCHS-A1a(代表品种:中国春)、TaCHS-A1b、TaCHS-A1c、TaCHS-A1d,其分布频率为85.72%,7.56%,3.36%,3.36%(表3)。4种TaCHS基因类型的DNA序列相似度为97.81%,氨基酸序列相似度为99.43%。TaCHS-A1a与TaCHS-A1b间仅存在1个G/A有义突变,导致1个天冬氨酸(Asp)突变为天冬酰胺(Asn)。TaCHS-A1a与TaCHS-A1c间存在48个SNP, 且在内含子上存在1个5 bp的Indel,其中有4个有义突变C/T、A/G、G/C和G/A,分别位于790,1 159,1 183,1 272 bp处。TaCHS-A1a与TaCHS-A1d间存在51个SNP, 且在内含子区域存在1个4 bp的Indel,其中有4个有义突变G/C、A/G、G/C和G/A,分别位于171,1 159,1 183,1 272 bp处(图6)。在4种TaCHS基因型中,TaCHS-A1a与TaCHS-A1b的DNA序列相似度最高,为99.92%;TaCHS-A1c与TaCHS-A1d的DNA序列相似度最高,为99.15%。

图5 不同TaCHS基因等位变异编码的氨基酸序列比较
Fig.5 Comparison of allelic amino acid sequences of TaCHS gene
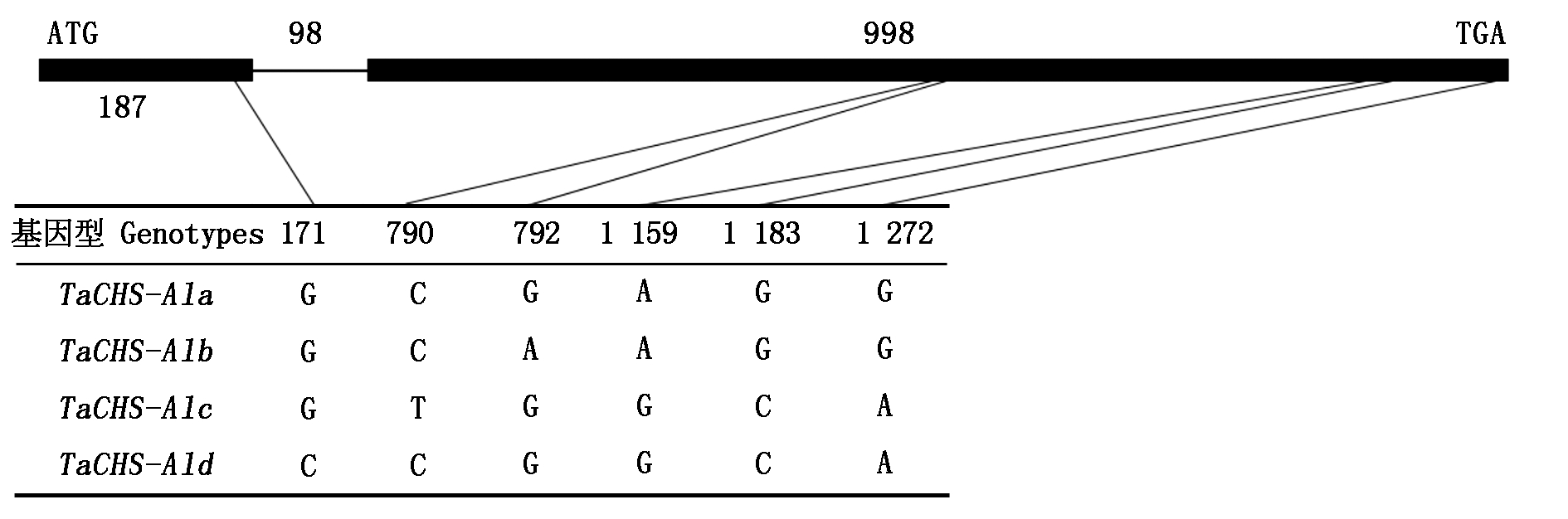
图6 TaCHS基因等位变异结构
Fig.6 Structure of allele variation in TaCHS gene
表3 TaCHS基因型分布
Tab.3 TaCHS genotype distribution

基因型Genotypes品种数量/个Number of varieties分布频率/%Distribution frequencyTaCHS-A1a10285.72TaCHS-A1b97.56TaCHS-A1c43.36TaCHS-A1d43.36
3 结论与讨论
CHS基因参与多种代谢调节活动,其主要功能是影响花色和育性,并在植物逆境胁迫中发挥作用[12-14]。CHS基因在黄酮代谢途径中调控着黄酮类成分的生物合成与积累,同时CHS的表达受各种外界环境的影响,植物在应激条件下,CHS基因表达量升高,促进黄酮类化合物积累[10]。对小麦TaCHS组织表达定量分析结果表明,TaCHS基因在茎中的表达水平最高,而在叶片中的表达量较低,这与报道的人参、苦荞和膜荚黄芪中CHS基因在叶片中的表达量高于茎中有一定差异[15-17]。这种差异的原因可能主要来源于不同物种的差异;另外,种植环境也对类黄酮合成酶基因的表达有显著的影响。前期的研究结果表明,叶片中TaCHS基因的表达量与小麦幼苗的抗旱性有显著相关性[18],因此,茎鞘中较高的TaCHS表达量是否与植株抗逆性有关还有待进一步的探讨。
黄酮类化合物被证实具有预防多种疾病的功能,因此,提高作物籽粒中黄酮类化合物含量对人体健康具有重要的意义[19-21]。灌浆期籽粒中TaCHS基因表达水平在花后14 d达到第一个峰值,花后21 d表达量急剧下降,这与Yang等[22]对CHS基因在蓝粒小麦中的定量分析结果基本一致。在灌浆期扬麦10号(红麦)的TaCHS基因表达量均高于周麦98165(白麦),这可能一定程度上有助于籽粒中积累较高的类黄酮化合物,提高其抗氧化性。研究表明,红粒小麦相对于白粒小麦具有较高的抗氧化性[23]。不同品种TaCHS基因在籽粒中的表达模式可能与籽粒中类黄酮类物质积累有关,因此,关于TaCHS基因表达量与籽粒类黄酮含量的积累及其抗氧化性的关系有待进一步的探讨。
通过对黄淮麦区119份小麦品种TaCHS基因序列分析,共鉴定到4种TaCHS基因类型, 85.71%的小麦品种的TaCHS基因序列与中国春的序列完全一致,表明TaCHS-A1a类型是目前育成品种的主要变异类型。TaCHS的序列较为保守,但依然在少数品种中存在等位变异,不仅在内含子区域存在片段的缺失,同时存在编码氨基酸差异的有义突变,明确这些变异尤其是位于结构域中的碱基差异与籽粒的类黄酮含量以及抗氧化性的关系,以及若能基于这些位点开发与类黄酮积累量高低相关的功能标记将对育种工作提供重要的参考,有待于下一步的研究。
[1] 薛保国,谢丽华,杨丽荣,孙润红,全鑫,杨艳艳,孙虎,雷振生,赵献林.小麦查尔酮合成酶基因及其启动子序列的克隆与分析[J].植物病理学报,2017,47(1):50-60. doi:10.13926/j.cnki.apps.000048.
Xue B G, Xie L H, Yang L R, Sun R H, Quan X, Yang Y Y, Sun H, Lei Z S, Zhao X L. Cloning and functional analysis of TaCHS and its promoter in wheat[J].Acta Phytopathologica Sinica, 2017, 47(1): 50-60.
[2] Han Y Y, Ming F, Wang J W, Guo B. Nested inverse polymerase chain reactions: an effective method for cloning of full-length sequences of chalcone synthase[J]. Plant Molecular Biology Reporter, 2007, 25(1-2): 63-69.doi:10.1007/s11105-007-0010-7.
[3] Heller W, Hahlbrock K. Highly purified ″flavanone synthase″ from parsley catalyzes the formation of naringenin chalcone[J]. Arch Biochem Biophys, 1980, 200(2):617-619. doi:10.1016/0003-9861(80)90395-1.
[4] Reimold U, Kröger M, Kreuzaler F, Hahlbrock K. Coding and 3′non-coding nucleotide sequence of chalcone synthase mRNA and assignment of amino acid sequence of the enzyme[J]. The EMBO Journal, 1983, 2(10):1801-1805. doi:10.1002/j.1460-2075.1983.tb01661.x.
[5] Trojan V, Musilová M, Vyhnánek T, Klejdus B, Hanácˇek P, Havel L. Chalcone synthase expression and pigments deposition in wheat with purple and blue colored caryopsis[J]. Journal of Cereal Science, 2014, 59(1): 48-55.doi:10.1016/j.jcs.2013.10.008.
[6] Mo Y, Nagel C, Taylor L P. Biochemical complementation of chalcone synthase mutants defines a role for flavonols in functional pollen[J]. Proceedings of the National Academy of Sciences, 1992, 89(15): 7213-7217. doi:10.1073/pnas.89.15.7213.
[7] Jiang C G, Schommer C K, Kim S Y, Suh D Y. Cloning and characterization of chalcone synthase from the moss,Physcomitrella patens[J]. Phytochemistry, 2006, 67(23):2531-2540. doi: 10.1016/j.phytochem.2006.09.030.
[8] Yang J, Huang J X, Gu H Y, Zhong Y, Yang Z H. Duplication and adaptive evolution of the chalcone synthase genes of Dendranthema (Asteraceae)[J]. Molecular Biology and Evolution, 2002, 19(10):1752-1759. doi:10.1093/oxfordjournals.molbev.a003997.
[9] 王凌云,宋小清,王晓明,范光宇,赵治海,赵艳,刘颖慧.不同品种谷子醇提物抗氧化活性比较[J].河南农业科学,2017,46(2):28-32.doi:10.15933/j.cnki.1004-3268.2017.02.006.
Wang L Y, Song X Q, Wang X M, Fan G Y, Zhao Z H, Zhao Y, Liu Y H.Comparative study on antioxidant activity of alcohol extracts from different millet cultivars[J]. Journal of Henan Agricultural Sciences, 2017,46(2):28-32.
[10] Dhiman A, Nanda A, Ahmad S. A quest for staunch effects of flavonoids: utopian protection against hepatic ailments[J]. Arabian Journal of Chemistry, 2012, 12(S2):1813-1823.doi: 10.1016/j.arabjc.2012.05.001.
[11] 张珊,郭启平,刘媛媛,党仁美,闻珊珊.小麦水通道蛋白TaTIP1-4DL基因的克隆及表达分析[J].华北农学报,2019,34(5):1-7. doi:10.7668/hbnxb. 201751747.
Zhang S, Guo Q P, Liu Y Y, Dang R M, Wen S S. Cloning and expression analysis of aquaporin TaTIP1-4DL gene in wheat[J]. Acta Agriculturae Boreali-Sinica, 2019, 34(5):1-7.
[12] 徐靖,朱家红,王效宁,韩义胜,唐力琼,朱红林.甘薯查尔酮合成酶基因IbCHS1的克隆和表达分析[J].分子植物育种,2018,16(6):1752-1757. doi:10.13271/j.mpb.016.001752.
Xu J, Zhu J H, Wang X N, Han Y S, Tang L Q, Zhu H L. Cloning and expression analysis of chalcone synthase gene IbCHS1 in Ipomoea batatas[J]. Molecular Plant Breeding, 2018, 16(6):1752-1757.
[13] 李苗,石磊,李国旗.植物查尔酮合成酶超基因家族组成及分子进化[J].分子植物育种,2016,14(6):1421-1429. doi:10.13271/j.mpb.014.001421.
Li M, Shi L, Li G Q. Molecular evolution and composition of the chalcone synthase (CHS) gene family in plant[J]. Molecular Plant Breeding, 2016,14(6):1421-1429.
[14] 赵志常,陈业渊,高爱平,黄建峰,党志国,罗睿雄.芒果CHS基因的克隆及其表达分析[J].华北农学报,2015,30(S1):200-203.doi:10.7668/hbnxb.2015. S1.036.
Zhao Z C, Chen Y Y, Gao A P, Huang J F, Dang Z G, Luo R X. Cloning and sequence analysis of CHS gene in mango[J]. Acta Agriculturae Boreali-Sinica, 2015, 30(S1):200-203.
[15] 张变玲,黄雪梅,刘心怡,谢彪,黄合庆,张儒.人参查尔酮合成酶基因PgCHS1的克隆与表达分析[J].中国细胞生物学学报,2018,40(12):2010-2017.doi:10.11844/cjcb.2018.12.0246.
Zhang B L, Huang X M, Liu X Y, Xie B, Huang H Q, Zhang R.Cloning and expression analysis of chalcone synthase gene PgCHS1 in Panax ginseng[J]. Chinese Journal of Cell Biology, 2018, 40(12):2010-2017.
[16] 郑涵予,袁元,金河延,于洋,全雪丽,吴松权.膜荚黄芪查尔酮合成酶基因的克隆与表达分析[J].延边大学农学学报,2019,41(2):9-14. doi:10.13478/j.cnki.jasyu.2019.02.002.
Zheng H Y, Yuan Y, Jin H Y, Yu Y, Quan X L, Wu S Q. Cloning and expression analysis of CHS gene in Astragalus membranaceus[J]. Agricultural Science Journal of Yanbian University, 2019, 41(2):9-14.
[17] 吴琦,李成磊,陈惠,邵继荣,赵海霞,苟琳.苦荞查尔酮合酶基因CHS的结构及花期不同组织表达量分析[J].中国生物化学与分子生物学报,2010,26(12):1151-1160. doi:10.13865/j.cnki.cjbmb.2010.12.016.
Wu Q, Li C L, Chen H, Shao J R, Zhao H X, Gou L. Molecular structure characterization of Fagopyrum tataricum chalcone synthase and its tissue-specific expression during florescence[J].Chinese Journal of Biochemistry and Molecular Biology, 2010, 26(12):1151-1160.
[18] Ma D Y, Sun D X, Wang C Y, Li Y G, Guo T C. Expression of flavonoid biosynthesis genes and accumulation of flavonoid in wheat leaves in response to drought stress[J].Plant Physiology and Biochemistry, 2014, 80:60-66. doi:10.1016/j.plaphy.2014.03.024.
[19] 张怀民,杨虹,郑海洲.天然黄酮类化合物防治心脑血管疾病的研究进展[J].中国新药与临床杂志,2016,35(10):704-708. doi:10.14109/j.cnki.xyylc.2016.10.005.
Zhang H M, Yang H, Zheng H Z. Research progress of natural flavonoids in prevention and treatment of cardiovascular and cerebrovascular diseases[J]. Chinese Journal of New Drugs and Clinical Remedies, 2016, 35(10):704-708.
[20] 曹秦,吴辉,张蓓蓓,吴晓俊.黄酮类化合物在防治神经退行性疾病中作用的研究进展[J].中国药理学与毒理学杂志,2015,29(3):457-463. doi:10.3867/j.issn.1000-3002.2015.03.018.
Cao Q, Wu H, Zhang B B, Wu X J. Progress in prevention and treatment of neurodegenerative diseases with flavonoids[J]. Chinese Journal of Pharmacology and Toxicology, 2015, 29(3):457-463.
[21] 曹剑锋,芦静波,滕树学,任朝辉,陈慧琴.密蒙花总黄酮的抗氧化及免疫调节作用[J].河南农业科学,2016,45(9):130-134. doi:10.15933/j.cnki.1004-3268.2016.09.026.
Cao J F, Lu J B, Teng S X, Ren C H, Chen H Q.Immune regulation function and antioxidant capacities of total flavonoids from Buddleja officinalis Maxim DC.[J].Journal of Henan Agricultural Sciences, 2016, 45(9):130-134.
[22] Yang G H, Li B, Gao J W, Liu J Z, Zhao X Q, Zheng Q, Tong Y P, Li Z S. Cloning and expression of two chalcone synthase and a flavonoid 3′5′-hydroxylase 3′-end cDNAs from developing seeds of blue-grained wheat involved in anthocyanin biosynthetic pathway[J]. Acta Botanica Sinica, 2004, 46(5):588-594.doi:10.3321/j.issn:1672-9072.2004.05.012.
[23] Ma D Y, Li Y G, Zhang J, Wang C Y, Qin H X, Ding H N, Xie Y X, Guo T C. Accumulation of phenolic compounds and expression profiles of phenolic acid biosynthesis-related genes in developing grains of white, purple, and red wheat[J]. Frontiers in Plant Science, 2016,7(528):1-10. doi:10.3389/fpls.2016.00528.
