光合作用是决定作物产量的最重要因素之一,作物中大约90%的干物质直接源于光合作用[1],因此,光合效率的高低直接关系到作物产量的高低。与C3作物相比,C4作物具有光合速率高,CO2补偿点低、光呼吸弱的优点,尤其是在强光、高温、干旱等不利条件下,C4植物更具有明显的生长优势[2]。丙酮酸磷酸双激酶(Pyruvate orthophos phate dikinase,PPDK)是C4植物光合途径中的一个关键限速酶[3-4],其催化固定CO2最初受体磷酸烯醇式丙酮酸(PEP)再生。PPDK主要有叶绿体型和胞质型2种类型,其中,叶绿体型PPDK催化的是一个可逆反应,在叶绿体中该反应主要向着形成PEP的方向进行;胞质型PPDK则在PEP传递、CO2呼吸、氨基酸互换及淀粉合成等过程中具有重要作用[5-6]。在非光合作用中,PPDK在保持植物体内的pH稳态、为生物合成和抗氧化系统提供氨基酸与其他化合物方面发挥积极作用[7]。PPDK在C4植物叶绿体基质中大量存在,主要在叶肉细胞的叶绿体中行使功能,而在维管束鞘细胞、根、茎等其他组织中表达量极低。在C3植物中,PPDK主要存在于未成熟种子、根、胚芽鞘和叶片中[8-9],但由于其中PPDK的表达量很低,对其功能还知之甚少[10-12]。前人关于C4植物中PPDK在非生物胁迫下的功能进行了一些研究,玉米体内的叶绿体型PPDK在温度低于12 ℃时会失去活性[13];苇子草PPDK在低温(14 ℃)下作为限速酶对维持其光合效率起主要作用[7]。另外,PPDK还具有一定的抗盐能力[14],在盐渍处理下玉米苗期叶肉细胞中PPDK的活性显著增强[15]。在阳光UV-B辐射及补充UV-B辐射的条件下,玉米PPDK表达量上升,但是在温室中补充UV-B,其表达量下调[7]。胞质型PPDK与胁迫的关系也有报道,但主要集中在低氧、缺水、高温及生物胁迫。此外,PPDK在基因结构[16-18]、进化关系[19]、功能[20-23]、调控机制[10,24-25]、基因工程[18,26-28]等方面也有一定的研究。
谷子(Setaria italica (L.) Beauv)是我国北方干旱和半干旱地区重要的粮食作物,具有抗旱、耐瘠、适应性广等特性。谷子基因组测序已完成并公布,为开展谷子抗逆分子生物学研究提供了极为便利的条件[29-30],但是,迄今为止,PPDK与非生物逆境胁迫的相关研究在C4作物谷子中还鲜有报道。
本研究为探索PPDK基因在谷子逆境应答中的功能,对SiPPDK2蛋白特征、氨基酸序列、功能、亚细胞定位、启动子区域顺势应答元件等参数进行分析和预测,进一步检测PPDK基因在苗期逆境胁迫下的动态表达模式及在拔节、抽穗、灌浆 3 个生育时期干旱胁迫和不同光照处理下的表达情况。研究结果可为进一步分析PPDK基因在谷子逆境应答信号途径中的功能和机制提供数据支持。
1 材料和方法
1.1 试验材料
选用谷子品种豫谷1号为试验材料,种子由山西省农业科学院谷子研究所谷子基因课题提供。
待植株长至三叶期时进行干旱(20% PEG6000)、盐 (250 mmol/L NaCl)、ABA(100 μmol/L)和低温(4 ℃)处理,分别处理0,1,3,6,12,24 h,整株取样,取样后液氮速冻,于-80 ℃冰箱中保存备用,分析非生物逆境胁迫下谷子苗期SiPPDK2的表达量差异。
旱棚设对照(正常浇水)和干旱(浇3次关键水)2个处理;旱棚光照试验设2个处理,光照Ⅰ(用黑色遮阳网遮挡1层)和光照Ⅱ(用黑色遮阳网遮挡2层),旱棚其他管理措施同大田。叶片取样分别在拔节期、抽穗期、灌浆期进行,设3次生物学重复。取样后液氮速冻,于-80 ℃冰箱中保存备用,分析不同生育期干旱胁迫及不同光照强度下的SiPPDK2的表达量差异。
1.2 试验方法
1.2.1 基因的结构与序列分析 氨基酸数目、相对分子量以及等电点等理化性质分析用EXPASY软件(http://web.expasy.org/protparam/)[31]进行;启动子顺式元件分析利用在线软件plantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)完成;亚细胞定位预测用Psort在线工具进行;基因序列比对使用ClustalX 1.83软件[32]完成;不同物种NJ系统进化树采用Mega 6.0软件邻接法构建[33]。
1.2.2 总RNA提取和实时荧光定量分析 植物RNA提取试剂盒购自生工生物工程(上海)有限公司,总RNA提取按照试剂盒说明书进行。SiPPDK2和内参基因SiGAPB特异性引物用PrimerPrimer 5.0软件设计。SiPPDK2上游引物:5′-GTGCTCAACCT GGGACTCAA-3′,下游引物:5′-AGGCAGTCAGGT CATTGTCG-3′;SiGAPB上游引物:5′-CAGTGGACG CACAACAGGTAT-3′,下游引物:5′-AGCAAGGT CAAGACGGAGAAT-3′。样品总RNA反转录合成cDNA,均一化后作为实时定量PCR模板。反应体系为20 μL:10 μL荧光染料、7.2 μL无菌水、0.4 μL正向引物、0.4 μL反向引物、2 μL cDNA。反应程序为:95 ℃ 3 min;95 ℃ 7 s,57 ℃ 10 s,72 ℃ 15 s,45个循环。每个处理3次重复,相对定量采用2-ΔΔCt 法计算[34]。
1.3 数据分析
采用Microsoft Excel 2007作图,数据统计分析采用SPSS 19.0软件进行。
2 结果与分析
2.1 SiPPDK2基因的生物信息学分析
2.1.1 SiPPDK2基因的鉴定 序列比对结果发现,SiPPDK2位于谷子3号染色体21 259 472-21 273 920区域。功能域分析显示,SiPPDK2蛋白含有2个N端的磷酸烯醇式丙酮酸结构域,位于蛋白的91-363位置和375-428位置;含有1个磷酸烯醇式丙酮酸结构域,位于蛋白的495-575 位置;含1个C端的磷酸烯醇式丙酮酸结构域,位于蛋白的590-943位置,这些结构都属于PPDK家族基因的基本特征。综合说明SiPPDK2基因是谷子PPDK家族成员。
2.1.2 SiPPDK2蛋白特性预测与亚细胞定位分析 采用EXPASY软件对SiPPDK2的蛋白特性进行分析,预测结果(表1)显示,T01和T02编码相同的氨基酸,分子量为1 024.249 4 ku,等电点为5.38,分子式为C4496H7188N1264O1375S46,不稳定指数为36.69,脂肪系数为85.26,平均疏水性指数为-0.168;T03分子量为957.603 5 ku,等电点为5.03,分子式为C4216H6709N1161O1291S45,不稳定指数为33.67,脂肪系数为86.70,平均疏水性指数为-0.136。利用 GSDS 在线软件对SiPPDK2基因结构进行了分析,结果如图1所示,SiPPDK2基因含有18个内含子(图1)。另外,检索发现,SiPPDK2有3个转录本,T01和T02编码相同的氨基酸,区别在于T01的5′端非翻译区比 T02 多一段,T03编码882个氨基酸,在N端比T01和T02少了63个氨基酸。在后续分析时将SiPPDK2同一基因位点出现的3个可变剪切体视为同一基因,仅以原始转录本对应的序列作为该基因的分析对象。利用Cello软件对谷子SiPPDK2蛋白的亚细胞定位进行了分析,结果预测其位于叶绿体中。
表1 SiPPDK2 蛋白质的理化性质
Tab.1 Physicochemical property of SiPPDK2 protein

染色体Chromosome转录Transcription转录序列/bpTranscriptsequence 氨基酸数目Number ofamino acid分子量/kuMolecularweight等电点pI分子式Formula不稳定指数Instabilityindex脂肪系数Aliphaticindex平均疏水指数Grand averageof hydrophobicity3T01、T023 3853 2049451 024.249 45.38C4496H7188N1264O1375S4636.6985.26-0.168T034 955882957.603 55.03C4216H6709N1161O1291S4533.6786.70-0.136
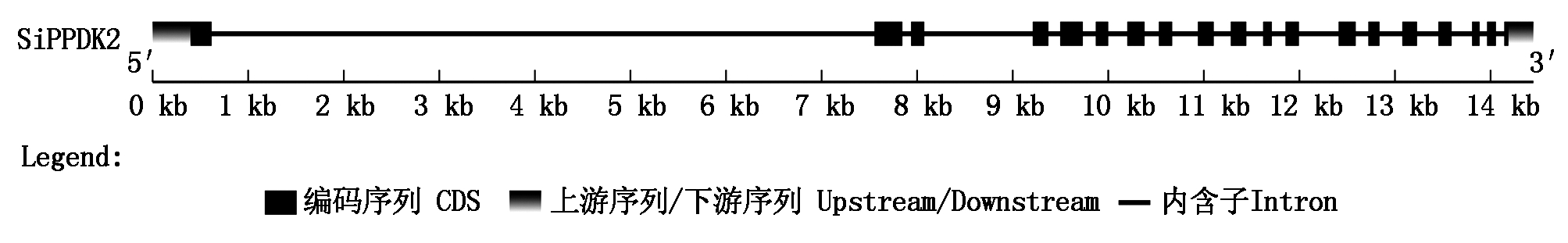
图1 SiPPDK2的基因结构
Fig.1 Gene structure of SiPPDK2 gene
2.2 PPDK序列比对及亲缘关系分析
通过NCBI数据库检索了不同物种PPDK氨基酸同源序列,包括玉米ZmPPDK1、ZmPPDK2、ZmPPDK3,高粱SbPPDK1、SbPPDK2、SbPPDK3,水稻OsPPDK1、OsPPDK2、OsPPDK3,小麦TaPPDK,拟南芥AtPPDK1、AtPPDK2、AtPPDK3,火炬松PtPPDK1、PtPPDK2、PtPPDK3,苔藓PpPPDK1、PpPPDK2、PpPPDK3。然后,对不同物种基因序列进行比对,构建了谷子SiPPDK2和其他物种PPDK蛋白成员的NJ进化树。氨基酸序列比对结果(图2)发现,SiPPDK2与玉米、高粱、水稻、拟南芥同源性非常高,相似性分别为94.15%,93.33%,91.85%和89.41%,而与其他物种(小麦、火炬松、苔藓)序列相似性则相对较低,分别仅为23.28%,9.72%和37.77%。
从图3可以看出,所有PPDK蛋白被分为三大类群,其中,类群Ⅰ中包含拟南芥AtPPDK1、AtPPDK2、AtPPDK3和小麦TaPPDK;类群Ⅱ中包含谷子SiPPDK2,玉米ZmPPDK1、ZmPPDK2、ZmPPDK3,高粱SbPPDK1、SbPPDK2、SbPPDK3和水稻OsPPDK1、OsPPDK2、OsPPDK3;类群Ⅲ中包含裸子植物中的火炬松PtPPDK1、PtPPDK2、PtPPDK3和苔藓PpPPDK1、PpPPDK2、PpPPDK3。在这些成员中,与谷子SiPPDK2的亲缘关系较近的是高粱、玉米和水稻的PPDK蛋白,所以推测这些 PPDK蛋白具有类似的生物功能;SiPPDK2与小麦、苔藓和火炬松的PPDK蛋白的亲缘关系较远,表明这些成员的生物功能可能与谷子SiPPDK2差异较大。

图中黑色背景氨基酸表示相同氨基酸残基;灰色背景氨基酸表示相似氨基酸残基(相似性≥60%)。
The black background amino acids represent the same amino acid residues; The gray background amino acids represent similar amino acid residues (≥60% similarity).
图2 SiPPDK2与其他已知PPDK氨基酸序列比对
Fig.2 Sequences alignment of SiPPDK2 and other known PPDKs
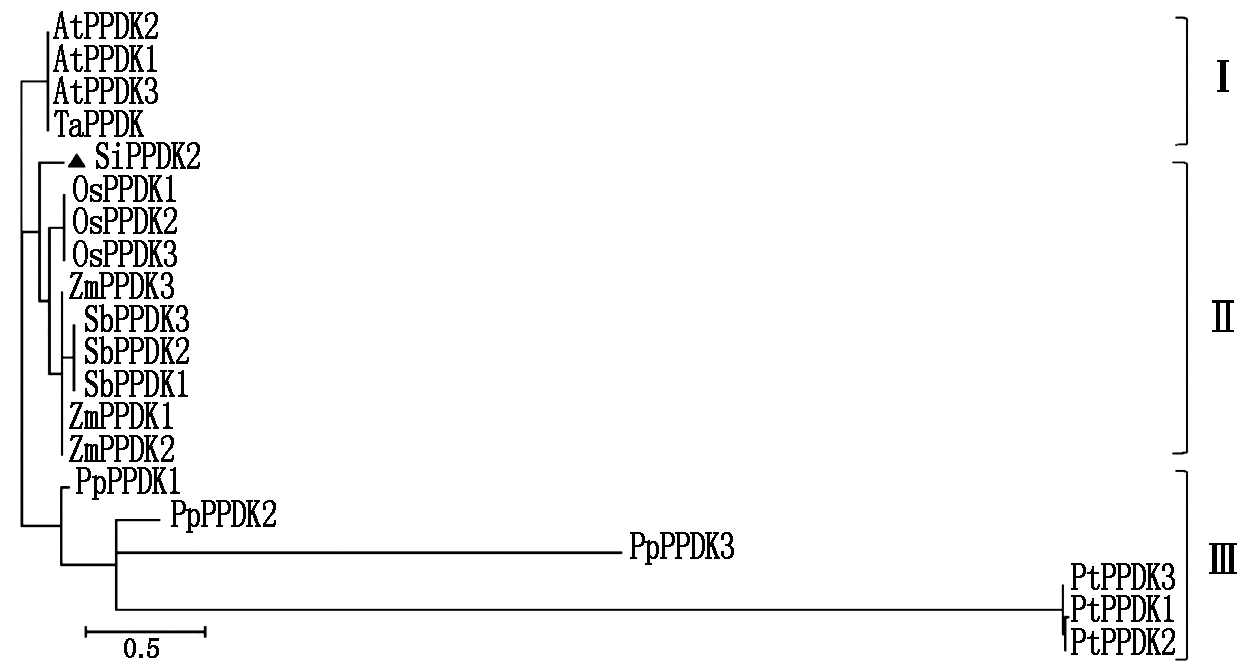
每个位点氨基酸替代值在底部用0.5刻度值标出。
Amino acid substitution values of each site were labeled with a 0.5 scale value at the bottom.
图3 不同物种PPDK 蛋白的进化关系(邻接法)
Fig.3 Phylogenetic relationships of PPDK proteins from different species (NJ method)
2.3 SiPPDK2基因的表达分析
2.3.1 苗期在非生物逆境胁迫下的表达分析 SiPPDK2基因苗期不同逆境胁迫下的表达分析(图4)表明,SiPPDK2 基因受不同逆境诱导,在苗期受ABA、低温、干旱和盐胁迫条件下,不同时间点呈现不同的表达模式。在低温胁迫时,该基因表达量受强烈诱导,随处理时间点增加,呈连续上升趋势,在24 h急剧上升达到最大,为对照的25.80倍,较对照差异达极显著水平;20% PEG胁迫时,该基因表达量在各处理时间点均极显著下降;ABA胁迫条件下,表达量呈现下降-上升-下降的趋势,在1 h和24 h显著或极显著下调表达,分别为对照的64%,52%,而3 h和6 h极显著上调表达,在6 h时表达量为对照的2.92倍;NaCl胁迫时,仅在6 h表达量稍有上调,为对照的1.37倍,但差异未达显著水平,在1,3,12,24 h的表达量均下调,分别为对照的41%,54%,99%,68%,其中1,3 h下调达极显著水平。综合表明,SiPPDK2参与了谷子苗期的非生物胁迫应答。
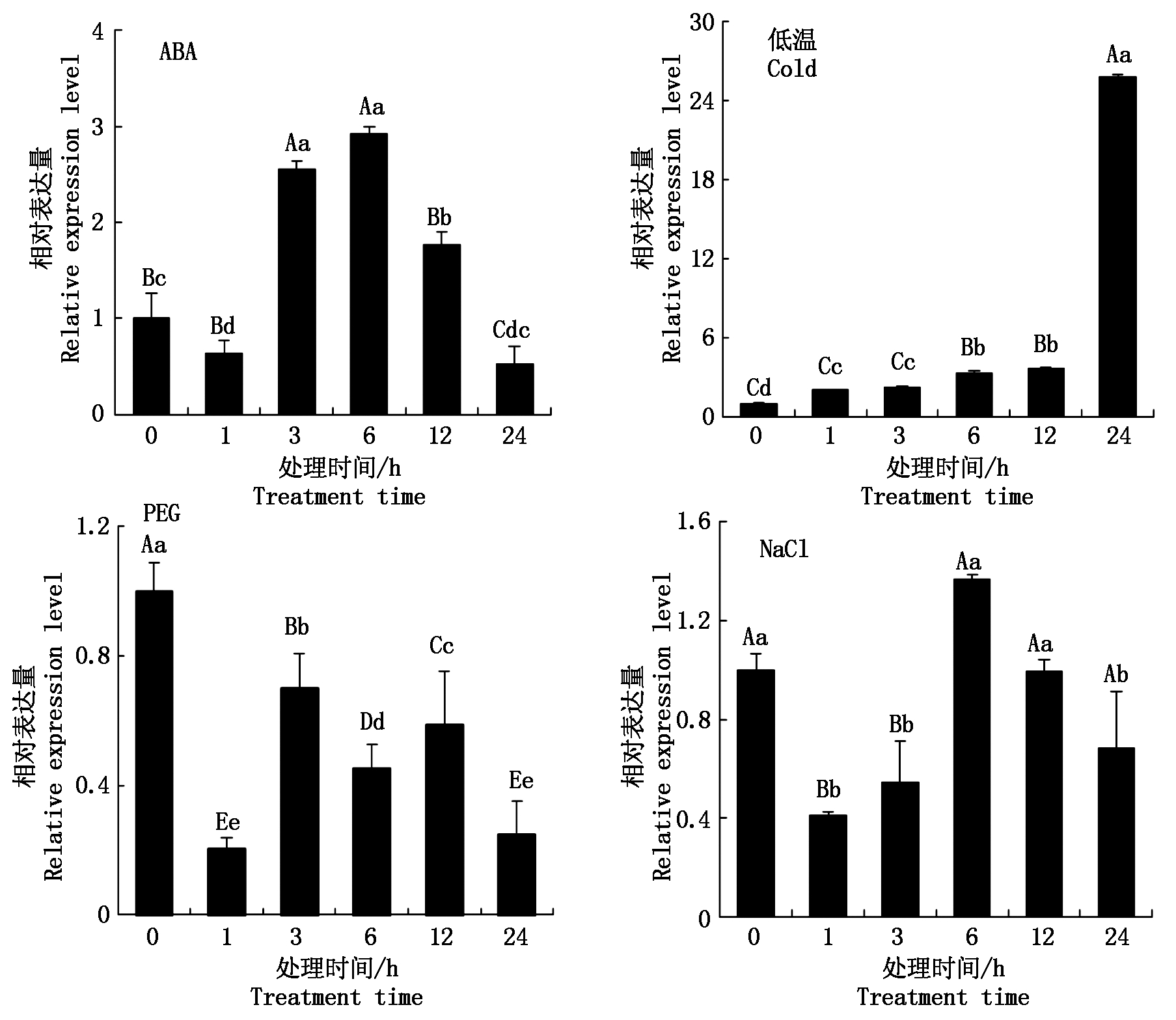
小写字母表示处理较对照(0 h)在0.05水平上差异显著;大写字母表示处理较对照(0 h)在0.01水平上差异显著。图5-6同。
Lowercase letters indicate significant difference at the level of 0.05 between the treatment and the control (0 h) ;Capital letters indicate significant difference at the level of 0.01 between the treatment and the control (0 h) . The same as Fig.5-6.
图4 非生物胁迫下 SiPPDK2 的相对表达量
Fig.4 Relative expression of SiPPDK2 under different abiotic stresses
2.3.2 不同生育期干旱胁迫及不同光照强度下的表达分析 SiPPDK2基因在谷子不同生育期干旱条件下的表达情况(图5)表明,正常条件下,SiPPDK2在抽穗期和灌浆期的表达量较拔节期均下调,分别为拔节期的81%和94%;干旱条件下,在抽穗期和灌浆期的表达量较拔节期均有极显著上调,分别为拔节期的6.03,4.58倍。由此可知,SiPPDK2在谷子不同生育时期干旱胁迫下其表达量均有明显增加,表明SiPPDK2基因参与了谷子不同生育时期对干旱胁迫的响应。SiPPDK2基因在谷子不同生育期干旱和光照强度条件下的表达情况(图6)表明,SiPPDK2在抽穗期和灌浆期干旱条件下的表达量较对照均有极显著上调,而拔节期较对照的表达量有所下降,为对照的86%,但差异未达显著水平;不同光照强度下,SiPPDK2在拔节期、灌浆期中等光照(光照Ⅰ)及抽穗期的中等光照(光照Ⅰ)和弱光照(光照Ⅱ)下的表达量趋于一致,较正常光照均有所下降,而在拔节期和灌浆期弱光照(光照Ⅱ)下的表达量均极显著上调,其中拔节期为正常光照的1.79倍,灌浆期为正常光照的2.14倍。由此可知,光照的强弱对SiPPDK2基因的表达具有极显著影响,表明SiPPDK2基因参与了对光照胁迫的响应。
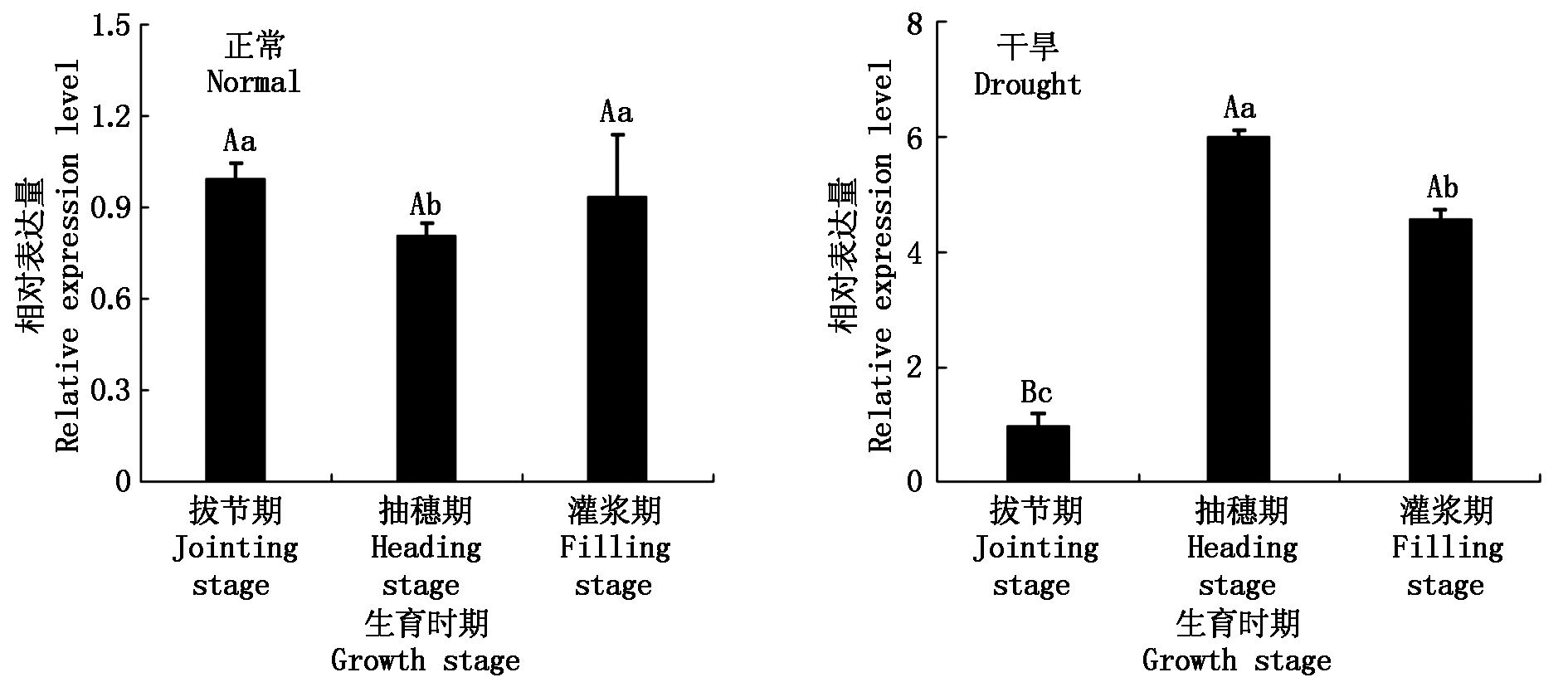
图5 正常和干旱条件下 SiPPDK2 在不同生育期的相对表达量
Fig.5 Relative expression of SiPPDK2 under normal and drought conditions during different growth tages
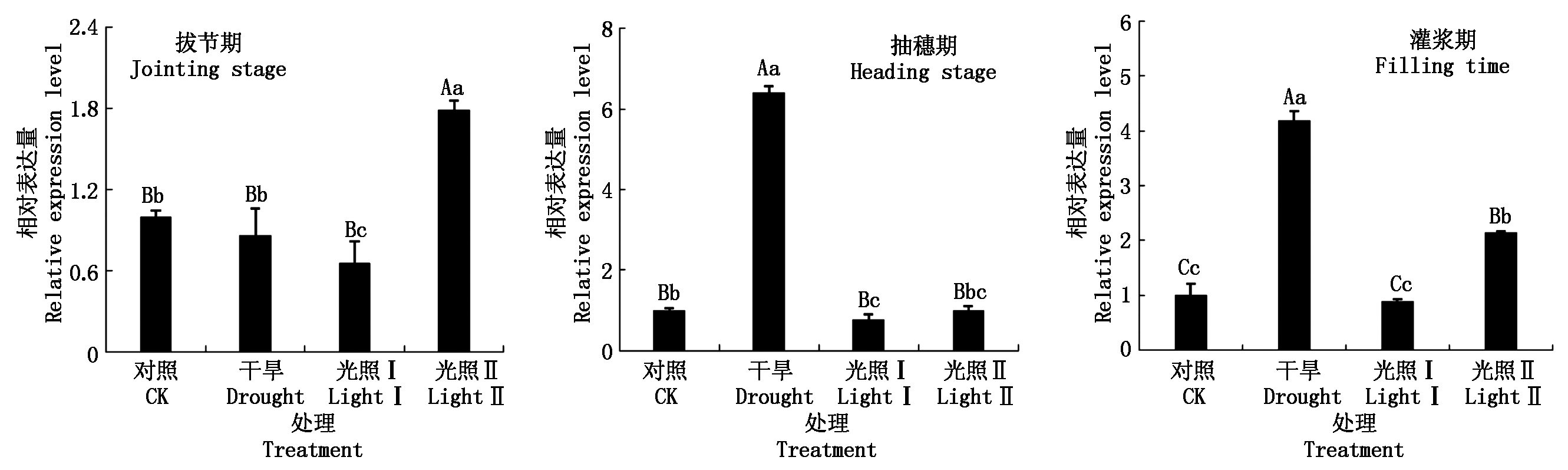
图6 干旱和光照条件下SiPPDK2的相对表达量
Fig.6 Relative expression of SiPPDK2 under light and drought conditions
为了进一步揭示SiPPDK2可能的生物功能,用plantCARE 在线软件对顺式作用元件进行了预测,结果(表2)显示,在SiPPDK2基因的启动子区域中包含了多个与逆境、激素和光应答相关的顺式作用元件,逆境(干旱、低温、防御和应激反应)应答元件有MBS、LTR和WUN-motif;植物激素类应答元件有生长素应答元件TGA-element、茉莉酸甲酯应答元件CGTCA-motif和TGACG-motif;光应答元件有I-box、SP1、GATA-motif。这些元件都暗示,SiPPDK2基因很可能参与了相应的信号途径。
表2 PlantCARE 预测 SiPPDK2启动子区域顺式作用元件
Tab.2 cis-acting regulatory elements in promoter of SiPPDK2 predicted by plant CARE

顺式元件cis-elements顺式元件预测Predicted cis-elements植物激素应答Auxin-respon-siveTGA-element、CGTCA-motif、TGACG-motif逆境应答Stress-responsiveMBS、LTR、WUN-motif光应答Light-responsiveI-box、SP1、GATA-motif其他应答Others responsiveCCAAT-box
3 结论与讨论
在自然界中,植物会遭受强光、极端温度、盐渍化、水分亏缺和大气干旱等各种环境因子的胁迫,其中,水分亏缺是影响干旱区植物生长发育和导致生理生化响应的主要因子和限制植物生长的关键因素[35]。初步研究表明,PPDK在植物逆境胁迫反应中具有重要作用[36-37]。本研究获得的SiPPDK2位于谷子3号染色体21 259 472-21 273 920区域,含有18 个内含子,具有3个转录本;具有磷酸烯醇式丙酮酸保守结构域,表明SiPPDK1基因是谷子PPDK家族成员;系统进化树表明,SiPPDK2蛋白与玉米、高粱、水稻有较近的亲缘关系,所以这些 PPDK蛋白在逆境应答或其他信号途径中有着相似的功能;亚细胞定位分析表明,SiPPDK2基因被定位在细胞的叶绿体中,这是植物光合作用的关键位置,揭示了它在植物光合作用信号途径中起比较重要的作用。
本研究中,SiPPDK2 基因在苗期对ABA、低温、干旱和盐胁迫均有响应,除在20% PEG胁迫时,SiPPDK2基因表达量在各处理时间点下调外,其他胁迫处理下都至少有1个时间点表现上调,其中以低温胁迫时,在24 h表达量最大。进一步试验结果发现,SiPPDK2基因在不同关键生育期干旱和光照胁迫下也有不同的响应。干旱条件下,在抽穗期和灌浆期表达量较对照均有极显著上调,以抽穗期时表达量最高;不同光照强度下,在拔节期和灌浆期弱光下表达量上调极显著。以上试验结果都表明,谷子SiPPDK2基因广泛参与了植物非生物逆境胁迫应答。前人也有相似的研究结果,脱落酸、聚乙二醇以及淹水显著地增加了水稻根中PPKD基因的表达;在干旱、低温、高盐以及甘露醇等缺水胁迫下,苗期水稻同样在根中诱导PPDK产生;在低氧胁迫(淹水培养或无氧环境培养)条件下,水稻幼苗中、幼苗根和胚芽鞘都可以诱导PPDK合成[38]。高温胁迫下,水稻籽粒中胞质型PPDK的转录水平下调[39]。上述研究均说明PPDK基因在植物逆境应答中起一定作用。顺式元件分析也表明,在SiPPDK2启动子区域含有ABA、低温、干旱和防御应激反应等顺式元件。
本研究从谷子全基因组中鉴定出1个PPDK基因家族成员,命名为SiPPDK2,其定位在谷子的3号染色体上,位于叶绿体,含有 18个内含子,有3个转录本,具有磷酸烯醇式丙酮酸高度保守结构域;SiPPDK2基因广泛参与了谷子苗期非生物逆境胁迫应答,在关键生育期光照和干旱逆境胁迫应答中起重要作用,而其中的分子机制还需进一步研究。
[1] 侯爱菊, 徐德昌. 植物髙光效基因工程育种[J]. 中国生物工程杂, 2005,25(9): 19-23. doi:10.3969/j.issn.1671-8135.2005.09.005.
Hou A J, Xu D C. Current advance of high photosynthetic efficiency breeding by gene engineering in plants[J]. China Biotechnology, 2005,25(9): 19-23.
[2] 腾胜, 钱前,黄大年. C4光合途径的分子生物学和基因工程硏究进展[J]. 农业生物技术学报,2001, 9(2): 198-201. doi:10.3969/j.issn.1674-7968.2001.02.025.
Teng S, Qian Q, Huang D N.Advances in molecular biology and genetic engineering of C4 photosynthesis pathway[J].Jounral of Agricultural Biotechnology,2001,9(2):198-201.
[3] Trevanion S J, Furbank R T, Ashton A R. NADP-Malate dehydrogenase in the C4 plant Flaveria bidentis (cosense suppression of activity in mesophyll and Bundle-sheath cells and consequences for photosynthesis)[J]. Plant Physiology, 1997, 113(4):1153-1165. doi:10.1104/pp.113.4.1153.
[4] Rees T A, Hill S A. Metabolic control analysis of plant metabolism[J]. Plant Cell and Environment, 1994, 17(5): 587-599. doi:10.1111/j.1365-3040.1994.tb00151.x.
[5] Chastain C J, Failing C J, Manandhar L, Zimmerman M A, Lakner M M, Nguyen T H T. Functional evolution of C4 pyruvate, orthophosphate dikinase[J]. Journal of Experimental Botany, 2011, 62(9): 3083-3091. doi:10.101093/jxb/err058.
[6] Meyer A O, Kelly G J, Latzko E. Pyruvate orthophosphate dikinase from the immature grains of cereal grasses[J]. Plant Physiology, 1982, 69(1):7-10. doi:10.1104/pp.69.1.7.
[7] Doubnerová V, Ryšlavá H. What can enzymes of C4 photosynthesis do for C3 plants under stress?[J]. Plant Science,2011, 180(4):575-583.doi:10.1016/j.plantsci.2010.12.005.
[8] Aoyagi K, Bassham J A. Pyruvate orthophosphate dikinase of C3 seeds and leaves as compared to the enzyme from maize[J]. Plant Physiology, 1984,75(2):387-392. doi:10.1104/pp.75.2.387.
[9] Parsley K, Hibberd J M. The Arabidopsis PPDK gene is transcribed from two promoters to produce differentially expressed transcripts responsible for cytosolic and plastidic proteins[J]. Plant Molecular Biology, 2006,62(3): 339-349. doi:10.1007/s11103-006-9023-0.
[10] Edwards G E, Nakamoto H, Burnell J N, Hatch M D. Pyruvate,pi dikinase and NADP-malate dehydrogenase in C4 photosynthesis: properties and mechanism of light/dark regulation[J]. Annual Review of Plant Physiology, 1985,36(1):255-286. doi:10.1146/annurev.pp.36.060185.001351.
[11] Chastain C J, Heck J W, Colquhoun T A, Voge D G, Gu X Y. Posttranslational regulation of pyruvate, orthophosphate dikinase in developing rice (Oryza sativa) seeds[J]. Planta,2006, 224(4): 924-934. doi:10.1007/s00425-006-0259-3.
[12] Burnell J N, Chastain C J. Cloning and expression of maize-leaf pyruvate, Pi dikinase regulatory protein gene[J]. Biochem Biophys Res Commun, 2006,345(2): 675-680. doi:10.1016/j.bbrc.2006.04.150.
[13] Ohta S,Ishida Y,Usami S.Expression of cold-tolerant pyruvate,orthophosphate dikinase cDNA,and heterotetramer formation in transgenic maize plants[J].Transgenic Research,2004,13(5): 475-485. doi:10.1007/s11248-004-1452-4.
[14] Alla M M N, Hassan N M. A possible role for C4 photosynthetic enzymes in tolerance of Zea mays to NaCl[J]. Protoplasma, 2012, 249(4):1109-1117. doi:10.1007/s00709-011-0356-4.
[15] Omoto E,Taniguchi M,Miyake H.Adaptation responses in C4 photosynthesis of maize under salinity[J].Plant Physiol,2012,169(5):469-477. doi:10.1016/j.jplph.2011.11.009.
[16] Matsuoka M. Structure, genetic mapping, and expression of the gene for pyruvate, orthophosphate dikinase from maize[J]. The Journal of Biological Chemistry, 1990,265(28):16772-16777.
[17] Matsuoka M. The gene for pyruvate,orthophosphate dikinase in C4 plants: structure, regulation and evolution[J]. Plant & Cell Physiology, 1995,36(6):937-943. doi:10.1093/oxfordjournals.pcp.a078864.
[18] Matsuok M, Ozeki Y, Yamamoto N, Hirano H, Kano-Murakumi Y, Tanaka Y. Primary structure of maize pyruvate, orthophosphate dikinase as deduced from cDNA sequence[J]. The Journal of Biological Chemistry, 1988,263(23):11080-11083.
[19] Kajala K, Brown N J, Williams B P, Borrill P, Taylor L E, Hibberd J M. Multiple Arabidopsis genes primed for recruitment into C4 photosynthesis[J]. The Plant Journal, 2012,69(1):47-56. doi:10.1111/j.1365-313x.2011.04769.x.
[20] 张建华, 姬虎太, 张定一, 王敏, 曹勇.植物DREB转录因子研究进展[J]. 山西农业科学, 2012, 40(1): 79-83. doi:10.3969/j.issn.1002-2481.2012.01.24.
Zhang J H,Ji H T,Zhang D Y,Wang M,Cao Y. Reasearch progress of DREB gene in plant[J].Journal of Shanxi Agricultural Sciences, 2012, 40(1): 79-83.
[21] Nakanishi T, Nakatsu T, Matsuoka M, Sakata K, Kato H. Crystal structures of pyruvate phosphate dikinase from maize revealed an alternative conformation in the swiveling-domain motion[J]. Biochemistry, 2005, 44(4): 1136-1144. doi:10.1021/bi0484522.
[22] 赵晋锋,王高鸿,杜艳伟,李颜方,王振华,赵根有,余爱丽. 谷子磷酸烯醇式丙酮酸羧化酶基因(PEPC)对逆境胁迫的响应[J]. 华北农学报,2019, 34(4): 67-74. doi:10.7668/hbnxb.201751614.
Zhao J F, Wang G H, Du Y W, Li Y F, Wang Z H, Zhao G Y, Yu A L. Response of phosphoenolpyruvate carboxylase (PEPC)gene to abiotic stresses in foxtail millet[J]. Acta Agriculturae Boreali-Sinica, 2019, 34(4): 67-74.
[23] Lim K, Read R J, Chen C C H, Tempczyk A, Wei M, Ye D, Wu C, Dunaway-Mariano, Herzberg O. Swiveling domain mechanism in pyruvate phosphate dikinase[J]. Biochemistry, 2007, 46(51): 14845-14853. doi:10.1021/bi7 01848w.
[24] Nakamoto H, Edwards G E. Light activation of pyruvate, Pi dikinase and NADP-malate dehydrogenase in mesophyll protoplasts of maize: effect of DCMU, antimycin a, CCCP, and phlorizin[J]. Plant Physiology, 1986, 82(1): 312-315. doi:10.1104/pp.82.1.312.
[25] Hatch M D, Slack C R. A new enzyme for the interconversion of pyruvate and phosphopyruvate and its role in the C4 dicarboxylic acid pathway of photosynthesiss[J]. The Biochemical Journal, 1968, 106(1): 141-146. doi:10.1042/bj1060141.
[26] Fukayama H, Tsuchida H, Agarie S, Nomura M, Onodera H, Ono K, Lee B H, Hirose S, Toki S, Ku M S B, Makino A, Matsuoka M, Miyao M. Significant accumulation of C4-specific pyruvate, orthophosphate dikinase in a C3 plant, rice[J]. Plant Physiology, 2001, 127(3): 1136-1146. doi:10.1104/pp.127.3.1136.
[27] 张建福, Datta K S, 王国英, 谢华安. 玉米高光效基因PPDK在籼稻IR64中的整合及其与光合作用相关的特性分析[J]. 分子植物育种, 2006, 4(6): 797-804. doi:10.3969/j.issn.1672-416X.2006.06.007.
Zhang J F, Datta K S, Wang G Y, Xie H A. Integration of C4-specific PPDK gene of maize to C3 rice IR64 and its photosynthesis characteristics analysis[J]. Molecular Plant Breeding,2006,4(6): 797-804.
[28] Kang H G, Park S, Matsuoka M, An G. White-core endosperm flour endosperm-4 in rice is generated by knockout mutations in the C4-type pyruvate orthophosphate dikinase gene (OsPPDKB)[J]. The Plant Journal, 2005, 42(6): 901-911. doc:10.1111/j.1365-313X.2005.02423.x.
[29] Zhang G Y, Liu X, Quan Z W, Cheng S F, Xu X, Pan S K, Xie M, Zeng P, Yue Z, Wang W L, Tao Y, Bian C, Han C L, Xia Q J, Peng X H, Cao R, Yang X H, Zhan D L, Hu J C, Zhang Y X, Li H N, Li H, Li N, Wang J Y, Wang C C, Wang R Y, Guo T, Cai Y J, Liu C Z, Xiang H T, Shi Q X, Huang P, Chen Q C, Li Y R, Wang J,Zhao Z H, Wang J. Genome sequence of foxtail millet (Setaria italica) provides insights into grass evolution and biofuel poten-tial[J]. Nature Biotechnology, 2012, 30(6): 549-554. doi:10.1038/nbt.2195.
[30] Bennetzen J L, Schmutz J, Wang H, Percifield R, Hawkins J, Pontaroli A C, Estep M, Feng L, Vaughn J N, Grimwood J, Jenkins J, Barry K, Lindquis E, Hellsten U, Deshpande S, Wang X W, Wu X M, Mitros T, Triplett J, Yang X H, Ye C Y, Mauro-Herrera M, Wang L, Li P H, Sharma M, Sharma R, Ronald P C, Panaud O, Kellogg E A, Brutnell T P, Doust A N, Tuskan G A , Rokhsar D, Devos K M . Reference genome sequence of the model plant setaria [J]. Nature Biotechnology, 2012, 30(6): 555-561. doi:10.1038/nbt.2196.
[31] Artimo P, Jonnalagedda M, Arnold K, Baratin D, Csardi G, de Castro E, Duvaud S, Flegel V, Fortier A, Gasteiger E, Grosdidier A, Hernandez C, Ioannidis V, Kuznetsov D, Liechti R, Moretti S, Mostaguir K, Redaschi N, Rossier G, Xenarios I, Stockinger H.Expasy: SIB bioinformatics resource portal[J]. Nucleic Acids Research, 2012,40(W11):597-603. doi:10.1093/nar/gks400.
[32] Larkin M A, Blackshields G, Brown N P, Chenna R, McGettigan P A, McWilliam H,Valentin F, Wallace I M, Wilm A, Lopez R, Thompson J D, Gibson T J, Higgins D G. Clustal W and Clustal X version 2.0[J]. Bioinformatics, 2007, 23(21): 2947-2948. doi:10.1093/bioinformatics/btm404.
[33] Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6: Molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30(12):2725-2729. doi:10.1093/molbev/mst197.
[34] Livak K J,Schmittgen T D. Analysis of relative gene expression data using Real-time quantitative PCR and the 2-ΔΔCt method[J].Methods, 2000, 25(4): 402-408. doi:10.1006/meth.2001.1262.
[35] Biehler K, Fock H. Evidence for the contribution of the mehler-peroxidase reaction in dissipating excess electrons in drought stressed wheat[J]. Plant Physiology, 1996,112(1): 265-272. doi:10.2307/4277325.
[36] Manicacci D, Camus-Kulandaivelu L, Fourmann M, Arar C, Barrault S, Rousselet A, Feminias N, Consoli L, Francès L, Méchin V, Murigneux A, Prioul J L, Charcosset A, Damerval C. Epistatic interactions between opaque2 transcriptional activator and its target gene cyppdk1 control kernel trait variation in maizet[J]. Plant Physiology, 2009, 150(1): 506-520. doi:10.2307/40537872.
[37] Hy'sková V D,Miedzińska L, Dobrá J, Vankova R,Ryšlavá H. Phosphoenolpyruvate carboxylase, NADP-malic enzyme, and pyruvate, phosphate dikinase are involved in the acclimation of Nicotiana tabacum L. to drought stress[J]. Journal of Plant Physiology, 2014, 171(5): 19-25. doi:10.1016/j.jplph.2013.10.017.
[38] Moons A, Valcke R, Van M, Montagu. Low-oxygen stress and water deficit induce cytosolic pyruvate orthophosphate dikinase(PPDK) expression in roots of rice, a C3 plant[J]. The Plant Journal, 1998,15(1): 89-98. doi:10.1046/j.1365-313X.1998.00185.x.
[39] Yamamoto E, Sugiyama T, Miyachi S. Action spectrum for light activation of pyruvate, phosphate dikinase in maize leaves[J]. Plant and Cell Physiology,1974, 15(6): 987-992. doi:10.1093/oxfordjournals.pcp.a075107.
