载脂蛋白(Apolipoprotein)是血浆中不溶于水可以与脂质结合参与脂类在血浆中转运的蛋白 [1]。目前已发现的载脂蛋白有20多种,按其组成分为APOA、B、C、D、E、H、M等[2]。其中在血浆中几乎与所有的脂蛋白颗粒相关的载脂蛋白E(Apolipoprotein E,APOE)是一种多态性蛋白,主要呈现3种同种型:E2、E3和E4,它们是单个基因位点上3个等位基因(ε2、ε3和ε4)的产物[3],APOE主要存在于大部分来源于肝细胞的乳糜微粒(Chylomicron,CM)、极低密度脂蛋白(Very low density lipoprotein,VLDL)和部分高密度脂蛋白(High density lipoprotein,HDL)中,其他外周来源有巨噬细胞、星形胶质细胞和脂肪细胞等,APOE既可以与低密度脂蛋白家族成员结合,也可与硫酸肝素蛋白聚糖结合,是影响脂代谢的重要遗传因素之一[4-6]。研究表明,APOE在脂质代谢中发挥重要作用,主要可总结为以下几个方面:①APOE在循环脂蛋白中起到脂蛋白摄取的配体作用,例如APOE对LDL受体家族成员的高亲和力可促进肝脏和肝外血浆脂蛋白的摄取,HDL调控动脉粥样硬化的功能可促进巨噬细胞中胆固醇的去除,因而APOE -/-小鼠已成为研究实验性小鼠动脉粥样硬化的优选模型[7-10];②APOE是甘油三酯(Triglycerides,TG)分解代谢的限速酶,能从血液中清除富含TG的VLDL和CM[11];③王艳梅等[12]研究发现,APOE基因的多态性与2型糖尿病周围神经病变有很大的相关性;④APOE是中枢神经系统(Central nervous system CNS)脂质代谢的主要载脂蛋白之一[13],其基因型对脑脂质稳态的影响可能是与APOE相关的阿尔兹海默病(Alzheimer disease,AD)风险的基础,并且APOE、APOA与CNS脂蛋白的脂质外排和递送功能有关[14]。以上关于人类疾病的研究均证明APOE基因在脂代谢中的重要作用,但APOE基因在反刍动物脂肪方面的研究尚未见报道,目前仅见GenBank上山羊APOE基因的预测序列,其序列的分子特征、组织表达规律及在脂肪细胞中的表达模式还不清楚,而阐明上述这些问题是最终揭示山羊APOE基因功能的重要基础。
因此,本试验首先利用RT-PCR及3′ RACE方法克隆山羊APOE基因序列,在获得基因序列基础上利用qPCR方法检测该基因在山羊各组织和皮下前体脂肪细胞分化过程中的表达模式,获得其组织和细胞表达谱。研究结果为阐明APOE基因在山羊代谢和脂肪细胞分化中的作用提供重要的基础数据。
1 材料和方法
1.1 试验材料
1.1.1 试验样品采集 本试验所用组织采自于四川省简阳大哥大牧业有限公司的1周岁健康大耳羊(Capra hircus)(n=4),清晨空腹屠宰放血后立即取其各个内脏和肌肉等组织,用已灭菌的DEPC水处理组织后再用灭过菌的锡箔纸包裹组织样品,迅速将样品置于冻存管中,标记好后于液氮中储存。
1.1.2 试验材料 RevertAid First Strand cDNA Synthesis Kit购自Thermo公司。I-5TM2×High-Fidelity Master Mix、TreliefTM5α感受态细胞和pClone007 Versatile Simple Vector Kit购自擎科梓熙生物技术有限公司,胶回收试剂盒购自天根生化科技有限公司。3′-Full RACE Core Set、TB GreenTM Premix Ex TaqTM Ⅱ和TRIzol均购自TaKaRa公司,DEPC购自Sigma公司,血清购自Gemini公司,胰蛋白酶和DEME/F12培养基购自Hyclone公司,油酸购自Sigma公司。
1.2 试验方法
1.2.1 山羊APOE基因克隆 以山羊肺脏组织为模板,利用TRIzol法提取总RNA,以1 μg RNA为模板合成cDNA,保存于-20 ℃。根据GenBank中牛(登录号:XM_005219148.4)的APOE基因预测序列,利用Primer premier 5.0设计克隆引物(表1),RT-PCR反应总体系:I-5TM2×High-Fidelity Master Mix 12.5 μL,模板cDNA 1 μL,10 μmol/L上、下游引物各1 μL,ddH2O 9.5 μL。RT-PCR扩增条件:预变性98 ℃ 2 min;变性98 ℃ 10 s,退火61 ℃ 15 s,延伸72 ℃ 15 s,延伸72 ℃ 5 min,变性至第1个延伸过程进行35个循环。利用胶回收试剂盒收获片段,连接到pClone007 Versatile Simple Vector载体后转化于TreliefTM5α感受态细胞,培养10~12 h后挑取单菌落于干净的EP管中,加入含AMP的LB液体培养基培养4~6 h,进行菌液PCR鉴定,将符合预期片段的菌液送至成都擎科梓熙生物技术有限公司测序。APOE基因3′UTR克隆根据3′-Full RACE Core Set试剂盒说明书,利用Primer premir 5.0设计引物(表1);按照说明书中使用实验样品RNA时的操作方法进行反转录、套氏PCR反应等步骤。套氏PCR采用扩增条件:预变性98 ℃ 2 min;变性98 ℃ 10 s,退火61 ℃ 15 s, 延伸72 ℃ 15 s,延伸72 ℃ 5 min,变性至第1个延伸过程进行30个循环。同上述克隆后续步骤,符合预期片段的菌液送至成都擎科梓熙生物技术有限公司测序。
表1 克隆和荧光定量PCR引物信息
Tab.1 Primers informations for reverse transcription PCR (RT-PCR) and quantitative Real-time PCR(qPCR)

基因Gene序列(5'-3')Sequence退火温度/℃Tm产物长度/bpProducts length用途PurposeAPOES:CGGTTTCTGGAGGCGAAG611 364克隆A:TGGGGGCACCAGAGACTCOuterS:CGACGCTGACGACCTGAAGAAG64615克隆A:TACCGTCGTTCCACTAGTGATTTInnerS:CCTCTCCGCCCAGTGAGAATCATT67199克隆A:CGCGGATCCTCCACTAGTGATTTCAC-TATAGGAPOES:CAGGAACTGACGGTGCTGATG62191qPCRA:TCGCTTCGGTACTGTGCCAUXTS:GCAAGTGGATTTGGGCTGTAAC60180内参A:ATGGAGTCCTTGGTGAGGTTGT
注:S.正义链引物;A.反义链引物;UXT.广泛表达转录子基因。
Note:S.Sense primer;A.Antisense primer;UXT.Ubiquitously-expressed transcript gene.
1.2.2 山羊APOE的生物信息学分析 使用DNAMAN进行氨基酸序列相似性比对;ExPASy ProtParam数据库推导APOE蛋白基本理化性质;利用NCBI中Conserved Domains预测蛋白结构域;Blast进行同源性分析;磷酸化位点、O糖基化位点和N糖基化位点分别由NetPhos 3.1、NetOGlyc 4.0和NetNGlyc 1.0预测;TMHMM预测跨膜结构域,SignalIP 4.1预测信号肽;使用NPSA预测蛋白质二级结构;蛋白三级结构使用Swiss-model进行预测分析;PSORT Ⅱ进行亚细胞定位;使用Mega 7.0软件,邻接法(Neighbor-Joining, NJ)进行系统进化树分析;利用Targetscan数据库预测靶标APOE基因的miRNA;利用Mirbase数据库查找miRNA序列。
1.2.3 山羊APOE基因组织表达模式分析 采用实时荧光定量PCR(qPCR)方法,检测该基因在山羊13种组织(心脏、肝脏、脾脏、肺脏、肾脏、背最长肌、股二头肌、臂三头肌、大肠、小肠、皮下脂肪、腹部脂肪、瘤胃)中的表达水平,根据克隆所得CDS区基因序列设计特异引物(表1)。qPCR反应体系:正向引物1 μL(10 μmol/L)、反向引物 1 μL(10 μmol/L)、cDNA 1 μL、TB GreenTM Premix Ex TaqTM Ⅱ 10 μL、ddH2O 7 μL。qPCR运行程序:预变性:95 ℃ 3 min;变性:95 ℃ 10 s,退火(APOE:62 ℃,UXT:60 ℃)10 s,延伸72 ℃ 15 s,38个循环。
1.2.4 APOE基因在山羊皮下脂肪细胞分化过程中的表达模式 取出冻存的山羊皮下脂肪细胞,37 ℃水浴锅复苏细胞后,与含有10%胎牛血清的DEME/F12培养基混合制成细胞悬液,接种于25 cm2培养瓶中,放置于37 ℃、5% CO2培养箱培养,每2 d换液进行传代培养,F3细胞传置12孔板,待细胞铺板到80%时换油酸诱导液诱导分化,分别在诱导0~96 h后加TRIzol收细胞,提取细胞RNA,利用RevertAid First Strand cDNA Synthesis Kit试剂盒反转录细胞cDNA,采用qPCR方法检测山羊皮下前体脂肪细胞分化不同时间段APOE基因的表达情况。qPCR引物、内参和反应条件同1.2.3。
1.2.5 数据统计分析 组织表达与细胞时序表达均选择UXT做内参基因,采用2-ΔΔCt法分析qPCR数据。利用SPSS 18.0中LSD法与Duncan法对数据进行单因素方差分析,使用GraphPad Prism 5绘制组织与时序表达谱,参照LSD与Duncan法分析结果对数据进行差异显著性标记。
2 结果与分析
2.1 山羊APOE基因克隆
以山羊肺脏组织cDNA为模板,RT-PCR扩增获得山羊APOE基因序列970 bp(图1-A),其中包括ORF 951 bp,5′端7 bp,3′UTR区12 bp;3′RACE获得山羊3′UTR 152 bp;编码316个氨基酸(图1-B)。提交GenBank获得登录号MN049956与MN049957;Conserved Domains结构域预测发现山羊APOE蛋白具有载脂蛋白家族典型结构域(图1-C)。

M.DL2000 DNA Marker;1.APOE 基因;信号肽剪切位点用方框表示;结构域用灰色底纹表示;*.终止密码子。A.山羊APOE基因扩增;B.山羊APOE基因核苷酸及推测的氨基酸序列;C.APOE蛋白质生物学功能预测。
M.DL2000 DNA Marker;1.APOE gene;Signal peptide cleavage site is indicated by the box;Domains are indicated by grey shading;*.The stop codon.A.Amplification of APOE gene in Capra hircus;B.The nucleotide sequence and the deduced amino acid sequence of APOE in goat;C.Prediction of biological function of APOE protein.
图1 山羊APOE基因克隆与结构域预测
Fig.1 Cloning and domain prediction of APOE in goat
2.2 生物信息学分析
2.2.1 基本理化性质分析 使用ProtParam、SignalIP 4.1、NetPhos 3.1、和TMHMM等在线预测软件预测山羊APOE蛋白的基本理化性质,预测结果见表2。
表2 山羊APOE基本理化性质
Tab.2 Basic physicochemical properties of goat APOE
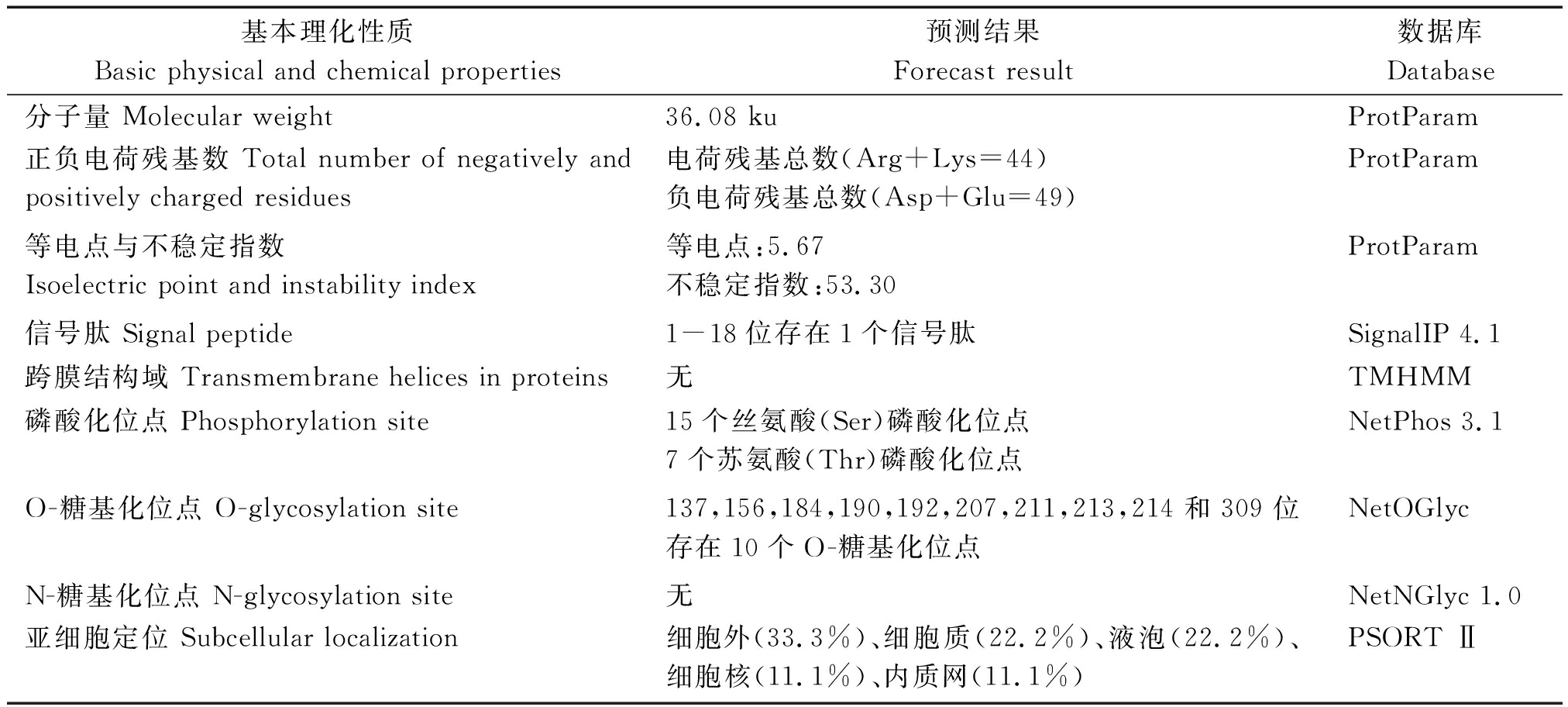
基本理化性质Basic physical and chemical properties预测结果Forecast result数据库Database分子量 Molecular weight36.08 kuProtParam正负电荷残基数 Total number of negatively and positively charged residues电荷残基总数(Arg+Lys=44)负电荷残基总数(Asp+Glu=49)ProtParam等电点与不稳定指数Isoelectric point and instability index等电点:5.67不稳定指数:53.30ProtParam信号肽 Signal peptide1-18位存在1个信号肽SignalIP 4.1跨膜结构域 Transmembrane helices in proteins无TMHMM磷酸化位点 Phosphorylation site15个丝氨酸(Ser)磷酸化位点7个苏氨酸(Thr)磷酸化位点NetPhos 3.1O-糖基化位点 O-glycosylation site137,156,184,190,192,207,211,213,214和309位存在10个O-糖基化位点NetOGlycN-糖基化位点 N-glycosylation site无NetNGlyc 1.0亚细胞定位 Subcellular localization细胞外(33.3%)、细胞质(22.2%)、液泡(22.2%)、细胞核(11.1%)、内质网(11.1%)PSORT Ⅱ
2.2.2 蛋白结构特征互作及靶标miRNA预测 NPSA二级结构预测显示,255(80.70 %)个氨基酸可能形成α螺旋(h),51(16.14 %)个氨基酸可能形成无规卷曲(c),10(3.16 %)个氨基酸可能形成延伸链(e)(图2-A);Swiss-model预测山羊APOE蛋白三级结构(图2-B);利用STRING进行蛋白互作分析构建蛋白互作网,发现APOE蛋白可能和APOB、APOA1、SCARB1、APP、LPL、LRP2、LRP1和LDLR等蛋白存在相互作用,构建蛋白相互作用网(图2-C);在Targetscan的牛数据库低保守的脊椎动物中预测发现可能靶标APOE基因的miR-22-3p(图2-D);Mirbase数据库查到chi-miR-22-3p,登录号:MIMAT0036076。

A.山羊APOE蛋白二级结构预测;B.山羊APOE蛋白三级结构;C.山羊APOE蛋白相互作用分析;D.APOE基因miRNA预测。
A.Prediction of APOE protein secondary structure in goats;B.Goat APOE protein tertiary structure;
C.Interaction analysis in APOE protein of goat;D.Prediction of APOE gene miRNA.
图2 山羊APOE蛋白功能与靶标miRNA预测结果
Fig.2 Goat APOE protein function and target miRNA prediction results
2.3 APOE系统进化树构建
本试验山羊APOE核苷酸和氨基酸序列比对结果见图3-A,山羊APOE基因核苷酸序列与绵羊、藏羚羊和牛的序列相似性较高,分别为98.42%,97.78%和92.05%。根据所选择的不同物种的APOE氨基酸序列绘制系统进化树(图3-B),由进化树可见,山羊与绵羊在同一分支亲缘关系最近,符合物种进化规律。试验引用序列均来自GenBank数据库。
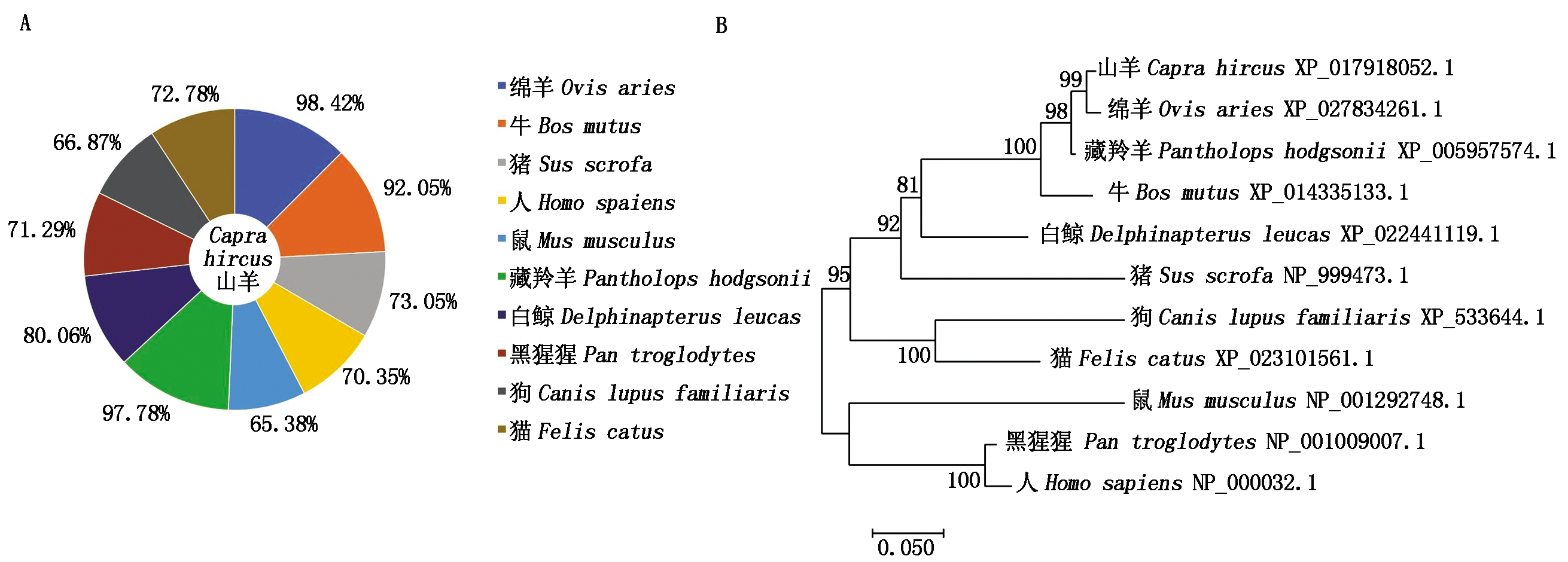
A.不同物种APOE蛋白相似性分析;B.不同物种间APOE氨基酸的进化树。
A.Similarity of APOE protein between different species;B.Phylogenetic tree of APOE of various species.
图3 不同物种APOE氨基酸序列分析
Fig.3 Amino acid sequence analysis of different species of APOE
2.4 山羊APOE基因组织表达谱构建
qPCR检测APOE基因在1周岁简州大耳山羊不同组织中的表达情况(图4),以UXT为内参基因,心脏表达水平做对照来分析试验结果,结果显示APOE基因在皮下脂肪表达量最高,极显著高于其他组织(P<0.01)。
2.5 山羊APOE基因细胞时序表达谱构建
基于APOE基因的组织表达谱得知其在脂肪组织中高表达,为了探究APOE基因在脂肪沉积中发挥的作用,本试验选择山羊皮下前体脂肪细胞为检测对象,分别在诱导分化0~96 h检测APOE的表达水平,以UXT基因为参照基因分析试验结果。结果显示,APOE基因在皮下前体脂肪细胞中表达量较高,在诱导分化60 h时表达量达到峰值,显著高于未分化前体脂肪细胞中的表达水平(P<0.05)(图5)。
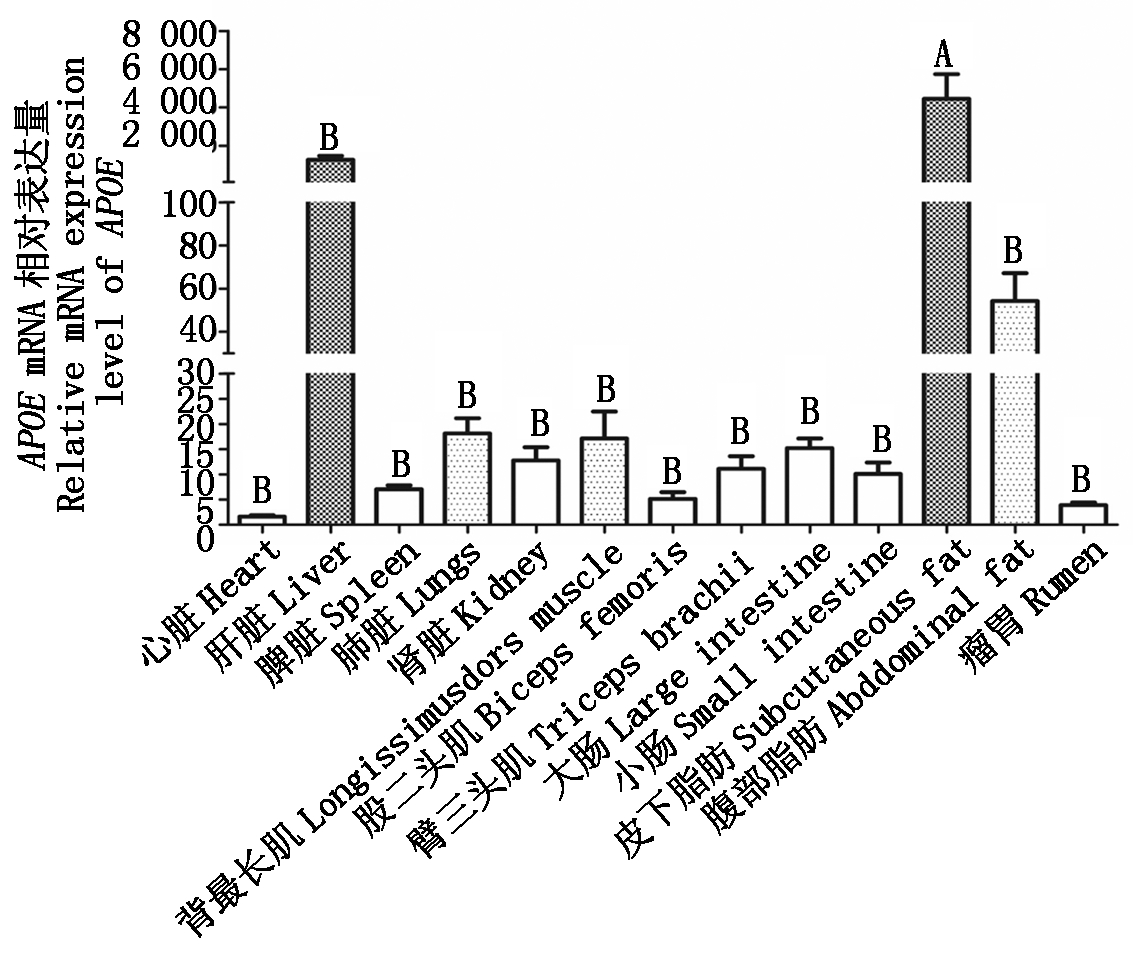
以心脏表达水平为对照,UXT为内参基因。不同字母表示组织间差异极显著(P<0.01)。
The expression level of the heart tissue was used as control,UXT was used as internal reference. The different letters indicated the extremely significant difference of each tissue(P<0.01).
图4 APOE在山羊不同组织中的相对表达水平
Fig.4 The tissues expression of APOE in goat
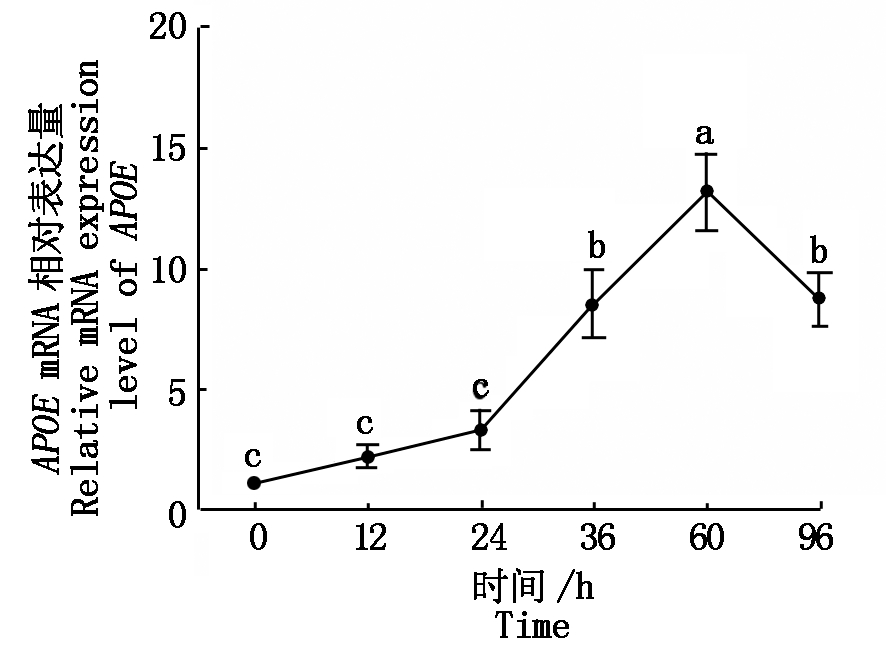
不同字母表示差异显著(P<0.05)。
Different letters indicated the significant difference(P<0.05).
图5 山羊APOE基因在皮下前体脂肪细胞分化过程中的相对表达量
Fig.5 Relative expression of APOE gene in goat during subcutaneous adipocyte differentiation
3 讨论与结论
载脂蛋白E(Apolipoprotein E, APOE)是血浆脂蛋白的重要组成成分,血浆脂蛋白包括极低密度脂蛋白(VLDL)、高密度脂蛋白(HDL)、中间密度脂蛋白(IDL)和乳糜微粒(CM)。相关研究表明,APOE主要存在于VLDL中并且在CM和HDL中也含有少量,APOE的主要功能是与脂蛋白、LDL-R和HSPG(包括肝素)相互作用从而介导CM、VLDL及其残粒在循环系统中的清除[15-16]。近年来研究发现,APOE 基因缺失糖尿病小鼠可能出现胰岛素抵抗,导致糖尿病小鼠血脂代谢进一步紊乱[17-18]。因此,关于其在脂代谢中的作用受到学者的广泛关注,但关于该基因在山羊脂肪沉积和脂代谢方面尚未见报道。本研究基于NCBI上APOE基因的预测序列克隆得到山羊APOE基因序列970 bp,通过在线数据库对山羊APOE进行生物信息学分析,山羊APOE编码316个氨基酸,蛋白预测发现该蛋白是一个不稳定的酸性蛋白;并利用STRING预测软件分析蛋白相互作用得知APOE与载脂蛋白B(APOB)、血清载脂蛋白A1(APOA1)、低密度脂蛋白受体相关蛋白1(LRP1)、淀粉样前体蛋白(APP)、低密度脂蛋白受体(LDLR)和脂蛋白脂酶(LPL)等蛋白存在相互作用,而以上蛋白均与脂代谢与脂肪沉积密切相关[19-24]。有研究证明,APOE可以和低密度脂蛋白受体(LDLR)和LDLR相关蛋白结合而促进脂蛋白颗粒的细胞摄取,并且APOE的3个等位基因E2、E3和E4的氨基酸序列不同会导致受体结合亲和力功能差异,即E2等位基因的氨基酸序列比E3和E4等位基因的结合亲和力低,导致肝脏VLDL和乳糜微粒残留物清除率降低,从而减少餐后脂蛋白颗粒的摄取[25-27]。Cheng等[28] 在APOE-|-小鼠中研究miR-182与脂质水平之间的潜在关联发现,在miR-182 agomir处理组,腹膜巨噬细胞中表达LPL mRNA和蛋白质显著增加,并且APOE-|-小鼠的血浆TC水平显著升高,血浆低密度脂蛋白胆固醇(LDL-C)浓度升高,但降低了血浆TG浓度。本研究在Targetscan牛数据库的低保守脊椎动物中预测到可能靶标APOE基因的miR-22-3p,并在Mirbase数据库发现存在chi-miR-22-3p,de Gonzalo-Calvo等[29]研究心外膜脂肪量(Epicardial fat volume,EFV)发现血浆miR-22-3p与EFV直接相关,并且高EFV患者血浆miR-22-3p表达水平显著高于底EFV患者,此外Colom等[30]针对Ⅰ型糖尿病(T1DM)研究,分析心外膜脂肪组织(Epicardial adipose tissue,EAT)与HDL成分之间的关系发现,EAT指数(iEAT)越高小HDL(HDL3)的比例越高,并且HDL中APOC-Ⅲ和APOA-Ⅱ的含量与iEAT呈正相关,而与ApoE含量为负相关。
为了进一步阐明山羊APOE基因的功能,本研究明确了其组织表达模式,结果发现该基因在皮下脂肪中的表达量极高,Hamlin等[31]报道APOE主要通过结合肝脏及肝外组织APOE.B100受体介导脂蛋白代谢。因APOE基因在山羊皮下脂肪组织中存在高水平表达,推测该基因与脂肪沉积有关,为了进一步证实这个想法,本研究利用qPCR方法明确了该基因在山羊皮下脂肪细胞分化前后的表达模式,结果发现该基因随着脂肪细胞分化的进行呈上升趋势,且在60 h时表达水平最高。推测APOE可能具有促进山羊肌内脂肪细胞分化的作用。姜红等[15]研究发现,在APOE敲除小鼠中,APOE缺失会使内脏脂肪素(Visfatin)基因表达增加,而Visfatin是重要的脂肪细胞因子。且Li等[32]指出,Visfatin融合蛋白可通过促进脂肪细胞分化标志基因PPARγ、aP2、C/EBPα和FAS的表达来促进鸡前脂肪细胞向成熟脂肪细胞的分化。但APOE基因在山羊肌内脂肪细胞分化中到底扮演着什么角色,则需要进一步利用超表达和干扰手段进行进一步研究。
本研究成功克隆获得山羊APOE基因序列(登录号:MN049956,MN049957),其中ORF为951 bp,编码316个氨基酸。 组织表达分析结果显示该基因在山羊皮下脂肪中存在高表达;时序表达分析结果显示,APOE基因在皮下脂肪细胞中诱导分化60 h时表达量最高,显著高于未分化中的表达水平。通过组织表达谱和时序表达谱可以推测APOE在山羊皮下脂肪中发挥主要作用,该结果为阐明APOE基因如何调控脂肪细胞分化、脂肪沉积及脂质代谢中的作用提供了基础理论数据。
[1] 杨开燕,钟栩,卢玉俊,蒙占权. 血浆载脂蛋白的研究进展[J]. 临床荟萃, 2012, 27(14): 1268-1271.
Yang K Y, Zhong Y, Lu Y J, Meng Z Q. Research progress of plasma apolipoprotein[J]. Clinical Focus, 2012, 27(14): 1268-1271.
[2] 谢印军,黄刚,邓峰美,迟晔虹. 载脂蛋白基因多态性与血脂关系研究进展[J]. 现代生物医学进展, 2010, 10(11): 2197-2200. doi:10.13241/j.cnki.pmb.2010.11.026.
Xie Y J, Huang G, Deng F M, Chi Y H. Relationship between apolipoprotein gene polymorphism and serum lipids[J]. Progress in Modern Biomedicine, 2010, 10(11): 2197-2200.
[3] Greenow K, Pearce N J, Ramji D P. The key role of apolipoprotein E in atherosclerosis[J]. J Mol Med, 2005, 83(5): 329-342. doi:10.1007/s00109-004-0631-3.
[4] Tudorache I F, Trusca V G, Gafencu A V. Apolipoprotein E-A multifunctional protein with implications in various pathologies as a result of its structural features[J]. Comput Struct Biotechnol J, 2017, 15:359-365.doi:10.1016/j.csbj.2017.05.003.
[5] Mahley R W, Rall S C Jr. Apolipoprotein E: far more than a lipid transport protein[J]. Annu Rev Genomics Hum Genet, 2000, 1: 507-537. doi:10.1146/annurev.genom.1.1.507.
[6] Lu J Y, Yu H Y, Mo Y F, Ma X J, Hao Y P, Lu W, Li H T, Bao Y Q, Zhou J, Jia W P. Patterns of circulating fibroblast growth factor 21 in subjects with and without type 2 diabetes[J]. PLoS One, 2015, 10(11): 207-209. doi:10.1371/journal.pone.0142207.
[7] Greenow K, Pearce N J, Ramji D P. The key role of apolipoprotein E in atherosclerosis[J]. J Mol Med, 2005, 83(5): 329-342. doi:10.1007/s00109-004-0631-3.
[8] Getz G S, Reardon C A. ApoE knockout and knockin mice: the history of their contribution to the understanding of atherogenesis[J]. Lipid Res, 2016, 57(5): 758-766. doi:10.1194/jlr.R067249.
[9] Getz G S,Reardon C A. Apoproteins E,A-I and SAA in macrophage pathobiology related to atherogenesis[J]. Front Pharmacol, 2019, 10: 536. doi:10.3389/fphar.2019.00536.
[10] Calabresi L, Gomaraschi M, Simonelli S, Bernini F, Franceschini G. HDL and atherosclerosis: Insights from inherited HDL disorders[J]. Biochim Biophys Acta, 2015, 1851(1):13-18. doi:10.1016/j.bbalip.2014.07.015.
[11] Liu H N, Li X, Song Y P, Wang Z B. MicroRNA-217 attenuates intima-media complex thickness of ascending aorta measured by ultrasound bio-microscopy and inhibits inflammation and lipid metabolism in atherosclerotic models of ApoE-/- mice[J]. Lipids Health Dis, 2018, 17(1): 170. doi:10.1186/s12944-018-0825-2.
[12] 王艳梅,严美花,徐远,李爱国,赵进喜. ApoE基因多态性与2型糖尿病周围神经病变及中医证型的关系[J]. 中华中医药杂志, 2018, 33(11): 4915.
Wang Y M, Yan M H, Xu Y, Li A G, Zhao J X. Relationship between ApoE gene polymorphism and peripheral neuropathy in type 2 diabetes and TCM syndrome[J]. China Journal of Traditional Chinese Medicine and Pharmacy, 2018, 33(11): 4915.
[13] Pitas R E, Boyles J K, Lee S H, Hui D, Weisgraber K H. Lipoproteins and their receptors in the central nervous system. Characterization of the lipoproteins in cerebrospinal fluid and identification of apolipoprotein B,E(LDL) receptors in the brain[J]. J Biol Chem, 1987, 262(29): 14352-14360.
[14] Rebeck G W. The role of APOE on lipid homeostasis and inflammation in normal brains[J]. J Lipid Res, 2017, 58(8): 1493-1499. doi:10.1194/jlr.R075408.
[15] 姜红,种艳莹,王东琦,凌志龙,方媛. ApoE缺失对糖尿病小鼠脂肪含量及脂代谢调节因子的影响[J]. 临床和实验医学杂志, 2018, 17(23): 2479-2483. doi:10.3969/j.issn.1671-4695.2018.23.005.
Jiang H, Chong Y Y, Wang D Q, Ling Z L, Fang Y. Effect of ApoE deficiency on fat content and lipid metabolism regulators in diabetic mice[J]. Journal of Clinical and Experimental Medicine, 2018, 17(23): 2479-2483.
[16] Getz G S, Reardon C A. Apoprotein E and reverse cholesterol transport[J]. Int J Mol Sci, 2018, 19(11): 3479. doi:10.3390/ijms19113479.
[17] Jiang Y W, Ma L, Han C W, Liu Q, Cong X, Xu Y P, Zhao T T, Li P, Cao Y T. Effects of apolipoprotein E isoforms in diabetic nephropathy of Chinese type 2 diabetic patients[J]. J Diabetes Res, 2017(3): 1-6. doi:10.1155/2017/3560920.
[18] Pang Y M, Gan L, Wang X Z, Su Q, Liang C, He P. Celecoxib aggravates atherogenesis and upregulates leukotrienes in ApoE-|- mice and lipopolysaccharide-stimulated RAW264.7 macrophages[J]. Atherosclerosis, 2019,284: 50-58. doi:10.1016/j.atherosclerosis.2019.02.017.
[19] 郑以漫,孙荣,李小英,高梅,赵列宾,许曼音,陈家伦. ApoE基因多态性与中国人非胰岛素依赖型糖尿病血脂水平的关系[J]. 中华医学遗传学杂志, 1997(3): 142-145.
Zheng Y M, Sun R, Li X Y, Gao M, Zhao L B, Xu M Y, Chen J L. Relationship between ApoE gene polymorphism and the serum lipid levels in Chinese with niddm[J]. Chinese Journal of Medical Genetics, 1997(3): 142-145.
[20] 谢君辉,张木勋. 载脂蛋白E基因多态性与高脂血症的相关性研究进展[J]. 医学综述, 2003, 9(10): 631-632. doi:10.3969/j.issn.1006-2084.2003.10.027.
Xie J H, Zhang M X. Research progress on the correlation between apolipoprotein E gene polymorphism and hyperlipidemia[J]. Medical Recapitulate, 2003, 9(10): 631-632.
[21] Zeng Z L, Cao B X, Guo X P, Li W J, Li S H, Chen J, Zhou W P, Zheng C S, Wei Y M. Apolipoprotein B-100 peptide 210 anti-body inhibits atherosclerosis by regulation of macrophages that phagocy-tize oxidized lipid[J]. Am J Transl Red, 2018, 10(6): 1817-1828.
[22] Lu Z, He X F, Ma B B,Zhang L, Li J L, Jiang Y, Zhou G H, Gao F. Increased fat synthesis and limited apolipoprotein B cause lipid accumulation in the liver of broiler chickens exposed to chronic heat stress[J]. Poult Science, 2019,98(9):3695-3704. doi:10.3382/ps/pez056.
[23] Lamantia V, Bissonnette S, Provost V, Devaux M, Cyr Y, Daneault C, Rosiers C D, Faraj M. The association of polyunsaturated fatty acid δ-5-Desaturase activity with risk factors for type 2 diabetes is dependent on plasma ApoB-Lipoproteins in overweight and obese adults[J]. The Journal of Nutrition, 2019, 149(1): 57-67. doi:10.1093/jn/nxy238.
[24] Xepapadaki E, Maulucci G, Constantinou C, Karavia E A, Zvintzou E, Daniel B, Sasson S, Kypreos K E. Impact of apolipoprotein A1-or lecithin: cholesterol acyltransferase-deficiency on white adipose tissue metabolic activity and glucose homeostasis in mice[J]. Biochim Biophys Acta Mol Basis Dis, 2019, 1865(6): 1351-1360. doi:10.1016/j.bbadis.2019.02.003.
[25] Eichner J E, Dunn S T, Perveen G, Thompson D M, Stewart K E, Stroehla B C. Apolipoprotein E polymorphism and cardiovascular disease:a HuGE review[J]. Am J Epidemiol, 2002, 155(6): 487-495. doi:10.1093/aje/155.6.487.
[26] Shatwan I M, Winther K H, Ellahi B, Elwood P, Ben-Shlomo Y, Givens I, Rayman M P, Lovegrove J A, Vimaleswaran K S. Association of apolipoprotein E gene polymorphisms with blood lipids and their interaction with dietary factors[J]. Lipids Health Dis, 2018, 17(1): 98. doi:10.1186/s12944-018-0744-2.
[27] 秦艳. 高脂饮食对ApoE/LDLR双缺失小鼠肝脏脂肪酸代谢相关因子表达的影响[D].济南:山东师范大学, 2010.
Qin Y. Effect of high-fat diet on the expression of fatty acid metabolism-related factors in the liver of ApoE/LDLR double-deficient mice[D].Jinan: Shandong Normal University, 2010.
[28] Cheng H P, Gong D, Zhao Z W, He P P, Yu X H, Ye Q, Huang C, Zhang X, Cen L Y, Xie W, Zhang M, Li L, Xia X D, Ouyang X P, Tan Y L, Wang Z B, Tian G P, Zheng X L, Yin W D, Tang C K. MicroRNA-182 promotes lipoprotein lipase expression and atherogenesisby targeting histone deacetylase 9 in apolipoprotein E-Knockout mice[J]. Circ J, 2017, 82(1): 28-38. doi:10.1253/circj.CJ-16-1165.
[29] de Gonzalo-Calvo D, Vilades D,Martínez-Camblor P, Vea  , Ferrero-Gregori A, Nasarre L, Bornachea O, Vega J S, Leta R, Puig N, Benítez S, Sanchez-Quesada J L, Carreras F, Llorente-Cortés V. Plasma microRNA profiling reveals novel biomarkers of epicardial adipose tissue: a multidetector computed tomography study[J]. J Clin Med, 2019, 8(6): 780. doi:10.3390/jcm8060780.
, Ferrero-Gregori A, Nasarre L, Bornachea O, Vega J S, Leta R, Puig N, Benítez S, Sanchez-Quesada J L, Carreras F, Llorente-Cortés V. Plasma microRNA profiling reveals novel biomarkers of epicardial adipose tissue: a multidetector computed tomography study[J]. J Clin Med, 2019, 8(6): 780. doi:10.3390/jcm8060780.
[30] Colom C, Viladés D, Pérez-Cuellar M, Leta R, Rivas-Urbina A, Carreras G, Ordó ez-Llanos J, Pérez A, S
ez-Llanos J, Pérez A, S nchez-Quesada J L. Associations between epicardial adipose tissue, subclinical atherosclerosis and high-density lipoprotein composition in type 1 diabetes[J]. Cardiovasc Diabetol, 2018, 17(1): 156. doi:10.1186/s12933-018-0794-9.
nchez-Quesada J L. Associations between epicardial adipose tissue, subclinical atherosclerosis and high-density lipoprotein composition in type 1 diabetes[J]. Cardiovasc Diabetol, 2018, 17(1): 156. doi:10.1186/s12933-018-0794-9.
[31] Hamlin A N, Chinnarasu S, Ding Y Y, Xian X D, Herz J, Jaeschke A, Hui D Y. Low-density lipoprotein receptor-related protein-1 dysfunction synergizes with dietary cholesterol to accelerate steatohepatitis progression[J]. The Journal of Biological Chemistry, 2018, 293(25): 9674-9684. doi:10.1074/jbc.RA118.001952.
[32] Li Z J, Wang Y C, Tian X X, Shang P F, Chen H, Kang X T, Tian Y D, Han R L. Characterization of the visfatin gene and its expression pattern and effect on 3T3-L1 adipocyte differentiation in chickens[J]. Gene, 2017, 632: 16-24. doi:10.1016/j.gene.2017.08.025.
