谷瘟病为灰梨孢菌(Magnaporthe oryzae)侵染谷子引起的一种真菌性病害,是目前谷子上重要的气传流行性病害[1],该病害流行时对谷子产量影响很大,发病严重时某些感病品种甚至绝收[2],抗谷瘟品种选育和种植是防治该病害最简单和有效的方法[3]。谷瘟病菌的无毒基因与谷子抗病基因互作与稻瘟病菌和水稻抗病基因互作机制相同,都遵循基因对基因假说[4]。当水稻所含有的抗病基因产物与稻瘟病菌无毒基因产物发生直接或间接的相互识别,可引发寄主发生过敏性坏死反应(HR),诱导水稻抗性产生起到抗病作用[5-6],但无毒基因经常发生变异[7],使得抗性品种在种植几年后抗病性衰退[8]。因此,对不同地区谷瘟病菌无毒基因进行检测,明确不同地区谷瘟病菌无毒基因类型及其变异机制,可为深入研究谷瘟病菌群体遗传结构及其无毒基因变异机制奠定基础。到目前为止,已经克隆了多个稻瘟病菌无毒基因,如Avr1-CO39[9]、Avr-pita[10]、ACE1[11]、AvrPiz-t[12]、Avr-pii[13]、Avr-pia和Avr-pik等,除Avr1-CO39分离自寄主弯叶画眉草外,其余均分离自水稻。PWL家族属于寄主特异性无毒基因,目前PWL1与PWL2被证明具有无毒基因功能,控制稻瘟病菌对弯叶画眉草的致病性[14-15]。穆慧敏等[16]通过设计PWL基因家族成员的特异性引物PCR扩增,证明PWL1基因仅存在牛筋草中,而寄主为水稻的稻瘟病菌中未发现PWL1基因,部分菌株存在PWL2基因。
至今,关于我国谷瘟病菌中PWL无毒基因家族的分布和变异的研究还未见报道,本研究通过设计PWL基因特异性引物对不同地区来源的252株谷瘟病菌进行扩增分析,以期了解PWL家族在谷瘟病菌群体的分布及变异,为进一步研究谷瘟病菌与谷子的互作机制奠定基础。
1 材料和方法
1.1 供试菌株
本试验中供试谷瘟病菌单孢菌株均分离自田间具有典型谷瘟病症状的样本,利用干燥滤纸片法在冰箱-20 ℃冷冻保存,菌株信息来源见表1。
1.2 谷瘟病菌活化培养和菌丝DNA提取
表1 252个谷瘟病菌供试菌株
Tab.1 252 isolates of Magnaportheoryzae from foxtail millet
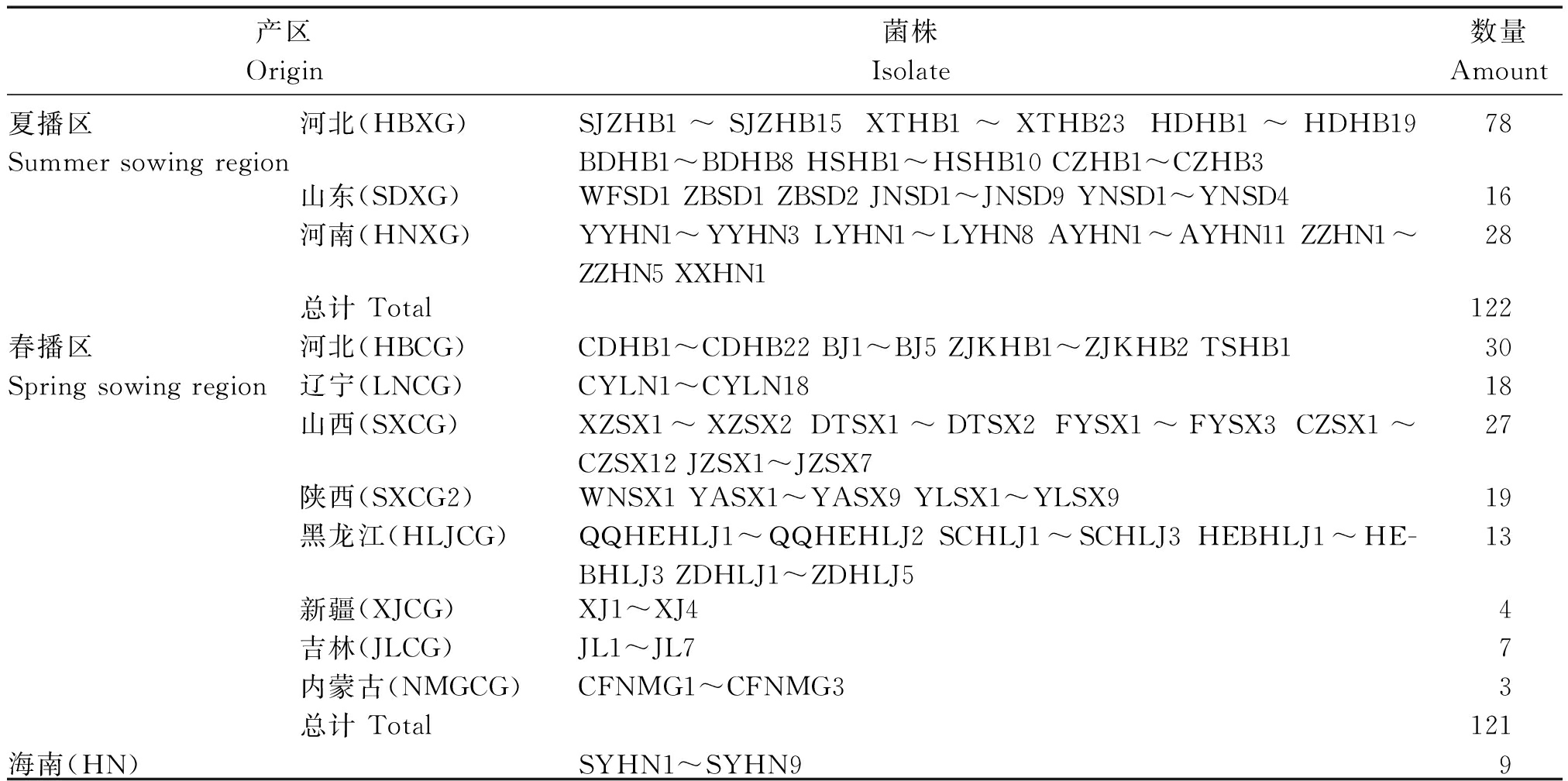
产区Origin菌株 Isolate 数量Amount夏播区Summer sowing region河北(HBXG)SJZHB1~SJZHB15 XTHB1~XTHB23 HDHB1~HDHB19 BDHB1~BDHB8HSHB1~HSHB10CZHB1~CZHB3 78山东(SDXG) WFSD1 ZBSD1 ZBSD2 JNSD1~JNSD9 YNSD1~YNSD416河南(HNXG) YYHN1~YYHN3 LYHN1~LYHN8 AYHN1~AYHN11 ZZHN1~ZZHN5 XXHN128总计 Total122春播区河北(HBCG) CDHB1~CDHB22 BJ1~BJ5 ZJKHB1~ZJKHB2 TSHB130Spring sowing region辽宁(LNCG) CYLN1~CYLN1818山西(SXCG) XZSX1~XZSX2 DTSX1~DTSX2 FYSX1~FYSX3 CZSX1~CZSX12 JZSX1~JZSX727陕西(SXCG2) WNSX1 YASX1~YASX9 YLSX1~YLSX919黑龙江(HLJCG) QQHEHLJ1~QQHEHLJ2 SCHLJ1~SCHLJ3 HEBHLJ1~HE-BHLJ3 ZDHLJ1~ZDHLJ513新疆(XJCG) XJ1~XJ44吉林(JLCG) JL1~JL77内蒙古(NMGCG) CFNMG1~CFNMG33总计 Total121海南(HN)SYHN1~SYHN99
将冻存的谷瘟病菌滤纸片放置到PDA平板上进行培养,培养7 d后挑取菌落边缘幼嫩菌饼接种到液体PDA培养基中,振荡培养7 d后用灭菌纱布收集菌丝体,用滤纸吸干多余水分。采用常规CTAB法提取谷瘟病菌菌丝体DNA,提取DNA样本检测合格后保存于-20 ℃备用。
1.3 引物设计
参考穆慧敏等[16]设计的用于扩增PWL1、PWL3和PWL4基因的特异性引物,从NCBI网站下载稻瘟病菌的PWL无毒基因家族相关基因核苷酸序列,根据PWL2与PWL3基因的核苷酸序列,通过Primer 5生物信息学软件设计这2个基因的检测引物,在上海生工合成相关引物。具体的引物信息见表2。
表2 PWL基因家族扩增引物序列
Tab.2 Primers sequences for amplification of PWL gene family
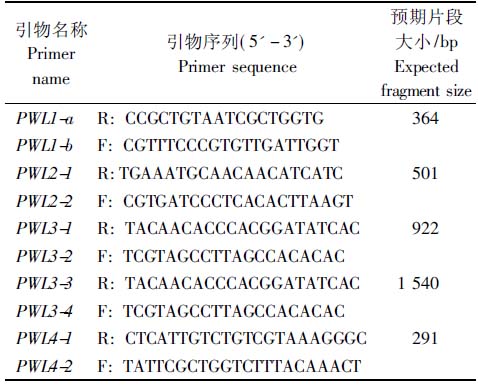
引物名称Primer name引物序列(5'-3')Primer sequence预期片段大小/bpExpected fragment sizePWL1-aR: CCGCTGTAATCGCTGGTG364PWL1-bF: CGTTTCCCGTGTTGATTGGTPWL2-1R:TGAAATGCAACAACAT-CATC501PWL2-2F: CGTGATCCCTCACACTTA-AGTPWL3-1R: TACAACACCCACGGATAT-CAC922PWL3-2F: TCGTAGCCTTAGCCACACACPWL3-3R: TACAACACCCACGGATAT-CAC1 540PWL3-4F: TCGTAGCCTTAGCCACACACPWL4-1R: CTCATTGTCTGTCGTA-AAGGGC291PWL4-2F: TATTCGCTGGTCTTTA-CAAACT
1.4 无毒基因扩增与测序分析
PCR反应总体系25 μL:2×Ex Taq Master Mix 12.5 μL(康为世纪),上游引物0.5 μL,下游引物0.5 μL,DNA模板 2 μL,ddH2O 9.5 μL。扩增步骤:94 ℃预变性5 min;94 ℃变性 30 s,55 ℃退火30 s,72 ℃延伸2 min,共35次循环;最后72 ℃充分延伸10 min。扩增产物经1.0%琼脂糖凝胶电泳后EB染色观察。用手术刀切取目的条带后用胶回收试剂盒纯化PCR产物,纯化后的产物与pMD19-T 载体 16 ℃连接过夜,通过热激法把连接产物转化到大肠杆菌DH5α感受态细胞中,复苏培养后涂布到氨苄青霉素LB平板上,挑取单克隆通过PCR法对克隆进行检测,把阳性克隆保存在甘油后邮寄到上海生工进行测序分析。采用DNAMAN 6.0软件对测序结果进行序列分析。
2 结果与分析
2.1 PWL基因家族的PCR扩增检测结果
利用5对PWL无毒基因引物对252株谷瘟病菌DNA进行检测,扩增结果表明:PWL基因家族在谷瘟病菌中存在变异,变异的形式主要为基因的缺失和插入。252株谷瘟病菌都未扩增出PWL1基因的特异性目的片段,说明谷瘟病菌菌株不含PWL1基因。65个谷瘟病菌菌株扩增出无毒基因PWL2的特异性目的片段,扩增率为25.8%。而无毒基因PWL4和PWL3扩增率均为100%,但无毒基因PWL3的扩增片段相较于目的片段超出849 bp,部分菌株无毒基因PWL家族扩增结果如图1所示。
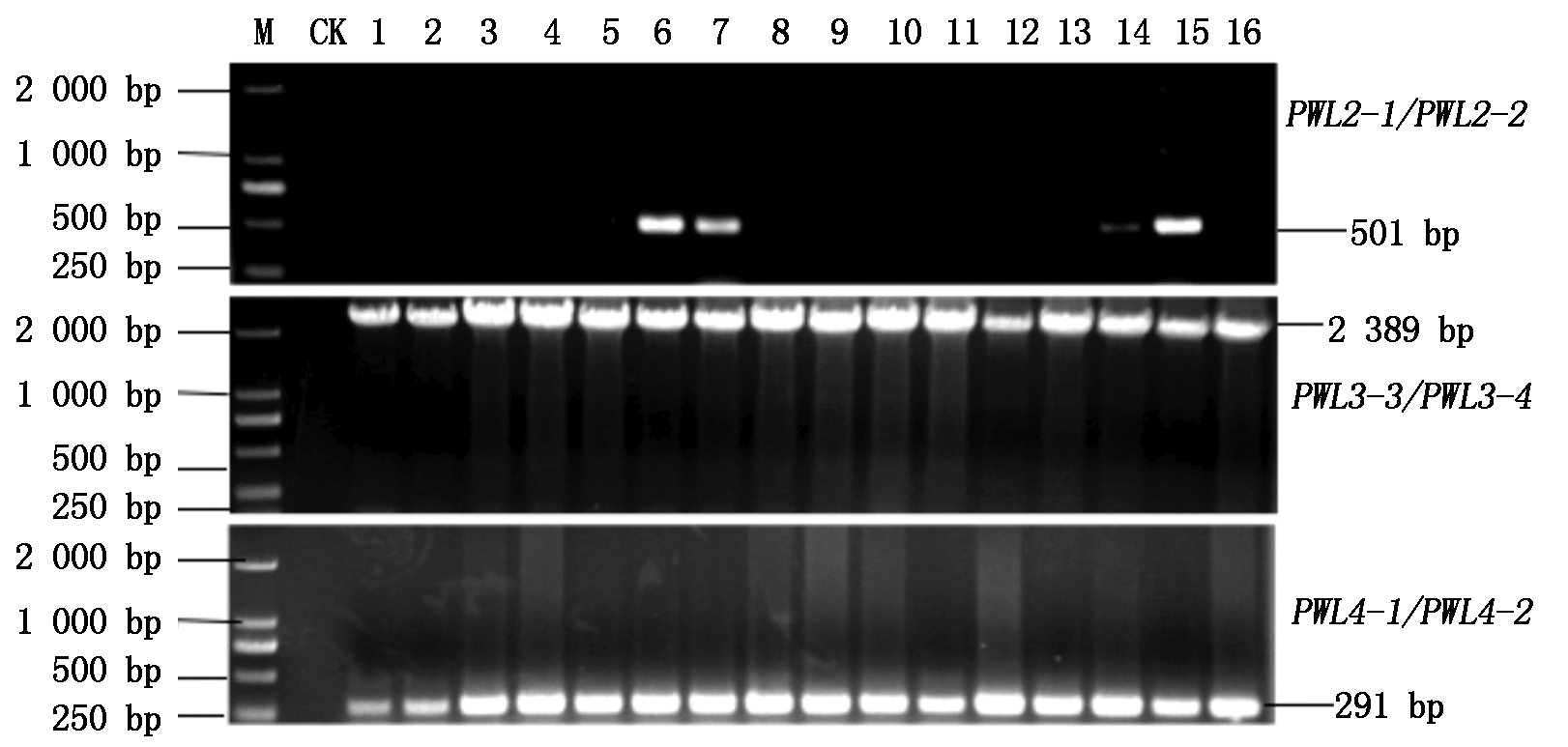
M.Marker;CK.阴性对照(ddH2O);1-16.谷瘟病菌分离物。
M.DNA size Marker; CK.Negative control(ddH2O); 1-16.Strain 1-16. Magnaporthe oryzae from foxtail millet.
图1 利用3对引物对PWL无毒基因检测
Fig.1 Three primer pairs detect the presence of Avr-genes of PWL
2.2 无毒基因PWL2序列变异分析
对65个谷瘟病菌菌株的无毒基因PWL2的扩增条带进行胶回收,并进行克隆测序。首先利用Chromas 2.6.4软件对测序结果进行人工比对校正,然后利用Clustalx 2.0软件对校正后的序列进行比对分析,比对结果通过DnaSP 5.0软件进行单倍型多样性分析,利用DNAMAN 6.0对不同单倍型的谷瘟病菌菌株无毒基因PWL2的核苷酸序列和编码的蛋白序列进行比对分析。结果表明(图2,3),谷瘟病菌无毒基因PWL2共划分为14个单倍型,分别命名为P1~P14(GenBank登录号:MG787153~MG787166)。以参考序列(U26313.1)为外群,利用NETWORK 5.0生物信息学软件构建谷瘟病菌PWL2的单倍型网络图,从图4可以看出优势单倍型为P1,共包含46个菌株,占65个谷瘟病菌菌株的70.8%,以此为基础衍生出其他主要单倍型,其次为单倍型P12共包含7株谷瘟病菌,黑龙江地区2株、河南地区4株及辽宁地区1株,其余单倍型均包含单一菌株,河北、河南、山西、陕西、辽宁、黑龙江等地区均有特异性单倍型,以河南地区单倍型种类最多,包含6种,说明总体上谷瘟病菌种群具有较高的遗传多样性。

图2 供试菌株的PWL2序列比对
Fig.2 Sequence comparison analysis of PWL2 gene among partial isolates

图3 不同菌株间PWL2编码氨基酸序列比对
Fig.3 Comparison of multiple amino acid sequences of PWL2 gene among different isolates
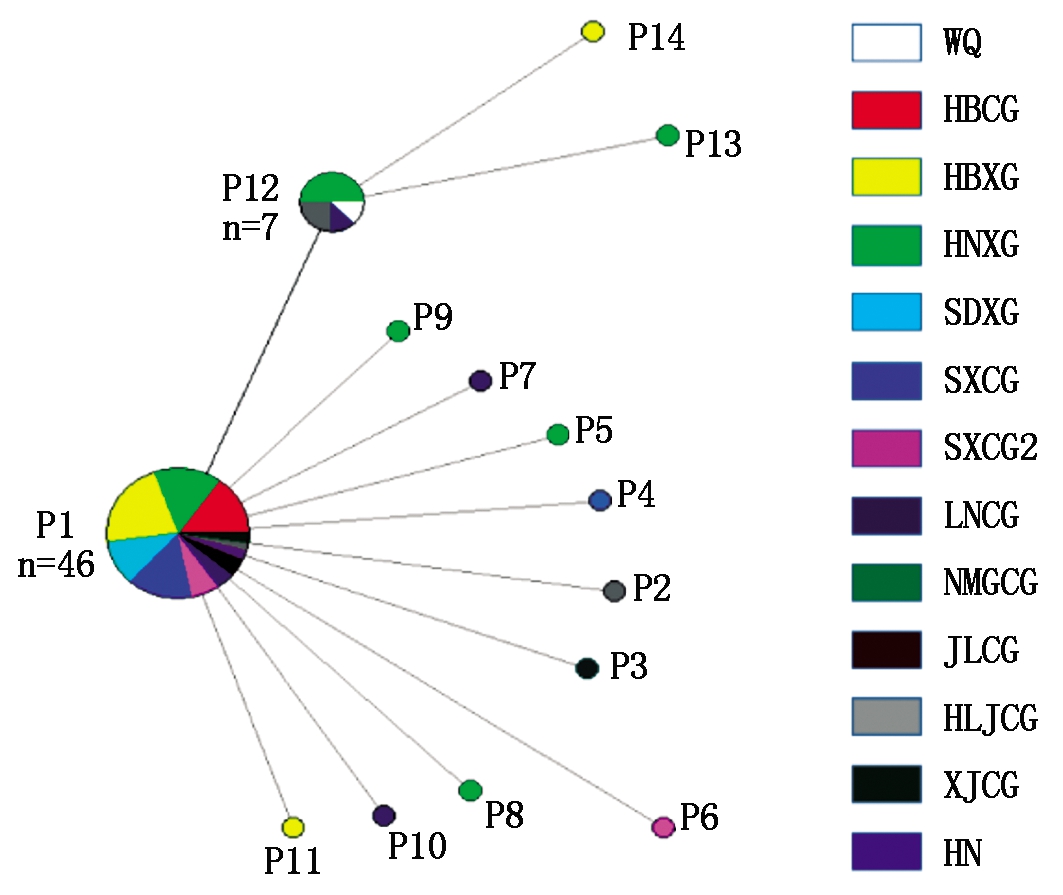
彩色饼图的面积反映了单倍型的数量;彩色段代表了在每个区域中发生相同单倍型的比例。
The area of the color pie indicates the number of haplotype and color segments reflects the proportion of different groups in each haplotype.
图4 谷瘟群体间PWL2单倍型网状图
Fig.4 Construction of haplotype network among different group of Magnaportheoryzae from foxtail millet based on PWL2 gene
2.3 谷瘟病菌与稻瘟病菌无毒基因PWL3序列差异
利用引物PWL3-1/PWL3-2对随机挑选的11株谷瘟病菌和11株稻瘟病菌进行扩增,电泳检测结果如图5所示。谷瘟病菌无毒基因PWL3序列比预期目的条带大850 bp左右,随机挑选6个菌株的无毒基因PWL3扩增序列进行回收克隆测序,通过NCBI在线比对分析表明,在基因编码区插入849 bp的核苷酸片段,与non-LTR retrotransposon:MGL的相似度达到96.7%。利用谷瘟病菌与稻瘟病菌的PWL3基因的差异,通过特异性引物可以从分子水平上快速区分谷瘟病菌与稻瘟病菌。
3 结论与讨论
在禾本科植物中,寄主为水稻的稻瘟病菌无毒基因的相关报道较多,其中以品种特异性无毒基因的变异及群体遗传多样性的研究居多,关于PWL无毒基因家族的变异分析较少,而谷子中谷瘟病菌无毒基因的研究更是鲜有报道。本研究通过5对特异性引物对谷瘟病菌PWL无毒基因家族成员进行扩增,探寻其在不同谷瘟病菌菌株的分布及变异情况。
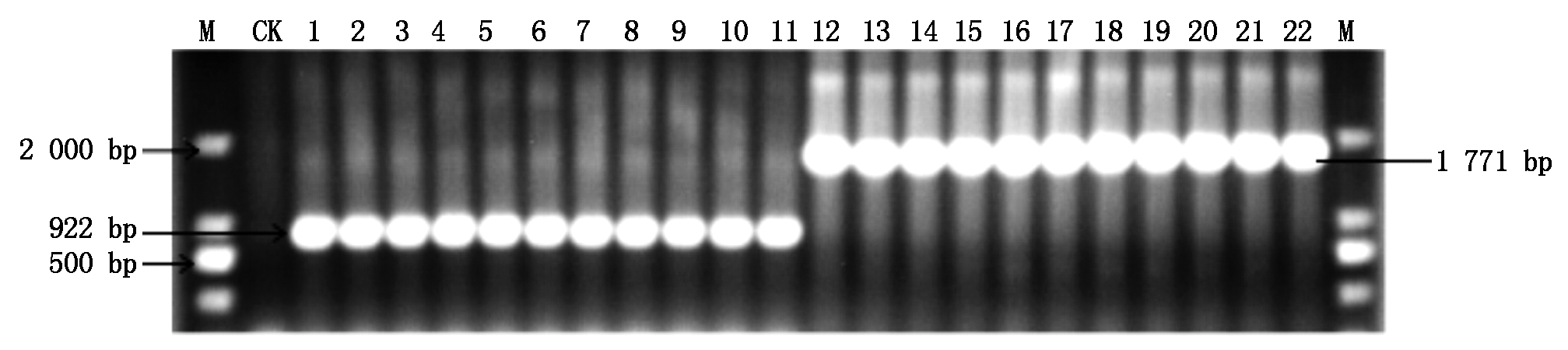
M.Marker;CK.阴性对照(ddH2O);1-11.稻瘟病菌菌株的DNA;12-22.谷瘟病菌菌株的DNA。
M.Marker; CK.Negative contro1(ddH2O); 1-11.Strain 1-11 of Magnaporthe oryzae from rices; 12-22.Strain 12-22 of Magnaporthe oryzae from foxtail millet.
图5 谷瘟病菌与稻瘟病菌中PWL3检测图
Fig.5 The detection of PWL3 gene in rice blast and foxtail millet blast by PCR
穆慧敏等[16]研究表明,稻瘟病菌中不存在PWL1无毒基因,而寄主为牛筋草的稻瘟病菌中却存在PWL1无毒基因,根据两者的差异,利用PCR扩增即可在分子水平上快速区分不同寄主的灰梨孢菌。本研究发现谷瘟病菌中同样不含有PWL1无毒基因,可以用于区分寄主分别为谷子和牛筋草的灰梨孢菌。任世龙等[17]研究不同谷瘟病菌菌株IGS序列时发现谷瘟病菌与稻瘟病菌的IGS序列存在差异,但因序列相差较小,普通琼脂糖电泳难以区分,必须通过测序才能从分子水平上进行区分。本研究发现,谷瘟病菌的PWL3无毒基因与稻瘟病菌PWL3无毒基因序列存在非常明显的差异,具体表现在谷瘟病菌PWL3基因在编码区插入849 bp的反转录转座子,PCR特异性扩增产物通过1.0%的普通琼脂糖凝胶电泳就能快速区分。
灰梨孢菌具有广泛的寄主范围,谷子为其寄主之一,除此,禾本科植物小麦、牛筋草、黑麦草以及水稻也都是其寄主[18]。关于不同寄主的灰梨孢菌相互之间能否互相侵染还有争议,Kato等[19]通过致病性、交配能力和DNA限制性片段长度多态性研究表明,每个植物属的灰梨孢菌分离物构成一个特定于宿主的子群,具体表现寄主为谷子的谷瘟病菌特异性侵染狗尾草属的植物,对水稻不具有侵染性。但杜新法等[20]采用分离自稻田周围常见的10种杂草上的梨孢菌与分离自水稻上的稻瘟病菌以涂布法做交叉接种,发现稻瘟病菌对田间的牛筋草、狗尾草等杂草具有较弱的致病性,同样分离自狗尾草的灰梨孢菌对丽江新团黑谷等少数品种具有致病性,并判断稻田周围杂草上的灰梨孢菌在稻瘟病菌的传播和积累过程中具有一定作用。传统谷子种植产区多分布于华北、西北及东北等地区,而东北地区和河南部分地区由于同时种植谷子与水稻,使得梨孢菌有可能在不同寄主间进行传播。所以快速区分不同寄主之间的灰梨孢菌对于研究该菌在不同作物间的传播及分布,阐明瘟病初侵染源及其寄主分布具有一定的意义。
Kang等[14]研究表明,无毒基因PWL1与PWL2控制着灰梨孢菌是否对弯叶画眉草具有侵染能力。 Sweigard等[15]研究发现,不同地理来源的稻瘟病菌菌株无毒基因PWL2具有较高的多态性,无毒基因PWL2易发生变异,单碱基的变异可造成无毒基因功能的丧失,进而恢复侵染弯叶画眉草的能力。Schneider等[21]发现一种PWL2基因的等位基因PWL2D,该基因与PWL2的主要区别为第90位的氨基酸由G转变为N,该位点变异可导致寄主植物对病原体丧失识别能力。
本研究结果表明,谷瘟病菌中均含有PWL2基因,并存在整个无毒基因的缺失和多处单核苷酸变异,通过DnaSP 5.0软件单倍型分析发现,谷瘟病菌中PWL2共包含14种单倍型,其中单倍型P1占比70.8%,为绝对优势单倍型,该类型与PWL2D相似,均在第90位氨基酸发生G到N的突变,但在142位的氨基酸并未发生C到S的突变,P1单倍型谷瘟病菌能否感染弯叶画眉草还需进一步验证。本研究尝试利用谷瘟病菌PWL2基因的核苷酸变异来分析不同地区谷瘟病菌群体的遗传结构,发现不同地区病菌群体之间具有共性,比如都包含P1单倍型,谷瘟病菌在各地区之间可能以某种形式存在一定的交流,同时不同地区也存在一些其他地区并不存在的单倍型,不同地理来源的谷瘟病菌群体具有差异性,这种共性与差异性的同时存在说明谷瘟病菌不同地区之间遗传差异性与地理来源之间存在较为复杂的关系。
综上所述,本研究以来自不同地区的252株谷瘟病菌单孢菌株为试验材料,通过PCR扩增及DNA测序分析发现,谷瘟病菌菌株不存在无毒基因PWL1,部分菌株存在PWL2基因,全部谷瘟病菌均存在PWL3与PWL42个基因。同时谷瘟病菌与稻瘟病菌的PWL3基因存在849 bp的差异,基于这种差异可通过PCR技术快速区分谷瘟病菌与稻瘟病菌。谷瘟病菌无毒基因PWL家族成员的分布及变异分析可为研究谷瘟病菌本身遗传多样性,及深入研究不同寄主灰梨孢菌的寄主特异性和灰梨孢菌自然条件下的积累与传播研究提供参考。
[1] 牛倩云, 刘丽青, 孙硕, 仪慧兰.谷瘟病菌拮抗放线菌的筛选、鉴定及抑菌活性[J]. 山西农业科学, 2017, 45(9): 1525-1529. doi: 10.3969/j.issn.1002-2481.2017.09.31.
Niu Q Y, Liu L Q, Sun S, Yi H L. Screening and identification of antagonistic actinomycetes against Pyricularia setariae nishik and analysis of its antipathogenic activity[J]. Journal of Shanxi Agricultural Sciences, 2017, 45(9): 1525-1529.
[2] 任世龙, 白辉, 王永芳, 全建章, 董志平, 李志勇, 邢继红. 谷瘟病菌无毒基因型鉴定及分析[J]. 中国农业科学, 2018, 51(6): 1079-1088. doi: 10.3864/j.issn.0578-1752.2018.06.007.
Ren S L, Bai H, Wang Y F, Quan J Z, Dong Z P, Li Z Y, Xing J H. Identification and analysis of Magnaporthe oryzae of foxtail millet avirulence genes[J]. Scientia Agricultura Sinica, 2018, 51(6): 1079-1088.
[3] Li W T, Chern M, Yin J J, Wang J, Chen X W. Recent advances in broad-spectrum resistance to therice blast disease[J]. Current Opinion in Plant Biology,2019, 50: 114-120. doi: 10.1016/j.pbi.2019.03.015.
[4] He M, Xu Y P, Chen J H, Luo Y, Lü Y, Su J, Kershaw M J, Li W T, Wang J, Yin J J, Zhu X B, Liu X H, Chern M, Ma B T, Wang J C, Qin P, Chen W L, Wang Y P, Wang W M, Ren Z L, Wu X J, Li P, Li S G, Peng Y L, Lin F C, Talbot N J, Chen X W. MoSnt2-dependent deacetylation of histone H3 mediates MoTor-dependent autophagy and plantinfection by the rice blast fungus Magnaporthe oryzae[J].Autophagy, 2018, 14(9): 1543-1561. doi: 10.1080/15548627.2018.1458171.
[5] Dangol S, Chen Y F, Hwang B K, Jwa N S. Iron-and reactive oxygen species-dependent ferroptotic cell death in rice-Magnaporthe oryzae interactions[J]. The Plant Cell, 2019,31(1): 189-209. doi: 10.1105/tpc.18.00535.
[6] Singh P K, Ray S, Thakur S, Rathour R, Sharma V, Sharma T R. Co-evolutionary interactions between host resistance and pathogen avirulence genes in rice-Magnaporthe oryzae pathosystem[J]. Fungal Genetics and Biology, 2018, 115: 9-19. doi: 10.1016/j.fgb.2018.04.005.
[7] Lopez A L C, Yli-Matilla T, Cumagun C J R. Geographic distribution of avirulence genes of the rice blast fungus Magnaporthe oryzae in the Philippines[J]. Microorganisms, 2019, 7(1): 23. doi: 10.3390/microorganisms7010023.
[8] Wang B H, Ebbole D J, Wang Z H. The arms race between Magnaporthe oryzae and rice: Diversity and interaction of Avr and R genes[J]. Journal of Integrative Agriculture, 2017, 16(12): 2746-2760. doi: 10.1016/S2095-3119(17)61746-5.
[9] Zhang X, He D, Zhao Y X, Cheng X L, Zhao W S, Taylor L A, Yang J, Liu J F, Peng Y L. A positive-charged patch and stabilized hydrophobic core are essential for avirulence function of AvrPib in the rice blast fungus[J]. The Plant Journal, 2018, 96(1): 133-146. doi: 10.1111/tpj.14023.
[10] Tranga H T T, Chau N N B, Thaoa N P, Jantasuriyaratc C, Quocd N B. Analyzing sequence variation of the avirulence Avr-Pita1 gene of rice blast isolates, Magnaporthe oryzae in Vietnam[J]. Agricultural andNatural Resources, 2019, 53(1): 20-25. doi: 10.34044/j.anres.2019.53.1.03.
[11] Law J W F, Ser H L, Khan T M, Chuan L H, Pusparajah P, Chan K G, Goh B H, Lee L H. The potential of streptomyces as biocontrol agents against the rice blast fungus, Magnaporthe oryzae(Pyricularia oryzae)[J]. Frontiers in Microbiology, 2017, 8: 3. doi: 10.3389/fmicb.2017.00003.
[12] Tang M Z, Ning Y S, Shu X L, Dong B, Zhang H Y, Wu D X, Wang H, Wang G L, Zhou B. The Nup98 homolog APIP12 targeted by the effector AvrPiz-t is involved in rice basal resistance against Magnaporthe oryzae[J]. Rice, 2017, 10(1): 5. doi: 10.1186/s12284-017-0144-7.
[13] Lu L, Wang Q, Jia Y, Bi Y Q, Li C Y, Fan H C, Li J B. Selection and mutation of the avirulence gene AVR-Pii of the rice blast fungus Magnaporthe oryzae[J]. Plant Pathology, 2019, 68(1): 127-134.doi: 10.1111/ppa.12935.
[14] Kang S, Sweigard J A, Valent B. The PWL host specificity gene family in the blast fungus Magnaporthe grisea[J]. Molecular Plant Microbe Interactions, 1995, 8(6):939-948.doi:10.1094/mpmi-8-0939.
[15] Sweigard J A, Carroll A M, Kang S, Farrall L, Chumley F G, Valent B. Identification, cloning, and characterization of PWL2, a gene for host species specificity in the rice blast fungus[J]. Plant Cell, 1995, 7(8):1221-1233.doi: 10.1105/tpc.7.8.1221.
[16] 穆慧敏,姜华,毛雪琴,柴荣耀,王艳丽,张震,王教瑜,邱海萍,杜新法.PWL基因家族在稻瘟病菌中的分布及变异初探[J].浙江农业学报,2013, 25(3):526-532. doi: 10.3969/j.issn.1004-1524.2013.03.20.
Mu H M, Jiang H, Mao X Q, Chai R Y, Wang Y L, Zhang Z, Wang J Y, Qiu H P, Du X F. A primary study on distribution and variability of PWL gene family in Magnaporthe grisea[J]. Acta Agriculturae Zhejiangensis, 2013, 25(3):526-532.
[17] 任世龙, 白辉, 董立, 董志平, 全建章, 李志勇, 邢继红. 中国不同地理来源谷瘟病菌rDNA-IGS序列分析[J]. 植物病理学报, 2017, 47(3): 305-312. doi: 10.13926/j.cnki.apps.000020.
Ren S L, Bai H, Dong L, Dong Z P, Quan J Z, Li Z Y, Xing J H. Sequence analysis of rDNA-IGS of Magnaporthe oryzae isolates from different geographical origins in China[J]. Acta Phytophylacica Sinica, 2017, 47(3): 305-312.
[18] Yoshida K, Saunders D G O, Mitsuoka C, Natsume S, Kosugi S, Saitoh H, Inoue Y, Chuma I, Tosa Y, Cano L M, Kamoun S,Terauchi R. Host specialization of the blast fungus Magnaporthe oryzae is associated with dynamic gain and loss of genes linked to transposable elements [J].BMC Genomics, 2016, 17(1): 1-18. doi: 10.1186/s12864-016-2690-6.
[19] Kato H, Yamamoto M, Yamaguchi-ozaki T, Kadouchi H, Iwamoto Y, Nakayashiki H, Tosa Y, Mayama S, Mori N. Pathogenicity, mating ability and DNA restriction fragment length polymorphisms of Pyricularia populations isolated from gramineae, bambusideae and zingiberaceae plants[J]. Journal of General Plant Pathology, 2000, 66(1):30-47. doi: 10.1007/PL00012919.
[20] 杜新法, 孙漱沅, 陶荣祥, 孙国昌, 张志明,郑重. 稻区杂草上的梨孢菌与稻瘟病发生的关系[J]. 植物病理学报, 1997, 27(4): 327-332.doi: 10. 13926 /j.cnki.apps.1997.04.010.
Du X F, Sun S R, Tao R X, Sun G C, Zhang Z M,Zheng Z.Effect of WEED-HOSTS Pyricularia incidence of rice blast disease [J]. Acta Phytopathologica Sinica, 1997, 27(4): 327-332.
[21] Schneider D R S, Saraiva A M, Azzoni A R, Miranda H R C A N,de Toledo M A S,Pelloso A C,Souza A P. Overexpression and purification of PWL2D, a mutant of the effector protein PWL2 from Magnaporthe grisea[J]. Protein Expression and Purification, 2010, 74(1):24-31. doi: 10.1016/j.pep.2010.04.020.
