水稻是人类赖以生存的主要粮食作物之一,稻米品质一直备受水稻育种家的关注,也是水稻品种改良的主要方向,其中香味是最受消费者欢迎的一个重要蒸煮品质。稻米的香味主要来源于一类挥发性物质-2-乙酰基-1-吡咯啉(2-acetyl-1-pyrroline, 2AP)[1],因此2AP含量成为衡量稻米香味的重要指标。研究表明,当甜菜醛脱氢酶基因Badh2功能缺失时,合成2AP的前体物质氨基丁醛积累,并转化成吡咯啉,与乙酰基团结合形成2-乙酰基-吡咯啉,从而使2AP含量增多[2-3]。因此,可通过对Badh2基因进行遗传改造,实现对水稻香味品质的遗传改良。
传统的杂交育种方法改良水稻的香味品质需要数年的杂交选育,而且选育过程中不可避免地会引入其他非目的性状,耗时费力。基因编辑技术的兴起和不断发展,为香稻育种提供了新途径,使得定向编辑基因、快速获得具有理想性状的品种成为可能。近年来,最主要的基因编辑技术包括人工介导的锌指核酸酶技术(Zinc finger nucleases, ZFNs)[4]、类转录激活因子效应核酸酶技术(Transcription activator-like effectors nucleases, TALENs)[5]和规律成簇的间隔回文重复相关蛋白技术(CRISPR/Cas9)[6-7],其中CRISPR/Cas9技术具有灵活高效的特点,成为最受欢迎的基因编辑工具,发展最为迅速,应用范围最广。高彩霞课题组利用TALEN技术成功获得badh2突变体,突变体的香味物质含量明显提高[8]。然而TALEN技术在构建载体时过程较为繁杂,需要将多个重复单元进行克隆并串联起来,构建难度大,成本高。因此,邵高能等[9]利用CRISPR/Cas9系统对Badh2基因进行编辑,也成功获得香味物质提高的突变体材料。目前所知,香稻的badh2基因突变类型有2种,一种是在第2个外显子上存在7 bp的碱基缺失,另一种是在第7个外显子上缺失8 bp,这2种碱基缺失均会造成移码突变,使Badh2不表达,功能缺失[10-11]。以上研究表明,通过对Badh2基因进行靶向编辑以改良水稻的香味品质是切实可行的。
超级稻品种龙粳31是由黑龙江省农业科学院育成,具有早熟、高产、抗稻瘟病、抗倒伏、耐寒等诸多优良性状[12-13],因其综合性状表现突出,近年来成为黑龙江省种植面积最大的水稻品种,但没有明显的香味,食味品质有提升空间。
本研究利用CRISPR/Cas9基因编辑系统,对龙粳31品种的Badh2基因进行靶向编辑,在T1选育出纯合的、无转基因序列遗留且香味品质明显改良的香稻株系,创制具有香味的龙粳31品种,不仅提高了龙粳31的食味品质,也为香稻品种育种提供了很好的育种材料,本研究具有非常重要的现实意义和经济价值。
1 材料和方法
1.1 植物材料
水稻转化受体材料是超级稻品种龙粳31,受体材料种植于湖北省武汉市实验基地,与武汉地区的早稻种植时间一致,常规田间种植管理。
1.2 载体构建和遗传转化
根据水稻基因组注释网址(http://rice.plantbiology.msu.edu/)提供的Badh2基因(登录号LOC_Os08g32870)序列信息,设计sgRNA-E2和sgRNA-E3 2个靶位点,靶位点sgRNA-E2跨Badh2基因的第2个内含子和第2个外显子(20380166-20380188),靶位点sgRNA-E3位于第3外显子(20381374-20381396)(图1),靶序列长20 bp,其后面是PAM序列(Protospacer adjacent motif)NGG,2个靶位点均位于现有香稻品种突变位置的上游,使获得有效突变的概率更大。载体构建参照Gao等[14]的方法,简述如下:以含U6表达框的载体pCXUN-Cas9/U6为模板进行2轮PCR扩增,第1轮用引物对sgRNA-E2-F和OsU6-R进行扩增,第2轮用sgRNA-E2-R和OsU6-F进行扩增;将2轮PCR扩增产物混合,以OsU6-F和OsU6-R为引物进行扩增,扩增产物连接到用KpnⅠ酶切后的pCXUN-Cas9载体骨架上,获得最终的表达载体pCXUN-Cas9-E2。sgRNA-E3的表达载体pCXUN-Cas9-E3构建方法同pCXUN-Cas9-E2。通过酶切和测序确定表达载体是否构建成功。将正确的表达载体转化农杆菌EHA105。植物受体材料的遗传转化由杭州百格生物技术有限公司完成。
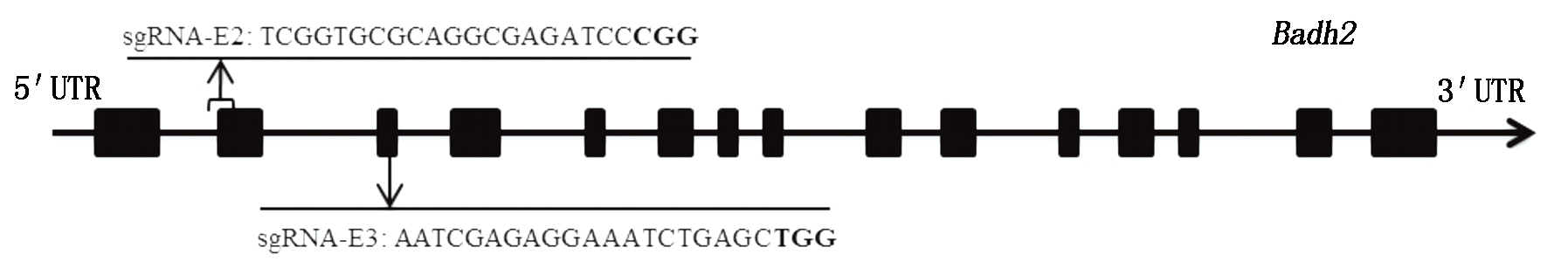
图1 2个靶位点在Badh2基因上的位置
Fig.1 Positions of the two target sites of the gRNA in Badh2
1.3 转基因阳性水稻株系检测及突变植株的筛选
用CTAB法提取野生型龙粳31和转基因T0植株叶片的基因组DNA,用潮霉素引物Hpt-F/Hpt-R(表1)进行PCR扩增,鉴定转基因阳性单株。
在2个gRNA的靶位点上下游分别设计引物GPE2-F/GPE2-R和GPE3-F/GPE3-R(表1),对2个靶位点分别进行PCR扩增,扩增产物连接到pEASY-T载体后,每个单株选10个单克隆用M13F通用引物进行Sanger测序。突变体和野生型的测序结果用MEGA 6软件进行序列比对。
1.4 突变体中Badh2基因表达情况
针对水稻Badh2基因的CDS序列,用在线引物设计工具Primer-Blast(https://www.ncbi.nlm.nih.gov/tools/primer-blast/)设计荧光定量PCR引物QBadh2-F/QBadh2-R(表1)。用TRIzol法提取野生型和突变株系成熟植株叶片的总RNA,用DNase Ⅰ消化总RNA中残留的DNA之后,以Oligo(dT)18为引物,按照M-MuLV反转录酶(NEB,英国)的使用说明书反转录为cDNA。用SYRB GREEN方法对Badh2在野生型和突变体中的表达进行相对定量,以水稻Ubiquitin基因作为内参基因,用TransStart®TipTop Green qPCR SuperMix reagent(全式金,北京)进行qRT-PCR反应,qPCR仪为ABI StepOne Plus。数据处理采用2-ΔΔCT法计算基因的相对表达量[15]。用Student′s t-test方法计算Badh2基因在野生型和突变体中表达的差异显著性。
表1 所用引物列表
Tab.1 List of primers used in this study

引物名称Primer name序列(5′-3′)Primer sequence用途UsagesgRNA-E2GCGGTGCGCAGGCGAGATCC靶序列合成sgRNA-E3GATCGAGAGGAAATCTGAGC靶序列合成CRISPR-E2-FTCGGTGCGCAGGCGAGATCCCGGGTTTTAGAGCTAGAAATAGCAAGTTA载体构建CRISPR-E2-RCCGGGATCTCGCCTGCGCACCGAAACCTGAGCCTCAGCGCAGC载体构建CRISPR-E3-FAATCGAGAGGAAATCTGAGCTGGGTTTTAGAGCTAGAAATAGCAAGTTA载体构建CRISPR-E3-RCCAGCTCAGATTTCCTCTCGATTAACCTGAGCCTCAGCGCAGC载体构建OsU6-FCCCCTTTCGCCAGGGGTACC载体构建OsU6-RTACGAATTCGAGCTCGGTACC载体构建Cas9-FTTATGAATTCATGGACTATAAGGACCACCas9基因Cas9-RCTATCCCGGGTTACTTTTTCTTTTTTGCCas9基因Hpt-FTAGGAGGGCGTGGATATGTC潮霉素磷酸转移酶基因扩增Hpt-RTACACAGCCATCGGTCCAGAGPE2-FCACTCCACACCTGACACCAC靶位点sgRNA-E2区域扩增GPE2-RATTGCGCGGAGGTACTTGGGPE3-FAGCAAAGCAGGGGATGCTA靶位点sgRNA-E3区域扩增GPE3-RCCCAACTACACCGATAGGCTCQBadh2-FGCCAAAAGCCAAGGTGCTACBadh2基因的荧光定量PCR扩增QBadh2-RTTCACACAGAGCACTGGACCUbiquitin-FTCCGAGAGATGGGTTTCATC水稻内参基因Ubiquitin扩增Ubiquitin-RGCCAAGATTGCCAAGAAGAC
1.5 香味物质测定
用气相色谱-质谱联用(GC-MS)技术测定香稻特征化合物2-乙酰-1-吡咯啉(2AP)的含量。方法参照Bergman等[16],用2AP和2,4,6-三甲基吡啶(2,4,6-trimethylpyridine, TMP)制作标准曲线。取2 g T1水稻种子脱壳,研磨成粉末置于顶空瓶中,加入TMP作为内标物,在Agilent 7890气相色谱质谱联用仪上检测。色谱柱为HP-INNOWax(32 m×250 μm×0.25 μm),色谱及质谱条件参照应兴华等[17],检测工作由上海百趣生物医学科技有限公司完成。
1.6 转基因T1突变体农艺性状调查
龙粳31是超级稻品种,在改良稻米香味的同时,不能破坏该品种原有的优良性状。因此,统计并分析了T0突变单株的自交后代群体的基本农艺性状,株高、有效分蘖数和千粒质量。每个株系选10个单株,测量株高,记录有效分蘖数、千粒质量的计算方式是随机选取300粒饱满的种子称质量,然后换算成千粒质量。
1.7 无转基因序列的突变株鉴定
对6个突变株系的T1单株用引物Hpt-F/Hpt-R和Cas9-F/Cas9-R(表1)进行PCR扩增,2对引物都不能扩增出载体目的片段的植株为无转基因序列的突变株。同时用靶位点扩增引物GPE2-F/GPE2-R和GPE3-F/GPE3-R(表1)检测T1植株的突变情况。
所有结果用Graphpad软件进行统计分析。
2 结果与分析
2.1 突变体的获得及靶位点突变类型分析
提取转基因植株叶片的基因组DNA,用潮霉素特异引物检测T0转基因植株,2个转化载体pCXUN-Cas9-E2和pCXUN-Cas9-E3分别获得转基因阳性单株7,4株(图2-A)。
进一步对转基因阳性植株的突变类型进行分析,现有报道将CRISPR/Cas9技术获得的突变体分为三类:纯合突变、复杂杂合突变和双等位突变[18]。结果发现(图2-B),在转基因T0获得2个纯合突变株badh2-E2-6和badh2-E2-11,4个双等位突变株badh2-E2-9、badh2-E2-13、badh2-E3-6和badh2-E3-14,这些株系的突变类型包括碱基缺失碱基插入、碱基突变。所有的突变均出现在gRNA区间,纯合突变株badh2-E2-6缺失7 bp,badh2-E2-11株插入8 bp;双等位突变株badh2-E2-9缺失7,2 bp,badh2-E3-6株插入1 bp(A/T),badh2-E3-14株缺失1 bp和插入1 bp。其中,badh2-E2-13出现不同程度的大片段缺失,在靶位点上下游分别缺失64,108 bp。
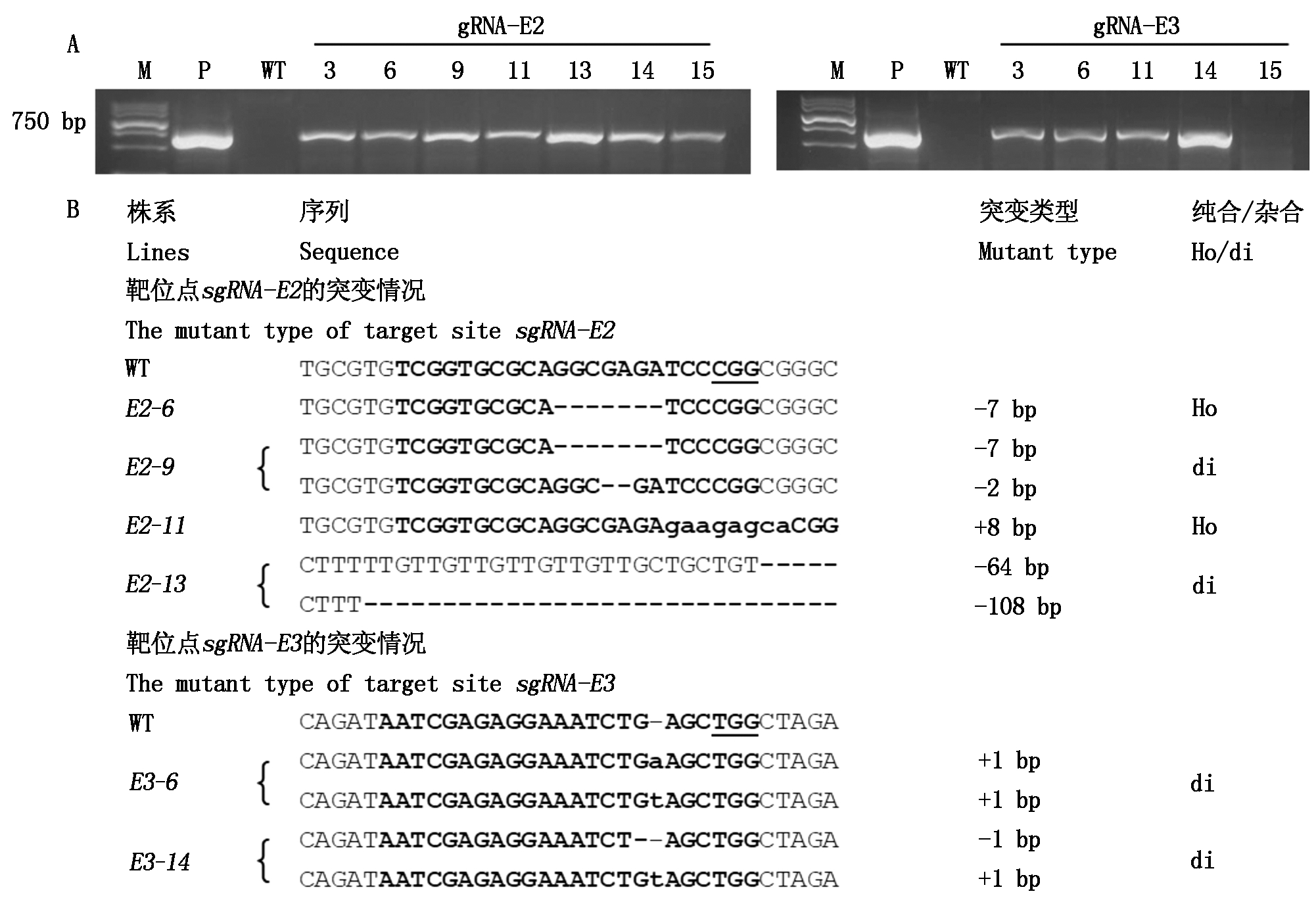
A.潮霉素基因PCR检测;B.T0突变位点序列分析。M.Marker DL2000;P.质粒;WT.野生型;E2-6、E2-9、E2-11、E2-13、E3-6、E3-14.Badh2的突变体。黑色粗体大写字母为靶位点序列,带下划线的字母为PAM序列;加粗小写字母.碱基插入或突变;-.碱基缺失。Ho.纯合突变;di.双等位突变。图3-4同。
A.PCR detecting Hpt; B.Sequence analysis of T0 mutation sites.M. Marker DL2000; P.Plasmid; WT.Wild type; E2-6, E2-9, E2-11, E2-13, E3-6, E3-14.Indicates mutant of Badh2. The target site is shown in bold capital letters; The underlined letter is the PAM sequence; Bold lowercase letter.Base insertion or mutation; Bolddashes.Deleted base. Ho.Homozygote; di.Di-allelic mutations.The same as Fig.3-4.
图2 T0突变位点序列分析
Fig.2 Sequence analysis of T0 mutation sites
2.2 突变体中Badh2基因的表达量显著降低
基因组水平上Badh2基因的突变体在靶位点处发生了碱基变化,为了检测这种变化在转录水平上是否延续,进一步检测了Badh2基因在突变体和野生型植株的表达情况。实时荧光定量PCR结果显示(图3-A):在突变体中,Badh2基因的表达量相对野生型显著下调。其中在badh2-E2-6、badh2-E2-9、badh2-E2-11、badh2-E2-13、badh2-E3-6和badh2-E3-14的表达相比野生型差异显著(P<0.05),表明2个靶点的突变能显著降低Badh2基因的表达。
2.3 香味物质含量的测定
利用气相质谱联用的方法测定部分突变体的香味物质2AP含量,结果发现(图3-B),这些突变体的2AP浓度在0.032~0.098 mg/kg,badh2-E2-6、badh2-E2-9、badh2-E2-11、badh2-E2-13、badh2-E3-6这5个株系较野生型差异显著(P<0.05),与应兴华等[17]测定的结果相符。其中,突变体badh2-E2-13相比野生型2AP的含量提高了4倍,相比对照香稻品种信香粳1号提高了1倍,显著提高了香味物质的含量。
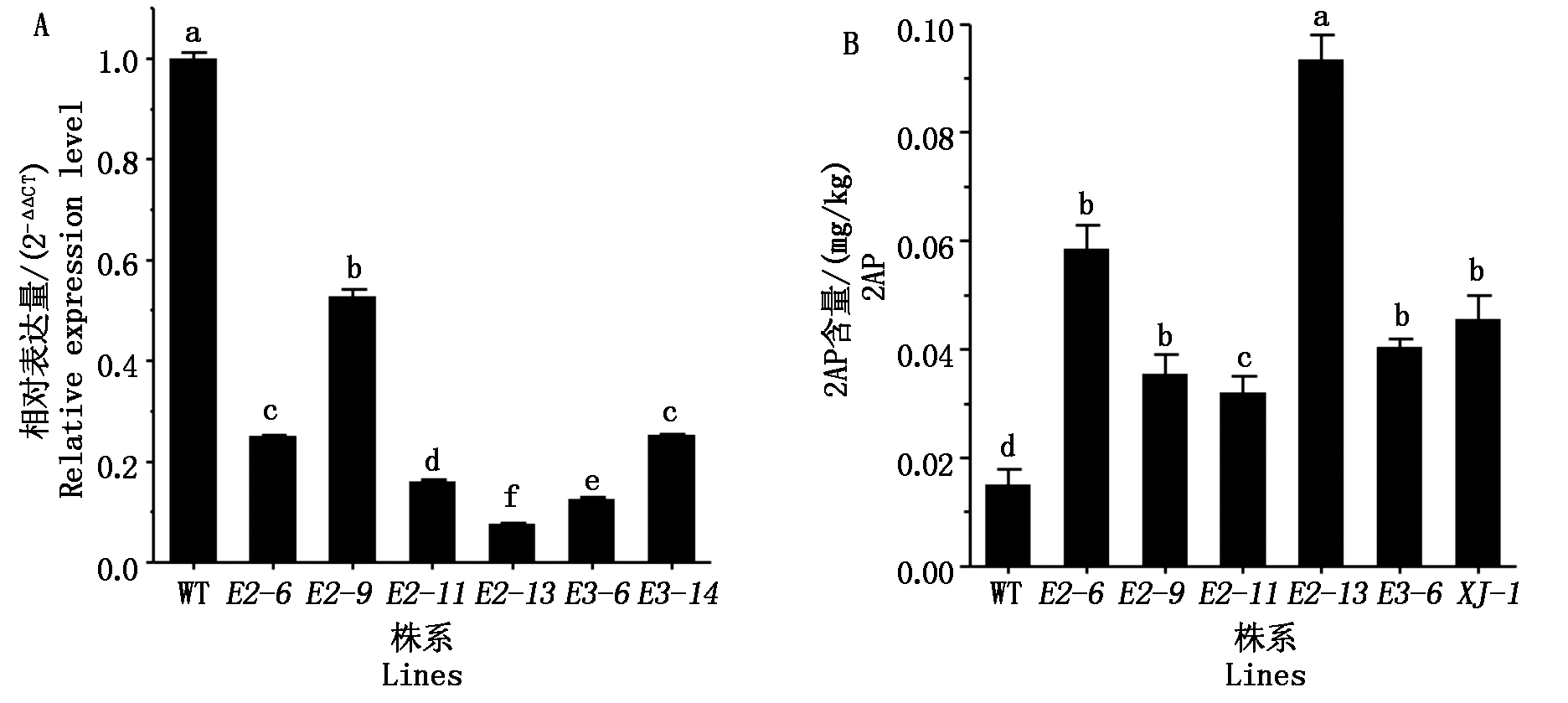
A.突变体Badh2基因的表达情况;B.突变体中香味物质(2AP)的含量测定。 XJ-1.信香粳1号。不同小写字母表示差异显著(P<0.05)。图4同。 A.The expression of Badh2 in the mutants; B.The content determination of aroma components (2AP). XJ-1.Xinxiangjing 1.Different lowercase letter indicate significant differences(P<0.05).The same as Fig.4.
图3 突变体中Badh2基因的表达情况及香味物质的含量测定
Fig.3 The expression of Badh2 in the mutants and the content determination of aroma components
2.4 突变体农艺性状调查
为了确定在编辑Badh2基因的同时是否对受体材料的其他农艺性状尤其是产量相关性状造成影响,调查了6个突变株系T1的株高、分蘖数和千粒质量。统计分析表明,株高和有效分蘖数没有差异,有3个株系的千粒质量在P<0.05水平差异显著(图4)。综合农艺性状的调查结果,筛选出3个株系(badh2-E2-6、badh2-E2-9、badh2-E3-6),在香味品质较野生型均显著改良的同时,其他主要农艺性状和受体保持一致,对这3个株系分单株套袋收种,用于后续的繁种和育种应用。
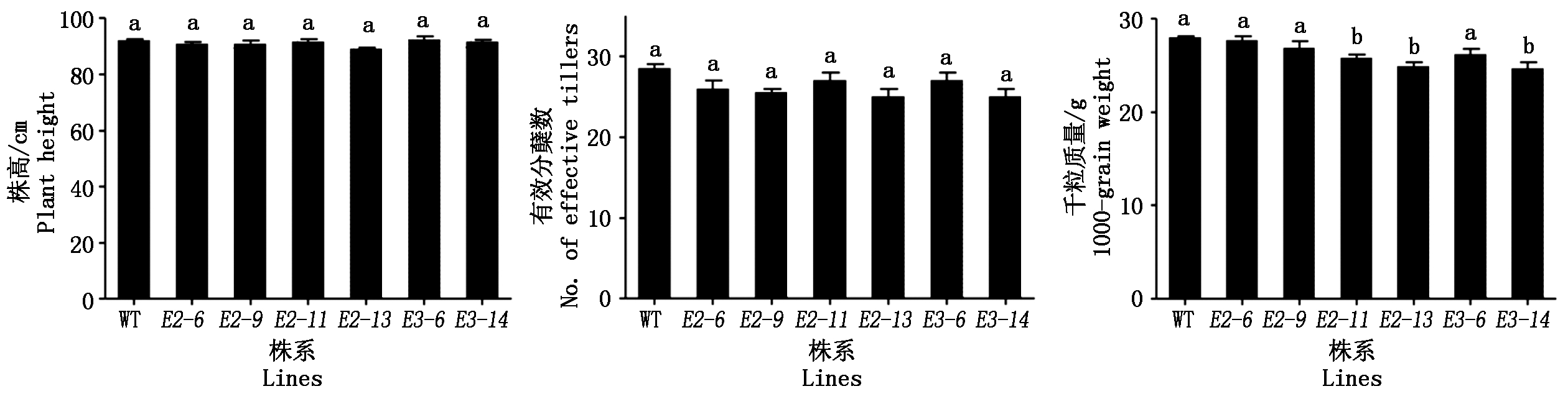
图4 T1突变体与野生型农艺性状调查
Fig.4 Investigation of agronomic traits in wild type and T1 mutants
2.5 无转基因序列遗留的突变体获得
转基因序列在突变体中的存在会对其继续进行基因编辑,为了得到能稳定遗传的转基因突变体,在T1中对无标记基因Hpt和载体骨架序列整合的单株进行筛选。总共调查了6个突变体的T1单株,绝大部分单株的转基因元件与突变靶点发生分离,共筛选到Cas9和Hpt基因检测阴性的单株10株(表2),badh2-E3-14株系没有获得无转基因序列的单株。进一步检测这些不含转基因序列的突变单株的突变情况,结果发现(图5),在T0表现为纯合突变的单株badh2-E2-6,其后代的突变与T0保持一致;突变株badh2-E2-9的T1 3个单株均为纯合突变(PCR产物测序峰图为单峰),其中2株表现为缺失7 bp,1株为缺失2 bp;badh2-E3-6的T1分别检测到2株纯合单株,均表现为插入1 bp(A/T),与T0突变类型保持一致。同时也发现突变株系badh2-E2-11和badh2-E2-13的T1与T0的突变没有保持一致,badh2-E2-11-3表现为缺失7 bp,badh2-E2-13检测到2种纯合单株,缺失19 bp和缺失50 bp,仍然存在大片段缺失(图5)。将10个筛选到的无转基因元件的纯合单株套袋收种。
表2 突变体的T1分离情况统计
Tab.2 Segregation of the T1 mutant
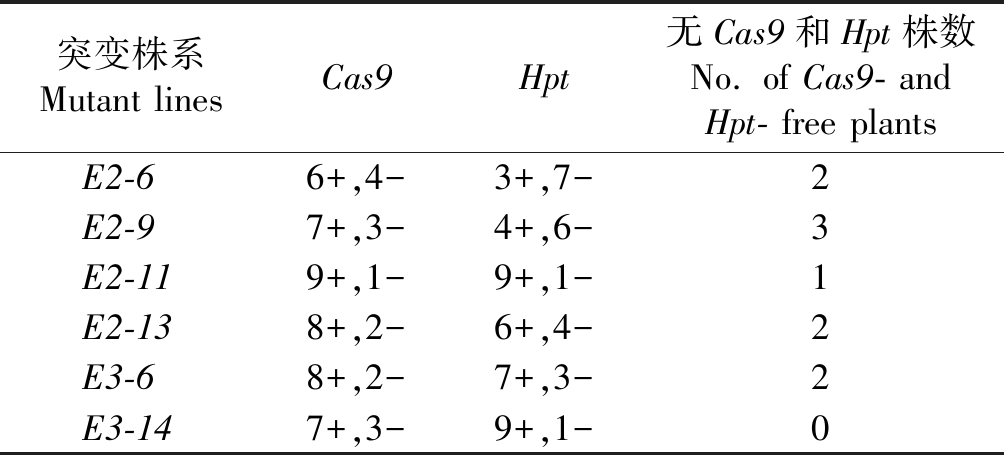
突变株系Mutant linesCas9Hpt无Cas9和Hpt株数No. of Cas9- and Hpt- free plantsE2-66+,4-3+,7-2E2-97+,3-4+,6-3E2-119+,1-9+,1-1E2-138+,2-6+,4-2E3-68+,2-7+,3-2E3-147+,3-9+,1-0
注:+.检测到;-.未检测到。
Note:+.Cas9 or Hpt detected;-.Cas9 or Hpt not detected.
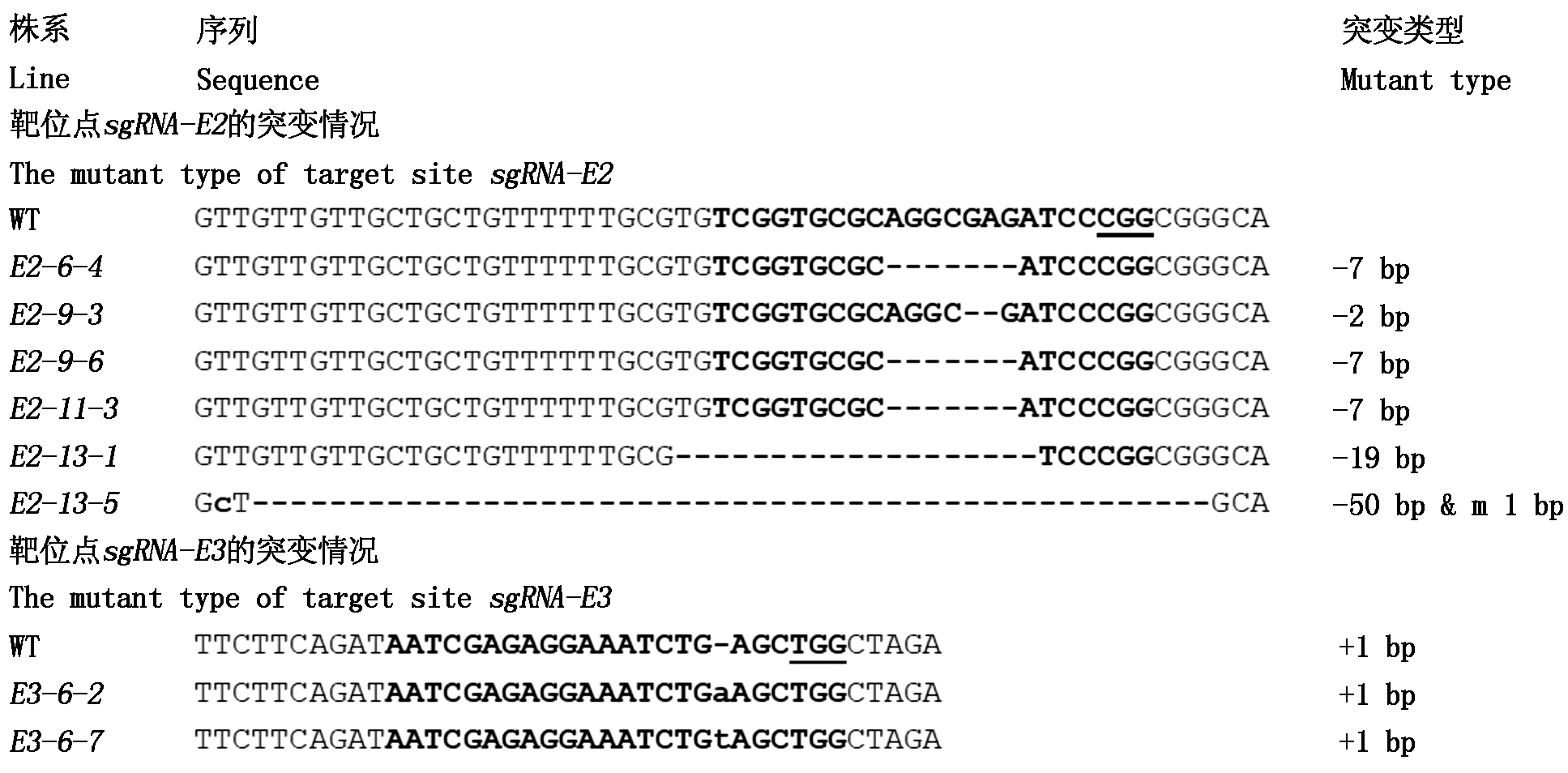
WT.野生型;E2-6-4、E2-9-3、E2-11-3、E2-13-1、E2-13-5、E3-6-2、E3-6-7.Badh2突变体的T1单株。m.碱基突变。 WT.Wild type; E2-6-4,E2-9-3,E2-11-3,E2-13-1,E2-13-5,E3-6-2,E3-6-7.T1 of Badh2 mutants. m.Base mutation.
图5 突变体T1靶位点测序结果
Fig.5 Sequencing results of target sites on T1 mutants
3 讨论与结论
稻米的蒸煮品质是水稻改良育种的一个主要方向,香味是其中一个最重要的食味品质。获得香稻的传统育种方法是通过与香稻品种进行杂交选育,再经过多年的回交获得性状稳定的纯合品种。此过程繁杂,而且费时费力,易与产量、抗病等性状产生连锁累赘,导致选育结果不理想。已有研究报道,利用RNAi和TALEN技术对水稻Badh2基因进行编辑[8, 19],有助于加快香稻育种进程。近年来兴起的CRISPR/Cas9技术能靶向编辑目标基因,快速稳定的获得可遗传的突变,可实现单碱基变异,可在转基因当代就可以获得纯合突变[20],该体系已经成功应用于动植物中,在水稻中的应用也日渐成熟[21]。
中国水稻育种经历高产、超高产到超级稻,单产不断突破,但是存在高产不优质的普通问题[22]。本研究中选用的受体材料是超级稻品种龙粳31,是我国年种植面积最大的超级稻品种,具有高产、抗病、抗倒伏等优良性状,但是稻米香味不明显。邵高能等[9]利用CRISPR/Cas9技术对中花11的Badh2基因进行编辑,获得在第1个外显子上突变1个碱基的突变体材料,香味物质含量显著提高。本研究通过CRISPR/Cas9技术对龙粳31的Badh2基因进行编辑,获得香味物质含量较受体显著提高的突变体,达到现有香稻的水平。本研究与邵高能等[9]的突变位点不同,分别在水稻Badh2基因的第2个内含子与第2个外显子间和第3个外显子上设计gRNA,以超级稻品种龙粳31为受体,进行靶向编辑,成功获得多种Badh2基因序列有变化的突变体,在转基因当代就获得纯合突变的单株badh2-E2-6和badh2-E2-11,且突变稳定遗传至T1。而突变体Badh2-E2-13在T0是双等位突变,表现为缺失64,108 bp,在T1则出现与T0不同的2种纯合突变株系,表现为19,50 bp的缺失,这与前人报道的结果一致,可能是T1仍存在gRNA的识别位点,从而再次对靶位点进行编辑[23-24],产生新的突变,到了T2以后转基因元件被剔除,突变即可稳定遗传[25]。因此,为了获得稳定的基因编辑系,有必要选择无转基因元件遗留的单株进行传代,在T1获得10个无转基因元件的株系以供后续研究。
通过改良香味基因来提高稻米香味物质含量已经被报道,但是提高的程度各不一样。有报道指出非香稻品种并非不含香味物质,而是香味不明显[26]。本研究中所用的受体材料本身的2AP含量有0.018 mg/kg,而突变体相对于受体提高了1~4倍,达到已报道的香稻水平[17],但没有邵高能、Shan等[8-9]报道的2AP含量高,这可能与受体材料本身特性有关。
突变Badh2基因可能会影响部分株系的产量,在本研究中调查了突变体T1植株的主要农艺性状,2AP含量最高的突变体Badh2-E2-13的千粒质量较野生型有显著降低,这可能与其大片段缺失有关,同时转基因过程也可能会影响突变株系的农艺性状。后期可以通过与野生型回交,结合香味物质含量和产量性状进行田间选育,优先地选择没有影响受体主要农艺性状的单株进行后续的育种应用,或者可以通过与野生型进行回交,选育出既保留龙粳31品种原有的优良性状,同时香味物质含量高的单株进行后续的应用。
本试验利用CRISPR/Cas9技术改良超级稻品种龙粳31的香味品质,对香味基因Badh2进行准确的基因编辑,获得了Badh2基因的表达量显著降低、香味物质含量显著提高且无转基因元件遗留的稳定突变株系,提高了龙粳31的经济价值,也为香稻品种选育提供了育种材料。
[1] Sakthivel K, Sundaram R M, Rani S N, Balachandran S M, Neeraja C N. Genetic and molecular basis of fragrance in rice[J]. Biotechnol Advances, 2009, 27(4): 468-473. doi: 10.1016/j.biotechadv.2009.04.001.
[2] Paule C M, Powers J J. Sensory and chemical examination of aromatic and nonaromatic rices[J]. Journal of Food Science, 1989, 54(2): 343-346. doi:10.1111/j.1365-2621.1989.tb03076.x.
[3] Buttery R G, Ling L C, Juliano B O, Turnbaugh J G. Cooked rice aroma and 2-acetyl-1-pyrroline[J]. Journal of Agricultural and Food Chemistry, 1983, 31(4): 823-826.doi:10.1021/jf00118a036.
[4] Gaj T, Gersbach C A, Barbas C F. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering[J]. Trends in Biotechnology, 2013, 31(7): 397-405. doi: 10.1016/j.tibtech.2013.04.004.
[5] Liu J, Gaj T, Patterson J T, Sirk S J, Barbas C F. Cell-penetrating peptide-mediated delivery of TALEN proteins via bioconjugation for genome engineering[J]. PLoS One, 2014, 9(1): e85755. doi:10.1371/journal.pone.0085755.
[6] Doudna J A, Charpentier E. The new frontier of genome engineering with CRISPR-Cas9[J]. Science, 2014, 346(6213): 1258096. doi: 10.1126/science.1258096.
[7] Bortesi L, Fischer R. The CRISPR/Cas9 system for plant genome editing and beyond[J]. Biotechnology Advances, 2015, 33(1): 41-52. doi: 10.1016/j.biotechadv.2014.12.006.
[8] Shan Q W, Zhang Y, Chen K L, Zhang K, Gao C X. Creation of fragrant rice by targeted knockout of the OsBADH2 gene using TALEN technology[J]. Plant Biotechnol J, 2015, 13(6):791-800. doi: 10.1111/pbi.12312.
[9] 邵高能,谢黎虹,焦桂爱,魏祥进,圣忠华,唐绍清,胡培松. 利用CRISPR-CAS9技术编辑水稻香味基因Badh2[J]. 中国水稻科学, 2017, 31(2): 216-222. doi: 10.16819/j.1001-7216.2017.6098.
Shao G N, Xie L H, Jiao G A, Wei X J, Sheng Z H, Tang S Q, Hu P S. CRISPR/CAS9-mediated editing of the fragrant gene Badh2 in rice[J]. Chinese Journal of Rice Science, 2017, 31(2): 216-222.
[10] Kovach M J, Calingacion M N, Fitzgerald M A, McCouch S R. The origin and evolution of fragrance in rice (Oryza sativa L.) [J]. Proc Natl AcadSci USA, 2009, 106(34): 14444-14449. doi: 10.1073/pnas.0904077106.
[11] Shao G N, Tang S Q, Chen M L, Wei X J, He J W, Luo J, Jiao G A, Hu Y C, Xie L H, Hu P S. Haplotype variation at Badh2, the gene determining fragrance in rice[J]. Genomics, 2012, 101(2): 157-162. doi: 10.1016/j.ygeno.2012.11.010.
[12] 李洪亮,柴永山,孙玉友,高春艳,魏才强,解忠,张巍巍,刘丹,程杜娟. 寒地超级稻龙粳31祖先亲本追溯及遗传基础解析[J]. 植物遗传资源学报, 2016, 17(3): 433-437. doi: 10.13430 /j.cnki.jpgr.2016.03.005.
Li H L,Chai Y S,Sun Y Y,Gao C Y,Wei C Q,Xie Z,Zhang W W,Liu D,Cheng D J. Ancestors tracking and analysis on genetic basis of super rice Longjing 31 in cold region[J]. Journal of Plant Genetic Resources, 2016, 17(3): 433-437.
[13] 刘殿宇,靳学慧,于连鹏,周戈力,国宝焕,张亚玲. 黑龙江省稻瘟病菌对主栽品种的致病性分析[J]. 黑龙江八一龙垦大学学报, 2018, 30(1): 8-11. doi: 10.3969/j.issn.1002-2090.2018.01.002.
Liu D Y,Jin X H,Yu L P,Zhou Y L,Guo B H,Zhang Y L. Analysis of the pathogenic of rice blast fungus on main rice varieties in Heilongjiang Province[J]. Journal of Heilongjiang Bayi Agricultural University, 2018, 30(1): 8-11.
[14] Gao Y B, Zhao Y D. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing[J]. Journal of Integrative Plant Biology, 2014, 56(4): 343-349.doi: 10.1111/jipb.12152.
[15] Livak K, Schmittgen T D. Analysis of relative gene expression data using Real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2002, 25(4): 402-408. doi: 10.1006/meth.2001.1262.
[16] Bergman C J, Delgado J T, Bryant R, Grimm C, Cadwallader K R, Webb B D. Rapid gas chromatographic technique for quantifying 2-acetyl-1-pyrroline and hexanal in rice (Oryza sativa L.) [J]. Cereal Chemistry, 2000, 77(4): 454-458. doi:10.1094/CCHEM.2000.77.4.454.
[17] 应兴华,徐霞,陈铭学,欧阳由男,朱智伟,闵捷.气相色谱、质谱技术分析香稻特征化合物2-乙酰基吡咯啉[J].色谱,2010,28(8):782-785.doi:10.3724/SP.J.1123.2010.00782.
Ying X H, Xu X, Chen M X, Ouyang Y N, Zhu Z W, Min J. Determination of 2-acetyl-1-pyrroline in aroma rice using gas chromatography-mass spectrometry[J]. Chinese Journal of Chromatography, 2010, 28(8): 782-785.
[18] Zhang H, Zhang J S, Wei P L, Zhang B T, Gou F, Feng Z Y, Mao Y F, Yang L, Zhang H, Xu N Y, Zhu J K. The CRISPR/Cas9 system produces specific and homozygous targeted gene editing in rice in one generation[J]. Plant Biotechnol J, 2014, 12(6): 797-807. doi: 10.1111/pbi.12200.
[19] Chen M L, Wei X J, Shao G N, Tang S Q, Luo J, Hu P S. Fragrance of the rice grain achieved via artificial microRNA-induced down-regulation of OsBADH2[J]. Plant Breeding, 2012, 131(5): 584-590. doi:10.1111/j.1439-0523.2012.01989.x.
[20] Zhang H, Zhang J S, Wei P L, Zhang B T, Gou F, Feng Z Y, Mao Y F,Yang L, Zhang H, Xu N F,Zhu J K. The CRISPR/Cas9 system produces specific and homozygous targeted gene editing in rice in one generation[J]. Plant Biotechnology Journal, 2014, 12(6): 797-807. doi:10.1111/pbi.12200.
[21] Miao J, Guo D S, Zhang J Z, Huang Q P, Qin G J, Zhang X, Wan J M, Gu H Y, Qu L J. Targeted mutagenesis in rice using CRISPR-Cas system[J]. Cell Research, 2013, 23(10): 1233-1236. doi:10.1038/cr.2013.123.
[22] Hsu Y C, Tseng M C, Wu Y P, Lin M Y, Wei F J, Hwu K K, Hsing Y I, Lin Y R. Genetic factors responsible for eating and cooking qualities of rice grains in a recombinant inbred population of an inter-subspecific cross[J]. Molecular Breeding, 2014, 34(2): 655-673. doi:10.1007/s11032-014-0065-8.
[23] Ma X L, Zhang Q Y, Zhu Q L, Liu W, Chen Y, Qiu R, Wang B, Yang Z F, Li H Y, Lin Y R,Xie Y Y,Shen R X,Chen S F,Wang Z,Chen Y L,Guo J X,Chen L T,Zhao X C,Dong Z C,Liu Y G. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants[J]. Molecular Plant, 2015, 8(8): 1274-1284. doi:10.1016/j.molp.2015.04.007.
[24] Xu R F, Li H, Qin R Y, Li J, Qiu C H, Yang Y C, Ma H, Li L, Wei P C, Yang J B. Generation of inheritable and ″transgene clean″ targeted genome-modified rice in later generations using the CRISPR/Cas9 system[J]. Scientific Reports, 2015, 5: 11491-11501. doi:10.1038/srep11491.
[25] Zhou H B,Liu B,Weeks D P, Spalding M H, Yang B. Large chromosomal deletions and heritable small genetic changes induced by CRISPR/Cas9 in rice[J]. Nucleic Acids Research, 2014, 42(17): 10903-10914. doi:10.1093/nar/gku806.
[26] Hinge V R, Patil H B, Nadaf A B. Aroma volatile analyses and 2AP characterization at various developmental stages in Basmati and Non-Basmati scented rice (Oryza sativa L.) cultivars[J]. Rice, 2016, 9(1): 1-22. doi:10.1186/s12284-016-0113-6.