肌钙蛋白(Troponin, Tn)是位于肌纤维蛋白细丝中的结构蛋白[1],也是一种与心脏和骨骼肌收缩相关的调节蛋白,能够影响家畜的肉质性状[2]。Tn可调节Ca2+介导的肌动蛋白和肌球蛋白之间的相互作用,由肌钙蛋白C(TnC)、肌钙蛋白T(TnT)和肌钙蛋白I(TnI)3个亚基组成。TnI是肌钙蛋白-原肌球蛋白调节复合物中的抑制性亚单位,可结合到肌动蛋白,阻止其与肌球蛋白的头部接触,也可抑制肌球蛋白酸的活性,使心肌和骨骼肌保持松弛状态[3]。TNNI 家族包括TNNI1、TNNI2和TNNI3 3种亚型,其表达具有组织特异性和发育调控模式[4]。杨华[5]研究表明,缓慢收缩的骨骼肌类型TnI(TNNI1)早期在心肌、慢收缩性骨骼肌纤维发育过程中均存在表达,但个体成年后,在慢收缩骨骼肌纤维中特异性表达;快收缩骨骼肌型TnI(TNNI2)在快速收缩的骨骼肌纤维中表达;心脏型TnI(TNNI3)是心肌表达的特异性基因。另外,在早期骨骼肌形成期间,TNNI1和TNNI2共同表达,但随着骨骼肌不断发育和成熟,TNNI1和TNNI2分别定向表达于骨骼肌慢肌纤维和快肌纤维。此外,TNNI1在氧化肌肉纤维中表达,常作为模型基因用于研究慢肌纤维的特异性表达机制[6-8]。
在人和鼠胚胎、胎儿期,TNNI1和TNNI3均能在心肌层表达,TNNI1的表达占主导地位[9]。个体出生后,TNNI3表达上调,TNNI1的表达下调。相关研究表明,TNNI1 mRNA和TNNI3 mRNA可通过RNA印迹在14~21日龄的大鼠心脏中检测到[10],但在出生后9个月的人心肌中仅能检测出TNNI3 mRNA[11]。动物体内试验表明,肌钙蛋白I基因家族与疾病密切相关。TNNI2蛋白是一种抑制血管生成活性和肿瘤转移的强特异性因子,且无明显的毒性作用[12]。与其他血管生成抑制因子相比,TNNI2蛋白具有更强的抑制活性。TNNI3基因异常与常见的3种遗传性心肌病有关,该基因序列中不同位点突变均可引起不同类型的心肌疾病,如扩张性心肌病(Dilated cardiomyopathy,DCM)由1处突变引起,限制性心肌病(Restrictive cardiomyopathy,RCM)由6处突变导致,肥厚性心肌病(Hypertrophicca cardiomyopathy,HCM)与26处突变有关[13]。此外,赵秀丽[14]研究发现,远端关节弯曲、弗里曼-谢尔登综合征(DA2A)、谢尔登·霍尔综合征(DA2B)及面部痉挛均由TNNI2位点突变导致,表明TNNI2对人骨骼和肌肉发育发挥重要作用,同时,TNNI1、TNNI2 在家畜肌肉脂肪沉积过程中也发挥重要调控作用[15-17]。TNNI1和TNNI3基因通过影响肌纤维直径直接影响肉质的嫩度[18]。
牦牛作为青藏高原农牧民主要的生产生活来源,是当地畜牧业经济发展不可缺少的重要畜种[19],深入研究牦牛肌钙蛋白的生物学特性及分子调控机制对高原畜牧业发展具有重要意义。本研究通过克隆类乌齐牦牛肌钙蛋白I基因家族的CDS区序列,运用多种生物信息学软件分析其蛋白质结构,预测其生物学功能,利用实时荧光定量PCR探究其在牦牛不同组织中的表达情况,旨在从分子水平初步分析肌钙蛋白I基因家族各成员的生物学特性,为深入研究其调控机制提供基础数据。
1 材料和方法
1.1 试验材料
选取西藏自治区昌都市类乌齐县0.5岁健康的类乌齐牦牛3头。屠宰后,迅速采集臀肌、心脏、肝脏、肺脏等组织样品,DEPC水冲洗干净后,用锡箔纸包装,迅速储存在液氮中。
TRIzol购自Invitrogen公司,DNA 聚合酶和dNTPs购自Thermo Scientific 公司,RNA 反转录试剂盒(PrimeScriptTM RT Reagent Kit with gDNA Eraser(Perfect Real Time))、pGEM-T Vector cloning kit、DH5α感受态细胞、2 000 bp Marker、TB Green Premix Ex Taq Ⅱ均购自TaKaRa 公司;DNA 纯化回收试剂盒购自北京天根生化科技有限公司;分析纯氯仿、异丙醇、无水乙醇均购自济南汇丰达化工有限公司。
1.2 试验方法
1.2.1 提取组织总RNA及cDNA合成 用TRIzol法提取各组织的总RNA,采用琼脂糖凝胶电泳检测RNA样品质量,NanoDrop 2000分光光度计测定其浓度及D260nm/280nm值。参照反转录试剂盒说明书将其反转录成cDNA,置于-20 ℃保存备用。
1.2.2 引物的设计与合成 根据GenBank上牛TNNI1、TNNI2和TNNI3基因序列,利用Primer 5.0 软件设计PCR扩增引物;基于得到的类乌齐牦牛TNNI基因家族CDS区序列及牦牛GAPDH 基因序列设计特异性荧光定量引物(表1)。引物由英潍捷基(上海)生物技术有限公司合成。
表1 肌钙蛋白Ⅰ基因家族引物序列信息
Tab.1 Troponin Ⅰ gene family primer sequence information
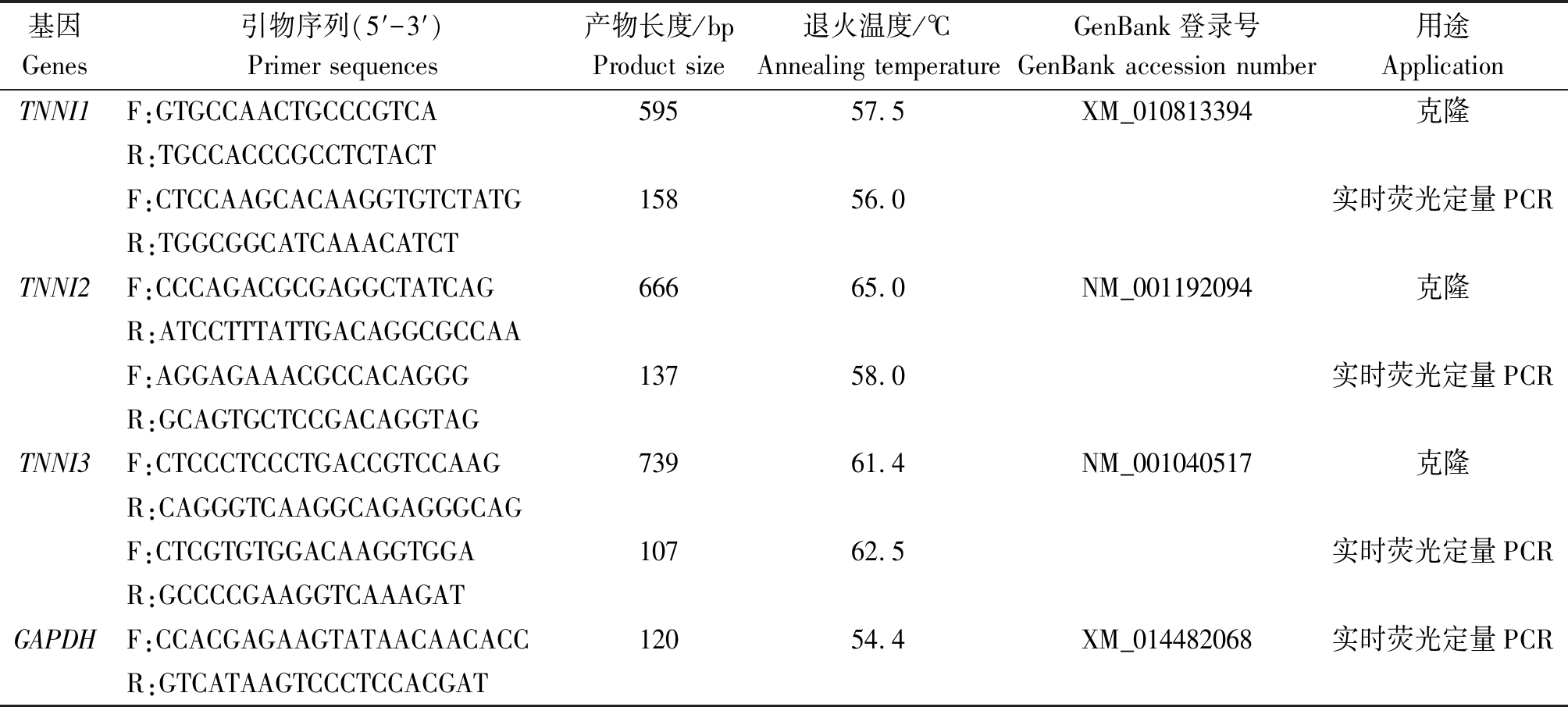
基因Genes引物序列(5′-3′)Primer sequences产物长度/bpProduct size退火温度/℃Annealing temperatureGenBank登录号GenBank accession number用途ApplicationTNNI1F:GTGCCAACTGCCCGTCA59557.5XM_010813394克隆R:TGCCACCCGCCTCTACTF:CTCCAAGCACAAGGTGTCTATG15856.0实时荧光定量PCRR:TGGCGGCATCAAACATCTTNNI2F:CCCAGACGCGAGGCTATCAG66665.0NM_001192094克隆R:ATCCTTTATTGACAGGCGCCAAF:AGGAGAAACGCCACAGGG13758.0实时荧光定量PCRR:GCAGTGCTCCGACAGGTAGTNNI3F:CTCCCTCCCTGACCGTCCAAG73961.4NM_001040517克隆R:CAGGGTCAAGGCAGAGGGCAGF:CTCGTGTGGACAAGGTGGA10762.5实时荧光定量PCRR:GCCCCGAAGGTCAAAGATGAPDHF:CCACGAGAAGTATAACAACACC12054.4XM_014482068实时荧光定量PCRR:GTCATAAGTCCCTCCACGAT
1.2.3 牦牛TNNI基因家族的克隆 以合成的cDNA为模板,通过PCR扩增目的基因。反应体系(25 μL):上下游引物各1 μL,cDNA模板1 μL,2×Taq PCR Master Mix 12.5 μL,ddH2O 9.5 μL。反应程序:94 ℃预变性5 min;94 ℃变性30 s,57.5 ℃退火30 s,72 ℃延伸35 s,共35个循环;72 ℃再延伸7 min,4 ℃保存。用1.0%琼脂糖凝胶电泳检测PCR产物,确认产物目的条带是所需的目的片段大小后,参照DNA纯化回收试剂盒说明书,回收PCR产物,储存在4 ℃备用。
按照pGEM-T载体试剂盒说明书进行连接,并转化至DH5α感受态细胞中,复苏后,均匀涂布在含Amp+的LB固体培养基表面,37 ℃倒置培养10~12 h;挑取白色单克隆菌落,筛选阳性菌液,送成都擎科梓熙生物技术有限公司测序。
1.2.4 牦牛TNNI基因家族的生物信息学分析 对类乌齐牦牛TNNI基因家族的CDS区进行序列分析、蛋白结构及功能预测(表2)。
表2 DNA和蛋白质序列数据分析工具
Tab.2 DNA and protein sequence data analysis tools
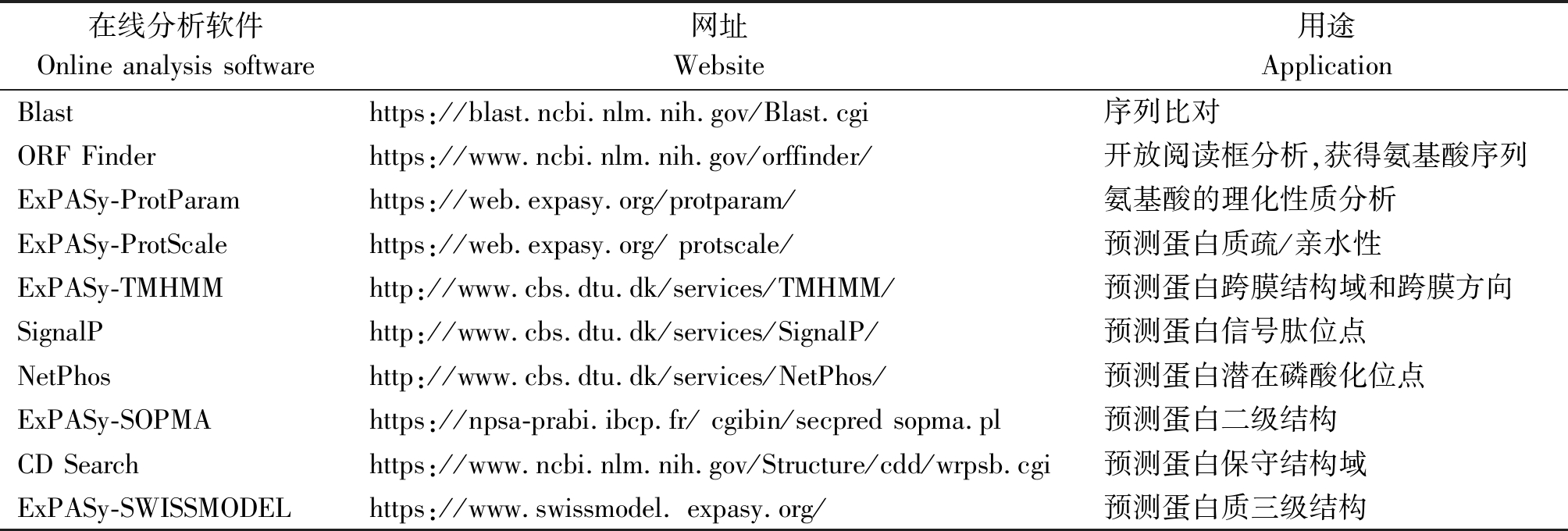
在线分析软件Online analysis software网址Website用途ApplicationBlasthttps://blast.ncbi.nlm.nih.gov/Blast.cgi序列比对ORF Finderhttps://www.ncbi.nlm.nih.gov/orffinder/开放阅读框分析,获得氨基酸序列ExPASy-ProtParamhttps://web.expasy.org/protparam/氨基酸的理化性质分析ExPASy-ProtScalehttps://web.expasy.org/ protscale/预测蛋白质疏/亲水性ExPASy-TMHMMhttp://www.cbs.dtu.dk/services/TMHMM/预测蛋白跨膜结构域和跨膜方向SignalPhttp://www.cbs.dtu.dk/services/SignalP/预测蛋白信号肽位点NetPhoshttp://www.cbs.dtu.dk/services/NetPhos/预测蛋白潜在磷酸化位点ExPASy-SOPMAhttps://npsa-prabi.ibcp.fr/ cgibin/secpred sopma.pl预测蛋白二级结构CD Searchhttps://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi预测蛋白保守结构域ExPASy-SWISSMODELhttps://www.swissmodel. expasy.org/预测蛋白质三级结构
1.2.5 牦牛TNNI基因家族的组织表达量检测 利用Primer 5.0软件设计肌钙蛋白I家族及内参基因GAPDH的实时荧光定量PCR特异性引物(表1)。反应体系(10 μL):TB Green Premix Ex Taq Ⅱ(2×)5 μL、上下游引物各0.4 μL、cDNA 模板1.0 μL、无RNAase 水3.2 μL。反应程序:95 ℃预变性30 s;95 ℃变性10 s,56 ℃退火30 s,65 ℃延伸5 s,共39 个循环;每个样品3个生物学重复,3个技术重复。以牦牛GAPDH为内参基因对组织进行定量结果分析,以溶解曲线来判定RT-qPCR 反应产物的特异性,得到每个样品的Ct值,利用2-ΔΔCt法计算各个样本的相对表达量,数据用平均值±标准误差表示,采用SPSS 18.0软件进行显著性分析,P<0.05表示差异显著,P>0.05表示差异不显著。
2 结果与分析
2.1 牦牛TNNI基因家族PCR扩增结果
以类乌齐牦牛臀肌组织cDNA为模板,扩增肌钙蛋白Ⅰ基因家族CDS区,经1%琼脂糖凝胶电泳检测,获得与预期牦牛TNNI1、TNNI2和TNNI3基因片段大小一致的单一清晰条带,大小分别为595,666,739 bp(图1)。
测序校对获得牦牛肌钙蛋白I基因家族TNNI1、TNNI2和TNNI3基因CDS区大小分别为564,549,639 bp,分别编码187,182,212个氨基酸残基(图2)。
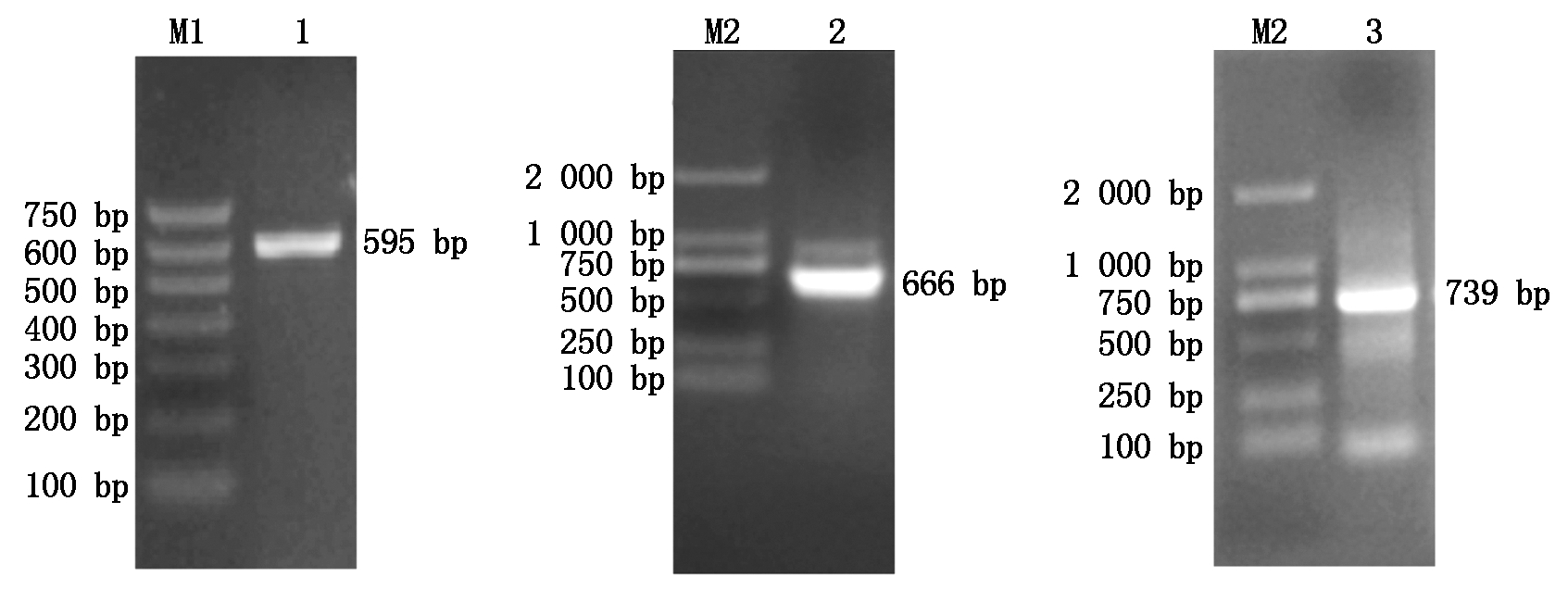
M1.DNA Marker DL750;M2.DNA Marker DL2000;1-3.TNNI1、TNNI2和TNNI3基因。 M1.DNA Marker DL750;M2.DNA Marker DL2000;1-3.TNNI1,TNNI2 and TNNI3 genes,respectively.
图1 肌钙蛋白I基因家族PCR扩增结果
Fig.1 PCR amplification results of the troponin I gene family

A-C.TNNI1、TNNI2和TNNI3基因。 A-C.TNNI1,TNNI2 and TNNI3 genes,respectively.
图2 肌钙蛋白I基因家族核苷酸、氨基酸序列
Fig.2 Troponin I gene family nucleotide and amino acid sequence
2.2 牦牛TNNI基因家族序列比对
从NCBI数据库中下载9个不同物种的肌钙蛋白I基因家族氨基酸序列,利用MegAlign软件对TNNI1、TNNI2和TNNI3基因氨基酸序列进行比对。结果显示,牦牛肌钙蛋白I基因家族在哺乳动物间高度保守(表3)。
2.3 牦牛TNNI基因家族系统进化分析
利用Mega 7.0软件对类乌齐牦牛、黄牛、水牛、人、小鼠、绵羊、黑猩猩、野猪、鸡和热带爪蟾的肌钙蛋白I基因家族氨基酸序列进行系统进化分析。结果表明,牦牛TNNI1基因与水牛的亲缘关系最密切,其次是绵羊,然后是黄牛,与野猪、人、黑猩猩等亲缘关系较远,与小鼠的亲缘关系最远;而TNNI2、TNNI3基因首先与黄牛聚在一起,其次是水牛,与其他亲缘关系较远(图3-5)。
表3 牦牛肌钙蛋白I基因家族与 其他物种序列相似性比对分析
Tab.3 Sequence homology analysis of yak troponin I gene family and other species %
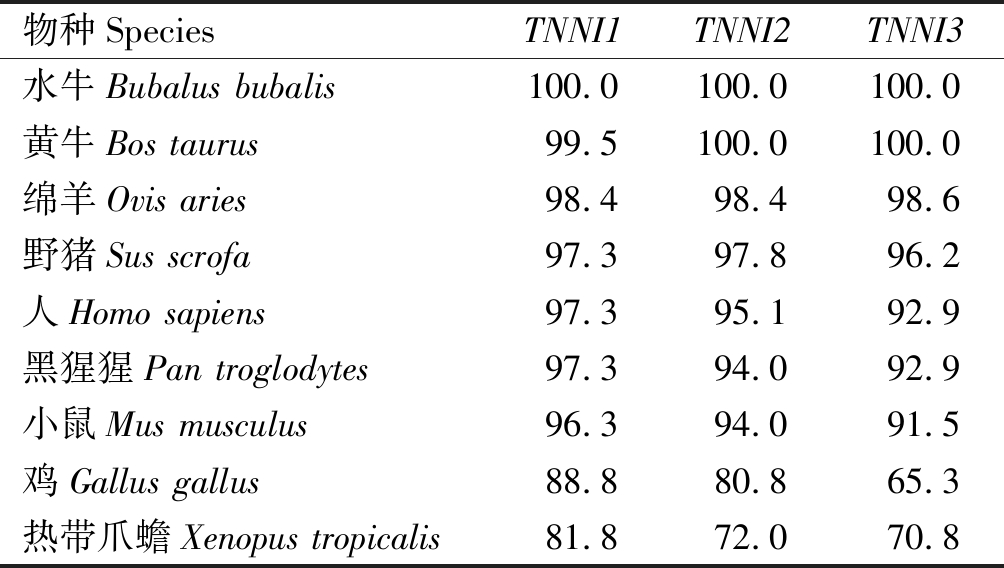
物种SpeciesTNNI1TNNI2TNNI3水牛Bubalus bubalis100.0100.0100.0黄牛Bos taurus99.5100.0100.0绵羊Ovis aries98.498.498.6野猪Sus scrofa97.397.896.2人Homo sapiens97.395.192.9黑猩猩Pan troglodytes97.394.092.9小鼠Mus musculus96.394.091.5鸡Gallus gallus88.880.865.3热带爪蟾Xenopus tropicalis81.872.070.8
2.4 牦牛TNNI基因家族蛋白结构和功能预测
2.4.1 蛋白质理化性质分析 利用在线软件ExPASy-ProtParam程序对牦牛肌钙蛋白I基因家族蛋白的理化性质进行预测。由表4可知,TNNI基因家族的蛋白等电点均大于 7,为偏碱性蛋白;TNNI1、TNNI2和TNNI3蛋白氨基酸出现频率最高的分别是赖氨酸(Lys)、谷氨酸(Glu)和精氨酸(Arg),最低的均是色氨酸(Trp);不稳定指数均大于40,说明TNNI家族蛋白结构不稳定;脂肪系数均大于60,说明TNNI基因家族蛋白流动性好。
2.4.2 疏水性、跨膜结构及信号肽预测 利用在线软件ExPASy-ProtScale对牦牛肌钙蛋白I基因家族的氨基酸序列进行亲/疏水性预测。牦牛TNNI基因家族蛋白中大多数氨基酸是亲水性的,结合ExPASy-ProtParam预测结果,TNNI1、TNNI2和TNNI3 蛋白均为亲水性蛋白。ExPASy-TMHMM 2.0预测表明TNNI1、TNNI2和TNNI3都不是跨膜蛋白。SignalP 4.1预测牦牛TNNI基因家族蛋白的信号肽位点,结果显示,TNNI基因家族蛋白没有信号肽,均为非分泌型蛋白。
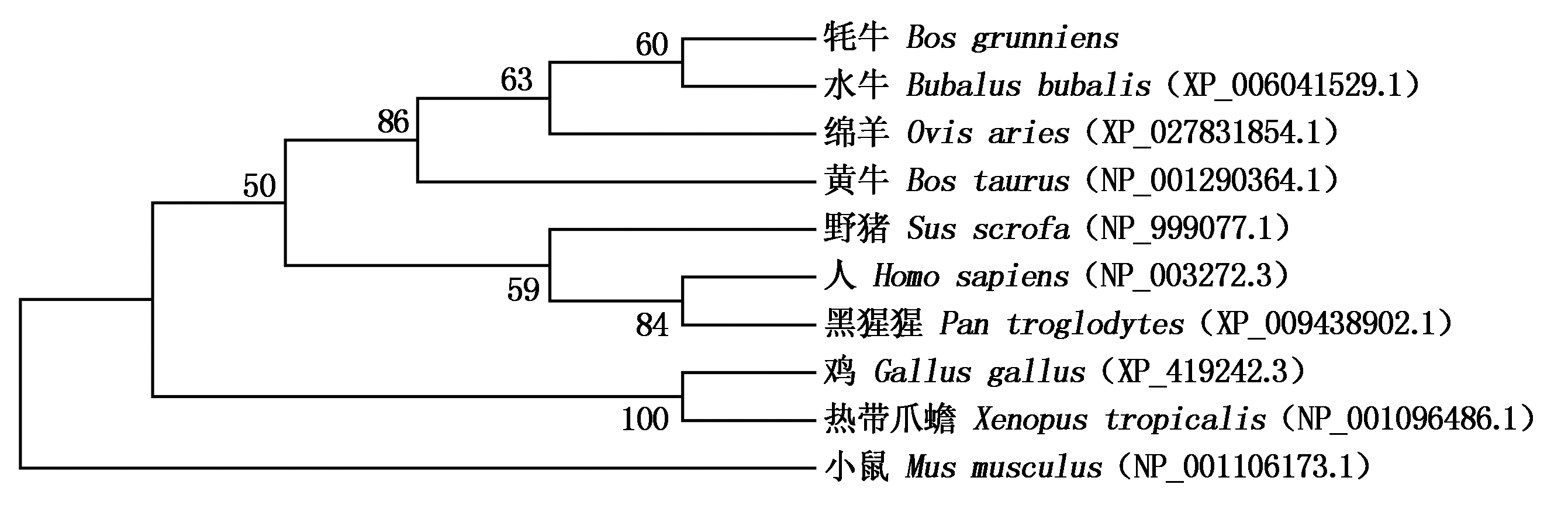
图3 TNNI1基因系统进化树
Fig.3 Phylogenetic tree of TNNI1

图4 TNNI2基因系统进化树
Fig.4 Phylogenetic tree of TNNI2
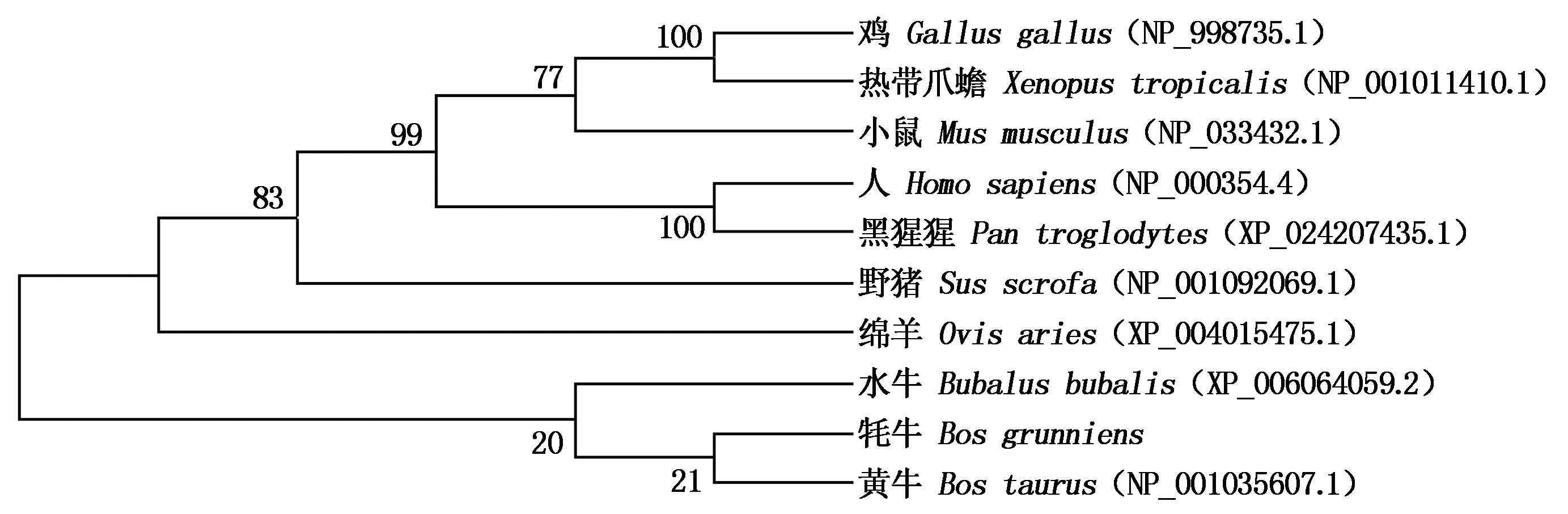
图5 TNNI3基因系统进化树
Fig.5 Phylogenetic tree of TNNI3
2.4.3 磷酸化位点及糖基化位点预测 磷酸化修饰是生物体内重要的翻译后修饰方式[20],蛋白质的磷酸化主要集中在肽链中的Tyr、Ser、Thr残基上。利用在线软件NetPhos 3.1预测牦牛肌钙蛋白I基因家族氨基酸上的磷酸化位点,显示阈值为0.5时,TNNI1蛋白有13个磷酸化位点,其中包括9个Ser磷酸化位点、3个Thr磷酸化位点和1个Tyr磷酸化位点;TNNI2蛋白预测到14个磷酸化位点,其中Ser磷酸化位点为7个,Thr磷酸化位点为5个,Tyr磷酸化位点为2个;TNNI3蛋白存在17个磷酸化位点,其中Ser磷酸化位点为10个(Ser24、Ser79、Ser152等),Thr磷酸化位点为6个,Tyr磷酸化位点为1个。
表4 牦牛肌钙蛋白Ⅰ家族蛋白的基本性质
Tab.4 Basic properties of yak troponin Ⅰ gene family proteins
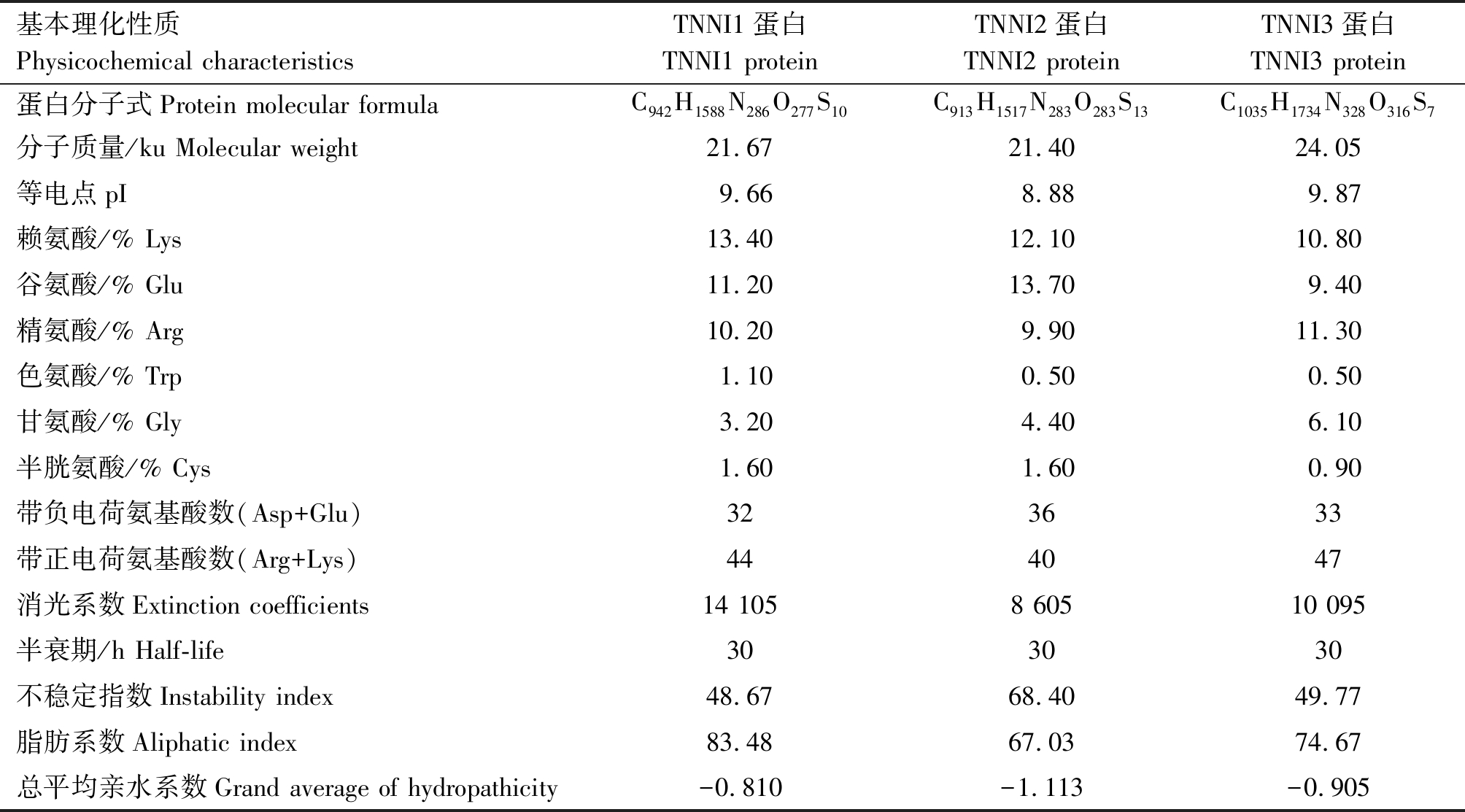
基本理化性质Physicochemical characteristicsTNNI1蛋白TNNI1 proteinTNNI2蛋白TNNI2 proteinTNNI3蛋白TNNI3 protein蛋白分子式Protein molecular formulaC942H1588N286O277S10C913H1517N283O283S13C1035H1734N328O316S7分子质量/ku Molecular weight21.6721.4024.05等电点pI9.668.889.87赖氨酸/% Lys13.4012.1010.80谷氨酸/% Glu11.2013.709.40精氨酸/% Arg10.209.9011.30色氨酸/% Trp1.100.500.50甘氨酸/% Gly3.204.406.10半胱氨酸/% Cys1.601.600.90带负电荷氨基酸数(Asp+Glu)323633带正电荷氨基酸数(Arg+Lys)444047消光系数Extinction coefficients14 1058 60510 095半衰期/h Half-life303030不稳定指数Instability index48.6768.4049.77脂肪系数Aliphatic index83.4867.0374.67总平均亲水系数Grand average of hydropathicity-0.810-1.113-0.905
蛋白质中有2种类型的糖基化位点,即N-糖基化位点和O-糖基化位点;Asn氨基上主要含N-糖基化位点,O-糖基化位点则主要存在于Ser和Thr的羟基上[21-22]。分别利用在线软件ExPASy中NetOGlyc 4.0和NetNGlyc 1.0进行O、N糖基化位点预测。分析结果表明,TNNI1蛋白存在5个O-糖基化位点,未检测到N-糖基化位点;TNNI2蛋白含有3个O-糖基化位点,未检测到N-糖基化位点;TNNI3蛋白存在13个O-糖基化位点(其中包括第152位)和1个N-糖基化位点。
2.4.4 保守结构域预测 蛋白质的保守结构域是在蛋白质的高级结构中,具有特殊遗传来源和功能的结构域[23-24]。利用NCBI-CD Search对牦牛肌钙蛋白I基因家族的氨基酸序列保守结构域进行预测(图6-8)。结果表明,TNNI1、TNNI2和TNNI3基因编码蛋白均含有肌钙蛋白超家族(Troponin superfamily)保守结构域;且TNNI3在1-32位含有肌钙蛋白I_N端超家族(Troponin-I_N superfamily)保守结构域,该超家族代表肌钙蛋白I的心脏N-端延伸,蛋白质的这个区域(1-32)可与cTnC的N-端相互作用,调节肌丝Ca2+的敏感性[25]。

图6 牦牛TNNI1基因编码蛋白保守结构域预测
Fig.6 Conserved domain prediction of yak TNNI1 gene encoding protein

图7 牦牛TNNI2基因编码蛋白保守结构域预测
Fig.7 Conserved domain prediction of yak TNNI2 gene encoding protein
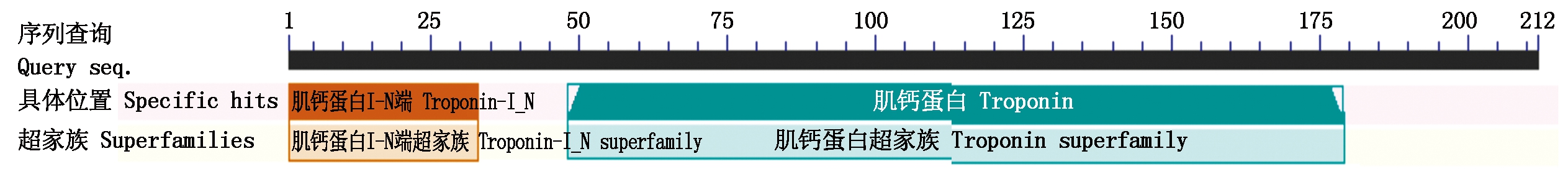
图8 牦牛TNNI3基因编码蛋白保守结构域预测
Fig.8 Conserved domain prediction of yak TNNI3 gene encoding protein
2.4.5 二级结构及三级结构预测 利用在线软件ExPASy-SOPMA预测牦牛肌钙蛋白I基因家族蛋白的二级结构(表5), TNNI1与TNNI3均由α-螺旋(Alpha helix)、无规则卷曲(Random coil)、β-转角(Beta turn)和延伸链(Extended strand)构成,且都以α-螺旋结构为主;而TNNI2蛋白的二级结构以α-螺旋为主,还有少量的无规则卷曲和β-转角。
利用在线软件ExPASy-SWISS-MODEL,基于PDB数据库预测牦牛肌钙蛋白I基因家族蛋白的三级结构模型(图9)。结果显示,牦牛TNNI家族蛋白三级结构均以α-螺旋为主,TNNI2蛋白还含有少量的无规则卷曲。
表5 牦牛TNNI基因家族蛋白的二级结构预测
Tab.5 Secondary structure prediction of yak TNNI gene family proteins %

二级结构类型Secondary structure typeTNNI1TNNI2TNNI3α-螺旋Alpha helix75.9478.5768.40延长链Extended strand0.530.001.89β-转角Beta turn2.673.853.30无规则卷曲Random coil20.8617.5826.42

A-C.TNNI1、TNNI2、TNNI3蛋白。 A-C.TNNI1,TNNI2 and TNNI3 proteins.
图9 牦牛TNNI基因家族蛋白的三级结构预测
Fig.9 Prediction of tertiary structure of yak TNNI gene family proteins
2.5 牦牛TNNI基因家族组织表达量分析
采用实时荧光定量PCR对TNNI家族各个基因进行不同组织间的差异表达分析,结合SPSS 18.0进行单因素方差分析(表6)。结果显示,在不同组织间,TNNI1基因的mRNA在臀肌、心脏中的表达量显著高于肺脏和肝脏(P<0.05);TNNI2 基因在臀肌中的表达量显著高于心脏、肝脏和肺脏(P<0.05);而TNNI3基因在心脏中表达量较高,而在肺脏、臀肌和肝脏中少量表达。在不同的基因中,TNNI1和TNNI2在臀肌中的表达量显著高于TNNI3(P<0.05),TNNI1在心脏、肝脏和肺脏中的表达量显著高于TNNI2(P<0.05),3个基因在肝脏中均有不同程度的表达,具体表现为TNNI1>TNNI2>TNNI3。
表6 牦牛肌钙蛋白Ⅰ家族基因在不同组织中的mRNA表达量
Tab.6 mRNA expression of yak troponin Ⅰ family genes in different tissues

基因Genes臀肌Gluteus心脏Heart肝脏Liver肺脏LungTNNI10.76±0.34Aa0.50±0.30Aa0.005 4±0.002 5Ab0.17±0.058AbTNNI20.82±0.24Aa8.43E-04±6.50E-04Bb2.15E-03±6.53E-04Bb0.021±0.002 4BbTNNI37.16E-05±6.52E-05Ba0.62±0.43Ab3.61E-05±1.54E-05Ca0.001 8±0.001 3Ba
注:同行不同小写字母表示组织间差异显著(P<0.05);同列不同大写字母表示基因间差异显著(P<0.05)。
Note:The different lowercase letters of the peer indicate significant difference among different tissues(P<0.05);The different uppercase letters in the same column indicate significant difference among different genes(P<0.05).
3 讨论与结论
目前,人类[6-7,26]、鸭[27]、猪[5]、羊[20,28]等多个物种的肌钙蛋白基因已被成功克隆,但对牦牛肌钙蛋白I基因家族的研究相对较少,本研究进行了牦牛肌钙蛋白I基因家族的克隆、生物信息学分析及组织表达分析。序列同源性分析、系统进化树在一定程度上反映了物种间的遗传关系[29]。通过对比类乌齐牦牛、黄牛、水牛、人、小鼠、绵羊、黑猩猩、野猪、鸡和热带爪蟾的氨基酸序列,发现牦牛肌钙蛋白I家族基因在哺乳动物间具有高度的保守性,表明TNNI 家族基因在Ca2+调控的心肌和骨骼肌收缩中具有功能性的保守性[5]。系统进化树结果表明,牦牛TNNI1基因先与水牛聚到一类,其次是绵羊,然后是黄牛,而TNNI2、TNNI3基因则与黄牛和水牛亲缘关系较近,与其他亲缘关系较远,综合说明,类乌齐牦牛TNNI基因家族与水牛和黄牛的亲缘关系较近。
谷氨酸在生物体内的蛋白质代谢过程中发挥重要作用,参与动物、植物和微生物中多种重要化学反应,同时是重要的鲜味剂,对香味具有增强作用。本研究显示牦牛TNNI 家族蛋白谷氨酸含量为11.20%,13.70%,9.40%,谷氨酸含量越高,肉品质越好,且易消化吸收。胶原蛋白是人体骨骼,尤其是软骨组织中的重要组成成分,只有摄入足够的胶原蛋白,钙才能更快地被人体消化吸收,并迅速到达骨骼部位进行沉积[30-32]。胶原蛋白的水解产物含有多种氨基酸,其中甘氨酸(Gly)含量最高,动物组织是人们获取天然胶原蛋白的主要途径,本研究显示,类乌齐牦牛TNNI 蛋白甘氨酸含量为3.2%,4.4%,6.1%,表明类乌齐牦牛是一个潜在的胶原蛋白源。跨膜蛋白基于多肽链以α-螺旋构象通过磷脂双层的次数,可分为单次和多次跨膜蛋白[23],且根据蛋白质的疏水性,也可帮助预测蛋白是否为跨膜螺旋。牦牛TNNI基因家族蛋白质中大多数氨基酸均属亲水性的,表明它们在膜内侧不稳定,不利于形成跨膜蛋白,可推测TNNI1、TNNI2和TNNI3均不是跨膜蛋白,与跨膜结构预测的结果一致。
蛋白质翻译后修饰在调节TNNI 家族基因的结构和功能中起主要作用[33]。丝氨酸磷酸化主要是变构蛋白质,影响蛋白质的生理活性和功能,这可能是TNNI蛋白在磷酸化过程中,改变构象和酶活性的原因。酪氨酸磷酸化则会促使蛋白质亚基的聚合,形成多蛋白复合体,是肌钙蛋白复合物行使功能的主要结构亚基[28]。本试验预测到TNNI1 蛋白有13个磷酸化位点,TNNI2 蛋白有14个磷酸化位点,TNNI3 蛋白有17个磷酸化位点。TNNI3 的磷酸化位点与其对心肌的收缩调节有关,当体内肾上腺素水平升高时,细胞内的蛋白激酶使TNNI3 蛋白磷酸化,从而调节心脏的收缩[28]。TNNI3是细胞内蛋白酶的底物,对μ-钙蛋白酶和m-钙蛋白酶具有敏感性,通过μ-钙蛋白酶的降解调控TNNI3的磷酸化,其中PKA的磷酸化降低了灵敏度,而PKC的磷酸化增加了对μ-钙蛋白酶蛋白水解的敏感性[34]。在小鼠心脏中,PKC-βⅡ对TNNI3的Thr 143的磷酸化增加了肌丝Ca2+的敏感性[35];但另一研究观察到Thr 143磷酸化不会改变Ca2+敏感性,而是会抑制细丝的协同激活[36]。本研究发现 TNNI3 Ser 152既是磷酸化位点,也是糖基化位点,该位点的糖基化与心肌型肌钙蛋白I对Ca2+敏感性有关[37]。由于AMPK是细胞能量学的关键调节因子,Ser 152的磷酸化可能与心脏和骨骼肌能量剥夺的适应机制有关。TNNI3蛋白有2个位点为半胱氨酸,易发生氧化或还原反应,这类反应可影响TNNI3蛋白的分子结构和抗原性,从而影响某些抗体的识别能力。
与TNNI1、TNNI2相比,类乌齐牦牛的TNNI3蛋白的N端延伸出32个氨基酸残基。成人心脏中TNNI3的特异性N末端延伸是一种调节结构,N末端延伸包含关键的PKA磷酸化位点,并在调节TNNI3的整体分子构象和功能中起作用[38]。相关研究表明,TNNI3的N末端延伸是针对舒张性心力衰竭靶向治疗的潜在位点[39]。本研究中二级结构预测结果表明,牦牛肌钙蛋白I家族基因蛋白都是α-螺旋为主,这有助于肌肉纤维肥大,从而有利于动物躯体肌肉增大、增粗[40];且含有较高的无规则卷曲和一定比例的β-转角,无规则卷曲被认为是构成酶活性部位以及其他功能部位的前提条件[20],TNNI1、TNNI2和TNNI3 3个蛋白的无规卷曲含量都较高,表明他们具有形成功能部位的条件。TNNI1和TNNI3还含有少量的延长链,而TNNI2没有延长链,这可能与其功能相关。
实时荧光定量PCR结果显示, TNNI1 基因在各组织中均有表达,依次为臀肌、心脏、肺脏、肝脏,与Yang等[41]发现TNNI1在猪小肠、肾脏组织中的表达量最高,都显著高于心脏;Sun 等[42]发现TNNI1在羊心脏中的表达量显著高于肌肉组织,而在肺脏、肝脏、肾脏中的表达量很低的研究结果存在一定差异,说明TNNI1 基因的组织表达具有物种特异性;也可能是因为类乌齐牦牛常年放养于海拔3 700 m以上的草甸草原地区,耐粗、耐劳、耐寒,其骨骼肌较为发达,以适应高寒生态环境,故臀肌等骨骼肌肌纤维含量非常丰富。TNNI2 基因在臀肌中的表达量显著高于心脏、肝脏和肺脏(P<0.05),与吴婷婷[20]、张阳[28]的研究结果相符。有研究报道,TNNI2 在小鼠血管中的平滑肌细胞中显著表达[43],在主动脉、膀胱、支气管的平滑肌中也会表达[44],但总体而言,TNNI2主要表达在快肌纤维中。TNNI3 基因在牦牛的心脏中高表达,在肺脏中微量表达,在臀肌和肝脏中几乎不表达,说明TNNI3 基因具有心脏表达特异性,这与Dhoot等[45]发表的人TNNI3 基因只在心肌内表达大致相符。
本研究首次克隆获得牦牛肌钙蛋白Ⅰ基因家族的CDS区序列,系统进化树分析显示,牦牛TNNI家族与黄牛、水牛的亲缘关系最近,实时荧光定量PCR结果表明,TNNI1和TNNI2在臀肌中的表达量最高,TNNI3在心脏中特异性表达,为研究肌钙蛋白Ⅰ基因家族在调控牦牛肌肉生长发育、致病机理方面提供了数据基础。
[1] Robaszkiewicz K, Ostrowska Z, Cyranka-Czaja A, Moraczewska J. Impaired tropomyosin-troponin interactions reduce activation of the actin thin filament[J]. Biochimica et Biophysica Acta (BBA)-Proteins and Proteomics, 2015, 1854(5):381-390. doi: 10.1016/j.bbapap.2015.01.004.
[2] Sepúlveda M, Gonano L, Viotti M, Morell M, Blanco P, Alarcón M L, Ramos I P, Carvalho A B, Medei E, Petroff M V. Calcium/calmodulin protein kinase Ⅱ-dependent ryanodine receptor phosphorylation mediates cardiac contractile dysfunction associated with sepsis[J]. Critical Care Medicine, 2017, 45(4):399-408. doi: 10.1097/CCM.0000000000002101.
[3] Wong S, Feng H Z, Jin J P. The evolutionarily conserved C-terminal peptide of troponin I is an independently configured regulatory structure to function as a myofilament Ca2+-desensitizer[J]. J Mol Cell Cardiol,2019,136:42-52. doi: 10.1016/j.yjmcc.2019.09.002.
[4] Sheng J J, Jin J P. TNNI1, TNNI2 and TNNI3: Evolution, regulation, and protein structure function relationships[J]. Gene, 2016, 576(1):385-394. doi: 10.1016/j.gene.2015.10.052.
[5] 杨华.猪肌钙蛋白Ⅰ基因家族的基因结构、表达谱和多态性分析[D].武汉:华中农业大学,2009.
Yang H. Porcine troponin I gene family: Genomic structure,expression profiling and polymorphism analysis[D].Wuhan:Huazhong Agricultural University,2009.
[6] Sun Y, Wang G Z, Ji Z B, Chao T L, Liu Z H, Wang X L, Liu G Q, Wu C H, Wang J M. Three slow skeletal muscle troponin genes in small-tailed Han sheep (Ovis aries): molecular cloning, characterization and expression analysis[J]. Molecular Biology Reports, 2016, 43(9):999-1010. doi: 10.1007/s11033-016-4027-6.
[7] De Matteis A, dell′Aquila M, Maiese A, Frati P, La Russa R, Bolino G, Fineschi V. The Troponin-I fast skeletal muscle is reliable marker for the determination of vitality in the suicide hanging[J].Forensic Science International, 2019, 301:284-288. doi: 10.1016/j.forsciint.2019.05.055.
[8] Anderson C M, Hu J X, Thomas R, Gainous T B, Celona B, Sinha T, Dickel D E, Heidt A B, Xu S M, Bruneau B G, Pollard K S, Pennacchio L A, Black B L. Cooperative activation of cardiac transcription through myocardin bridging of paired MEF2 sites[J]. Development, 2017, 144(7):1235-1241. doi: 10.1242/dev.138487.
[9] Kracklauer M P, Feng H Z, Jiang W R, Lin J L, Lin J J, Jin J P. Discontinuous thoracic venous cardiomyocytes and heart exhibit synchronized developmental switch of troponin isoforms[J]. FEBS Journal, 2013, 280(3):880-891. doi: 10.1111/febs.12076.
[10] Murphy A M, Jones L, Sims H F, Strauss A W. Molecular cloning of rat cardiac troponin I and analysis of troponin I isoform expression in developing rat heart[J]. Biochemistry, 1991, 30(3):707-712. doi: 10.1021/bi00217a018.
[11] Bhavsar P K, Brand N J, Yacoub M H, Barton P J. Isolation and characterization of the human cardiac Troponin I gene (TNNI3)[J]. Genomics, 1996, 35(1):11-23. doi: 10.1006/geno.1996.0317.
[12] Moses M A, Wiederschain D, Wu I M, Fernandez C A, Ghazizadeh V, Lane W S, Flynn E, Sytkowski A, Tao T, Langer R. Troponin I is present in human cartilage and inhibits angiogenesis[J]. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(6):2645-2650. doi: 10.1073/pnas.96.6.2645.
[13] Ahmad F, Seidman J G, Seidman C E. The genetic basis for cardiac remodeling[J].Annu Rev Genomics Hum Genet,2005, 6(6):185-216.doi: 10.1146/annurev.genom.6.080604.162132.
[14] 赵秀丽.不同肢端畸形中 HOXD13、TNNI2 和TPM2 基因的突变鉴定[D].北京:中国协和医科大学,2006.
Zhao X L. Molecular genetics of limb malformations:mutation identification in HOXD13,TNNI2 and TPM2[D].Beijing: Peking Union Medical College, 2006.
[15] 杨树猛,张海滨,刘汉丽,杨勤,余群力.蕨麻猪血清中激素含量的分析及其与肉质相关性研究[J]. 中国畜牧兽医文摘,2007,1:79-80.
Yang S M, Zhang H B, Liu H L, Yang Q, Yu Q L. Analysis of hormone content in serum of Juerna pig and its correlation with meat quality[J]. Chinese Abstracts of Animal Husbandry and Veterinary Medical,2007,1:79-80.
[16] 朱贵明.秦川牛肉质性状的系统研究[D].杨凌:西北农林科技大学,2003.
Zhu G M. The research on meat quality of Qinchuan cattle[D]. Yangling:Northwest A&F University, 2003.
[17] Li M Z, Li X W, Zhu L, Teng X K, Xiao H H, Shuai S R, Chen L, Li Q, Guo Y J. Differential expression analysis and regulatory network reconstruction for genes associated with muscle growth and adipose deposition in obese and lean pigs[J]. Progress in Natural Science, 2008, 18(4):387-399. doi: 10.1016/j.pnsc.2007.10.011.
[18] 孙艳.小尾寒羊三个慢型骨骼肌肌钙蛋白的基因克隆、结构特征及组织表达分析[D].泰安:山东农业大学,2016.
Sun Y. Molecular cloning,characterization and tissue expression analysis of three slow skeletal muscle troponin genes in small-tailed Han sheep[D].Taian:Shandong Agricultural University,2016.
[19] Song Q Q, Chai Z X, Xin J W, Zhao S J, Ji Q M, Zhang C F, Ma Z J, Zhong J C. Genetic diversity and classification of Tibetan yak populations based on the mtDNA COⅢ gene[J]. Genetics and Molecular research, 2015, 14(1):1763-1770. doi: 10.4238/2015.March.13.3.
[20] 吴婷婷. 天府肉羊TNNI1和TNNI3基因的克隆、表达及其与肌肉组织学性状相关性研究[D]. 雅安:四川农业大学, 2013.
Wu T T. Cloning and expression of Tianfu goat TNNI1 and TNNI3 gene and their relationship with muscle tissue traits[D]. Yaan:Sichuan Agricultural University, 2013.
[21] Karabulut N P, Frishman D. Sequence-and structure-based analysis of tissue-specific phosphorylation sites[J]. PLoS One, 2016, 11(6):e0157896. doi: 10.1371/journal.pone.0157896.
[22] Yang H Y, Zhu Q, Zhou N D, Tian Y P. Optimized expression of prolyl aminopeptidase in Pichia pastoris and its characteristics after glycosylation[J]. World Journal of Microbiology and Biotechnology, 2016, 32(11):176. doi: 10.1007/s11274-016-2135-z.
[23] 赵伯阳. 山羊CAPN1基因和CAST基因Ⅱ型转录本克隆及在不同组织中的表达[D]. 雅安:四川农业大学, 2011.
Zhao B Y. cDNA cloning and tissue expression of CAPN1 gene and CAST transcript Ⅱ in goat[D]. Yaan:Sichuan Agricultural University,2011.
[24] 苏强强. 绵羊MYOZ2和MYOZ3基因的克隆及在凉山半细毛羊组织的时空表达[D]. 雅安:四川农业大学, 2011.
Su Q Q. Cloning of MYOZ2,MYOZ3 gene in Ovine and tissues with time expression in Liangshan semi-fine wool sheep[D]. Yaan:Sichuan Agricultural University,2011.
[25] Wei H G, Jin J P. NH2-terminal truncations of cardiac troponin I and cardiac troponin T produce distinct effects on contractility and calcium homeostasis in adult cardiomyocytes[J]. Am J Physiol Cell Physiol, 2015, 308(5):397-404. doi: 10.1152/ajpcell.00358.2014.
[26] Bollen I A E, Schuldt M, Harakalova M, Vink A, Asselbergs F W, Pinto J R, Krüger M, Kuster D W D, van der Velden J. Genotype-specific pathogenic effects in human dilated cardiomyopathy[J]. The Journal of Physiology, 2017, 595(14):4677-4693. doi: 10.1113/JP274145.
[27] 姬改革, 束婧婷, 单艳菊, 章明, 屠云洁, 巨晓军, 刘一帆. 高邮鸭TNNI1基因克隆及组织表达分析[J]. 农业生物技术学报, 2018,26(6): 970-977. doi: 10.3969/j.issn.1674-7968.2018.06.007.
Ji G G, Shu J T, Shan Y J, Zhang M, Tu Y J, Ju X J, Liu Y F. Molecular cloning and tissue expression analysis of TNNI1 gene in Gaoyou duck (Anas platyrhynchos)[J]. Journal of Agricultural Biotechnology, 2018,26(6): 970-977.
[28] 张阳. 小尾寒羊TNNI2和TNNI3基因的克隆、结构分析及组织表达分析[D].泰安:山东农业大学,2016.
Zhang Y. Molecular cloning,characterization and tissue expression analysis of the TNNI2 and TNNI3 genes from small-tailed Han Sheep[D]. Taian:Shandong Agricultural University, 2016.
[29] 李伯江, 李平华, 吴望军, 李齐发, 黄瑞华, 刘红林. 骨骼肌肌纤维形成机制的研究进展[J]. 中国农业科学, 2014,47(6):1200-1207. doi:10.3864/j.issn.0578-1752.2014.06.016.
Li B J, Li P H, Wu W J, Li Q F, Huang R H, Liu H L. Progresses in research of the mechanisms of skeletal muscle fiber formation[J]. Scientia Agricultura Sinica,2014,47(6):1200-1207.
[30] Pang X C, Lin L J, Tang B. Unraveling the role of calcium ions in the mechanical properties of individual collagen fibrils[J]. Scientific Reports, 2017, 7:46042. doi: 10.1038/srep46042.
[31] Moreno H M, Jacq C, Montero M P, Gómez-Guillén M C, Borderías A J, Mørkøre T. Effect of selective breeding on collagen properties of Atlantic salmon (Salmo salar L.)[J]. Food Chemistry, 2016, 190:856-863. doi: 10.1016/j.foodchem.2015.06.022.
[32] Lesiow T, Rentfrow G K, Xiong Y L. Polyphosphate and myofibrillar protein extract promote transglutaminase-mediated enhancements of rheological and textural properties of PSE pork meat batters[J]. Meat Science, 2017, 128:40-46. doi: 10.1016/j.meatsci.2017.02.002.
[33] Luu B E, Tessier S N, Duford D L, Storey K B. The regulation of Troponins I, C and ANP by GATA4 and Nkx2-5 in heart of hibernating thirteen-lined ground squirrels, Ictidomys tridecemlineatus[J]. PLoS One, 2015,10(2):e0117747. doi: 10.1371/journal.pone.0117747.
[34] Martin-Garrido A, Biesiadecki B J, Salhi H E, Shaifta Y, Dos Remedios C G, Ayaz-Guner S, Cai W X, Ge Y, Avkiran M, Kentish J C. Monophosphorylation of cardiac troponin-I at Ser23/24 is sufficient to regulate cardiac myofibrillar Ca2+, sensitivity and calpain-induced proteolysis[J]. Journal of Biological Chemistry, 2018, 293(22):8588-8599. doi: 10.1074/jbc.RA117.001292.
[35] Wang H, Grant J E, Doede C M, Sadayappan S, Robbins J, Walker J W. PKC-betaII sensitizes cardiac myofilaments to Ca2+ by phosphorylating troponin I on threonine-144[J].Journal of Molecular and Cellular Cardiology, 2006, 41(5):823-833.doi: 10.1016/j.yjmcc.2006.08.016.
[36] Lu Q W, Hinken A C, Patrick S E, Solaro R J, Kobayashi T. Phosphorylation of cardiac troponin I at protein kinase C site threonine 144 depresses cooperative activation of thin filaments[J]. Journal of Biological Chemistry, 2010, 285(16):11810-11817. doi: 10.1074/jbc.M109.055657.
[37] Messer A E, Bayliss C R, El-Mezgueldi M, Redwood C S, Ward D G, Leung M C, Papadaki M, Dos Remedios C, Marston S B. Mutations in troponin T associated with hypertrophic cardiomyopathy increase Ca2+-sensitivity and suppress the modulation of Ca2+-sensitivity by troponin I phosphorylation[J]. Archives of Biochemistry and Biophysics, 2016, 601:113-120. doi: 10.1016/j.abb.2016.03.027.
[38] Akhter S,Zhang Z L, Jin J P. The heart-specific NH2-terminal extension regulates the molecular conformation and function of cardiac troponin I[J]. AJP Heart and Circulatory Physiology, 2012, 302(4):923-933. doi: 10.1152/ajpheart.00637.2011.
[39] Zhao Y, Feng Y, Zhang Y M, Ding X X, Song Y Z, Zhang A M, Liu L, Zhang H, Ding J H, Xia S. Targeted next-generation sequencing of candidate genes reveals novel mutations in patients with dilated cardiomyopathy[J]. International Journal of Molecular Medicine, 2015, 36(6):1479-1486. doi: 10.3892/ijmm.2015.2361.
[40] 曹晨, 马堃. 蛋白质二级结构指定[J]. 生物信息学, 2016,14(3):181-187. doi:10.3969/j.issn.1672-5565.2016.03.09.
Cao C, Ma K. Protein secondary structure assignment[J]. Chinese Journal of Bioinformatics, 2016,14(3):181-187.
[41] Yang H, Xu Z Y, Ma Z X, Xiong Y Z, Deng C Y, Zuo B. Molecular cloning and comparative characterization of the porcine troponin I family[J]. Animal Biotechnology, 2009, 21(1): 64-76. doi: 10.1080/10495390903385983.
[42] Sun Y, Wang G Z, Ji Z B, Chao T L, Liu Z H, Wang X L, Liu G Q, Wu C H, Wang J M. Three slow skeletal muscle troponin genes in small-tailed Han sheep (Ovis aries):molecular cloning, characterization and expression analysis[J]. Molecular Biology Reports, 2016,43(9): 999-1010. doi: 10.1007/s11033-016-4027-6.
[43] Moran C M, Garriock R J, Miller M K, Heimark R L, Gregorio C C, Krieg P A. Expression of the fast twitch troponin complex, fTnT, fTnI and fTnC, in vascular smooth muscle[J]. Cell Motility and the Cytoskeleton, 2008, 65(8):652-661. doi: 10.1002/cm.20291.
[44] Ju Y W, Li J, Xie C, Ritchlin C T, Xing L P, Hilton M J, Schwarz E M. Troponin T3 expression in skeletal and smooth muscle is required for growth and postnatal survival: Characterization of Tnnt3tm2α(KOMP)Wtsi mice[J]. Genesis, 2013, 51(9):667-675. doi: 10.1002/dvg.22407.
[45] Dhoot G K, Gell P G H, Perry S V. The localization of the different forms of troponin I in skeletal and cardiac muscle cells[J]. Experimental Cell Research, 1978, 117(2):357-370.doi:10.1016/0014-4827(78)90149-0.