青藏高原作为世界海拔最高的高原,平均海拔为4 000~5 000 m,因此造成了高寒、低氧、紫外线极强等恶劣多变的高原气候。寒冷和氧气含量低等原因给恒温脊椎动物的生存带来了严峻的挑战,由于氧气分压的降低减缓了呼吸组织细胞对氧气的供应,因此限制了能量的代谢而抑制了热量的产生,研究高原动物如何抵抗低氧与寒冷环境有助于了解低氧适应性进化进程[1]。藏黄牛是青藏高原地区的传统饲养品种,主要分布在海拔3 000~4 500 m的农区、林区及半农半牧区,以放牧饲养为主,可肉、乳、役兼用,是经长期自然选择而逐渐适应高原气候的小型地方原始品种[2]。由于高海拔地区独特的环境特点,藏黄牛进化出与低氧环境相适应的生理特征,例如独特的心脏结构、单位面积内肺泡数增多等[3-9]。藏黄牛作为高原地区所特有的原始品种,既是促进当地经济发展的重要动物,也是作为研究黄牛低氧适应性的理想物种。三江黄牛原产于四川省阿坝藏族自治州汶川县,具有良好的役用性能、躯干较长、肉质好、适应性与抗病性强等特点,是遗传资源中一个极为珍贵的基因库[10]。
据报道,长期生活在低温、低氧及高辐射的高海拔地区的动物进化出与低海拔地区动物不同的呼吸系统,例如支气管管壁变厚、肺泡内毛细血管丰富且呈扩张状态、心血管系统发达等[11-12]。齐晓园等[13]对牦牛与藏黄牛血液生理生化指标的研究发现,随着海拔高度的升高,牦牛通过使血液中红细胞和血红蛋白含量不断增加、增大血细胞比容等方式来适应高原低氧环境,黄牛通过增加红细胞的体积、增加血液中血红蛋白含量与血红蛋白浓度等方式来适应低氧环境,从而发现牦牛与黄牛对于应对低氧适应性产生了不同的机制;王志敏等[14]对藏鸡低氧适应性机制研究发现,随着海拔高度的升高,红细胞数、血红蛋白含量及平均红细胞体积随之升高,而平均血红蛋白量与红细胞平均血红蛋白浓度随着海拔的升高而降低,并且发现处在同一海拔高度的公母鸡之间的血液生理生化指标也存在差异,公鸡的各项指标均高于母鸡。
心脏是脊椎动物身体中对氧含量变化较敏感的重要器官之一,为血液流动至全身各部位提供动力,使细胞能够正常的代谢及运作,研究发现藏黄牛进化出与三江黄牛不同的心血管系统,且在低氧情况下能够通过增加呼吸频率,加强心泵功能等方式进行自身调控来适应低氧环境[15-16],因此,本研究拟通过对藏黄牛和三江黄牛心脏组织进行转录组测序分析,以期获得高原低氧适应性相关的基因及其涉及的信号通路,比较不同海拔高度黄牛心脏组织中差异表达转录本,为高海拔环境胁迫下产生的适应性进化机制研究奠定基础。
1 材料和方法
1.1 样品采集
分别选取健康的4.5岁、位于西藏自治区昌都市类乌齐县的藏黄牛(海拔高度:3 810 m)与四川省成都市郫都区的三江黄牛(海拔高度:500 m)各3头,采集其心脏组织,经DEPC水冲洗后,用锡箔纸包裹置于液氮中带回实验室保存备用。
1.2 总RNA提取、cDNA文库构建及Illumina测序
取出液氮中放置的各黄牛心脏组织,加入液氮于研钵中研磨,研磨至粉末状后移入1.5 mL无RNase的离心管,加入1 mL TRIzol试剂(购自Invitrogen公司,美国)提取总RNA,提取步骤参照 TRIzol试剂说明书。提取的总RNA经紫外分光光度计测试其浓度与OD260nm/280nm值,再利用1%的琼脂糖凝胶电泳(BIORAD)检测其质量。将提取的总RNA反转录构建cDNA文库,建好的测序文库用Illumina HiSeqTM 4000平台进行测序。
1.3 数据处理及分析
为了保证数据质量,在生物信息学分析前对数据进行质量控制与过滤,步骤如下:先将初步过滤得到的干净数据序列(Clean reads)进行进一步更严格的过滤,去除含有接头的reads、全部都是A碱基的reads、含N比例大于10%的reads和低质量的reads(质量值Q≤20的碱基数占整条reads的50%以上),最终得到高质量数据用于后续的信息分析。
使用比对软件Tophat2(2.1.1)将数据对比到黄牛参考基因组(ARS-UCD1.2)进行分析。利用FPKM(Fragments Per Kilobase of transcript per Million mapped reads)法计算转录本表达量并进行统计。根据FPKM定量结果,使用R语言包(http://www.r-project.org/)进行热图绘制,计算出所有样品两两之间的相关性。样品间基因表达水平相关性是检验试验可靠性和样本选择是否合理的重要指标,相关系数越接近1,表明样品表达模式的相似度越高[13,17]。
使用 Cufflink(http://cufflinks.cbcb.umd.edu/ howitworks.html)软件根据比对结果来重构转录本,对比重构转录本与参考基因组(ARS-UCD1.2)序列(筛选转录本长度≥200 bp且外显子数目≥2),如果新转录本与参考基因组对比上,则得到样本已知的转录本和新的转录本。重构转录本可能由于延长了转录本注释的5′或者3′端,因此优化了转录本结构[18]。 利用 rMATS 软件(http://rnaseq-mats.sourceforge.net/index.html)进行可变剪切事件的分析。rMATS软件可以将可变剪切事件分成5类:外显子跳跃(Skipped exon,SE)、可变5′端剪切(Alternative 5′ splice site,A5SS)、可变3′端剪切(Alternative 3′ splice site,A3SS)、外显子选择性跳跃(Mutually exclusive exon,MXE)、内含子保留(Retained intron,RI)[19-20]。
1.4 差异表达mRNA的 GO富集与KEGG信号通路分析
使用edgeR软件以RPKM(Reads per kilobase transcriptome per million mapped reads)法计算表达量,对藏黄牛与三江黄牛心脏组织mRNA表达量进行差异分析,FDR(False discovery rate)是多重检验校正P值的一种方法,以筛选条件为:FDR<0.05 且 |log2FC|>1(FC为3个样品表达量的平均值)来筛选差异转录本。同时利用皮尔森算法计算样品间的距离,使用皮尔逊相关系数r(Pearson′s correlation coefficient)来表示生物学重复相关性,r2 越接近1就表示2个样品间相关性越强,对同一条件下每一对生物学重复样品的基因表达量的相关性作图,根据距离大小建立聚类图。
对得到的差异转录本进行GO功能分析将差异表达转录本向GO数据库(http://www.geneontology.org/)的各term(词条、节点)映射,并计算每个条目的转录本数 [21]。GO功能分析主要包括分子功能(Molecular function)、细胞组分(Cellular component)、生物过程(Biological process)。应用超几何检验,找出与整个转录本组背景相比,在差异表达转录本中显著富集的GO条目。P值通过FDR校正之后得到Q值,以Q≤0.05为阈值,满足此条件的GO条目定义为在差异表达转录本中显著富集的GO条目。
通过KEGG(Kyoto encyclopedia of genes and genomes)数据库进行比对[22],应用超几何检验,找出差异表达转录本中显著性富集的通路,确定差异表达转录本参与的主要生化代谢途径和信号转导途径[23]。
1.5 RT-qPCR检测
为验证测序结果是否准确,在差异表达基因中随机选取6个基因,以GADPH为内参基因,用SYBR® Premix Ex TaqTM Ⅱ试剂,以反转录的cDNA为模板利用CFX96 实时定量PCR 系统(美国Bio-Rad 公司)进行实时荧光定量PCR(Real-time PCR)验证。使用Primer Premier 5.0软件设计荧光定量引物(表1)并由赛默飞世尔(上海)科技有限公司合成。10 μL反应体系:2×SYBR 5 μL;上下游引物各0.4 μL;cDNA模板0.8 μL;无RNAase水3.4 μL。定量反应程序:95 ℃预变性30 s;95 ℃变性5 s,60 ℃退火30 s,39个循环,同时记录溶解度曲线用来鉴定引物的特异性,同一样品设置3个技术重复。利用2-ΔΔCT法计算各个样本的相对表达量,结果以“平均值±标准差”来表示。
表1 荧光定量PCR引物
Tab.1 RT-qPCR primers
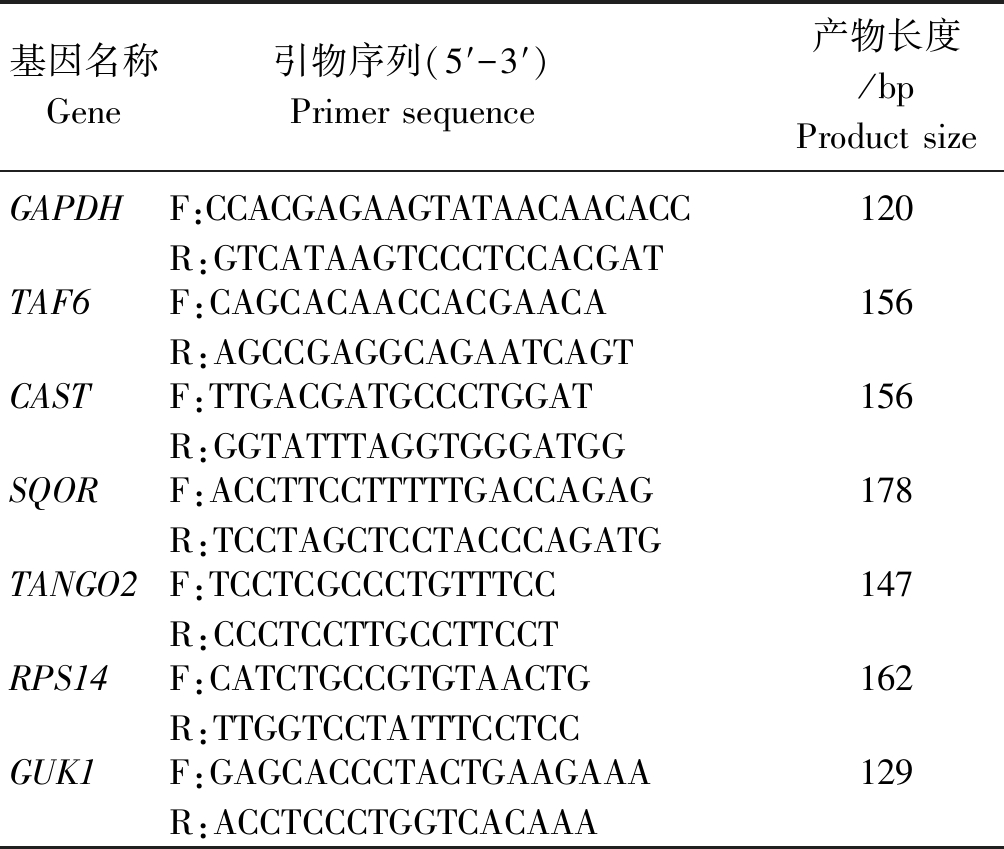
基因名称Gene引物序列(5′-3′) Primer sequence 产物长度/bpProduct sizeGAPDHF:CCACGAGAAGTATAACAACACCR:GTCATAAGTCCCTCCACGAT120TAF6F:CAGCACAACCACGAACAR:AGCCGAGGCAGAATCAGT156CASTF:TTGACGATGCCCTGGATR:GGTATTTAGGTGGGATGG156SQORF:ACCTTCCTTTTTGACCAGAGR:TCCTAGCTCCTACCCAGATG178TANGO2F:TCCTCGCCCTGTTTCCR:CCCTCCTTGCCTTCCT147RPS14F:CATCTGCCGTGTAACTGR:TTGGTCCTATTTCCTCC162GUK1F:GAGCACCCTACTGAAGAAAR:ACCTCCCTGGTCACAAA129
2 结果与分析
2.1 测序数据质量评估及分析
本研究共构建藏黄牛与三江黄牛心脏组织转录组文库6个,测序得到的原始数据经过滤,高质量数据在测序数据中所占比例均高于98%(表2)。经过过滤后样品GC含量均不小于50%,从碱基组成与质量分析图可知,过滤后样品的碱基分布稳定,Q30(数据质量达到Q30即测序准确率为99.9%的碱基数目及百分比)值均大于95.9%,证明测序质量可靠,可以用于后续试验分析[24]。
表2 经过过滤数据统计
Tab.2 Statistics of filtered data
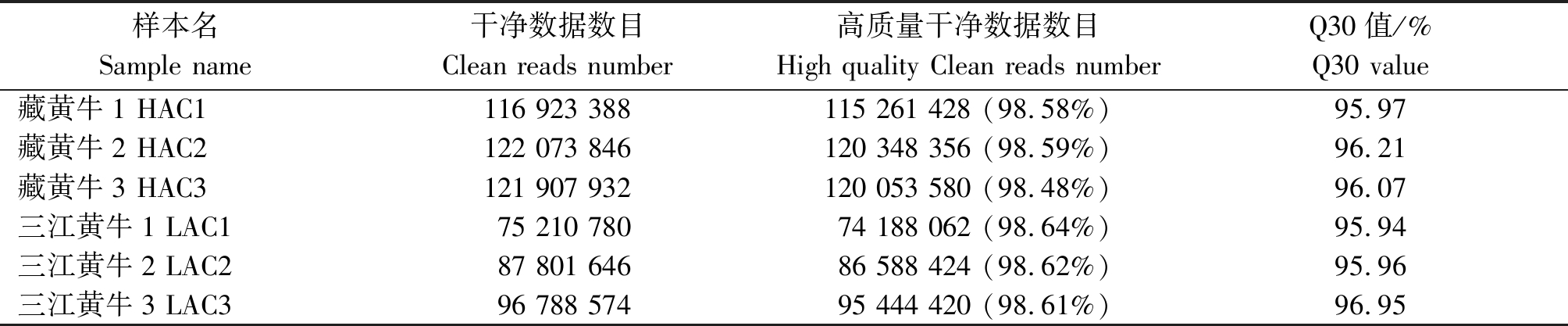
样本名Sample name干净数据数目Clean reads number高质量干净数据数目High quality Clean reads numberQ30值/%Q30 value藏黄牛1 HAC1116 923 388115 261 428 (98.58%)95.97藏黄牛2 HAC2122 073 846120 348 356 (98.59%)96.21藏黄牛3 HAC3121 907 932120 053 580 (98.48%)96.07三江黄牛1 LAC175 210 78074 188 062 (98.64%)95.94三江黄牛2 LAC287 801 64686 588 424 (98.62%)95.96三江黄牛3 LAC396 788 57495 444 420 (98.61%)96.95
注:HAC1、HAC2、HAC3分别是藏黄牛的3个生物学重复;LAC1、LAC2、LAC3是三江黄牛的3个生物学重复。表3-4同。
Note: HAC1, HAC2, and HAC3 are three biological replicates of Tibetan cattle;LAC1, LAC2, and LAC3 are three biological replicates of Sanjiang cattle. The same as Tab.3-4.
2.2 转录组测序数据对比分析
比对黄牛参考基因组(ARS-UCD1.2)结果表明,唯一比对率均在86%以上,对比率均高于87%(表3),表明转录组测序数据质量稳定[25-26]。结果可知唯一能对比到的reads数在3个藏黄牛转录组中平均有81 930 524条,3个三江黄牛转录组中平均有79 135 363条,藏黄牛转录本数高于三江黄牛,而三江黄牛转录组对比率(Mapping ratio)高于藏黄牛。
表3 去除核糖体RNA数据对比到参考基因组
Tab.3 Removal rRNA data to the reference genome
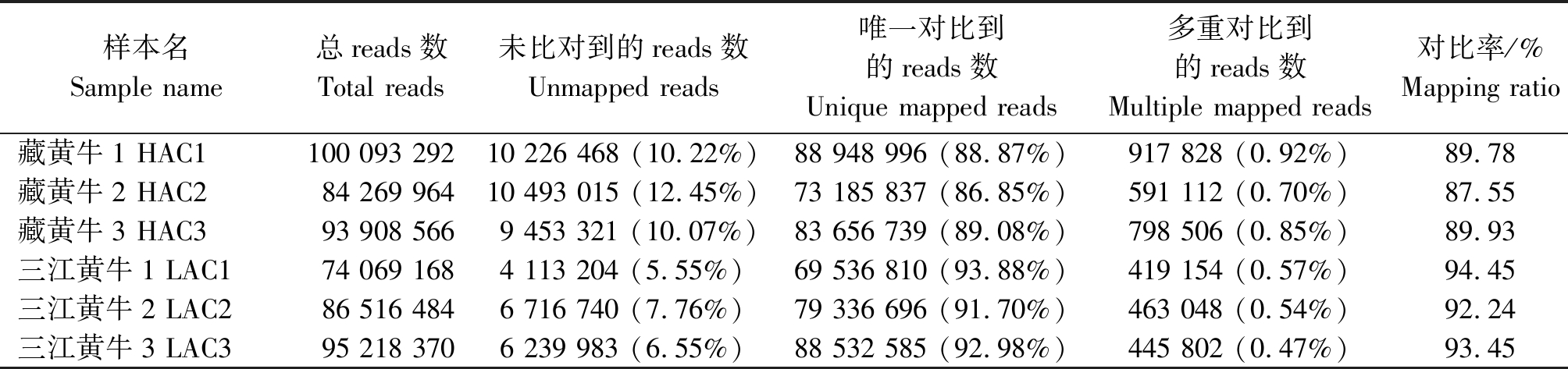
样本名Sample name总reads数Total reads未比对到的reads数Unmapped reads唯一对比到的reads数Unique mapped reads多重对比到的reads数Multiple mapped reads对比率/%Mapping ratio藏黄牛1 HAC1100 093 29210 226 468 (10.22%)88 948 996 (88.87%)917 828 (0.92%)89.78藏黄牛2 HAC284 269 96410 493 015 (12.45%)73 185 837 (86.85%)591 112 (0.70%)87.55藏黄牛3 HAC393 908 5669 453 321 (10.07%)83 656 739 (89.08%)798 506 (0.85%)89.93三江黄牛1 LAC174 069 1684 113 204 (5.55%)69 536 810 (93.88%)419 154 (0.57%)94.45三江黄牛2 LAC286 516 4846 716 740 (7.76%)79 336 696 (91.70%)463 048 (0.54%)92.24三江黄牛3 LAC395 218 3706 239 983 (6.55%)88 532 585 (92.98%)445 802 (0.47%)93.45
2.3 新转录本预测
使用Cufflink软件根据比对结果来重构转录本,将重构结果与黄牛参考转录本(ARS-UCD1.2)序列进行比较,得到已知的转录本和新的转录本(表4),并对新转录本的基因组信息进行功能注释。结果可知,3个藏黄牛转录本中平均预测出新mRNA 5 238个,三江黄牛转录本中预测出新mRNA 3 331个。
表4 转录本统计汇总
Tab.4 Transcript statistics summary
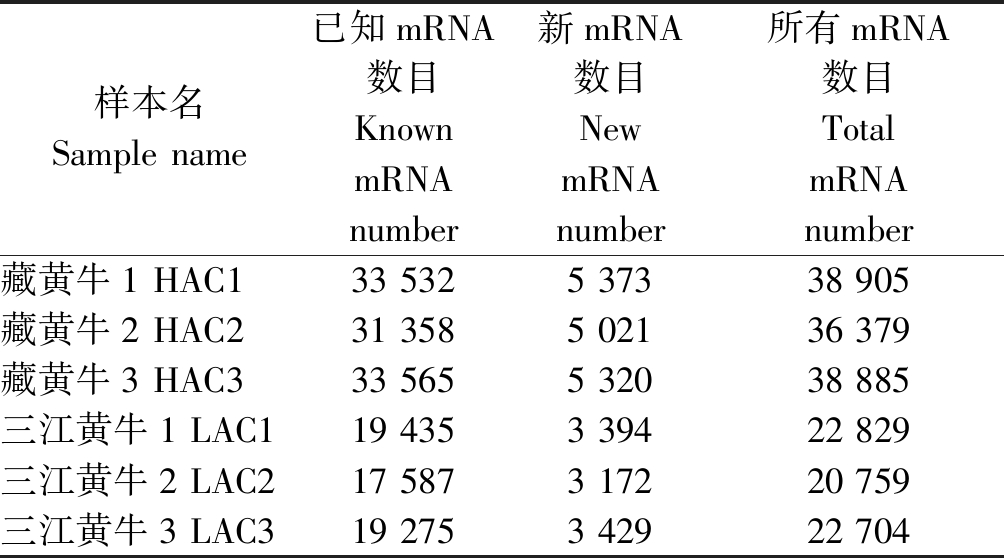
样本名Sample name已知mRNA数目Known mRNA number新mRNA数目NewmRNA number所有mRNA数目TotalmRNA number藏黄牛1 HAC133 5325 37338 905藏黄牛2 HAC231 3585 02136 379藏黄牛3 HAC333 5655 32038 885三江黄牛1 LAC119 4353 39422 829三江黄牛2 LAC217 5873 17220 759三江黄牛3 LAC319 2753 42922 704
藏黄牛与三江黄牛样品可变剪切结果可知(图1),共得到差异可变剪切事件4 742个。在5类可变剪切事件模型中以SE模型为主,占所有差异可变剪切事件的62.5%;RI模型在差异可变剪切中所占比例最小,为3.5%。
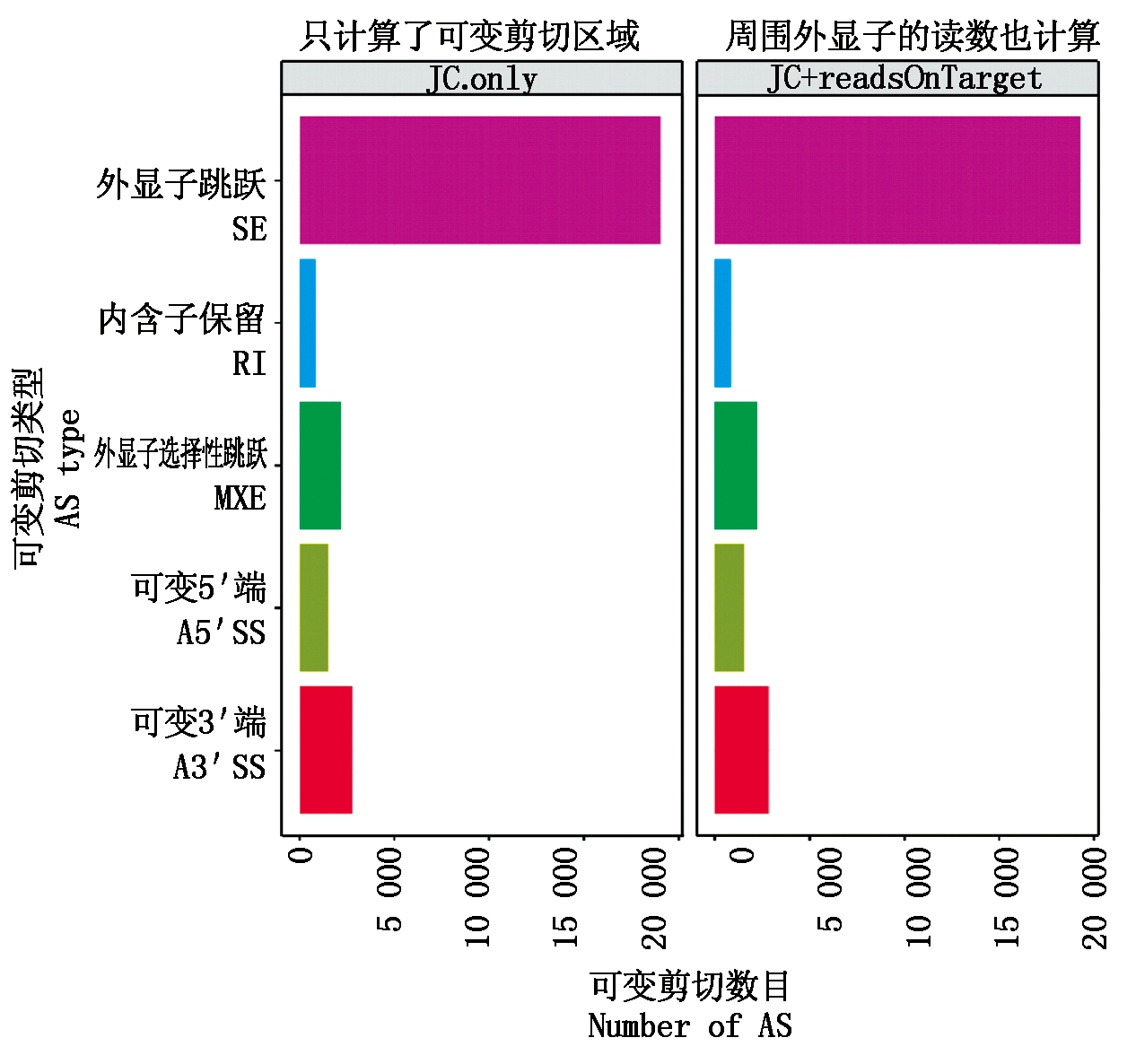
图1 差异可变剪切分类和数量统计图
Fig.1 Alternative splicing and quantity statistics
2.4 不同海拔高度黄牛心脏mRNA表达水平比较
对所有样本转录本的表达量丰度进行比较,其中横坐标为log10(FPKM),数值越高表示转录本表达量越高;纵坐标为转录本的丰度,即为对应横轴表达量的转录本数 / 检测已表达转录本的总数。结果如图2显示,三江黄牛3个样本的转录本表达量丰度明显高于藏黄牛样本转录本的表达量。
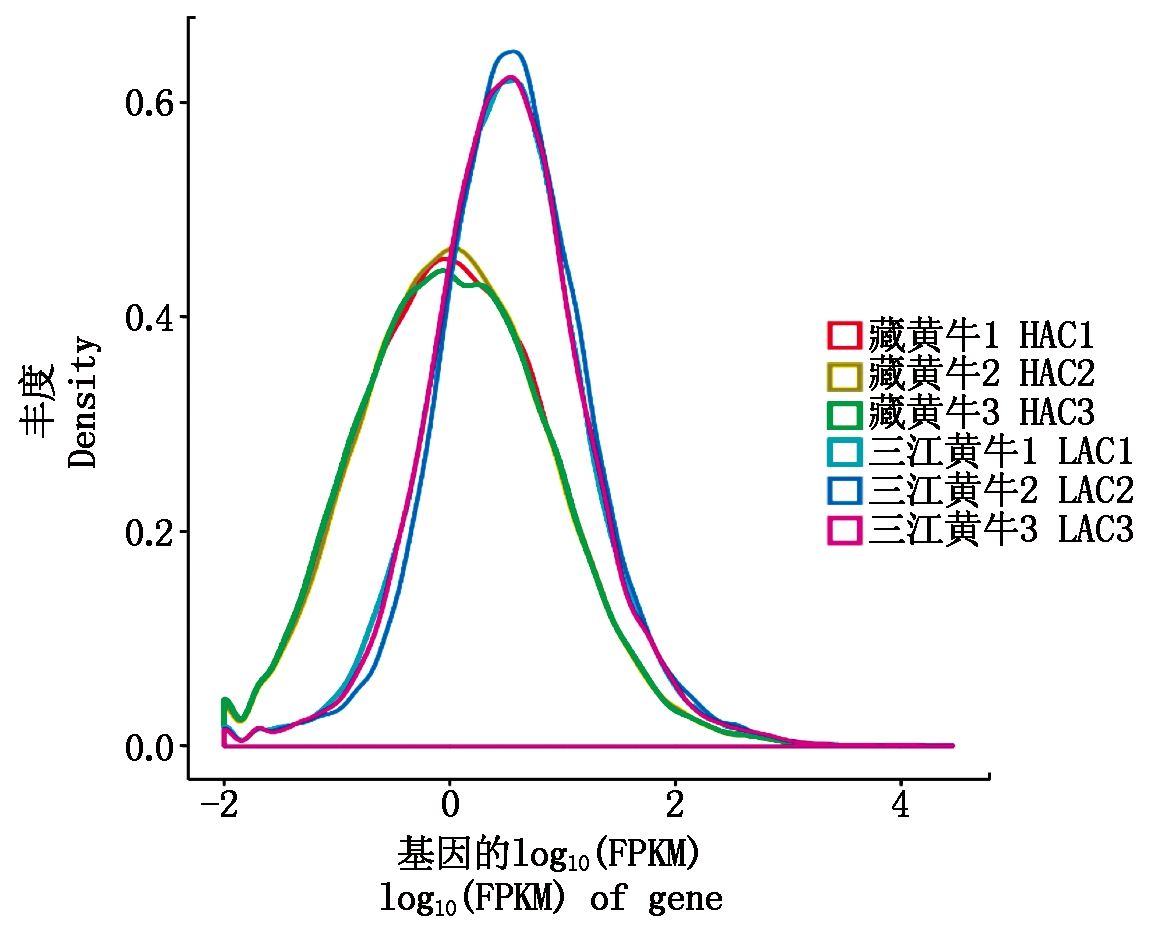
图2 所有样本表达量丰度分布图
Fig.2 FPKM distribution of all samples
根据FPKM计算结果对转录本表达量进行计算,根据结果计算出各样品之间的相关性(图3)。以三江黄牛样本1与2组比较为例,r2值为0.973 7,表明其重复性高,数据准确且稳定。藏黄牛样本的相关性系数r2平均值为0.889 0,三江黄牛样本的相关性系数r2平均值为0.972 6。
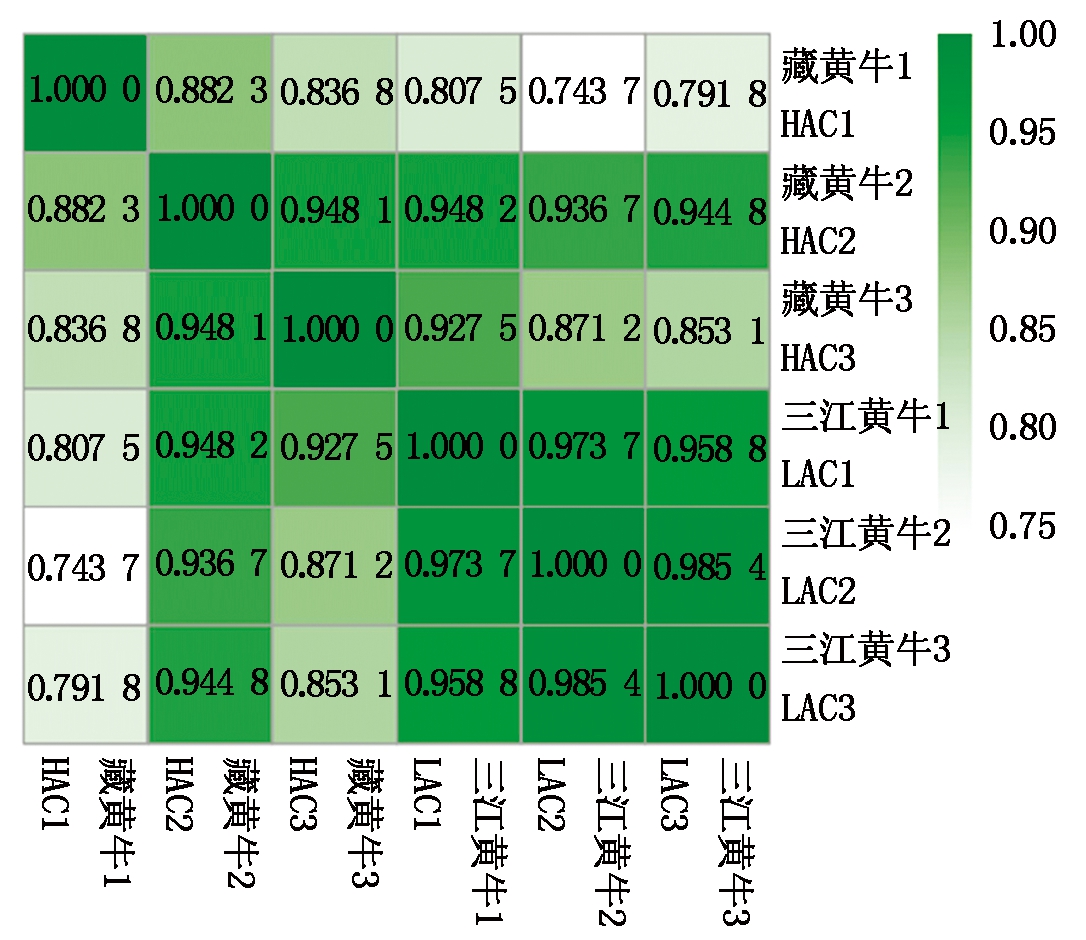
图3 样品间表达量相关性热图
Fig.3 Correlation heat map of gene expression level in samples
2.5 mRNA差异表达分析
以FDR 与 log2FC 来筛选差异转录本结果表明,藏黄牛心脏组织转录组文库相比于三江黄牛心脏组织转录组文库分析,共发现显著差异表达基因608个,其中上调基因467个,下调基因141个(图4)。在藏黄牛与三江黄牛中表达量最高且表达差异倍数较大的基因分别是β2微球蛋白基因(Beta-2-microglobulin,B2M)与细胞色素C氧化酶7C亚基基因(Cytochrome c oxidase subunit 7C,COX7C)。另外,活化T细胞核因子1基因(Nuclear factor of activated T cells 1,NFATC1)、内皮素转化酶1基因(Endothelin converting enzyme 1,ECE1)、钙蛋白酶抑制蛋白基因(Calpastatin,CAST)在藏黄牛与三江黄牛中表达量也较高,差异倍数较大,且FDR值较低。
对筛选出的差异表达本进行聚类分析,每列代表一个样品,每行代表一个转录本,颜色越红表示表达量越高,颜色越蓝表示表达量越低。结果如图所示(图5)。在纵向来看,藏黄牛(HAC)与三江黄
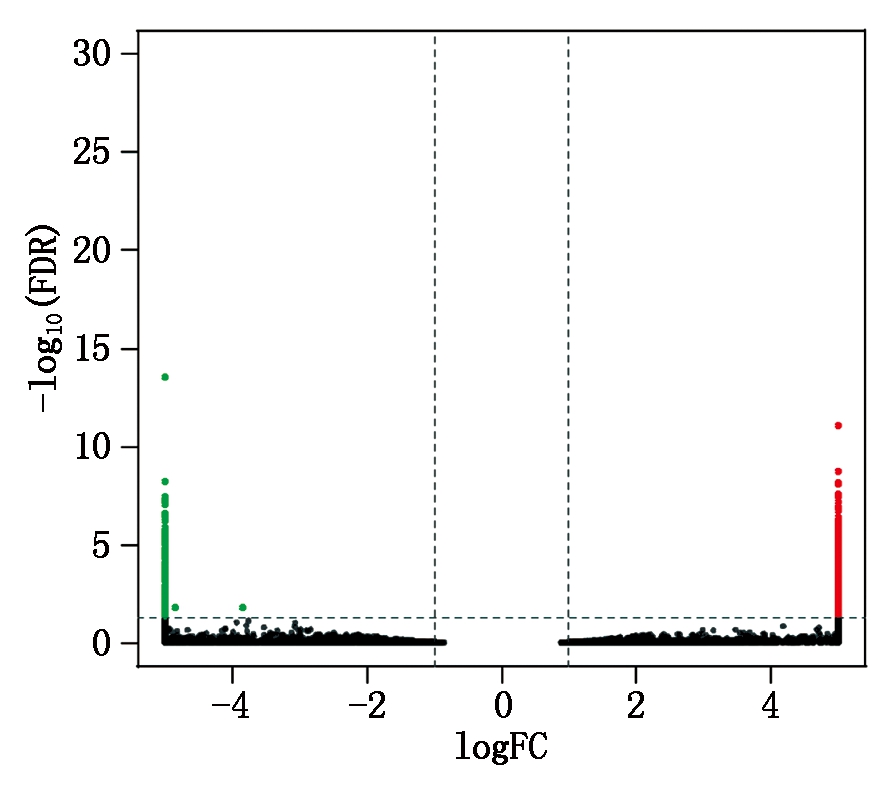
图中的每个点代表一个基因,红色和绿色的点分别表示上调与下调的显著差异表达基因,黑色的点表示没有差异。
Each point in the figure represents a gene, and the red and green dots represent significant up-regulated expressed genes and down-regulated genes, the black dots indicate no difference.
图4 差异表达基因火山图
Fig.4 Volcano plot of differentially expressed genes
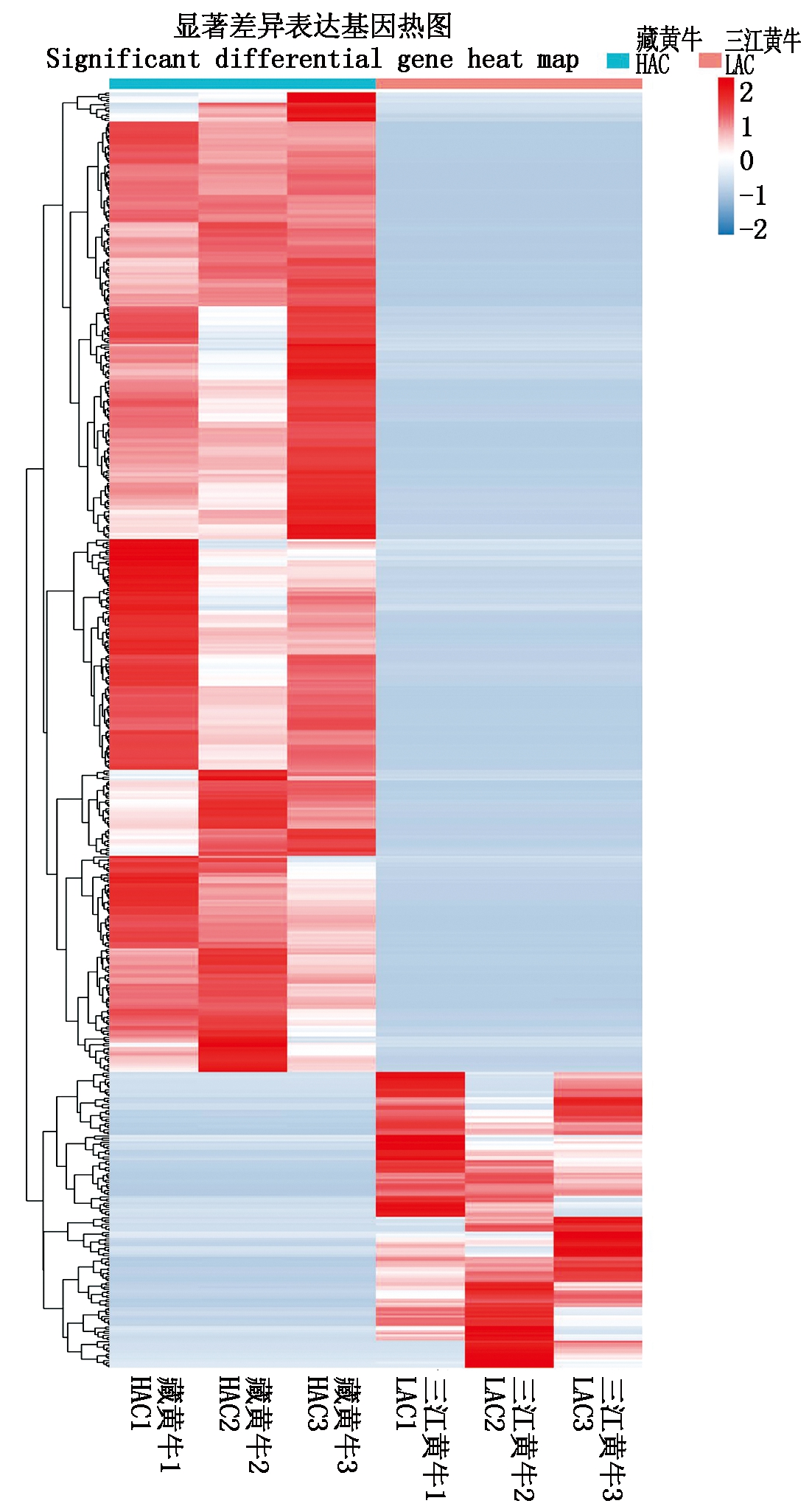
图5 差异表达基因聚类分析图热图
Fig.5 Differential expression gene clustering analysis heat map
牛(LAC)的3个生物学重复样本聚类在一起,说明生物学重复样品间具有很高的相关性。从纵向来看显著性差异基因可以分为上下2个基因簇,结果显示上调与下调基因的聚类是成功的,与筛选出的差异表达基因结果相符合,并且上调基因之间及下调基因间相关性均很高[27]。
2.6 差异表达基因的GO功能富集分析
608个显著差异表达mRNA经过功能富集分析在分子功能、细胞组分、生物学过程分别富集到327,253,2 191个条目。结果显示,在生物学过程本体中,细胞发育过程条目富集程度最高;在分子功能本体中,结合与转录因子活性条目富集程度最高;在细胞组分本体中,细胞前缘条目富集程度最高。由图6可知,以上下调基因进行分类可以富集在50个条目(Term),其中参与分子功能、细胞组分、生物过程的条目分别为11,18,21个。参与分子功能的条目中,涉及参与基因数目最多的3个条目分别为:细胞过程(Cellular process)条目,其上调基因274个,下调基因80个;单一生物过程(Single-organism process)条目其上调基因239个,下调基因72个;代谢过程(Metabolic process)条目,其上调基因190个,下调基因56个。参与细胞组分的条目中,涉及参与基因最多的条目分别为:核酸结合转录因子活性(Nucleic acid binding transcription factor activity条目,其上调基因27个,下调基因7个;结合(Binding)条目,上调基因256个,下调基因71个;催化活性(Catalytic activity)条目,其上调基因103个,下调基因有34个。参与生物过程的条目中细胞器(Organelle)、细胞(Cell)、细胞部分(Cell part)条目参与基因数目最多,其参与基因数目分别为上调基因212个,下调基因65个,上调基因258个,下调基因77个,上调基因258个,下调基因77个。
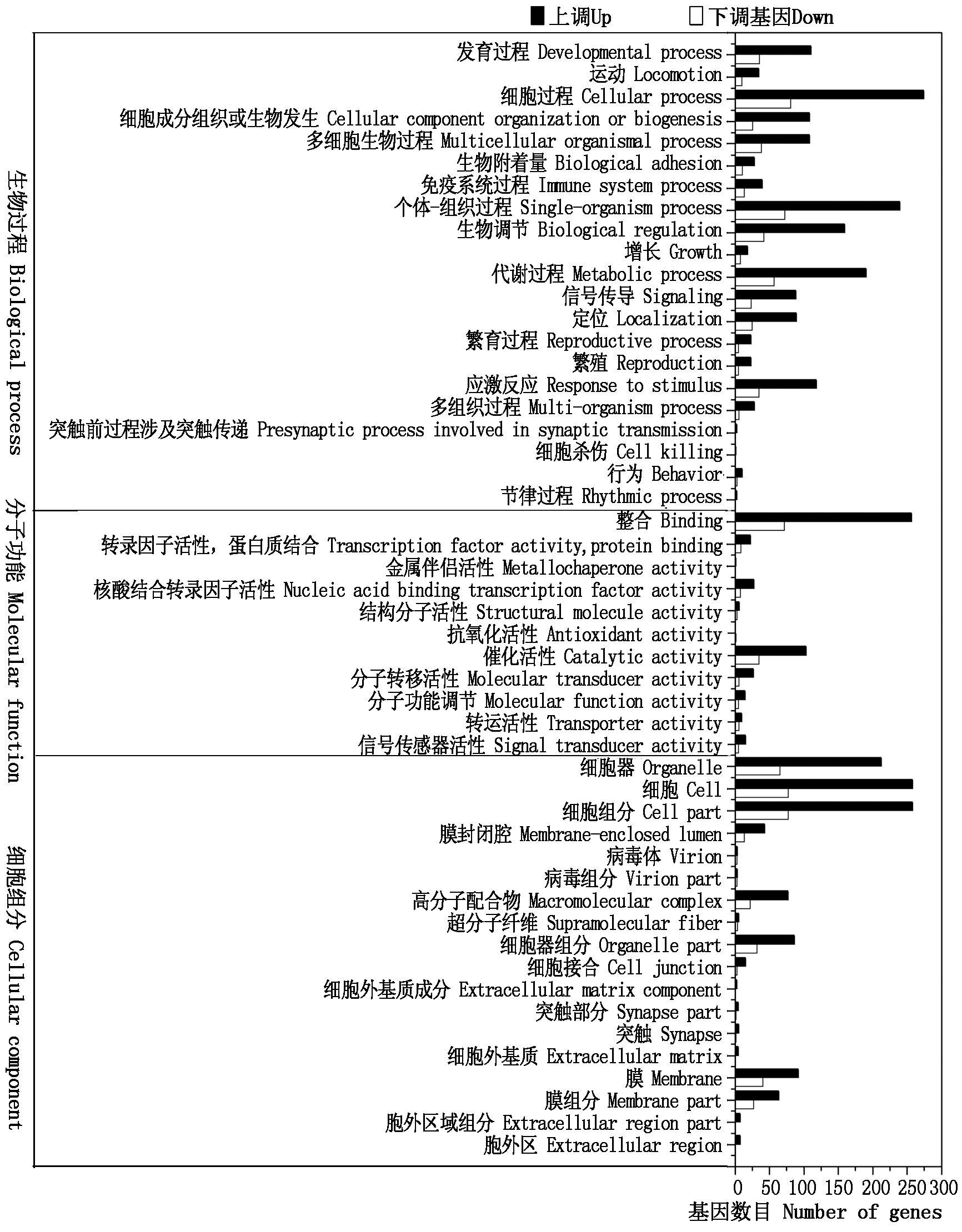
图6 差异基因的GO富集分析柱状图
Fig.6 GO enrichment analysis histogram of differential genes
2.7 差异mRNA的KEGG通路富集分析
经过KEGG通路富集分析,差异表达转录本中显著富集通路共有8条,分别是cGMP-PKG信号通路、T细胞受体信号通路、FcγR介导的吞噬作用、甲状腺激素信号通路、轴突指导、磷脂酶D信号通路、白细胞跨内皮迁移、肌动蛋白细胞骨架调节通路(图7)。其中差异表达基因NFATC1显著富集在cGMP-PKG信号通路与T细胞受体信号通路中(图8)。
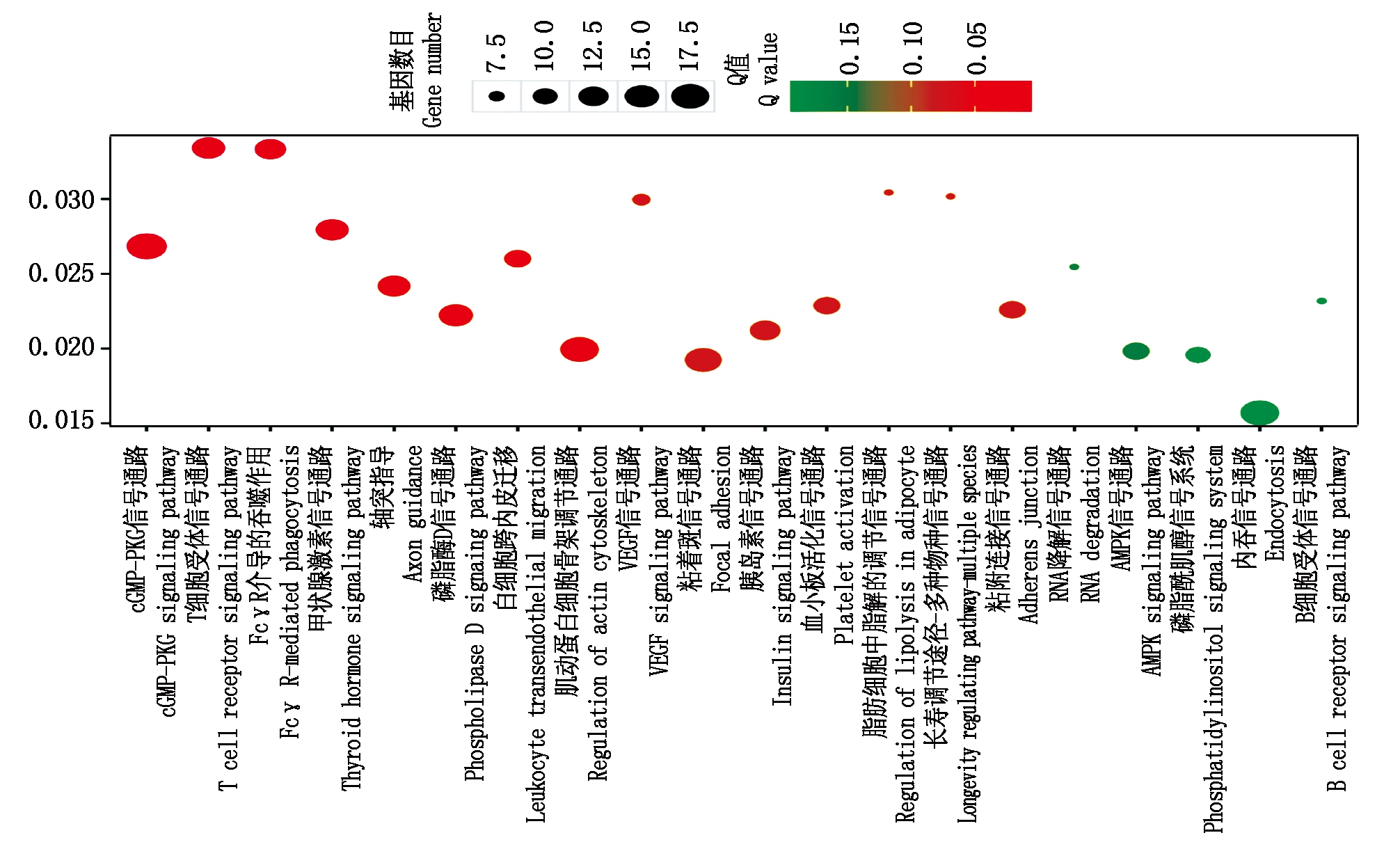
图7 KEGG富集气泡图
Fig.7 KEGG enriched bubble chart

图8 cGMP-PKG信号通路
Fig.8 cGMP-PKG signaling pathway
2.8 RT-qPCR验证
随机选取6个差异表达基因,分别是CAST、TANGO2、GUK1、TAF6、SQRDL、RPS14做RT-qPCR验证,根据结果(图9)可知,转录组测序数据与荧光定量结果基因的表达水平变化趋势一致,判定转录组测序数据可靠。
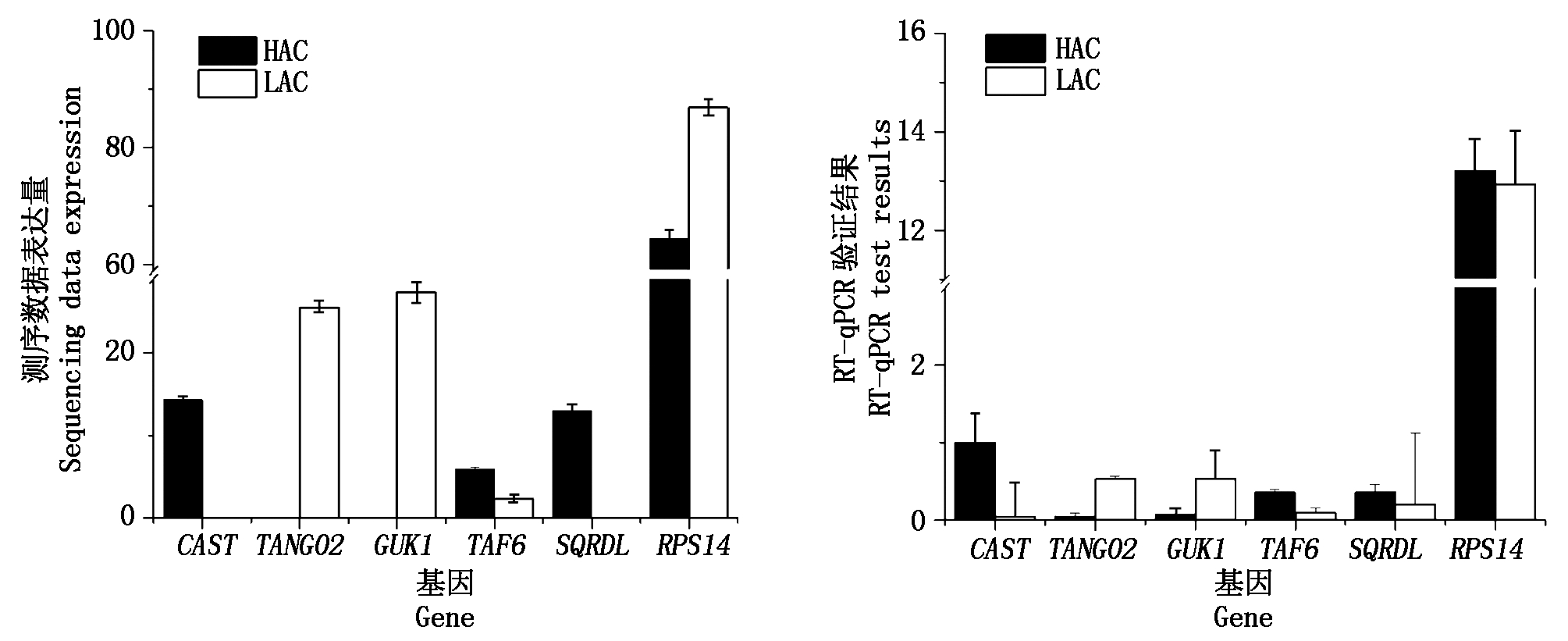
图9 转录组测序结果与荧光定量结果比较分析
Fig.9 Comparison analysis of the RNA-Seq and RT-PCR result
3 讨论与结论
近年来随着高通量测序技术地迅速发展,转录组测序技术被广泛地应用在基因的表达及调控研究中[27-32]。通过转录组技术对高原低氧环境中动物适应性的研究已经在藏猪、藏鸡[14]、田鼠和牦牛[33]等动物中有所研究。本试验首次采用转录组测序技术对藏黄牛与三江黄牛心脏组织中差异表达转录本进行研究,为研究藏黄牛低氧适应性机制提供理论基础。
3.1 转录组测序数据分析
本研究使用Illumina HiSeqTM 4000测序平台构建了藏黄牛与三江黄牛心脏组织转录组本库6个,经过数据处理后获得了大量的高质量数据。对比到参考基因组后藏黄牛获得唯一比对的reads比例大于86%,且对比率均大于87%;三江黄牛唯一比对的reads比例大于91.5%,对比率均大于92%,三江黄牛与参考基因组对比率高于藏黄牛。通过对mRNA表达水平的丰度研究,发现三江黄牛mRNA表达量要高于藏黄牛,各个样品间相关性热图可以看出各个样品间的相关性较好,同样三江黄牛样品的相关性高于藏黄牛,重复性散点图得到的结果与其一致,各结果均表明测序数据稳定且质量较高。
3.2 差异表达mRNA分析
本研究对藏黄牛与三江黄牛心脏组织mRNA差异表达分析,发现在藏黄牛与三江黄牛中表达量最高,且表达差异倍数较大的B2M、COX7C、NFATC1、ECE1、CAST基因可能是与低氧适应性相关的基因。研究发现缺氧环境会导致肌纤维生长缓慢[34], CAST基因是与肉质性状相关的基因,推测藏黄牛可能通过增大心肌面积,增加心肌的密度,减小心肌纤维直径等组织学改变来适应高原低氧环境。
细胞色素C氧化酶 (Cytochrome c oxidase,Cox)是线粒体中呼吸链电子传递的终末复合物,在能量的产生与代谢中起重要作用[35],COX7C基因是发挥能量代谢作用中一个起调节作用的重要亚基。研究发现其上有与线粒体基因表达转录因子作用的位点,许多鱼类在低温环境时通过增加细胞色素C氧化酶活性来抵御寒冷[36],该基因功能与线粒体活动密切相关。本研究中COX7C基因在藏黄牛与三江黄牛中显著差异,而且在三江黄牛中下调并且表达量最高,这与藏黄牛通过增强能量代谢来抵御寒冷的特征相符[34],间接说明了藏黄牛对线粒体的调度与利用率高于三江黄牛。
活化T细胞核因子1基因(Nuclear fator of activated T cell 1,NFATC1)是Rel转录因子家族中的一员,其通过与Ca2+结合的途径进入靶细胞核内从而影响DNA的复制与转录,诱导心内组织的分化,从而形成心内膜和主肺动脉瓣等。研究证明NFATC1与心脏的房室分隔形成有关[37],进一步研究证明NFATC1可以通过降低血管内皮生长因子表达而使心内膜细胞分化,从而最终形成心脏瓣膜[38]。本研究中NFATC1基因在藏黄牛与三江黄牛中差异显著且表达量较高,可以推测其是与低氧适应性相关的基因,并且它还参与cGMP-PKG信号通路与T细胞受体信号通路,进一步证明其可能是与低氧适应性相关的基因。
内皮素转化酶 1(Endothelin-converting enzyme-1,ECE)是生物体内合成过程中重要的限速酶,其基因CEC1是与低氧适应性相关的重要候选基因之一,ECE主要位于心血管系统内,属于中性肽键内切酶(NEP)家族,它有3种异构体并且广泛分布于组织内,研究表明,体内在低氧诱导情况下可以通过上调ECE1的表达从而促进内皮素-1的合成,这与本研究中ECE1基因在藏黄牛与三江黄牛中差异表达且在藏黄牛中上调的结果相符[39]。王延东[40]对ECE1基因在藏猪、莱芜猪、大白猪的研究中发现,ECE1基因在藏猪中的表达明显高于其他2个平原猪,在藏猪10个组织中均有表达并且在心脏组织与肺脏组织的表达中最高。心肺组织是机体对抗低氧环境的重要器官,本研究中ECE1基因在藏黄牛心脏转录组中显著表达且上调,更进一步证明了ECE1基因可能是黄牛与低氧适应性相关的候选基因。
3.3 GO功能显著富集分析
据报道,在生物体内氧气的运输主要由红细胞与血红蛋白负责,同样在细胞内低氧环境时可以诱导线粒体发生自噬以适应低氧环境[41],牦牛、藏猪、藏鸡等高海拔动物,在低氧环境时通过不断增加血液中红细胞和血红蛋白含量[2]、增大血红细胞体积[30]等生理生化指标的改变来适应低氧环境。本研究发现,GO功能富集分析结果共涉及3大类别的50个功能条目,在生物学过程、分子功能、细胞组分3个本体中富集程度最高的条目分别是细胞发育过程、结合与转录因子活性和细胞前缘条目,说明富集程度高的条目都是与细胞过程有关的功能条目;差异表达mRNA功能主要富集在与细胞各项功能相关的条目上,因此,推测藏黄牛血红蛋白在长期低氧环境中有独特的携带氧气的能力来适应低氧环境。
3.4 KEGG富集通路分析
经过多重效验校正后的KEGG分析显著富集的通路有8条,其中最显著富集的通路是cGMP-PKG信号通路(cGMP-PKG signaling pathway)是与心血管疾病相关的通路。cGMP:环磷酸鸟苷酸(cyclic guanosine 3′,5′ monophosphate)为一种第二信使,由鸟苷酸环化酶作用于GTP(三磷酸鸟苷)而形成,可以在多种组织细胞中发挥生物功能;PKG:环磷酸鸟苷酸依赖的蛋白激酶,是一种真核细胞中广泛存在的丝/苏氨酸蛋白激酶[42]。在高海拔地区由于氧气含量低,容易引起心脏衰竭等临床症状。研究表明,在心脏发生衰竭时,可以通过β3-AR(肾上腺素能受体)作用于cGMP-PKG-P38信号通路使心肌细胞产生巨噬细胞移动抑制因子(Macrophage migrationinhibitory factor)而避免心肌细胞凋亡[43]。心肌在缺血时可以通过再灌注治疗使其可以较快的恢复心脏血液供应,但是不可避免的对心脏产生损伤。有研究表明,NO-cGMP-PKG通路可能与保护作用相关,其可以通过脑内神经产生特异性结合物质激活cGMP使心肌内浓度升高进而激活PKG,此活动是通过NO-cGMP-PKG通路实现的[44]。
KEGG富集通路中,肌动蛋白细胞骨架调节通路(Regulation of actin cytoskeleton)显著富集。研究表明,它们是与心血管发育、肌肉组织结构、肌肉细胞分化等相关的通路,肌动蛋白骨架在维持细胞形态、调节细胞粘连等方面具有重要功能,而粘着斑结则参与细胞膜受体与肌动蛋白骨架之间的结构连接[21]。赵启南等[45]在研究蒙古马高负荷运动后肌肉组织转录组差异分析发现,差异表达基因被富集于扩张型心肌病、心肌收缩、钙信号通路、肌动白骨架调节等与运动相关的多条通路中。此结果与本研究相似,高强度运动时由于肺部气体交换不及时,会在体内形成缺氧症状,对机体各组织器官造成损伤,高海拔地区由于环境恶劣,氧分压低造成氧气含量少,高海拔地区动物逐渐形成了独特的低氧适应机制,因此认为肌动蛋白细胞骨架调节通路可以作为与低氧适应性相关的通路。
本试验通过高通量测序技术对藏黄牛与三江黄牛心脏组织转录组测序分析,获得转录组测序数据稳定且质量良好;经过筛选共得到显著差异表达基因608个,其中上调基因467,下调基因141个,其中表达量较高且差异倍数较大的COX7C、ECE1、B2M、NFATG、CAST等基因可能是与低氧适应性相关的基因;经过GO富集与KEGG Pathway信号通路分析,GO富集结果可知细胞发育过程(Cellular developmental process)、结合(Binding)、细胞前缘(Cell leading edge)等条目显著富集;KEGG富集分析得到多条相关通路,多重效验后显著富集的通路共有8条,其中cGMP-PKG信号通路与肌动蛋白细胞骨架调节通路可能是与低氧适应性相关的通路。本研究为探究动物低氧适应性机制提供理论基础。
[1] 李菁. 不同海拔高度来源山羊心肌、肺和背最长肌的转录组比较分析[D]. 雅安:四川农业大学,2016.
Li J. Comparative analysis of transcriptomes of myocardium,lung and longissimus dorsi of goats at different altitudes [D]. Yaan: Sichuan Agricultural University,2016.
[2] Gao Y H,Gautier M,Ding X D,Zhang H,Wang Y H,Wang X,Faruque M D O,Li J Y,Ye S H,Gou X,Han J L,Johannes A L,Zhang Y. Species composition and environmental adaptation of indigenous Chinese cattle[J]. Scientific Reports,2017,7(1):16196. doi:10.1038/s41598-017-16438-7.
[3] Moore L G. Human genetic adaptation to high altitudes: current status and future prospects [J]. Quat Int,2017,461:4-13. doi:10.1016/j.quaint.2016.09.045.
[4] Julian C G. Epigenomics and human adaptation to high altitude[J]. Journal of Applied Physiolog,2017,123(5):1362-1370. doi:10.1152/japplphysiol.00351.2017.
[5] He Y X,Qi X B,Ouzhu L B,Liu S M,Li J,Zhang H,Baima K Z,Bai C J,Zheng W S,Guo Y B,Duoji Z M,Baima Y J,Deji Q Z,Bian B,Gongga L Z,Pan Y Y,Qu L, Kang M,Ciren Y J,Guo W,Yang L,Peng Y,Zhang X M,Xiang K,Yang Z H,Wang L B,Geng D,Zhang Y F,Wu T Y,Su B,Cui C Y. Blunted nitric oxide regulation in Tibetans under high-altitude hypoxia[J].National Science Review,2018,5(4):516-529. doi:10.1093/nsr/nwy037.
[6] Qiu Q, Zhang G J, Ma T, Qian W B, Wang J Y, Ye Z Q, Cao C C, Hu Q J, Kim J, Larkin D M, Auvil L, Capitanu B, Ma J, Lewin H A, Qian X J, Lang Y S, Zhou R, Wang L Z, Wang K, Xia J Q, Liao S G, Pan S K, Lu X, Hou H L, Wang Y, Zang X T, Yin Y, Ma H, Zhang J, Wang Z F, Zhang Y M, Zhang D W, Yonezawa T, Hasegawa M, Zhong Y, Liu W B, Zhang Y, Huang Z Y, Zhang S X, Long R. The yak genome and adaptation to life at high altitude[J].Nature Genetics,2012,44(8):946-949. doi:10.1038/ng.2343.
[7] Cao X F,Bai Z Z,Ma L,Ma S,Ge R L. Metabolic alterations of Qinghai-Tibet plateau pikas in adaptation to high altitude [J]. High Altitude Medicine & Biology,2017,18(3):219-225. doi:10.1089/ham.2016.0147.
[8] Zhang Z R,Du H R,Bai L J,Yang C W,Li Q Y,Li X C,Qiu M H,Yu C L,Jiang Z R,Jiang X Y,Liu L,Hu C M,Xia B,Xiong X,Song X Y,Jiang X S. Whole genome bisulfite sequencing reveals unique adaptations to high-altitude environments in Tibetan chickens [J]. PLoS One,2018, 13(3):e0193597. doi:10.1371/journal.pone.0193597.
[9] Jin L,Mao K,Li J,Huang W Y,Che T D,Fu Y H,Tang Q Z,Liu P L,Song Y,Liu R,Lin X X,Shang D Z,Liu Y H,Liu Y K,Ma J D,Gu Y R,Li X W,Li M Z. Genome-wide profiling of gene expression and DNA methylation provides insight into low-altitude acclimation in Tibetan pigs[J]. Gene,2018,642(5):522-532. doi:10.1016/j.gene.2017.11.074.
[10] 宋娜娜,钟金城,柴志欣,汪琦,何世明,吴锦波,蹇尚林,冉强,蒙欣,胡红春. 三江黄牛全基因组数据分析[J].中国农业科学,2017,50(1):183-194. doi: 10.3864/j.issn.0578-1752.2017.01.016.
Song N N,Zhong J C,Chai Z X,Wang Q,He S M,Wu J B,Jian S L,Ran Q,Meng X,Hu H C. The whole genome data analysis of Sanjiang cattle [J]. Scientia Agricultura Sinica,2017,50(1):183-194.
[11] Yadav B S,Swati S, Srivastava S, Singh N K, Mani A. Whole transcriptome expression profiling and biological network analysis of chickpea during heavy metal stress[J]. Journal of Plant Biochemistry and Biotechnology,2019,28(3):345-352.doi:10.1007/s13562-019-00486-3.
[12] Pang Y Y,Zhang C,Xu M J,Huang G Y,Cheng Y X,Yang X Z. The transcriptome sequencing and functional analysis of eyestalk ganglions in Chinese mitten crab (Eriocheir sinensis) treated with different photoperiods[J]. PLoS One,2019,14(1):e0210414. doi:10.1371/journal.pone.0210414.
[13] 齐晓园,马黎,杨舒黎,孔小艳,严达伟. 牦牛、藏黄牛低氧适应血液生理表征研究[J].黑龙江畜牧兽医,2017(1):9-14.doi: 10.13881/j.cnki.hljxmsy.2017.0116.
Qi X Y,Ma L,Yang X L,Kong X Y,Yan D W. Study on the blood physiological representation for hypoxia adaptation in yaks and Tibetan cattle[J].Heilongjiang Animal Science and Veterinary Medicine,2017(1):9-14.
[14] 王志敏,徐亚欧,杨磊,徐珑洋,王英明.不同海拔地区藏鸡生理生化指标的研究[J].畜牧与饲料科学,2018,39(2):1-7. doi: 10.16003/j.cnki.issn1672-5190.2018.02.001.
Wang Z M, Xu Y O,Yang L,Xu L Y,Wang Y M. A studies on physiological and biochemical indicaters of Tibetan chickens in different altitude areas [J]. Animal Husbandry and Feed Science,2018,39(2): 1-7.
[15] Hérault F, Houée-Bigot M, Baeza E, Bouchez O, Esquerré D, Klopp C, Diot C. RNA-seq analysis of hepatic gene expression of common Pekin,Muscovy,mule and hinny ducks fed ad libitum or overfed[J]. BMC Genomics, 2019,20(1):13. doi:10.1186/s12864-018-5415-1.
[16] Stainier D Y R. Zebrafish genetics and vertebrate heart formation [J]. Nature Reviews Genetics, 2001,2(1):39-48. doi:10.1038/35047564.
[17] Trapnell C, Williams B A, Pertea G, Mortazavi A, Kwan G, van Baren M J, Salzberg S L, Wold B J, Pachter L. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation[J]. Nature Biotechnology,2010,28(5):511-515. doi:10.1038/nbt.1621.
[18] 李丽,缪中纬,辛清武,朱志明,章琳俐,庄晓东,郑嫩珠. 半番鸭与番鸭精巢组织差异表达转录组测序分析[J].中国农业科学,2017,50(18):3608-3619. doi: 10.3864/j.issn.0578-1752.2017.18.016.
Li L,Miao Z W,Xin Q W,Zhu Z M,Zhang L L,Zhuang X D,Zheng N Z. Transcriptome analysis of differential gene expression associated with testis tissue in mule duck and muscovy duck[J]. Scientia Agricultura Sinica,2017,50(18):3608-3619.
[19] Nie H Y,Geng H Y,Lin Y,Xu S P,Li Z G,Zhao Y Z,Su S K. Genome-Wide identification and characterization of Fox genes in the Honeybee, Apis cerana and comparative analysis with other Bee Fox genes[J]. International Journal of Genomics, 2018, 2018:1-12. doi:10.1155/2018/5702061.
[20] 字向东,罗斌,夏威,郑玉才,熊显荣,李键,钟金城,朱江江,张正帆.基于RNA-Seq技术的牦牛体外受精胚胎发育转录组分析[J].中国农业科学,2018,51(8):1577-1589. doi: 10.3864/j.issn.0578-1752.2018.08.015.
Zi X D,Luo B,Xia W,Zheng Y C,Xiong X R,Li J,Zhong J C,Zhu J J,Zhang Z F. Transcriptomic analysis of IVF embryonic development in the Yak (Bos grunniens) via RNA-Seq [J]. Scientia Agricultura Sinica,2018,51(8):1577-1589.
[21] Li Y,Yuan Y. Alternative RNA splicing and gastric cancer[J]. Mutation Research/Reviews in Mutation Research,2016,773:263-273. doi: 10.1016/j.mrrev.2016.07.011.
[22] Zhang J Q,Liu F Z, Cao J T,Liu X L. Skin transcriptome profiles associated with skin color in chickens[J]. PLoS One,2015,10(6): e0127301. doi: 10.1371/journal.pone.0127301.
[23] Cao S Y,Han J Y,Wu J,Li Q Y,Liu S C,Zhang W,Pei Y L,Ruan X A,Liu Z H,Wang X M,Lim B,Li N. Specific gene-regulation networks during the pre-implantation development of the pig embryo as revealed by deep sequencing[J]. BMC Genomics,2014,15(1): 4. doi:10.1186/1471-2164-15-4.
[24] Grafa A,Krebs K, Heininen-Brown M, Zakhartchenko V, Blum H, Wolf E. Genome activation in bovine embryos: review of the literature and new insights from RNA sequencing experiments[J]. Animal Report Science,2014,149 (1-2):46-58. doi: 10.1016/j.anireprosci.2014.05.016.
[25] Jiang Z L,Sun J W,Dong H,Luo O,Zheng X B, Obergfell C,Tang Y,Bi J B, O′Neill R,Ruan Y J,Chen J B,Tian X C. Transcriptional profiles of bovine in vivo pre-implantation development[J]. BMC Genomics,2014,15(1): 756. doi: 10.1186/1471-2164-15-756.
[26] Picelli S, Björklund Å K , Faridani O R , Sagasser S, Winberg G, Sandberg R. Smart-Seq2 for sensitive full-length transcriptome profiling in single cells[J]. Nature Methods,2013,10(11): 1096-1098.doi: 10.1038/NMETH.2639.
[27] Bai X,Zheng Z Q,Liu B,Ji X Y,Bai Y S,Zhang W G. Whole blood transcriptional profiling comparison between different milk yield of Chinese Holstein cows using RNA-seq data [J]. BMC Genomics,2016,17 (7): 512. doi: 10.1186/s12864-016-2901-1.
[28] Zhang B,Qiangba Y Z,Shang P,Lu Y F,Yang Y Z,Wang Z X,Zhang H. Gene expression of vascular endothelial growth factor A and hypoxic adaptation in Tibetan pig[J].Journal of Animal Science and Biotechnology,2016,7(4):21.doi:10.1186/s40104-016-0082-z.
[29] Wang Y,Luo M R,Jiang M F,Lin Y Q,Zhu J J. Comparative analysis of tissue expression and methylation reveals the crucial hypoxia genes in hypoxia-resistant animals[J].Revue Canadienne de Science Animal,2018,98(1):204-212. doi: 10.1139/CJAS-2017-0021.
[30] Zhao Y J,Wang X,Milody N,Hou M. Nitric oxide in red blood cell adaptation to hypoxia[J]. Acta Biochimica et Biophysica Sinica, 2018,50(7):621-634. doi: 10.1093/abbs/gmy055.
[31] Bai D P,Lin X Y,Wu Y,Zhou S Y,Huang Z B,Huang Y F,Li A,Huang X H. Isolation of blue-green eggshell pigmentation-related genes from Putian duck through RNA-seq[J]. BMC Genomics,2019,20(1):66.doi: 10.1186/s12864-019-5436-4.
[32] 张然然,刘华淼,王磊,邢秀梅,苏伟林,高兵. 不同生长时期梅花鹿鹿茸转录组分析[J].畜牧兽医学报,2017,48(2):235-242. doi: 10.11843/j.issn.0366-6964.2017.02.006.
Zhang R R,Liu H M,Wang L,Xing X M,Su W L,Gao B. Transcriptome analysis of sika deer antlers at different stages[J]. Acta Veterinaria et Zootechnica Sinica,2017,48(2):235-242.
[33] 杨琦玥,陈通,兰道亮,熊显荣,候定超,李键. 牦牛肺转录组图谱绘制及特有正选择遗传进化基因探究[J].畜牧兽医学报,2017,48(7):1202-1211. doi: 10.11843/j.issn.0366-6964.2017.07.004.
Yang Q Y,Chen T,Lan D L,Xiong X Y,Hou D C,Li J. Mapping of transcriptome and identification of positively selected genes in lung in Yak[J]. Acta Veterinaria et Zootechnica Sinica,2017,48(7):1202-1211.
[34] Vander Z S,Brocherie F,Kom B L G,Millet G P,Deldicque L,Vander L W J,Girard O,Jaspers R T. Adaptations in muscle oxidative capacity, fiber size, and oxygen supply capacity after repeated-sprint training in hypoxia combined with chronic hypoxic exposure[J]. Journal of Applied Physiology,2018,124(6):1403-1412. doi: 10.1152/japplphysiol.00946.2017.
[35] Zhao T S,Huang X T,Xia Y H. Human heart cell proteins interacting with a C-terminally truncated 2A protein of coxsackie B3 virus: identification by the yeast two-hybrid system[J]. Virus Genes,2016,52(2):172-178. doi: 10.1007/s11262-015-1270-1.
[36] Duggan A T,Kocha K M,Monk C T,Bremer K,Moyes C D. Coordination of cytochrome c oxidase gene expression in the remodelling of skeletal muscle[J]. Journal of Experimental Biology,2011,214(11):1880-1887. doi: 10.1242/jeb.053322.
[37] Sharma A, Halu A, Decano J L, Padi M, Liu Y Y, Prasad R B, Fadista J, Santolini M, Menche J, Weiss S T, Vidal M, Silverman E K, Aikawa M, Barabási A L, Groop L, Loscalzo J. Controllability in an islet specific regulatory network identifies the transcriptional factor NFATC4 which regulates Type 2 Diabetes associated genes[J]. NPJ Systems Biology and Applications,2018,4(1):25-32. doi: 10.1038/s41540-018-0057-0.
[38] Goettsch C,Rauner M,Hamann C,Sinningen K,Hempel U,Bornstein S R,Hofbauer L C. Nuclear factor of activated T cells mediates oxidised LDL-induced calcification of vascular smooth muscle cells[J]. Diabetologia,2011,54(10):2690-2701. doi: 10.1007/s00125-011-2219-0.
[39] Wang W,Yen H T,Chen C H,Soni R,Jasani N,Sylvestre G,Reznik S E. The endothelin-converting Enzyme-1/Endothelin-1 pathway plays a critical role in inflammation-associated premature delivery in a mouse model[J]. The American Journal of Pathology,2008,173(4): 1077-1084. doi: 10.2353/ajpath.2008.080257.
[40] 王延东. 西藏藏猪耐低氧基因ECE1的分子克隆及其表达分析[D]. 泰安:山东农业大学, 2016.doi:107666/dD83391.
Wang Y D. Molecular cloning and expression analysis of the Xizang Tibetan Pig anti-hypoxia gene ECE1[D].Taian:Shandong Agricultural University,2016.
[41] Ferri A, Panariti A, Miserocchi G, Rocchetti M,Comani G B, Rivolta I, Bishop D J. Tissue specificity of mitochondrial adaptations in rats after 4 weeks of normobaric hypoxia[J]. European Journal of Applied Physiology,2018,118(8): 1641-1652. doi: 10.1007/s00421-018-3897-9.
[42] Persoon S,Paulus M,Hirt S,Jungbauer C,Dietl A,Luchner A,Schmid C,Maier L S, Birner C. Cardiac unloading by LVAD support differentially influences components of the cGMP-PKG signaling pathway in ischemic and dilated cardiomyopathy[J]. Heart and Vessels,2018,33(8):948-957. doi: 10.1007/s00380-018-1149-x.
[43] Yu L M,Di W C,Dong X,Li Z,Zhang Y,Xue X D,Xu Y L,Zhang J,Xiao X,Han J S,Liu Y,Yang Y,Wang H S. Melatonin protects diabetic heart against ischemia-reperfusion injury, role of membrane receptor-dependent cGMP-PKG activation[J]. Biochimica et Biophysica Acta-Molecular Basis of Disease,2017,1864(2):563-578. doi: 10.1016/j.bbadis.2017.11.023.
[44] Wu B,Jiang H,Lin R,Cui B,Wen H Z,Lu Z B. Pretreatment with B-type natriuretic peptide protects the heart from ischemia-reperfusion injury by inhibiting myocardial apoptosis[J]. The Tohoku Journal of Experimental Medicine,2009,219(2):107-114. doi: 10.1620/tjem.219.107.
[45] 赵启南,芒来,白东义,赵一萍,李蓓,乌尼尔夫,乌英嘎,苏日嘎,敖乳嘎. 蒙古马高负荷运动训练前后miRNA差异表达分析[J].中国农业大学学报,2018,23(3):61-68. doi: 10.11841 /J. issn.2007-4333.320.2018.3.008.
Zhao Q N,Mang L,Bai D Y,Zhao Y P,Li B,Wuni E F,Wu Y G,Su G R,Ao R G. miRNA analysis of Mongolian horses before and after high-load training[J]. Journal of China Agricultural University,2018,23(3):61-68.