水稻是世界上最重要的粮食作物之一,全球约有一半以上人口以水稻为主食。随着我国人口数量的不断增加,耕地面积因气候环境变化逐年减少,导致粮食安全问题日益严峻。只有不断提高粮食产量,才能从根本上解决粮食问题。水稻千粒质量作为重要的产量性状一直是育种家关注的重点,同时也会间接影响到品质相关性状。水稻千粒质量是多基因控制的数量性状,是由主效基因和一些分布不均衡的微效多基因共同作用的,所以进一步定位和克隆新的控制粒质量的基因是十分必要的[1]。水稻千粒质量由籽粒的体积和饱满度决定,其中水稻籽粒体积一般由粒长、粒宽和粒厚决定,饱满度一般由灌浆速率、活跃灌浆期时间等指标衡量[2]。近年来已经有很多控制粒型的基因被克隆,目前克隆的大部分控制水稻粒型的基因是通过转录调节因子途径、油菜素内酯、G-蛋白信号、生长素以及泛素连接酶途径调控水稻粒型性状。其中转录调节因子调控粒型途径如GS2(GL2、LGS1)编码蛋白质可以与DELLA蛋白产生互作,作用于赤霉素信号传导途径,可以促进氮元素的吸收和在植物体内的转运,同时调控植物的光合作用、碳水化合物的代谢和细胞内转运等,通过促进水稻细胞分裂和膨大调节水稻粒长和粒质量[3-4]。最新研究表明,GS2基因突变可能影响OsGRF-miR396胁迫响应应答,从而增强水稻苗期的耐冷性[5],也表明了GS2基因存在一因多效。GW8编码包含SBP结构域的转录因子,能够与GW7启动子结合并抑制其表达,导致水稻籽粒变短圆[6]。GS9编码转录激活因子与OFPs家族蛋白(转录抑制家族)OsOFP14和OsOFP8相互作用通过影响细胞分裂调节水稻粒长、粒宽和外观品质[7]。编码G-蛋白信号转导途径,如GS3负调节水稻粒质量和粒长,蛋白表达主要在水稻幼穗,随着幼穗的发育表达量逐渐减少,在其他组织如叶片、胚芽、茎端分生组织中有微弱表达,在根中大量表达[8]。泛素连接酶途径,如GW2编码环型E3泛素连接酶,通过在细胞质中调节底物与受体酶的结合降解,从而负调节水稻细胞的分裂,影响粒宽和粒质量[9]。GW2等位基因WY3因功能缺失,无法促使底物被识别降解,从而可以激活颖壳外稃的细胞分裂,增加了灌浆速率,显著增加水稻的粒宽和千粒质量,同时也正调控水稻单株穗数、延长生育期,并显著地降低每穗粒数和主穗长度[10]。调控生长素转导途径,如TGW6编码IAA-葡萄糖水解酶,控制IAA-葡萄糖的水解,通过影响细胞分裂从而负调控籽粒质量[11]。TGW3(GL3.3)编码蛋白激酶OsSK41能够磷酸化生长素应答因子OsARF4(生长素途径中的转录抑制因子)。OsSK41-OsARF4通过负调控生长素信号,调节水稻颖壳中细胞的大小和数量,进而负调控水稻粒长和粒质量[12-13]。同时TGW3与之前发现的GS3存在上位性效应,GS3与TGW3同时存在可以导致水稻粒型的显著增大[14]。油菜素内酯调控途径,如GW5编码蛋白调节油菜素内酯信号传导,可以通过与GSK2激酶相互作用从而抑制GSK2激酶活性。GSK2激酶可以磷酸化OsBZR1和DLT蛋白,GW5的表达会增加细胞核中未磷酸化的OsBZR1和DLT蛋白的积累,从而调节油菜素内酯响应基因的表达水平和生长响应[15]。QTL-seq是近年来发展起来的一种新型的QTL定位手段,是将传统的混合分组分析法(Bulk segregant analysis,BSA)和高通量测序结合起来快速定位到性状相关QTL,节约了大量构建群体的时间,Park 等[16]通过QTL-seq和RNA-seq技术定位了辣椒中影响辣椒素含量的主效基因,Luo等[17]通过QTL-seq和KASP分型技术定位了花生脱壳率的主效QTL。QTL-seq目前已经在水稻粒长[18]、水稻耐寒性[19]、大白菜抗TuMV[20]、盐胁迫下大麦的籽粒育性[21]与抗病性、西瓜枯萎病[22]、油菜有限花序[23]、鹰嘴豆粒质量[24]、卷心菜和西兰花开花时间[25]等研究中得到了广泛的应用,证明了QTL-seq在基因定位中的可行性。现阶段大部分水稻粒型定位研究大都是以高世代的RIL群体或染色体单片段代换系为材料,以分子标记或高密度遗传图谱作为主要的定位手段进行QTL定位,构建群体时间长,研究成本较高。通过QTL-seq定位的遗传位点报道较少。对影响粒质量的关键表型性状进行鉴定分析,进一步挖掘可能控制粒质量的候选基因,丰富粒质量性状多基因调控的理论基础,以期可以在实际育种中加以应用。
1 材料和方法
1.1 试验材料与田间处理
材料为国家植物航天育种工程技术研究中心前期收集的优良种质资源重粒亲本B233和轻粒亲本B91以及杂交繁育的F2群体。于2017年早造进行杂交,2017年晚造种植F1(6株×6株小区),于2018年早造双垄种植F2 506株全部收获并进行表型考察。
所有的田间处理试验均在华南农业大学国家植物航天育种工程技术中心试验田完成,田间种植由实验室统一管理。
1.2 B91及B233产量相关性状的考察
收获2 a四造亲本种子在温室自然风干,对亲本进行粒长、粒宽、千粒质量、单株穗数、穗粒数等表型性状的考察。
1.3 B91及B233籽粒扫描电镜观察
1.3.1 颖壳表面扫描电镜观察 水稻颖壳用2.5%~5.0%的戊二醛(采用0.1~0.2 mol/L的PBS(pH值6.8)稀释25%戊二醛至浓度为2.5%~5.0%)固定。为了让固定液充分浸入材料,当颖壳放入固定液后进行真空处理,然后在4 °C的条件下固定1~3 h后吸出固定液,加入0.1 mol/L pH值6.8的PBS漂洗1 h,期间换漂洗液3~4次;吸出PBS后用乙醇梯度干燥,即30%-50%-70%-80%-90%-100%-100%,样品在每级乙醇中停留30 min;经无水乙醇干燥后的颖壳用解剖针剖开,使用导电双面胶粘贴在载物台上,喷金后使用XL-30-ESEM型号扫描电镜观察。
1.3.2 籽粒胚乳淀粉粒扫描电镜观察 收获已经完成灌浆的水稻籽粒(本研究为开花40 d后),将成熟籽粒自然风干后剥除颖壳,用镊子掰开,留出中部胚乳腹部作为观察样本,将观察样本的截面用导电胶粘贴置于载物台上,喷金后使用XL-30-ESEM型号扫描电镜观察。
1.4 B233及B91灌浆速率的测定
在水稻孕穗期选取长势整齐的单株挂牌标记,当穗部露出外叶鞘3 cm时,在此穗的剑叶上标记抽穗日期,统一于15:00开始标记,取样时间也统一在15:00进行。对标记后的穗子进行跟踪调查,在穗子中部颖花开放的时间进行定穗,记为开花0 d,并在开花的颖壳上用防水笔标记。自开花的第5天起,每5 d取样一次,直到开花的第40天粒质量不再发生变化为止。每次每个材料取100粒,取样均取水稻穗的优势粒进行测量,保证数据的准确性。取回的穗子立即放入冰袋并带回实验室测量鲜质量,在60 ℃的烘箱内烘72 h至恒质量测量干质量。灌浆动态用Richards方程拟合。
1.5 QTL-seq分析
对F2种子进行粒质量考察,在F2群体中筛选出极端轻粒单株30株和极端重粒单株30株,各取叶片2 g,用CTAB法提取叶片DNA,提取后测量DNA浓度和纯度,根据浓度等量混合F2群体的DNA。
2个混合池DNA和2个亲本DNA建库进行30×的全基因组重测序,测序得到的原始数据经过过滤后进行SNPs和InDels注释。以对照亲本作为参考,计算每个子代混合池在所有的SNP分离位点的亲本基因型频率,即SNP-index。采用滑动窗口策略来绘制2个子代混合池的SNP-index图(以2 Mb为窗口,100 kb为步长),并过滤掉2 M范围内小于10个SNP位点的区域。为了减少测序错误和比对错误的影响,同时过滤掉子代SNP-index都小于0.3,SNP深度都小于6的位点。得到2个子代混合池的SNP-index图后,计算分离位点在子代2个极端池中基因频率的差值(△SNP-index),然后采取滑动窗口的策略绘图。并进行1 000次置换检验,选取99%置信水平作为选取的阈值,超过阈值外的区域为潜在的QTL候选区域。对候选SNPs所在的编码基因进行统计,发掘潜在的与性状相关的候选基因。
1.6 注释基因功能及表达位置分析
通过国家水稻数据中心对于注释基因的蛋白结构域注释和预测功能及NCBI网站对候选区域内注释基因的转录组分析进一步筛选候选基因。
1.7 数据处理
表型数据的录入、整理与柱状图由Excel 2016完成,使用SPSS 20完成数据相关性及方差分析等。
2 结果与分析
2.1 亲本表型性状的鉴定和扫描电镜观察
2.1.1 B233和B91的表型性状对比 2 a共四造对B233和B91籽粒形态进行调查(图1),B91与B233粒长、粒宽、粒厚、千粒质量均具有显著差异,其中以千粒质量差异最大,所以在F2群体中以千粒质量作为主要考察性状进行极端单株的筛选,构建混合池。
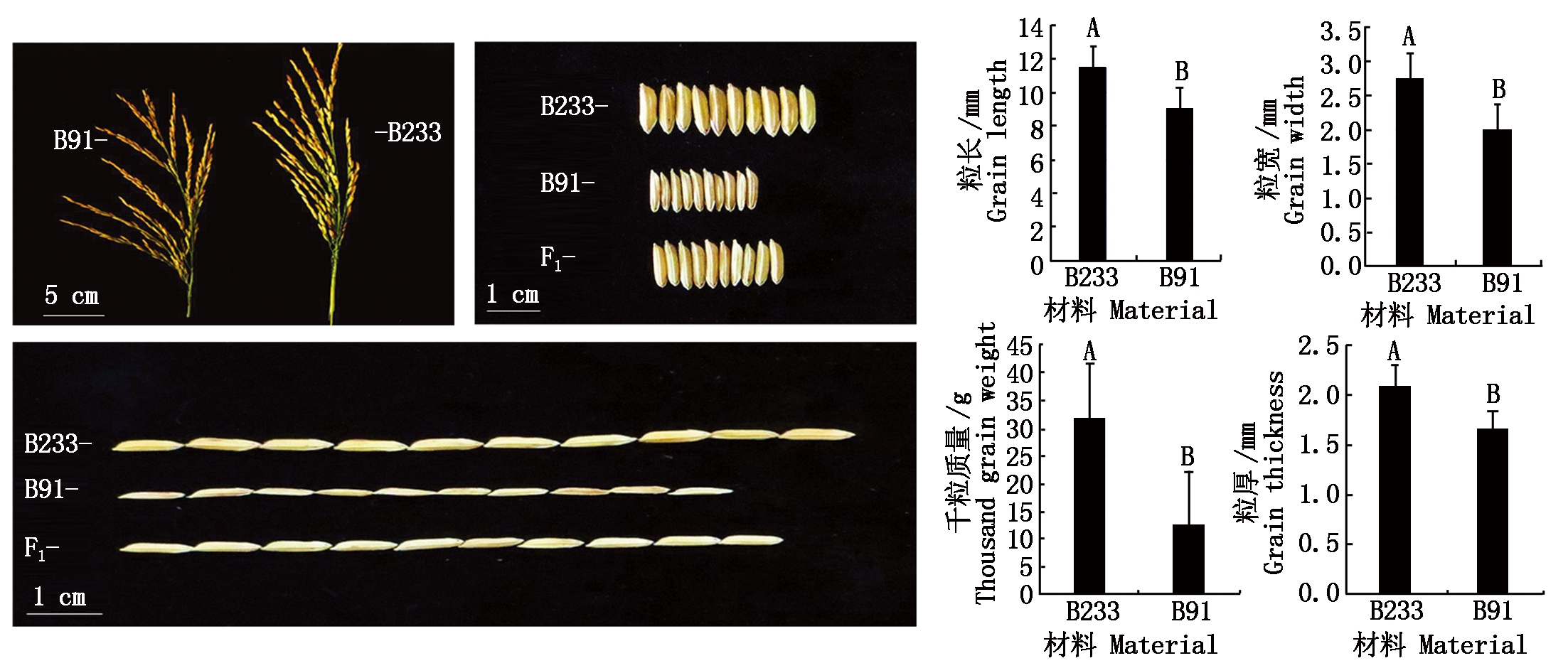
不同字母表示亲本间差异极显著(P<0.01)。 Different letters indicate extremely significant difference (P<0.01)between B233 and B91.
图1 B233、B91及F1穗部及籽粒形态对比
Fig.1 Comparison of the morphology of the panicles and grains of B233, B91 and F1
2.1.2 B91与B233灌浆速率对比 灌浆速率等相关指标也会直接影响籽粒充实度并影响到最终粒质量,所以本研究对比了B91及B233灌浆速率(图2)的相关指标。B91的起始生长势、活跃灌浆期时间、平均灌浆速率、最大灌浆速率等指标均小于B233(表1)。其中平均灌浆速率、活跃灌浆期等指标都直接或间接影响最终籽粒质量。灌浆动态指标也是导致B233和B91粒质量差异和品质差异的重要因素之一。
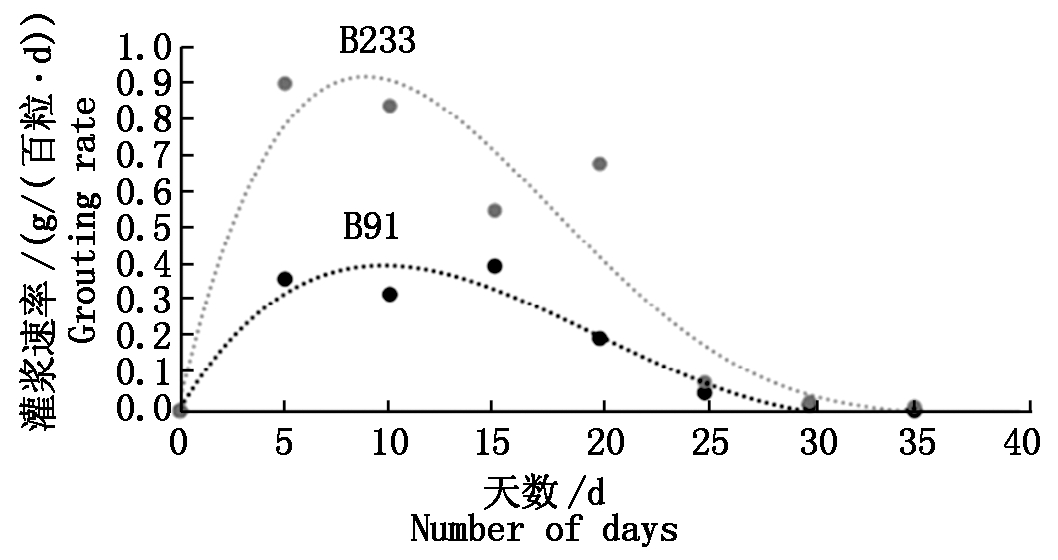
图2 B91和B233灌浆速率的比较
Fig.2 Comparison of the filling rates of B91 and B233
表1 Richards方程拟合灌浆参数
Tab.1 Richards equation fitting grout parameters

样品SampleA/gR0RmaxRmeanDmaxD/dWmaxR2B911.335 60.1970.3970.0619.5719.60.630.997 5B2333.059 90.3540.8840.13610.0720.21.640.983 4
注:A.籽粒终极生长量;R0.起始生长势;Rmax.最大灌浆速率;Rmean.平均灌浆速率;Dmax.达到最大灌浆速率的时间;Wmax.灌浆速率最大时的籽粒质量;D.活跃灌浆期;R2.方程系数。
Note: A.Grain final growth; R0.Initial growth potential; Rmax.Maximum grouting rate; Rmean.Average grouting rate; Dmax.Time to reach the maximum grouting rate; Wmax.Grain weight when the grouting rate is maximum; D.Active grouting period; R2.Coefficient of equation.
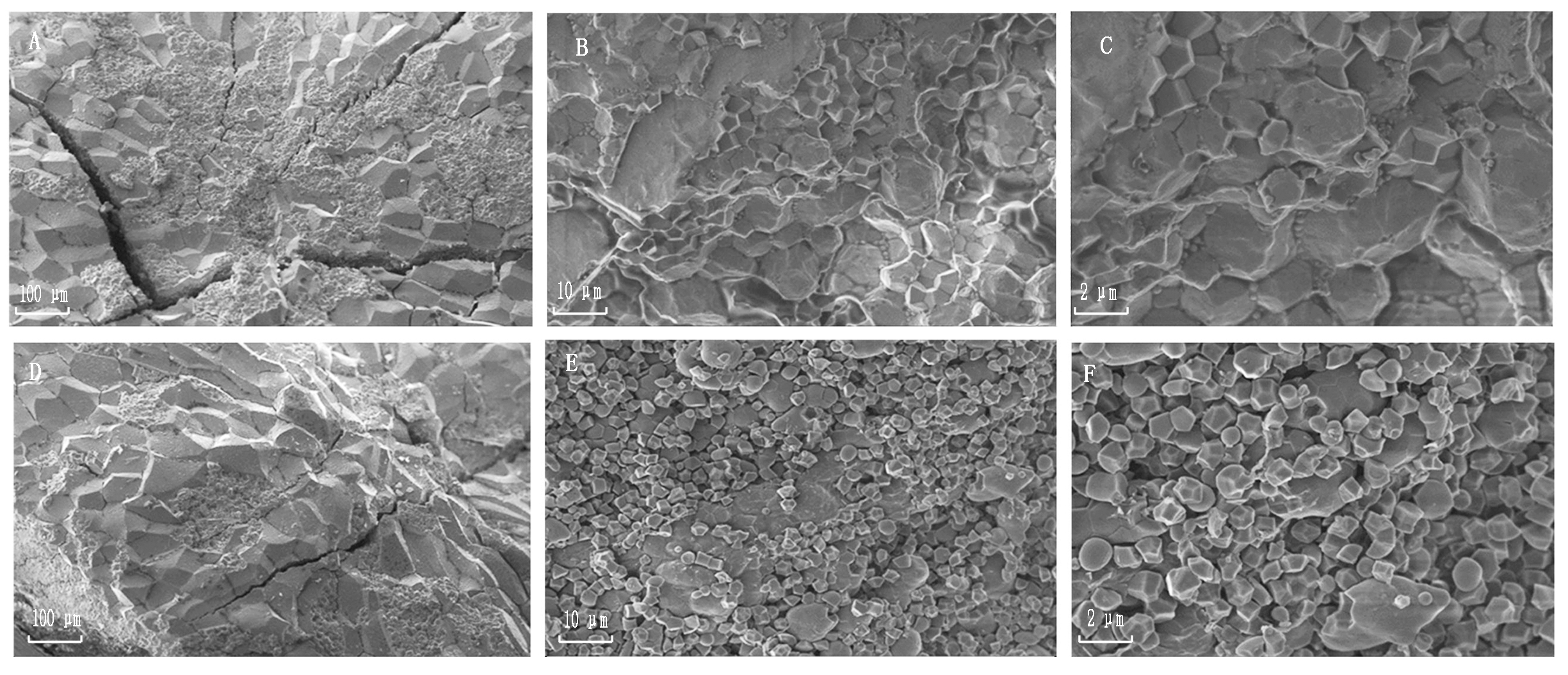
2.1.3 B233和B91颖壳表面和胚乳的显微观察和扫描电镜观察 由150×、500×和1 000×扫描电镜视图(图3)可以看到,细胞大小和细胞数量在亲本间均有明显差异,表明二者为B91和B233粒型差异的决定因素。根据300×扫描电镜观察,单位面积内B91纵向细胞列数与B233纵向细胞列数相比有显著差异,横向细胞行数没有显著差异;颖壳细胞长和细胞宽均有极显著差异(表2)。
B91精米细长,米质坚硬,米粒亮透明如玻璃状,米粒大小均匀,胚乳整齐美观,无腹白心白,对B91成熟籽粒腹部胚乳细胞的扫描电镜观察淀粉粒呈明显的多边形、排列紧密、颗粒间基本没有空隙,

A-C.B91扫描电镜视图;D-F.B233扫描电镜视图;A,D.观察倍数150×;B,E.观察倍数500×;C,F.观察倍数1 000×。 A-C.B91 scanning electron microscope view; D-F.B233 scanning electron microscope view; A, D. Observation multiple 150 ×;B, E. Observation multiple 500×; C, F. Observation multiple 1 000×.
图3 B91、B233颖壳扫描电镜观察
Fig.3 Scanning electron microscope observation of B91 and B233
表2 300×扫描电镜视图颖壳单位面积下细胞长宽和细胞数量对比
Tab.2 Comparison of cell length and width and cell number per unit area in 300× scanning electron microscope

样品Sample 细胞长(平均)/μmCell length 细胞宽(平均)/μmCell width横向细胞行数(平均)/个 Horizontal cell number 纵向细胞列数(平均)/个Longitudinal cell numberB9121.875±1.567A22.768±2.301A13±0.816A18±0.943AB23331.964±1.413B30.213±1.889B12±0.943A12±0.471B
注:同列不同大写字母表示样品间差异极显著。
Nota:Different capital letter represents extremely significant difference between the two samples.
A-C.B91扫描电镜视图;D-F.B233扫描电镜视图;A,D.观察倍数300×;B,E.观察倍数3 000×;C,F.观察倍数6 000×。 A-C.B91 scanning electron microscope view; D-F.B233 scanning electron microscope view; A,D. Observation multiple 300×; B,E. Observation multiple 3 000×; C,F. Observation multiple 6 000×.
图4 B91、B233胚乳扫描电镜观察
Fig.4 Scanning electron microscope observation of B91 and B233 endosperm
籽粒填充度较好(图4-A-C)。B233具腹白或心白,稻米品质比B91稍差,对B233成熟籽粒腹部胚乳细胞进行扫描电镜观察籽粒淀粉粒多面体的棱角不明显,部分淀粉粒有椭圆形、排列疏松、颗粒间存在较大空隙、透光性稍差(图4-D-F)。
2.2 QTL-seq分析
2.2.1 F2群体粒质量分布及遗传分析 水稻轻粒亲本B91和重粒亲本B233杂交获得F1,自交得F2,分别考察粒型相关性状。F1及F2群体平均粒长、粒宽、粒厚和千粒质量介于两亲本之间(表3),对F2群体共506个单株进行粒质量的考察,粒质量分布呈标准正态分布(图5),表明在B233和B91中控制亲本粒质量性状的为数量位点基因,F2群体粒质量极端值明显,是良好的QTL-seq作图群体。挑选了粒质量极值单株各30株,方差分析显示,选取的单株粒质量具有极显著差异,可以用作测序样本。分别提取单株叶片DNA并留种。
表3 水稻亲本、F1(mean)、F2(mean) 粒长、粒宽、粒厚、千粒质量
Tab.3 B91 (mean), B233 (mean), F1 (mean), F2 (mean) grain length, grain width, grain thickness, thousand grain weight
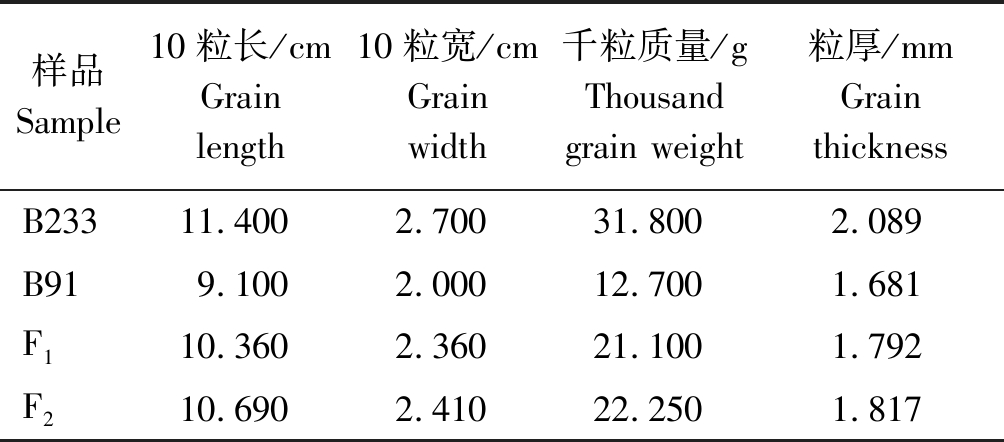
样品Sample10粒长/cmGrain length10粒宽/cmGrain width 千粒质量/gThousand grain weight粒厚/mmGrain thicknessB23311.4002.70031.8002.089B919.1002.00012.7001.681F110.3602.36021.1001.792F210.6902.41022.2501.817
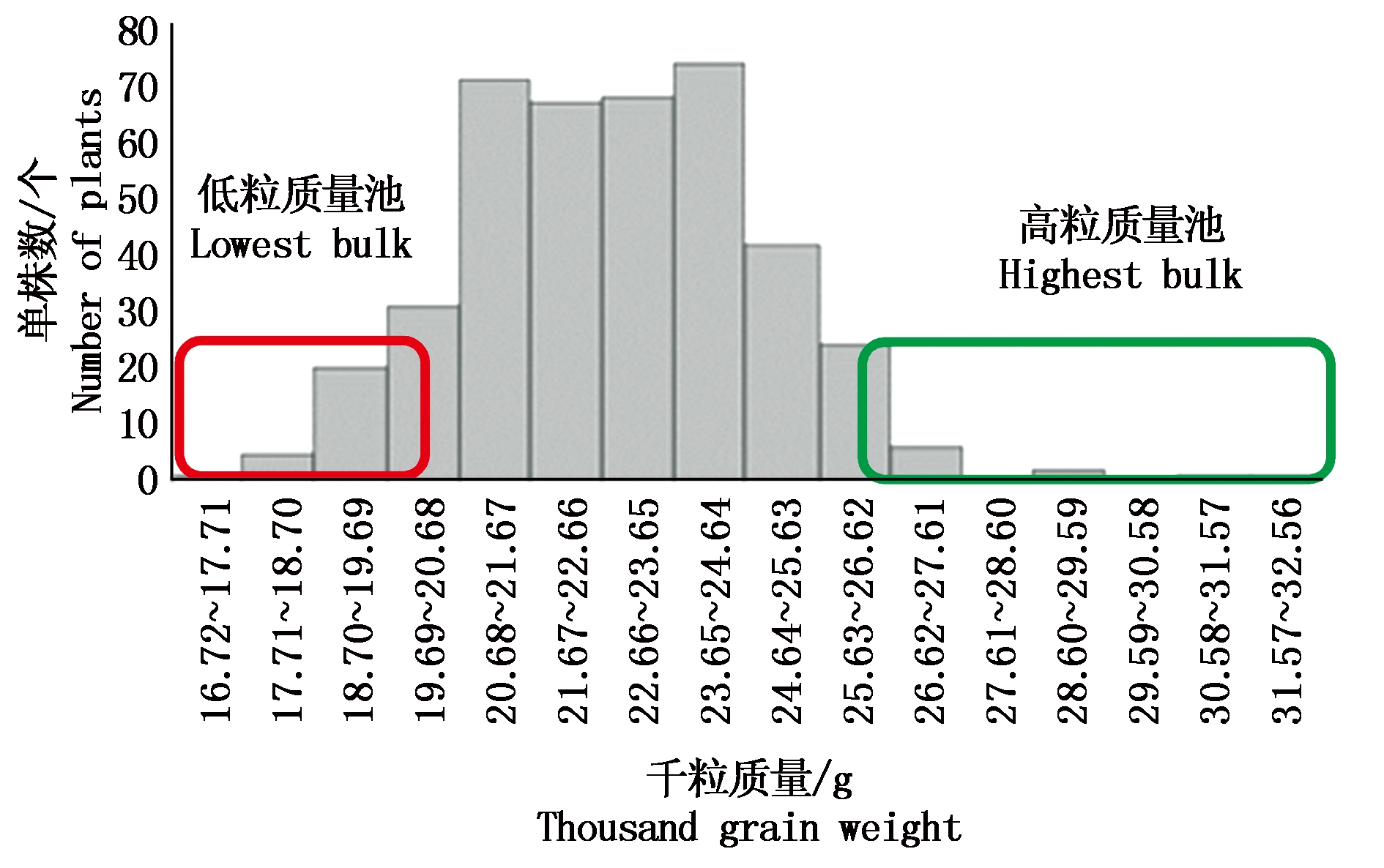
图5 F2群体粒质量分布
Fig.5 Grain weight distribution in the F2
2.2.2 基于双亲分离SNP标记的BSA 将从群体中筛选出双亲分离的SNP作为标记,以便后续进行BSA法QTL定位(QTL-seq)。过滤获得可以用于后续QTL-seq分析的有效SNP位点共430 762个。SNP窗口总数量为3 620个,SNP数量 ≥ 10的窗口数量为3 234个,占SNP窗口总数量的89.34%。根据双亲分离的SNP标记定位粒质量相关的QTL(图6)。
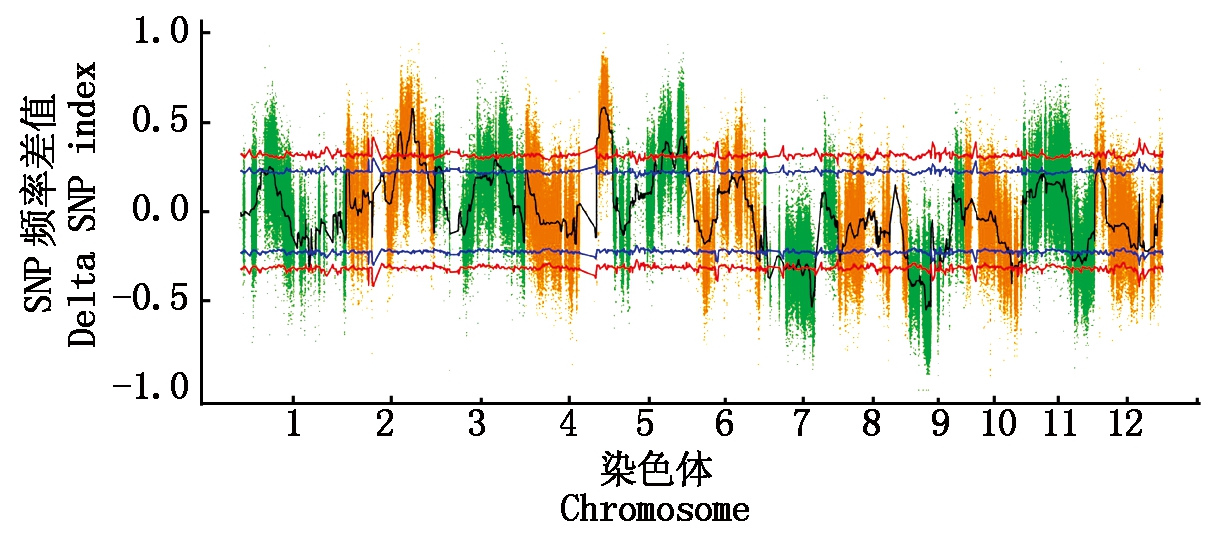
以2个子代SNP-index作差,计算△(SNP-index)。计算过程进行1 000次置换检验,获得各个位点以及窗口的△(SNP index)95%置信区间(蓝色阈值线,即相当于P =5%),以及99%置信区间(红色阈值线,即相当于P=1%)。
With the difference of two daughter SNP-index, calculate △ (SNP-index). The calculation process performs 1 000 displacement tests to obtain a △ (SNP index) 95% confidence interval for each site and window (blue threshold line, which is equivalent to P=5%), and a 99% confidence interval (red threshold line, That is equivalent to P=1%).
图6 Δ(SNP index)总分布图
Fig.6 Total distribution of Δ(SNP index)
因为两亲本遗传背景差异较大,前期未对两亲本遗传背景进行人为纯化,故本次QTL-seq分析定位到的粒质量QTL数量较多,且本次QTL-seq分析结果比较理想。为了更有针对性的对粒质量性状进行分析,因为调控大粒的QTL在育种中更具有研究价值,所以在本研究主要关注超过正阈值区99%置信区间的QTL位置,并进一步在这些QTL区间内筛选可能调控粒质量的基因。可以看到在水稻的2,4,5号染色体上共有5个与粒质量性状相关的QTL区间(图6),其中2号染色体和4号染色体的2个QTL峰值最为明显。在正阈值区内定位到的5个QTL最小的物理区间为2.1 M(Chr2:20.9~23.0 M),最大的物理区间6.3 M(Chr4:28.9~35.2 M)(表4)。
表4 水稻粒质量QTL物理位置
Tab.4 Physical location of rice grain weight QTL
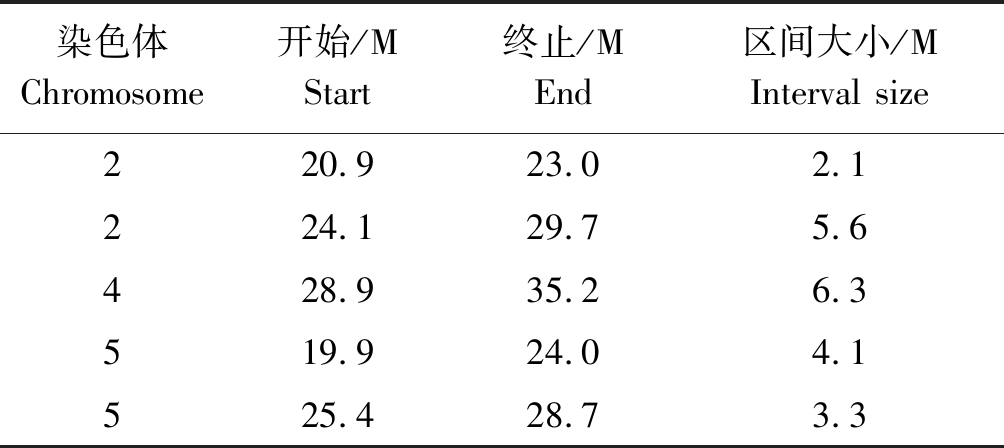
染色体Chromosome开始/MStart终止/MEnd区间大小/MInterval size 220.923.02.1224.129.75.6428.935.26.3519.924.04.1525.428.73.3
2.3 正阈值区内候选基因的筛选
2.3.1 正阈值区△(SNP-index)>0.7 SNP位点和InDel位点的筛选 对正阈值区QTL位点内的基因进行△(SNP-index)分析,重点关注在子代混合池内△(SNP-index)在0.7以上的SNP位点,选出在23个基因发生非同义突变及剪接位点上的突变(表5),同时选出11个基因的Indel位点进行进一步分析(表6)。共筛选出34个候选基因,其中32个基因未报道克隆,这些位点可能对应影响粒型性状的候选基因。
表5 正阈值区内候选基因的注释信息
Tab.5 Annotation information of candidate genes in the positive threshold region
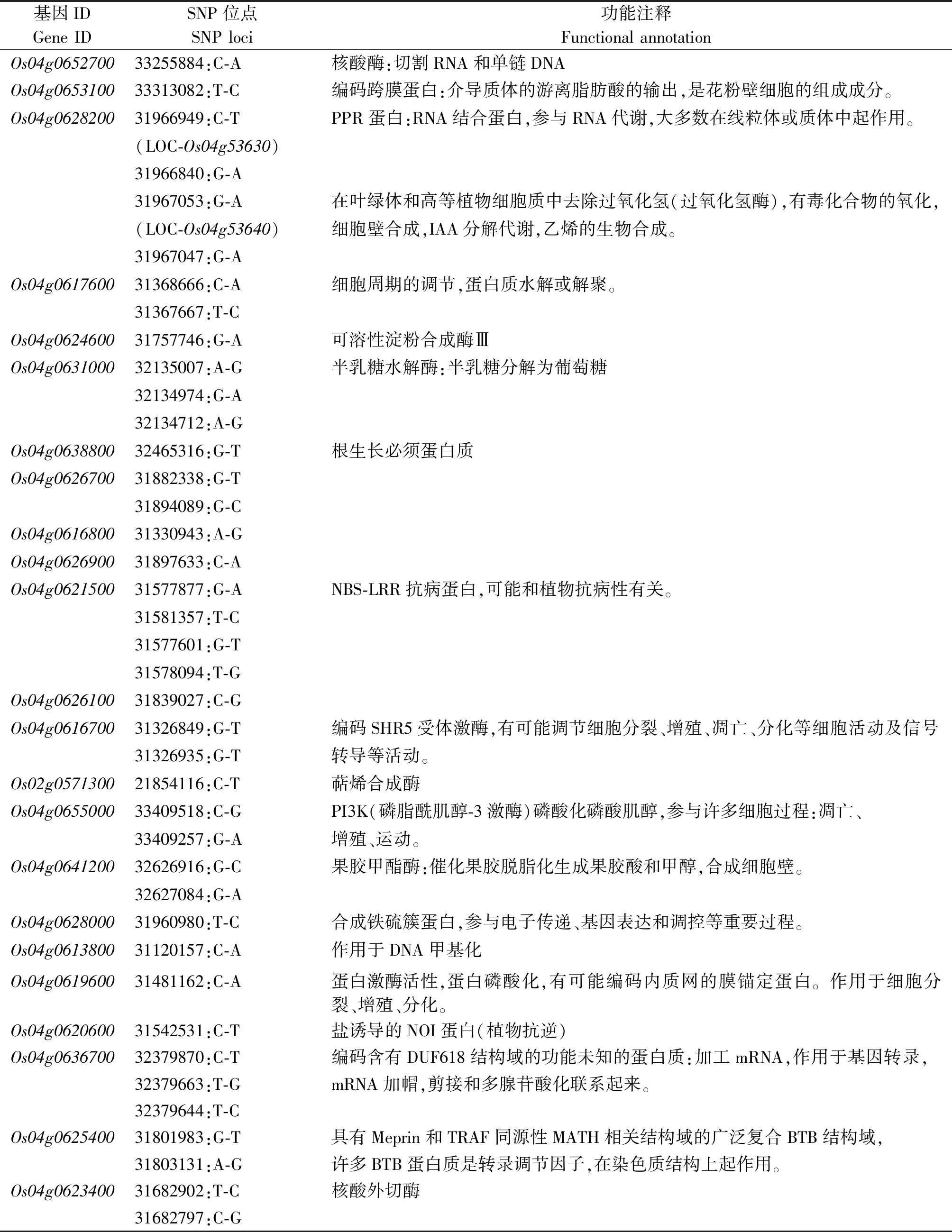
基因IDGene IDSNP位点SNP loci功能注释Functional annotationOs04g065270033255884:C-A核酸酶:切割RNA和单链DNAOs04g065310033313082:T-C编码跨膜蛋白:介导质体的游离脂肪酸的输出,是花粉壁细胞的组成成分。Os04g062820031966949:C-TPPR蛋白:RNA结合蛋白,参与RNA代谢,大多数在线粒体或质体中起作用。(LOC-Os04g53630)31966840:G-A31967053:G-A在叶绿体和高等植物细胞质中去除过氧化氢(过氧化氢酶),有毒化合物的氧化,(LOC-Os04g53640)细胞壁合成,IAA分解代谢,乙烯的生物合成。31967047:G-AOs04g061760031368666:C-A细胞周期的调节,蛋白质水解或解聚。31367667:T-COs04g062460031757746:G-A可溶性淀粉合成酶ⅢOs04g063100032135007:A-G半乳糖水解酶:半乳糖分解为葡萄糖32134974:G-A32134712:A-GOs04g063880032465316:G-T根生长必须蛋白质Os04g062670031882338:G-T31894089:G-COs04g061680031330943:A-GOs04g062690031897633:C-AOs04g062150031577877:G-ANBS-LRR抗病蛋白,可能和植物抗病性有关。31581357:T-C31577601:G-T31578094:T-GOs04g062610031839027:C-GOs04g061670031326849:G-T编码SHR5受体激酶,有可能调节细胞分裂、增殖、凋亡、分化等细胞活动及信号31326935:G-T转导等活动。Os02g057130021854116:C-T萜烯合成酶Os04g065500033409518:C-GPI3K(磷脂酰肌醇-3激酶)磷酸化磷酸肌醇,参与许多细胞过程:凋亡、33409257:G-A增殖、运动。Os04g064120032626916:G-C果胶甲酯酶:催化果胶脱脂化生成果胶酸和甲醇,合成细胞壁。32627084:G-AOs04g062800031960980:T-C合成铁硫簇蛋白,参与电子传递、基因表达和调控等重要过程。Os04g061380031120157:C-A作用于DNA甲基化Os04g061960031481162:C-A蛋白激酶活性,蛋白磷酸化,有可能编码内质网的膜锚定蛋白。作用于细胞分裂、增殖、分化。Os04g062060031542531:C-T盐诱导的NOI蛋白(植物抗逆)Os04g063670032379870:C-T编码含有DUF618结构域的功能未知的蛋白质:加工mRNA,作用于基因转录,32379663:T-GmRNA加帽,剪接和多腺苷酸化联系起来。32379644:T-COs04g062540031801983:G-T具有Meprin和TRAF同源性MATH相关结构域的广泛复合BTB结构域,31803131:A-G许多BTB蛋白质是转录调节因子,在染色质结构上起作用。Os04g062340031682902:T-C核酸外切酶31682797:C-G
注:SNP位点为△(SNP-index)>0.7的位点。
Note: The SNP locus is a locus with △(SNP-index)>0.7.
表6 Indel位点筛选
Tab.6 Indel site screening
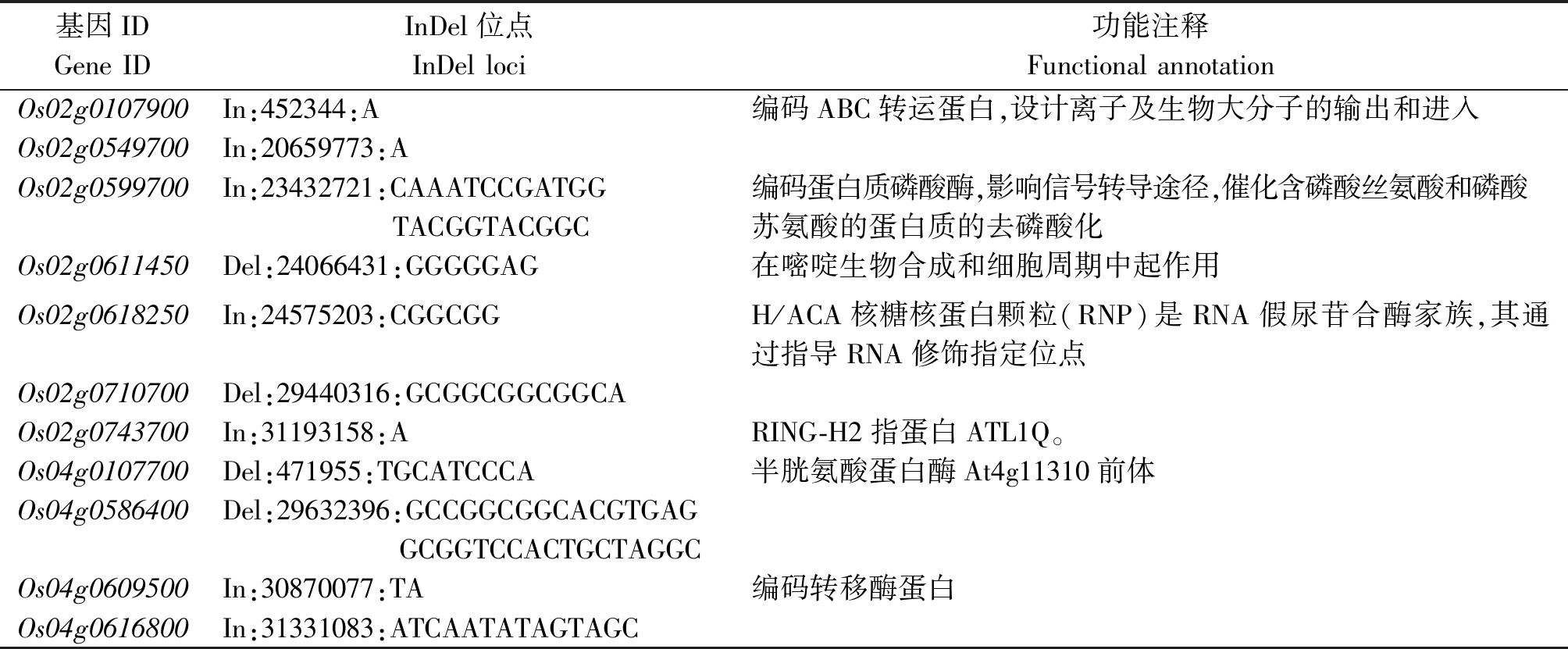
基因IDGene IDInDel位点InDel loci功能注释Functional annotationOs02g0107900In:452344:A编码ABC转运蛋白,设计离子及生物大分子的输出和进入Os02g0549700In:20659773:AOs02g0599700In:23432721:CAAATCCGATGG编码蛋白质磷酸酶,影响信号转导途径,催化含磷酸丝氨酸和磷酸TACGGTACGGC苏氨酸的蛋白质的去磷酸化Os02g0611450Del:24066431:GGGGGAG在嘧啶生物合成和细胞周期中起作用Os02g0618250In:24575203:CGGCGGH/ACA核糖核蛋白颗粒(RNP)是RNA假尿苷合酶家族,其通过指导RNA修饰指定位点Os02g0710700Del:29440316:GCGGCGGCGGCAOs02g0743700In:31193158:ARING-H2指蛋白ATL1Q。Os04g0107700Del:471955:TGCATCCCA半胱氨酸蛋白酶At4g11310前体Os04g0586400Del:29632396:GCCGGCGGCACGTGAG GCGGTCCACTGCTAGGCOs04g0609500In:30870077:TA编码转移酶蛋白Os04g0616800In:31331083:ATCAATATAGTAGC
2.3.2 注释基因的转录组信息筛选及预测功能分析 查询NCBI网站注释基因的转录组信息,主要筛选出在种子和花中特异表达的基因(图7),同时根据基因的预测功能对定位到的基因进行筛选,共获得6个可能与粒质量性状相关的候选基因:Os04g0653100、Os04g0617600、Os04g0624600、Os04g0631000、Os04g0619600、Os02g0611450。其中,Os04g0617600、Os04g0619600编码蛋白激酶或转录调节因子作用,调控细胞凋亡、分化、增殖等细胞周期和生物合成等过程;Os04g0624600、Os04g0631000与碳水化合物的合成与分解相关,可能调控稻米的粒型与品质;Os04g0653100编码蛋白控制生物大分子的运输,同时也是花粉壁细胞的重要组成成分;Os02g0611450编码的蛋白质可能调控生物大分子的合成和细胞周期。
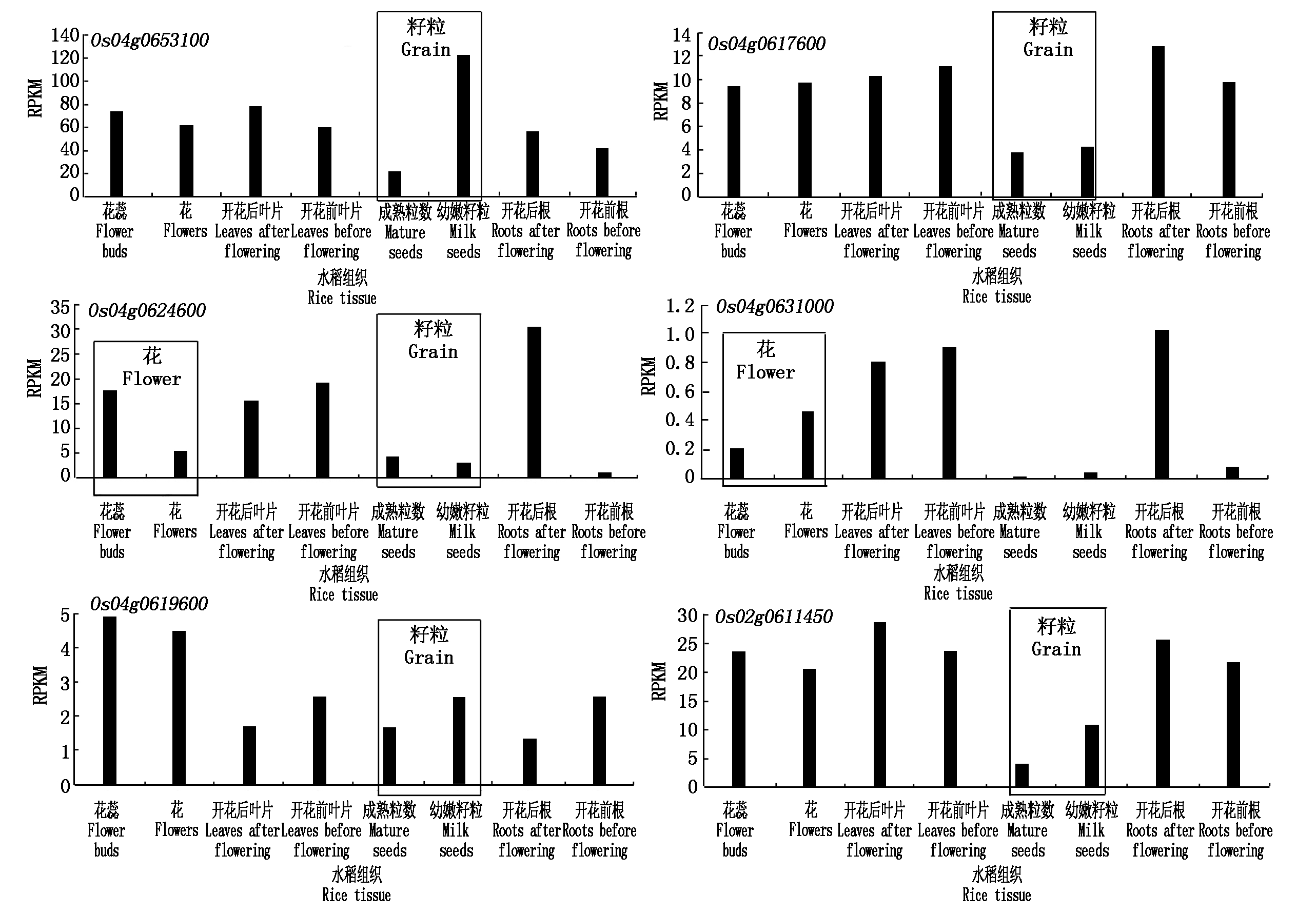
图7 NCBI网站中注释基因的转录组信息
Fig.7 Transcriptome information of annotated genes in the NCBI website
3 讨论与结论
3.1 粒长、粒宽、粒厚和灌浆指标是影响粒质量的关键生理性状
千粒质量既是决定水稻单产的重要指标之一,也是衡量水稻品种优良与否的重要因素。本研究通过扫描电镜观察水稻颖壳和胚乳,旨在细胞层面解释亲本粒质量之间的差异。根据水稻颖壳扫描电镜观察,B233与B91粒长、粒宽、粒厚之间的差异由细胞大小和细胞数量共同决定[26]。水稻籽粒垩白的形成主要是由于水稻胚乳中存在淀粉颗粒区域性的排列松散而导致胚乳腹部存在一些空腔,而这些空腔的存在除了会影响稻米品质之外,因为灌浆不充分,也会影响到水稻籽粒质量等相关性状[27]。通过Richards对灌浆速率曲线进行拟合,导出相关灌浆参数,B233最大灌浆速率,活跃灌浆时期等指标均大于B91,这些指标直接影响到籽粒质量。根据朱庆森等[28]的方法,因为N>1,所以(W/A)N>W/A,可以视为库容对灌浆速率较小,所以B91和B233均属于源限制性品种,所以受播种环境灌浆物质影响较大,边缘效应体现的较为明显,所以在粒质量调查中应尽量保证在同一生长条件下进行考察。因此,水稻籽粒长、宽、厚以及胚乳形态灌浆速率等是考察水稻粒质量的重要指标,对控制这些性状遗传位点的挖掘具有重要意义。
3.2 QTL-seq是鉴定粒质量遗传位点的有效手段
目前,大多数QTL定位研究采用传统的遗传标记来定位遗传位点,如SSR标记、RFLP标记等,但是传统遗传标记开发成本较高,在水稻染色体上覆盖密度低,分布不均匀等问题制约了相关遗传定位研究[29]。同时开发高密度遗传图谱的成本也较高,同时材料选择也十分重要,构建群体的时间也较长,不适宜大规模使用[30]。本研究是基于全基因组重测序的高密度SNP标记进行QTL-seq分析,而QTL-seq技术的发展很好地解决了构建群体时间长,测序费用昂贵等问题。在本研究通过使用2个籼稻亲本B91及B233构建的F2群体进行QTL-seq分析,在正阈值区共定位到了5个与粒质量相关的QTL,通过分析测序数据筛选出△(SNP-index)>0.7的位点和InDel位点,共在正阈值区筛选出34个候选基因(SNP突变基因23个,Indel突变基因11个),其中包括2个已经克隆的基因:NAL1(Chr4)[31]、OsJAZ1(Chr4)[32]。NAL1主要在叶鞘、叶片、茎秆和幼穗中表达,主要影响水稻叶片的宽窄[31]、OsJAZ1是一个茉莉酮酸酯ZIM结构域蛋白。OsJAZ1和OsCOI1b之间相互作用影响水稻小穗发育,OsJAZ1主要在幼穗和花中表达[32],这些基因在一定程度上影响水稻的产量。这些共定位结果一方面验证了本研究的可靠性,另一方面在前人的研究中均未提及这2个基因对于水稻粒质量及相关性状的影响,也暗示了这些基因可能存在一因多效的情况。同时通过注释基因功能和共筛选出32个没有报道过的基因,为后续粒质量基因的验证及功能分析提供了基础。
3.3 水稻粒型性状可能受到蛋白激酶和转录因子相关基因的调控
目前,克隆与水稻粒型和产量相关基因很多与转录调节因子、泛素连接酶和蛋白激酶磷酸化相关。本研究筛选到6个可能与粒质量相关的基因中有2个基因编码蛋白质起到蛋白激酶或转录调节因子作用,Os04g0617600主要编码AAA家族的ATP酶。AAA家族的ATP酶在植物细胞中参与许多活动,包括细胞周期调节、蛋白质水解与合成、细胞内大分子转运等,同时也充当DNA解旋酶和转录因子。Os04g0619600编码的蛋白质为蛋白激酶作用,蛋白激酶按照底物的不同可分为三类,丝氨酸/苏氨酸蛋白激酶、酪氨酸蛋白激酶及双特异性蛋白激酶。蛋白磷酸化在大多数细胞作用中都起到重要作用,如细胞分裂分化、细胞增殖、细胞凋亡等。Os04g0624600编码蛋白质可能调控淀粉合成与分解,起到可溶性淀粉合成酶的作用,水稻中与淀粉合成相关的基因主要分为两类,一类主要在胚乳中特异性表达,另外一类在其他组织和早期胚乳发育中表达[33],本研究认为Os04g0624600可能是第二类群淀粉合成基因,但同样具有进一步的研究价值。
水稻粒质量与粒长、粒宽、粒厚及籽粒饱满度呈显著正相关。通过灌浆指标和显微观察可以反映水稻籽粒质量和稻米品质。通过QTL-seq分析及分析注释基因的预测功能和表达位置可以显著提高候选基因的筛选效率。水稻粒质量可能受到蛋白激酶和转录因子相关基因控制。
[1] 张亚东,张颖慧,董少玲,陈涛,赵庆勇,朱镇,周丽瑟,姚姝,赵凌. 特大粒水稻材料粒型性状的QTL检测[J]. 中国水稻科学,2013, 27(2):122-128.doi:10.3969/j.issn.1001-7216.2013.02.003.
Zhang Y D,Zhang Y H,Dong S L,Chen T,Zhao Q Y, Zhu Z, Zhou L H, Yao S, Zhao L. Identification of QTL for rice grain traits based on an extra-large grain material [J].Chinese Journal of Rice Science,2013,27(2):122-128.
[2] 高志强,占小登,梁永书,程式华,曹立勇. 水稻粒形性状的遗传及相关基因定位与克隆研究进展[J]. 遗传,2011,33(4): 314-321.doi:10.3724/SP.J.1005.2011.00314.
Gao Z Q,Zhan X D,Liang Y S,Cheng S H,Cao L Y. Progress on genetics of rice grain shape trait and its related gene mapping and cloning [J]. Hereditas,2011,33(4):314-321.
[3] Li S, Tian Y H, Wu K, Ye Y F, Yu J P, Zhang J Q, Liu Q, Hu M Y, Li H, Tong Y P, Harberd N P, Fu X D. Modulating plant growth metabolism coordination for sustainable agriculture[J]. Nature,2018, 560(7720): 595-600. doi: 10.1038/s41586-018-0415-5.
[4] Sun P Y, Zhang W H, Wang Y H, He Q, Shu F, Liu H, Wang J, Wang J M, Yuan L P, Deng H F. OsGRF4 controls grain shape, panicle length and seed shattering in rice[J]. Journal of Integrative Plant Biology,2016, 58(10): 836-847. doi: 10.1111/jipb.12473.
[5] Chen X L, Jiang L R, Zheng J S, Chen F Y, Wang T S, Wang M L, Tao Y, Wang H C, Hong Z L, Huang Y M, Huang R Y. A missense mutation in Large Grain Size 1 increases grain size and enhances cold tolerance in rice [J]. Journal of Experimental Botany,2019,70(15):3851-3866. doi:10.1093/jxb/erz192.
[6] Wang S K, Li S, Liu Q, Wu K, Zhang J Q,Wang S S, Wang Y, Chen X B, Zhang Y, Gao C X, Wang F, Huang H X, Fu X D. The OsSPL16-GW7 regulatory module determines grain shape and simultaneously improves rice yield and grain quality[J]. Nature Genetics,2015, 47(8): 949-954. doi: 10.1038/ng.3352.
[7] Zhao D S, Li Q F, Zhang C Q, Zhang C, Yang Q Q, Pan L X, Ren X Y, Lu J, Gu M H, Liu Q Q. GS9 acts as a transcriptional activator to regulate rice grain shape and appearance quality[J]. Nature Communications,2018, 9(1):1240. doi: 10.1038/s41467-018-03616-y.
[8] Mao H L, Sun S Y, Yao J L, Wang C R, Yu S B, Xu C G, Li X H, Zhang Q F. Linking differential domain functions of the GS3 protein to natural variation of grain size in rice[J]. Proceedings of the National Academy of Sciences,2010, 107(45): 19579-19584. doi: 10.1073/pnas.1014419107.
[9] Song X J, Huang W, Shi M, Zhu M Z, Lin H X. A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase[J]. Nature Genetics,2007, 39(5): 623-630. doi: 10.1038/ng2014.
[10] Yan S, Zou G H, Li S J, Wang H, Liu H Q, Zhai G W, Guo P, Song H M, Yan C G, Tao Y Z. Seed size is determined by the combinations of the genes controlling different seed characteristics in rice[J]. Theoretical and Applied Genetics,2011,123(7): 1173-1181. doi: 10.1007/s00122-011-1657-x.
[11] Ishimaru K, Hirotsu N, Madoka Y, Murakami N, Hara N, Onodera H, Kashiwagi T, Ujiie K, Shimizu B, Onishi A, Miyagawa H, Katoh E. Loss of function of the IAA-glucose hydrolase gene TGW6 enhances rice grain weight and increases yield[J]. Nature Genetics,2013, 45(6): 707-711. doi: 10.1038/ng.2612.
[12] Hu Z J, Lu S J, Wang M J, He H H,Sun L,Wang H R, Liu X H, Jiang L,Sun J L, Xin X Y, Kong W, Chu C C, Xue H W, Yang J S, Luo X J, Liu J X. A novel QTL qTGW3 encodes the GSK3/SHAGGY-Like Kinase OsGSK5/OsSK41 that interacts with OsARF4 to negatively regulate grain size and weight in rice[J]. Molecular Plant,2018, 11(5): 736-749. doi: 10.1016/j.molp.2018.03.005.
[13] Ying J Z, Ma M, Bai C, Huang X H, Liu J L, Fan Y Y, Song X J. TGW3, a major QTL that negatively modulates grain length and weight in rice[J]. Molecular Plant,2018, 11(5): 750-753. doi: 10.1016/j.molp.2018.03.007.
[14] Xia D, Zhou H, Liu R J, Dan W H, Li P B, Wu B, Chen J X, Wang L Q, Gao G J, Zhang Q L, He Y Q. GL3.3, a novel QTL encoding a GSK3/SHAGGY-like kinase, epistatically interacts with GS3 to produce extra-long grains in rice[J]. Mol Plant,2018, 11(5): 754-756. doi: 10.1016/j.molp.2018.03.006.
[15] Liu J F, Chen J, Zheng X M, Wu F Q, Lin Q B, Heng Y Q, Tian P, Cheng Z J, Yu X W, Zhou K N, Zhang X, Guo X P, Wang J L, Wang H Y, Wan J M. GW5 acts in the brassinosteroid signalling pathway to regulate grain width and weight in rice[J]. Nature Plants,2017, 3: 17043. doi: 10.1038/nplants.2017.43.
[16] Park M, Lee J H, Han K, Jang S, Han J, Lim J H, Jung J W, Kang B C. A major QTL and candidate genes for capsaicinoid biosynthesis in the pericarp of Capsicum chinense revealed using QTL-seq and RNA-seq[J]. Theoretical and Applied Genetics,2019, 132(2): 515-529. doi: 10.1007/s00122-018-3238-8.
[17] Luo H Y, Pandey M K, Khan A W, Guo J B, Wu B, Cai Y, Huang L, Zhou X J, Chen Y N, Chen W G, Liu N, Lei Y, Liao B S, Varshney R K, Jiang H F.Discovery of genomic regions and candidate genes controlling shelling percentage using QTL-seq approach in cultivated peanut (Arachis hypogaea L.)[J]. Plant Biotechnology Journal,2019,17(7):1248-1260. doi:10.1111/pbi.13050.
[18] Qin Y B, Cheng P, Cheng Y C, Feng Y, Huang D R, Huang T X, Song X J, Ying J Z. QTL-Seq identified a major QTL for grain length and weight in rice using near isogenic F2 population[J]. Rice Science,2018,25(3):121-131. doi: 10.1016/j.rsci.2018.04.001.
[19] Luo X D, Liu J L, Zhao J, Dai L F, Chen Y L, Zhang L, Zhang F T, Hu B L, Xie J K. Rapid mapping of candidate genes for cold tolerance in Oryza rufipogon Griff. by QTL-seq of seedlings[J].Journal of Integrative Agriculture,2018,17(2):265-275.doi: 10.1016/S2095-3119(17)61712-X.
[20] 李国亮,张淑江,钱伟,李菲,章时蕃,张慧,方智远,孙日飞. 大白菜抗TuMV显性位点F2的QTL-seq分析[J]. 园艺学报,2019,46(2): 307-316. doi:10.16420/j.issn.0513-353x.2018-0517.
Li G L,Zhang S J,Qing W,Li F,Zhang S F,Zhang H,Fang Z Y,Sun R F. Genetic analysis of dominant locus involved in resistance to Turnip mosaic virus by QTL-seq in Chinese cabbage[J]. Acta Horticulture Sinica,2019, 46(2): 307-316.
[21] Hisano H, Sakamoto K, Takagi H, Terauchi R, Sato K. Exome QTL-seq maps monogenic locus and QTLs in barley[J]. BMC Genomics,2017, 18(1):125. doi:10.1186/s12864-017-3511-2.
[22] Branham S E, Patrick Wechter W, Lambel S, Massey L, Ma M, Fauve J, Farnham M W, Levi A. QTL-seq and marker development for resistance to Fusarium oxysporum f. sp. niveum race 1 in cultivated watermelon[J]. Molecular Breeding,2018, 38(11):139. doi: 10.1007/s11032-018-0896-9.
[23] 张尧锋,张冬青,余华胜,林宝刚,华水金,丁厚栋,傅鹰. 基于极端混合池(BSA)全基因组重测序的甘蓝型油菜有限花序基因定位[J]. 中国农业科学,2018,51(16): 3029-3039. doi:10.3864/j.issn.0578-1752.2018.16.001.
Zhang Y F,Zhang D Q,Yu H S,Lin B G,Hua S J,Ding H D,Fu Y. Location and mapping of the determinate growth habit of Brassica napus by bulked segregant analysis (BSA) using whole genome re-sequencing[J].Scientia Agricultura Sinica,2018,51(16):3029-3039.
[24] Das S, Upadhyaya H D, Bajaj D, Kujur A, Badoni S, Laxmi, Kumar V, Tripathi S, Gowda C L L, Sharma S, Singh S, Tyagi A K, Parida S K. Deploying QTL-seq for rapid delineation of a potential candidate gene underlying major trait-associated QTL in chickpea[J]. DNA Research, 2015, 22(3): 193-203.doi: 10.1093/dnares/dsv004.
[25] Shu J S, Liu Y M, Zhang L L, Li Z S, Fang Z Y, Yang L M, Zhuang M, Zhang Y Y, Lü H H. QTL-seq for rapid identification of candidate genes for flowering time in broccoli×cabbage[J]. Theoretical and Applied Genetics, 2018, 131(4): 917-928. doi: 10.1007/s00122-017-3047-5.
[26] 蔡建秀,陈伟. 水稻穗上发芽生理生化及颖壳扫描电镜观察[J]. 中国农学通报,2007, 23(8): 207-211.doi:10.3969/j.issn.1000-6850.2007.08.045.
Cai J X,Chen W. Study on the physiological biochemistry of pre-harvest sprouting and scanning electron microscopy of glume in rice[J]. Chinese Agricultural Science Bulletin,2007, 23(8): 207-211.
[27] 陆彦,张晓敏,祁琰,张昌泉,凌裕平,刘巧泉.不同透明度水稻籽粒横断面扫描电镜分析[J]. 中国水稻科学,2018,32(2): 189-199.doi:10.16819/j.1001-7216.2018.7107.
Lu Y,Zhang X M,Qi Y,Zhang C Q,Ling Y P,Liu Q Q. Scanning electron microscopic analysis of grain cross-section from rice with different transparency[J].Rice Science,2018,32(2):189-199.
[28] 朱庆森,曹显祖,骆亦其. 水稻籽粒灌浆的生长分析[J]. 作物学报, 1988(3): 182.
Zhu Q S,Cao X Z,Luo Y Q. Growth analysis of grain filling in rice[J]. The Crop Journal,1988(3):182.
[29] 董骥驰,杨靖,郭涛,陈立凯,陈志强,王慧. 基于高密度Bin图谱的水稻抽穗期QTL定位[J]. 作物学报,2018, 44(6): 938-946.doi:10.3724/SP.J.1006.2018.00938.
Dong J C,Yang J,Guo T,Chen L K,Chen Z Q,Wang H.QTL mapping for heading date in rice using high-density Bin map[J].Acta Agronomica Sinica,2018, 44(6): 938-946.
[30] Zhu Z Z, Li X Q, Wei Y, Guo S B, Sha A H. Identification of a novel QTL for panicle length from wild rice (Oryza minuta) by specific locus amplified fragment sequencing and high density genetic mapping[J]. Frontiers in Plant Science,2018,9:1492.doi:10.3389/fpls.2018.01492.
[31] Jiang D, Fang J J, Lou L M, Zhao J F, Yuan S J, Yin L, Sun W, Peng L X, Guo B T, Li X Y. Characterization of a null allelic mutant of the rice NAL1 gene reveals its role in regulating cell division[J]. PLoS One, 2015, 10(2): e118169. doi: 10.1371/journal.pone.0118169.
[32] Cai Q, Yuan Z, Chen M J, Yin C S, Luo Z J, Zhao X X, Liang W Q, Hu J P, Zhang D B. Jasmonic acid regulates spikelet development in rice[J]. Nature Communications,2014, 5(1):3476. doi: 10.1038/ncomms447.
[33] 潘晓雪,姜华武,闫洪波,李美茹,吴国江.水稻ADP-葡萄糖焦磷酸化酶小亚基Ⅰ基因的启动子分析[J]. 热带亚热带植物学报,2008,16(3): 189-194.
Pan X X,Jiang H W,Yan H P,Li M R,Wu G J. Promoter analysis of the gene encoding ADP-glucose pyrophosphorylase small subunit in rice[J]. Journal of Tropical and Subtropical Botany,2008,16(3): 189-194.