矮秆紧凑型玉米具有抗倒伏、耐密和适宜机械化生产等特点[1-4],但目前可供利用的玉米矮秆基因资源比较单一,遗传基础狭窄,主要集中在br2[5-7]。进一步发掘新的玉米矮秆资源、加强矮化基因的鉴定与利用,为矮秆、紧凑、耐密优良玉米杂交种的选育提供物质和技术储备,对玉米矮化育种具有十分重要的意义[8-13]。根据MaizeGDB数据库资料,控制玉米株高的有近250个QTLs,分布在10 条染色体上;已报道玉米矮秆单基因如br、br-2、bv和D8等60多个,大部分为隐性基因,但基因D8、D9、D(t)[14]、D*-10[15]、D11[16]等为显性单基因,基因br-2[17]、an1[18]、D8[19]、D9[20]、d3[21]和D(t) [22]等已成功克隆。本研究以矮秆突变体K125d为主要研究材料,从主要性状表现、矮秆性状的遗传模式、控制该矮秆性状的基因定位与克隆等方面,进行系统的遗传评价,以期明确该突变体在玉米矮化育种方向的应用价值,为其应用研究提供参考依据。
1 材料和方法
1.1 供试材料
矮秆突变体K125d(P1),7个测验种(P2),以及构建的正反交F1、B1、B2和F2共6个世代群体。7个测验种名称及主要特征为,K211(K125d同源高秆)、K123d和626(矮秆)、K365和K363(中秆)、K305和K236(高秆)。以上材料均由四川农大正红生物技术有限责任公司(以下简称公司)提供。
1.2 试验设计
1.2.1 K125d与K211形态差异比较 在公司四川双流育种基地种植K125d和K211,选取有代表性的30个单株考察株高等主要农艺性状;成熟时,在各材料小区中间收获有代表性的30个果穗晒干,于室内考查主要经济性状;对植株进行拍照对比。
1.2.2 K125d矮秆性状遗传模式分析 试验在四川双流和雅安2个生态点进行。其中,每个点种植P1和P2群体各36株,正反交F1群体各84株;将籽粒最多的B1和B2果穗平均分成2份,在2个点全播;F2群体每试验点均播种336粒。对分离群体进行株高鉴定,用χ2检验株高分离比例适合性,确定其遗传模式。
1.2.3 K125d矮化基因定位 定位群体为(K236×K125d)F2,在公司四川双流育种基地播种1 400粒,为扩大定位群体规模,将上述用于遗传模式分析的群体也用于定位使用。单株编号挂牌,分别判定高、矮株类型并登记。采用集团分离分析法(Bulked segregation analysis,BSA) [23]进行基因定位,在定位群体中各选取10株极高和极矮单株提取DNA并等量混合,构建高秆和矮秆基因池。选取由上海生工生物工程股份有限公司合成的458对SSR引物(查自Maize Genetics and Genomics Database,http://www.maizegdb.org),用于亲本K236和K125d间的多态性引物筛选;然后在高秆和矮秆基因池间,进一步筛选多态性标记,筛选出可能与矮秆连锁的分子标记;再用定位群体中的典型高秆和典型矮秆植株各10株进行验证;最后用定位群体中的255个矮秆单株进行扩增。用2×CTAB法提取DNA并纯化,PCR反应体系用25 μL,PCR扩增产物用3% 琼脂糖凝胶进行电泳,用凝胶成像系统成像后观察统计带型,用MAPMAKER 3.0软件进行连锁分析,用MAPDraw V2.1[24]绘制遗传连锁图谱。
2 结果与分析
2.1 K125d与K211主要性状比较
K125d与K211的植株对比见图1,可见K125d植株叶片密集重叠,节间短、株高和穗位高较矮。考种结果表明,它们的果穗均为筒型、白轴、籽粒半马齿型,K211籽粒黄色,K125d籽粒白色。将它们主要农艺和经济性状比较结果列于表1,可见,与K211相比,K125d生育期极显著变长;株高、穗位高和叶夹角分别降低53.07%,71.46%和21.81%,均达极显著水平;但叶片数和叶宽均极显著增加,其增加比例分别为22.75%和15.15%,其余农艺性状差异不显著;就主要经济性状来看,K125d虽然穗长、穗粗和穗行数差异不显著,但行粒数和百粒质量却显著或极显著降低,造成单株产量极显著降低34.41%。

A.植株;B.茎秆;h.K211;d.K125d;标尺为10 cm。
A.Plant;B.Stem;h.K211;d.K125d;Bar=10 cm.
图1 K211与K125d 植株比较
Fig.1 Comparisons of the plants between K211 and K125d
表1 K125d与K211形态差异比较
Tab.1 Comparisons of the morphological difference between the K125d and K211
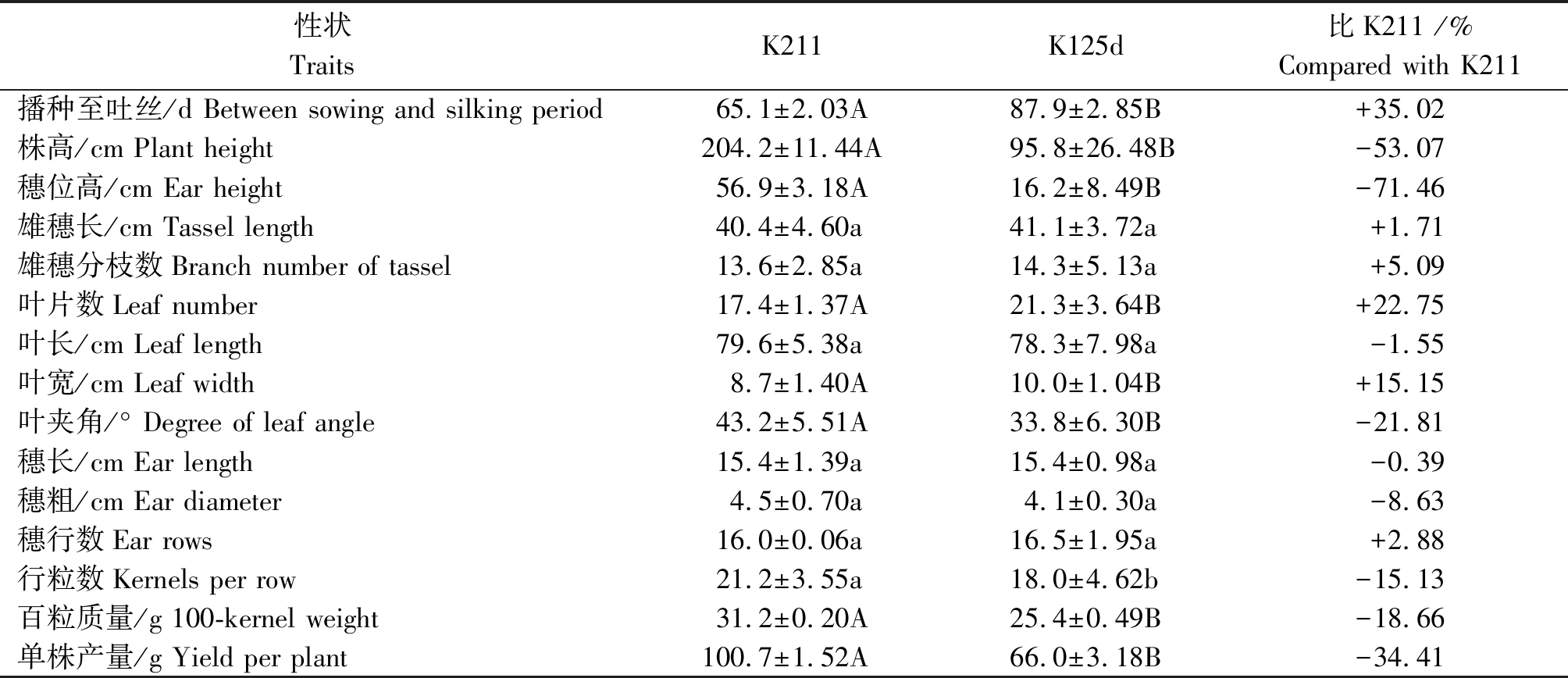
性状TraitsK211K125d比K211 /%Compared with K211播种至吐丝/d Between sowing and silking period65.1±2.03A87.9±2.85B+35.02株高/cm Plant height204.2±11.44A95.8±26.48B-53.07穗位高/cm Ear height56.9±3.18A16.2±8.49B-71.46雄穗长/cm Tassel length40.4±4.60a41.1±3.72a+1.71雄穗分枝数Branch number of tassel13.6±2.85a14.3±5.13a+5.09叶片数Leaf number17.4±1.37A21.3±3.64B+22.75叶长/cm Leaf length79.6±5.38a78.3±7.98a-1.55叶宽/cm Leaf width8.7±1.40A10.0±1.04B+15.15叶夹角/° Degree of leaf angle43.2±5.51A33.8±6.30B-21.81穗长/cm Ear length15.4±1.39a15.4±0.98a-0.39穗粗/cm Ear diameter4.5±0.70a4.1±0.30a-8.63穗行数Ear rows16.0±0.06a16.5±1.95a+2.88行粒数Kernels per row21.2±3.55a18.0±4.62b-15.13百粒质量/g 100-kernel weight31.2±0.20A25.4±0.49B-18.66单株产量/g Yield per plant100.7±1.52A66.0±3.18B-34.41
注:±.标准误; 2 组处理数据后的英文字母不同,则表示这2 组处理间的数据差异显著,小写字母代表P=0.05水平差异显著;大写字母代表P=0.01水平差异显著。
Note:±.SE; If the English letters of the two groups were different,the difference between the two groups was significant difference, lowercase letters means significantly different at P=0.05;Capital letters means significantly different at P=0.01.
2.2 突变体K125d株高的遗传分析
2.2.1 F1正反交群体株高表现 将K125d配制的7个F1正反交群体株高表现列于表2。可见,所有正反交F1群体在2个试验点均表现为高秆,且正、反交群体平均株高无显著差异,说明K125d株高遗传表现无细胞质效应,即该矮秆性状可能由核基因调控。另外,除用测验种K211配制的组合K125d×K211外,测验种株高越高,其相应的F1群体株高也有增高趋势,即用高秆测验种配制的组合其平均株高高于中秆、中秆高于矮秆;雅安点F1株高比双流点有增高的趋势,表明F1株高由双亲遗传背景共同控制,并受生态环境影响。
表2 正反交F1群体的株高表现
Tab.2 Plant height of orthogonal and reciprocal cross F1 groups cm
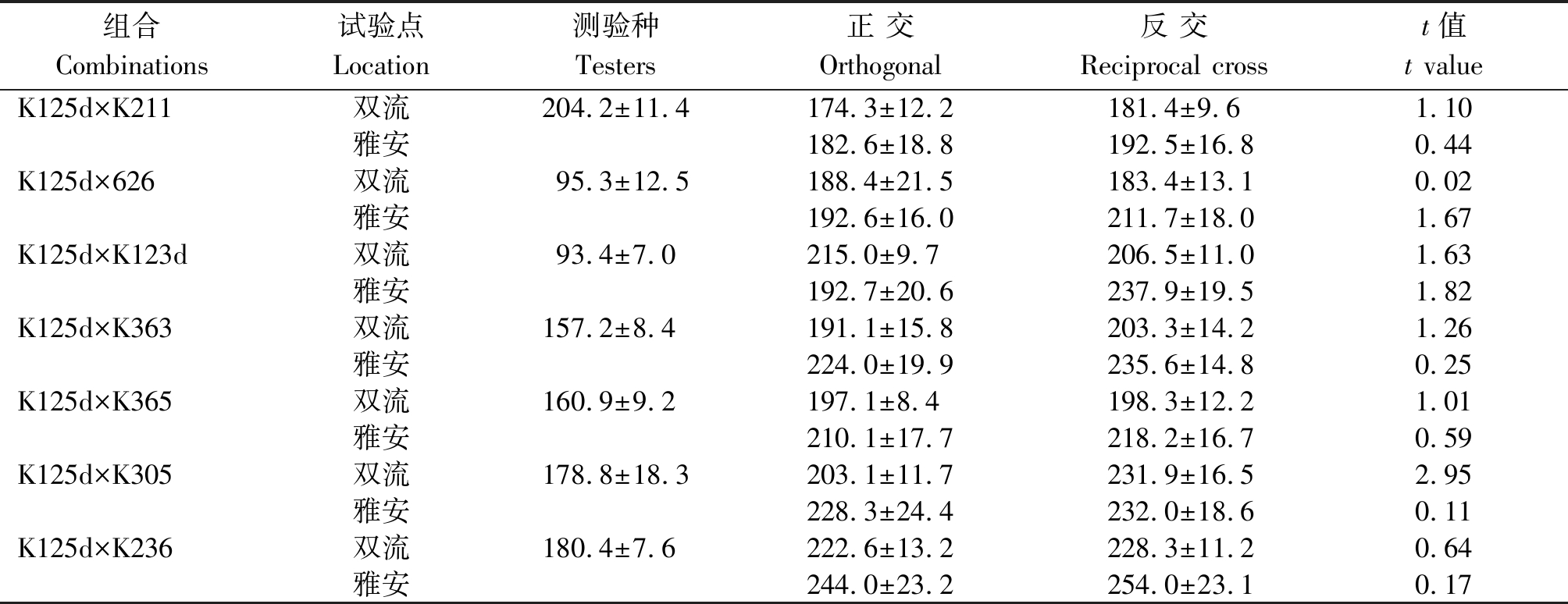
组合Combinations试验点Location测验种Testers正 交Orthogonal反 交Reciprocal crosst值t valueK125d×K211双流204.2±11.4174.3±12.2181.4±9.61.10雅安182.6±18.8192.5±16.80.44K125d×626双流95.3±12.5188.4±21.5183.4±13.10.02雅安192.6±16.0211.7±18.01.67K125d×K123d双流93.4±7.0215.0±9.7206.5±11.01.63雅安192.7±20.6237.9±19.51.82K125d×K363双流157.2±8.4191.1±15.8203.3±14.21.26雅安224.0±19.9235.6±14.80.25K125d×K365双流160.9±9.2197.1±8.4198.3±12.21.01雅安210.1±17.7218.2±16.70.59K125d×K305双流178.8±18.3203.1±11.7231.9±16.52.95雅安228.3±24.4232.0±18.60.11K125d×K236双流180.4±7.6222.6±13.2228.3±11.20.64雅安244.0±23.2254.0±23.10.17
2.2.2 F2和回交群体的株高频次分析 将F2和回交群体的植株高度作分布频次分析,结果为用K125d回交的所有B1和F2群体的植株高度均表现为双峰分布;B2群体(用K123d回交的B2除外)均表现高秆,表明K125d矮秆性状由多基因控制的可能性不大。
2.2.3 回交群体的株高分离比例分析 将用K125d构建的7个B1回交群体和用K123d构建的B2回交群体的株高分离比例结果列于表3。可见,在四川双流和雅安,7个B1回交群体均出现高矮分离,分离比例符合1∶1;用626、K211、K236、K305、K363和K365等测验种构建的6个B2回交群体在2个生态点均表现为高秆,表明可能由1对隐性核基因控制K125d的矮秆性状。至于用K123d构建的B2回交群体出现高矮分离,分离比例符合1∶1,其原因可能是控制K125d和K123d矮秆性状的基因不等位。
表3 回交群体的株高表现χ2检验
Tab.3 The χ2 test of plant height of backcross groups
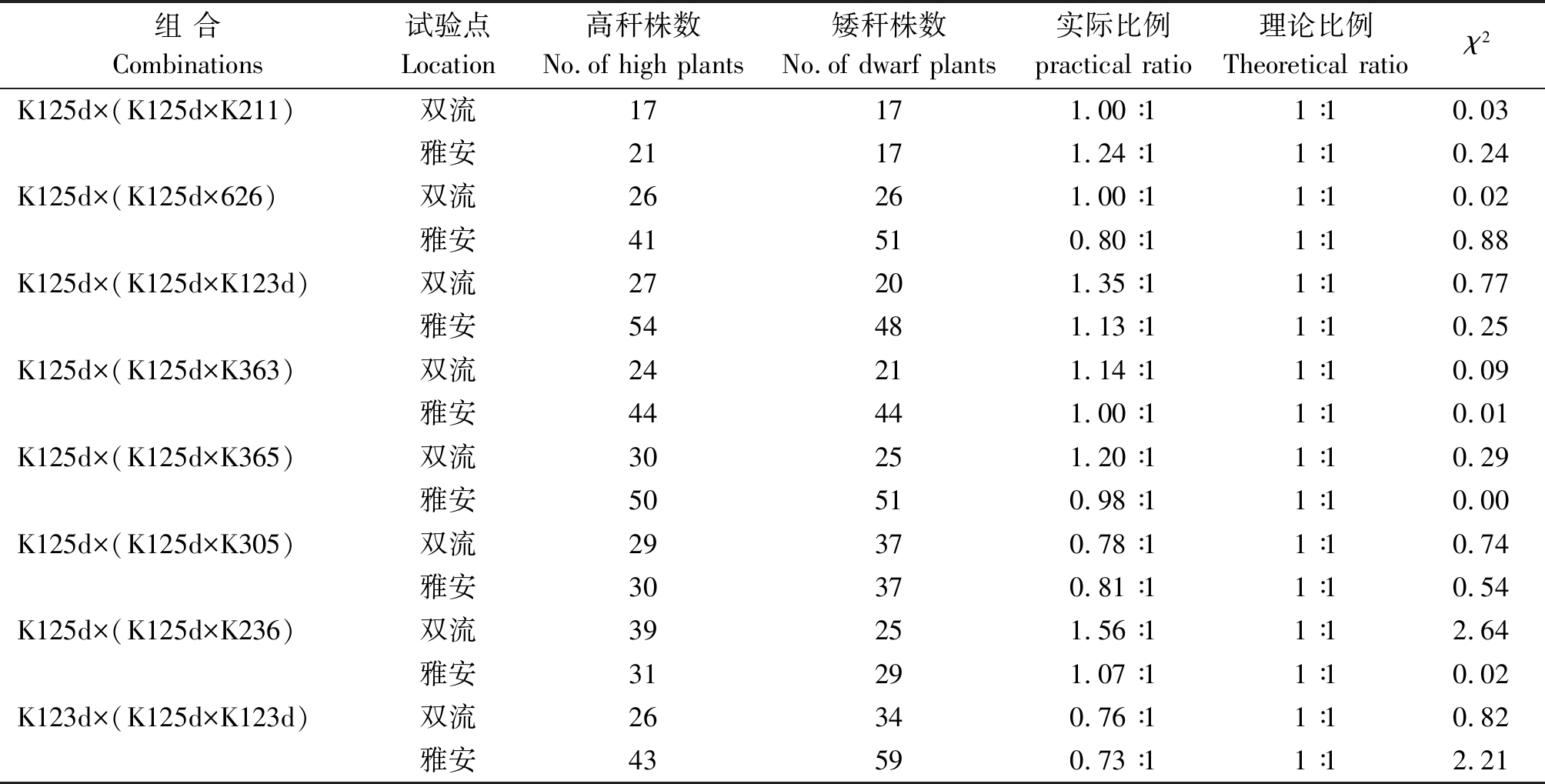
组 合Combinations试验点Location高秆株数No.of high plants矮秆株数No.of dwarf plants实际比例practical ratio理论比例Theoretical ratioχ2K125d×(K125d×K211)双流17171.00∶11∶10.03雅安21171.24∶11∶10.24K125d×(K125d×626)双流26261.00∶11∶10.02雅安41510.80∶11∶10.88K125d×(K125d×K123d)双流27201.35∶11∶10.77雅安54481.13∶11∶10.25K125d×(K125d×K363)双流24211.14∶11∶10.09雅安44441.00∶11∶10.01K125d×(K125d×K365)双流30251.20∶11∶10.29雅安50510.98∶11∶10.00K125d×(K125d×K305)双流29370.78∶11∶10.74雅安30370.81∶11∶10.54K125d×(K125d×K236)双流39251.56∶11∶12.64雅安31291.07∶11∶10.02K123d×(K125d×K123d)双流26340.76∶11∶10.82雅安43590.73∶11∶12.21
注:临界t值, χ20.05(1)=3.84, χ20.01(1)=6.63。表4同。
Note: The critical t value, χ20.05(1)=3.84, χ20.01(1)=6.63.The same as Tab.4.
2.2.4 F2群体的株高分离比例分析 将用K125d构建的7个F2群体的株高分离结果列于表4。可见,所有F2群体在雅安和双流2个生态点株高分离规律表现一致,除(K125d×K123d)F2群体外,其余6个F2群体植株高矮分离符合3∶1,进一步表明由1对隐性核基因控制K125d的矮秆性状。而(K125d×K123d)F2群体植株高矮分离比例为9∶7,符合2对隐性单基因控制同一性状分离比例,表明控制K125d和K123d矮秆性状的隐性单基因不等位。
综合分析表明,K125d与7个不同株高自交系配制的F1正反交群体,在2个试验点均表现为高秆,其对应正、反交群体间的株高差异不明显,证明控制K125d矮秆性状的基因无胞质效应;除K123d外(br2类型)[11],与其余6个不同株高自交系配制的B1回交群体,高、矮秆分离比例为1∶1,B2回交群体全为高秆,F2群体高、矮秆分离比例为3∶1,充分证明K125d的矮秆性状受1对隐性核基因控制,暂命名为d125。用K125d和K123d构建的回交群体B1、B2及自交F2群体,其群体植株高矮分离,比例分别为1∶1、1∶1和9∶7,符合由2对互补隐性基因控制株高遗传表现的分离比例规律,表明基因d125与br2可能不等位。
表4 F2群体株高表现的χ2检验
Tab.4 The χ2 test of plant height of F2 groups
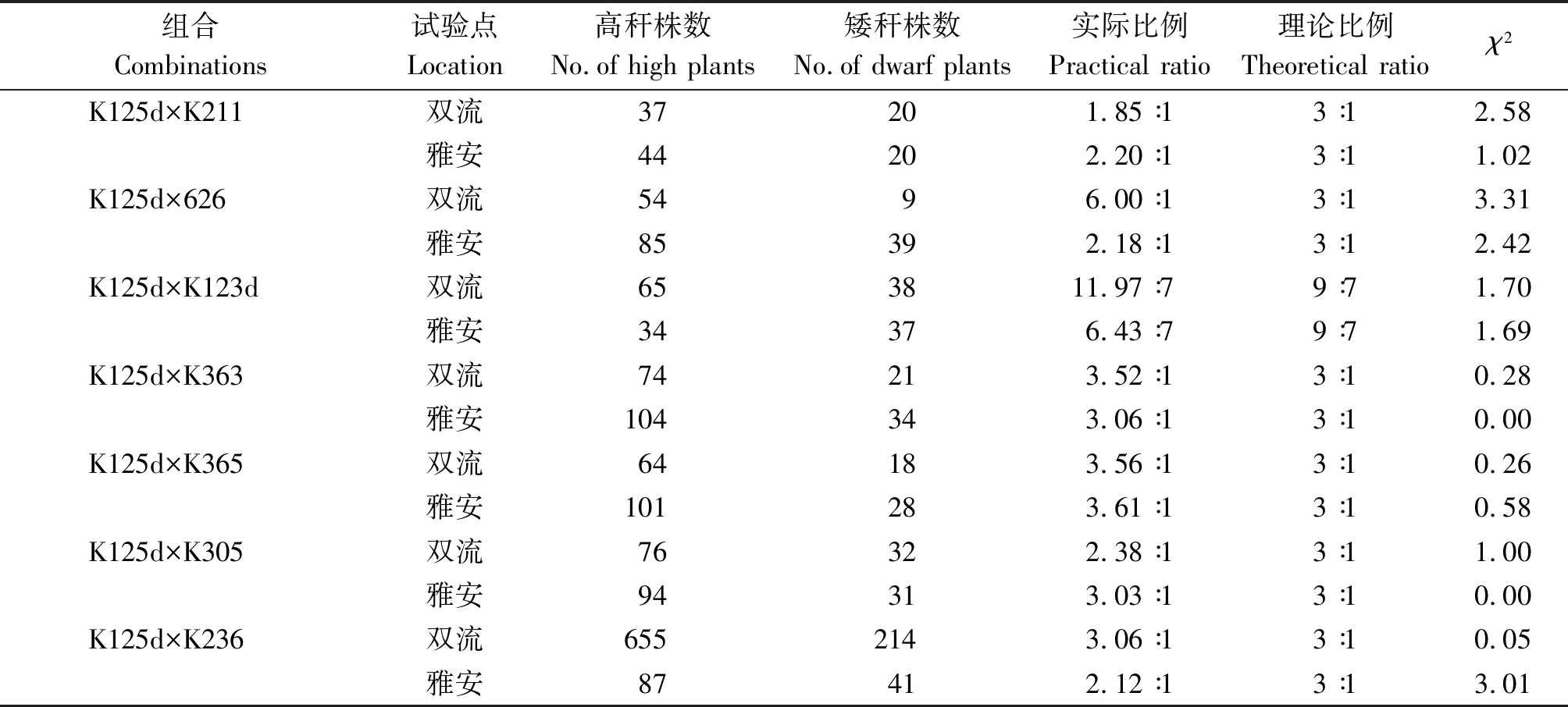
组合Combinations试验点Location高秆株数No.of high plants矮秆株数No.of dwarf plants实际比例Practical ratio理论比例Theoretical ratioχ2K125d×K211双流37201.85∶13∶12.58雅安44202.20∶13∶11.02K125d×626双流5496.00∶13∶13.31雅安85392.18∶13∶12.42K125d×K123d双流653811.97∶79∶71.70雅安34376.43∶79∶71.69K125d×K363双流74213.52∶13∶10.28雅安104343.06∶13∶10.00K125d×K365双流64183.56∶13∶10.26雅安101283.61∶13∶10.58K125d×K305双流76322.38∶13∶11.00雅安94313.03∶13∶10.00K125d×K236双流6552143.06∶13∶10.05雅安87412.12∶13∶13.01
2.3 矮秆基因d125的定位
基因定位群体为(K125d×K236)F2,选取均匀覆盖10条玉米染色体上的SSR引物458对,筛选出在双亲间多态性较好的引物109对;进一步在高、矮秆基因池间对这109对引物进行多态性筛选,其中多态性表现较好的引物为umc2235。以引物umc2235分布的位置为参考,选择并合成位于玉米第一条染色体1.05~1.09位置区间的SSR引物153对继续进行多态性筛选,结果发现bnlg1619、umc1085、umc1278、umc1601、umc1689、umc1035、umc2560、umc2569和umc2387等9对引物在高、矮秆基因池间均表现出多态性,至此,共筛选出在高、矮基因池间具有多态性的SSR引物10对。
用上述10对SSR引物,对255个矮秆单株DNA,分别进行PCR扩增,用琼脂糖凝胶电泳检测扩增产物,用凝胶成像系统对电泳结果照相,统计10对引物对定位群体中所有矮秆单株DNA扩增带型。扩增结果连锁分析用作图软件MAPMAKER 3.0,将重组率转化为遗传距离用Kosambi函数转化,引物与矮秆基因d125的连锁关系用“Group”命令(LOD值大于3.0,重组率小于50%)判断,各标记间的图距用MAP命令计算,用MAPDraw V2.1 构建遗传连锁图谱。结果为,矮秆基因d125,初步定位于1号染色体上,介于umc2569和umc1278 2个SSR分子标记之间,其遗传距离分别为6.6,5.1 cM(图2)。
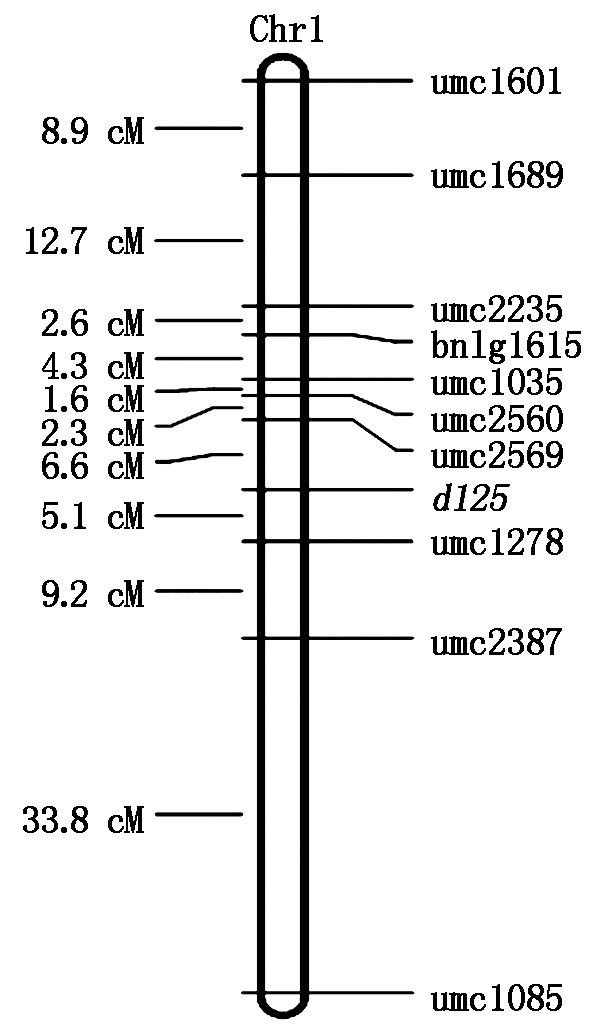
图2 矮秆基因d125的遗传连锁图谱
Fig.2 Genetic linkage map of dwarf mutant d125
2.4 矮秆基因d125的克隆
根据定位结果,位于标记umc2569和umc1278附近的已知矮秆基因有5个,br2(bin 1.06)、br1(bin 1.07)、d*-N1352B(bin 1.06)、smp*-N706A(bin 1.06)和d*-N454A(bin 1.06),在MaizeGDB中搜索相关信息发现仅有br2关联有基因模型,因此以br2作为候选基因进行克隆。
根据br2基因模型GRMZM2G315375设计了8对重叠引物(图3-A),用于扩增br2的5个外显子区域。克隆结果如图3-B所示,可见引物d125-8从K125d中扩增产物比K211约小250 bp(图3-B中的箭头所示);对扩增产物的测序结果如图3-C所示,可见d125在第1 651个碱基处有一个9 bp片段的插入,在第6 438碱基处有一个232 bp片段的缺失。通过DNAMAN对编码序列进行翻译显示,d125插入的9个碱基编码谷氨酸(E)、天冬氨酸(D)和甘氨酸(G),该位置不在功能结构域内(图3-D中三角形所示);而缺失导致从第1 288个氨基酸后产生移码突变(如图3-D的箭头所示),该突变区域在SMART(http://smart.embl-heidelberg.de)的注释为与各种细胞活动有关的ATP酶,而在P-糖蛋白(P-glycoprotein,PGP)中为核酸结合域(Nucleotide binding domain,NBD),该功能域功能主要是为PGP转运提供能量。
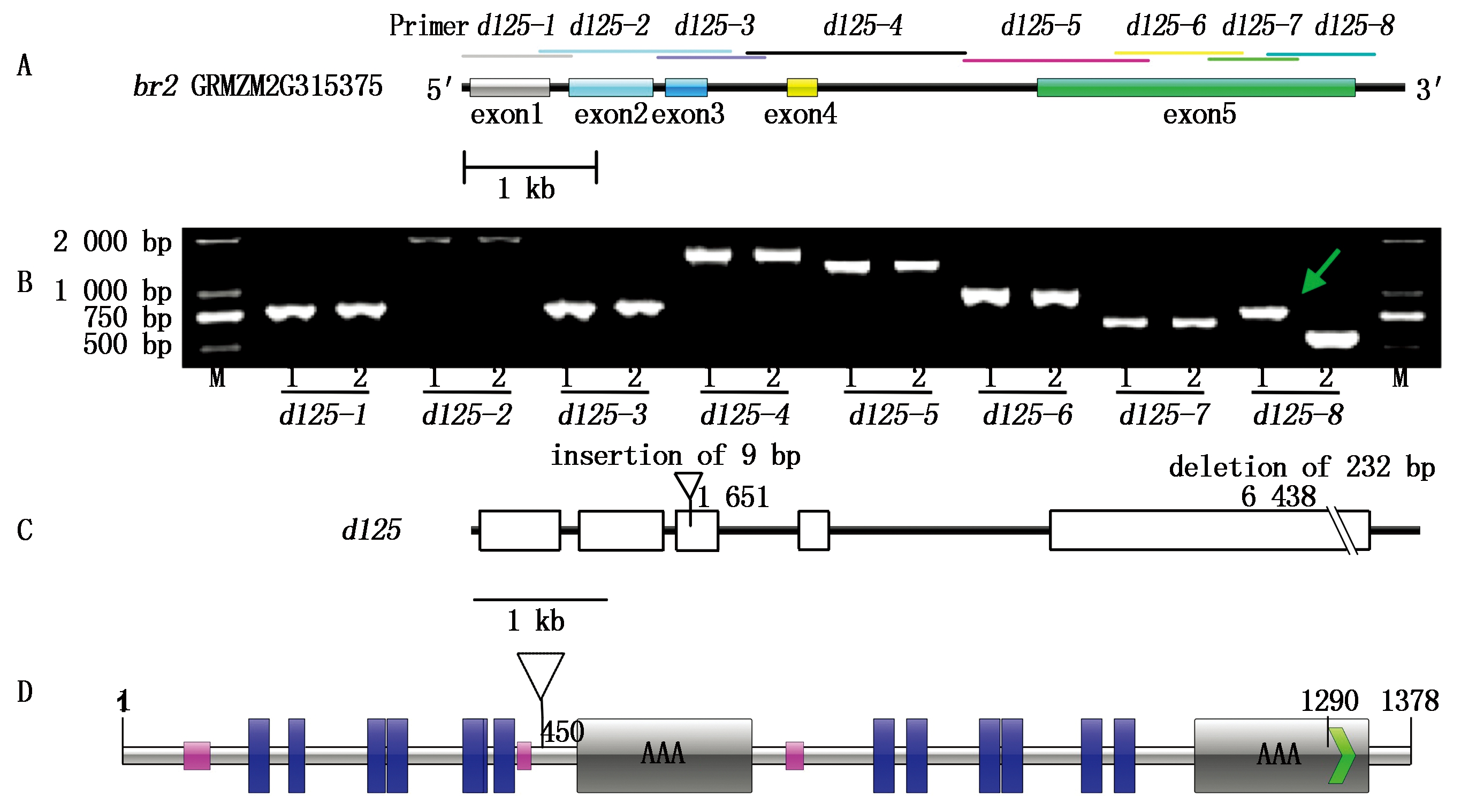
图3 矮秆基因d125克隆结果
Fig.3 Cloning results of dwarf mutant d125
3 讨论与结论
3.1 矮秆突变体K125d株高的遗传模式
玉米矮秆基因的遗传一般分为单基因和多基因2种遗传模式[25-26],针对一个新的矮秆资源,常用与其他自交系构建的F1、B1、B2和F2群体的株高分离表现情况,来分析判断其遗传模式,如张素梅等[14]于2007年就做过类似研究。本研究用矮秆突变体K125d与同源自交系K211以及2个矮秆、中秆和高秆自交系,构建的正反交F1,回交B1、B2和自交F2群体分析其遗传模式,结果为K125d矮秆性状受1对隐性核基因控制,命名为d125。
3.2 矮秆基因d125初步定位结果
MaizeGDB已收录矮秆基因60多个,定位在玉米1号染色体上的有11个。基因br2是发现较早且研究较多的玉米矮秆基因,该基因与生长素的合成相关,已被成功克隆[17]。笔者将矮秆基因d125初步定位在1号染色体umc2569和umc1278 SSR分子标记之间,其距离分别为6.6,5.1 cM,在染色体上的位置与矮秆基因br2(bin 1.06)和br1(bin 1.07)等相近。
3.3 矮秆基因d125序列及功能预测
br2是目前应用较多的玉米矮秆基因,编码1个PGP蛋白,由两部分组成,各部分均包含了6个假定的跨膜结构域和一个细胞内的ATP结合域,并具有各自的功能[27]。已克隆得到的br2等位基因中,br2-6、br2-7和br2-9均在外显子1有插入,br2-3在内含子4中有插入[17],br2-23在第6 667个碱基处有8 bp缺失[28],株高QTL-qph1中SNP5259(T)编码的氨基酸带点性质改变可能使蛋白与底物结合能力下降[29],高粱中与br2同源的dw3,其在外显子5上有一个882 bp单元的直接重复,使得该基因功能缺失[17],致使编码蛋白功能散失。d125在第6 438碱基处缺失232 bp,包含了br2-23中的缺失部分,缺失产生移码突变,这部分位于PGP蛋白和ABC转运蛋白中的NBD内。NBD的功能主要是为PGP转运提供充足的能量,而保守的结构域保证ATP结合与水解[30],2个Walker结构中任一个突变导致功能完全丧失[31],而D125蛋白中也具有这个保守结构。此外,NBD含有约215个氨基酸构成的核心区,核心区内的部分氨基酸是保守的,对整个蛋白质的功能具有极其重要的作用[32]。Walker B附近的小片段可能直接或间接地参与底物相互作用和NBD到跨膜域间的信号转导,是PGP蛋白中底物结合的主要位点[17]。d125在Walker B后移码,造成的功能位点缺失可能是导致转运功能丧失的主要原因。
[1] 曹庆军, 杨粉团, 姜晓莉, 陈莫军, 李贺, 于洪浩, 鲁建华, 张兆琴, 薄晓杰, 李刚. 玉米抗茎倒能力评价及理想株型[J]. 东北农业科学, 2017, 42(2): 17-21. doi:10.16423/j.cnki.1003-8701.2017.02.005.
Cao Q J, Yang F T, Jiang X L, Chen M J, Li H, Yu H H, Lu J H, Zhang Y Q, Bo X J, Li G. Evaluation of stem lodging resistance and ideal plant type of lodging resistant maize varieties[J]. Journal of Northeast Agricultural Sciences, 2017, 42(2): 17-21.
[2] 何川. 抗倒伏玉米矮生系的选育与利用[J]. 杂粮作物, 2010, 30(6): 387-388. doi:10.3969/j.issn.2095-0896.2010.06.004.
He C. Breeding and utilizing of dwarf maize lodging[J]. Rain Fed Crops, 2010, 30(6): 387-388.
[3] 周颖, 顾万荣, 赵猛, 佟桐, 刘笑鸣, 李彩凤, 李晶, 魏湜. 黑龙江省不同熟期春玉米品种茎秆特性及机收指标差异[J]. 华北农学报, 2017, 32(S1): 140-146. doi:10.7668/hbnxb.2017.S1.025.
Zhou Y, Gu W R, Zhao M, Tong T, Liu X M, Li C F, Li J, Wei T. Differences of stalk characteristics and grain mechanically harvesting qualities of different maturing-type spring maize in Heilongjiang Province[J]. Acta Agriculturae Boreali-Sinica, 2017, 32(S1): 140-146.
[4] 彭长俊, 崔士友. 玉米育种技术体系的构建及有关问题的讨论[J]. 农学学报, 2018, 8(4): 1-7.
Peng C J, Cui S Y. The construction of technology system of maize breeding and the discussion on relevant problems[J]. Journal of Agriculture, 2018, 8(4): 1-7.
[5] 崔绍平, 孙世珍, 徐有, 郑积德, 李洪杰. 玉米br-2矮生基因利用的研究[J]. 华北农学报, 1990, 5(2): 7-12. doi:10.3321/j.issn:1000-7091.1990.02.002.
Cui S P, Sun S Z, Xu Y, Zheng J D, Li H J. A study on util ization of a dwarf gene br-2 in maize breeding[J]. Acta Agriculturae Boreali-Sinica, 1990, 5(2): 7-12.
[6] 阎淑琴. 矮生玉米的遗传与育种[J]. 玉米科学, 2000, 8(2): 36-37, 45. doi:10.3969/j.issn.1005-0906.2000.02.009.
Yan S Q. Inheritance and breeding of dwarf type maize[J]. Journal of Maize Sciences, 2000, 8(2): 36-37, 45.
[7] 何川, 郑祖平, 谢树果, 李钟, 刘代惠. 隐性单基因br-2玉米矮生系的选育[J]. 中国农业科学, 2009, 42(8): 2978-2981. doi:10.3864/j.issn.0578-1752.2009.08.042.
He C, Zheng Z P, Xie S G, Li Z, Liu D H. Breeding of the maize monogenic br-2 dwarf lines[J]. Scientia Agricultura Sinica, 2009, 42(8): 2978-2981.
[8] 樊景胜. 玉米矮生基因遗传及其利用[J]. 黑龙江农业科学, 1999(1): 29-30.doi:10.11942/j.issn1002-2767.1999.01.0029.
Fan J S. Inheritance and utilization of dwarf gene in maiz[J].Heilongjiang Agricultural Sciences, 1999(1): 29-30.
[9] 田齐建, 乔治军, 董存吉, 穆志新. 玉米矮化育种研究进展及发展前景[J]. 山西农业科学, 2003, 31(2): 23-26. doi:10.3969/j.issn.1002-2481.2003.02.006.
Tian Q J, Qiao Z J, Dong C J, Mu Z X. Review on research of maize breeding for dwarfness and its development prospect[J]. Journal of Shanxi Agricultural Sciences, 2003, 31(2): 23-26.
[10] 邱正高, 杨华, 袁亮, 张亚勤, 张采波, 汤玲, 荣廷昭, 曹墨菊. 一份新选玉米矮秆突变体的鉴定与遗传分析[J]. 华北农学报, 2015, 30(6): 112-118. doi:10.7668/hbnxb.2015.06.017.
Qiu Z G, Yang H, Yuan L, Zhang Y Q, Zhang C B, Tang L, Rong T Z, Cao M J. Identification and genetic analysis of a new dwarf mutant in maize[J]. Acta Agriculturae Boreali-Sinica, 2015, 30(6): 112-118.
[11] 徐敏, 石海春, 余学杰, 谭义川, 柯永川, 赵长云, 柯永培. 一个玉米矮秆突变体K123d的遗传鉴定[J]. 植物遗传资源学报, 2017, 18(1): 155-163. doi:10.13430/j.cnki.jpgr.2017.01.020.
Xu M, Shi H C, Yu X J, Tan Y C, Ke Y C, Zhao C Y, Ke Y P. Genetic identification of a dwarf mutant K123d in maize(Zea mays L.)[J]. Journal of Plant Genetic Resources, 2017, 18(1): 155-163.
[12] 牛群凯, 杨聪, 时子文, 曹墨菊. 玉米太空诱变核不育突变体矮化性状的QTL定位及分析[J]. 四川农业大学学报, 2018, 36(4): 429-435, 480. doi:10.16036/j.issn.1000-2650.2018.04.002.
Niu Q K, Yang C, Shi Z W, Cao M J. QTL mapping of dwarf-associated traits in the maize male sterile mutant obtained by space flight[J]. Journal of Sichuan Agricultural University, 2018, 36(4): 429-435, 480.
[13] 刘忠祥, 杨梅, 殷鹏程, 周玉乾, 何海军, 邱法展. 玉米株高主效QTL qPH3.2精细定位及遗传效应分析[J]. 作物学报, 2018, 44(9): 1357-1366.doi:10.3724/SP.J.1006.2018.01357.
Liu Z X, Yang M, Yin P C, Zhou Y Q, He H J, Qiu F Z. Fine mapping and genetic effect analysis of a major QTL qPH3.2 associated with plant height in maize(Zea mays L.)[J]. Acta Agronomica Sinica, 2018, 44(9): 1357-1366.
[14] 张素梅, 刘凤军, 刘保申, 王立静, 董树亭. 新的玉米显性矮秆基因的发现及初步分析[J]. 玉米科学, 2007, 15(3): 15-18. doi:10.3969/j.issn.1005-0906.2007.03.004.
Zhang S M, Liu F J, Liu B S, Wang L J, Dong S T. Discovery of a new dominant dwarf gene in maize and its preliminary study[J]. Journal of Maize Sciences, 2007, 15(3): 15-18.
[15] 王益军, 苗楠, 施亚婷, 邓德祥, 卞云龙. 一份玉米显性矮秆突变体的遗传分析[J]. 华北农学报, 2010, 25(5): 90-93. doi:10.7668/hbnxb.2010.05.019.
Wang Y J, Miao N, Shi Y T, Deng D X, Bian Y L. Genetic analysis of a dominant dwarf mutant in maize[J]. Acta Agriculturae Boreali-Sinica, 2010, 25(5): 90-93.
[16] Wang Y J, Deng D X, Ding H D, Xu X M, Zhang R, Wang S X, Bian Y L, Yin Z T, Chen Y. Gibberellin biosynthetic deficiency is responsible for maize dominant dwarf11(D11) mutant phenotype: physiological and transcriptomic evidence[J]. PLoS One, 2013, 8(6): e66466. doi:10.1371/journal.pone.0066466.
[17] Multani D S, Briggs S P, Chamberlin M A, Blakeslee J J, Murphy A S, Johal G S. Loss of an MDR transporter in compact stalks of maize br2 and sorghum dw3 mutants[J]. Science, 2003, 302(5642): 81-84. doi:10.1126/science.1086072.
[18] Bensen R J, Johal G S, Grane V C, Tossberg J T, Schnable P S, Meeley R B, Briggs S P. Cloning and characterization of the maize An1 gene[J]. The Plant Cell,1995, 7(1): 75-84. doi:10.1105/tpc.7.1.75.
[19] Peng J R, Rivhards D E, Hartley N M, Murphy G P, Devos K M, Flintham J E, Beales J, Fish L J, Worland A J, Pelica F, Sudhakar D, Christou P, Snape J W, Gale M D, Harberd N P. Green revolution genes encode mutant gibberellin response modulators[J]. Nature, 1999, 400(6741): 256-261. doi:10.1038/22307.
[20] Lawit S J, Wych H M, Xu D P, Kundu S, Tomes D T. Maize DELLA proteins dwarf plant8 and dwarf plant9 as modulators of plant development[J]. Plant & Cell Physiology, 2010, 51(11): 1854-1868.doi:10.1093/pcp/pcq153.
[21] Winkler R G, Helentjaris T. The maize Dwarf3 gene encodes a cytochrome P450-mediated early step in gibberellin biosynthesis[J]. The Plant Cell, 1995, 7(8): 1307-1317.doi:10.1105/tpc.7.8.1307.
[22] 王立静. 玉米矮秆基因Dt和坏死基因net-t的图位克隆与功能分析[D]. 泰安: 山东农业大学, 2012. doi:10.7666/d.d224451.
Wang L J. Map-based cloning and functional analysis of dwarf gene Dt and necrotic gene net-t in maize[D].Taian:Shandong Agricultural University, 2012.
[23] Michelmore R W, Paran I, Kesseli R V. Identification of markers linked to disease-resistance genes by bulked segregant analysis:a rapid method to detect markers in specific genomic regions by using segregating populations[J]. Proceedings of the National Academy of Sciences, 1991, 88(21): 9828-9832. doi:10.2307/2357899.
[24] 刘仁虎, 孟金陵. MapDraw, 在Excel中绘制遗传连锁图的宏[J]. 遗传, 2003, 25(3): 317-321. doi:10.3321/j.issn:0253-9772.2003.03.019.
Liu R H, Meng J L. MapDraw:a microsoft excel macro for drawing genetic linkage maps based on given genetic linkage data[J]. Hereditas, 2003, 25(3): 317-321.
[25] 李钟, 郑祖平, 张国清, 何川. 矮生玉米自交系的选育和利用[J]. 玉米科学, 2006, 14(1): 76-78. doi:10.3969/j.issn.1005-0906.2006.01.023.
Li Z, Zheng Z P, Zhang G Q, He C. Breeding and application on the inbred line of dwarf maize[J]. Journal of Maize Sciences, 2006, 14(1): 76-78.
[26] 李忠南, 王克伟, 王越人, 邬生辉, 李光发. 玉米品种先玉335的血缘系谱及主要农艺性状遗传分析[J]. 玉米科学, 2018, 26(3): 32-39. doi:10.13597/j.cnki.maize.science.20180307.
Li Z N, Wang K W, Wang Y R, Wu S H, Li G F. Genetic analysis on pedigree and agronomic characters of maize variety Xianyu 335[J]. Journal of Maize Sciences, 2018, 26(3): 32-39.
[27] S nchez-fern
nchez-fern ndez R, Davies T G E, Coleman J O D, Rea P A. The Arabidopsis thaliana ABC protein superfamily, a complete inventory[J]. Journal of Biological Chemistry, 2001, 276(32): 30231-30244. doi:10.1074/jbc.M103104200.
ndez R, Davies T G E, Coleman J O D, Rea P A. The Arabidopsis thaliana ABC protein superfamily, a complete inventory[J]. Journal of Biological Chemistry, 2001, 276(32): 30231-30244. doi:10.1074/jbc.M103104200.
[28] Pilu R, Cassani E, Villa D, Curiale S, Panzeri D, Badone F C, Landoni M. Isolation and characterization of a new mutant allele of brachytic 2 maize gene[J]. Molecular Breeding, 2007, 20(2): 83-91. doi:10.1007/s11032-006-9073-7.
[29] Xing A Q, Gao Y F, Ye L F, Zhang W P, Cai L C, Ade C, Liaca V, Johnson B, Liu L, Yang X H, Kang D M, Yan J B, Li J S. A rare SNP mutation in Brachytic2 moderately reduces plant height and increases yield potential in maize[J]. J Exp Bot, 2015, 66(13): 3791-3802. doi:10.1093/jxb/erv182.
[30] Jamie S,Guillaume T, Houry W A. The AAA+ superfamily of functionally diverse proteins[J]. Genome Biology, 2008, 9(4): 1-8. doi:10.1186/gb-2008-9-4-216.
[31] Azzaria M, Schurr E, Gros P. Discrete mutations introduced in the predicted nucleotide-binding sites of the mdr1 gene abolish its ability to confer multidrug resistance[J]. Molecular & Cellular Biology, 1989, 9(12): 5289-5297. doi:10.1128/MCB.9.12.5289.
[32] Beaudet L, Gros P. Functional dissection of P-glycoprotein nucleotide-binding domains in chimeric and mutant proteins. Modulation of drug resistance profiles[J]. Journal of Biological Chemistry, 1995, 270(29): 17159-17170. doi:10.1074/jbc.270.29.17159.