植物叶色突变与光合作用密切相关,叶色突变体的获得为研究植物的光合作用途径以及生理代谢过程提供了关键材料[1]。ALB3即ALBINO3,可能参与叶绿体的生物发生,在植物的衰老过程中发挥重要作用。其编码产物与植物叶绿体蛋白相关的细菌膜蛋白YidC以及酵母菌OXA1蛋白具有序列相似性,具有高度保守的结构域,位于叶绿体类囊体膜上,参与叶绿体酶复合体的组装[2]。因此,ALB3的缺失可使拟南芥叶绿体发育异常,导致拟南芥叶片白化。
拟南芥ALB3突变体于1993年由Long等[3]首次得到,其利用玉米转座子系统Ac/Ds作为诱变剂,Ds的插入导致拟南芥产生白化现象,从而得到ALB3纯合突变体,利用此种方法获取突变体耗时且过程繁琐,纯合突变体幼苗生长在土壤中会致死,但当培养在萌发培养基(GM培养基)时,ALB3纯合突变体可以长出大约12片叶子,偶然会开花,但依旧不育[2]。随后,证明ALB3是叶绿体分化重要的核基因,突变可导致类囊体不能正常发生[2]。近些年来,关于拟南芥ALB3的研究极少,这可能与难以获得ALB3突变体且突变体容易致死有关,但是关于ALB3的研究相对较多,比如ALB3膜插入酶的C端可以聚集cpSRP43到类囊体膜[4];拟南芥ALB3与亚碲酸钾抗性C蛋白共同参与光系统Ⅱ蛋白合成[5];拟南芥ALB3不需要保守的带正电荷的残基来功能性地补充大肠杆菌YidC消耗菌株或促进YidC依赖性膜蛋白的插入[6];植物和藻类叶绿素合成酶在集胞藻中起作用并可与YidC/Alb3膜插入酶相互作用[7]; ALB3转位酶的基质结构域以高亲和力结合并调节cpSRP54·cpFtsY复合物中的GTP水解等[8]。上述报道都为进一步研究ALB3的功能奠定了基础。
为了更高效地对拟南芥ALB3进行定点编辑,建立一种更高效、安全的基因编辑体系,本研究利用CRISPR/Cas9基因编辑技术构建一种带有红色荧光蛋白基因mCherry且可敲除拟南芥ALB3的载体,利用可视化与分子生物学相结合的方法快速筛选突变幼苗,大大节省了操作步骤,提高了工作效率,为快速获取不含外源转化元件的基因编辑后代提供借鉴与参考。
1 材料和方法
1.1 主要试验材料
本试验所用的带有荧光筛选标记基因mCherry的质粒pHDE-mCherry由加州大学(圣地亚哥分校)Professor Zhao赠送,DH5α大肠杆菌感受态细胞和GV3101农杆菌感受态细胞分别购自广州鼎国生物技术有限公司和上海维地生物技术有限公司,Col-0野生型拟南芥生长于广东石油化工学院植物培养室。
1.2 主要试验试剂
质粒小提试剂盒、琼脂糖凝胶DNA回收试剂盒、Taq DNA Polymerase(ET101)以及DNA Marker购于北京天根生化科技有限公司;卡那霉素和DNA Loading Buffer于上海生工生物工程股份有限公司购置,Epicentre T5 exonuclease、NEB Taq DNA Ligase、Bsa Ⅰ限制性内切酶以及NEB Phusion DNA polymerase均购于New England Biolabs公司(美国),二硫苏糖醇(DTT)购于北京碧云天生物技术公司。
1.3 靶位点的设计
从NCBI上下载拟南芥ALB3基因序列(NC_003071.7),并以此为目标序列,通过CRISPR/Cas target在线设计网站(http://crispr.dbcls.jp/)设计ALB3基因的2个Target位点,Target位点共包含23个碱基,形式如G-N19-NGG,第一个碱基G为小RNA转录起始位点,G-N19为插入载体的gRNA序列,NGG为PAM(Proto-spacer adjacent motif)序列,是Cas9基因的识别位点,即下划线位置。Target1:5′-GTCCGTCACAGTGGAGTAGGAGG-3′;Target2:5′-CCTCTGCCGAGCAGCTATAGTGG-3′。
1.4 引物设计
试验所用引物在Primer primer 5软件上设计,由上海生工生物工程股份有限公司进行合成(表1)。
表1 试验所用引物序列
Tab.1 Primer sequence used in the experiment
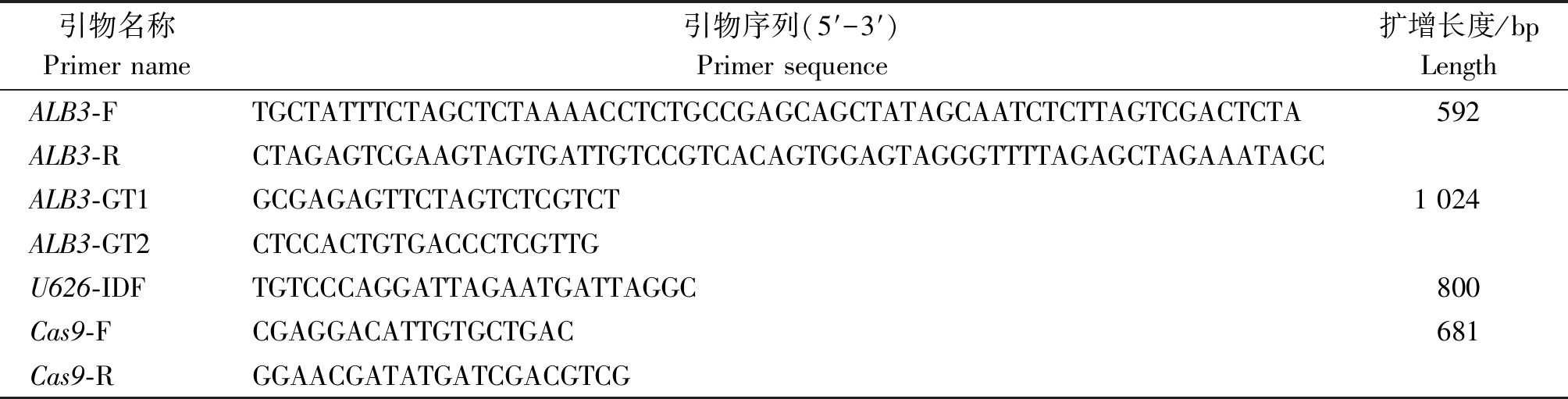
引物名称Primer name引物序列(5′-3′)Primer sequence扩增长度/bp LengthALB3-FTGCTATTTCTAGCTCTAAAACCTCTGCCGAGCAGCTATAGCAATCTCTTAGTCGACTCTA592ALB3-RCTAGAGTCGAAGTAGTGATTGTCCGTCACAGTGGAGTAGGGTTTTAGAGCTAGAAATAGCALB3-GT1GCGAGAGTTCTAGTCTCGTCT1 024ALB3-GT2CTCCACTGTGACCCTCGTTGU626-IDFTGTCCCAGGATTAGAATGATTAGGC800Cas9-FCGAGGACATTGTGCTGAC681Cas9-RGGAACGATATGATCGACGTCG
1.5 pHDE-mCherry-ALB3表达载体的构建
1.5.1 ALB3 sgRNA片段的扩增 以pCBC质粒(含gRNA)为模板,ALB3-F和ALB3-R为引物,进行PCR扩增,反应程序:预变性94 ℃ 4 min,35个循环(变性94 ℃ 30 s,退火56 ℃ 30 s,延伸72 ℃ 45 s),总延伸72 ℃ 2 min,4 ℃ 保存。用1.0%的琼脂糖凝胶电泳对PCR产物进行检验,然后将检验成功的PCR产物(目的条带)用琼脂糖凝胶DNA回收试剂盒进行回收。
1.5.2 质粒pHDE-mchenry的酶切及同源重组 用Bas Ⅰ酶对pHDE-mCherry质粒进行酶切,20 μL体系,37 ℃反应4 h,以未酶切质粒为对照,用1.0%的琼脂糖凝胶电泳对酶切产物进行验证。
将1.5.1纯化回收所得到的ALB3 sgRNA片段与酶切产物进行同源重组,4.5 μL体系(重组酶3 μL,酶切质粒0.3 μL,ALB3 sgRNA片段1.2 μL),50 ℃水浴1 h。
1.5.3 重组产物转化大肠杆菌以及菌液PCR鉴定 利用热击法将重组产物导入DH5感受态细胞,方法参照广州鼎国生物技术有限公司DH5α感受态使用说明(产品编号MCC001),将转化后的DH5α感受态细胞在LB固体培养基上(含卡那霉素)培养12 h,挑选单菌落并进行PCR鉴定,反应程序同1.5.1。
1.5.4 重组质粒pHDE-mCherry-ALB3的提取与阳性重组子鉴定 将菌液PCR鉴定结果为阳性的菌液在LB液体培养基中(含50 mg/mL卡那霉素,卡那霉素与LB液体培养基的体积比为1/1000)扩大培养,并于37 ℃摇菌12 h,用质粒小提试剂盒提取重组质粒,进行质粒PCR鉴定,反应条件同菌液PCR。
挑选质量较好的阳性重组质粒送至生工生物工程股份有限公司(广州)测序,并用Mega 6.06软件进行序列比对验证。
1.6 拟南芥的转化
1.6.1 重组质粒转化农杆菌及浸染液的配制 将测序成功的质粒转入农杆菌感受态细胞,转化方法参照上海唯地生物技术有限公司说明书(产品编号AC1001),将转化后的农杆菌GV3103感受态细胞于LB固体培养基上(含卡那霉素和利福平)于28 ℃培养48 h,挑取单菌落并进行PCR鉴定。反应程序同1.5.1。
经琼脂糖凝胶电泳验证,挑选条带质量最好的菌液于LB液体培养基(含卡那霉素和利福平)中扩繁培养,28 ℃摇菌16 h。收集菌体,重悬于100 mL浸染缓冲液(5%的蔗糖溶液,0.02%的Silwet L-77)中,浸染液即配制完成。
1.6.2 拟南芥的浸染 将Col-0野生型拟南芥种子进行种植,出现花蕾后打顶,待侧枝出现花蕾后,于浸染前1 d将所有开花的花朵以及荚果剪除。
用农杆菌浸染液浸染花苞,轻轻搅动1~2 min后取出,将叶片以及茎上的残留液体用吸水纸吸除,暗培养48 h后正常培养,待新一批花苞长出时再次浸染。
1.7 转化后代的筛选与鉴定
1.7.1 转化T0种子的可视化筛选 收取转化后的拟南芥种子,命名为T0种子,利用倒置荧光显微镜在T0种子中筛选带有红色荧光的种子并种植,得到T1植株。
1.7.2 突变植株的筛选及鉴定 观察T1植株的表型是否与野生型植株有所异同,并剪取T1植株(尤其是表型特殊的)幼嫩叶片,提取植株的DNA,以此为模板,利用ALB3-GT1和ALB3-GT2这对鉴定引物对目标序列进行PCR扩增,反应程序:预变性94 ℃ 4 min,35个循环(变性94 ℃ 30 s,退火55 ℃ 30 s,延伸72 ℃ 45 s),总延伸72 ℃ 2 min,4 ℃ 保存。用1.0%的琼脂糖凝胶电泳对目的条带的大小进行验证,确认是否将目标基因ALB3敲除成功。然后以Cas9-F和Cas9-R这对引物对已敲除ALB3的突变植物进行PCR扩增,反应程序:预变性94 ℃ 4 min,35个循环(变性94 ℃ 30 s,退火56 ℃ 30 s,延伸72 ℃ 45 s),总延伸72 ℃ 2 min,4 ℃ 保存。验证其是否携带外源转化元件Cas9蛋白。
利用倒置荧光显微镜筛选T1种子中不带荧光的种子进行种植,并对其幼苗(T2幼苗)进行ALB3敲除验证以及是否携带外源转化元件Cas9蛋白鉴定,方法同T1植株。
2 结果与分析
2.1 pHDE-mCherry-ALB3表达载体的构建结果
Bsa Ⅰ酶切pHDE-mCherry质粒(图1-A),酶切后因质粒线性化,电泳迁移率慢于酶切前。经PCR扩增目的基因ALB3 sgRNA(图1-B),条带大小为500~750 bp,与预期扩增长度592 bp相一致,切胶回收后可与酶切产物同源重组。
重组产物转化DH5α感受态细胞后,菌液PCR结果(图1-C)显示,扩增条带大小为500~750 bp,与预期(592 bp)相符,且阳性率为100%。
挑选目的条带质量较好的菌液扩繁培养,提取质粒并对质粒进行PCR鉴定(图1-D),条带大小符合预期,初步判定pHDE-mCherry-ALB3表达载体构建成功。送样测序后,通过序列比对(图2),进一步确认pHDE-mCherry-ALB3表达载体构建成功。
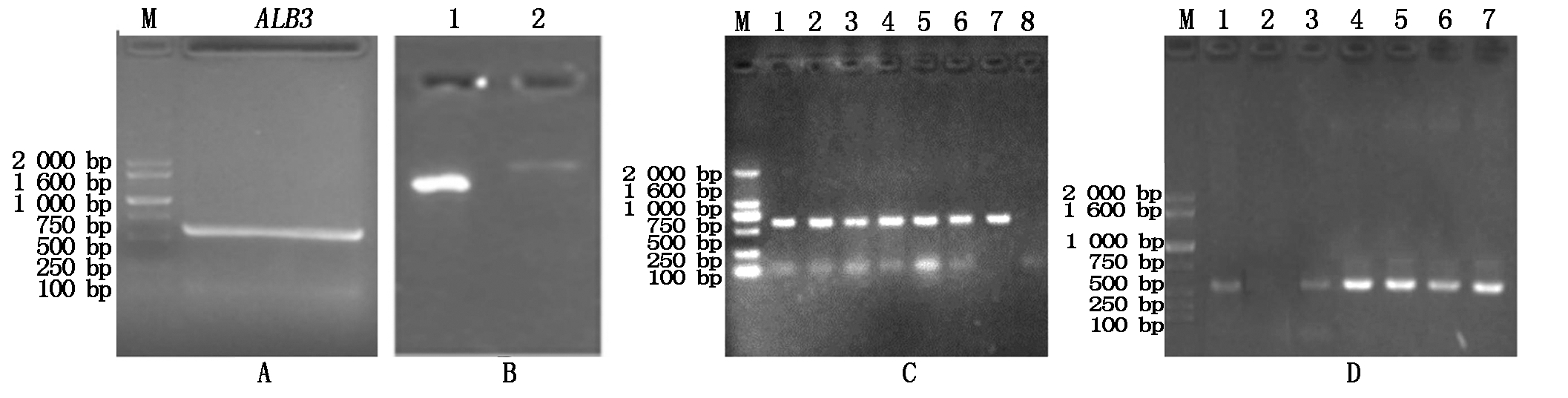
A.ALB3sgRNA片段的PCR扩增:M.DL2000 DNA Marker;B.Bsa Ⅰ酶切pHDE-mCherry质粒电泳结果: 1.未酶切的pHDE-mCherry质粒;2.Bsa Ⅰ酶切的pHDE-mCherry质粒;C.pHDE-mCherry-ALB3重组质粒菌液PCR鉴定结果:M.DL2000 DNA Marker; 1-6.重组质粒转化菌液 PCR结果;7.阳性对照; 8.阴性对照;D.重组载体pHDE-mCherry-ALB3 PCR鉴定结果:M.DL2000 DNA Marker;1.阳性对照; 2.阴性对照;3-7.重组质粒pHDE-mCherry-ALB3 PCR结果。
A.The PCR amplification result of ALB3sgRNA sequence:M.DL2000 DNA Marker; B.The Bsa Ⅰ digestion result of pHDE-mCherry plasmid: 1. Original pHDE-mCherry plasmid;2.pHDE-mCherry plasmid digested with Bsa Ⅰ enzyme; C.The bacterial so lution PCR identification results of pHDE-mCherry-ALB3 recombinant plasmid:M.DL2000 DNA Marker; 1-6. PCR result of recombinant plasmid transforming bacterial solution;7.Positive control; 8. Negative control; D.The PCR identification result of recombinant vector pHDE-mCherry-ALB3:M. DL2000 DNA Marker;1.Positive control; 2.Negative control;3-7. PCR result of recombinant plasmid pHDE- mCherry-ALB3.
图1 pHDE-mCherry-ALB3表达载体的构建
Fig.1 Construction of pHDE-mCherry-ALB3 expression vector
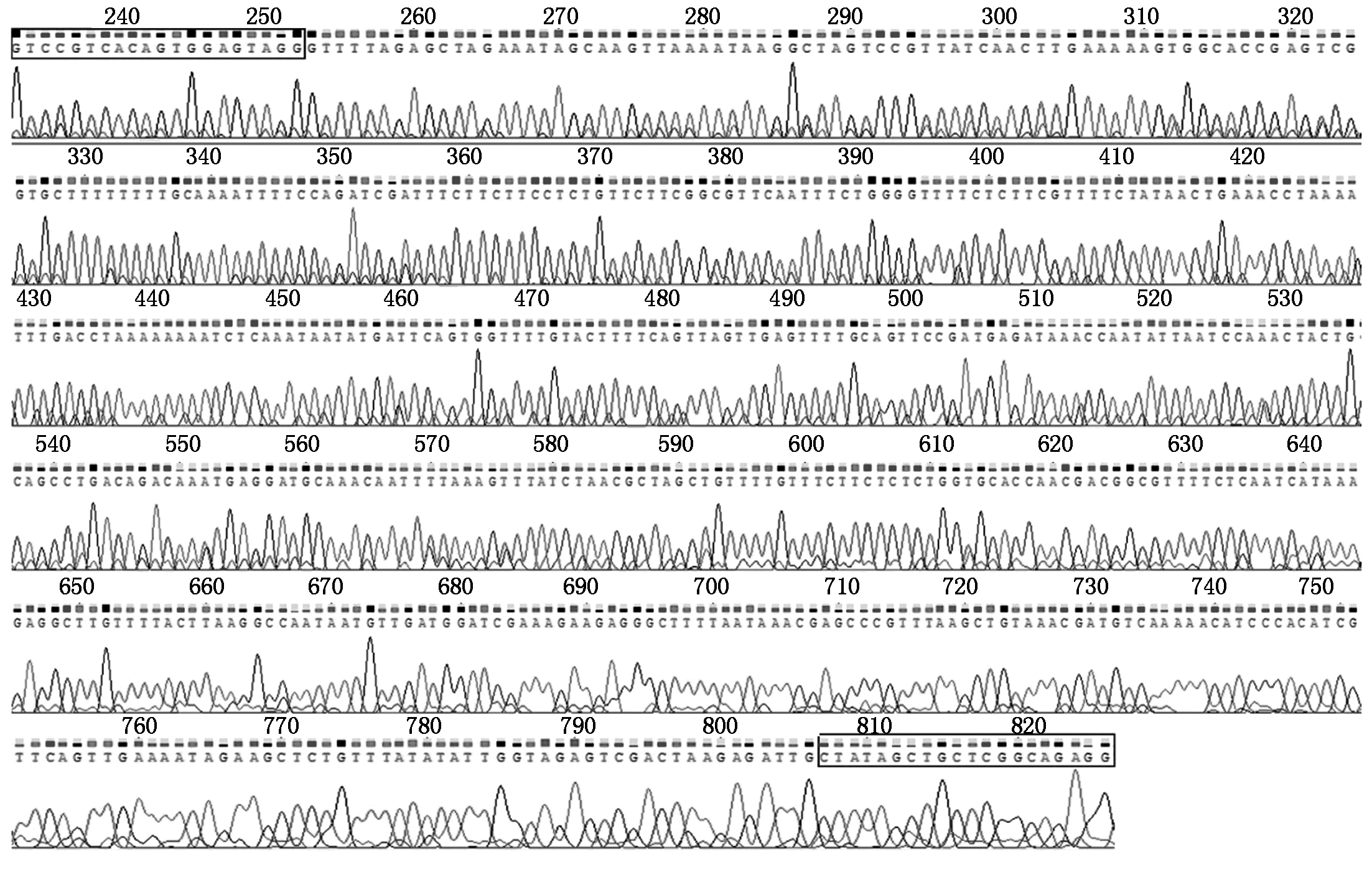
图2 序列比对结果
Fig.2 The result of sequence alignment
2.2 重组质粒pHDE-mCherry-ALB3转化农杆菌结果
重组质粒转化农杆菌GV3103感受态细胞后,为了验证是否成功转化,对其菌液进行PCR鉴定,鉴定结果(图3)显示条带大小在500~750 bp,符合预期大小,说明工程菌转化成功。
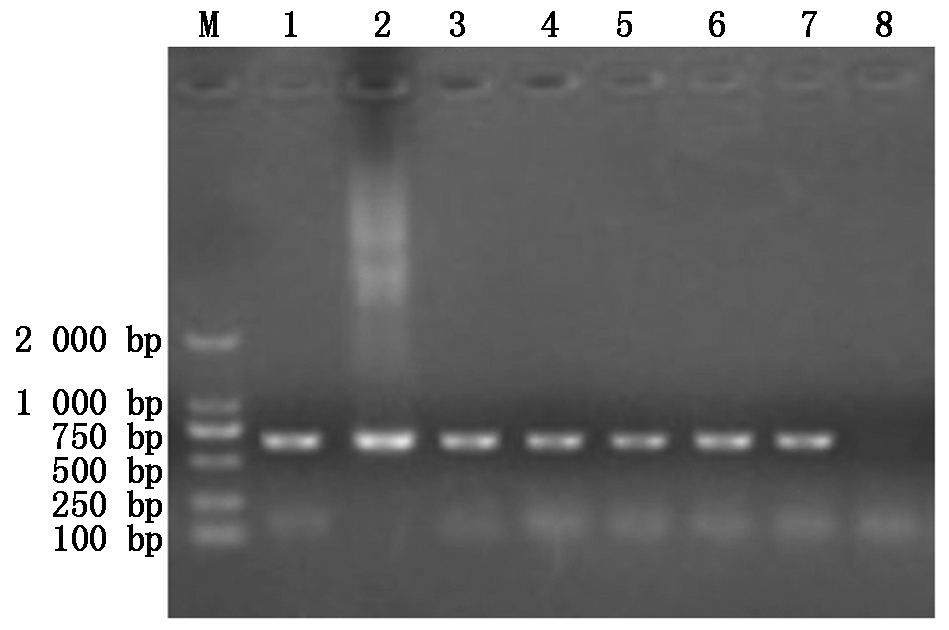
M.DL2000 DNA Marker;1-6.重组质粒pHDE-mCherry-ALB3转化农杆菌 PCR结果;7.阳性对照;8.阴性对照。
M.DL2000 DNA Marker;1-6. PCR result of recombinant plasmid pHDE-mCherry-ALB3 transfer Agrobacterium;7.Positive control;8.Negative control.
图3 重组质粒pHDE-mCherry-ALB3转化农杆菌PCR鉴定结果
Fig.3 The PCR identification result of recombinant vector pHDE-mCherry-ALB3 transfer Agrobacterium
2.3 转化后代的可视化筛选
2.3.1 阳性转化种子筛选 本试验所用表达载体中含有红色荧光蛋白mCherry基因,成功转化的种子在580 nm激发光的照射下可激发出红色荧光(图4),没有转化成功的种子没有荧光,极大地提高了阳性转化后代的筛选效率。
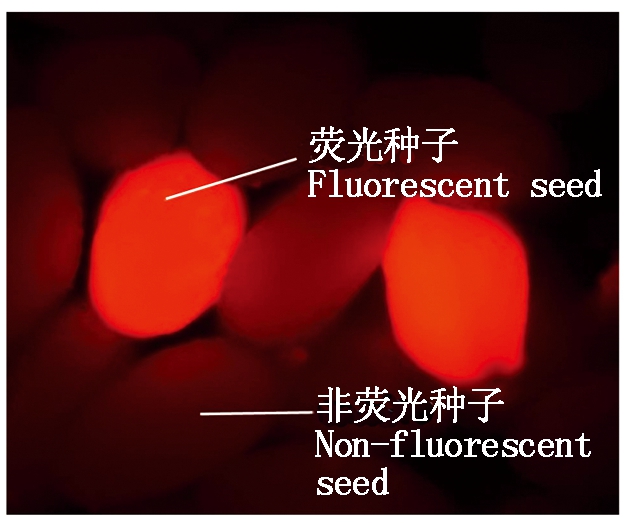
图4 倒置荧光显微镜下种子显色情况
Fig.4 Seed color development under inverted fluorescence microscope
2.3.2 编辑后代的表型及分子鉴定结果 将筛选得到的红色荧光种子种植在培养基上,萌发后观察到一些幼苗的叶子极淡,为浅黄色(图5),将这些幼苗移植到土壤中生长一段时间,叶子颜色更淡,为白色(图6),表现为ALB3基因缺失的表型。
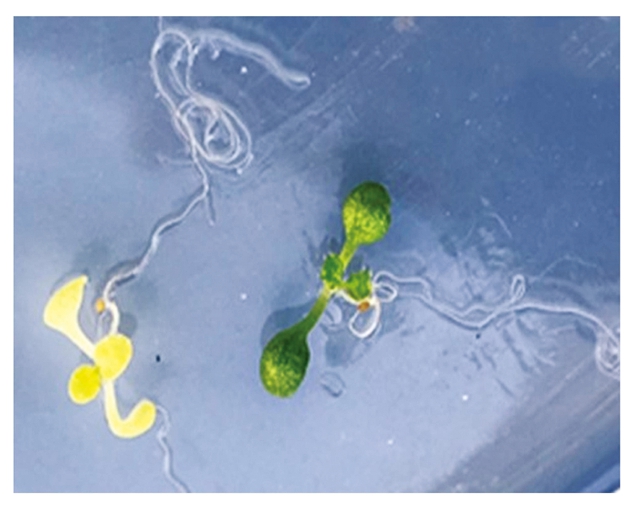
图5 培养基中突变幼苗与野生幼苗生长情况
Fig.5 Growth of mutant seedlings and wild seedlings in culture medium
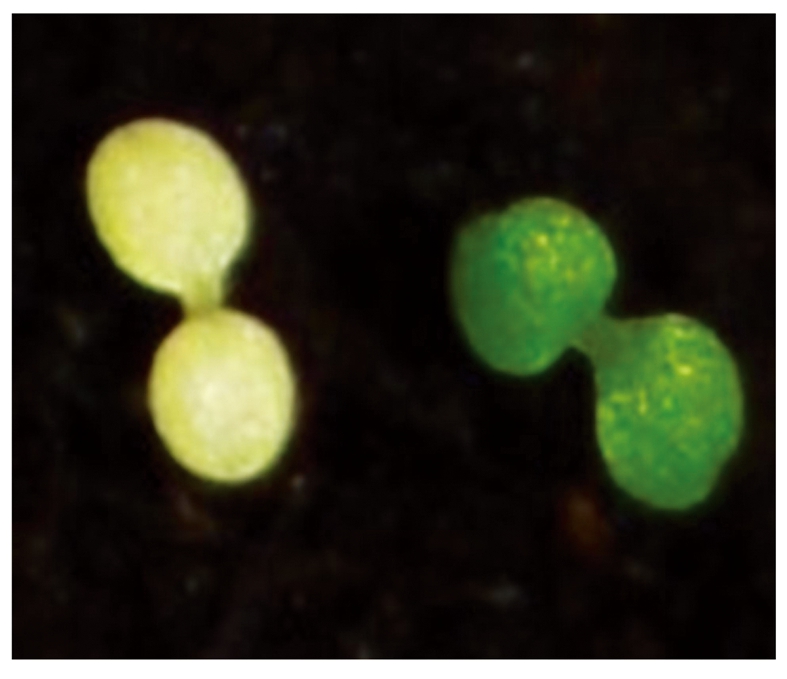
图6 土壤中突变幼苗与野生幼苗生长情况
Fig.6 Growth of mutant seedlings and wild seedlings in soil
为验证ALB3基因编辑情况,对上述白化植株进行 PCR鉴定(图7-A)。由于本试验中对ALB3基因设计了2个编辑位点,从而实现大片段的敲除,在2个编辑位点上下游设计引物进行PCR扩增时,野生型对照(泳道9)扩增得到一个大片段,而泳道8扩增片段大小明显小于野生植株,为ALB3基因中间敲除后扩增得到的小片段,即为纯合突变,泳道1和5扩增得到一大一小2个片段,表明这2个植株为杂合突变。对含红色荧光种子的T1植株进行外源转化元件Cas9蛋白的检验(图7-B),均能扩增得到Cas9基因片段,说明T1植株均含外源转化元件Cas9蛋白。
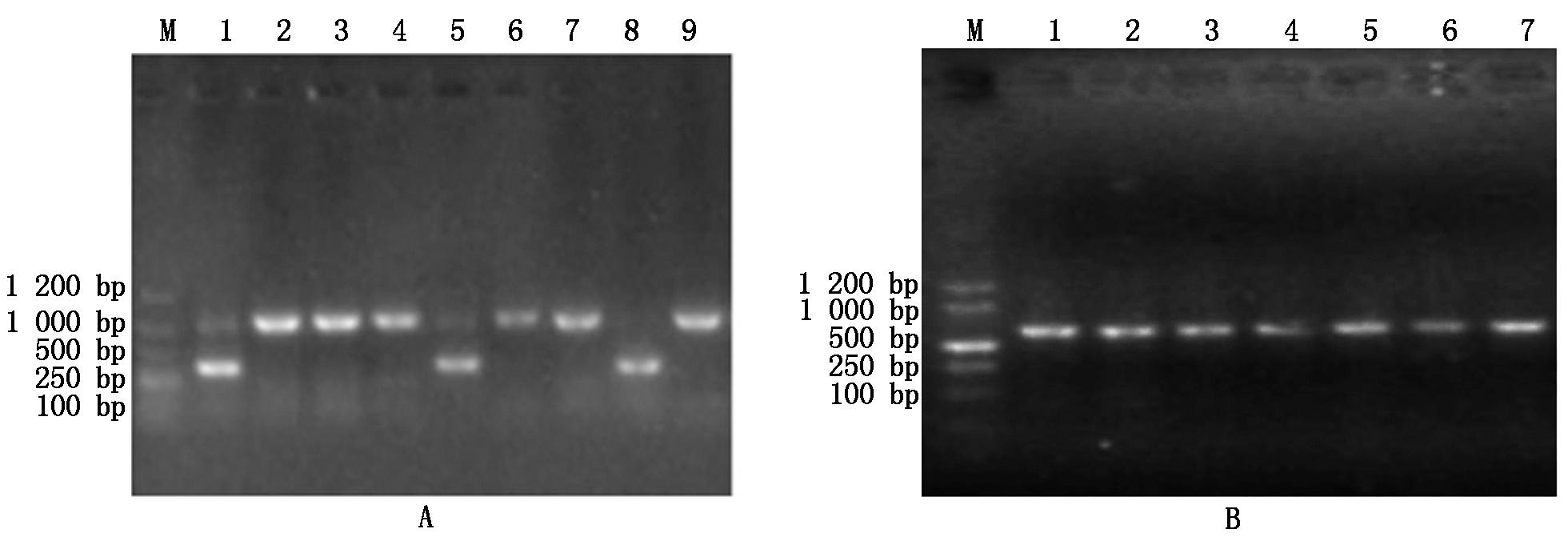
A.T1突变体植株的基因敲除鉴定:M.DL1200 DNA Marker;1和5.T1杂合突变幼苗; 8.T1纯合突变幼苗;2-4.T1未发生突变幼苗;9.野生幼苗(对照组); B.T1突变体植株的Cas9检验:M.DL1200 DNA Marker; 1-6.T1突变幼苗; 7.野生幼苗(对照组)。
A. Gene knockout identification of T1 mutant plants:M. DL1200 DNA Marker; 1 and 5.T1 generation homozygous mutant seedlings; 8.Heterozygote mutants in the T1 generation;2-4. Mutants of T1 generation without mutation;9.Wild seedlings (control group); B. Identification of T1 generation mutant plants with Cas9:M.DL1200 DNA Marker;1-6. T1 generation mutant seedlings;7.Wild seedlings (control group).
图7 T1突变体植株分子鉴定结果
Fig.7 Molecular identification results of T1 mutant plants
对T2幼苗进行ALB3敲除鉴定(图8-A)和外源转化元件Cas9蛋白检验(图8-B),发现纯合突变幼苗都不含Cas9基因,由此成功获得不含外源转化元件Cas9蛋白的纯合突变植株。但是突变植株经过一段时间的培养后,一直处于只有2片叶子的状态,无法存活,这可能是ALB3基因的缺失引起的,需深入研究。
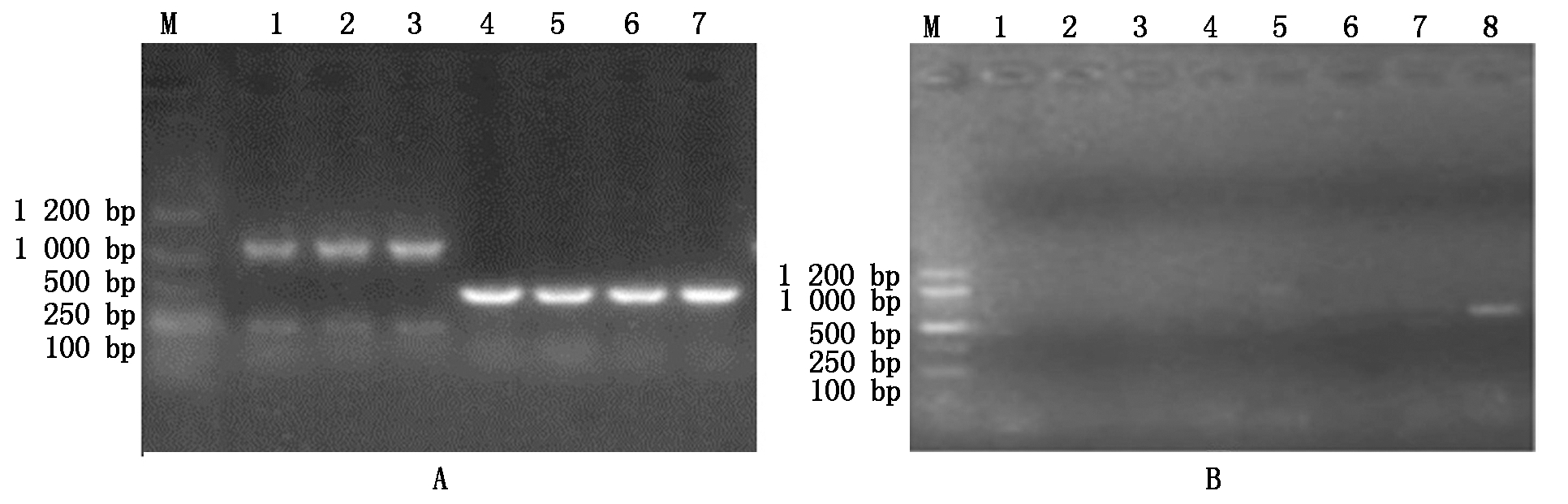
A. T2突变体植株的基因敲除鉴定:M.DL1200 DNA Marker;1.野生幼苗(对照组);2-3.T2未发生突变幼苗;4-7. T2纯合突变幼苗; B. T2突变体植株的Cas9检验: M.DL1200 DNA Marker; 1-7.T2突变幼苗;8.野生幼苗(对照组)。
A.Gene knockout identification of T2 mutant plants:M.DL1200 DNA Marker;1.Wild seedlings (control group);2-3. Mutants of T2 generation without mutation;4-7. T2 generation homozygous mutant seedlings;B.Identification of T2 generation mutant plants with Cas9:M. DL1200 DNA Marker;1-7. T2 generation mutant seedlings;8.Wild seedlings (control group).
图8 T2突变体植株分子鉴定结果
Fig.8 Molecular identification results of T2 mutant plants
3 讨论
3.1 mCherry在筛选突变种子中的应用优势
mCherry为一种红色荧光蛋白基因,来自于蘑菇珊瑚(Mushroom coral),所编码的蛋白具有很强的光稳定性[9]。本研究在所构载体中引入mCherry,利用种皮特异性启动子At2S3促使其表达,在荧光显微镜特定波长的激发下,应用外源转化元件中带有荧光蛋白的标记特性,筛选拟南芥转化初期带有荧光的种子,实现对突变体的可视化筛选,提高获得转化后代阳性植株比例,提高筛选效率。这种通过可视化荧光筛选标记来判断种子中是否含有CRISPR/Cas9表达系统的方法简单易行,准确性高,对基因编辑事件的检测在经过可视化筛选后只需要进行简单的PCR验证,即可确定是否获得Cas9-Free的突变后代,极大地提高了工作效率,为有性繁殖作物基因编辑后代中获得新种质资源提供了一种全新的技术方法,具有一定的创新性。
3.2 CRISPR/Cas9基因编辑技术的应用优势
CRISPR/Cas9技术作为一种有效的基因编辑工具,具有操作简单、高效性和特异性等其他基因编辑工具不可比拟的优势[10-14]。利用TALEN和CRISPR/Cas9 2种基因编辑技术分别对同一种基因进行改造,效率分别为0~34%,51%~79%,充分证明了CRISPR/Cas9技术的高效性,且相较于前者,CRISPR/Cas9技术操作简单[15]。目前,CRISPR/Cas9技术已在大豆[16]、水稻[17-18]、亚麻[19]、伴矿景天[20]、木薯[21]、甘蓝[22]等植物中实现了基因编辑,应用广泛,为植物特定基因的功能探究以及新种质资源的创建提供了技术参考。
本研究设计2个sgRNA,使其同时识别靶基因ALB3 的2个不同位点,构建一种带有荧光筛选标记且可实现靶基因敲除的基因编辑体系。通过转化拟南芥,挑选转化后种皮上带有红色荧光标记的T0阳性种子进行种植,得到T1幼苗,经观察植株表型以及PCR鉴定得到纯合突变幼苗,但此时纯合突变幼苗存在Cas9表达核且会致死,为了得到不携带Cas9表达核的纯合突变体,需进一步挑选T1植株中不带荧光的种子进行培育,得到的T2幼苗,经表型观察以及PCR鉴定,得到纯合突变幼苗,利用PCR技术对Cas9基因进行检测,Cas9基因已去除,说明外源转化元件Cas9蛋白已随着配子产生过程中染色体的分离而去除,由此得到无Cas9蛋白外源转化元件的纯合突变体幼苗,可稳定遗传。本技术成功分离出外源转化元件Cas9蛋白,获得可稳定遗传的纯合突变体。为探索ALB3在光合作用中的作用提供了理想材料,并为研究ALB3在植物生长发育的作用以及新种质资源的建立奠定了基础。
[1] 郭彦,乔清华,刘立科,吕山花,张文会.拟南芥白化突变体的叶绿体分析及突变基因克隆[C]// 山东植物生理学会第七次代表大会暨植物生物学与现代农业研讨会论文集,2012:64-68.
Guo Y,Qiao Q H,Liu L Q,Lü S H,Zhang W H. Chloroplast analysis and mutation gene cloning of Arabidopsis whitening mutant [C]// Proceedings of the 7th Congress of the Plant Physiology Society of Shandong and the Symposium on Plant Biology and Modern Agriculture,2012:64-68.
[2] Sundberg E,Slagter J G,Fridborg I,Cleary S P,Robinson C,Coupland G. ALBINO3,an Arabidopsis nuclear gene essential for chloroplast differentiation,encodes a chloroplast protein that shows homology to proteins present in bacterial membranes and yeast mitochondria[J]. The Plant Cell,1997,9:717-730. doi: 10.1105/tpc.9.5.717.
[3] Long D,Martin M,Sundberg E,Swinburne J,Puangsomlee P,Coupland G. The maize transposable element system Ac/Ds as a mutagen in Arabidopsis:identification of an albino mutation induced by Ds insertion[J]. Proceedings of the National Academy of Sciences of the United States of America,1993,90(21): 10370-10374. doi: 10.1073/pnas.90.21.10370.
[4] Dünschede B,Bals T,Funke S,Schünemann D. Interaction studies between the chloroplast signal recognition particle subunit cpSRP43 and the full-length translocase Alb3 reveal a membrane-embedded binding region in Alb3 protein[J].Journal of Biological Chemistry,2011,286(40): 35187-35195. doi: 10.1074/jbc.M111.250746.
[5] Schneider A,Steinberger I,Strissel H,Kunz H H,Manavski N,Meurer J,Burkhard G,Jarzombski S,Schünemann D,Geimer S,Flügge U I,Leister D.The Arabidopsis tellurite resistance C protein together with ALB3 is involved in photosystem Ⅱ protein synthesis[J]. The Plant Journal,2014,78(2): 344-356. doi: 10.1111/tpj.12474.
[6] Chen Y Y,Soman R,Shanmugam S K,Kuhn A,Dalbey R E. The role of the strictly conserved positively charged residue differs among the gram-positive,gram-negative,and chloroplast YidC homologs[J]. Journal of Biological Chemistry,2014,289(51): 35656-35667. doi: 10.1074/jbc.M114.595082.
[7] Proctor M S,Chidgey J W,Shukla M K,Jackson P J,Sobotka R,Hunter C N,Hitchcock A. Plant and algal chlorophyll synthases function in Synechocystis and interact with the YidC/Alb3 membrane insertase[J]. FEBS Lett,2018,592(18):3062-3073. doi: 10.1002/1873-3468.13222.
[8] Chandrasekar S, Shan S O. Anionic phospholipids and the Albino3 translocase activate signal recognition particle-receptor interaction during light-harvesting chlorophyll a/b-binding protein targeting[J]. J Biol Chem,2017,292(1): 397-406. doi: 10.1074/jbc.M116.752956.
[9] 欧阳乐军,袁玉梅,李莉梅,陈凯钊,戴发.巨桉miR156 CRISPR/Cas9载体构建[J].森林与环境学报,2018,38(4): 106-111. doi: 10.13324/j.cnki.jfcf.2018.04.016.
Ouyang L J,Yuan Y M,Li L M,Chen K Z,Dai F.Construction of Eucalyptus grandis miR156 family CRISPR/Cas9 vector [J]. Journal of Forest and Environment,2018,38(4): 106-111.
[10] 赵山山,邸一桓,郝光飞. CRISPR-Cas9基因编辑技术在基因功能和作物育种中的研究进展[J].分子植物育种,2019,17(21):7087-7093.doi:10.13271/j.mpb.017.007087.
Zhao S S,Di Y H,Hao G F. Research progress of CRISPR-Cas9 gene editing technology in gene function and crop breeding [J]. Molecular Plant Breeding,2019,17(21):7087-7093.
[11] Ali Z,Zaidi S S,Tashkandi M,Mahfouz M M. A simplified method to engineer CRISPR/Cas9-mediated geminivirus resistance in plants[J]. Antiviral Resistance in Plants,2019,2028:167-183. doi: 10.1007/978-1-4939-9635-3_10.
[12] Osmani Z,Jin S,Mikami M,Endo M,Atarashi H,Fujino K,Yamada T,Nakahara K S. CRISPR/Cas9-mediated editing of genes encoding rgs-CaM-like proteins in transgenic potato plants[J]. Antiviral Resistance in Plants,2019,2028:153-165. doi: 10.1007/978-1-4939-9635-3_9.
[13] Schuster M,Kahmann R. CRISPR-Cas9 genome editing approaches in filamentous fungi and oomycetes[J]. Fungal Genet Biol,2019,130: 43-53. doi: 10.1016/j.fgb.2019.04.016.
[14] Ma K,Han J L,Hao Y,Yang Z F,Chen J Y,Liu Y G,Zhu Q L,Chen L T. An effective strategy to establish a male sterility mutant mini-library by CRISPR/Cas9-mediated knockout of anther-specific genes in rice[J]. Journal of Genetics and Genomics,2019,46(5): 273-275. doi: 10.1016/j.jgg.2019.03.005.
[15] Ding Q R,Regan S N,Xia Y L,Oostrom L A,Cowan C A,Musunuru K. Enhanced efficiency of human pluripotent stem cell genome editing through replacing TALENs with CRISPRs[J]. Cell Stem Cell,2013,12(4):393-394. doi: 10.1016/j.stem.2013.03.006.
[16] 侯智红,吴艳,程群,董利东,芦思佳,南海洋,甘卓然,刘宝辉.利用CRISPR/Cas9技术创制大豆高油酸突变系[J].作物学报,2019,45(6): 839-847. doi: 10.3724/SP.J.1006.2019.84157.
Hou Z H,Wu Y,Cheng Q,Dong L D,Lu S J,Nan H Y,Gan Z R,Liu B H. Creation of high oleic acid soybean mutation plants by CRISPR/Cas9 [J]. Acta Agronomica Sinica,2019,45(6): 839-847.
[17] Yang C H,Zhang Y,Huang C F. Reduction in cadmium accumulation in japonica rice grains by CRISPR/Cas9-mediated editing of OsNRAMP5[J]. Journal of Integrative Agriculture,2019,18(3): 688-697. doi: 10.1016/S2095-3119(18)61904-5.
[18] 孙慧宇,宋佳,王敬国,刘化龙,孙健,莫天宇,徐善斌,郑洪亮,邹德堂.利用CRISPR/Cas 9技术编辑Badh2基因改良粳稻香味[J].华北农学报,2019,34(4):1-8.doi:10.7668/hbnxb.201751503.
Sun H Y.Song J,Wang J G,Liu H L,Sun J,Mo T Y,Xu S B,Zheng H L,Zou D T.Editing Bach2 gene to improre the fragrance of japonica rice by CIRSPR/cas9 technology[J].Acta Agricuturae Boreali-Sinica,2019,34(4):1-8.
[19] 张喻,江海霞,闫文亮,郭栋良,杨亮杰,叶佳丽,王玥,谢丽琼. CRISPR-Cas9系统敲除亚麻FAD2基因表达载体的构建[J]. 分子植物育种,2019,17(7): 2185-2192. doi: 10.13271/j.mpb.017.002185.
Zhang Y,Jiang H X,Yan W L,Guo D L,Yang L J,Ye J L,Wang Y,Xie L Q. Construction of CRISPR-Cas9 system knockout flax FAD2 gene expression vector[J]. Molecular Plant Breeding,2019,17(7): 2185-2192.
[20] Zhao H X,Wang L S,Zhao F J,Wu L H,Liu A N,Xu W Z. SpHMA1 is a chloroplast cadmium exporter protecting photochemical reactions in the Cd hyperaccumulator Sedum plumbizincicola[J]. Plant,Cell & Environment,2019,42(4): 1112-1124. doi: 10.1111/pce.13456.
[21] 谢龙莲.利用CRISPR/Cas9基因编辑技术培育抗褐条病木薯[J].世界热带农业信息,2018(11):36-37.
Xie L L. Cultivating anti-browning cassava with CRISPR/Cas9 gene editing technology [J]. World Tropical Agriculture Information,2018(11): 36-37.
[22] 孙勤富,刘东晓,林俐,吴德伟,吴健,王幼平. 甘蓝型油菜和甘蓝CRISPR/Cas9编辑效果的快速检测[J].中国油料作物学报,2018,40(6):737-744. doi:10.7505/j.issn.1007-9084.2018.06.001.
Sun Q F,Liu D X,Lin L,Wu D W,Wu J,Wang Y P. Rapid detection of CRISPR/Cas9-mediated genome editing efficiency in Brassica napus and B. oleracea[J]. Chinese Journal of Oil Crop Sciences,2018,40(6): 737-744.