梭梭(Haloxylon ammodendron)属黎科(Chenopodiaceae),为超旱生小乔木,呈高大灌木状,植株高度一般在1~4 m,个别植株可高达8~10 m,亦称盐木、琐琐树、梭梭柴。梭梭是肉苁蓉(Cistanche deserticola Y.C.Ma)的寄主,肉苁蓉是名贵的中药材,在医学上具有抗肿瘤、保肝、补肾、通便、抗老年痴呆、抗辐射等独特功能,其寄生在梭梭的根端[1-2]。梭梭长期生长在荒漠环境中,形成了抗旱、抗风沙、耐高温、耐寒、耐盐碱和耐贫瘠等特性。因此,梭梭是荒漠化地区主要的防风固沙树种,也可作为骆驼、羊适口性饲料,是荒漠草场中主要的木本饲料树种之一[3]。梭梭具有重要的经济价值、生态价值和饲用价值,是我国的二级保护植物,国内外许多学者已从梭梭抗逆的形态学、解剖学、生理学、生态学及分子生物学等方面进行了相关研究。
梭梭的生存环境极其严酷,其主要分布地区常年干旱少雨,年降水量不足50 mm,相对湿度低于40%,蒸发量高于降水量的30~100倍,生存环境的极端温度最高可达40 ℃以上,最低为-40 ℃以下,地表温度可达70 ℃以上[4]。为了适应极端环境,梭梭逐步形成了自身的极端抗旱机制,但梭梭的抗旱机理、抗旱遗传机制和抗旱相关组学的研究相对较少,前人研究发现,部分转录因子及与干旱胁迫相关,例如,AP2-EREBP转录因子(TINY、CBF1、Ptis、AtEBP、DREB1、DREB2)主要调节植物对低温、干旱及高盐[5]等的分子应答反应;bZIP转录因子在拟南芥基因组、豆科植物基因组、水稻基因组中参与对各种逆境胁迫的响应[6-7];拟南芥中3个NAC基因(ANAC019、ANAC055 和 ANAC072)的过表达能够显著提高植株对干旱的抗性[8];许多NAC基因过表达能够显著提高水稻植株的胁迫抗性,却不会减缓植株的正常生长[9-10];bHLH基因也参与水稻应对干旱胁迫的分子调控过程[11]。在改良植物的抗逆分子育种中,导入或改良转录因子较导入或改良个别功能基因在增强植物抗逆性方面更为有效。因此,本研究主要对干旱胁迫和复水处理下梭梭的转录因子进行分析,挖掘与干旱相关的转录因子,为阐明梭梭抗旱机理奠定理论基础,保护及开发我国抗逆植物基因资源。
1 材料和方法
1.1 材料及处理条件
梭梭种子收集于内蒙古自治区阿拉善左旗。将梭梭种植在塑料花盆中(直径:15 cm,高:18 cm),盆内土壤均匀,湿度及质量均一致。梭梭幼苗在植物培养室种植,室内温度为24/20 ℃(16 h光照 / 8 h 暗处理),相对湿度为30%,光子通量密度为350 μmol/(m2·s)。本研究随机单株种植25盆梭梭幼苗,包括1个正常浇水组(对照组,CK)、干旱胁迫组(土壤相对含水量为30%~45%,LMS;土壤相对含水量为20%~25%,MWS;土壤相对含水量为10%~15%,SWS)和旱后复水处理组(土壤相对含水量为在重度水分胁迫组上增加10%~15%,RSWS)。干旱胁迫组、复水处理组和对照组的种植均设置5个生物学重复,每天在固定时间测质量、控水、补水并记录待幼苗生长90 d之后,分别从CK、LWS、MWS、SWS 以及重度胁迫90 d之后复水处理7 d的RSWS的植株上采集等量的根、茎和叶混合,液氮速冻,置于-80 ℃冰箱中储存备用,提取各处理的总RNA。
1.2 总 RNA 提取
材料总RNA的提取采用 RNAprep Pure Plant Plus KIT(DP441,TIANGEN北京生化科技有限公司)试剂盒。用 DNase酶(2212,大连宝生物工程有限公司)对样品中的 gDNA 进行消化处理。RNA完整性和纯度质量经1.2%非变性琼脂糖凝胶电泳和 Agilent 2100 分析系统检测。
1.3 RNA测序和分析方法
总RNA提取按照文献[12]的方法,利用 Solexa/Illumina进行测序及深圳华大基因公司的Illumina系统软件进行数据分析。RPKM值用于基因表达水平的分析,利用Cluster软件[13],以欧氏距离为距离矩阵计算公式,对基因和试验条件同时进行等级聚类分析,聚类结果用Java Treeview显示,利用WEGO软件对差异基因做GO功能分类统计[14],pathway显著性富集分析以KEGG pathway为单位,应用超几何检验,找出与整个基因组相比后差异表达基因中显著性富集的pathway基因和基因组的代谢途径[15]。
1.4 de novo 拼接及序列分析
用Trinity[16]做转录组 de novo 组装,并通过 reads overlap 关系得到 Contig。然后,将 clean reads 比对回Contig,通过paired-end reads 能确定来自同一转录本的不同 Contig 以及这些 Contig 之间的距离,Trinity 将 Contig 连在一起,最后得到两端不能再延长的序列,即为 Unigene。组装得到的 Unigene,首先使用 Tgicl 将其去冗余并进一步拼接,然后再对这些序列进行同源转录本聚类,最终得到非冗余 Unigene。用 Tophat v2.0.9(Broad Institute)将拼接得到的非冗余 Unigene 与已公布的同种属基因组信息和转录组数据进行比对、注释,用已获取各测序样本中包含的转录因子编码基因信息。
1.5 差异表达基因的筛选
样本中 Unigene 的表达量以公式化校正的 FPKM(Fragments per kb per million fragments)[17] 值衡量。2个样本间差异表达基因(Differential expression genes,DEGs)的筛选及分析使用DEGSeq R package软件,以FDR(False discovery rate)≤0.001,且Log2(fold change,FC)≥1作为筛选阈值进行差异表达基因的筛选。
1.6 差异表达基因 Real-time PCR 验证
从测序结果中随机挑选 9 个转录因子编码基因,用荧光实时定量 PCR 法验证转录组测序数据的有效性和准确性。引物设计用 Primer 5 软件,序列见表1。以18S为内参基因,用2-ΔΔCt计算,并校正目标基因在不同处理下的表达量,每个基因3次生物学重复。10 μL反应体系中包括5 μL SYBR Premix Ex Taq(TaKaRa,TaKaRa Clontech,Japan),5 μmol/L上游引物,5 μmol/L下游引物和20 ng 模板cDNA。所有反应体系加样都在96孔板进行,并在Lightcycle-480(罗氏)荧光定量PCR仪上完成,反应程序:95 ℃ 5 min;后按95 ℃ 15 s,55 ℃ 2 min,72 ℃ 1 min,共40个循环;72 ℃延伸2 min。18S基因为扩增体系的内参基因,扩增产物的表达水平按 2-ΔΔCt 方法计算[18]。每个样品都有3次技术重复,结果都以平均值±标准误(s)(n= 3)计算。
表1 实时定量PCR引物
Tab.1 Selected DEGs of Haloxylon ammodendron and their DNA primer sequences for Real-time PCR
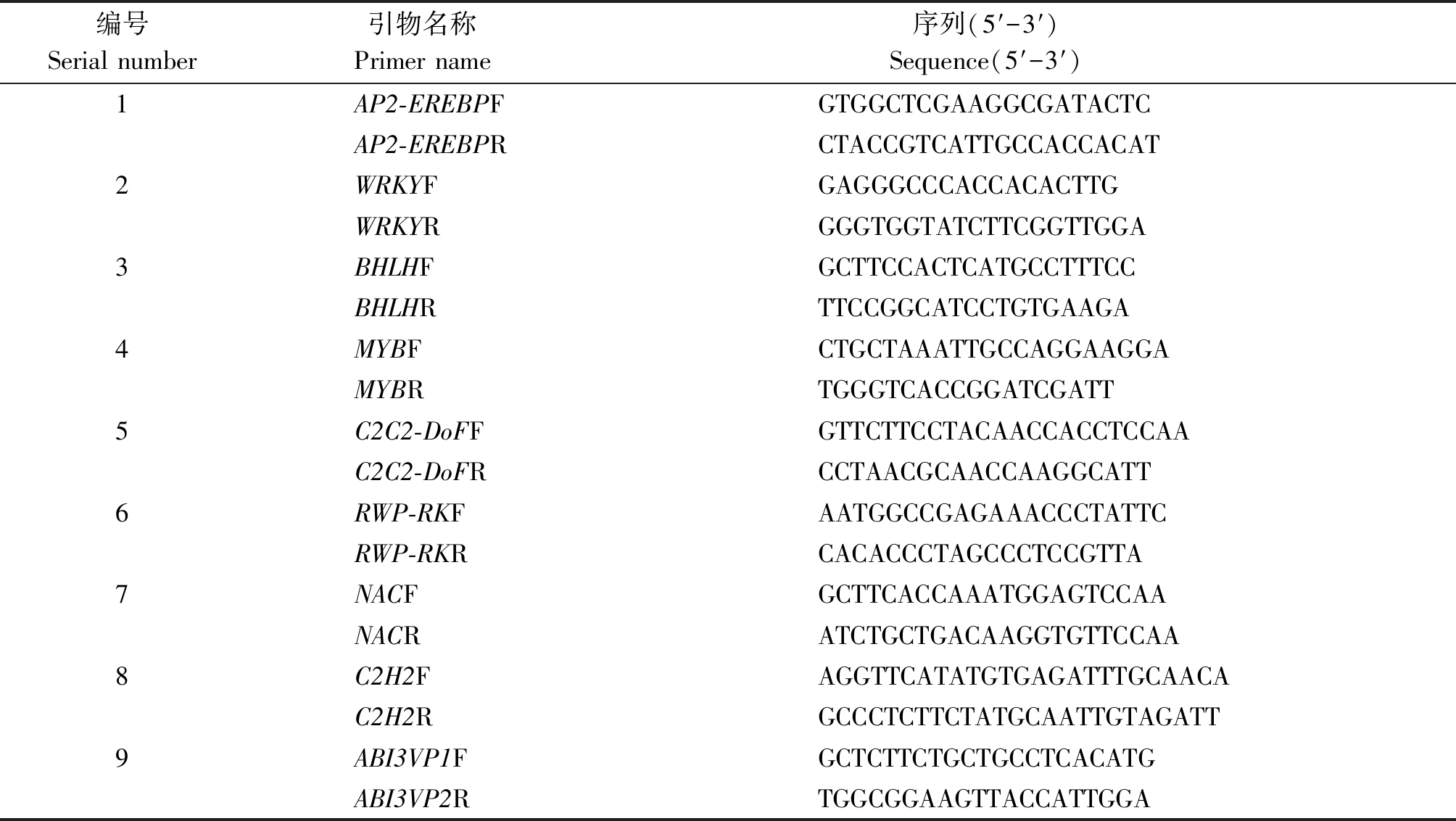
编号Serial number引物名称 Primer name 序列(5′-3′) Sequence(5′-3′) 1AP2-EREBPFGTGGCTCGAAGGCGATACTCAP2-EREBPRCTACCGTCATTGCCACCACAT2WRKYFGAGGGCCCACCACACTTGWRKYRGGGTGGTATCTTCGGTTGGA3BHLHFGCTTCCACTCATGCCTTTCCBHLHRTTCCGGCATCCTGTGAAGA4MYBFCTGCTAAATTGCCAGGAAGGAMYBRTGGGTCACCGGATCGATT5C2C2-DoFFGTTCTTCCTACAACCACCTCCAAC2C2-DoFRCCTAACGCAACCAAGGCATT6RWP-RKFAATGGCCGAGAAACCCTATTCRWP-RKRCACACCCTAGCCCTCCGTTA7NACFGCTTCACCAAATGGAGTCCAANACRATCTGCTGACAAGGTGTTCCAA8C2H2FAGGTTCATATGTGAGATTTGCAACAC2H2RGCCCTCTTCTATGCAATTGTAGATT9ABI3VP1FGCTCTTCTGCTGCCTCACATGABI3VP2RTGGCGGAAGTTACCATTGGA
2 结果与分析
2.1 转录组数据统计分析
以梭梭幼苗为材料,对CK、LWS、MWS、SWS和RSWS处理组进行转录组测序和表达谱分析。对测序数据进行拼接、比对和整理,经de novo 拼接、去冗余、同源转录本聚类后,最终在CK、LWS、MWS、SWS和RSWS 5个组中得到74 641条非冗余Unigene,平均长度780 bp,N50长度1 537 bp。 将CK、LWS、MWS、SWS和RSWS混合样本拼接得到的非冗余Unigene进行比对、注释,共检测到56个转录因子家族,1 307个转录因子编码基因;在CK、LWS、MWS、SWS和RSWS组分别检索到 934,978,994,1 003,991 条转录因子编码基因。LWS、MWS、SWS分别与CK组相比,RSWS与SWS相比,差异表达转录因子均以下调表达方式为主,CK-vs-SWS组中下调转录因子数目最多,达112条,约占该组差异表达转录因子编码基因总数的59.3%(表2)。
表2 差异表达转录因子
Tab.2 Differentially expressed transcription factors
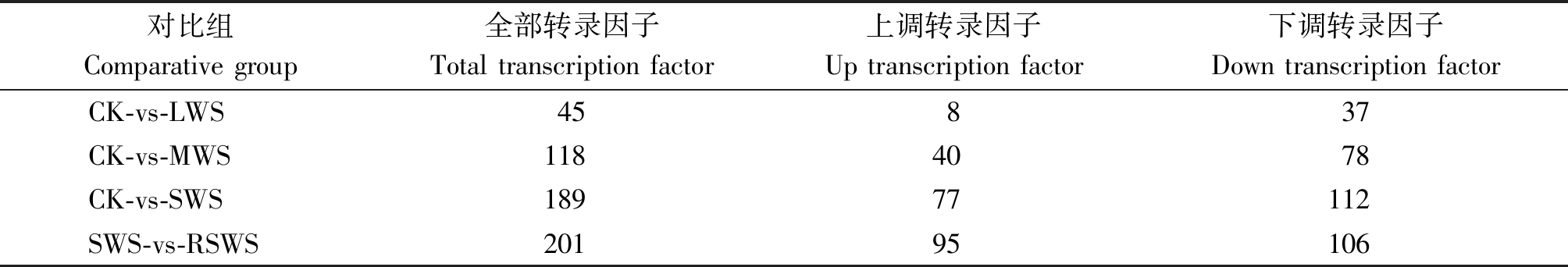
对比组Comparative group全部转录因子Total transcription factor上调转录因子Up transcription factor下调转录因子Down transcription factorCK-vs-LWS45837CK-vs-MWS1184078CK-vs-SWS18977112SWS-vs-RSWS20195106
2.2 差异表达转录因子家族统计分析
获得4个对比组转录因子基本数量信息后,进一步统计分析转录因子家族及其包含成员的数量关系。LWS、MWS、SWS分别与CK组对比,RSWS与SWS相比,表3分析表明,其中,AP2-EREBP、WRKY、bHLH、MYB、NAC和ABI3VP1 家族富集的DEGs 超过该组差异表达基因总数的50%以上,其中AP2-EREBP、WRKY、MYB和bHLH家族转录因子较多。由图1-A-C结果显示,AP2-EREBP家族包含的下调差异表达基因,分别为6个(CK-vs-LWS)、17个(CK-vs-MWS)和 16个(CK-vs-SWS)DEGs,约占各组DEGs总数的75%,81%,59%;WRKY家族包含的下调差异表达基因,分别为6个(CK-vs-LWS)、12个(CKvs-MWS)和 2个(CK-vs-SWS)DEGs,约占各组DEGs总数的100%,100%,33%;bHLH家族包含的下调差异表达基因,分别为5个(CK-vs-LWS)、4个(CK-vs-MWS)和 16 个(CK-vs-SWS)DEGs,约占各组DEGs总数的100%,57%,84%;MYB家族包含的下调差异表达基因,分别为5个(CK-vs-LWS)、9个(CK-vs-MWS)和 10 个(CK-vs-SWS),约占各组DEGs总数的100%,64%,45%。图1-D结果显示,AP2-EREBP家族、NAC家族和WRKY家族包含的上调差异表达基因分别占各自DEGs总数的67%,86%,75%。以上表达规律显示,MYB和WRKY家族基因在LWS组中没有上调表达,但在MWS和SWS中其上调表达数逐渐增加。综合说明,RSWS组中偏向通过增加 AP2-EREBP、NAC家族和WRKY家族编码基因的上调表达数来实现梭梭幼苗对水分刺激的应答调节;而干旱组(LWS、 MWS和 SWS)更偏向于下调AP2-EREBP和bHLH 的DEGs数量来实现其对干旱胁迫的响应。
表3 干旱和复水组各转录因子家族中的基因表达数
Tab.3 Gene expression numbers in each transcription factor family in drought and rehydration groups
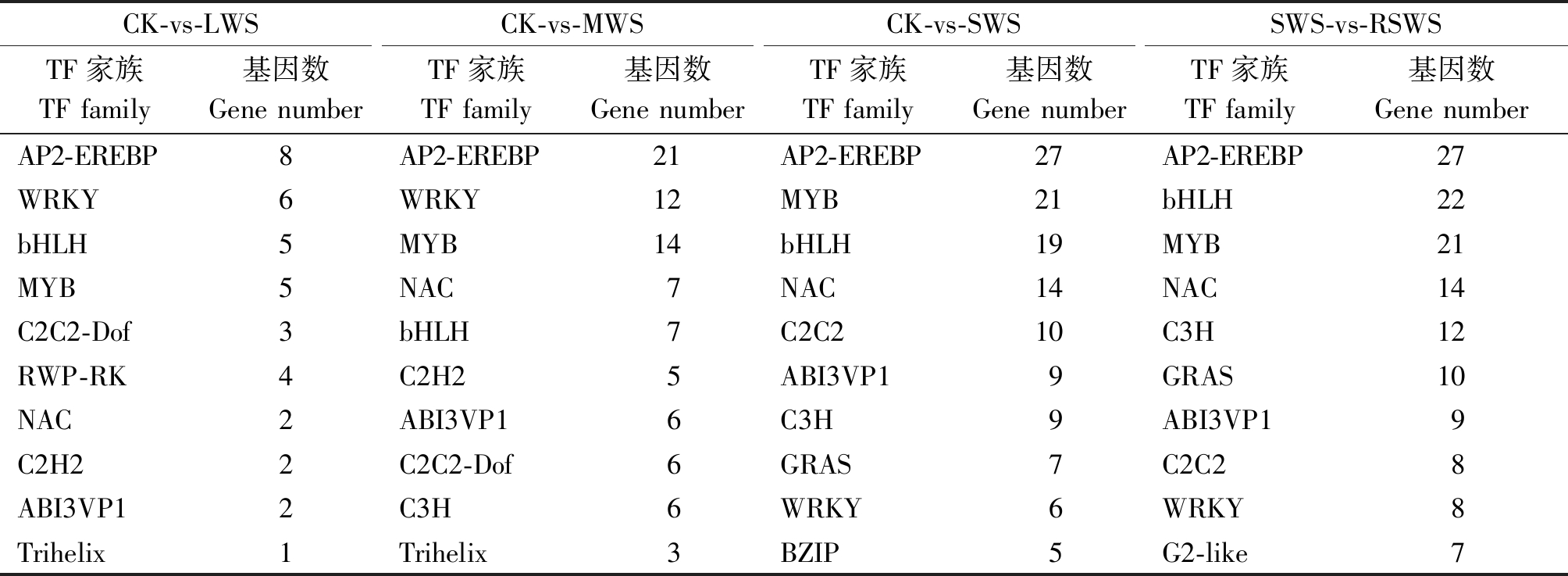
CK-vs-LWSCK-vs-MWSCK-vs-SWSSWS-vs-RSWSTF家族TF family基因数Gene numberTF家族TF family基因数Gene numberTF家族TF family基因数Gene numberTF家族TF family基因数Gene numberAP2-EREBP8AP2-EREBP21AP2-EREBP27AP2-EREBP27WRKY6WRKY12MYB21bHLH22bHLH5MYB14bHLH19MYB21MYB5NAC7NAC14NAC14C2C2-Dof3bHLH7C2C210C3H12RWP-RK4C2H25ABI3VP19GRAS10NAC2ABI3VP16C3H9ABI3VP19C2H22C2C2-Dof6GRAS7C2C28ABI3VP12C3H6WRKY6WRKY8Trihelix1Trihelix3BZIP5G2-like7
2.3 差异表达转录因子表达量分析
从4个对比组中挑选了表达量(FC)变化幅度最大的10个上调和下调基因,并对这些基因的表达规律进行统计分析。结果表明:差异表达基因Tify(CL5087)和NAC(Unigene28108)在干旱胁迫组(LWS、MWS和SWS)和复水组(RSWS)中均上调表达,表达量(FC)变幅为1.23~12.24,2.89~11.37;AP2-EREBP(Unigene2016)、AP2-EREBP(Unigene442)和AP2-EREBP(Unigene160)在干旱胁迫组(LWS、 MWS和SWS)中下调,在复水组(RSWS)中又上调表达。表达量变幅为-4.65~12.22,-8.31~7.38,-6.99~6.85。bHLH(CL2529)和E2F-DP(CL4870)在干旱胁迫组(LWS、 MWS和SWS)中下调表达,变化幅度分别为-14.17~-3.73,-11.89~-4.56;在复水组中却不表达。RWP-RK(CL8167)、DBP(CL6537),在干旱胁迫组(LWS、MWS和SWS)和复水组(RSWS)中均上调表达。BZIP(Unigene23554)在干旱胁迫组(LWS、 MWS和SWS)和复水组(RSWS)中均下调表达。综合统计数据结果显示,AP2-EREBP、NAC、E2F-DP、bHLH 和BZIP等5类转录因子对干旱和水分刺激应答显著(表 4)。
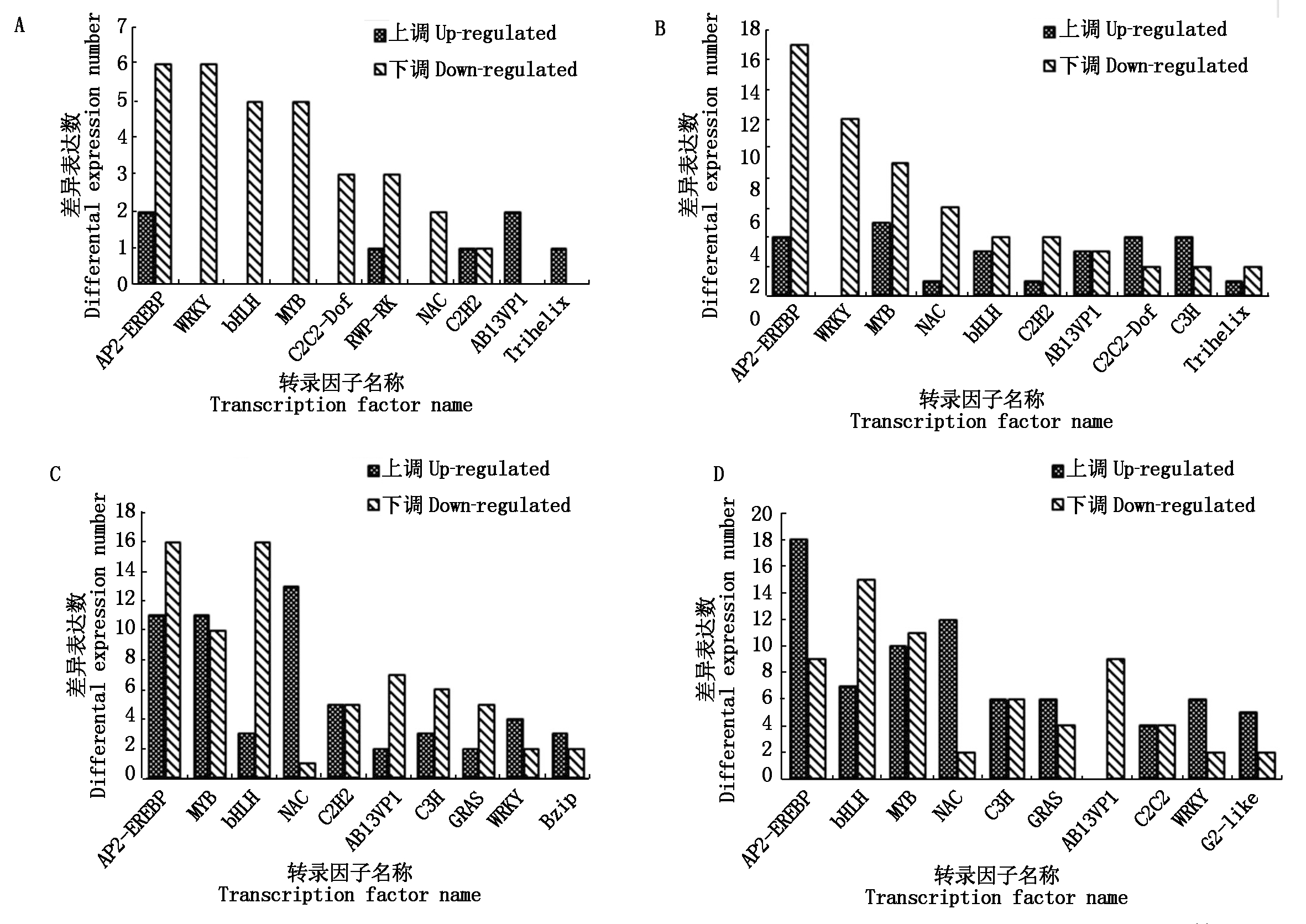
A.CK-vs-LWS的差异表达基因; B.CK-vs-MWS的差异表达基因;C.CK-vs-SWS的差异表达基因; D.RSWS-vs-SWS的差异表达基因。
A.DEGs between CK and LWS;B.DEGs between CK and MWS;C.DEGs between CK and SWS; D.DEGs between SWS and RSWS.
图1 不同比较组间差异表达基因的统计结果
Fig.1 Statistical result of DEGs in different comparable group
表4 对比组差异表达转录因子表达量分析
Tab.4 Analysis of differentially expressed transcription factors in the different comparison group
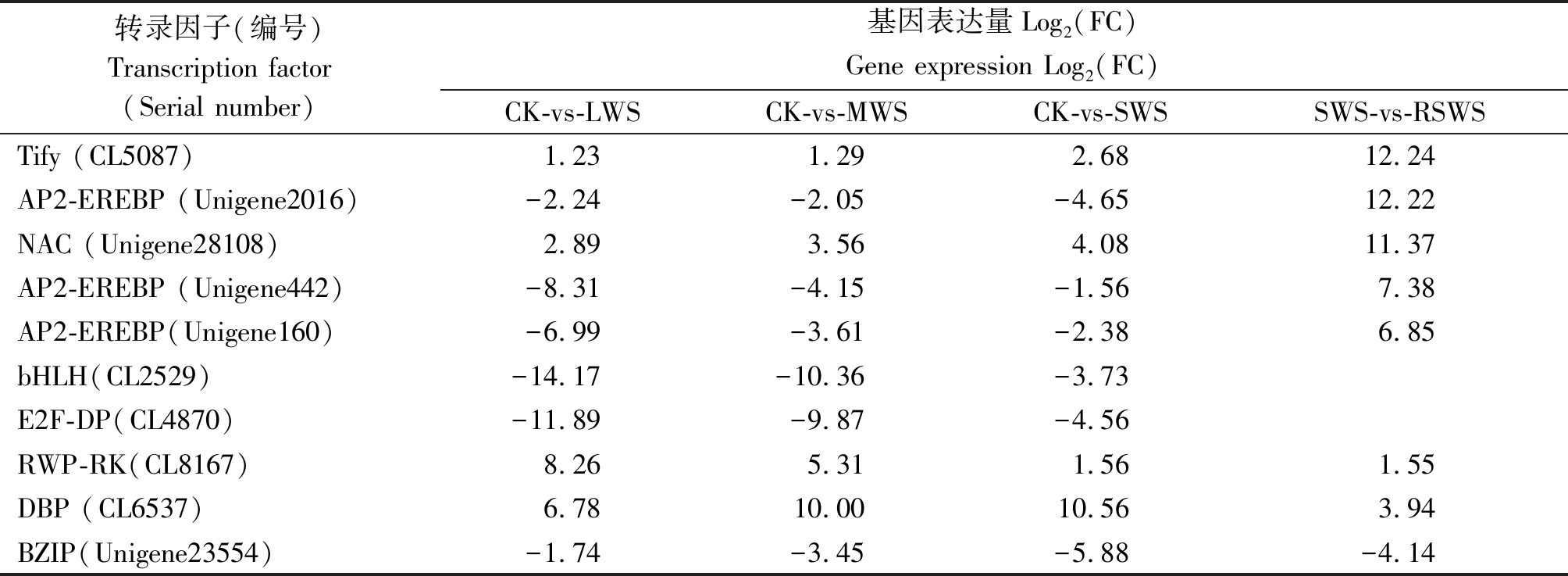
转录因子(编号)Transcription factor(Serial number)基因表达量Log2(FC)Gene expression Log2(FC)CK-vs-LWSCK-vs-MWSCK-vs-SWSSWS-vs-RSWSTify (CL5087)1.231.292.6812.24AP2-EREBP (Unigene2016)-2.24-2.05-4.6512.22NAC (Unigene28108)2.893.564.0811.37AP2-EREBP (Unigene442) -8.31-4.15-1.567.38AP2-EREBP(Unigene160) -6.99-3.61-2.386.85bHLH(CL2529)-14.17-10.36-3.73E2F-DP(CL4870)-11.89-9.87-4.56RWP-RK(CL8167)8.265.311.561.55DBP (CL6537)6.7810.0010.563.94BZIP(Unigene23554)-1.74-3.45-5.88-4.14
注:Log2(FC)≥1时,基因表达显著。
Note:Gene expression is significant when Log2(FC)≥1.
2.4 Real-time PCR 验证
从测序数据中随机选取了9个转录因子编码基因,用Real-time PCR 方法验证测序数据的有效性。图2显示,干旱和水分刺激应答相关的AP2-EREBP、WRKY、bHLH、MYB、C2C2-Dof、RWP-RK、NAC、C2H2和ABI3VP1编码基因在处理前后表达量变化均极显著。干旱胁迫组和复水组测序数据与用荧光定量PCR 法得到的基因表达数据之间的相关性分别为0.947 8(P<0.01),0.967 2(P<0.01),说明干旱胁迫和复水处理后产生的差异表达基因数据有效。
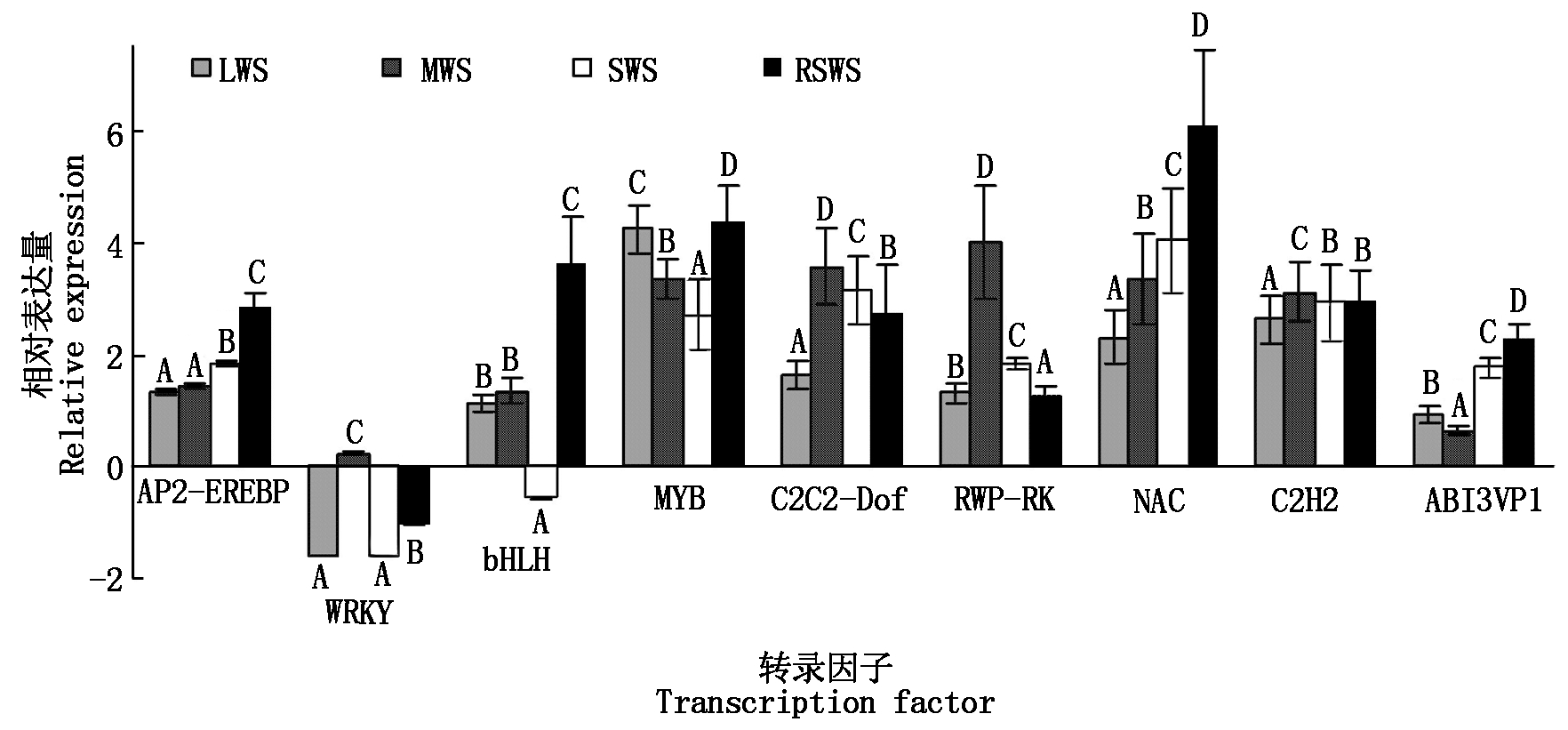
均值±s,样本均为3个重复;不同大写字母表示统计结果在1%水平上有显著差异。
Means±s,n=3;Bars with different letters are significantly different at 1% level.
图2 梭梭转录因子在不同干旱胁迫组和复水组中的实时表达情况
Fig.2 Real-time PCR analysis on DEGs of H. ammodendron in response to drought stress and water stimulation
3 讨论与结论
本研究通过对干旱胁迫和复水处理下梭梭转录因子应答的分子机理进行研究,发掘其中的关键转录因子,对培育抗旱能力强、水分利用效率高的新作物品种具有重要的意义。研究表明,转录因子(Transcription factors,TFs)受胁迫刺激诱导产生,并将信号传递、放大,能够通过调控靶基因转录效率从多个层面降低胁迫对植物的伤害,对植物在逆境下的生长发育起到了至关重要的调节作用,尤其是AP2-EREBP、 E2F/DP、MYB、NAC、WRKY、bHLH和BZIP等家族,是信号转导通路上的关键调控因子,对干旱胁迫响应和水分刺激应答发挥了重要的调节作用[19-23]。本试验测序结果中,各对比组中 AP2-EREBP、MYB、bHLH、NAC、WRKY和ABI3VP1等转录因子编码基因的数量占主要比例,说明这几类转录因子对梭梭干旱胁迫和水分刺激应答调控作用极显著,其中,AP2-EREBP、WRKY、MYB和bHLH是所有56个家族中差异表达转录因子成员最多的蛋白家族。AP2-EREBP转录因子要调节植物对低温、干旱及高盐[23]等的分子应答反应。本研究中 AP2-EREBP是干旱和复水处理下数量丰富、表达量变化最显著的一类转录因子,且AP2-EREBP(Unigene2016)、AP2-EREBP(Unigene442)和AP2-EREBP(Unigene160),在干旱胁迫下显著下调,而在复水组中显著上调,与 bHLH 转录因子类似,干旱胁迫处理下诱导更多的下调ERF转录因子表达,说明这类转录因子发挥调控作用的模式可能与bHLH类似。本研究结果还表明,干旱处理和复水处理后下调的bHLH编码基因数量均大于上调数,推测bHLH编码基因主要通过下调其表达调控机体应答胁迫过程。张子佳等[11]对水稻 22 个bHLH 基因在PEG 和 ABA 胁迫下的表达谱进行了分析,结果表明,其中一些bHLH 基因在响应干旱胁迫过程中受ABA负调控。此外,不同的 bHLH 基因参与应答环境胁迫的调节方式不同,表明参与 PEG 和 ABA 胁迫应答的 bHLH基因具有不同的分子路径或模式。因此,梭梭中bHLH转录因子的进一步研究已在该课题组将继续进行。
Liao等[24]从研究的大豆基因中获得了 156 个 MYB 家族基因,利用酵母单杂交的方法筛选出了40个左右与逆境相关的基因,将这些基因转入拟南芥进行功能验证,发现转 GmMYB177 提高了拟南芥耐旱性。丁震乾等[25]在陆地棉中克隆了 MYB 转录因子基因 GhRAX3,发现其表达在干旱胁迫 0.5 h 后即表现为显著上调,且 48 h 内持续高表达水平;而抑制 GhRAX3的表达,则加快了棉花植株的失水率、细胞质膜过氧化和细胞受损程度,降低了棉花对干旱胁迫的耐受性。目前,发现的大多数 MYB 类转录因子基因均表现为提高植物抗旱能力,但亦有例外,如 Zhou 等[26]的研究任务,将JcMYB001转入拟南芥后,其过表达反而增加了拟南芥对干旱的敏感性。本研究中的MYB转录因子在干旱胁迫组与对照组相比,其下调差异表达基因较其他转录因子多,MYB家族中上调基因数随着干旱胁迫程度的增加而增加。说明本研究结果也证实了MYB家族的转录因子对干旱胁迫具有正调控和负调控性。
综合以上结果推测,发挥主要调控作用的转录因子家族或成员间可能存在一定的共表达性或相互关联性,各转录因子间协同作用对干旱和水分刺激做出精细应答调控。植物对干旱胁迫和水分刺激应答过程受众多分子和细胞通路的调控[27]。转录因子能特异与顺式作用元件结合激活或抑制下游靶基因表达,参与胁迫信号传递网络,其“阀门作用”的重要性不言而喻。本研究借助高通量测序技术,从整体层面概要性的分析了干旱和干旱后复水处理下梭梭幼苗转录因子种类及初步的表达规律,结果将为干旱胁迫应答调控网络、有目的地筛选关键调控基因和分子设计育种等研究提供基础理论数据。
[1] Mckee E D.世界沙海的研究[M].赵兴梁,译.银川:宁夏人民出版社,1993:440-441.
Mckee E D.A study of global sand seas[M].Zhao Xingliang,translated.Yinchuan:Ningxia People′ Publishing House,1993:440-441.
[2] 赵小军,刘宇.梭梭的生态价值与经济价值[J].内蒙古林业,2003(3): 39-45.
Zhao X J,Liu Y. The value of ecological and economic in Haloxylon ammodendron[J].Journal of Inner Mongolia Forestry,2003(3): 39-45.
[3] 姚云峰,王林和,姚洪林.沙漠学[M].呼和浩特:内蒙古人民出版社,1998: 263-264.
Yao Y F,Wang L H,Yao H L.Eremology[M].Hohhot:Inner Mongolia People Publishing House,1998: 263-264.
[4] 何江峰,房永雨,赵小庆,刘红葵.梭梭的研究进展[J].畜牧与饲料科学,2015,36(12):94-98. doi:10.3969/j.issn.1672-5190.2015.12.035.
He J F,Fang Y Y,Zhao X Q,Liu H K. Research progress on Haloxylon[J].Animal Husbandry and Feed Science,2015,36(12):94-98.
[5] Liu Q, Kasuga M, Sakuma Y, Abe H, Miura S, Yamaguchi-Shinozaki K, Shinozaki K. Two transcription factors,DREB1 and DREB2,with an EREBP/AP2 DNA-binding domain separate two cellular signal transduction pathways in drought-and low-temperature-responsive gene expression in Arabidopsis[J]. Plant Cell,1998,10(8):1391-1406. doi:10.1105/tpc.10.8.1391.
[6] Rodriguez-Uribe L,O′onnell M A. A root-specific bZIP transcription factor is responsive to water deficit stress in tepary bean(Phaseolus acutifolius)and common bean(P. vulgaris)[J]. Journal of Experimental Botany,2006,57(6):1391-1398. doi:10.1093/jxb/erj118.
[7] Hossain M A,Lee Y,Cho J I,Ahn C H,Lee S K,Jeon J S,Kang H,Lee C H,An G,Park P B. The bZIP transcription factor OsABF1 is an ABA responsive element binding factor that enhances abiotic stress signaling in rice[J].Plant Molecular Biology,2010,72(4-5):557-566.doi:10.1007/s11103-009-9592-9.
[8] Tran L S P,Nakashima K,Sakuma Y,Simpson S D,Fujita Y,Maruyama K,Fujita M,Seki M,Shinozaki K,Yamaguchi-Shinozaki K. Isolation and functional analysis of Arabidopsis Stress-Inducible NAC transcription factors that bind to a Drought-Responsive cis-Element in the early responsive to dehydration stress 1 Promoter[J]. The Plant Cell,2004,16(9): 2481-2498.doi:10.1105/tpc.104.022699.
[9] Liu Q L,Xu K D,Zhao L J,Pan Y Z,Jiang B B,Zhang H Q,Liu G L. Overexpression of a novel Chrysanthemum NAC transcription factor gene enhances salt tolerance in tobacco[J].Biotechnology Letters,2011,33(10):2073-2082.doi:10.1007/s10529-011-0659-8.
[10] Takasaki H,Maruyama K,Kidokoro S,Ito Y,Fujita Y,Shinozaki K,Yamaguchi-Shinozaki K,Nakashima K. The abiotic stress-responsive NAC-type transcription factor NAC5 regulates stress-inducible genes and stress tolerance in rice[J].Molecular Genetics and Genomics,2010,284:173-183.doi:10.1007/s00438-010-0557-0.
[11] 张子佳,王迪,傅彬英.水稻转录因子bHLH家族基因响应环境胁迫表达谱分析[J].分子植物育种,2008,6(3):425-431.doi:10.3969/j.issn.1672-416X-2008-03-003.
Zhang Z J,Wang D,Fu B Y. Expression patterns bHLH genes responsive to environmental stresses[J].Molecular Plant Breeding,2008,6(3):425-431.
[12] He J F,Goyal R,Laroche A,Zhao M G,Lu Z X.Effects of salinity stress on starch morphology,composition and thermal properties during grain development in Triticale[J]. Canadian Journal of Plant Science,2013,93(5):765-771. doi:10.4141/cjps2013-065.
[13] De Hoon M J L,Imoto S,Nolan J,Miyano S. Open source clustering software[J]. Bioinformatics,2004,20(9):1453-1454. doi:10.1093/bioinformatics/bth078.
[14] Ye J, Fang L, Zheng H K, Zhang Y, Chen J, Zhang Z J, Wang J, Li S T, Li R Q, Bolund L, Wang J. WEGO: a web tool for plotting GO annotations[J]. Nucleic Acids Research,2006,34(2):293-297.doi:10.1093/nar/gkl031.
[15] Kanehisa M, Araki M, Goto S, Hattori M, Hirakawa M,Itoh M, Katayama T,Kawashima S, Okuda S,Tokimatsu T, Yamanishi Y. KEGG for linking genomes to life and the environment[J]. Nucleic Acids Research,2007,36(1):480-484. doi:10.1093/nar/gkm882.
[16] Garber M,Grabherr,M G,Guttman M,Trapnell C. Computational methods for transcriptome annotation and quantification using RNA-seq[J].Nature Methods,2011,8(6):469-477. doi:10.1038/nmeth.1613.
[17] Mortazavi A,Williams B A,McCue K,Schaeffer L,Wold B. Mapping and quantifying mammalian transcriptomes by RNA-Seq[J]. Nature Methods,2008,5(7):621-628.doi:10.1038/nmeth.1226.
[18] Livak K J,Schmittgen T D. Analysis of relative gene expression data using Real-time quantitative PCR and the 2-ΔΔCTMethod[J]. Methods,2001,25(4):402-408. doi:10.1006/meth.2001.1262.
[19] Seki M, Narusaka M, Ishida J,Nanjo T,Fujita M,Oono Y,Kamiya A, Nakajima M,Enju A,Sakurai T,Satou M, Akiyama K,Taji T,Yamaguchi-Shinozaki K, Carninci P, Kawai J, Hayashizaki Y, Shinozaki K. Monitoring the expression profiles of 7000 Arabidopsis genes under drought,cold and high-salinity stresses using a full length cDNA microarray[J]. The Plant Journal,2002,31(3): 279-292. doi:10.1046/j.1365-313X.2002.01359.x.
[20] Jeong J S, Kim Y S, Baek K H, Jung H, Ha S H,Choi Y D, Kim M,Reuzeau C, Kim J K.Root-specific expression of OsNAC10 improves drought tolerance and grain yield in rice under field drought conditions[J]. Plant Physiology,2010,153(1):185-197.doi:10.2307/25680838.
[21] Takasaki H,Maruyama K,Kidokoro S,Ito Y,Fujita Y,Shinozaki K, Yamaguchi-Shinozaki K, Nakashima K. The abiotic stress-responsive NAC-type transcription factor OsNAC5 regulates stress-inducible genes and stress tolerance in rice[J].Molecular Genetics and Genomics,2010,284: 173-183. doi:10.1007/s00438-010-0557-0.
[22] Hao Y J,Wei W,Song Q X,Chen H W,Zhang Y Q,Wang F,Zou H F,Lei G,Tian A G,Zhang W K,Ma B,Zhang J S,Chen S Y. Soybean NAC transcription factors promote abiotic stress tolerance and lateral root formation in transgenic plants[J].The Plant Journal: for Cell and Molecularbiology,2011,68(2):302-313.doi:10.1111/j.1365-313x.2011.04687.x.
[23] Ying S,Zhang D F,Fu J,Shi Y S,Song Y C,Wang T Y,Li Y. Cloning and characterization of a maize bZIP transcription factor,ZmbZIP72,confers drought and salt tolerance in transgenic Arabidopsis [J].Planta,2012,235(2):253-266. doi:10.1007/s00425-011-1496-7.
[24] Liao Y, Zou H F, Wang H W, Zhang W K,Ma B,Zhang J S,Chen S Y.Soybean GmMYB76, GmMYB92,and GmMYB177 genes confer stress tolerance in transgenic Arabidopsis plants[J].Cell Research,2008,18(10):1047-1060. doi:10.1038/cr.2008.280.
[25] 丁震乾,陈天子,刘廷利,刘小双,张保龙,周兴根.棉花干旱诱导MYB类转录因子GhRAX3 的功能分析[J].中国农业科学,2015,48(18):3569-3579.doi: 10.3864/j.issn.0578-1752.2015.18.001.
Ding Z Q,Chen T Z,Liu T L,Liu X S,Zhang B L,Zhou X G. Function analysis of a drought stress induced myb transcription factor GHRAX3 in cotton[J].Scientia Agricultura Sinica,2015,48(18):3569-3579:10.3864/j.issn.0578-1752.2015.18.001.
[26] Zhou C P,Chen Y B,Wu Z Y, Lu W J, Han J L, Wu P Z, Chen Y P, Li M R, Jiang H W, Wu G J. Genome-wide analysis of the MYB gene family in physicnut(Jatropha curcas L.)[J].Gene,2015,572(1):63-71.doi:10.1016/j.gene.2015.06.072.
[27] 朱长保,刘志,徐辰峰,刘仁梅,林中正,成立瑞.全基因组分析盐胁迫下水稻转录因子的表达[J].北方农业学报,2018,46(6):38-43.doi: 10.3969/j.issn.2096-1197.2018.06.08.
Zhu C B,Liu Z,Xu C F,Liu R M,Lin Z Z,Cheng L R.Global genome expression analysis of transcription factors in rice under salt stress[J].Journal of Northern Agriculture,2018,46(6):38-43.