牦牛(Bos grunniens)是分布于青藏高原及其毗邻的高山、亚高山地区的特有且珍贵畜种,能适应高寒、低氧、强紫外线等高原恶劣生态环境以生存和繁衍后代,能充分利用高寒牧草资源为高原牧民提供肉、乳、毛、皮、役用等生产生活资料[1],是当地牧民主要经济来源。因此,开展牦牛遗传资源保护与利用具有重要的科学和经济价值,研究其生长发育及其调控机理对充分发挥牦牛品种优良特性和促进牧区畜牧产业发展具有重要意义。
α肌动蛋白1(Actin alpha 1,ACTA1)是肌动蛋白的一类异构形式,是构成骨骼肌收缩蛋白的重要成分,广泛参与肌细丝组装、骨骼肌纤维发育以及细胞和细胞器的运动等生理活动[2]。研究表明,ACTA1基因突变与人类多种肌肉疾病密切相关,如骨骼肌先天性肌病、横纹肌疾病、杆状体肌病与盖肌病等[3-7]。近年来,国内外许多研究也对畜禽ACTA1基因进行了报道。Swery等[8]研究发现,ACTA1基因突变与斑马的肌无力相关。Sibut等[9]研究表明,12月龄韩牛背最长肌中ACTA1基因表达水平显著高于27月龄韩牛,ACTA1基因可能是调控韩牛肉质性状的重要基因。Shin等[10]研究显示,ACTA1基因在韩牛低等级大理石花纹牛肉中表达量显著高于高等级大理石花纹个体,ACTA1基因可能是影响肉质性状的候选基因。目前,有关ACTA1基因调控牦牛肉质性状的分子机制尚未见报道。
DNA甲基化即在DNA甲基化转移酶(DNA methyltransferases,DNMTs)作用下,将S-腺苷甲硫氨酸(S-adenosylmethionine,SAM)提供的甲基转移到第5位胞嘧啶,而形成5-甲基胞嘧啶(5-mC)。发生DNA甲基化修饰的主要位点是CpG二核苷酸序列的胞嘧啶上[11]。在基因核苷酸序列不发生改变的情况下,基因表达却能发生可遗传的变化[12]。DNA甲基化与生物体内重要基因表达调节密切相关,目前相关研究表明,DNA甲基化与基因的表达呈负相关,启动子区和结构基因的高甲基化与基因的表达存在一定的负相关,而启动子区低甲基化与转录活性正相关[13]。经证实,DNA甲基化在家畜生长发育过程中主要参与细胞新陈代谢活动以及众多生长代谢和调控过程,与家畜肌肉生长发育和脂肪沉积等经济性状密切相关[14]。
本试验以类乌齐牦牛、麦洼牦牛为研究对象,采用实时荧光定量PCR(qRT-PCR)技术检测ACTA1基因在这2个牦牛群体各组织的表达差异;利用重亚硫酸氢盐测序法(Bisulfite-sequencing PCR,BSP)检测ACTA1基因在各组织的甲基化状态,从表观遗传学角度分析ACTA1基因与牦牛肉质性状的相关性,旨在为进一步揭示ACTA1基因对牦牛生长发育、肌肉发育等的调控作用,并为建立有效可靠的分子标记等提供理论依据。
1 材料和方法
1.1 试验动物与样品采集
于西藏自治区昌都市类乌齐县和四川省红原县分别选取3头4.5岁类乌齐牦牛和麦洼牦牛,分别采集臀大肌、心脏、肾脏、肺脏、肝脏、脂肪6种组织,DEPC水清洗干净,锡箔纸包装后迅速置于液氮中保存,用于提取基因组DNA和总RNA。
1.2 主要试剂
TRIzol RNA提取试剂盒(Thermo);血液/细胞/组织基因组DNA提取试剂盒TIANamp Genomic DNA Kit(TIANGEN);DNA纯化回收试剂盒(TIANGEN);反转录试剂盒RevertAid First Strand cDNA Synthesis(Thermo Scientific);琼脂糖BIOWEST REGULAR AGAROSE G-10(GENE COMPANYLTD);DL2000 DNA Ladder(TaKaRa);Loading Buffer(TaKaRa);pMD19-T载体(TaKaRa);DH5α大肠杆菌感受态细胞(TIANGEN);质粒DNA提取试剂盒Plasmid Miniprep Plus Purification(GeneMark);DNA甲基化处理试剂盒ZYMO EZ DNA Methylation-GoldTM Kit(The Epigenetics companyTM);Dream TaqTM Green PCR Master Mix(2X)(Thermo Scientific);Zymo TaqTM Premix(Epigenetics companyTM);RNase-Free ddH2O(Thermo Scientific)。
1.3 组织样核酸提取
1.3.1 组织总RNA提取及cDNA合成 利用TRIzol法提取组织总RNA,紫外分光光度计测定RNA样品质量和浓度(吸光度OD260/280),通过1.5%琼脂糖凝胶电泳检验其完整性,置于-80 ℃保存备用。将提取的总RNA稀释成200 ng/μL,分别取5 μL,反转录获得cDNA,-20 ℃保存备用。
1.3.2 基因组DNA提取与重亚硫酸氢盐处理 利用DNA提取试剂盒提取麦洼牦牛、类乌齐牦牛各组织基因组DNA,紫外分光光度计测定DNA样品的质量和浓度(吸光度OD260/280),通过1.5%琼脂糖凝胶电泳检测后于-80 ℃保存备用。严格按照DNA甲基化试剂盒说明书对基因组DNA 20 μL(DNA质量为200 ~ 500 ng最适宜,不足20 μL应加ddH2O补齐)进行重亚硫酸氢盐处理,-20 ℃保存备用。
1.4 引物设计与合成
根据西南民族大学青藏高原研究院动物遗传育种与繁殖实验室已有的牦牛ACTA1基因CDS区序列设计荧光定量引物ACTA1-qPCR,以β-actin作为内参基因。根据ACTA1基因序列(GenBank: AC_000185)设计启动子区PCR引物ACTA1-PCR,引物具体信息见表1。引物由Thermo(上海)公司合成。
表1 ACTA1-PCR引物
Tab.1 ACTA1-PCR primers

引物Primers引物序列(5′-3′) Sequences(5′-3′) 退火温度/ ℃Tm片段大小/bpAmplified lengthACTA1-PCRF: TTCGTGCGTGCCGAGGTAGAT63.01 028R: GCGGTTAGCCTTGGGGTTGAGACTA1-qPCRF: CGTGGCTACTCCTTCGTG60.0152R: TGACCGTCGGGCAACT Cβ-actinF: TGATGATATTGCTGCGCTCG60.0153R: TACGAGTCCTTCTGGCCCATACTA1-PCRF: GTTTTGAGTTTTTAGATGGAGAAA58.5273R: ACAAAAACAAAAATACCATAAAAAC
1.5 ACTA1基因启动子区克隆、序列分析
以基因组DNA为模板,以ACTA1-PCR为引物进行ACTA1基因启动子区扩增(25 μL体系)。凝胶电泳检测扩增结果,试剂盒纯化PCR阳性扩增产物。取1 μL胶回收产物与PUC载体pMD-19T于16 ℃过夜连接,然后转化至DH5α感受态细胞,并涂布于Ampr/LB固体培养基平板;挑取单个菌落接种于含Ampr/LB液体培养基中。经PCR鉴定后,将阳性重组质粒送擎科生物技术公司(成都)测序。
启动子区克隆测序结果经NCBI-Blast比对后,从NCBI数据库搜集12个物种ACTA1基因启动子区序列,利用MEGA 7.0软件构建不同物种间ACTA1基因启动子区核苷酸序列进化树。在线软件Methprimer(http://www.urogene.org/cgi-bin/methprimer/methprimer.cgi预测CPG岛(图1)。选择长度为230 bp的 CpG1(-202~-431 bp、包含16个CG位点)作为研究对象,并筛选ACTA1基因BSP引物ACTA1-P1(图2)。如图2所示,第一排为目的基因原序列,第二排为重亚硫酸氢盐转化后的序列,除CG位点外的C均转化为T,P1-F/R为BSP引物。
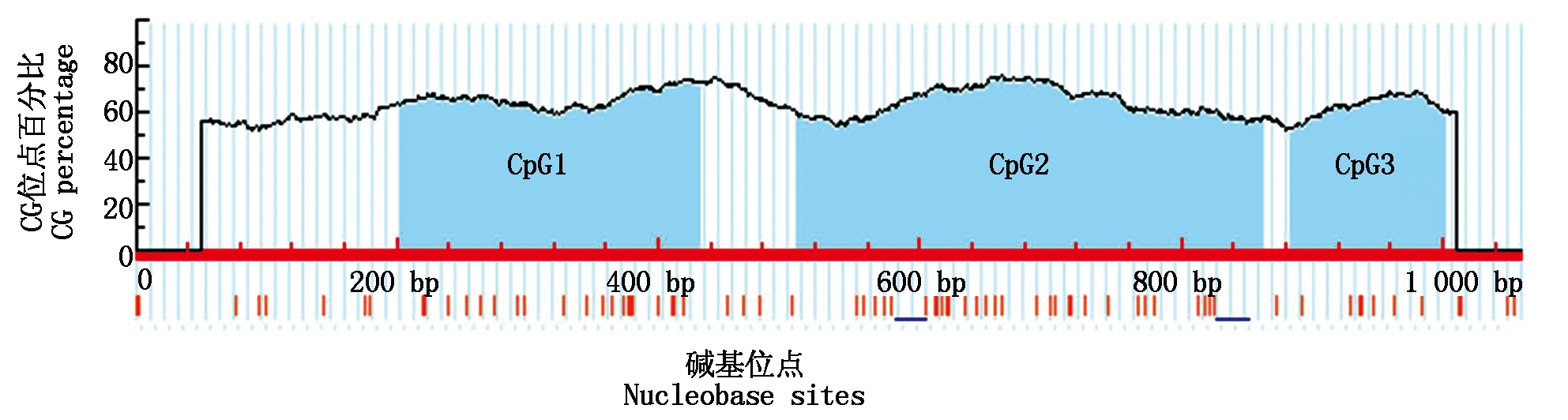
图1 ACTA1基因启动子区CpG岛预测
Fig.1 The CpG island prediction of ACTA1 gene promoter region
1.6 重亚硫酸盐测序及分析
取重亚硫酸盐处理后的DNA作为模板进行BSP反应,PCR反应体系(10 μL):Zymo TaqTM Premix 25 μL,上、下游引物各1 μL(10 μmol/L),模板4 μL,ddH2O 19 μL。反应程序:94 ℃ 10 min;94 ℃ 30 s,最佳Tm 30 s,72 ℃ 30 s,35个循环;72 ℃ 7 min。将PCR产物连接到载体PMD19-T,并转化至DH5α感受态细胞,从平板上挑取单克隆进行菌液PCR鉴定,每个转化板挑取至少10个阳性克隆,送擎科(成都)生物技术公司测序。使用Bio Analyzer 软件分析测序结果;利用在线软件MSR calculate(http://www.msrcall.com/ MSRcalcalate.aspx)绘制ACTA1基因甲基化位点棒棒糖模型。

图2 ACTA1基因启动子区序列及BSP扩增区域示意图
Fig.2 ACTA1 gene promoter region sequence and BSP amplification area schematic
1.7 qRT-PCR定量分析类乌齐牦牛、麦洼牦牛ACTA1 mRNA表达
实时荧光定量 PCR以β-actin为内参基因,对类乌齐牦牛、麦洼牦牛ACTA1基因mRNA在臀大肌、心脏、肝脏、肺脏、肾脏、脂肪6个组织中的表达水平进行定量检测。反应体系(10 μL):SYBR premix Dimer Eraser(2x)5 μL,ddH2O 3.2 μL,上下游引物(10 μmol/L)各 0.4 μL,cDNA 1 μL。反应程序: 95 ℃ 30 s; 95 ℃ 5 s,60 ℃ 30 s,72 ℃ 10 s,39个循环;添加熔解曲线。设置以ddH2O为模板的阴性对照,每个组织进行3个技术重复,使用Bio-Rad 公司荧光定量PCR仪进行定量分析。
1.8 数据分析与处理
采用2-ΔΔCt法对ACTA1基因mRNA表达水平进行相对定量分析,所得数据用SPSS 19.0软件进行分析,ACTA1基因mRNA相对表达丰度采用ANOVA方差分析和Turkey′s LSD检验,所有结果当P<0.05时,具有统计学意义。最后使用GraphPad Prism 5绘制ACTA1基因在类乌齐牦牛、麦洼牦牛不同组织的mRNA表达柱状图;甲基化程度和mRNA相对表达量的所有数据均采用“平均值±标准误差(mean±SEM)”表示。
2 结果与分析
2.1 ACTA1基因序列分析
经克隆及测序获得牦牛ACTA1基因转录起始位点上游及第一外显子区域部分序列,琼脂糖凝胶电泳检测结果如图3-A所示,总长为1 028 bp,经NCBI-Blast分析,与黄牛序列一致性为99.7%,其中A、T、G、C残基含量分别为19.4%(199),17.5%(180),30.9%(318),32.2%(331),A+T的含量(36.9%)小于G+C(63.1%)。以重亚硫酸盐处理后的DNA作为模板进行ACTA1启动子区,CpG1 BSP克隆及测序,琼脂糖凝胶电泳检测结果如图3-B所示。
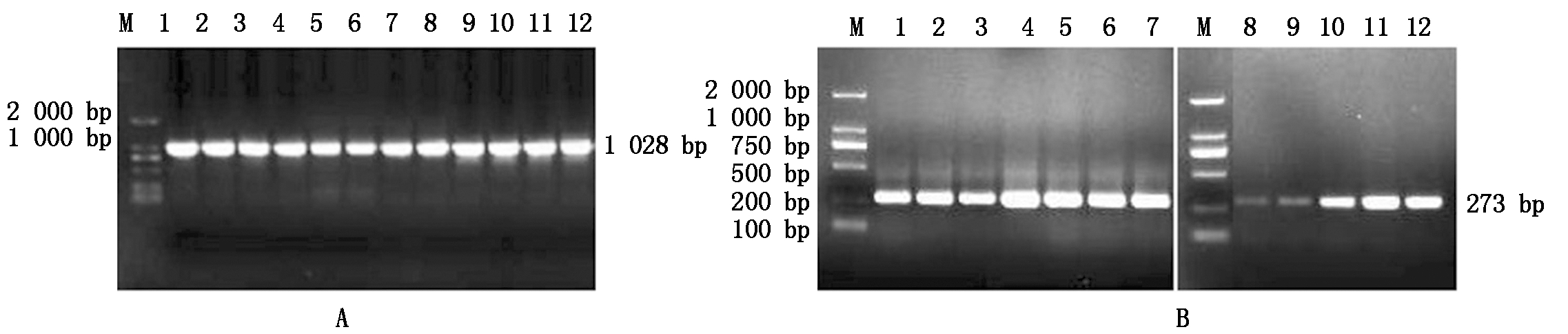
M.DL2000分子质量标记; A(1-12).ACTA1基因启动子PCR扩增; B(1-12).ACTA1基因CpG1 PCR扩增。
M.DL2000 molecular Marker; A(1-12).ACTA1 gene promoter PCR amplification; B(1-12).ACTA1 gene CpG1 PCR amplification.
图3 PCR 扩增结果
Fig.3 Results of PCR-amplified fragments
2.2 系统发育树构建
以NJ法构建ACTA1基因启动子区系统发育树,结果显示,牦牛与黄牛聚为一支,亲缘关系最近;水牛、绵羊、猪及犬各单独占据一支,与牦牛亲缘关系较近;两栖纲的热带爪蟾与牦牛亲缘关系最远,说明哺乳动物ACTA1基因在长期生物进化过程中高度保守(图4)。
2.3 CpG岛甲基化水平分析
利用在线软件MSR Calcalate绘制各组织甲基化状态的棒棒糖模型(图5)。图示每行代表一个阳性克隆测序和比对后的甲基化状态,其中白色圆圈表示未被甲基化的CG位点,黑色圆圈表示被甲基化修饰的CG位点。结果显示,ACTA1启动子区CpG岛在成年类乌齐牦牛和麦洼牦牛各组织中均表现为较低的甲基化状态,且除肺脏组织外,其他各组织甲基化水平无显著差异(P>0.05),类乌齐牦牛臀大肌、心脏、肾脏、肺脏、肝脏和脂肪的甲基化概率分别为6.25%,6.88%,20.00%,16.25%,26.25%,29.38%,麦洼牦牛臀大肌、心脏、肾脏、肺脏、肝脏和脂肪的甲基化概率分别为5.00%,7.50%,18.13%,20.00%,26.25%,28.75%(表2);,其中臀大肌的甲基化水平最低,脂肪组织的甲基化水平最高。统计分析发现,第3,4,11,13位CG位点甲基化频率分别为34.17%,42.50%,35.83%,35.83%,远高于其他CG位点。
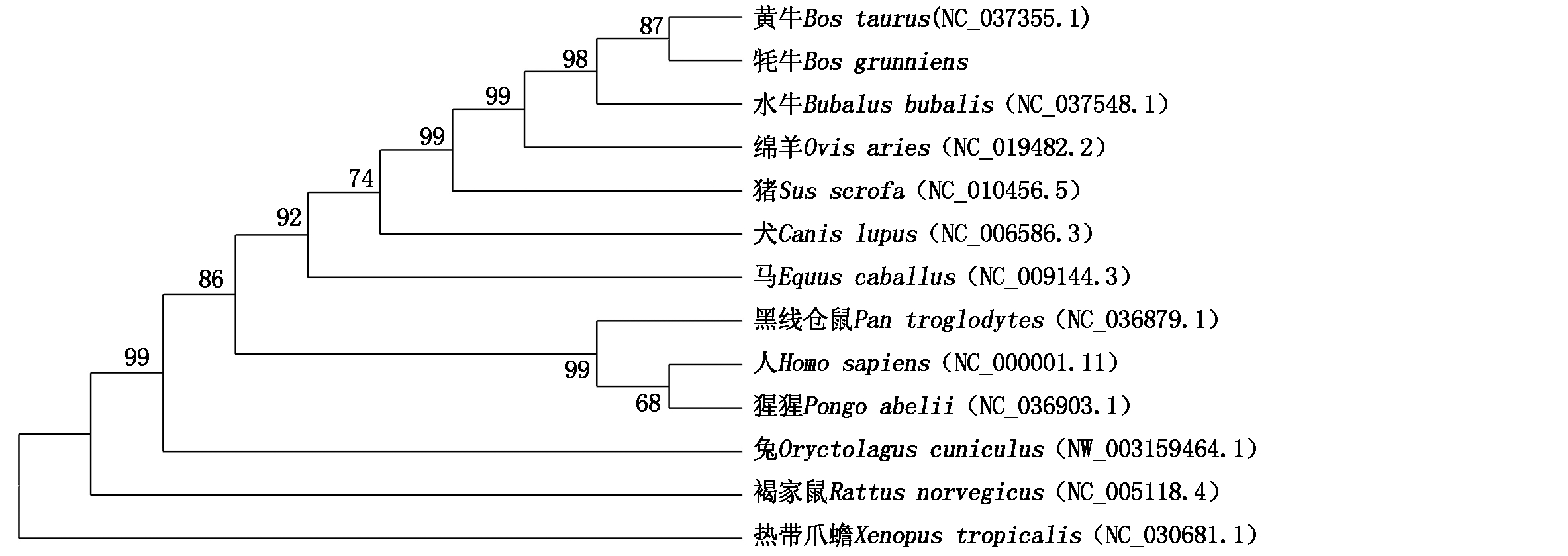
图4 ACTA1基因启动子序列系统发育树
Fig.4 ACTA1 gene promoter sequence phylogenetic tree

A-F.类乌齐牦牛不同组织(臀大肌、心脏、肾脏、肺脏、肝脏、脂肪); G-L.麦洼牦牛不同组织(臀大肌、心脏、肾脏、肺脏、肝脏、脂肪)。
A-F. Different tissues of Leiwuqi yak(gluteus maximus,heart,kidney,lung,liver,adipose); G-L.Different tissue of Maiwa yak(gluteus maximus,heart,kidney,lung,liver,adipose).
图5 ACTA1基因在不同组织甲基化水平
Fig.5 The level of methylation of ACTA1 gene in different tissues
2.4 qRT-PCR定量分析牦牛ACTA1 mRNA表达
荧光定量PCR结果显示,ACTA1基因在类乌齐牦牛、麦洼牦牛各组织中均有表达,表达量存在一定差异。由表2可知,2个群体在臀大肌均呈现最高表达,显著高于其他组织表达量(P<0.05);其次在心脏的表达量较高,显著高于肾脏、肺脏、肝脏、脂肪组织表达量(P<0.05);而在肾脏、肺脏、肝脏、脂肪组织中的表达量依次降低,其中肺脏与肾脏和肝脏差异不显著(P>0.05),但三者均显著高于脂肪(P<0.05)。相对表达量与DNA甲基化相关性分析发现,ACTA1基因各组织表达量与DNA甲基化水平显著负相关(r=-0.797,P=0.002)(表2)。
表2 ACTA1基因在麦洼牦牛和类乌齐牦牛各组织的相对表达量及其CpG岛甲基化程度
Tab.2 Relative expression of ACTA1 gene in Maiwa yak and Leiwuqi yak and their CpG island methylation status
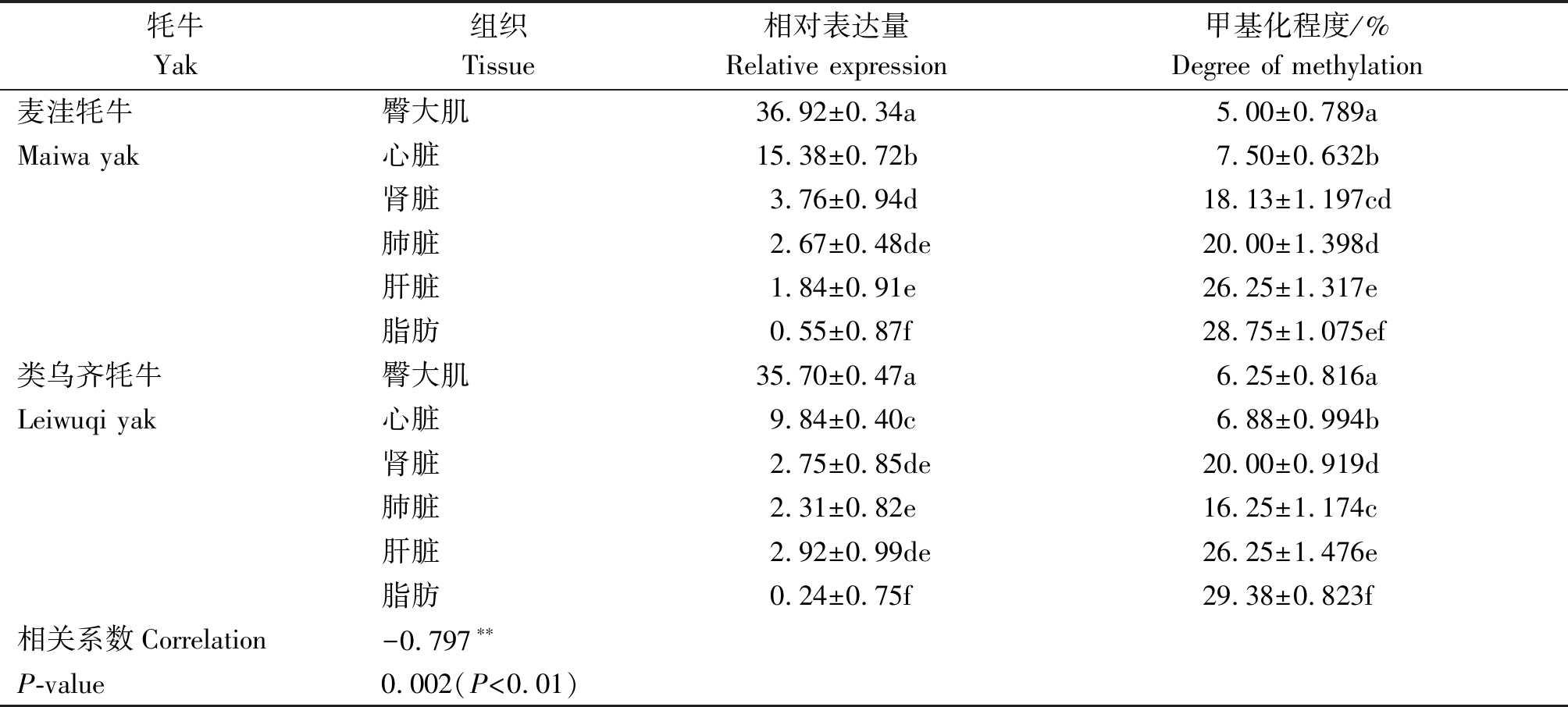
牦牛Yak组织Tissue相对表达量Relative expression甲基化程度/%Degree of methylation麦洼牦牛臀大肌36.92±0.34a5.00±0.789aMaiwa yak心脏15.38±0.72b7.50±0.632b肾脏3.76±0.94d18.13±1.197cd肺脏2.67±0.48de20.00±1.398d肝脏1.84±0.91e26.25±1.317e脂肪0.55±0.87f28.75±1.075ef类乌齐牦牛臀大肌35.70±0.47a6.25±0.816aLeiwuqi yak心脏9.84±0.40c6.88±0.994b肾脏2.75±0.85de20.00±0.919d肺脏2.31±0.82e16.25±1.174c肝脏2.92±0.99de26.25±1.476e脂肪0.24±0.75f29.38±0.823f相关系数Correlation-0.797∗∗P-value0.002(P<0.01)
注:**表示在0.01水平(双侧)上显著相关(P<0.01)。相同字母代表无显著差异(P>0.05);相异字母代表差异显著(P<0.05)。
Note:** Indicates significant correlation at the 0.01 level (both sides) (P<0.01). Identical letters represent no significant differences (P>0.05); Distinct letters represent significant differences (P<0.05).
3 结论与讨论
在动物生长发育过程中,DNA甲基化修饰对基因表达发挥重要作用[15]。最初,DNA甲基化在动物育种工作中的研究主要集中在杂交育种[16-17]、雄性不育[18-20]、胚胎移植[21-22]以及泌乳性能等方面[23],近几年,出现了少量有关DNA甲基化影响肌肉发育、脂肪沉积的研究报道。韩玉娇[24]发现 6月龄秦川牛肌肉和脂肪组织中甲基化水平显著低于24月龄秦川牛肌肉和脂肪组织中甲基化水平(P<0.01),且甲基化水平与 mRNA表达水平呈负相关。房希碧[25]利用全基因组甲基化测序法和转录组测序法联合分析发现,日本和牛与草原红牛背最长肌的147个基因DMRs甲基化水平与基因表达水平呈负相关,其中大部分基因可能通过 DNA 甲基化调控基因的表达水平,从而影响肉质性状。姚力丹等[26]分析5月龄巴什拜羊肌肉组织MSTN基因DNA甲基化和mRNA表达量的关系,发现MSTN基因DNA甲基化与其表达量呈负相关。上述研究均证实了基因启动子区域CpG岛的甲基化程度与该基因的表达水平呈负相关 [13]。DNA甲基化具有组织特异性、时间和空间特异性,即使同一个体在不同时期不同组织DNA甲基化状态可能会相差迥异[27]。本研究在前期克隆了ACTA1基因编码区及启动子区的基础上,检测了各组织ACTA1 mRNA在类乌齐牦牛和麦洼牦牛臀大肌、心脏、肾脏、肺脏、肝脏、脂肪各组织间表达水平差异,并利用重亚硫酸氢盐测序法分析了各组织器官DNA甲基化水平,试图寻找ACTA1基因甲基化模式与mRNA表达量间的联系,为牦牛的表观遗传领域提供一定的数据支持。
相关研究证明,哺乳动物体内的组织特异性基因CpG岛通常呈现较低的甲基化状态,其甲基化的CG位点往往为随机分布,CpG岛区域DNA甲基化修饰程度与mRNA表达量呈负相关[28],本研究结果显示,ACTA1启动子区CpG岛在牦牛的各个组织中甲基化的CG位点随机分布,且均表现为较低的甲基化水平。其中ACTA1基因在脂肪组织DNA甲基化水平最高,肌肉组织最低,其中类乌齐牦牛甲基化水平依次为:臀大肌<心脏<肾脏<肺脏<肝脏<脂肪,而麦洼牦牛甲基化水平依次为:臀大肌<心脏<肺脏<肾脏<肝脏<脂肪。ACTA1基因mRNA表达水平定量分析显示在6个组织中均有表达,其中类乌齐牦牛和麦洼牦牛在臀大肌和心脏组织中的表达量均最高,在脂肪的表达量均最低,其表达量趋势均为:臀大肌>心脏>肾脏(肺脏)>肝脏>脂肪。牦牛常年生活在平均海拔3 500 m以上的高寒草甸草场,饲养方式以自然放牧为主,牦牛善走陡坡险路、雪山沼泽、能渡江河等,其骨骼肌为适应高强度运动及代谢需求,臀大肌等骨骼肌肌纤维含量非常丰富且发达,因此,ACTA1 mRNA表达量极显著高于其他组织器官。此外,牦牛心脏ACTA1 mRNA表达量显著高于其他组织,肌纤维含量丰富,有利于氧气运输及气体交换,在氧含量极低的高原环境也可为机体代谢活动供应充足的氧气。利用SPSS 19.0分析各组织ACTA1 mRNA表达量与DNA甲基化水平相关性极显著性,结果显示,各组织表达量与DNA甲基化水平存在显著负相关(r=-0.796,P<0.01),表明ACTA1基因启动子区甲基化组织特异性较强。各组织DNA甲基化模式在类乌齐牦牛与麦洼牦牛间无显著差异,推测是因为ACTA1基因在长期生物进化过程中高度保守,故其甲基化率在同一物种不同品种间无显著差异,相关推测有待进一步验证。ACTA1基因启动子区CpG岛区域具有一定的转录活性,且各组织间甲基化水平与组织特异性表达存在显著负相关,2个品种间各组织甲基化水平无显著差异,证明启动子区域DNA甲基化对肌肉发育具有一定的调控作用,但影响基因表达的因素很多,可能还会涉及染色质重塑、非编码RNA、蛋白质修饰等其他调控方式,因此,本研究结果仅可在表观遗传领域为牦牛遗传育种提供部分数据支持。
本试验成功克隆了ACTA1基因启动子区并构建了13个物种间系统发育树,发现牦牛与黄牛、羊的亲缘关系最近,证明ACTA1基因在长期生物进化过程中高度保守;对ACTA1基因启动子区CpG1进行DNA甲基化差异分析和mRNA表达水平检测,结果发现,ACTA1基因DNA甲基化与其表达量呈显著负相关,臀大肌甲基化水平低于其他组织,其ACTA1 mRNA表达量显著高于其他组织,脂肪组织与之相反,表明DNA甲基化对肌肉和脂肪发育具有一定的调控作用,可作为牦牛遗传育种的表观遗传标记的参考。
[1] 施奇静,王静,孙军平,薛堃,孟秀祥.中国牦牛资源保护及可持续利用:驯化与品种培育[J].家畜生态学报,2016,37(1): 81-86. doi: 10.3969/j.issn.1673-1182.2016.01.017.
Shi Q J,Wang J,Sun J P,Xue K,Meng X X. Conservation and sustainable utilization of Yak resources of China: domestication and breed[J].Journal of Domestic Animal Ecology,2016,37(1): 81-86.
[2] 朱筱娟,曾宪录,宋朝霞,郝水.细胞核内肌动蛋白及其功能研究进展[J].科学通报,2004,49(11): 1031-1035. doi:10.3321/j.issn:0023-074X.2004.11.003.
Zhu Y J,Zeng X L,Song Z X,Hao S. Research progress on actin and its function in nucleus[J]. Chinese Science Bulletin,2004,49(11): 1031-1035.
[3] Atencia R,Asumendi A,García-Sanz M. Role of cytoskeleton in apoptosis[J].Vitamins and Hormones,2000,58: 267-297. doi:10.1016/S0083-6729(00)58028-5.
[4] Sparrow J C,Nowak K J,Durling H J,Beggs A H,Wallgren-Pettersson C,Romero N,Nonaka I,Laing N G. Muscle disease caused by mutations in the skeletal muscle alpha-actin gene(ACTA1)[J].Neuromuscular Disorders,2003,13(7-8): 519-531. doi:10.1016/S0960-8966(03)00101-9.
[5] Gavin R H. Microtubule-microfilament synergy in the cytoskeleton[J].International Review of Cytology,1997,173: 207-242. doi:10.1016/S0074-7696(08)62478-X.
[6] Kao J C,Liewluck T,Milone M. A novel ACTA1 mutation causing progressive facios capulo peroneal myopathy in an adult[J].Journal of Clinical Neuroscience,2018,53: 261-262. doi: 10.1016/j.jocn.2018.04.044.
[7] Ueda K,Serajee F,Huq A M. A mutation in the ACTA1 gene manifesting nemaline myopathy with central nervous system lesions[J]. Journal of Clinical Neurology,2017,13(3): 300-302. doi: 10.3988/jcn.2017.13.3.300.
[8] Swery C A,Holton J L,Dick D J,Muntoni F,Hanna M G. Zebra body myopathy is caused by a mutation in the skeletal muscle actin gene(ACTA1)[J].Neuromuscul Disord,2015,25(5): 388-391. doi: 10.1016/j.nmd.2015.02.003.
[9] Sibut V,Hennequet-Antier C,Le Bihan-Duval E,Marthey S,Duclos M J,Berri C. Identification of differentially expressed genes in chickens differing in muscle glycogen content and meat quality[J].BMC Genomics,2011,12(1):112. doi: 10.1186/1471-2164-12-112.
[10] Shin S C,Chung E R. Identification of differentially expressed genes between high and low marbling score grades of the Longissimus dorsi muscle in Hanwoo(Korean cattle)[J].Meat Sci,2016,121(5):114-118. doi:10.1016/j.meatsci.2016.05.018.
[11] Guintivano J,Arad M,Gould T D,Payne J L,Kaminsky Z A. Antenatal prediction of postpartum depression with blood DNA methylation biomarkers[J].Molecular Psychiatry,2014,19(5):560-567. doi:10.1038/mp.2013.62.
[12] Dahl C,Guldberg P. DNA methylation analysis techniques[J].Biogerontology,2003,4(4): 233-250. doi: 10.1023/a:1025103319328.
[13] Vaissière T,Sawan C,Herceg Z. Epigenetic interplay between histone modifications and DNA methylation in gene silencing[J]. Mutat Res,2008,659(1-2):40-48.doi:10.1016/j.mrrev.2008.02.004.
[14] Wilkinson M F. Evidence that DNA methylation engenders dynamic gene regulation[J].Proceedings of the National Academy of Sciences of the United States of America,2015,112(17):E2116. doi: 10.1073/pnas.1502195112.
[15] Huang Y Z,Sun J J,Zhang L Z,Li C J,Womack J E,Li Z J,Lan X Y,Lei C Z,Zhang C L,Zhao X,Chen H. Genome-wide DNA methylation profiles and their relationships with mRNA and the microRNA transcriptome in bovine muscle tissue(Bos taurine)[J]. Scientific Reports,2014,4(1): 6546. doi: 10.1038/srep06546.
[16] 邱峥艳,郭将,张永宏,李传民,赵志辉.莱芜猪与大白猪杂交一代甲基化程度差异分析[J].中国兽医学报,2013,33(5): 780-784. doi:10.16303/j.cnki.1005-4545.2013.05.031.
Qiu Z Y,Guo J,Zhang Y H,Li C M,Zhao Z H. Differential analysis of DNA methylation contents in hybirds Yorkshinex Lai wu pig[J].Chinese Journal of Veterinary Science,2013,33(5): 780-784.
[17] Wang S,Pan Z,Zhang Q,Xie Z,Liu H,Li Q. Differential mRNA expression and promoter methylation status of SYCP3 gene in testes of Yaks and Cattle-Yaks[J].Reproduction in Domestic Animals,2012,47(3): 455-462. doi: 10.1111/j.1439-0531.2011.01902.x.
[18] 刘振山,李齐发,张庆波,屈旭光,董丽艳,强巴央宗,谢庄,刘红林.犏牛及其亲本IGF2基因mRNA表达水平、DNA甲基化状态差异分析[J]. 自然科学进展,2009,19(4): 380-385. doi:10.3321/j.issn:1002-008X.2009.04.003.
Liu Z S,Li Q F,Zhang Q B,Qu X G,Dong L Y,Qiang B Y Z,Xie Z,Liu H L. Analysis of mRNA expression level and DNA methylation status of IGF2 gene in yak and its parents[J].Advances in Natural Science,2009,19(4): 380-385.
[19] 潘增祥,刘振山,李隐侠,于莎莉,李明桂,谢庄,李齐发.犏牛及其亲本睾丸组织中印记基因SNRPN DMR甲基化与mRNA表达差异研究[J].中国农业科学,2010,43(22): 4709-4716.doi:10.3864/j.issn.0578-1752.2010.22.019.
Pan Z X,Liu Z S,Li Y X,Yu S L,Li M G,Xie Z,Li Q F. Differences of SNRPN methylation status and Its mRNA expression in testes between Cattle-Yaks and their parents[J].Scientia Agricultura Sinica,2010,43(22): 4709-4716.
[20] 周阳,骆骅,李伯江,贾超,谢庄,赵兴波,钟金城,李齐发.牦牛和犏牛睾丸组织DDX4基因mRNA表达水平与启动子区甲基化[J]. 中国农业科学,2013,46(3): 630-638.doi:10.3864/j.issn.0578-1752.2013.03.020.
Zhou Y,Luo H,Li B J,Jia C,Xie Z,Zhao X B,Zhong J C,Li Q F. mRNA expression level and promoter methylation of DDX4 gene in testis of yak and cattle-yak[J].Scientia Agricultura Sinica,2013,46(3): 630-638.
[21] 陈洁,李冬杰,刘艳琴,张萃,戴蕴平,李世杰,李宁. 体细胞核移植牛肺脏中H19和Xist基因的DNA甲基化状态[J].科学通报,2008,53(11): 1305-1310.
Chen J,Li D J,Liu Y Q,Zhang C,Dai Y P,Li S J,Li N. DNA methylation status of H19 and Xist genes in bovine lungs after somatic cell nuclear transfer[J].Chinese Science Bulletin,2008,53(11): 1305-1310.
[22] Zhou X Y,Liu L L,Jia W C,Pan C Y. Methylation profile of bovine Oct4 gene coding region in relation to three germ layers[J].Journal of Integrative Agriculture,2016,15(3): 618-628. doi: 10.1016/S2095-3119(15)61100-5.
[23] 赵海谕.奶山羊PITX1、PITX2基因的遗传变异、mRNA表达及DNA甲基化修饰对泌乳性能的影响[D].杨凌:西北农林科技大学,2014.
Zhao H Y. Effects of genetic variation,mRNA expression and DNA methylation of PITX1 and PITX2 genes on lactation performance in dairy goats[D]. Yangling: Northwest A&F University,2014.
[24] 韩玉娇.秦川牛LCORL基因的多态性、mRNA表达及启动子区甲基化研究[D]. 杨凌: 西北农林科技大学,2016.
Han Y J. Polymorphism,mRNA expression and promoter methylation of LCORL gene in Qinchuan cattle[D]. Yangling: Northwest A&F University,2016.
[25] 房希碧.基于转录组和全基因组甲基化联合分析的牛肉质性状候选基因的鉴定与功能验证[D].长春:吉林大学,2017.
Fang X B. Identification and functional verification of beef trait candidate genes based on transcriptome and genome-wide methylation analysis [D].Changchun:Jilin University,2017.
[26] 姚力丹,祖玲玲,马晓燕,依明·苏来曼,决肯·阿尼瓦什.新疆巴什拜羊肌肉生长抑制素基因DNA甲基化与mRNA表达量分析[J].中国畜牧兽医,2017,45(10):2987-2993.doi:10.16431/j.cnki.1671-7236.2017.10.022.
Yao L D,Zu L L,Ma X Y,Yi M·Su L M,Jue K·A N W. Analysis of DNA methylation and mRNA expression level of MSTN gene in Xinjiang Bashiby sheep[J].China Animal Husbandry & Veterinary Medicine,2017,45(10): 2987-2993.
[27] 赵海谕,蓝贤勇,雷初朝,陈宏. 表观遗传学: 家畜遗传育种的新挑战[J].家畜生态学报,2014,35(8):1-5.doi: 10.3969/j.issn.1673-1182.2014.08.001.
Zhao H Y,Lan X Y,Lei C C,Chen H. Epigenetics: new challenges in animal genetics and breeding[J].Acta Ecologae Animalis Domastici,2014,35(8): 1-5.
[28] Ichimura T,Watanabe S,Sakamoto Y,Aoto T,Fujita N,Nakao M. Transcriptional repression and heterochromatin formation by MBD1 and MCAF/AM family proteins[J].Journal of Biological Chemistry,2005,280(14):13928-13935.doi: 10.1074/jbc.M413654200.