叶片是植物进行光合作用的主要器官,叶片颜色与光合作用及产量密切相关[1-4],谷子是C4植物的模式研究作物之一,它具有基因组小、二倍体植物、生育期短、抗旱性和耐瘠薄性极强等特点,因此,谷子叶色突变体已成为探究叶绿体发育机制和C4光能利用率的理想材料之一[5-7]。目前,在谷子中公开研究报道的叶色突变体非常少,Luo等[8]鉴定出14个具有异常“花环型”结构的叶色突变体,可用于今后“花环型”结构发育和C4光合作用研究相关基因的定位和克隆。在已经克隆的谷子叶色突变体中,SiYGL1编码Mg-螯合酶的一个D亚基(CHLD)蛋白,该蛋白属于编码叶绿素合成途径关键酶基因之一,该基因突变后叶片表现为黄绿色[9];在叶绿体发育方面,SiSTL1[10]和SiSTL2[11]分别编码核糖核苷酸还原酶(Ribonucleotide reductase,RNR)大亚基蛋白和脱氧胞苷单磷酸脱氨酶(Deoxycytidine monophosphate deaminase,DCD)蛋白,其突变体表现出不同程度的条纹叶[10-11]。
与拟南芥、水稻和玉米等相比,目前,关于谷子叶色突变体及叶绿体相关分子机制方面的进展不能满足对其C4光合和叶绿体发育机制的研究,迫切需要利用更多的叶色突变体并发掘鉴定相关基因及其功能。
条纹叶表型受多个基因调控,其分子生物学机理较为复杂。影响叶绿体生物合成相关基因突变后造成叶片白条纹表型,研究表明,水稻V3和St1基因分别编码核糖核苷酸还原酶大亚基蛋白(RNRL1)和核糖核苷酸还原酶小亚基蛋白(RNRS1),这类基因在三叶期茎基部组织中高表达,即叶绿体发育第一阶段,这2个基因突变后影响质体DNA的合成,阻碍叶绿体分化[12];水稻V1[13]、V2[14-15]和VYL[16]基因分别编码NUS1、pt/mtGK和OsClpP6,该类基因在叶绿体发育的第二阶段高度积累,其突变影响叶绿体遗传机制的建立,阻碍叶绿体的形成,造成水稻白条纹表型[13-16]。WSL[17]、WSL4[18]和WSL5[19]属于三角状五肽(Pentatricopeptide Repeat,PPR)家族蛋白,与叶绿体中RNA代谢有关,其基因突变造成rpl2、rps12、atpA、atpF和ndhA等叶绿体转录本不能被正常编辑或剪切,导致叶片产生条纹[17-19];还有一类定位于叶绿体类核中的蛋白,这些蛋白的主要功能是参与质体转录,调控丙酮酸羧化酶(PEP)和RNA聚合酶有关基因的表达,如WLP2[20]、WSL3[21]和YSS1[22],其突变后损坏PEP活性,造成白条纹表型[20-22];WLP1基因编码一个50S核糖体大亚基蛋白L13[23],而WP1基因编码一个缬氨酸-tRNA合成酶1[24],它们主要参与调控叶绿体核糖体的发育,其突变体叶片表现为条纹表型,抽穗后幼穗表现白化[23-24];而YSS2[25]和WSL12[26]编码核苷二磷酸激酶(Nucleoside diphosphate kinase 2,NDPK2)蛋白,参与叶绿素的合成和光合作用,其基因突变后表现为条纹叶[25-26]。此外,编码钾离子通道蛋白的基因AM1突变后亦可造成叶片白条纹表型[27]。
在前期工作中,山西省农业科学研究院谷子研究所谷子分子育种课题组从谷子主推品种长农35号的甲基磺酸乙酯(EMS)诱变库中筛选鉴定到一个条纹叶突变体wsl2[28],有关该突变体的遗传机制尚不清楚。
本研究以谷子品种长农35号条纹叶突变体wsl2为主要研究材料,以wsl2×野生型杂交自交F2分离群体为定位群体,采用MutMap技术进行wsl2基因精细定位[29-30],同时,开发、鉴定共分离标记,并进行wsl2候选基因预测,旨在揭示wsl2基因在谷子叶绿素含量和光合作用中的功能,为解析C4植物叶绿体的发育机制奠定一定理论基础。
1 材料和方法
1.1 试验材料
以谷子材料长农35号为原始材料,经EMS诱变获得条纹叶色突变体wsl2。试验所用谷子材料均种植于山西省农业科学院谷子研究所试验田。
1.2 试验方法
1.2.1 wsl2突变体表型、遗传背景、农艺性状及遗传分析 于谷子苗期、拔节期、抽穗期、灌浆期和成熟期分别对wsl2和野生型进行叶片颜色表型调查;同时,选取谷子分子育种实验室现有的50个谷子分子标记(表1)对wsl2和野生型以及另外4份谷子材料品资39号、09K65、高88和K5-496进行分子标记检测,验证该突变体的遗传背景。选取突变体wsl2与野生型各10株于成熟期进行调查,调查的农艺性状包括节数、株高、穗下节长、节间粗、穗长、穗粗、穗质量及穗粒质量等。突变体wsl2同野生型长农35号杂交获得F1,观察F1叶色情况。F1自交构建F2分离群体,苗期(3~6叶)调查F2分离群体叶色分离情况,统计野生表型和条纹叶表型的植株数目,并进行卡方测试,进行突变体wsl2的遗传分析。
1.2.2 通过MutMap方法克隆候选基因 本研究采用MutMap方法对候选基因进行克隆[29-30]。wsl2与野生型长农35号杂交分离获得F2 群体,在苗期取30株条纹叶表型植株的叶片,提取DNA后等量混合并进行全基因组测序,同时,野生型长农35号作为对照进行基因组重测序(百迈克生物科技有限公司)。测序完成后,wsl2的混池数据以及野生型的数据分别同参考基因组豫谷1号比对,得到SNPs数据集。然后,对染色体上的SNP 位点进行过滤,去掉长农35号和混池DNA都有的SNPs,从而挑选出对于混池DNA特有的SNPs。最后计算每个SNPs的SNP-index,即SNP-index为该位点测到的突变序列的频次除以该位点的总深度,而理论上由于候选SNP同表型完全连锁,其SNP-index为1。挑选SNP-index接近1的区域为关联区间,最后对关联区间内基因注释后进一步得到候选基因。
1.2.3 候选基因的进一步确定 根据候选SNPs所在区域的DNA序列,采用软件Primer Premier 5.0设计引物(表1),分别以wsl2和野生型基因组DNA为模板,特异PCR产物回收后直接测序,根据峰图判断突变体中是否存在候选SNP。进一步根据突变位点设计dCAPS分子标记或共分离标记(表1),对wsl2和野生型进行检测,从而确定突变位点是否真实存在。PCR扩增总体积10 μL:模板1 μL,10× Buffer 1 μL,2.5 mmol/L dNTPs 0.8 μL,4 μmol/L特异引物1 μL,rTaq DNA聚合酶0.1 μL和ddH2O 6.1 μL。PCR扩增程序为:95 ℃ 5 min;95 ℃ 30 s,55~60 ℃ 30 s,72 ℃ 45~90 s,35个循环;72 ℃ 10 min,4 ℃保存。引物SIsv0296b1、SIsv0078b2、SIsv1045b1、SIsv0546b1、MRI501-3和MRI498-1(Cac8Ⅰ)用8%非变性PAGE胶电泳,经快速银染后照相统计。引物MRI413和MRI414的PCR产物在1%琼脂糖凝胶上进行电泳检测。
表1 所用引物信息
Tab.1 List of used primers

引物名称 Primer name 引物序列(5′-3′) Primer sequence 用途 Purpose SIsv0296b1FCCGGACAGATTTTGAAACACT突变体遗传背景检测部分标记SIsv0296b1RGGGTTGGTAATTGTGGCTGASIsv0078b2FTGGATTATGTGGAGCCATGT突变体遗传背景检测部分标记SIsv0078b2RGCATTGGTCTTCTTCCAAGGSIsv1045b1FCGGCATGGTGCATGTATTGT突变体遗传背景检测部分标记SIsv1045b1RTTGGAGGACGGATGCCTAAGSIsv0546b1FCTGTTTTGTCGTTCCCCTCT突变体遗传背景检测部分标记SIsv0546b1RCCCAGTCTGTGTCCAAAGTTMRI501-3FAGCACATGGCTGTGGGAAGG检测突变位点的共分离标记MRI501-3RCCAAACAGTGGCAATGAAAMRI498-1F(cac8Ⅰ)CTCGGATGGAGGAGAAACAGG检测突变位点的CAPS标记MRI498-1R(cac8Ⅰ)GCTAAAGACAGACGCTCGGAAGMRI413FAACGAAGCGAAATCCTACTC扩增基因Seita.9G561800前半部分MRI413RGATTAAACGTGGAAACCAGGMRI414FTGTGCCTTTACTAATTTGCG扩增基因Seita.9G561800后半部分MRI414RTTTGAACATGAGGGATTAAGC
利用Phytozome(https:// phytozome.jgi.doe.gov/pz/portal.html#!info?alias= Org_Sitalica)和PlantGDB(http://www.plantgdb.org/SiGDB/)数据库查找定位区间内的基因,对区间内基因进行功能注释;同时结合SIFGD(http://structuralbiology.cau. edu.cn/SIFGD/)数据库进行候选基因的转录模式分析[31]。
2 结果与分析
2.1 wsl2突变体表型、遗传背景及农艺性状分析
通过EMS诱变品种长农35号,筛选得到一个条纹叶突变体,命名为wsl2。在山西省农业科学院谷子研究所试验田大田生长情况下,wsl2在苗期表现为条纹叶表型,但从拔节期开始恢复为正常叶片表型,而野生型长农35号全生育期叶片表现均正常(图1-A)。选用50对分子标记对wsl2、野生型及另外4个谷子材料进行检测,突变体与野生型对照的条带在凝胶上的相对位置一致,DNA片段大小相同,说明突变体与野生型遗传背景相同,不是其他品种混杂;而其余4个谷子品种的条带与野生型在部分标记间显示出差异性(图1-B-E)。在农艺性状方面,突变体wsl2与野生型相比,节数、株高、穗下节长、节间粗、穗长、穗粗、穗质量及穗粒质量等性状上均无显著差异(表2)。
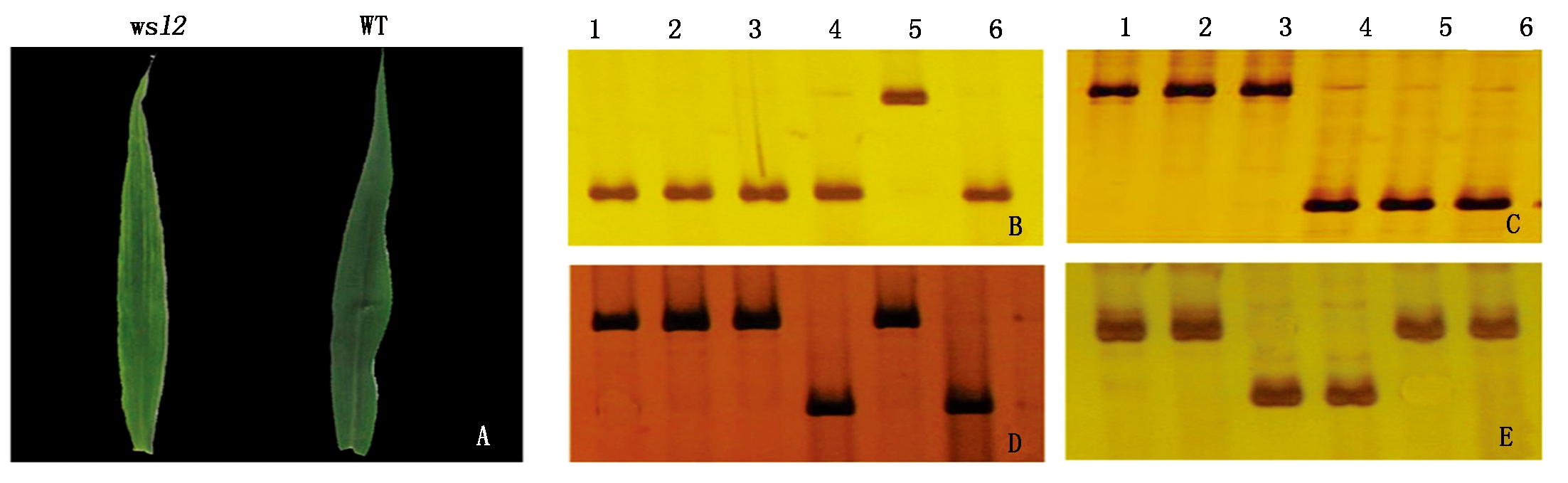
A.wsl2和野生型苗期第4叶表型;B-E.分别为部分标记SIsv0296b1、SIsv0078b2、SIsv1045b1和SIsv0546b1检测突变体遗传背景的电泳图片;1-6.分别为WT(长农35号)、wsl2、品资39号、09K65、高88和K5-496。
A.Phenotype of the 4th leaf between wsl2 and wild type in seedling stage; B-E.Electrophoretic images of partial markers SIsv0296b1,SIsv0078b2,SIsv1045b1 and SIsv0546b1 used to detect the genetic background of wsl2;1-6.WT(Changnong 35),wsl2,Pinzi 39,09K65,Gao 88 and K5-496.
图1 突变体wsl2苗期表型和遗传背景检测
Fig.1 Phenotype at seeding stage and genetic background detection of mutant wsl2
表2 wsl2和WT的农艺性状比较
Tab.2 Comparison of agronomic traits between wsl2 and WT
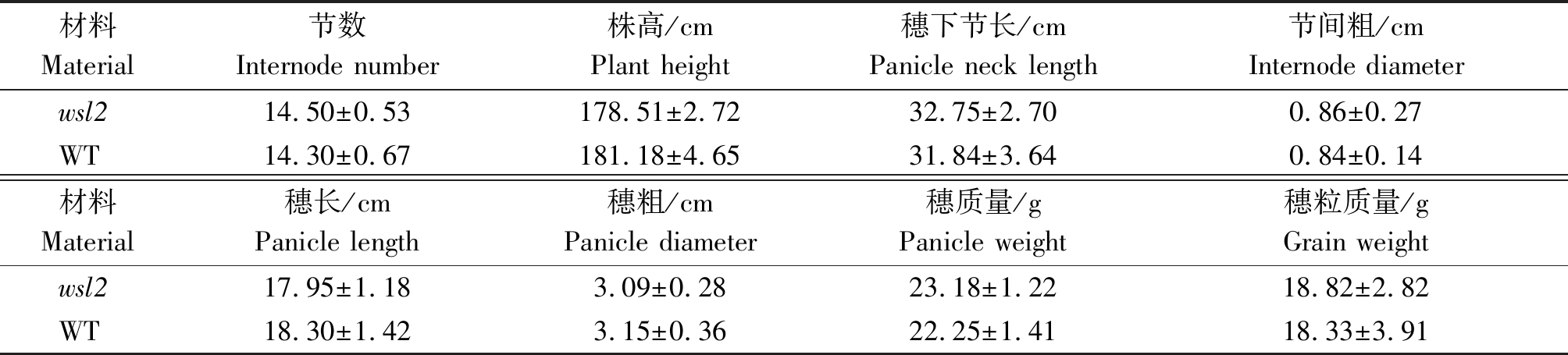
材料Material节数Internode number株高/cmPlant height穗下节长/cmPanicle neck length节间粗/cmInternode diameterwsl214.50±0.53178.51±2.7232.75±2.700.86±0.27WT14.30±0.67181.18±4.6531.84±3.640.84±0.14材料Material穗长/cmPanicle length穗粗/cmPanicle diameter穗质量/gPanicle weight穗粒质量/gGrain weightwsl217.95±1.183.09±0.2823.18±1.2218.82±2.82WT18.30±1.423.15±0.3622.25±1.4118.33±3.91
2.2 wsl2的遗传学分析
为了研究wsl2突变体的遗传特性,将wsl2同野生型长农35号杂交,F1植株的表型同野生型长农35号相同,没有条纹叶形成。在F2群体中,植株表现出明显的分离。苗期对植株表型进行观察,统计表明,wsl2×长农35号的F2群体中正常植株(289)和突变表型植株(94)数目的比例接近3∶1。卡方测试![]() 进一步说明wsl2的表型由单一隐性基因控制。
进一步说明wsl2的表型由单一隐性基因控制。
2.3 基于MutMap的候选基因克隆
为了进一步研究wsl2条纹叶形成的分子机制,本研究利用MutMap方法对突变体wsl2的候选基因进行克隆[29-30]。从突变体与野生型长农35号杂交的F2群体中挑选出30株条纹叶表型植株,混池测序,同时对野生型长农35号进行测序作为对照,然后进行SNP分析。挑选SNP-index接近1的SNPs为候选SNPs。对染色体上的SNP位点进行过滤,突变体wsl2共检测到12 585个混池特异SNP,得到突变体混池SNP-index的分布图(图2)。
利用拟合后index的95百分位数即0.95,得到该染色体上包含基因的关联区域共2个(表3),区间总长度为1.35 Mb,共包含184个基因,据统计,亲本与混池间存在非同义突变的SNP共18个,分布于9个基因上,即非同义突变的基因共9个,这些SNP或基因很有可能与突变性状直接相关(表4)。
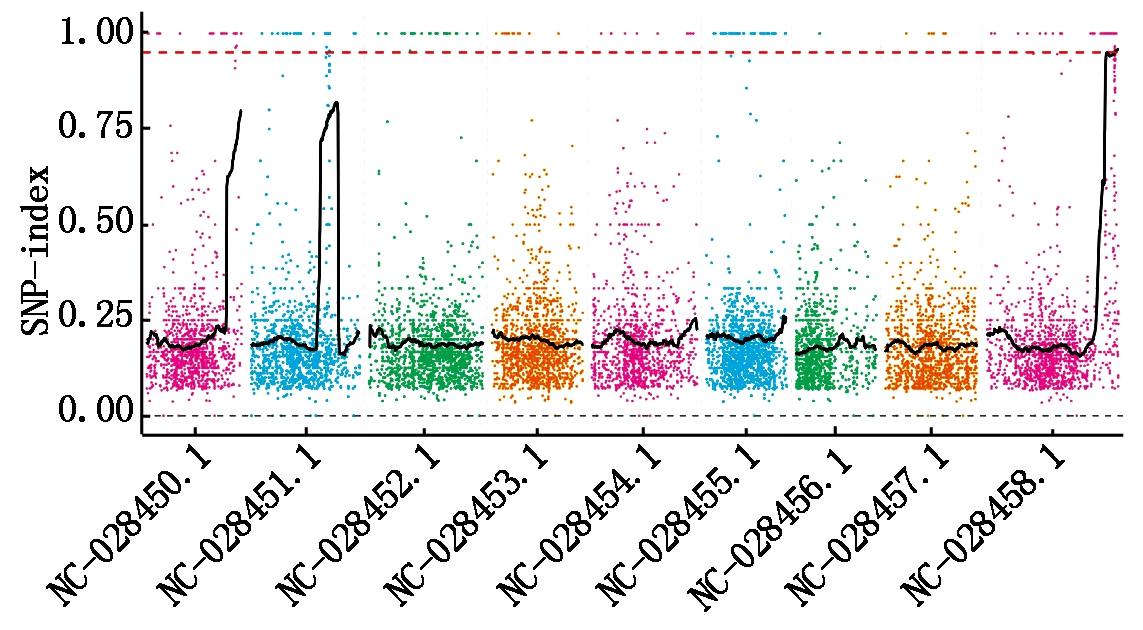
图2 SNP-index值在染色体上的分布
Fig.2 Distribution map of SNP-index value on chromosome
表3 MutMap关联区域信息统计
Tab.3 Statistical table of association region information by MutMap

染色体Chromosome_ID起始位点Start终止位点End大小/ MbSize基因数量Gene numberNC_028458.157548540575769590.032NC_028458.157627686589492471.32182总计 Total1.35184
表4 突变体wsl2非同义突变位点分析
Fig.4 Analysis of non-synonymous mutation sites for the mutant wsl2
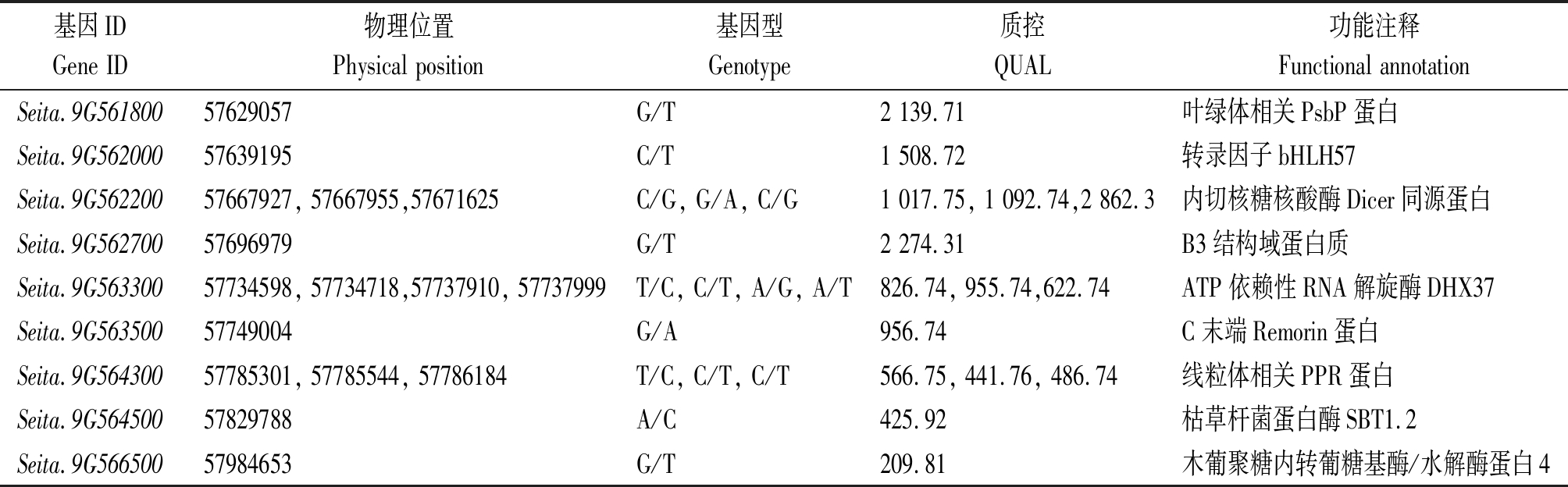
基因IDGene ID物理位置Physical position基因型Genotype质控QUAL功能注释Functional annotation Seita.9G56180057629057G/T2 139.71叶绿体相关PsbP蛋白Seita.9G56200057639195C/T1 508.72转录因子bHLH57Seita.9G56220057667927, 57667955,57671625C/G, G/A, C/G1 017.75, 1 092.74,2 862.3内切核糖核酸酶Dicer同源蛋白Seita.9G56270057696979G/T2 274.31B3结构域蛋白质Seita.9G56330057734598, 57734718,57737910, 57737999T/C, C/T, A/G, A/T826.74, 955.74,622.74ATP依赖性RNA解旋酶DHX37Seita.9G56350057749004G/A956.74C末端Remorin蛋白Seita.9G56430057785301, 57785544, 57786184T/C, C/T, C/T566.75, 441.76, 486.74线粒体相关PPR蛋白Seita.9G56450057829788A/C425.92枯草杆菌蛋白酶SBT1.2Seita.9G56650057984653G/T209.81木葡聚糖内转葡糖基酶/水解酶蛋白4
通过Phytozome(https://phytozome.jgi.doe.gov/pz/portal.html#!info?alias= Org_Sitalica)和PlantGDB(http://www.plantgdb.org/SiGDB/)数据库对非同义突变基因进行功能注释,发现Seita.9G561800是一个编码与叶绿体相关的PsbP蛋白基因(表4);同时,用SIFGD(http://structuralbiology.cau.edu.cn/SIFGD/)数据库对这9个非同义突变基因的转录模式进行了分析[31],根据基于FPKM的转录水平,发现Seita.9G561800在叶部的表达明显高于其他部位(图3)。因此,该基因可能为wsl2的关键候选基因。
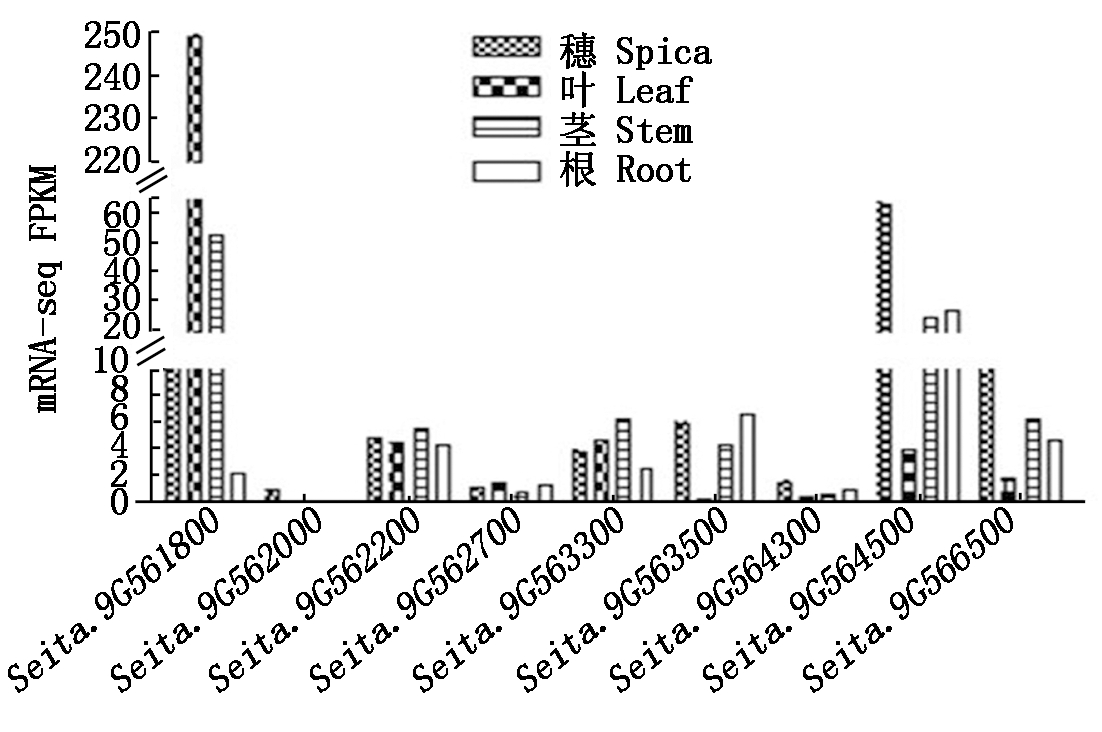
数据来源于SIFGD(http://structuralbiology.cau.edu.cn/SIFGD/)数据库[31]。
Data comes from SIFGD(http://structuralbiology.cau.edu.cn/SIFGD/)database.
图3 9个非同义突变基因在不同组织器官中的FPKM值
Fig.3 FPKM values of nine non-synonymous mutation genes in different tissues
2.4 突变位点的进一步验证
为了进一步验证突变体wsl2的候选突变位点,在突变位点上下游设计引物(表1)。PCR 扩增及测序显示,在野生型长农35号中,该位置为G,而在wsl2中,该位置为T(图4-A)。因此,在wsl2中确实发生了突变。为了进一步验证wsl2突变体的候选位点,根据突变碱基设计了dCAPS 标记MRI498-1(cac8 Ⅰ)(突变位点造成限制性内切酶Cac8 Ⅰ的差异)和共分离标记MRI501-3(表1)。利用分子标记检测野生型长农35号和wsl2突变体,结果显示,wsl2 与野生型条带存在差异,与预期结果相符合(图4-B)。因此,wsl2中确实存在G-T突变,wsl2的条纹叶表型很可能是由基因Seita.9G561800的突变导致。
2.5 基因位点的功能分析
查询谷子基因注释网站(https: //phytozome.jgi.doe. gov/pz/portal.html#!info? alias= Org_ Sita lica),Seita.9G561800含有6个外显子和5个内含子。其中,wsl2突变发生在第1外显子77 bp处,导致一个精氨酸(R)变成亮氨酸(L)。Seita.9G561800编码叶绿体相关的类PsbP蛋白,已被报道参与植物光合作用等生物学过程。Seita.9G561800是拟南芥AT3G553330基因的同源基因,其编码的类PsbP蛋白PPL1能够在高强度光照下有效修复PSⅡ;是水稻LOC-Os 10g32400基因的同源基因,其编码PsbP蛋白,研究发现,PsbP表达下调的植株叶片轻微发白,生长迟缓,光合作用受到抑制。因此,结合候选基因的分析和功能注释,wsl2应该是谷子的一个新条纹叶突变体,初步推测其是由一个编码类PsbP蛋白的Seita.9G561800基因发生突变所致,但还需进一步进行基因功能验证。
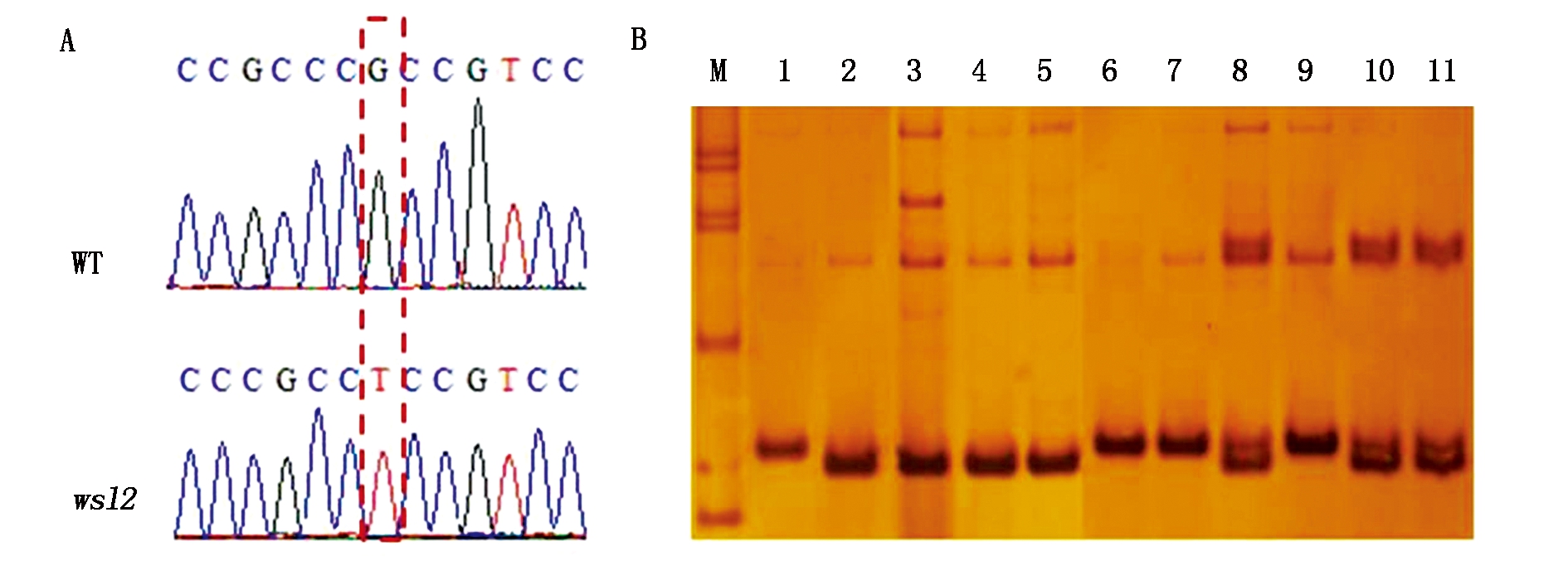
A.突变体和野生型测序峰图;B.紧密连锁标记MRI501-3电泳图片。M.DNA Marker;1.野生型长农35号;2.突变体wsl2;3-5.wsl2×野生型杂交自交F2分离群体中的部分突变表型单株;6-11.wsl2×野生型杂交自交F2分离群体中的部分正常表型单株。
A.Sequencing peak maps of WT and wsl2;B.Electrophoretic images of linkage marker MRI501-3. M.DNA Marker;1.WT Changnong 35;2.wsl2;3-5.Partial mutant phenotypic individuals in F2 populations of wsl2×WT;6-11.Normal phenotypic individuals in F2 populations of wsl2×WT.
图4 基因突变位点验证及紧密连锁标记的开发
Fig.4 Verification of gene mutation site and development of linkage marker
3 结论与讨论
3.1 MutMap在突变基因定位和克隆中的应用
MutMap方法是Abe等[29-30]2012年提出的一种基因克隆新方法,相较于图位克隆方法,MutMap方法克隆基因更快捷、效率更高、成本更低,该方法是以二代测序技术为基础,通过对F2分离群体中隐性表型个体的DNA pool的深度测序,然后把突变体群体测序结果与野生型基因组序列相比对,找出造成突变体性状的基因。MutMap方法在快速克隆质量性状的隐性核基因和数量性状的QTL定位方面均有应用[29-30,32-34]。本研究中,以wsl2和野生型杂交自交的F2分离群体为定位群体,采用MutMap技术进行wsl2突变基因精细定位,取30株条纹叶表型植株的叶片,同时野生型作为对照进行基因组重测序,筛选SNP-index接近于1的区域作为与目标性状关联的染色体区域,在该区间中共包含9个非同义突变基因,进一步根据基因功能注释及表达模式分析,快速推测候选基因。
3.2 PsbP蛋白家族的功能分析
PsbP蛋白是叶绿体光系统Ⅱ中放氧复合体的外周蛋白之一,是保证PSⅡ功能完整性不可缺少的组成部分,在光合作用中能够利用光能氧化水产生氧气[35-36]。光合系统相关基因的研究对叶绿体发育机制和提高植物光合效率具有重要的理论意义和研究价值[37]。
高等植物中的PsbP蛋白来源于蓝细菌的cyanoP蛋白[35]。在高等植物中发现了大量的PsbP同源基因,拟南芥中有2个PsbP基因分别编码PsbP1和PsbP2,而烟草中据报道有4个PsbP同源基因,这些基因编码蛋白的结构功能相对比较保守[36,38]。除此之外,高等植物中还编码了一些与cyanoP序列同源性较高的类PsbP蛋白(PsbP-like protein,PPL)[39-40]和一些具有PsbP结构域的PPD(PsbP-domain protein,PPD)蛋白[41]。PsbP蛋白家族含有保守的C端结构域。利用RNAi方法不同程度地沉默烟草的4个同源基因(PsbP-RNAi),使PsbP的表达受到抑制,结果发现,PsbP的积累量影响了PSⅡ的活性。突变体在生长过程中与野生型相比叶色偏白,植株生长迟缓,叶绿体结构紊乱[42-43]。拟南界中发现了2个类PsbP蛋白,PPL1(AT3G553330)能够在高强度光照下有效修复PSⅡ[39],而PPL2(AT2G39470)是叶绿体NAD(P)H脱氢酶复合体稳定积累所必需的[40]。
本研究中,通过MutMap方法克隆到一个与叶绿体相关的类PsbP蛋白基因,Seita. 9G 561800是拟南芥PPL1(AT3G553330)基因的同源基因,其编码的类PsbP蛋白PPL1能够在高强度光照下有效修复PSⅡ;是水稻Os10g32400基因的同源基因,其编码PsbP蛋白,研究发现,PsbP表达下调的植株叶片轻微发白,生长迟缓,光合作用受到抑制。因此,结合候选基因的分析和功能注释,该编码类PsbP蛋白的Seita.9G561800基因发生的突变可能是导致wsl2叶色发生突变的原因,该基因尚需进一步进行功能验证。
[1] Zhao S L,Long W H,Wang Y H,Liu L L,Wang Y L,Niu M,Zheng M,Wang D,Wan J M. A rice White-stripe leaf3 (wsl3)mutant lacking an HD domain-containing protein affects chlorophyll biosynthesis and chloroplast development[J]. Journal of Plant Biology,2016,59(3):282-292. doi:10.1007/s12374-016-0459-8.
[2] van Rooijen R, Kruijer W, Boesten R, van Eeuwijk F A, Harbinson J, Aarts M G M.Natural variation of YELLOW SEEDLING 1 affects photosynthetic acclimation of Arabidopsis thaliana[J]. Nature Communications,2017,8(1): 1424. doi:10.1038/s41467-017-01576-3.
[3] Wang N,Liu Z Y,Zhang Y,Li C Y,Feng H. Identification and fine mapping of a stay-green gene(Brnye1)in pakchoi(Brassica campestris L. ssp. chinensis)[J]. Theoretical and Applied Genetics,2018,131(3):673-684. doi: 10.1007/s00122-017-3028-8.
[4] 邹金财,张维林,夏明辉,邱洋松,王长春,杨玲,张小明.水稻阶段性温敏白化转绿突变体stgra254的特征和基因定位[J].华北农学报,2017,32(3): 1-6. doi:10.7668/hbnxb.2017.03.001.
Zou J C,Zhang W L,Xia M H,Qiu Y S,Wang C C,Yang L,Zhang X M. Characterization and gene mapping of stage thermo-sensitive green-revertible albino mutant stgra254 in rice[J].Acta Agriculturae Boreali-Sinica,2017,32(3): 1-6.
[5] Junqueira N E G, Ortiz-Silva B, Leal-Costa M V, Alves-Ferreira M, Dickinson H G, Langdale J A, Reinert F.Anatomy and ultrastructure of embryonic leaves of the C4 species Setaria viridis[J]. Annals of Botany,2018,121(6):1163-1172. doi: 10.1093/aob/mcx217.
[6] Jia G Q,Huang X H,Zhi H,Zhao Y,Zhao Q,Li W J,Chai Y,Yang L F,Liu K Y,Lu H Y,Zhu C R,Lu Y Q,Zhou C C,Fan D L,Weng Q J,Guo Y L,Huang T,Zhang L,Lu T T,Feng Q,HaO H F,Liu H K,Lu P,Zhang N,Li Y H,Guo E H,Wang S J,Wang S Y,Liu J R,Zhang W F,Chen G Q,Zhang B J,Li W, Wang Y F,Li H Q,Zhao B H,Li J Y,Diao X M,Han B. A haplotype map of genomic variations and genome-wide association studies of agronomic traits in foxtail millet(Setaria italica)[J]. Nature Genetics,2013,45(8): 957-961. doi:10.1038/ng.2673.
[7] 贾冠清,刁现民.谷子(Setaria italica(L.)P. Beauv.)作为功能基因组研究模式植物的发展现状及趋势[J].生命科学,2017,29(3): 292-301. doi: 10.13376/j.cbls/2017039.
Jia G Q,Diao X M. Current status and perspectives of researches on foxtail millet(Setaria italica (L.)P. Beauv): A potential model of plant functional genomics studies[J]. Chinese Bulletin of Life Sciences,2017,29(3): 292-301.
[8] Luo M Z,Zhang S,Tang C J,Jia G Q,Tang S,Zhi H,Diao X M.Screening of mutants related to the C4 photosynthetic kranz structure in foxtail millet[J]. Frontiers in Plant Science,2018,9:1650. doi:10.3389/fpls.2018.01650.
[9] Li W,Tang S,Zhang S,Shan J G,Tang C J,Chen Q N,Jia G Q,Han Y H,Zhi H,Diao X M. Gene mapping and functional analysis of the novel leaf color gene SiYGL1 in foxtail millet(Setaria italica(L.)P. Beauv)[J]. Physiologia Plantarum,2016,157(1): 24-37. doi:10.1111/ppl.12405.
[10] Tang C J,Tang S,Zhang S,Luo M Z,Jia G Q,Zhi H,Diao X M. SiSTL1 encoding a large subunit of ribonudeotide reductase,is crucial for plant growth,chloroplast biogenesis,and cell cycle progression in Setaria italica[J]. Journal of Experimental Botany,2019,70(4): 1167-1182. doi:10.1093/jxb/ery429.
[11] Zhang S, Tang S, Tang C J, Luo M Z, Jia G Q, Zhi H, Diao X M. SiSTL2 is required for cell cycle,leaf organ development,chloroplast biogenesis,and has effects on C4 photosynthesis in Setaria italica(L.)P. Beauv[J]. Frontiers in Plant Science,2018,9: 1103. doi: 10.3389/fpls.2018.01103.
[12] Yoo S C,Cho S H,Sugimoto H,Li J J,Kusumi K,Koh H J,Iba K,Paek N C. Rice virescent3 and stripe1 encoding the large and small subunits of ribonucleotide reductase are required for chloroplast biogenesis during early leaf development[J]. Plant Physiol,2009,150(1): 388-401. doi:10.1104/pp.109.136648.
[13] Kusumi K,Mizutani A,Nishimura M,Iba K. A virescent gene V1 determines the expression timing of plastid genes for transcription/translation apparatus during early leaf development in rice[J]. The Plant Journal,1997,12(6): 1241-1250. doi:10. 1046/j.1365-313x. 1997. 12 0 61241.x.
[14] Sugimoto H,Kusumi K,Tozawa Y,Yazaki J,Kishimoto N,Kikuchi S,Iba K. The virescent-2 mutation inhibits translation of plastid transcripts for the plastid genetic system at an early stage of chloroplast differentiation[J]. Plant and Cell Physiology,2004,45(8): 985-996. doi: 10.1093/pcp/pch111.
[15] Sugimoto H,Kusumi K,Noguchi K,Yano M,Yoshimura A,Iba K. The rice nuclear gene,VIRESCENT 2,is essential for chloroplast development and encodes a novel type of guanylate kinase targeted to plastids and mitochondria[J]. The Plant Journal,2007,52(3):512-527. doi:10.1111/j.1365-313X.2007.03251.x.
[16] Dong H,Fei G L,Wu C Y,Wu F Q,Sun Y Y,Chen M J,Ren Y L,Zhou K N,Cheng Z J,Wang J L,Jiang L,Zhang X,Guo X P,Lei C L,Su N,Wang H Y,Wan J M. A rice virescent-yellow leaf mutant reveals new insights into the role and assembly of plastid caseinolytic protease in higher plants[J]. Plant Physiol,2013,162(4):1867-1880. doi:10.1104/pp.113.217604.
[17] Tan J J,Tan Z H,Wu F Q,Sheng P K,Heng Y Q,Wang X H,Ren Y L,Wang J L,Guo X P,Zhang X,Cheng Z J,Jiang L,Liu X M,Wang H Y,Wan J M. A novel chloroplast-localized pentatricopeptide repeat protein involved in splicing affects chloroplast development and abiotic stress response in rice[J]. Molecular Plant,2014,7(8):1329-1349. doi:10.1093/mp/ssu054.
[18] Wang Y,Ren Y L,Zhou K N,Liu L L,Wang J L,Xu Y,Zhang H,Zhang L,Feng Z M,Wang L W,Ma W W,Wang Y L,Guo X P,Zhang X,Lei C L,Cheng Z J,Wan J M. WHITE STRIPE LEAF4 encodes a novel P-Type PPR protein required for chloroplast biogenesis during early leaf development[J]. Frontiers in Plant Science,2017,8:1116. doi:10.3389/fpls.2017.01116.
[19] Liu X,Lan J,Huang Y S,Cao P H,Zhou C L,Ren Y K,He N Q,Liu S J,Tian Y L,Thanhliem N,Jiang L,Wan J M. WSL5,a pentatricopeptide repeat protein,is essential for chloroplast biogenesis in rice under cold stress[J]. Journal of Experimental Botany,2018,69(16): 3949-3961. doi:10.1093/jxb/ery259.
[20] Lü Y S,Shao G N,Qiu J H,Jiao G A,Sheng Z H,Xie L H,Wu Y W,Tang S Q,Wei X J,Hu P S. White Leaf and Panicle 2,encoding a PEP-associated protein,is required for chloroplast biogenesis under heat stress in rice[J]. J Exp Bot,2017,68(18):5147-5160. doi:10.1093/jxb/erx332.
[21] Wang L W,Wang C M,Wang Y H,Niu M,Ren Y L,Zhou K N,Zhang H,Lin Q B,Wu F Q,Cheng Z J,Wang J L,Zhang X,Guo X P,Jiang L,Lei C L,Wang J,Zhu S S,Zhao Z C,Wan J M. WSL3,a component of the plastid-encoded plastid RNA polymerase,is essential for early chloroplast development in rice[J]. Plant Molecular Biology,2016,92(4-5):581-595. doi:10.1007/s11103-016-0533-0.
[22] Zhou K N, Ren Y L, Zhou F, Wang Y, Zhang L, Lyu J, Zhao S L, Ma W W, Zhang H, Wang L W, Wang C M, Wu F Q, Zhang X, Guo X P, Cheng Z J, Wang J L, Lei C L, Jiang L, Li Z F, Wan J M. Young Seedling Stripe1 encodes a chloroplast nucleoid-associated protein required for chloroplast development in rice seedlings[J]. Planta,2017,245(1):45-60.doi:10.1007/s00425-016-2590-7.
[23] Song J,Wei X J,Shao G N,Sheng Z H,Chen D B,Liu C L,Jiao G A,Xie L H,Tang S Q,Hu P S. The rice nuclear gene WLP1 encoding a chloroplast ribosome L13 protein is needed for chloroplast development in rice grown under low temperature conditions[J].Plant Molecular Biology,2014,84(3):301-314. doi:10.1007/s11103-013-0134-0.
[24] Wang Y L,Wang C M,Zheng M,Lyu J,Xu Y,Li X H,Niu M,Long W H,Wang D,Wang H Y,Terzaghi W,Wang Y H,Wan J M. WHITE PANICLE1,a Val-tRNA synthetase regulating chloroplast ribosome biogenesis in rice,is essential for early chloroplast development[J]. Plant Physiology,2016,170(4):2110-2123. doi:10.1104/pp.15.01949.
[25] Zhou K N,Xia J F,Wang Y L,Ma T C,Li Z F. A Young Seedling Stripe2 phenotype in rice is caused by mutation of a chloroplast-localized nucleoside diphosphate kinase 2 required for chloroplast biogenesis[J]. Genetics and Molecular Biology,2017,40(3): 630-642. doi:10.1590/1678-4685-GMB-2016-0267.
[26] Ye W J,Hu S K,Wu L W,Ge C W,Cui Y T,Chen P,Wang X Q,Xu J,Ren D Y,Dong G J,Qian Q,Guo L B. White stripe leaf 12(WSL12),encoding a nucleoside diphosphate kinase 2(OsNDPK2),regulates chloroplast development and abiotic stress response in rice(Oryza sativa L.)[J]. Molecular Breeding: New Strategies in Plant Improvement,2016,36:57. doi: 10. 1007/ s11 032-016-0479-6.
[27] Sheng P K,Tan J J,Jin M N,Wu F Q,Zhou K N,Ma W W,Heng Y Q,Wang J L,Guo X P,Zhang X,Cheng Z J,Liu L L,Wang C M,Liu X M,Wan J M. Albino midrib 1,encoding a putative potassium efflux antiporter,affects chloroplast development and drought tolerance in rice[J]. Plant Cell Reports,2014,33(9):1581-1594. doi:10.1007/s00299-014-1639-y.
[28] Wang J,Yuan F,Yang H Q,Li Y H,Fan H P,Wang L X,Zhang A Y,Guo R H. Identification and analysis of mature plant type mutants of M1 generation of foxtail millet Changnong 35 treated with EMS mutagenesis[J]. Agricultural Science & Technology,2011,12(11):1628-1632.doi:10.6175/j.cnki.1009-4229.2011.11.032.
[29] Abe A, Kosugi S, Yoshida K, Natsume S, Takagi H, Kanzaki H, Matsumura H, Yoshida K, Mitsuoka C, Tamiru M, Innan H, Cano L, Kamoun S, Terauchi R. Genome sequencing reveals agronomically important loci in rice using MutMap[J]. Nature Biotechnology,2012,30(2):174-178. doi: 10.1038/nbt.2095.
[30] James G V, Patel V, Nordström K J, Klasen J R, Salomé P A, Weigel D, Schneeberger K.User guide for mapping-by-sequencing in Arabidopsis[J].Genome Biology,2013,14(6): R61. doi: 10.1186/gb-2013-14-6-r61.
[31] You Q,Zhang L W,Yi X,Zhang Z H,Xu W Y,Su Z. SIFGD: Setaria italica functional genomics database[J]. Molecular Plant,2015,8(6): 967-970. doi:10.1016/j.molp.2015.02.001.
[32] 陈竹锋,严维,王娜,张文辉,谢刚,卢嘉威,简智华,刘东风,唐晓艳.利用改进的MutMap方法克隆水稻雄性不育基因[J].遗传,2014,36(1):85-93.doi:10.3724/SP.J.1005.2014.00085.
Chen Z F,Yan W,Wang N,Zhang W H,Xie G,Lu J W,Jian Z H,Liu D F,Tang X Y. Cloning of a rice male sterility gene by a modified MutMap method[J]. Hereditas,2014,36(1): 85-93.
[33] 袁金红,李俊华,袁娇娇,贾克利,李书粉,邓传良,高武军.基于全基因组测序的MutMap方法在正向遗传学研究中的应用[J].遗传,2017,39(12):1168-1177. doi: 10.16288/j.yczz.17-095.
Yuan J H,Li J H,Yuan J J,Jia K L,Li S F,Deng C L,Gao W J. The application of MutMap in forward genetic studies based on whole-genome sequencing[J]. Hereditas,2017,39(12): 1168-1177.
[34] 郭广君,刁卫平,刘金兵,潘金宝,戈伟,王述彬.辣椒抗黄瓜花叶病毒病研究进展[J].华北农学报,2014,29(S1): 77-84. doi: 10.7668/hbnxb2014.S1.017.
Guo G J,Diao W P,Liu J B,Pan J B,Ge W,Wang S B. Research process of resistance to Cucumber mosaic virus in pepper[J]. Acta Agriculturae Boreali-Sinica,2014,29(S1):77-84.
[35] Ifuku K,Ishihara S,Shimamoto R,Ido K,Sato F. Structure,function,and evolution of the PsbP protein family in higher plants[J]. Photosynthesis Research,2008,98(1-3):427-437. doi: 10.1007/s11120-008-9359-1.
[36] Bricker T M,Roose J L,Zhang P P,Frankel L K.The PsbP family protiens[J].Photosynthesis Research,2013,116(2-3):235-250. doi:10.1007/s11120-013-9820-7.
[37] Tsai C C,Wu Y J,Sheue C R,Liao P C,Chen Y H,Li S J,Liu J W,Chang H T,Liu W L,Ko Y Z,Chiang Y C. Molecular basis underlying leaf variegation of a moth orchid mutant(Phalaenopsis aphrodite subsp. formosana)[J]. Frontiers in Plant Science,2017,8:1333. doi:10.3389/fpls.2017.01333.
[38] Ifuku K,Yamamoto Y,Ono T A,Ishihara S,Sato F. PsbP protein,but not PsbQ protein,is essential for the regulation and stabilization of photosystem Ⅱ in higher plants[J]. Plant Physiology,2005,139(3):1175-1184. doi:10.1104/pp.105.068643.
[39] Ishihara S, Takabayashi A, Ido K, Endo T, Ifuku K, Sato F.Distinct functions for the two PsbP-like proteins PPL1 and PPL2 in the chloroplast thylakoid lumen of Aarabidopsis[J]. Plant Physiology,2007,145(3):668-679. doi:10.1104/pp.107.105866.
[40] Ishihara S,Takabayashi A,Endo T,Ifuku K,Sato F. Functional analysis of two PsbP-like(PPL)proteins in Arabidopsis thaliana[M]// Allen J F,Gantt E,Golbeck J H,Osmond B.Photosynthesis. Energy from the Sun Dordrech: Springer,2008: 1091-1094. doi:10.1007/978-1-4020-6709-9_237.
[41] Liu J,Yang H X,Lu Q T,Wen X G,Chen F,Peng L W,Zhang L X,Lu C M. PsbP-domain protein1,a nuclear-encoded thylakoid lumenal protein,is essential for photosystem I assembly in Arabidopsis[J]. The Plant Cell,2012,24(12):4992-5006. doi:10.1105/tpc.112.106542.
[42] Ifuku K,Yamamoto Y,Sato F. Specific RNA interference in psbP genes encoded by a multigene family in Nicotiana tabacum with a short 3′untranslated sequence[J]. Bioscience,Biotechnology,and Biochemistry,2003,67(1):107-113. doi:10.1271/bbb.67.107.
[43] Ishihara S,Yamamoto Y,Ifuku K,Sato F. Functional analysis of four members of the PsbP family in photosystem Ⅱ in Nicotiana tabacum using differential RNA interference[J]. Plant and Cell Physiology,2005,46(12):1885-1893. doi: 10.1093/pcp/pci207.