成花转变作为植物开花的第一步,是植物从营养生长向生殖生长转变的重要转折点。前人研究表明,MADS-box基因家族在控制开花时间方面起着非常重要的作用[1-4]。SOC1(SUPPRESSOR OF OVEREXPRESSION OF CONSTANS 1)基因属于MADS-box基因家族中的SOC1/Tomato MADS-box gene 3(TM3)亚家族,它是作用于开花时间调控路径的关键整合子,能够整合来自光周期途径、春化途径、自主途径和赤霉素途径的多种开花信号[5-6],促进植物的营养分生组织向花分生组织转变,进而促进开花。目前已从矮牵牛[7](Petunia hybrida)、非洲菊[8](Gerbera hybrida)、石斛兰属[9](Dendrobium)、洋桔梗[10](Eustoma grandiflorumin)、甘菊[11](Chrysanthemum lavandulifolium)、美洲山杨[12](Populus tremuloides)、葡萄[13](Vitis vinifera)、紫斑牡丹[14](Paeonia rockii)、芒果[15](Mangifera indica)、水曲柳[16](Fraxinus mandschurica)、蒺藜苜蓿[17](Medicago truncatula)、水仙[18](Narcissus tazetta)、红掌[19](Anthurium andraeanum)和火龙果[20](Hylocereus polyrhizus)等多个物种中分离到SOC1的同源基因。近年来的研究发现SOC1基因除了具有促进开花的功能,在很多物种中还表现出影响花发育的功能[7-9,21-22];因此,为探明SOC1在不同植物中的功能,还需开展进一步的研究。
梅花(Prunus mume Sieb. et Zucc.)是中国传统名花,在中国有3 000多年的栽培历史[23]。了解梅花的开花转变过程对调控梅花花期、加速品种改良进程具有十分重要的意义。然而,有关梅花SOC1基因的功能分析未见报道,笔者前期从梅花中克隆获得了3个SOC1同源基因PmSOC1-1(GenBank ID:JF806632.1)、PmSOC1-2(GenBank ID:KP938964)和PmSOC1-3(GenBank ID:KP938965)的cDNA全长,并对其进行了时空表达模式分析[24]。本研究通过构建表达载体将克隆得到的3个SOC1同源基因在拟南芥中异源表达,分析这些基因在拟南芥成花过程中的功能,希望能为探明梅花成花转变的分子机理及利用基因工程加速梅花育种进程提供理论依据。
1 材料和方法
1.1 试验材料与试剂
试验用拟南芥为生态型Col-0。拟南芥种子播于培养基质中(营养土∶蛭石=1∶1),放入光照培养箱中进行培养,温度为(22±1)℃,相对湿度60%~80%,每天16 h光照/8 h黑暗,光强约120 μmol/(m2·s)。
植物表达载体pCAMBIA 1304和土壤农杆菌(Agrobacterium tumefaciens) EHA105由花卉种质创新与分子育种北京市重点实验室保存;限制性内切酶Bgl Ⅱ和BstE Ⅱ购自NEB公司;PrimeSTAR HS DNA Polymerase(R010A)、Solution Ⅰ连接液购自大连宝生物有限公司。
1.2 表达载体构建及转化拟南芥
根据前期克隆得到的梅花SOC1同源基因PmSOC1-1、PmSOC1-2和PmSOC1-3的cDNA全长序列和pCAMBIA 1304载体上的酶切位点分别设计特异性引物(表1),在限制性内切酶Bgl Ⅱ和BstE Ⅱ的处理下,将目的条带连入pCAMBIA1304植物表达载体上。采取花序浸染法[25]将含有目的基因的表达载体转化到野生型拟南芥中,利用潮霉素筛选转基因植株。以转基因拟南芥的DNA为模板,分别利用PmSOC1-1、PmSOC1-2和PmSOC1-3的检测引物(表1)对转基因拟南芥进行PCR检测,分别以含PmSOC1-1、PmSOC1-2和PmSOC1-3基因的质粒和野生型拟南芥作为阳性和阴性对照。收获阳性转基因株系的T3种子用于后期表型分析。
1.3 转基因拟南芥表型分析
将T3拟南芥转基因株系种子播于含潮霉素(50 mg/L)培养基进行筛选,培养14 d后分别移栽至长日照(16 h光照/8 h黑暗)和短日照(8 h光照/16 h黑暗)条件下养护,环境温度为(22±1)℃,光强约120 μmol/(m2·s)。采用RT-PCR和qRT-PCR对梅花PmSOC1-1、PmSOC1-2和PmSOC1-3基因在转基因拟南芥中是否表达及表达量高低进行分析。对T3转基因植株和野生型对照开花时的莲座叶数量、开花需要天数和表型变化进行记录。开花定义标准:花序下方第一朵花或花序顶端合生的Terminal flower(TF)开放的时间。采用SPSS 16.0软件进行数据处理和分析,每个株系以及野生型对照均统计20株以上。
1.4 转基因拟南芥中基因表达分析
为了分析转基因拟南芥中AtAGL24、AtLFY、AtAP1、AtFUL等基因的表达水平,以拟南芥AtTUB2为内参基因,剪取播种后6,12,16 d转基因拟南芥的全株进行实时荧光定量(qRT-PCR)分析,所用引物序列见表1。
表1 引物序列
Tab.1 Primers sequence used in this study
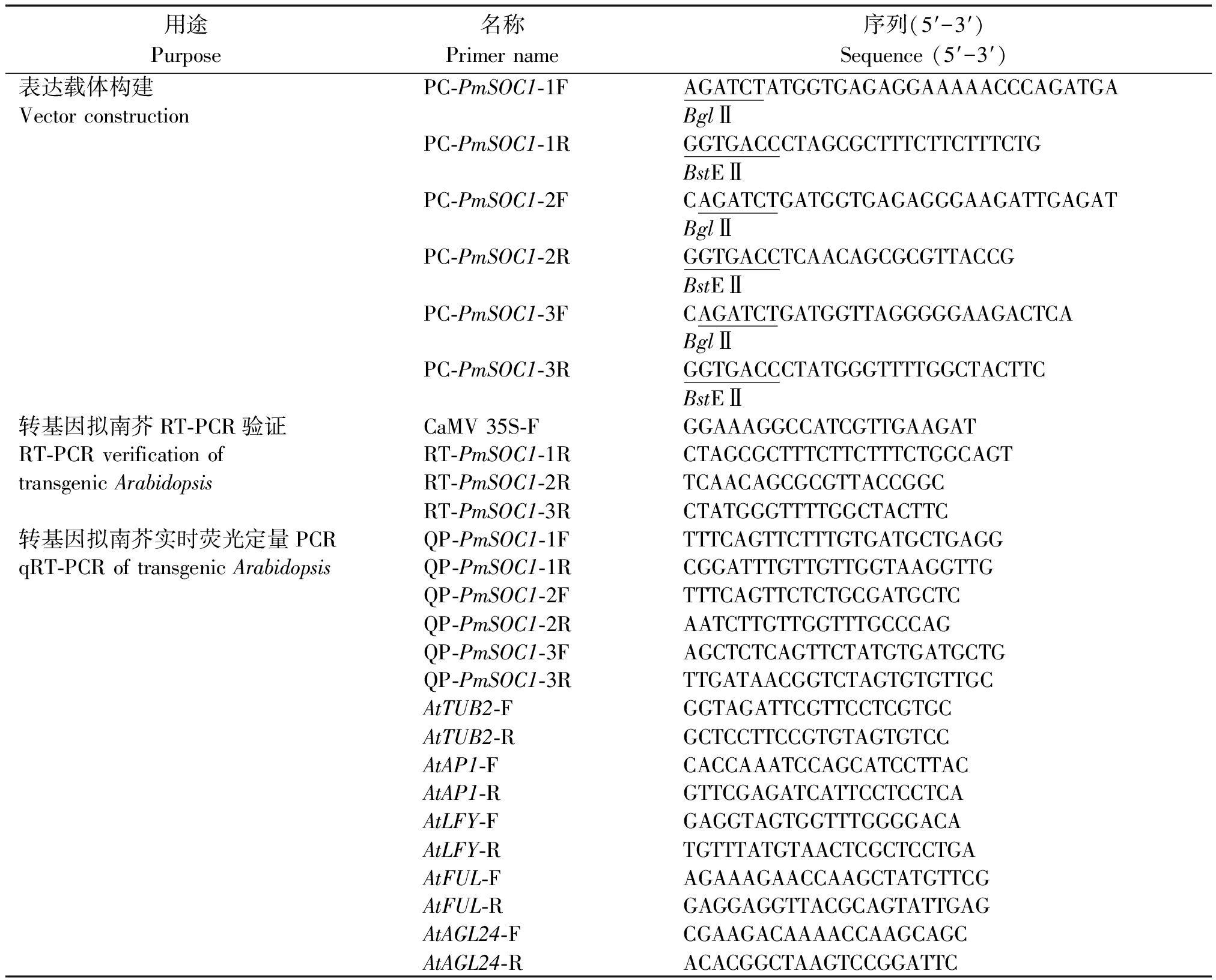
用途Purpose名称Primer name序列(5′-3′)Sequence (5′-3′)表达载体构建PC-PmSOC1-1FAGATCTATGGTGAGAGGAAAAACCCAGATGAVector constructionBglⅡPC-PmSOC1-1RGGTGACCCTAGCGCTTTCTTCTTTCTGBstEⅡPC-PmSOC1-2FCAGATCTGATGGTGAGAGGGAAGATTGAGATBglⅡPC-PmSOC1-2R GGTGACCTCAACAGCGCGTTACCGBstEⅡPC-PmSOC1-3FCAGATCTGATGGTTAGGGGGAAGACTCABglⅡPC-PmSOC1-3RGGTGACCCTATGGGTTTTGGCTACTTCBstEⅡ转基因拟南芥RT-PCR验证CaMV 35S-F GGAAAGGCCATCGTTGAAGATRT-PCR verification of RT-PmSOC1-1RCTAGCGCTTTCTTCTTTCTGGCAGTtransgenic ArabidopsisRT-PmSOC1-2RTCAACAGCGCGTTACCGGCRT-PmSOC1-3RCTATGGGTTTTGGCTACTTC转基因拟南芥实时荧光定量PCRQP-PmSOC1-1FTTTCAGTTCTTTGTGATGCTGAGGqRT-PCR of transgenic ArabidopsisQP-PmSOC1-1RCGGATTTGTTGTTGGTAAGGTTGQP-PmSOC1-2FTTTCAGTTCTCTGCGATGCTCQP-PmSOC1-2RAATCTTGTTGGTTTGCCCAGQP-PmSOC1-3FAGCTCTCAGTTCTATGTGATGCTGQP-PmSOC1-3RTTGATAACGGTCTAGTGTGTTGCAtTUB2-FGGTAGATTCGTTCCTCGTGCAtTUB2-RGCTCCTTCCGTGTAGTGTCCAtAP1-FCACCAAATCCAGCATCCTTACAtAP1-RGTTCGAGATCATTCCTCCTCAAtLFY-FGAGGTAGTGGTTTGGGGACAAtLFY-RTGTTTATGTAACTCGCTCCTGAAtFUL-FAGAAAGAACCAAGCTATGTTCGAtFUL-RGAGGAGGTTACGCAGTATTGAGAtAGL24-FCGAAGACAAAACCAAGCAGCAtAGL24-RACACGGCTAAGTCCGGATTC
2 结果与分析
2.1 转基因拟南芥中的外源基因表达检测
采用农杆菌介导的花器官浸染法将PmSOC1-1、PmSOC1-2和PmSOC1-3基因分别转入拟南芥。经过潮霉素筛选,共获得转PmSOC1-like基因的T1拟南芥108株,其中转PmSOC1-1基因的拟南芥35株,转PmSOC1-2基因的拟南芥47株,转PmSOC1-3基因的拟南芥26株。对这些转基因拟南芥进行了PCR检测,结果显示,转基因植株中均能扩增出与阳性对照同样大小的目的片段,而野生型拟南芥中未扩增出该片段(图1),说明PmSOC1-1、PmSOC1-2和PmSOC1-3基因已经分别导入拟南芥植株中。
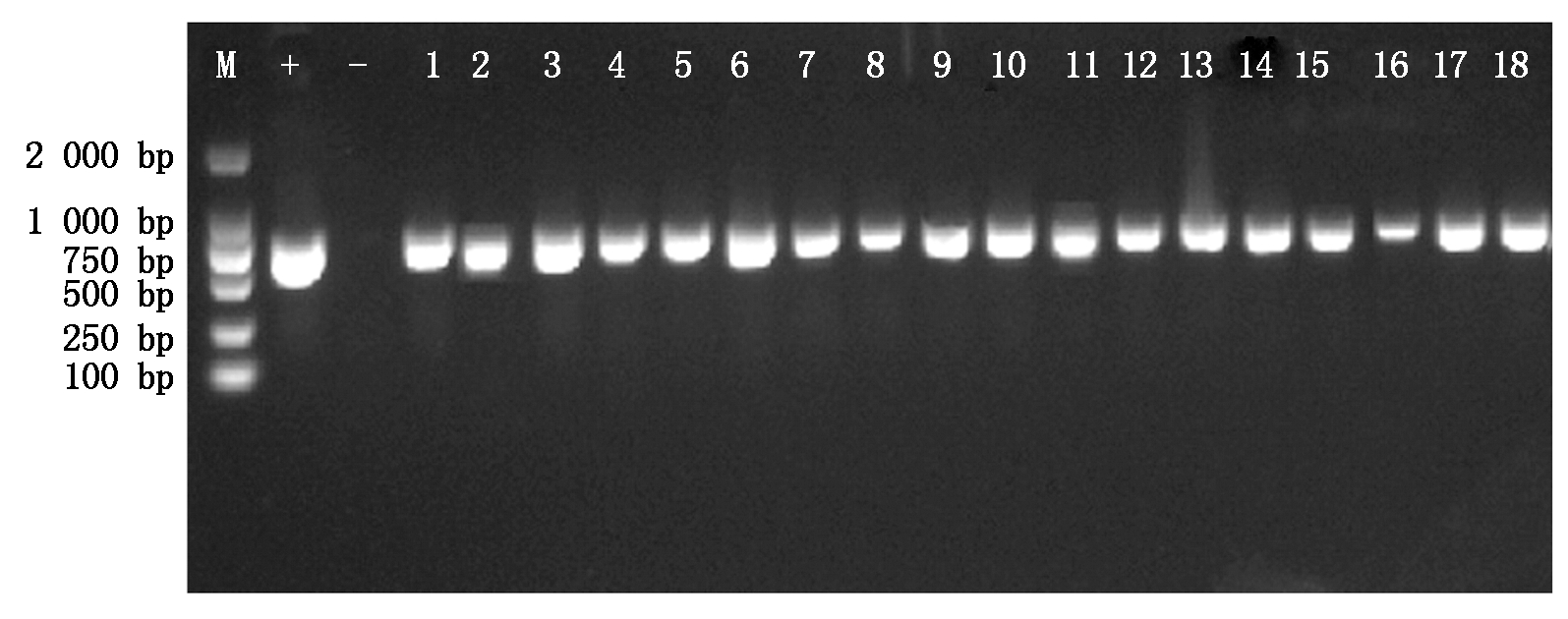
M.DNA Marker DL2000;+.以质粒为阳性对照;-.以野生型对照DNA为阴性对照;1-6.转PmSOC1-1基因拟南芥植株;7-12.转PmSOC1-2基因拟南芥植株;13-18.转PmSOC1-3基因拟南芥植株。
M.DNA Marker DL2000;+.Positive control;-.Negative control;1-6.Transgenic Arabidopsis plants overexpressing PmSOC1-1;7-12.Transgenic Arabidopsis plants overexpressing PmSOC1-2;13-18.Transgenic Arabidopsis plants overexpressing PmSOC1-3.
图1 拟南芥转基因株系的筛选和鉴定
Fig.1 Selection and identification of transgenic lines of Arabidopsis
采用RT-PCR和qRT-PCR对PmSOC1-1、PmSOC1-2和PmSOC1-3基因在转基因拟南芥中是否表达及表达量高低进行了分析。由图2可以看出,每个转入拟南芥的基因在6个株系中都有表达;但是荧光定量的结果显示,不同转基因株系的外源基因表达量存在差异。在转PmSOC1-1基因和PmSOC1-2基因的株系中,根据转入基因的表达量明显可以分为2组,表达量达到显著差异(P<0.05)。转PmSOC1-3的6个株系中,除#12株系外,其他株系的表达量无显著差异。
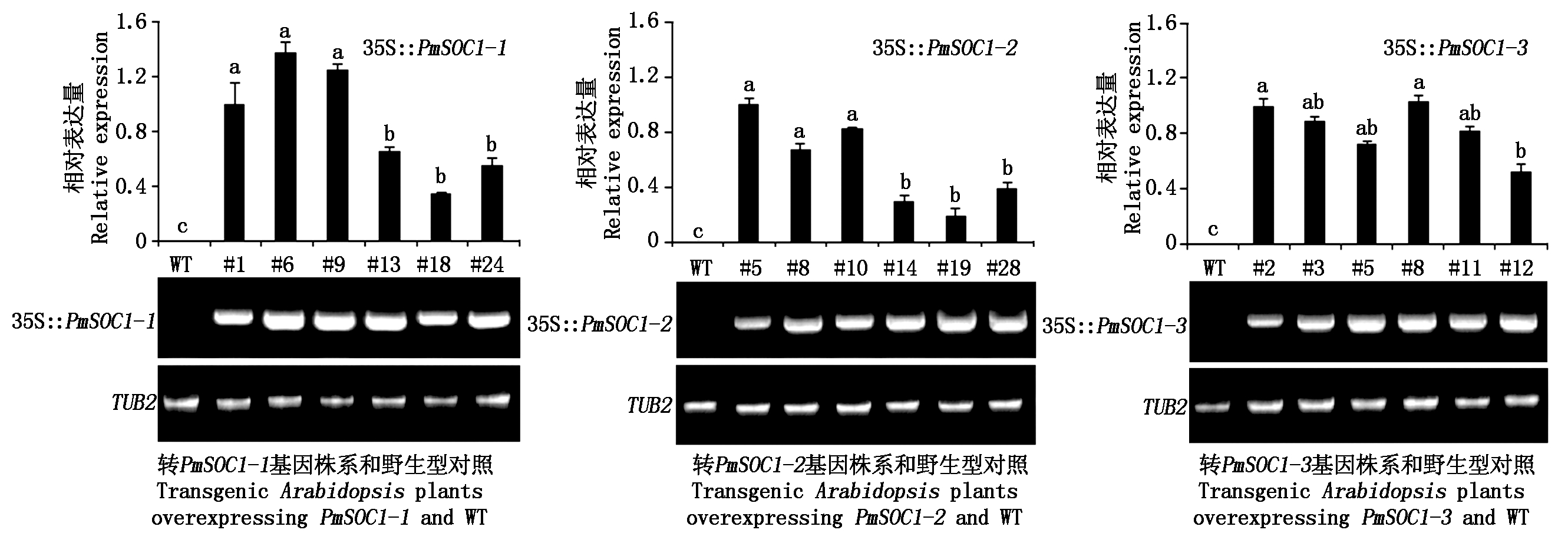
qRT-PCR检测(上);RT-PCR检测(下);不同小写字母表示差异达显著水平(P<0.05);WT.野生型拟南芥。
qRT-PCR detection(above); RT-PCR detection(below); Different lowercase letters indicate a significant difference at P<0.05; WT.Wild-type Arabidopsis.
图2 梅花PmSOC1-like基因在转基因拟南芥中的表达
Fig.2 Expression patterns of floral genes in transgenic Arabidopsis
2.2 过表达PmSOC1-like基因促使拟南芥提前开花
通过对转基因拟南芥T3和野生型的表型观察,除35S::PmSOC1-3的转基因株系表型较为一致外,35S::PmSOC1-1和35S::PmSOC1-2的转基因株系表型在花期提前的程度和表型变异的强弱方面表现出了强表现型和弱表现型2种表型。其中,35S::PmSOC1-1中的#1、#6、#9为强表现型,#13、#18、#24为弱表现型;35S::PmSOC1-2中的#5、#8、#10为强表现型,#14、#19、#28为弱表现型。这与之前利用qRT-PCR检测转入基因的表达量的结果相一致。即PmSOC1-1和PmSOC1-2的强表现型转基因株系其转入基因的表达量也高(图2),这说明转入基因的表达丰度与转基因植株的表型变异存在正相关关系。
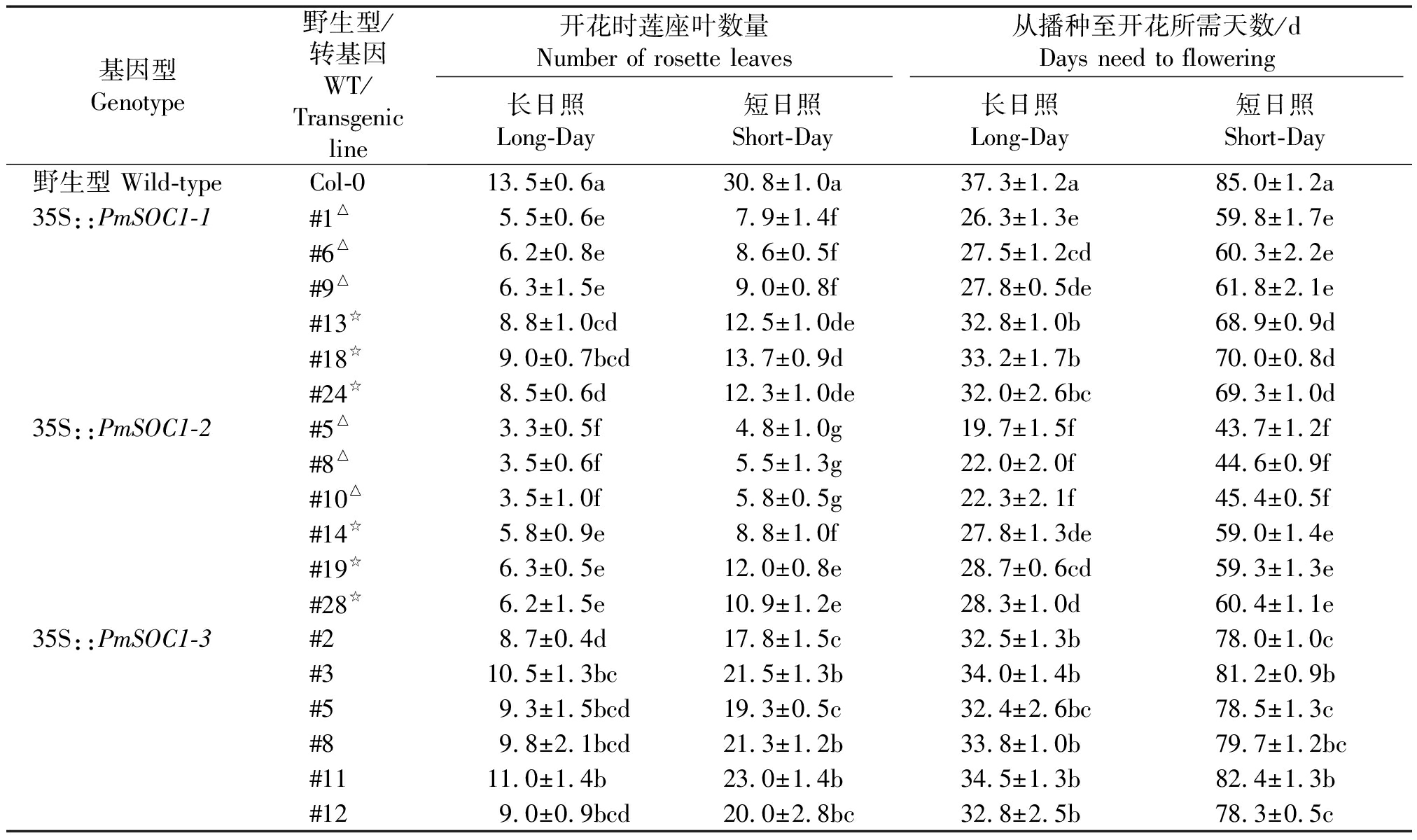
表2 转PmSOC1-like基因拟南芥开花表型
Tab.2 Flowering phenotypes of PmSOC1-like transgenic Arabidopsis
基因型Genotype野生型/转基因WT/Transgenicline开花时莲座叶数量Number of rosette leaves 从播种至开花所需天数/dDays need to flowering 长日照Long-Day短日照Short-Day长日照Long-Day短日照Short-Day野生型 Wild-typeCol-013.5±0.6a30.8±1.0a37.3±1.2a85.0±1.2a35S::PmSOC1-1#1△5.5±0.6e7.9±1.4f26.3±1.3e59.8±1.7e#6△6.2±0.8e8.6±0.5f27.5±1.2cd60.3±2.2e#9△6.3±1.5e9.0±0.8f27.8±0.5de61.8±2.1e#13☆8.8±1.0cd12.5±1.0de32.8±1.0b68.9±0.9d#18☆9.0±0.7bcd13.7±0.9d33.2±1.7b70.0±0.8d#24☆8.5±0.6d12.3±1.0de32.0±2.6bc69.3±1.0d35S::PmSOC1-2#5△3.3±0.5f4.8±1.0g19.7±1.5f43.7±1.2f#8△3.5±0.6f5.5±1.3g22.0±2.0f44.6±0.9f#10△3.5±1.0f5.8±0.5g22.3±2.1f45.4±0.5f#14☆5.8±0.9e8.8±1.0f27.8±1.3de59.0±1.4e#19☆6.3±0.5e12.0±0.8e28.7±0.6cd59.3±1.3e#28☆6.2±1.5e10.9±1.2e28.3±1.0d60.4±1.1e35S::PmSOC1-3#28.7±0.4d17.8±1.5c32.5±1.3b78.0±1.0c#310.5±1.3bc21.5±1.3b34.0±1.4b81.2±0.9b#59.3±1.5bcd19.3±0.5c32.4±2.6bc78.5±1.3c#89.8±2.1bcd21.3±1.2b33.8±1.0b79.7±1.2bc#1111.0±1.4b23.0±1.4b34.5±1.3b82.4±1.3b#129.0±0.9bcd20.0±2.8bc32.8±2.5b78.3±0.5c
注: 表中数据为开花所需天数和莲座叶数量的平均值±标准差;同一列的不同小写字母为P<0.05的显著性差异;Col-0.野生型拟南芥哥伦比亚Columbia-0;△.强表现型;☆.弱表现型。
Note:Values indicate the mean ± standard deviation of days need to flower and number of rosette leaves when flowering; Different lowercase letters in the same colume indicate a significant difference at P<0.05;Col-0.Wild-type A. thaliana ecotype Columbia-0; △.Strong phenotype; ☆.Weak phenotype.
在长日照条件下,与野生型拟南芥植株需长出13片莲座叶才能抽薹相比,35S::PmSOC1-1转基因株系强表现型的植株开花提前明显,在5片莲座叶即可开花(图3-A1),而弱表现型的植株在长出8片莲座叶也开始抽薹开花。35S::PmSOC1-2转基因株系的强表现型植株在所有转基因株系中开花提前最为明显,从表2可以看出,无论是在长日照条件还是短日照条件下,均能使植株开花时间大幅提前,个别转基因株系在长日照条件下长出3片莲座叶时即已开花(图3-A2)。弱表现型植株在平均长出6片莲座叶时也能够开花。35S::PmSOC1-3转基因株系在长日照条件下需要32.4~34.5 d开花,与野生型需要大约37.3 d开花相比略有提前(表2)。以上结果说明3个PmSOC1-like基因都可以促使拟南芥提前开花,但作用强度存在差异。

A1~I1.转PmSOC1-1拟南芥表型;A2~I2.转PmSOC1-2拟南芥表型;A3~G3.转PmSOC1-3拟南芥表型;A0~G0.野生型拟南芥表型;A1,A2,A3.长日照条件下与野生型拟南芥(A0)相比,播种30 d时的35S::PmSOC1-1,35S::PmSOC1-2和35S::PmSOC1-3的早花表型;B1和B2.35S::PmSOC1-1和35S::PmSOC1-2花瓣细丝状;B3~C3,B0~C0.35S::PmSOC1-3和野生型拟南芥的正常花瓣;C1和C2.35S::PmSOC1-1和35S::PmSOC1-2雌蕊伸出花被外;D3和D0.35S::PmSOC1-3和野生型拟南芥的正常雌蕊;D1~E1,D2~E2.35S::PmSOC1-1和35S::PmSOC1-2花萼叶片状;G3和G0.35S::PmSOC1-3和野生型拟南芥的正常花萼;F1和F2.35S::PmSOC1-1和35S::PmSOC1-2的花萼、花瓣在果荚伸长期宿存不脱落;E3和E0.35S::PmSOC1-3和野生型拟南芥正常果荚,花萼和花瓣均已脱落;G2.35S::PmSOC1-2植株一级侧枝向水平方向生长;H1~I1.35S::PmSOC1-1花瓣变为绿色;H2~I2.35S::PmSOC1-2 顶生花的不同形态。
A1-I1.Phenotypes of 35S::PmSOC1-1 transgenic A. thaliana plant; A2-I2.Phenotypes of 35S::PmSOC1-2 transgenic A.thaliana plant; A3-G3.Phenotypes of 35S::PmSOC1-3 transgenic A. thaliana plants; A0-G0.Wild-type A.thaliana plants; A1, A2, A3.35S::PmSOC1-1, 35S::PmSOC1-2, and 35S::PmSOC1-3 transgenic A.thaliana plants, early flowering in comparison with A0 at 30 d after sowing under long-day conditions; B1 and B2.Flower of 35S::PmSOC1-1 and 35S::PmSOC1-2 transgenic A.thaliana, respectively, with petals into filament-like structures; B3-C3, B0-C0.Flower from 35S::PmSOC1-3 transgenic and wild-type A. thaliana, respectively, with the normal petal; C1and C2.Flower of 35S::PmSOC1-1 and 35S::PmSOC1-2 transgenic A. thaliana, respectively, with large carpel out of perianth; D3 and D0.Flower from 35S::PmSOC1-3 transgenic and wild-type A.thaliana, respectively, with the normal carpel; D1-E1, D2-E2.Flower of 35S::PmSOC1-1 and 35S::PmSOC1-2 transgenic A. thaliana, respectively, with sepals into leaf-like plant; G3 and G0.Flower from 35S::PmSOC1-3 transgenic and wild-type A.thaliana, respectively, with the normal sepals; F1 and F2.Flower of 35S::PmSOC1-1 and 35S::PmSOC1-2 transgenic A. thaliana, respectively, with petals and sepals abscission inhibited at silique elongation stage; E3 and E0.Flower from 35S::PmSOC1-3 transgenic and wild-type A.thaliana, respectively, with all organs falling from green silique at the same development stage with F1 and F2; G2.35S::PmSOC1-2 transgenic A.thaliana plants, horizontal growth of branches in comparison with G1, F3, and F0; H1-I1.Flower of 35S::PmSOC1-1 transgenic A. thaliana with greenish sepaloid petal; H2-I2.Flower of 35S::PmSOC1-2 transgenic A.thaliana with different morphologies of terminal flowers.
图3 转PmSOC1-like基因拟南芥表型
Fig.3 Phenotypes of transgenic Arabidopsis overexpressing PmSOC1-like genes
2.3 过表达PmSOC1-1和PmSOC1-2基因影响拟南芥花器官发育
在获得的35S::PmSOC1-3拟南芥转基因株系中,与野生型拟南芥相比(图3-A0-G0),除花期略有提前外,未发现其他明显差异(图3-A3-G3)。qRT-PCR分析表明,PmSOC1-3转基因株系中有强弱不同的表达(图2),暗示35S::PmSOC1-3在转基因株系中有稳定表达。35S::PmSOC1-1和35S::PmSOC1-2的转基因株系在诱导拟南芥提前开花中表现出的强弱2种表型,在花型、花色和株型等方面与野生型拟南芥相比也相应地表现出强弱2种不同表型。与野生型相比,35S::PmSOC1-1和35S::PmSOC1-2转基因株系的弱表现型除35S::PmSOC1-2的花瓣细化更明显外,其他变异基本一致,都发生了花瓣细丝状,花瓣檐部直立,不能平展成十字形(图3-B1,B2);雌蕊心皮长度变长,伸出花被外(图3-C1,C2);花萼叶片状(图3-D1,E1,D2,E2),花瓣和花萼在果荚伸长期宿存(图3-F1,F2)等变异。它们的强表现型则表现出不同的变异。35S::PmSOC1-1转基因株系的强表现型植株在发生了弱表现型的变异基础上,与野生型相比,还发生了花瓣变为绿色的变异(图3-H1,I1)。35S::PmSOC1-2转基因株系的强表现型植株除了发生弱表现型的变异外,还在植株形态建成方面发生了明显变化,即所有主茎上的分枝(茎生分枝)都向水平方向生长(图3-G2),从而使拟南芥的株型彻底发生了改变。此外,35S::PmSOC1-2强表现型植株的部分花序顶端发生了由2~4朵花合生形成顶生花(Terminal flower,简称TF)的现象,花序顶端TF的花器官数目不定,且通常最先开放(图3-H2,I2)。以上表型说明,PmSOC1-1和PmSOC1-2可能具有调节花器官发育的功能。除此之外,PmSOC1-2还具有将花序顶端分生组织转变为花分生组织的功能。
2.4 转PmSOC1-like基因对拟南芥植株中相关开花基因的调控
通过对拟南芥的研究表明,SOC1基因整合了多条开花途径的开花信号,并通过上调其下游花分生组织特性基因来促进植物开花[26-27]。由于将3个PmSOC1-like基因导入拟南芥都表现出了早花表型,为了验证这种早花表型是否是由导入的PmSOC1-like基因上调了拟南芥中的下游花分生组织特性基因引起的。利用qRT-PCR技术检测了LFY、AP1、AGL24和FUL基因在强表现型转基因拟南芥种子萌发后的第6,12,16天的表达情况。结果表明,LFY基因在35S::PmSOC1-2转基因株系中的表达量显著高于野生型拟南芥和其他2个PmSOC1-like转基因株系(35S::PmSOC1-1和35S::Pm-SOC1-3)(P<0.05),并一直维持较高水平(图4)。与此同时,AP1基因的表达量也表现出了与LFY基因较为一致的变化趋势(图4)。而与SOC1基因功能相似的AGL24基因和另一个促进花起始发育的FUL基因[28-30]的表达水平则表现出了随着植株生长发育逐步上升的相似规律(图4),特别是在种子萌发后的第16天,AGL24和FUL基因的表达量明显上升,且在不同转基因株系中的表达量存在显著差异(P<0.05)(图4-C,D)。
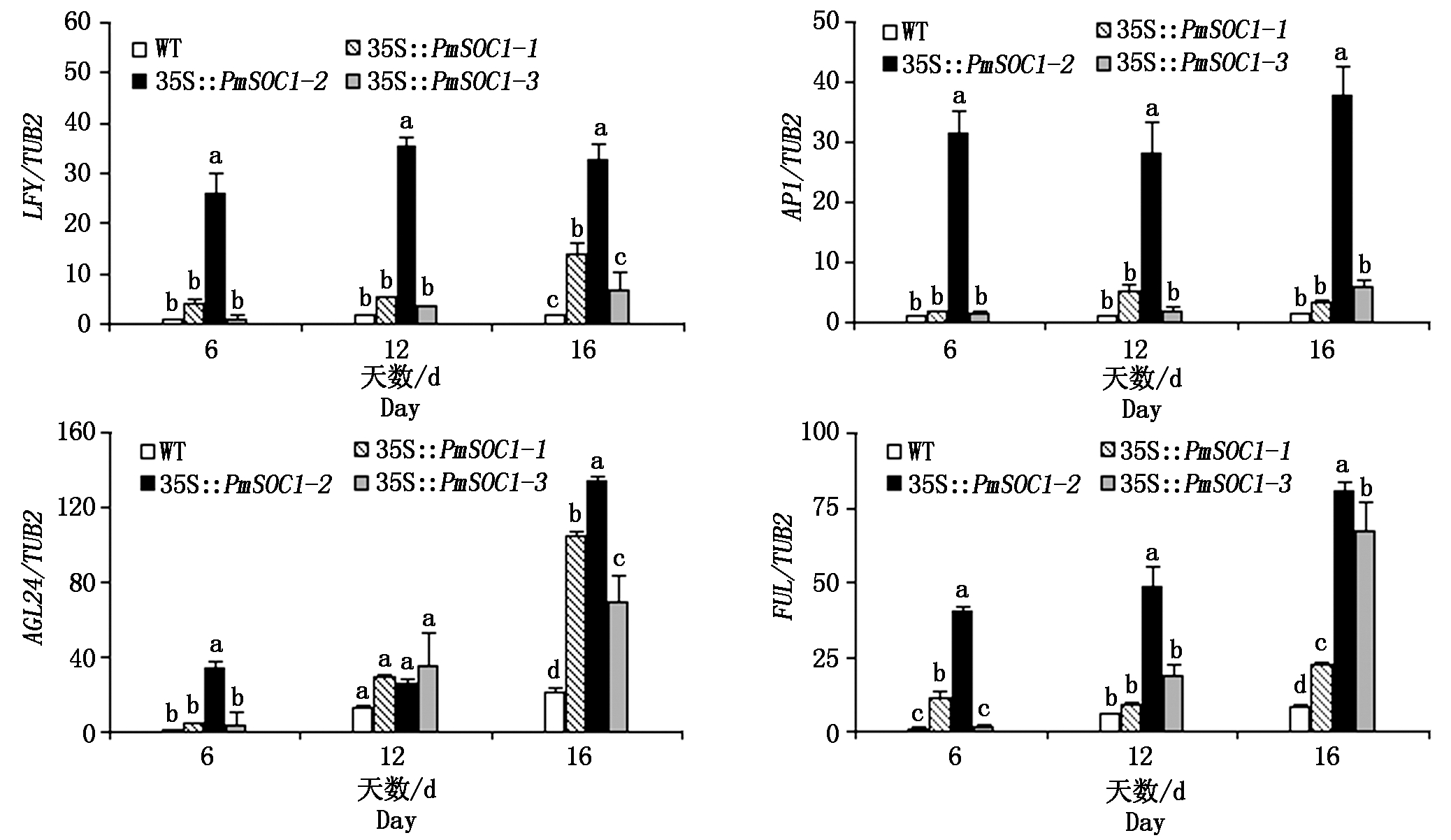
以拟南芥TUB2为内参基因;误差线代表标准误;柱形图上方的不同小写字母表示差异达显著水平(P<0.05)。
Relative expression levels were normalized to A. thaliana TUB2 expression; Error bars indicate the standard errors;
Different lowercase letters above the column diagram indicate a significant difference at P<0.05.
图4 PmSOC1-like转化拟南芥植株在播种后6,12,16 d基因表达qRT-PCR分析
Fig.4 Expression analyses of different flowering time genes in PmSOC1-like transgenic
A.thaliana seedlings grown for days 6, 12, and 16 in long-day conditions.
3 结论与讨论
在拟南芥成花转变过程中SOC1是最早在分生组织中出现表达量上调的基因,而这种表达有效地促进了早花。目前在其他物种中鉴定到的SOC1同源基因在拟南芥中的过表达也都一致地表现出了早花表型。如菊花(Chrysanthemum)中的ClSOC1-1和ClSOC1-2[11]、葡萄(Grapevine)中的VvMADS8[13]、桉树(Eustoma grandiflorumin)中的EgSOC1[10]、柑橘(Citrus)中的CsSL1[31]在拟南芥中的过表达都能够使拟南芥提前开花。大豆(Glycine max)中GmGAL1和兰花(Orchid)中的DOSOC1的过表达不但能够使野生型拟南芥的开花时间提前,而且还能够互补soc1突变体的部分晚花表型[9,32]。在本研究中,转PmSOC1-like基因拟南芥无论在长日照还是短日照条件下,都表现出早花,暗示着SOC1/TM3家族基因在促进开花方面具有保守性。证明了SOC1基因在各个物种中功能的保守性。
SOC1基因除了具有促进开花的功能,在很多物种中还表现出影响花发育的功能。拟南芥中AtSOC1的过表达会产生绿色的花萼状花瓣和长度伸长的雌蕊[21]。非洲菊中GhSOC1的过表达没有促进植物开花,但是却导致了花瓣颜色和形状的改变[8]。矮牵牛PhUNS基因在拟南芥中的过表达导致花器官中出现了本应在叶片和茎中生长的表皮毛,并致使花瓣转变为类似叶片的结构[7]。在石斛兰中,SOC1的同源基因DOSOC1的过表达影响了花器官的正常发育,造成了顶端花芽只能发育出不健全的初始花被[9]。同样的,本研究中PmSOC1-1和PmSOC1-2在拟南芥中的过表达导致拟南芥花器官出现了花瓣细丝状,花萼叶片状等表型。其中PmSOC1-1的过表达还产生了绿色的花瓣和长度伸长的雌蕊,这与AtSOC1过表达产生的表型一致。而PmSOC1-2的过表达则表现出花序顶端出现2~3朵花合生的表型。以上结果表明,PmSOC1-like基因与拟南芥中SOC1基因一样,不但能够促进开花,还具有影响花器官发育的功能。
前人研究表明,SOC1基因主要是通过上调其下游花分生组织特性基因来促进植物开花。如在成花转变过程中,SOC1与AGL24之间能够形成异质二聚体激活LFY基因的表达,从而促进开花[27]。类似的作用机理也出现在FUL和SOC1之间,有研究发现,FUL和SOC1能够在细胞核中形成二聚体,并且二者通过绑定到LFY启动子的相同区域发挥作用。因此SOC1,AGL24和FUL之间可以形成冗余二聚体,或更高阶的分子复合物,以确保通过启动LFY基因来激活其他花分生组织基因[33]。菊花中的ClSOC1和石斛兰中的DOSOC1在拟南芥中的过表达能够上调AGL24和LFY等开花促进因子的表达[9,11]。本研究中,利用qRT-PCR技术检测转基因拟南芥中SOC1下游基因LFY、AP1、AGL24和FUL的结果显示,与野生型相比,PmSOC1-like的引入使LFY、AP1、AGL24和FUL的表达量在不同阶段都有不同程度提高。其中AGL24和FUL的表达趋势相似,均呈现逐步上升趋势(从种子萌发后的第6天到第12天),暗示SOC1与AGL24和FUL之间可能形成了二聚体来激活LFY基因表达从而促进开花。这从转基因拟南芥中LFY的表达水平中可以得到验证;另外,在拟南芥中,FUL基因具有调节果实的发育和伸长的功能,本研究中转PmSOC1-1和PmSOC1-2拟南芥中雌蕊均有所伸长,可能与激活FUL表达有关。提前开花最显著、表型出现TF的转PmSOC1-2株系,其LFY和AP1的表达量最高,说明PmSOC1-2基因可能通过与AGL24和FUL作用有效地激活了LFY基因的表达,从而促进花序分生组织转变为花分生组织。然而需要更多的证据来验证这一假设。
本研究结果为利用SOC1基因改变梅花花期提供了理论依据,为采用转基因技术实现梅花成花阶段转变的人为控制奠定了基础。但是,花期转变是由很多因素共同调控的,单个基因的研究不能脱离整体,如何构建以梅花SOC1基因为中心的多方向的成花调控体系,研究梅花SOC1基因与其他开花相关基因的作用关系,阐明梅花成花机理将是今后的重点研究方向。
[1] Distelfeld A, Li C, Dubcovsky J.Regulation of flowering in temperate cereals[J].Current Opinion in Plant Biology,2009,12(2):178-184. doi:10.1016/j.pbi.2008.12.010.
[2] Greenup A, Peacock W J, Dennis E S, Trevaskis B. The molecular biology of seasonal flowering-responses in Arabidopsis and the cereals[J].Annals of Botany,2009,103(8):1165-1172.doi:10.1093/aob/mcp063.
[3] Trevaskis B, Hemming M N, Dennis E S,Peacock W J.The molecular basis of vernalization-induced flowering in cereals[J].Trends in Plant Science,2007,12(8):352-357. doi:10.1016/j.tplants.2007.06.010.
[4] 胡丽芳,金志强,徐碧玉.MADS-box基因对花的发育及开花早晚的影响[J].生命科学研究,2004,8(4):7-12.doi:10.16605/j.cnki.1007-7847.2004.s2.002.
Hu L F, Jin Z Q, Xu B Y. The effect of MADS-box gene to the development of flower and flowering-time[J]. Life Science Research, 2004,8(4):7-12.
[5] Moon J, Suh S S, Lee H,Choi K R, Hong C B, Paek N C, Kim S G, Lee I.The SOC1 MADS-box gene integrates vernalization and gibberellin signals for flowering in Arabidopsis[J].The Plant Journal, 2003,35(4):613-623. doi:10.1046/j.1365-313X.2003.01833.x.
[6] Samach A, Onouchi H, Gold S E, Ditta G S, Schwarz-Sommer Z, Yanofsky M F, Coupland G. Distinct roles of CONSTANS target genes in reproductive development of Arabidopsis[J]. Science, 2000,288(5471):1613-1616. doi:10.1126/science.288.5471.1613.
[7] Ferrario S, Busscher J, Franken J, Gerats T, Vandenbussche M, Angenent G C, Immink R G H.Ectopic expression of the petunia MADS box gene UNSHAVEN accelerates flowering and confers leaf-like characteristics to floral organs in a dominant-negative manner[J]. The Plant Cell, 2004,16(6):1490-1505. doi:10.1105/tpc.019679.
[8] Ruokolainen S, Ng Y P, Albert V A, Elomaa P, Teeri T H. Over-expression of the Gerbera hybrida At-SOC1-like1 gene Gh-SOC1 leads to floral organ identity deterioration[J]. Annals of Botany, 2011, 107(9):1491-1499. doi:10.1093/aob/mcr112.
[9] Ding L H, Wang Y W, Yu H.Overexpression of DOSOC1, an ortholog of Arabidopsis SOC1, promotes flowering in the Orchid dendrobium Chao Parya Smile[J].Plant and Cell Physiology, 2013,54(4):595-608. doi:10.1093/pcp/pct026.
[10] Nakano Y, Kawashima H, Kinoshita T, Yoshikawa H, Hisamatsu T. Characterization of FLC, SOC1 and FT homologs in Eustoma grandiflorum:effects of vernalization and post-vernalization conditions on flowering and gene expression[J]. Physiologia Plantarum, 2011, 141(4):383-393. doi:10.1111/j.1399-3054.2011.01447.x.
[11] Fu J X, Qi S, Yang L W, Dai Y, Dai S L.Characterization of chrysanthemum ClSOC1-1 and ClSOC1-2, homologous genes of SOC1[J].Plant Molecular Biology Reporter, 2013,32(3):740-749. doi:10.1007/s11105-013-0679-8.
[12] Cseke L J, Zheng J, Podila G K.Characterization of PTM5 in aspen trees:a MADS-box gene expressed during woody vascular development[J].Gene, 2003,318:55-67. doi:10.1016/s0378-1119(03)00765-0.
[13] Sreekantan L, Thomas M R. VvFT and VvMADS8, the grapevine homologues of the floral integrators FT and SOC1, have unique expression patterns in grapevine and hasten flowering in Arabidopsis[J].Functional Plant Biology, 2006,33(12):1129-1139. doi:10.1071/FP06144.
[14] 刘传娇,王顺利,薛璟祺,朱富勇,任秀霞,李名扬,张秀新.牡丹开花调控转录因子基因PrSOC1的克隆与表达分析[J].园艺学报,2014,41(11):2259-2267. doi:10.16420/j.issn.0513-353x.2014.11.013.
Liu C J, Wang S L, Xue J Q, Zhu F Y, Ren X X, Li M Y, Zhang X X. Molecular cloning and expression analysis of the flowering-regulating transcription factor PRSOC1 gene in tree peony[J]. Acta Horticulturae Sinica, 2014,41(11):2259-2267.
[15] 魏军亚,唐杰,刘国银,刘德兵,陈业渊. 芒果MSOC1基因的克隆与表达分析[J]. 西北植物学报, 2015, 35(6):1092-1097. doi:10.7606/j.issn.1000-4025.2015.06.1092.
Wei J Y, Tang J, Liu G Y, Liu D B, Chen Y Y. Cloning and expression analysis of MSOCl gene in mango (Mangifera indica L.)[J]. Acta Botanica Boreali-Occidentalia Sinica, 2015, 35(6):1092-1097.
[16] 付德山,朱珠,崔丽婷,詹亚光,齐凤慧. 水曲柳SOC1基因表达载体的构建及生物信息学分析[J]. 西南林业大学学报, 2015,35(6):6-13. doi:10.11929/j.issn.2095-1914.2015.06.002.
Fu D S, Zhu Z, Cui L T, Zhan Y G, Qi F H. Construction of SOC1 gene expression vector and bioinformatic analysis in Fraxinus mandschurica[J]. Journal of Southwest Forestry University, 2015,35(6):6-13.
[17] 贾丝俏, 穆致远,李思峰,袁建波,韩烈保,晁跃辉. 蒺藜苜蓿SOC1基因克隆及转化[J]. 中国草地学报, 2018, 40(5):25-30. doi:10.16742/j.zgcdxb.2018-05-04.
Jia S Q, Mu Z Y, Li S F, Yuan J B, Han L B, Chao Y H. Cloning and transformation of SOC1 gene in Medicago truncatula[J]. Chinese Journal of Grassland, 2018, 40(5):25-30.
[18] 武丹,吴菁华,张志忠. 中国水仙NtSOC1基因克隆及亚细胞定位[J]. 西北植物学报, 2017,37(10):1889-1895. doi:10.7606/j.issn.1000-4025.2017.10.1889.
Wu D, Wu J H, Zhang Z Z. Cloning and sukrcellular localization of NtSOCl gene from Narcissus tazetta var. chinensis[J]. Acta Botanica Boreali-Occidentalia Sinica, 2017,37(10):1889-1895.
[19] 马广莹,朱开元,史小华,邹清成,刘慧春,詹菁,田丹青. 红掌2个SOC1基因的克隆、序列与表达分析[J]. 浙江大学学报(农业与生命科学版), 2017, 43(3):289-297. doi:10.3785/j.issn.1008-9209.2016.10.111.
Ma G Y, Zhu K Y, Shi X H, Zou Q C, Liu H C, Zhan J, Tian D Q. Cloning, sequence and expression analysis of two SOC1 genes from Anthurium[J]. Journal of Zhejiang University (Agric and Life Sci), 2017,43(3):289-297.
[20] 严佳文,解璞,袁启凤,肖图舰,毛永亚,马玉华. 火龙果开花调控转录因子基因HpSOC1的克隆与表达分析[J]. 植物生理学报, 2018, 54(10):1561-1568. doi:10.13592/j.cnki.ppj.2018.0341.
Yan J W, Xie P, Yuan Q F, Xiao T J, Mao Y Y, Ma Y H. Molecular cloning and expression analysis of the flowering regulating transcription factor HpSOC1 gene in Hylocereus polyrhizus[J]. Plant Physiology Journal, 2018, 54(10):1561-1568.
[21] Borner R, Kampmann G, Chandler J, Gleissner R, Wisman E, Apel K, Melzer S. A MADS domain gene involved in the transition to flowering in Arabidopsis[J]. The Plant Journal, 2000, 24(5):591-599. doi:10.1046/j.1365-313x.2000.00906.x.
[22] Jaudal M, Zhang L L, Che C, Li G F, Tang Y H, Wen J Q, Mysore K S, Putterill J. A SOC1-like gene MtSOC1a promotes flowering and primary stem elongation in Medicago[J]. Journal of Experimental Botany, 2018,69(20):4867-4880. doi:10.1093/jxb/ery284.
[23] 陈俊愉. 中国梅花[M]. 海口:中国海南出版社, 1996:12-58.
Chen J Y. Chinese mei flowers[M].Haikou: Hainan Publishing House,China,1996:12-58.
[24] 李玉舒,杨炜茹,程堂仁,王佳,张启翔. 梅花PmSOC1-like基因的克隆与表达分析[J]. 华北农学报, 2016, 31(5):78-85. doi:10.7668/hbnxb.2016.05.012.
Li Y S, Yang W R, Cheng T R, Wang J, Zhang Q X. Molecular cloning and expression analysis of flowering-regulating transcription factor PmSOC1-like gene in Prunus mume[J]. Acta Agriculturae Boreali-Sinica, 2016, 31(5):78-85.
[25] Clough S J, Bent A F. Floral dip:a simplified method for Agrobacterium-mediated transformation of Arabidopsis thaliana[J]. The Plant Journal, 1998, 16(6):735-743. doi:10.1046/j.1365-313x.1998.00343.x.
[26] Lee J, Lee I. Regulation and function of SOC1, a flowering pathway integrator[J]. Journal of Experimental Botany, 2010, 61(9):2247-2254. doi:10.1103/PhysRevA.57.2379.
[27] Lee J, Oh M, Park H, Lee I. SOC1 translocated to the nucleus by interaction with AGL24 directly regulates LEAFY[J].The Plant Journal, 2008, 55(5):832-843. doi:10.1111/j.1365-313X.2008.03552.x.
[28] Liu C, Chen H Y, Er H L, Soo H M, Kumar P P, Han J H, Liou Y H, Yu H. Direct interaction of AGL24 and SOC1 integrates flowering signals in Arabidopsis[J]. Development, 2008, 135:1481-1491. doi:10.1242/dev.020255.
[29] Michaels S D, Ditta G, Gustafson-Brown C, Pelaz S, Yanofsky M, Amasino R M. AGL24 acts as a promoter of flowering in Arabidopsis and is positively regulated by vernalization[J].The Plant Journal, 2003, 33(5):867-874.doi:10.1046/j.1365-313X.2003.01671.x.
[30] Yu H, Xu Y F, Tan E L, Kumar P P. AGAMOUS-LIKE 24, a dosage-dependent mediator of the flowering signals[J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(25):16336-16341. doi:10.1073/pnas.212624599.
[31] Tan F C, Swain S M. Functional characterization of AP3, SOC1 and WUS homologues from citrus (Citrus sinensis)[J]. Physiologia Plantarum, 2007, 131(3):481-495. doi:10.1111/j.1399-3054.2007.00971.x.
[32] Zhong X F, Dai X, Xü J H, Wu H Y, Liu B, Li H Y. Cloning and expression analysis of GmGAL1, SOC1 homolog gene in soybean[J]. Molecular Biology Reports, 2012, 39(6):6967-6974. doi:10.1007/s11033-012-1524-0.
[33] Balanzà V, Martí nez-Fernández I, Ferrándiz C. Sequential action of FRUITFULL as a modulator of the activity of the floral regulators SVP and SOC1[J]. Journal of Experimental Botany, 2014, 65(4):1193-1203. doi:10.1093/jxb/ert482.