糖是植物光合作用的产物,是植物碳和能量代谢的重要载体,对植物生长和发育起着非常重要的作用。同时,糖也可作为信号转导的信号分子,参与和调控植物的各种代谢途径,如参与调控植物组织渗透压和植物逆境胁迫[1]。在植物中,糖主要以蔗糖、多元醇或低聚糖的形式从源组织(如叶子)输送到库组织(如根、种子和其他生殖组织)。蔗糖作为植物中的主要碳水化合物,主要在叶肉细胞中合成,进入韧皮部后,通过长距离运输系统运输到库组织邻近的韧皮部,通过胞间连丝形成的共质体途径或通过蔗糖转运蛋白、细胞壁转化酶和单糖转运体介导的非共质体的途径进入库组织中[2]。植物组织获取由蔗糖在质外体中水解成的己糖(如葡萄糖、果糖)需要由一组转运蛋白参与调控,如拟南芥中的糖转运蛋白、水稻中的单糖转运蛋白(Monosaccharide transporters,MSTs)、葡萄中的己糖转运蛋白(Hexose transporters,HTs),这些蛋白家族通过参与糖转运进而调控植物碳分配、花器官发育、作物产量和环境适应性[3-5]。
STP蛋白属于主要协助转运蛋白超家族(Major facilitator superfamily,MFS),是一类通常由12个跨膜结构域组成的H+/糖或Na+/糖协同转运蛋白[6-8]。自1989年从小球藻中克隆出第一个STP基因以来[9],研究者先后在拟南芥、水稻、葡萄、西红柿、梨、木薯、甘蓝中发现了一系列STP基因[10-17]。STP蛋白的主要特征是定位于细胞膜上进行等离子转运,且具有广谱的单糖,如葡萄糖、果糖、半乳糖、木糖和甘露醇运载能力[10,18]。已有研究表明,STP基因家族在植物不同生长阶段和不同的胁迫下表现出不同的表达模式。如在拟南芥中,AtSTP1主要在萌发的种子、幼苗和保卫细胞中表达[19-20];AtSTP2、AtSTP6、 AtSTP9、AtSTP11特异性地在花粉中表达,而AtSTP4在根中高表达[10, 21];AtSTP13定位于叶脉和叶肉细胞,可能参与蔗糖从细胞质到叶脉的运输[22]。在水稻中,STP基因OsMST3在根木质部特异性表达,参与积累植物细胞壁增厚阶段细胞壁合成所需的单糖[13]。除此之外,STP基因家族也会对环境压力,如机械损伤、线虫和细菌侵染,做出响应[21-24]。
高粱(Sorghum bicolor)是全世界种植的第五大禾谷类作物,是重要的经济作物、能源作物和饲料作物,特别在干旱和半干旱地区农业生产中具有重要地位。高粱是C4植物,具有高光效、易栽培、适应性强、营养价值高、适口性好及抗旱、耐盐碱等多重抗逆的特性。自高粱基因组测序以来[25],大量基于基因组信息的基因研究得以开展。鉴于STP基因家族在植物的生长发育和逆境胁迫中的重要作用,本研究利用生物信息学手段在全基因组水平上分析和鉴定高粱STP 基因家族,解析STP家族基因的染色体定位、分类与表达模式,为进一步研究该家族成员的功能及高粱高产、广适分子改良提供科学依据。
1 材料和方法
1.1 试验材料
高粱全基因组数据、蛋白质序列、cDNA数据均来自于Phytozome数据库(https://phytozome.jgi.doe.gov/pz/portal.html),水稻、拟南芥STP蛋白序列分别来自RGAP 数据库(http://rice.plantbiology.msu.edu/index.shtml)、TAIR数据库(http://www.Arabidopsis.org)。
1.2 高粱STP基因家族的鉴定
从Pfam数据库(http://pfam.xfam.org/)中下载STP 基因家族的隐马尔科夫模型文件Sugar_tr结构域(PF00083)。利用文件中高粱STP 基因家族的蛋白保守序列,使用HMMER 软件(http://www.hmmer.org/)和NCBI(http://www.ncbi.nlm.nih.gov/)数据库tBlastN功能获取高粱STP 基因家族的蛋白保守序列。对获取的序列使用Pfam和Smart(http://smart.embl-heidelberg.de/)程序检测STP核心序列,除去无STP典型结构域、结构域不完整和冗余序列,之后与14个拟南芥STP基因蛋白序列和28个水稻STP基因蛋白序列进行比对,获得高粱STP基因家族序列。
1.3 高粱STP基因家族蛋白理化性质分析、亚细胞定位和跨膜结构分析
利用Expasy 数据库(https://web.expasy.org/protparam/)蛋白质的氨基酸数目、理论等电点、分子质量、疏水性、脂肪族氨基酸指数等理化性质信息。利用 Cell-PLoc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)对家族成员进行亚细胞定位,利用TMHMM 2.0(http://www.cbs.dtu.dk/services/TMHMM/)进行跨膜结构区域分析。
1.4 高粱STP基因家族基因定位
利用高粱STP基因在染色体上的位置信息,并从SYMAP数据库(http://www.agcol.arizona.edu/software/symap/)下载高粱基因组重复片段位置信息,利用RCircos工具对STP基因在染色体上的位置及高粱染色体重复片段进行可视化[26]。
1.5 高粱STP基因家族多序列比对、基因分组和进化树分析
利用高粱STP基因家族的保守域序列,使用ClustalW软件采用默认参数进行多序列比对。所有鉴定出来的高粱STP基因通过保守域序列比对和蛋白质序列比对结果进行分组。利用拟南芥、水稻和高粱STP蛋白,使用ClustalW进行多序列比对,然后利用MEGA 6.0程序,采用邻接法(Neighbor-joining method, NJ)构建系统进化树,参数设置如下:Bootstrap method, 1 500 bootstrap replications, Poisson model, pairwise deletion。
1.6 高粱STP基因家族基因结构和保守结构域分析
利用MEME 4.12.0(http://meme-suite.org/tools/meme)对STP基因家族的保守基序进行分析,并用SMART的Squence analysis在线软件比对数据库,确认基序的种类。利用 TBtools软件Gene Viewer工具绘制高粱STP基因保守域和基因结构示意图[27]。
1.7 高粱STP基因重复、共线性分析
使用MCScanX(Multiple collinearity scan toolkit)软件,采用默认参数来分析高粱STP基因的复制事件,并分析高粱与拟南芥和高粱与水稻之间的基因同源性。根据高粱与拟南芥和高粱与水稻STP基因的直系同源关系,利用TBtools软件中的Multiple synteney plotter工具绘制高粱、拟南芥和水稻STP基因的共线性比较图谱[27]。使用DnaSP 5.1软件对发生复制事件的基因进行非同义替换(Non-synonymous substitution, ka)和同义替换(Synonymous substitution, ks)分析[28]。
1.8 高粱STP基因家族的表达分析
高粱RNA-Seq数据来源于Davidson等[29]2012年公开发表的数据和GEO数据库中的RNA-Seq数据(GEO登录号:GSE30249)[30]。Davidson等[29]发表的高粱RNA-Seq数据包括高粱叶片(Leaves)、原生花序(Primodial inflorescences)、花序(Inflorescences)、花药(Anther)、雌蕊(Pistil)、种子(Seed)、胚(Embryo)、胚乳(Endosperm)共8个组织的RNA-Seq数据;GSE30249数据集包括不同浓度ABA和PEG处理条件下的根(Root)和茎(Shoot)的RNA-Seq数据。下载以上数据集中RNA-Seq 数据,利用TBtools软件中Table ID Extractor and Filter工具提取STP基因的表达量(Fragments per kilobase of exon per million fragments mapped, FPKM)值[24],对STP 基因表达量进行标准化,然后利用TBtools软件中The Amazing Heatmap 工具绘制STP基因表达热图[27]。
表1 高粱STP 基因家族理化性质分析
Tab.1 Analysis of physical and chemical characteristics of STP gene family in Sorghum bicolor

名称Name基因序列号Gene ID染色体Chromosome氨基酸数目/个Number of amino acid分子质量/kuMolecular weight理论等电点Theoretical pI脂肪族氨基酸指数Aliphatic index蛋白质疏水性GRAVYSTP保守域位置STP domain location跨膜结构域Transmembrane domainsSbSTP1Sobic.001G190200153356.728.90106.470.6329-49511SbSTP2Sobic.001G297600152658.029.29105.080.4930-49512SbSTP3Sobic.001G454100151756.989.04108.380.4930-48412SbSTP4Sobic.001G543500153858.539.12105.110.5030-49611SbSTP5Sobic.002G004900252056.899.25106.130.5027-48711SbSTP6Sobic.002G073600251256.069.13101.190.5028-4909SbSTP7Sobic.002G201900251855.779.30106.780.5631-48811SbSTP8Sobic.002G338500253157.649.03105.760.5831-48811SbSTP9Sobic.003G192200350955.739.39109.570.6924-48611SbSTP10Sobic.004G049300452157.248.9199.600.5828-49011SbSTP11Sobic.004G332700452355.639.12102.750.5830-49011SbSTP12Sobic.005G171200552456.069.40101.660.5128-49812SbSTP13Sobic.006G101900654257.739.77101.730.6060-52111SbSTP14Sobic.006G102300651155.339.40105.280.6125-48811SbSTP15Sobic.006G102400652155.699.2599.620.5336-49910SbSTP16Sobic.006G102800651354.049.18104.890.6429-49110SbSTP17Sobic.007G061800752156.268.0698.080.5527-48311SbSTP18Sobic.009G152800948051.538.73101.770.4229-44910SbSTP19Sobic.010G0306001052154.589.81104.490.5928-49612
2 结果与分析
2.1 高粱STP基因的鉴定和理化性质分析
通过HMMER软件对高粱全部蛋白序列进行搜索并在NCBI数据库中进行tBlastN比对,鉴定STP基因蛋白核心结构域,并剔除冗余序列和无完整读码框序列,最终获得19个高粱STP基因(表1)。根据STP基因在基因组上的位置,命名为SbSTP1~SbSTP19。
高粱STP基因理化性质分析表明:SbSTP基因编码蛋白氨基酸序列的长度较为接近,其中最长的是SbSTP13,为542个氨基酸(Amino acid, aa),最短的是SbSTP18,为480个aa,平均长度为520.1个aa。STP蛋白分子质量为51.53(SbSTP18)~58.53 ku(SbSTP4)。蛋白质理论等电点均大于7,表明高粱STP家族含有大量碱性氨基酸,其中SbSTP19等电点最高,为9.81。STP家族的脂肪族氨基酸指数分布在98.08(SbSTP17)~109.57(SbSTP9),平均为103.91,表明该家族蛋白质热稳定性较为一致。蛋白质疏水性均大于0,表明该家族蛋白均为疏水性蛋白质。STP保守域长度集中在461~467个aa,平均为460.68个aa,其中SbSTP18保守域最短(421个aa),SbSTP12保守域最长(471个aa)。SbSTP蛋白含有丰富的跨膜结构域,为9(SbSTP6)~12个,其中有11个蛋白含有11个跨膜结构域。
2.2 高粱STP基因家族基因定位
高粱STP基因不均匀分布在除第8号染色体外的其余9条染色体上,且多分布于染色体两端(图1)。其中第3,5,7,9,10号染色体上均含有1个STP基因;第4号染色体上含有2个STP基因;其余3条染色体均含有4个STP基因,其中第6号染色体上的4个STP基因在染色体上成簇分布。比较高粱STP基因在染色体上的位置和高粱基因组重复区域的位置发现,SbSTP2、SbSTP11、SbSTP16和SbSTP18位于基因组上的4个重复区域,但在基因组重复区均只有1个拷贝。
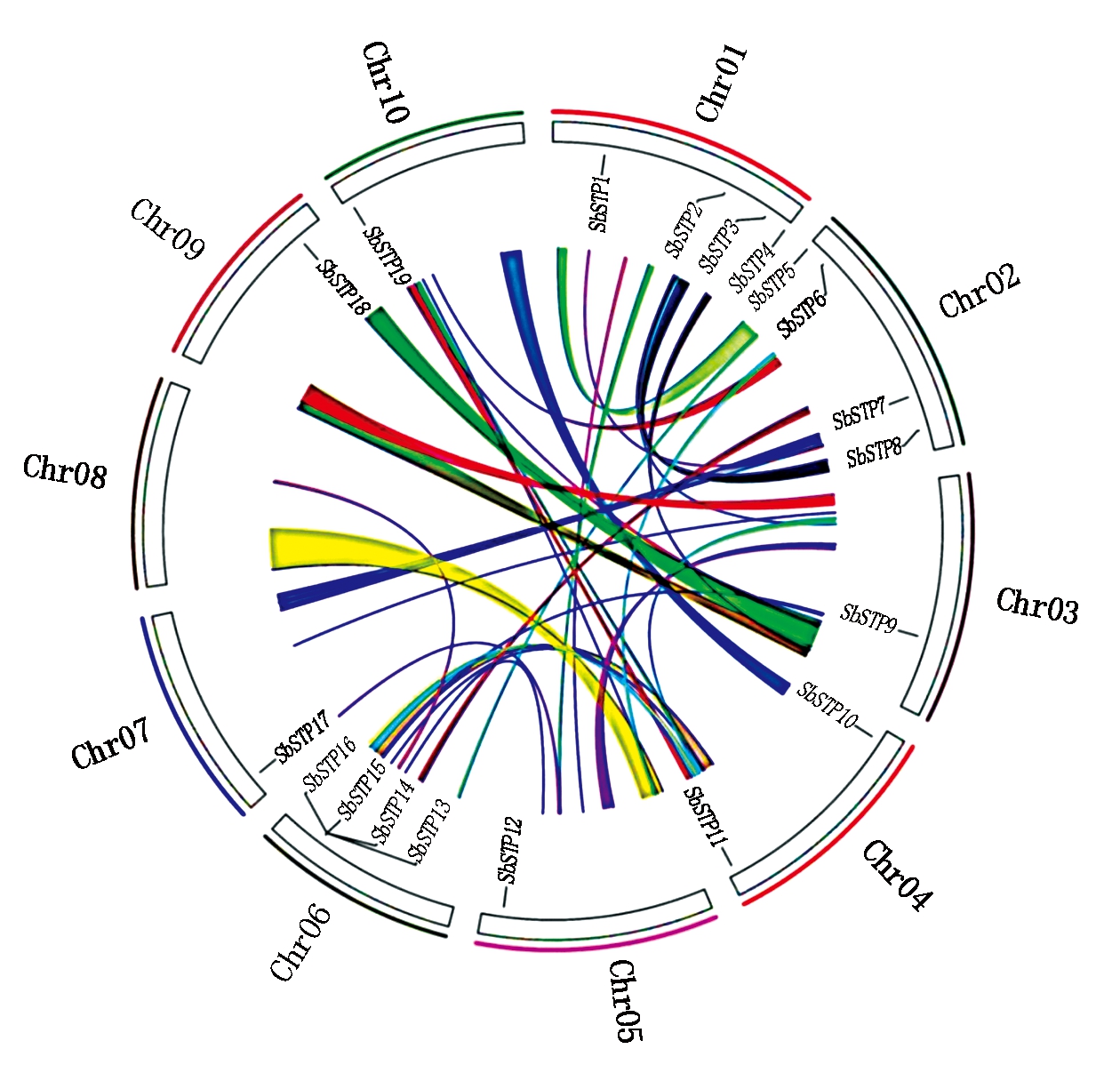
图1 高粱STP 基因的染色体定位
Fig.1 Chromosome location of STP genes in Sorghum bicolor genome
2.3 高粱STP基因家族多序列比对、基因分组和进化树分析
利用高粱STP蛋白的STP保守域序列和全长序列分别进行序列比对和进化分析,结果表明,采用2种方法高粱STP基因的进化关系一致,这表明高粱STP基因家族高度保守。根据高粱STP基因家族的进化树,可将其分为5组,其中Ⅰ组成员最多,为7个,所有成员均含有11个跨膜结构域,Ⅱ组成员最少,仅有1个,SbSTP7(图2)。

图2 高粱STP基因家族系统进化树
Fig.2 Phylogenetic analysis of STP gene family in Sorghum bicolor
利用高粱、拟南芥和水稻的STP蛋白序列进行系统进化分析,结果表明,STP基因家族可以分为6组,其中高粱STP成员分布于Ⅰ~Ⅴ组,与高粱STP家族进化树分组一致;Ⅰ~Ⅴ组中均含有SbSTP、AtSTP和OsSTP,其中Ⅰ组由7个SbSTP(SbSTP1、SbSTP5、SbSTP8、SbSTP9、SbSTP10、SbSTP11、SbSTP17)、8个OsSTP和6个AtSTP, Ⅱ组中含有SbSTP(SbSTP7)、AtSTP和OsSTP各1个,Ⅲ组中含有2个SbSTP(SbSTP3、SbSTP6)、4个AtSTP和2个OsSTP,Ⅳ组中含有4个SbSTP(SbSTP13、SbSTP14、SbSTP15、SbSTP16)、1个AtSTP和9个OsSTP,Ⅴ组中含有5个SbSTP(SbSTP2、SbSTP4、SbSTP12、SbSTP18、SbSTP19)、1个AtSTP和7个OsSTP ;Ⅵ组中仅含有AtSTP和OsSTP各1个。这表明,STP基因的形成早于拟南芥、水稻和高粱的分化,在高粱形成过程中可能发生过STP基因的丢失事件,且在Ⅰ~Ⅴ组中,高粱STP与水稻STP的遗传距离更近。
2.4 高粱STP家族基因结构及蛋白基序分析
基因结构分析表明,SbSTP11、SbSTP18不存在上下游调控区,SbSTP1不存在上游调控区,SbSTP6、SbSTP12、SbSTP16、SbSTP19不存在下游调控区,其余12个成员均含有上下游调控区(图3)。在内含子和外显子数量上高粱STP基因间存在较大差异:内含子方面,除SbSTP17不含有内含子外,其余成员内含子数量为1~4个,其中SbSTP6、SbSTP8、SbSTP11、SbSTP16、SbSTP19均含有1个内含子,SbSTP18含有4个内含子;外显子方面,大部分成员含有2~4个外显子,其中SbSTP17仅含有1个外显子,SbSTP18外显子数量最多为5个。
蛋白基序分析表明:共有12个蛋白基序被鉴定出来,长度为15~80个aa,其中基序1、2、3、6、7是STP蛋白核心结构域的组成部分,在19个高粱STP蛋白中高度保守(图3、表2)。其中,SbSTP18缺失了基序6,含有11个基序;其余成员均含有12个蛋白基序,且进化树上同组的家族成员大部分具有相同或类似的基序类型和排列顺序。
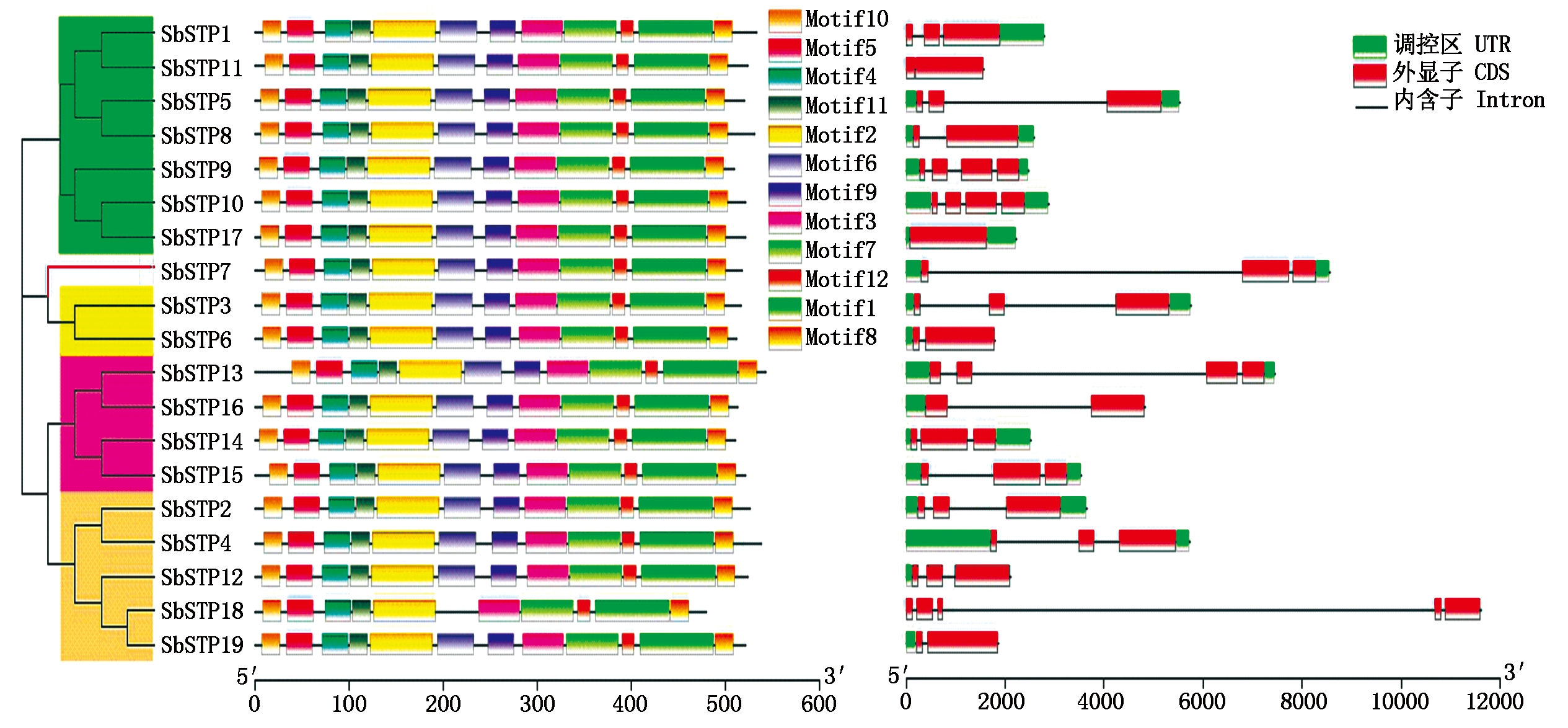
图3 高粱STP基因结构及蛋白基序分布
Fig.3 Intron-exon structure and protein motif compositions of STP gene family in Sorghum bicolor
表2 高粱STP蛋白中鉴定出来的12个基序
Tab.2 Twelve motifs observed in Sorghum bicolor STP proteins

基序Motif序列Sequence长度/个WidthPfam 结构域Pfam domain1WSWGPLGWLVPSEIFPLEIRSAGQSITVAVNMLFTFVIAQAFLSMLCHLKFGJFFFFAAWVVVMTLFVALFLPETKGVPI80Sugar_tr2GAALNGAAVNVAMLIVGRILLGVGVGFANQAVPLYLSEMAPARLRGALNIGFQLMITIGILAANLINY68Sugar_tr3RYRPQLVMAVLIPFFQQLTGINVIMFYAPVLFRTIGFGSDASLMS45Sugar_tr4NNYCKFDSQLLTAFTSSLYLAGLVASLFA295GLIFGYDIGISGGVTSMDPFLEKFFPSVY296KIIGGWGWRLSLGLAAVPAAIJTLGALFLPDTPNSLIZRGR41Sugar_tr7AVITGLVNVFSTLVSIVTVDRVGRRKLFLQGGVQMJVSQVAVGAIIAAKFGDSGEAT57Sugar_tr8EZMGRVWKKHWFWKRFVADDA219RGTADVDAELDDJVAASEAARAVEHPWRN2910SAGGGKDYPGRVTLFVVLTCJ2111SSVTRRLGRKWSMLIGGVLFL2112RGYAAAVVVLICVYV15
2.5 高粱STP基因重复、共线性分析
在小于200 kb的染色体片段上包含2个或者多个同一家族基因的事件被定义为串联重复事件(Tandem duplications event)[31]。分析发现,6号染色体上存在1个串联重复事件区,基因SbSTP14和SbSTP15为1对串联重复基因。除此之外,1号染色体上还存在1个片段重复事件(Segmental duplication event),基因SbSTP2和SbSTP4为1对片段重复基因,可能由基因重组事件使2个基因重组到同一条染色体上。
为进一步推断高粱STP基因家族的系统发育机制,利用双子叶植物拟南芥、单子叶植物水稻2种模式作物基因组信息,构建了高粱与拟南芥、水稻的基因共线性比较图谱(图4)。共有2个SbSTP基因与拟南芥STP显示出共线性,14个SbSTP基因与水稻STP显示出共线性,与拟南芥和水稻形成的直系同源基因对分别为3,16对。一些SbSTP还与2个基因形成同源基因对,如SbSTP8与2个拟南芥基因AtSTP1和AtSTP12直系同源,SbSTP4、SbSTP13分别与2个水稻STP形成同源基因对,推测这些基因可能在基因家族进化过程中发挥着重要作用。大部分SbSTP仅与水稻形成直系同源基因对,可能表明这些基因在单子叶和双子叶植物分化后形成;而SbSTP2和SbSTP8与拟南芥和水稻STP都存在着直系同源基因,推测它们的形成可能早于单子叶和双子叶的分化,在进化中具有高度的保守性,可能在糖转运中发挥着某些特定的功能。此外,高粱和水稻之间鉴定出来的一些STP同源基因对位于超过100个基因的高度保守的共线性区块上;而在高粱和拟南芥STP基因位于的共线性区块上,同源基因对少于30对,这可能与高粱和水稻、拟南芥的亲缘关系有关。
ka/ks值是基因异义替换(ka)和同义替换(ks)的比值,可用于判断选择压力在基因进化中的作用[32]。高粱STP的串联重复和片段重复基因对及大多数高粱与水稻、拟南芥的STP直系同源基因对的ka/ks值均小于1,这表明高粱STP基因家族在进化过程中可能受到了强烈的纯化选择压力。
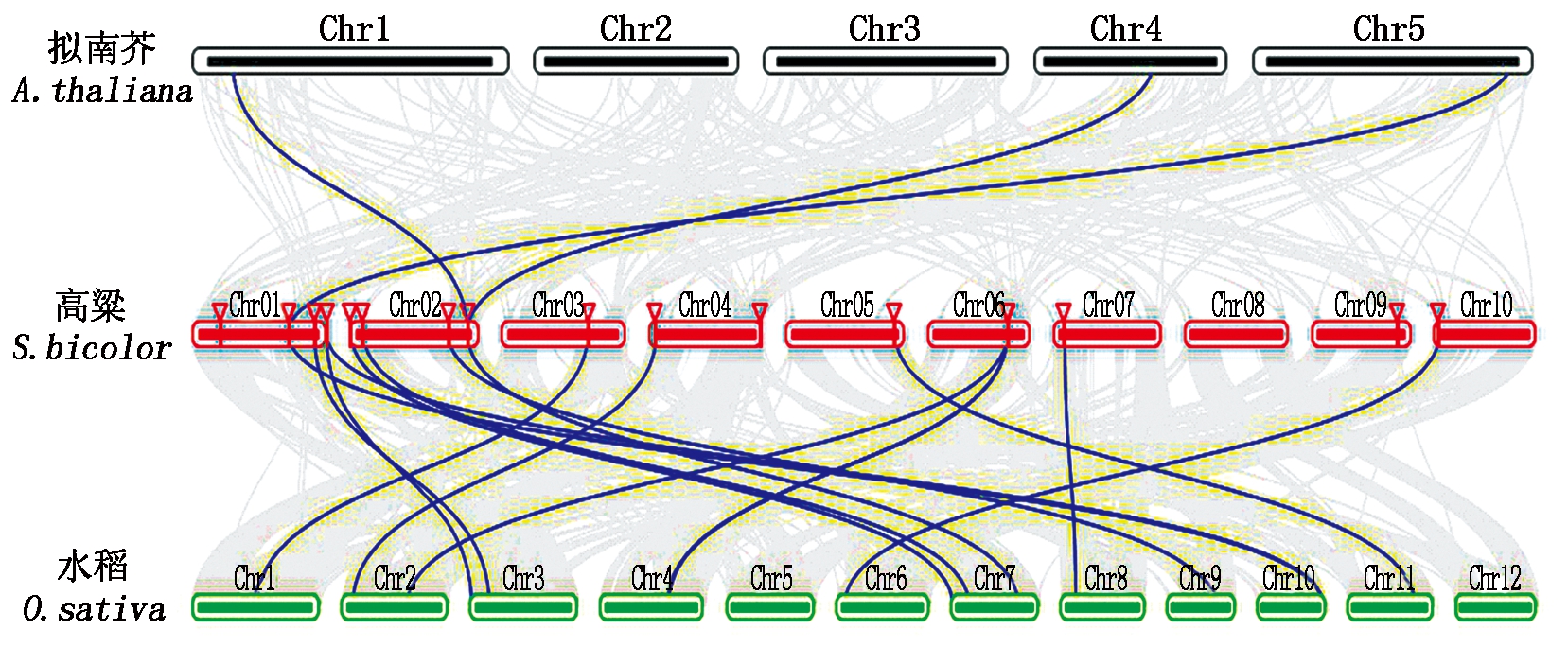
图4 高粱与拟南芥和水稻的共线性图谱
Fig.4 Synteny analysis of STP genes between Sorghum bicolor and Arabidopsis thaliana, Oryza sativa
2.6 高粱STP基因表达分析
来源于GEO数据库的10个高粱组织RNA-Seq数据[29-30]被用来分析高粱STP基因的表达模式。在所有高粱STP基因中,至少有18个SbSTP基因可能表达,其中SbSTP18在所有检测样本中均未表达,推测该基因为假基因或者具备特殊的表达时序。分析结果表明,高粱STP基因在不同的组织中表现出不同的表达方式(图5)。有4个基因(SbSTP2、SbSTP4、SbSTP7、SbSTP8)在所有样本中都有表达(FPKM>0),其中有3个(SbSTP2、SbSTP4、SbSTP7)为组成型表达(在所有样本中FPKM>1)。SbSTP11特异性在花中表达;SbSTP7在叶片中高表达;4个基因(SbSTP6、SbSTP9、SbSTP10、SbSTP17)在花序中高表达;2个基因(SbSTP3、SbSTP5)在不同浓度ABA胁迫下的根中高表达,2个基因(SbSTP5、SbSTP13)在PEG处理下的根中高表达,SbSTP4在ABA和PEG处理的茎秆中高表达。
3 结论与讨论
STP基因在模式植物,如拟南芥和水稻中,已经被证明在糖转运中发挥着重要作用[10-11, 33]。然而,STP基因家族在其他作物,如高粱中的全基因组的表达模式还缺乏研究。利用高粱基因组数据,本研究鉴定了高粱STP基因家族,对基因理化性质、染色体上的定位、结构、基因的共线性和重复进行了分析,并对SbSTP的表达模式进行了初步研究。
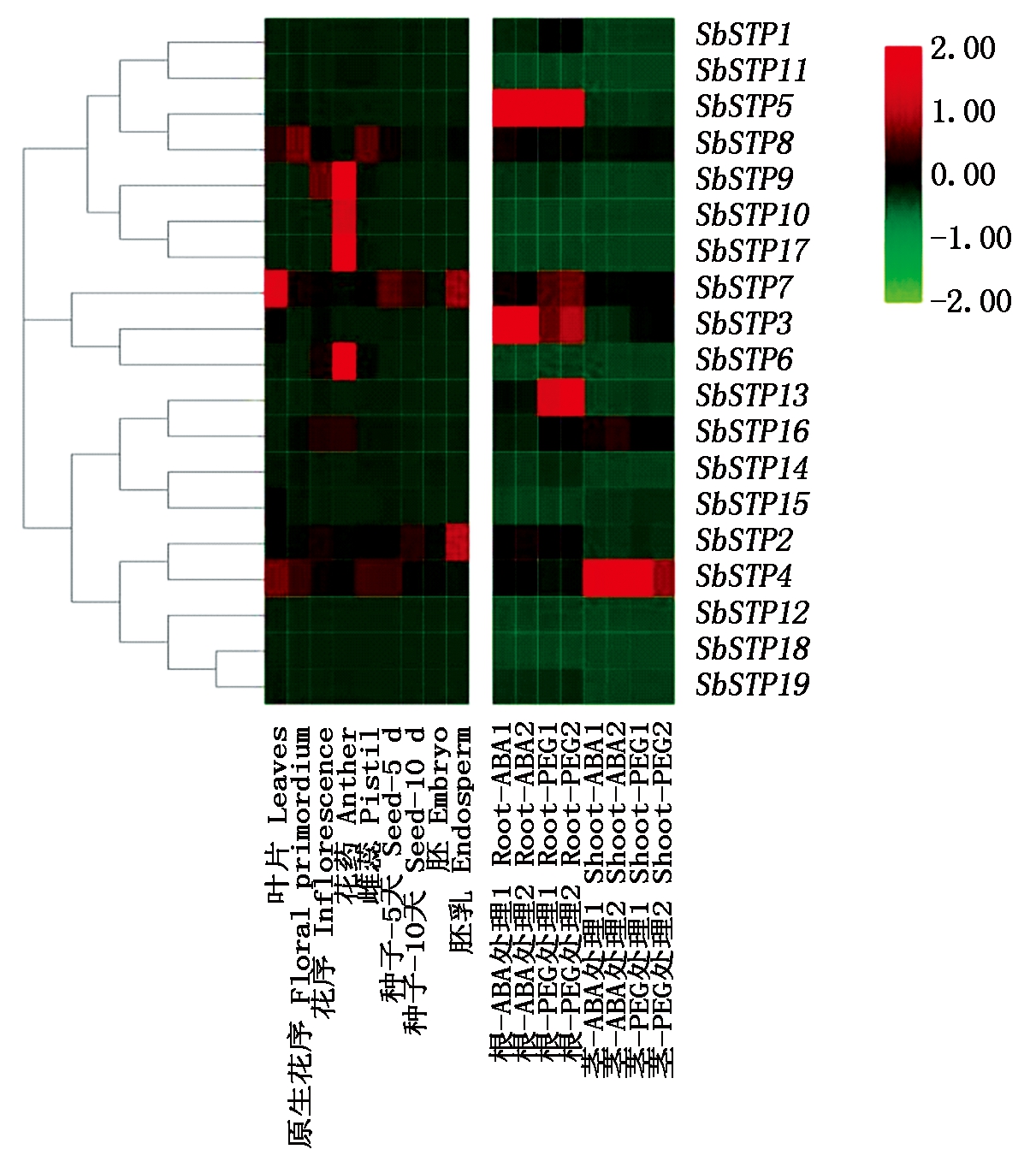
图5 高粱STP基因的表达模式
Fig.5 The expression profile of SbSTP genes in Sorghum bicolor
利用HMMER软件和Blast搜索,从高粱基因组中共鉴定了19个不均匀分布于9条染色体上的SbSTP。所有的高粱STP蛋白都含有1个属于MFS超家族的Sugar_tr结构域(PF00083)。植物中的MFS载体通常具有12个跨膜结构域(TMD1-TMD12),可以被细分为N端跨膜结构域(TMD1-TMD6)和C端跨膜结构域(TMD7-TMD12)[6, 34]。而在本研究中,仅有4个SbSTP具有12个TMDs,大部分SbSTP含有11个TMDs,缺失N端第4个或C端第9个TMD;其中SbSTP6最少,含有9个TMDs,在N端缺失了第4和第6个TMD,在C端缺失了第9个TMD。这些结果表明,在进化过程中,大部分SbSTP蛋白可能在N端或(和)C端区域发生了TMD缺失事件,并且在高粱中的TMD缺失要明显高于木薯、葡萄和番茄中STP蛋白TMD的缺失[13-14, 16]。在基因结构上,SbSTP外显子数量为1~5个,以2~4个居多,数量上表现出丰富的变异性,在拟南芥、葡萄、梨STP基因中也有类似的现象[10, 12-13, 15],由 Xu等[35]对基因结构研究结果推测,STP基因结构的差异性可能是进化过程中内含子和外显子的缺失或插入等变异造成基因结构分化的结果。
系统进化分析表明,高粱、拟南芥和水稻的全部STP蛋白可被分为6组,其中SbSTP位于Ⅰ~Ⅴ组,每组中都含有AtSTP和OsSTP,表明在这3个物种中STP蛋白关系密切,具有高度保守的特点。此外,在进化树上,每个SbSTP都与1个OsSTP组成1个最小的进化分支,并且在SbSTP形成的19个最小分支中,13个分支上的 SbSTP和OsSTP同时为直系同源基因对,这表明高粱STP与水稻具有更近的亲缘关系,这与高粱全基因组研究结果一致[25]。在STP成员数量上,高粱STP成员数量明显少于由同一祖先分化而来的水稻,表明禾本科祖先在由水稻向高粱进化过程中STP基因发生了大量的基因丢失事件[25]。在木薯、梨、水稻的进化过程中,片段复制和串联重复在STP家族的扩张和功能分化中发挥了重要作用[11, 15-16],而在高粱的STP基因进化上片段复制和串联重复各仅有一次,且在进化过程中SbSTP家族成员呈现丢失缩减的特征,这对SbSTP功能的影响还有待进一步探讨。
系统进化上位于同一分支的STP蛋白可能具有相似的生化特性[16, 36]。在Ⅰ组中,SbSTP8的直系同源基因,AtSTP1特异性在保卫细胞中表达,参与拟南芥碳素获取,并且可能参与保卫细胞的渗透调节[19];AtSTP11是一种在花粉管上特异性表达的单糖载体,参与花粉管发育过程中的单糖运输[37]。在Ⅲ组中,AtSTP6与SbSTP6在同一分支上,是一种高亲和性的可转运多种单糖H+/糖转运蛋白,主要在花粉发育后期表达[38];同样的AtSTP13与SbSTP3位于同一分支,被认为是在维管束组织中表达的特异性己糖/ H+转运蛋白,可能通过参与碳、氮代谢,来调节植物的生物产量[39]。在Ⅴ组中,AtSTP3是SbSTP2的直系同源基因,是由绿色叶片损害诱导的低亲和力、能量依赖的H+同向转运载体[40]。拟南芥中这些已知功能的STP,能够为进化上较近的高粱STP基因功能预测和研究提供一定的参考。
STP基因植物单糖分配中发挥着重要作用,进而参与到植物生长和发育过程中的不同代谢途径。本研究利用公共数据库中的高粱RNA-Seq数据对SbSTP的表达模式进行了研究[26-27],SbSTP在不同高粱组织中表现出不同的表达模式。同样,AtSTP在不同的拟南芥组织中也表现出不同的表达模式,如AtSTP1主要在根中表达,是从胞外吸收己糖的主要载体[10,19];AtSTP3在叶中特异性表达,可能参与到从细胞质渗漏胞外糖的转运[36]。在本研究中,一些基因也表现出组织特异性表达,如SbSTP11特异性在花中表达;一些基因还在某些组织中表现出高表达,如SbSTP7在叶片中高表达,SbSTP6、SbSTP9、SbSTP10、SbSTP17在花序中高表达;此外,SbSTP5在ABA和PEG诱导的根中高表达,SbSTP4在ABA和PEG诱导的茎秆中高表达,推测这些基因可能参与到由ABA参与的或干旱引起的逆境应答。SbSTP表达模式的分析为这些基因在特定组织中的潜在功能提供了证据。
综上所述,本研究对高粱STP基因组家族进行了综合分析,家族成员可以分为5组,在同一组上的家族成员具有相同或类似的基序类型和排列顺序。对3个物种的系统进化分析和共线性分析,为高粱STP基因家族的进化特征提供了有价值的线索。系统发育和基因表达分析,有助于揭示高粱STP基因的功能。SbSTP基因在不同组织和诱导处理下的表达模式表明这些基因可能在高粱的生长发育过程中发挥着重要作用。研究结果为进一步研究单个SbSTP基因的生物学功能,挖掘SbSTP家族的应用潜力奠定了基础。
[1] Guo C Y, Li H Y, Xia X Y, Liu X Y, Yang L.Functional and evolution characterization of SWEET sugar transporters in Ananas comosus[J].Biochemical and Biophysical Research Communications,2018,496(2):407-414.doi:10.1016/j.bbrc.2018.01.024.
[2] Rennie E A, Turgeon R.A comprehensive picture of phloem loading strategies[J].Proceedings of the National Academy of Sciences,2009,106(33):14162-14167. doi: 10.1073/pnas.0902279106.
[3] Ludewig F, Flüegge U I. Role of metabolite transporters in source-sink carbon allocation[J].Frontiers in Plant Science,2013,4:231.doi:10.3389/fpls.2013.00231.
[4] Rottmann T, Fritz C, Sauer N, Stadler R.Glucose uptake via STP transporters inhibits in vitro pollen tube growth in a HEXOKINASE1-Dependent manner in Arabidopsis thaliana[J].The Plant Cell,2018,30(9):2057-2081.doi:10.1105/tpc.18.00356.
[5] 胡丽萍,张峰,徐惠,刘光敏,王亚钦,何洪巨.植物SWEET 基因家族结构,功能及调控研究进展[J].生物技术通报,2017,33(4):27-37. doi:10.13560/j.cnki.biotech.bull.
Hu L P, Zhang F, Xu H, Liu G M, Wang Y Q, He H J. Research advances in the structure,function and regulation of SWEET gene family in plants[J].Biotechnology Bulletin, 2017,33(4):27-37.
[6] Yan N.Structural advances for the major facilitator superfamily(MFS)transporters[J].Trends in Biochemical Sciences,2013,38(3):151-159.doi:10.1016/j.tibs.2013.01.003.
[7] Schulz A, Beyhl D, Marten I, Wormit A, Neuhaus E, Poschet G, Büettner M, Schneider S, Sauer N, Hedrich R.Proton-driven sucrose symport and antiport are provided by the vacuolar transporters SUC4 and TMT1/2[J].Plant Journal,2011,68(1):129-136.doi:10.1111/j.1365-313X.2011.04672.x.
[8] 李纯,孙春玉,陈静,林彦萍,王义,张美萍.主要协同转运蛋白超家族的研究进展[J].生物技术通报,2018,34(8):43-49. doi:10.13560/j.cnki.biotech.bull.1985.2018-0069.
Li C, Sun C Y, Chen J, Lin Y P, Wang Y, Zhang M P.Research advances in the major facilitator superfamily[J].Biotechnology Bulletin,2018,34(8):43-49.
[9] Sauer N, Tanner W.The hexose carrier from Chlorella. cDNA cloning of a eucaryotic H+-cotransporter[J].FEBS Letters,1989,259(1):43-46. doi: 10.1016/0014-5793(89)81489-9.
[10] Büettner M.The Arabidopsis sugar transporter(AtSTP)family: an update[J].Plant Biology,2010,12(1):35-41.doi:10.1111/j.1438-8677.2010.00383.x.
[11] Toyofuku K,Kasahara M, Yamaquchi J. Characterization and expression of monosaccharide transporters.(OsMSTs)in rice[J].Plant and Cell Physiology,2000,41(8):940-947. doi: 10.1093/pcp/pcd016.
[12] Johnson D A, Thomas M A.The monosaccharide transporter gene family in Arabidopsis and rice: A history of duplications, adaptive evolution, and functional divergence[J].Molecular Biology and Evolution,2007,24(11):2412-2423.doi:10.1093/molbev/msm184.
[13] Afoufa-Bastien D, Medici A, Jeauffre J A, Coutos-Tévenot P, Lemoine R, Atanassova R, Laloi M. The vitis vinifera sugar transporter gene family: phylogenetic overview and macroarray expression profiling[J].BMC Plant Biology,2010,10:245.doi:10.1186/1471-2229-10-245.
[14] Reuscher S, Akiyama M, Yasuda T, Makino H, Aoki K, Shibata D, Shiratake K.The sugar transporter inventory of tomato: genome-wide identification and expression analysis[J].Plant and Cell Physiology,2014,55(6):1123-1141.doi:10.1093/pcp/pcu052.
[15] Li J M, Zheng D M, Li L T, Qiao X, Wei S W, Bai B, Zhang S L, Wu J.Genome-Wide function, evolutionary characterization and expression analysis of sugar transporter family genes in pear(Pyrus bretschneideri Rehd)[J].Plant and Cell Physiology,2015,56(9):1721-1737.doi:10.1093/pcp/pcv090.
[16] Liu Q, Dang H J, Chen Z J, Wu J Z, Chen Y, Chen S B, Luo L J.Genome-Wide identification,expression,and functional analysis of the sugar transporter gene family in cassava(Manihot esculenta)[J].International Journal of Molecular Sciences,2018,19(4):10. doi: 10.3390/ijms19040987.
[17] Zhang W, Wang S, Yu F, Tang J, Yu L, Wang H, Li J.Genome-Wide identification and expression profiling of sugar transporter protein(STP)family genes in cabbage(Brassica oleracea var.capitata L.)reveals their involvement in clubroot disease responses[J].Genes,2019,10(1):71. doi: 10.3390/genes10010071.
[18] Paulsen P A, Custódio T F, Pedersen B P.Crystal structure of the plant symporter STP10 illuminates sugar uptake mechanism in monosaccharide transporter superfamily[J].Nature Communications,2019,10(1):10. doi: 10.1038/s41467-018-08176-9.
[19] Otori K, Tanabe N, Tamoi M, Shigeoka S.Sugar transporter protein 1(STP1)contributes to regulation of the genes involved in shoot branching via carbon partitioning in Arabidopsis[J].Bioscience Biotechnology and Biochemistry,2019,83(3):472-481.doi:10.1080/09168451.2018.1550355.
[20] Cordoba E, Aceves-Zamudio D L,Hernández-Bernal A F,Ramos-Vega M,León P.Sugar regulation of SUGAR TRANSPORTER PROTEIN 1(STP1)expression in 1026 Arabidopsis thaliana[J].Journal of Experimental Botany,2015,66(1):147-159. doi: doi.org/10.1093/jxb/eru394.
[21] Truernit E, Schmid J, Epple P, Illig J, Sauer N.The sink-specific and stress-regulated Arabidopsis STP4 gene: enhanced expression of a gene encoding a monosaccharide transporter by wounding, elicitors, and pathogen challenge[J].The Plant Cell,1996,8(12):2169-2182.doi:10.1105/tpc.8.12.2169.
[22] Nørholm M H H,Nour-Eldin H H,Brodersen P,Mundy J,Halkier B A.Expression of the Arabidopsis high-affinity hexose transporter STP13 correlates with programmed cell death[J].FEBS Letters,2006,580(9):2381-2387. doi: 10.1016/j.febslet.2006.03.064.
[23] Mendgen K, Hahn M.Plant infection and the establishment of fungal biotrophy[J].Trends in Plant Science,2002,7(8):352-356.doi:10.1016/S1360-1385(02)02297-5.
[24] Yamada K, Saijo Y, Nakagami H, Takano Y.Regulation of sugar transporter activity for antibacterial defense in Arabidopsis[J].Science,2016,354(6318):1427-1430.doi:10.1126/science.aah5692.
[25] Paterson A H, Bowers J E, Bruggmann R, Dubchak I, Grimwood J, Gundlach H, Haberer G, Hellsten U, Mitros T, Poliakov A, Schmutz J, Spannagl M, Tang H, Wang X, Wicker T, Bharti A K, Chapman J, Feltus F A, Gowik U, Grigoriev I V, Lyons E, Maher C A, Martis M, Narechania A, Otillar R P, Penning B W, Salamov A A, Wang Y, Zhang L, Carpita N C, Freeling M, Gingle A R, Hash C T, Keller B, Klein P, Kresovich S, McCann M C, Ming R, Peterson D G, Mehboobur-Rahman, Ware D, Westhoff P, Mayer K F, Messing J, Rokhsar D S.The Sorghum bicolor genome and the diversification of grasses[J].Nature,2009,457(7229):551-556. doi: 10.1038/nature07723.
[26] Zhang H, Meltzer P, Davis S.RCircos:an R package for Circos 2D track plots[J].BMC Bioinformatics,2013,14:244. doi: 10.1186/1471-2105-14-244.
[27] Chen C J, Xia R, Chen H, He Y H. TBtools, a Toolkit for biologists integrating various HTS-data handling tools with a user-friendly interface[J]. bioRxiv, 2018: 289660. doi: 10.1101/289660.
[28] Librado P, Rozas J.DnaSP v5: a software for comprehensive analysis of DNA polymorphism data[J].Bioinformatics,2009,25(11):1451-1452.doi:10.1093/bioinformatics/btp187.
[29] Davidson R M, Gowda M, Moghe G, Lin H N, Vaillancourt B, Shiu S H, Jiang N, Buell C R.Comparative transcriptomics of three Poaceae species reveals patterns of gene expression evolution[J].The Plant Journal,2012,71(3):492-502. doi: 10.1111/j.1365-313X.2012.05005.x.
[30] Dugas D V, Monaco M K, Olsen A, Klein R R, Kumari S, Ware D, Klein P E.Functional annotation of the transcriptome of Sorghum bicolor in response to osmotic stress and abscisic acid[J].BMC Genomics,2011,12:514. doi: 10.1186/1471-2164-12-514.
[31] Holub E B.The arms race is ancient history in Arabidopsis,the wild flower[J].Nature Reviews Genetics,2001,2(7):516-527. doi: 10.1038/35080508.
[32] Hurst L D.The Ka/Ks ratio: diagnosing the form of sequence evolution[J].Trends in Genetics,2002,18(9):486-487.doi:10.1016/S0168-9525(02)02722-1.
[33] Slewinski T L.Diverse functional roles of monosaccharide transporters and their homologs in vascular plants: a physiological perspective[J].Molecular Plant,2011,4(4):641-662.doi:10.1093/mp/ssr051.
[34] Hirai T, Heymann J A W, Shi D, Sarker R, Maloney P A, Subramaniam S.Three-dimensional structure of a bacterial oxalate transporter[J].Nature Structural Biology,2002,9(8):597-600.doi:10.1038/nsb821.
[35] Xu G U, Guo C, Shan H Y,Kong H Z.Divergence of duplicate genes in exon-intron structure[J].Proceedings of the National Academy of Sciences,2012,109(4):1187-1192. doi: 10.1073/pnas.1109047109.
[36] Chen S K, Niu X, Guan Y X, Li H F.Genome-Wide analysis and expression profiles of the MYB genes in Brachypodium distachyon[J].Plant and Cell Physiology,2017,58(10):1777-1788.doi:10.1093/pcp/pcx115.
[37] Schneidereit A, Scholz-Starke J, Sauer N, Büttner M.AtSTP11,a pollen tube-specific monosaccharide transporter in Arabidopsis[J].Planta,2005,221(1):48-55.doi: 10.1007/s00425-004-1420-5.
[38] Scholz-Starke J, Büttner M, Sauer N.AtSTP6,a new pollen-specific H+-monosaccharide symporter from Arabidopsis[J].Plant Physiology,2003,131(1):70-77.doi: 10.1104/pp.012666.
[39] Schofield R A, Bi Y M, Kant S, Rothstein S J.Over-expression of STP13, a hexose transporter, improves plant growth and nitrogen use in Arabidopsis thaliana seedlings[J].Plant Cell and Environment,2009,32(3):271-285.doi:10.1111/j.1365-3040.2008.01919.x.
[40] Büttner M, Truernit E, Baier K,Scholz-starke J, Sontheim M, Lauterbach C,Huss V A R,Sauer N.AtSTP3,a green leaf-specific,low affinity monosaccharide-H+symporter of Arabidopsis thaliana[J].Plant, Cell & Environment,2000,23(2):175-184. doi: 10.1046/j.1365-3040.2000.00538.x.