干旱是引起土地荒漠化、农业减产最主要的自然灾害之一[1-2]。植物在漫长的进化过程中形成了多种抵御干旱的机理,其中最有效的抗旱机理之一就是分泌蜡质到植物角质层,形成一层严密的保水层[3]。植物角质层蜡质合成途径已基本清楚,其脂肪族成分是由链长C26~C32的超长链饱和脂肪酸在表皮细胞中合成,存在2条基本的合成途径:酰基还原途径和脱羰基途径[4-5]。在拟南芥(Arabidopsis thaliana)中,脱羰基途径产生的蜡质成分占其蜡质总量的80%以上,是合成植物角质层蜡质的主要途径[6],醛脱羰基酶(ECERIFERUM1 ,CER1)是该途径的关键酶。对拟南芥cer1突变体的研究表明,其茎表面没有蜡质结晶,体内醛大量积累,而链长C29烷烃、C29醇和C29酮含量均下降99%以上[6-7],表明CER1可能编码醛脱羰基酶,催化长链偶数碳的醛转化为长链奇数碳的烷烃,与C29烷烃及其衍生物的合成密切相关。但是上述结果中,醛也可能只是该途径的副产物,作为后备物在链烷烃合成受干扰时引入到该途径,因此,醛增加的表型并不能直接证明CERl就是醛脱羰基酶。进一步构建的CER1基因酵母异源表达系统没有得到有效的结果,可能是因为CER1专一性催化产生链长大于C27的烷烃产物,而酵母只能合成链长小于C26超长链饱和脂肪酸,所以酵母异源表达系统不适于研究植物CER1基因[6]。CER1基因与植物的抗旱性密切相关[6,8]。因此,在多种植物中克隆CER1基因并验证其功能,将为系统理解植物的抗旱适应机制,挖掘其相关功能基因奠定理论基础,对我国西北干旱地区生态环境建设和栽培作物的抗旱性遗传改良具有十分重要的理论意义和实践价值。
扁果枸杞(Lycium barbarum ssp. Bianguo)是宁夏枸杞(Lycium barbarum)的一个品种,其干燥果实具有较高的营养和药用价值。对10个宁夏枸杞品种干燥果实的主要营养成分分析表明,扁果枸杞的枸杞多糖和还原型VC含量均最高,分别达0.11,12.35 mg/g,类胡萝卜素含量达0.46 mg/g,甜菜碱含量1.05%,品质综合排名第二,仅次于宁杞0901[9-10]。扁果枸杞也是一种表皮蜡质极为发达的抗旱耐盐植物[11],是研究植物抗逆机理和挖掘抗逆基因资源用于作物遗传育种的良好材料。
本研究用逆转录PCR(Reverse transcription PCR,RT-PCR)法和RACE法克隆扁果枸杞CER1基因全长,进行生物信息学分析,并用荧光实时定量PCR(Real-time fluorescence quantitative PCR ,qRT-PCR)法初步探讨了渗透胁迫处理下,扁果枸杞CER1基因的表达模式,为深入研究CER1基因功能奠定分子基础。
1 材料和方法
1.1 试验材料与处理
扁果枸杞种子于2014年8月采自甘肃省白银市景泰县玉杰枸杞引种基地。幼苗培养方法参考文献[11]:挑选籽粒饱满的种子,先用2%次氯酸钠溶液消毒8~10 min,蒸馏水冲洗后,均匀铺在湿润滤纸上,避光培养4~5 d,待种子露白后种在蛭石中并加入1/2 Hoagland营养液,置于昼夜温度(26±2)℃/(23±2)℃,光照16 h/d,光强约200 μmol/(m2·s),空气相对湿度40%~60%的温室中,每2~3 d换一次营养液。待幼苗长至3周龄,用含有80 mmol/L D-山梨醇的1/2Hoagland营养液渗透处理24 h后,取80~100 mg叶置于液氮中速冻用于基因克隆;再分别用含有0(对照),80,160 mmol/L D-山梨醇的1/2Hoagland营养液处理幼苗0,6,12 h后,取根、茎、叶提取总RNA用于qRT-PCR分析。每处理3个重复,每个重复包括2~3株幼苗。
扁果枸杞总RNA提取和反转录参照上海生工UNIQ-10柱式TRIzol总RNA抽提试剂盒说明书和SanPrep柱式DNA胶回收试剂盒说明书进行;PrimeScriptTM Ⅱ第一链cDNA合成试剂盒、PCR扩增试剂盒、T-载体PCR产物克隆试剂盒购自TaKaRa公司。DNA Marker购自北京天根生化科技有限公司。琼脂糖购自Sigma。5′和3′-RACE-ready cDNA用宝生物的SMARTerTMRACE cDNA试剂盒制备;qRT-PCR参照TaKaRaPrimeScriptTMRT reagent Kit with gDNA Eraser (Perfect Real Time)反转录试剂盒说明书,合成第一链cDNA,并根据SYBR®Premix Ex TaqTMⅡ(Tli RNaseH Plus)试剂盒说明书以第一链cDNA为模板在ABI公司的7500 fast系统中建立反应体系,所用引物均由北京华大基因合成。
1.2 试验方法
1.2.1 LbCER1基因的克隆 在NCBI中下载拟南芥(Arabidopsis thaliana)、亚麻荠(Camelina sativa)、番茄(Solanum lycopersicum)、烟草(Nicotiana tabacum)等植物已知的CER1基因序列,根据其高度同源的保守区序列,利用DNAMAN 6.0和Primer 5.0生物软件设计1对CER1基因的引物P1、P2(表1),用于扩增CER1基因核心片段,推测PCR产物长度为828 bp。
根据已知的LbCER1核心片段序列设计扩增5′端的特异性引物GSP1、NGSP1和3′端的特异性引物GSP2、NGSP2(表1)。这些引物与SMARTerTMRACE cDNA试剂盒所带的通用引物UPM、UPM short共同使用,用于外侧、内侧PCR扩增。
根据上述试验获得的序列设计开放阅读框引物,扩增后测序验证,再用DNAMAN软件进行序列拼接,得到LbCER1基因全长。
1.2.2 LbCER1基因的生物信息学分析 用DNAMAN 6.0软件分析CER1核苷酸和氨基酸序列;用TMHMM在线工具预测跨膜区[12];功能结构域分析用InterPro:Protein sequence analysis & classification在线软件[13];基本理化性质用Expasy的ProtParam工具[12,14];亲水性/疏水性特征分析用Expasy的ProtScale工具[13];蛋白质二级结构和三级结构预测分别用SOPMA[14]和Swiss-Model软件[12];信号肽预测采用 SignalP 4.0 Server[13];亚细胞定位用CELLO[13]和SOPMA在线软件。用MEGA 5.0软件基于邻位相连法构建系统进化树。
1.2.3 LbCER1基因的表达模式分析 根据已经得到的扁果枸杞肌动蛋白(Actin,ACT) 基因片段的序列和LbCER1的全长序列设计实时定量引物QC1和QC2(表1),以不同处理时间和不同强度渗透胁迫处理的扁果枸杞根、茎、叶cDNA为模板,以扁果枸杞ACT基因片段(引物为QA1、QA2)为内参,按照SYBR®Premix Ex TaqTMⅡ(Tli RNaseH Plus)试剂盒说明书,建立qRT-PCR体系,然后置于ABI Onestep实时定量PCR仪中进行扩增,程序如下:95 ℃预变性 30 min;95 ℃变性 5 s,60 ℃退火30 s,40个循环。采用2-ΔΔCT法计算LbCER1的相对表达量,并用SPSS 17.0和Excel进行分析和作图。
表1 LbCER1克隆和qRT-PCR所用的引物序列
Tab.1 Primers for LbCER1 gene cloning and qRT-PCR
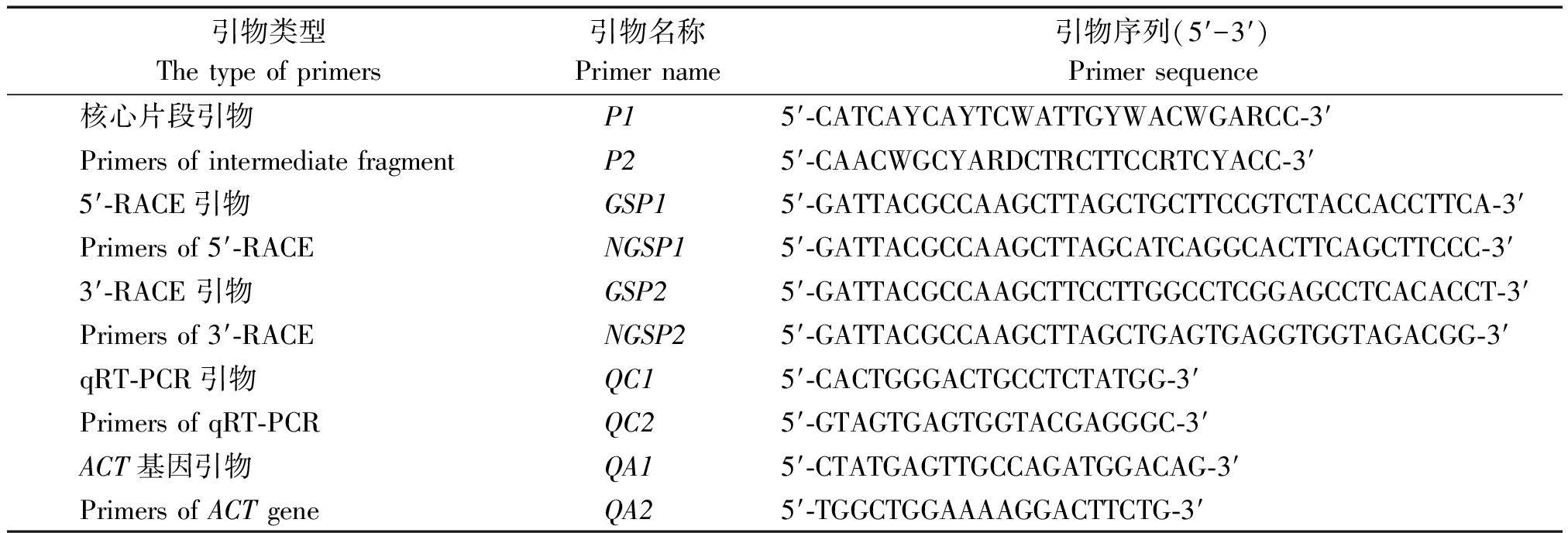
引物类型The type of primers引物名称Primer name引物序列(5′-3′)Primer sequence核心片段引物P15′-CATCAYCAYTCWATTGYWACWGARCC-3′Primers of intermediate fragmentP25′-CAACWGCYARDCTRCTTCCRTCYACC-3′5′-RACE引物GSP15′-GATTACGCCAAGCTTAGCTGCTTCCGTCTACCACCTTCA-3′Primers of 5′-RACENGSP15′-GATTACGCCAAGCTTAGCATCAGGCACTTCAGCTTCCC-3′3′-RACE 引物GSP25′-GATTACGCCAAGCTTCCTTGGCCTCGGAGCCTCACACCT-3′Primers of 3′-RACENGSP25′-GATTACGCCAAGCTTAGCTGAGTGAGGTGGTAGACGG-3′qRT-PCR引物QC15′-CACTGGGACTGCCTCTATGG-3′Primers of qRT-PCRQC25′-GTAGTGAGTGGTACGAGGGC-3′ACT基因引物QA15′-CTATGAGTTGCCAGATGGACAG-3′Primers of ACT geneQA25′-TGGCTGGAAAAGGACTTCTG-3′
2 结果与分析
2.1 LbCER1基因的克隆
根据其他植物的CER1序列设计简并性引物,采用RT-PCR方法从扁果枸杞叶中克隆到828 bp的CER1基因核心片段序列(图1-A)。Blast分析表明,该片段与已知的植物CER1的同源性在70%以上。通过5′-RACE和3′-RACE方法,克隆到5′和3′端序列,长度分别为992 bp(图1-B)和786 bp(图1-C),最后通过片段拼接得到长度2 168 bp的LbCER1核苷酸全长序列。该序列包含1 881 bp的开放阅读框(ORF),107 bp的5′非翻译区(UTR)和180 bp的3′-UTR,将其命名为LbCER1。
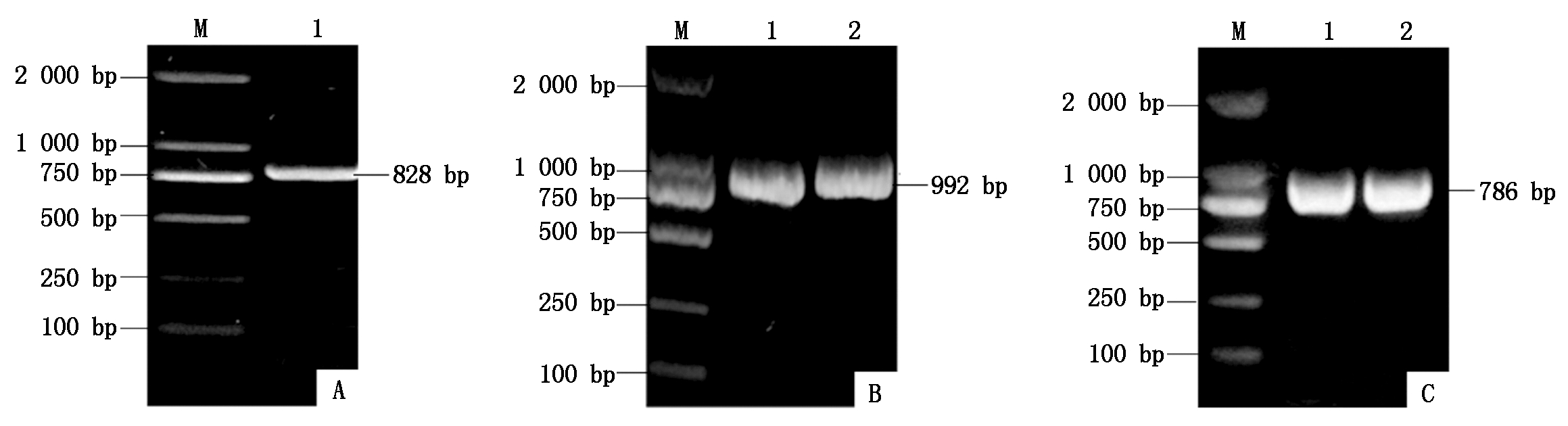
A. LbCER1基因核心片段;B.5′-RACE的PCR扩增产物;C.3′-RACE的PCR扩增产物。
A.The intermediate fragment of LbCER1; B.5′-RACE fragment; C.3′-RACE fragment.
图1 LbCER1基因克隆扩增产物电泳
Fig.1 Agarose gel electrophoresis of PCR products about LbCER1 gene
2.2 LbCER1的生物信息学分析
根据核苷酸全长序列分析,LbCER1蛋白包含626个氨基酸残基(图2)。跨膜区分析和功能结构域分析表明,该蛋白有4个跨膜区(图2、图3-A),一个脂肪酸羟化酶结构域(Fatty acid hydroxylase domain,图3-B)和1个位于C-末端的Wax2结构域(Wax2 domain of C-terminal,图3-B)。
蛋白质理化性质分析表明,LbCER1由20种氨基酸组成,其中亮氨酸(Leu)含量最高,达10.4%,半胱氨酸(Cys) 含量最低,为1.3%;分子质量为72.47 ku,等电点为7.40,分子式为C3338H5057N851O902S29;带正电荷的氨基酸(Arg+Lys)与带负电荷的氨基酸(Asp+Glu)等量,均为58个。该蛋白脂肪指数为91.28,不稳定系数为27.88,表明LbCER1是一种耐高温,性质稳定的偏碱性蛋白。
亲水性/疏水性分析表明,LbCER1平均亲水性(Grand average of hydropathicity, GRAVY) 为-0.070,是一种亲水性蛋白,其中第95位的天冬酰胺(Asn)残基分值最小(-3.433),是肽链中亲水性最强的位点;第50位的异亮氨酸(Ile) 残基分值最大(2.878),是疏水性最强的位点(图3-C)。

TM1、TM2、 TM3 和TM4分别表示跨膜区。
TM1, TM2, TM3 and TM4 represents the transmembrane regions respectively.
图2 LbCER1氨基酸序列
Fig.2 Amino acid residues sequence of LbCER1
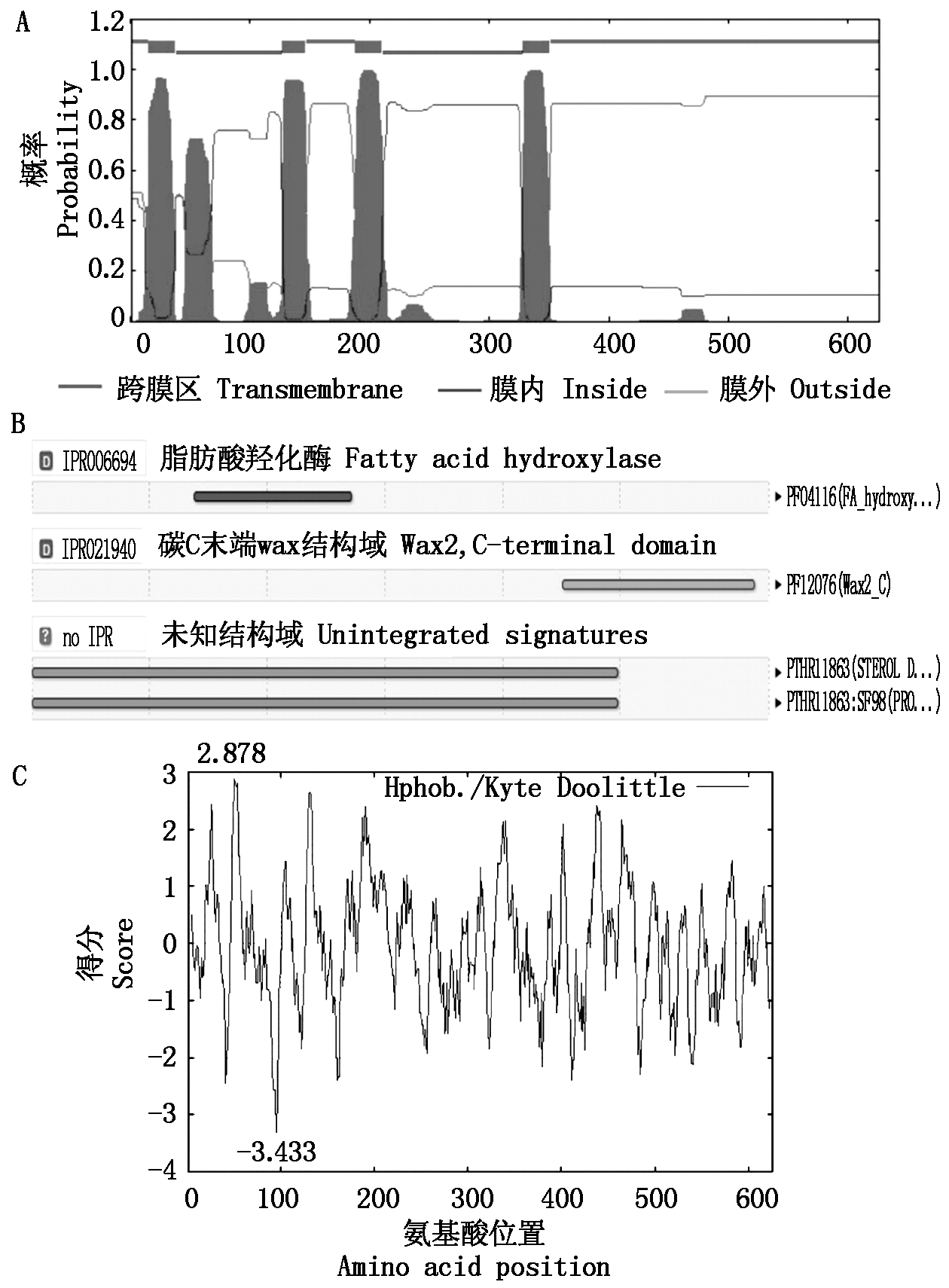
A.跨膜区预测;B.功能结构域预测;C.亲水/疏水性分析。
A.Transmembrane prediction; B.Functional domain prediction;
C.Hydrophilicity/hydrophobicity prediction.
图3 LbCER1跨膜区、功能结构域和亲水/疏水性的预测分析
Fig.3 Transmembrane regions, functional domain and hydrophilicity/hydrophobicity prediction of LbCER1
对LbCER1蛋白二级结构预测结果表明,LbCER1蛋白中69.65%的氨基酸残基埋在膜内,仅21.88%氨基酸残基暴露在膜外(图4-A),推测该蛋白是一个稳定的跨膜蛋白,与跨膜区结构预测结果相符(图4-A)。该蛋白中α-螺旋(α-Helix)占44.09%,环(Loop)占45.21%,延伸链(Strand)占10.7%(图4-A),与三级结构预测结果相符(图4-B)。
信号肽分析表明,LbCER1蛋白无信号肽(图4-C),不是分泌蛋白。用CELLO软件分析亚细胞定位表明,该蛋白定位在内膜上(表2),SOPMA软件分析显示LbCER1定位在内质网上(GO term ID:GO:0005789)。
表2 LbCER1的亚细胞定位分析
Tab.2 Subcellular localization analysis of LbCER1 protein
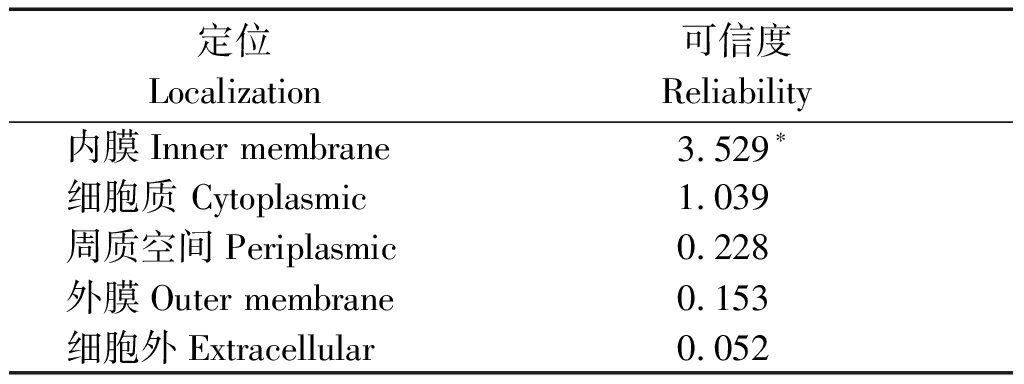
定位Localization可信度Reliability内膜Inner membrane3.529∗细胞质 Cytoplasmic1.039周质空间Periplasmic0.228外膜Outer membrane0.153细胞外Extracellular0.052
2.3 LbCER1与其他植物CER1氨基酸序列系统进化分析
系统进化树分析表明, LbCER1与烟草(Nicotiana tabacum)、番茄(Solanum lycopersicum)、辣椒(Capsicum annuum)等茄科植物亲缘关系极近,同源性达100%,与可可(Theobroma cacao)、油菜(Brassica napus)、拟南芥(Arabidopsis thaliana)、亚麻荠(Camelina sativa)等双子叶植物的同源性也在72%以上,但与小麦(Triticum aestivum)、玉米(Zea mays)等单子叶植物则亲缘关系较远(图5)。
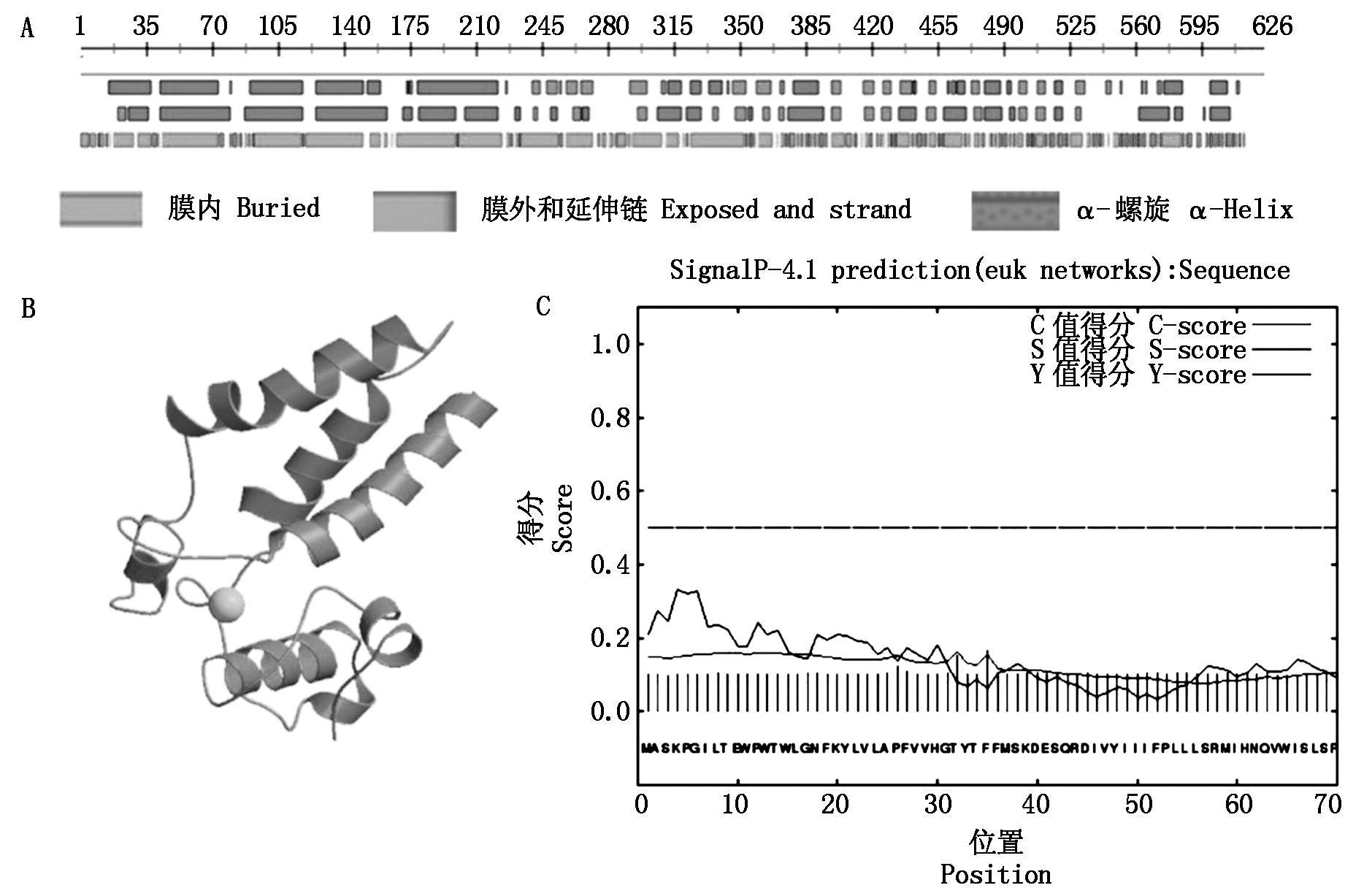
A.二级结构预测;B.三级结构预测;C.信号肽预测。
A.The secondary structure prediction; B.The tertiary structure prediction; C.Signal peptide prediction.
图4 LbCER1蛋白二级结构、三级结构和信号肽预测
Fig.4 The secondary structure, tertiary structure and signal peptide prediction of LbCER1
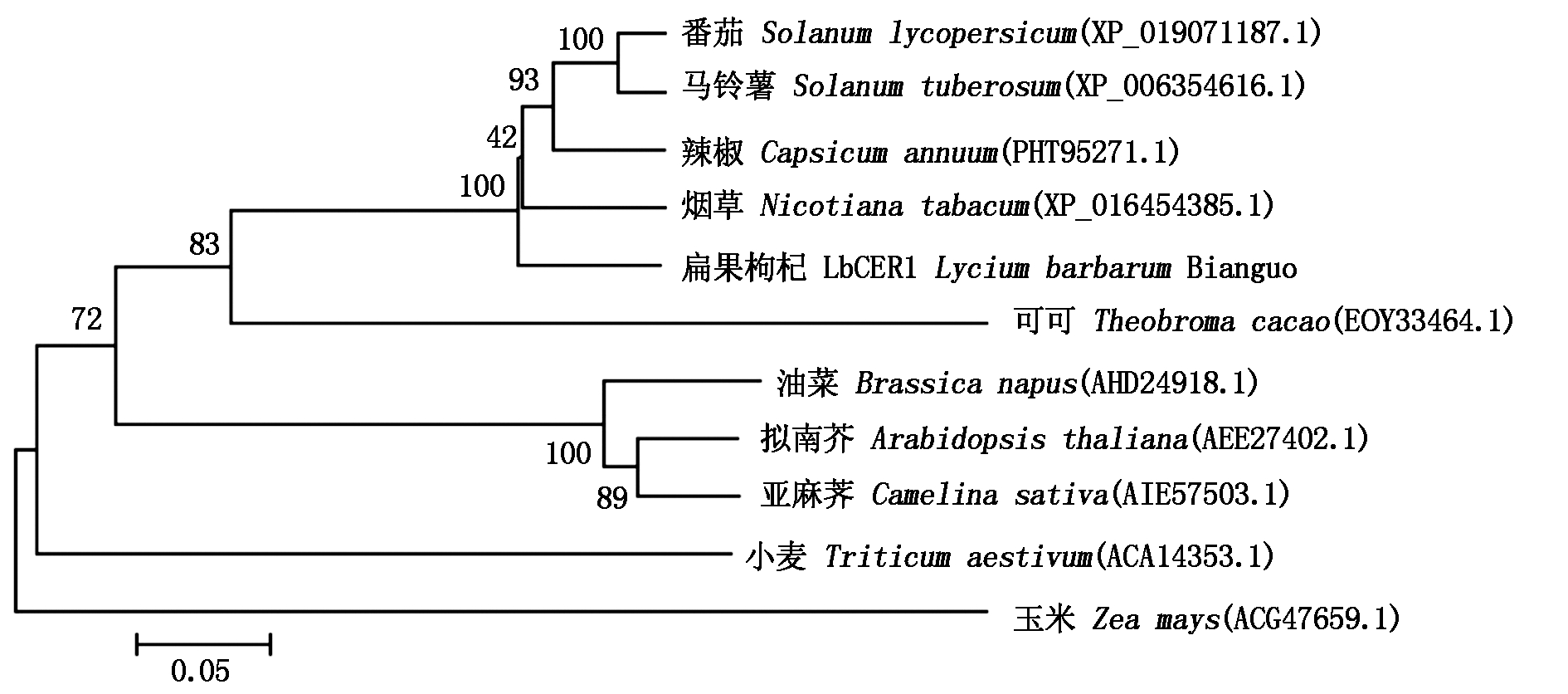
图5 LbCER1与其他植物CER1氨基酸序列系统进化分析
Fig.5 Phylogenetic analysis of LbCER1 and CER1 from other plants
2.4 LbCER1在渗透胁迫下的表达特征分析
qRT-PCR分析表明, LbCER1基因在叶、茎、根中均表达(图6)。与对照相比,160 mmol/L山梨醇渗透胁迫处理6 h时,LbCER1基因在叶中的相对表达量达到最高,比对照增加1.8倍,处理12 h时增加1倍;在茎中,LbCER1的相对表达量有波动,6 h时表达量增加21%,12 h时降低14%;在根中,160 mmol/L山梨醇渗透胁迫处理6,12 h时,LbCER1的相对表达量达分别增加1.1倍和32%。80 mmol/L山梨醇渗透胁迫处理6 h时,LbCER1基因在叶中的相对表达量增加66%,而12 h时降低61%;在茎中,80 mmol/L山梨醇渗透胁迫处理12 h时LbCER1表达量降低42%;在根中,80 mmol/L山梨醇渗透胁迫处理6 h时LbCER1表达量降低32%(图6)。这些结果表明,LbCER1的表达受渗透胁迫的诱导。
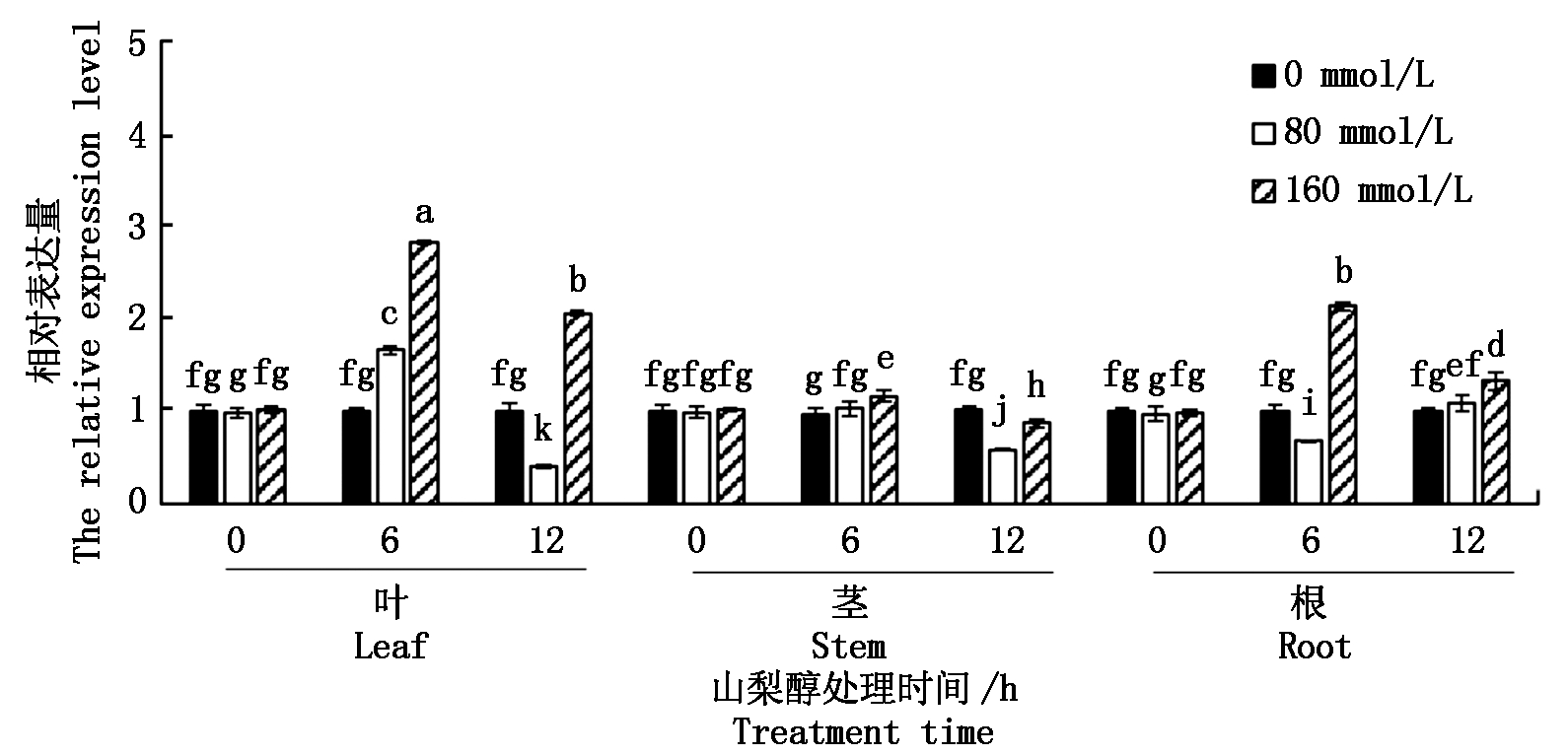
不同处理间不同小写字母表示差异显著(P<0.05)。
Different treatments with different lowercase mean significant difference at 0.05 level (P<0.05).
图6 LbCER1在扁果枸杞不同组织中的相对表达量
Fig.6 The relative expression levels of LbCER1 in different tissues of Lycium barbarum Bianguo
3 结论与讨论
高等植物中编码醛脱羰基酶的CER1基因功能研究最早由Aarts等[15]在拟南芥中进行,随后在千里光(Senecio scandens Buch.-Ham. ex D. Don)[16]、辣椒[17]、亚麻荠[18]、可可[19]、油菜[20]、小麦[21]、玉米[22]等多种植物中被报道。本研究从表皮蜡质发达的旱生植物扁果枸杞中克隆得到编码醛脱羰基酶的基因LbCER1,该基因包含1 881 bp的开放阅读框(ORF),编码626个氨基酸,与千里光CER1的ORF框大小相似[16]。序列结构分析表明,拟南芥AtCER1[15]和扁果枸杞蛋白LbCER1均存在多个跨膜区和一个脂肪酸羟化酶保守区;在序列相似性上,LbCER1与茄科植物亲缘关系最近,其次与油菜、亚麻荠等双子叶植物亲缘关系叶达72%以上,但与小麦、玉米等单子叶植物关系较远,符合植物进化的一般趋势[23],上述特征均说明CER1蛋白序列在进化上相对保守。
拟南芥AtCER1蛋白定位在内质网膜上[24],由CER1编码的醛脱羰基酶和由CER3/WAX2/YRE编码的醛合成酶及一种细胞色素b6亚型(Cytochrome b6 isoforms ,CYTB5s)催化构成一个多酶复合物催化两步反应,将长链脂酰-CoA转化成长链烷烃[5]。对扁果枸杞LbCER1的亚细胞定位分析与表明,LbCER也定位在内质网上,且扁果枸杞LbCER1 蛋白的C-端包含Wax2结构域,可能该结构域与蛋白的催化功能密切相关。
拟南芥AtCER1基因只在幼苗、茎、叶、花及荚果的表皮专一性表达,在根中不表达[6]。但本研究表明, LbCER1基因在叶、茎、根中均表达。AtCER1对干旱胁迫十分敏感,低湿条件下处理3 h, AtCER1表达丰度达到最大,为对照组的40倍[6]。水稻Glossyl(GL1) 基因与AtCER1基因同源,其表达也受渗透胁迫的诱导[8]。本研究中,LbCER1受一定强度渗透胁迫的诱导而上调,表明该基因可能与植物抗逆响应密切相关。
在拟南芥中,WAX INDUCER1/SHINE1 (WIN1/SHN1)是第一个被报道的能在干旱处理下通过上调蜡质合成相关基因KCS1、CER1和 CER2促进表皮蜡质合成的转录因子[25]。降低蜡质合成基因(Decrease wax biosynthesis, DEWAX)是目前发现的唯一在黑暗诱导下通过调控蜡质合成相关基因CER1、FAR6、LACS2、ACLA2和ECR抑制表皮蜡质沉积的转录因子[25]。旱生植物中影响表皮蜡质沉积及LbCER1基因表达的上下游调控因子还需进一步鉴定,进而深入研究LbCER1功能及其调控机制,以期为作物的抗旱育种奠定分子基础。
[1] Peleg Z, Apse M P, Blumwald E. Engineering salinity and water-stress tolerance in crop plants:getting closer to the field [J]. Advances in Botanical Research, 2011, 57:405-443. doi:10.1016/b978-0-12-387692-8.00012-6.
[2] Baldoni E, Genga A, Cominelli E. Plant MYB transcription factors:their role in drought response mechanisms [J]. International Journal of Molecular Sciences, 2015, 16(7):15811-15851. doi:10.3390/ijms160715811.
[3] Riederer M, Schreiber L. Protecting against water loss:analysis of the barrier properties of plant cuticles [J]. Journal of Experimental Botany, 2001, 52(363):2023-2032.doi:10.1093/jexbot/52.363.2023.
[4] 倪郁, 郭彦军. 植物超长链脂肪酸及角质层蜡质生物合成相关酶基因研究现状[J]. 遗传, 2008, 30(5):561-567. doi:10.3724/SP.J.1005.2008.00561.
Ni Y, Guo Y J. Progress in the study on genes encoding enzymes involved in biosynthesis of very long chain fatty acids and cuticular wax in plants [J]. Hereditas, 2008, 30(5):561-567.
[5] Bernard A, Domergue F, Pascal S,Jetter R, Renne C, Faure J D, Haslam R P, Napier J A, Lessire R, Joubè s J. Reconstitution of plant alkane biosynthesis in yeast demonstrates that Arabidopsis ECERIFERSUM1 and ECERIFERUM3 are core components of a very long-chain alkane synthesis complex [J].The Plant Cell, 2012, 24(7):3106-3118. doi:10.1105/tpc.112.099796.
[6] Bourdenx B, Bemard A, Domergue F, Pascal S, Léger A, Roby D, Pervent M, Vile D, Haslam R P, Napier J A, Lessire R, Joubès J. Overexpression of Arabidopsis ECERIFERUM1 promotes wax very-Long-Chain alkane biosynthesis and influences plant response to biotic and abiotic stresses [J]. Plant Physiology, 2011, 156(1):29-45. doi:10.1104/pp.111.172320.
[7] Hannoufa A, McNevin J, Lemieux B. Epicuticular waxes of eceriferum mutants of Arabidopsis thaliana[J]. Phytochemistry, 1993, 33(4):851-855. doi:10.1016/0031-9422(93)85289-4.
[8] 周玲艳, 姜大刚, 李静, 周海, 曹伟炜, 庄楚雄. 逆境处理下水稻叶角质层蜡质积累及其与蜡质合成相关基因OsGL1表达的关系[J]. 作物学报, 2012, 38(6):1115-1120. doi:10.3724/SP.J.1006.2012.01115.
Zhou L Y, Jiang D G, Li J, Zhou H, Cao W W, Zhuang C X. Effect of stresses on leaf cuticular wax accumulation and its relationship to expression of OsGL1-Homologous genes in rice[J]. Acta Agronomica Sinica, 2012, 38(6):1115-1120.
[9] 王益民, 张珂, 许飞华, 王玉, 任晓卫, 张宝琳. 不同品种枸杞子营养成分分析及评价[J]. 食品科学, 2014, 35(1):34-38. doi:10.7506/spkx1002-6630-201401007.
Wang Y M, Zhang K, Xu F H, Wang Y, Ren X W, Zhang B L. Chemical analysis and nutritional evaluation of different varieties of Goji berries (Lycium barbarum L.) [J]. Food Science, 2014, 35(1):34-38.
[10] Yao R Y, Huang C, Chen X F, Yin Z Q, Fu Y P, Li L X, Feng B, Song X, He C L, Yue G Z, Jing B, Lǖ C, Su G, Ye G, Zou Y F. Two complement fixing pectic polysaccharides from pedicel of Lycium barbarum L. promote cellular antioxidant defense [J]. International Journal of Biological Macromolecules, 2018, 112:356-363. doi:10.1016/j.ijbiomac.2018.01.207.
[11] 袁惠君, 刘轲, 王春梅, 谢辉灿, 李虎军, 贾鸿震. 两个宁夏枸杞品种的耐渗透胁迫和耐盐特征比较[J]. 草业科学, 2016, 33(4):681-690. doi:10.11829/j.issn.1001-0629.2015-0526.
Yuan H J, Liu K, Wang C M, Xie H C, Li H J, Jia H Z. The differences between two cultivars of Lycium barbarum in osmotic stress tolerance and salt tolerance [J]. Pratacultural Science, 2016, 33(4):681-690.
[12] 阎文飞, 程凡升, 姜新强, 刘翠霞, 朱丹. 野大豆盐碱胁迫相关GsTIFY6B基因克隆及表达特性分析[J]. 华北农学报, 2018, 33(4):82-89. doi:10.7668/hbnxb.2018.04.012.
Yan W F, Cheng F S, Jiang X Q, Liu C X, Zhu D. Cloning and expression analysis of GsTIFY6B associated with saline and alkali stress in Glycine soja [J]. Acta Agriculturae Boreali-Sinica, 2018, 33(4):82-89.
[13] 范琪, 马彦妮, 陈佰鸿, 左存武, 毛娟. 葡萄CO4基因的克隆及其对光质响应的表达分析[J]. 华北农学报, 2018, 33(4):90-97. doi:10.7668/hbnxb.2018.04.013.
Fan Q, Ma Y N, Chen B H, Zuo C W, Mao J. Cloning and expression analysis of CO4 gene in the different light qualities and light transfers of grapevine [J]. Agriculturae Boreali-Sinica, 2018, 33(4):82-89.
[14] 王俊斌, 杨文丽, 丁博, 吴天文, 王海凤, 谢晓东. 小麦TaWRKY71a基因克隆、生物信息学及表达分析[J]. 华北农学报, 2018, 33(3):7-13. doi:10.7668/hbnxb.2018.03.002.
Wang J B, Yang W L, Ding B, Wu T W, Wang H F, Xie X D. Cloning,bioinformatics and expression analysis of TaWRKY71a gene in wheat [J]. Agriculturae Boreali-Sinica, 2018, 33(3):7-13.
[15] Aarts M G M, Keijzer C J, Stiekema W J, Pereira A. Molecular characterization of the CER1 gene of Arabidopsis involved in epicuticular wax biosynthesis and pollen fertility[J]. The Plant Cell, 1995, 7(12):2115-2127. doi:10.2307/3870155.
[16] 汤贤春, 钱倩, 罗才林, 王蕾, 钱刚. 千里光脂肪醛脱羰基酶(CER1蛋白)的保守基序(CSM)与功能结构域分析[J]. 生命科学研究, 2016, 20(5):395-400. doi:10.16605/j.cnki.1007-7847.2016.05.004.
Tang X C, Qian Q, Luo C L, Wang L, Qian G. Analysis of conserved sequence Motif (CSM) and functional/structural domains of fatty-aldehyde decarbonylase (CER1 Protein) in Senecio scandens Buch.-Ham. ex D. Don[J]. Life Science Research, 2016, 20(5):395-400.
[17] Kim S, Park M, Yeom S I, Kim Y M, Lee J M, Lee H A, Seo E, Choi J, Cheong K, Kim K T, Jung K, Lee G W, Oh S K, Bae C, Kim S B, Lee H Y, Kim S Y, Kim M S, Kang B C, Jo Y D, Yang H B, Jeong H J, Kang W H, Kwon J K, Shin C, Lim J Y, Park J H, Huh J H, Kim J S, Kim B D, Cohen O, Paran I,Suh M C,Lee S B, Kim Y K, Shin Y, Noh S J, Park J, Seo Y S, Kwon S Y, Kim H A, Park J M, Kim H J, Choi S B, Bosland P W, Reeves G, Jo S H, Lee B W, Cho H T, Choi H S, Lee M S, Yu Y, Do Choi Y, Park B S, van Deynze A, Ashrafi H, Hill T, Kim W T, Pai H S, Ahn H K, Yeam I, Giovannoni J J, Rose J K C, Sørensen I, Lee S J, Kim R W, Choi I Y, Choi B S, Lim J S, Lee Y H, Choi D. Genome sequence of the hot pepper provides insights into the evolution of pungency in Capsicum species [J]. Nature Genetics, 2014, 46(3):270-278. doi:10.1038/ng.2877.
[18] Lee S B, Kim H, Kim R J, Suh M C. Overexpression of Arabidopsis MYB96 confers drought resistance in Camelina sativa via cuticular wax accumulation [J]. Plant Cell Reports, 2014, 33(9):1535-1546. doi:10.1007/s00299-014-1636-1.
[19] Motamayor J C, Mockaitis K, Schmutz J, Haiminen N, Livingstone D, Cornejo O, Findley S D, Zheng P, Utro F, Royaert S, Saski C, Jenkins J, Podicheti R, Zhao M X, Scheffler B E, Stack J C, Feltus F A, Mustiga G M, Amores F, Phillips W, Marelli J P, May G D, Shapiro H, Ma J X, Bustamante C D, Schnell R J, Main D, Gilbert D, Parida L, Kuhn D N. The genome sequence of the most widely cultivated cacao type and its use to identify candidate genes regulating pod color [J]. Genome Biology, 2013, 14(6):1-25. doi:10.1186/gb-2013-14-6-r53.
[20] Pu Y Y, Gao J, Guo Y L, Liu T T, Zhu L X, Xu P, Yi B, Wen J, Tu J X, Ma C Z, Fu T D, Zou J T, Shen J X. A novel dominant glossy mutation causes suppression of wax biosynthesis pathway and deficiency of cuticular wax in Brassica napus [J]. BMC Plant Biology, 2013, 13(1):215-229. doi:10.1186/1471-2229-13-215.
[21] Hu X J, Zhang Z B, Li W Q, Fu Z Y, Zhang S X, Xu P. cDNA cloning and expression analysis of a putative decarbonylase TaCer1, from wheat (Triticum aestivum L.) [J]. Acta Physiologiae Plantarum, 2009, 31(6):1111-1118. doi:10.1007/s11738-009-0329-9.
[22] Alexandrov N N, Brover V V, Freidin S, Troukhan M E, Tatarinova T V, Zhang H Y, Swaller T J, Lu Y P, Bouck J, Flavell R B. Insights into corn genes derived from large-scale cDNA sequencing[J]. Plant Molecular Biology, 2009, 69(1/2):179-194. doi:10.1007/s11103-008-9415-4.
[23] Soltis P S, Soltis D E. The origin and diversification of angiosperms [J]. American Journal of Botany, 2004, 91(10):1614-1626. doi:10.3732/ajb.91.10.1614.
[24] Kamigaki A, Kondo M, Mano S, Hayashi M, Nishimura M. Suppression of peroxisome biogenesis factor 10 reduces cuticular wax accumulation by disrupting the ER network in Arabidopsis thaliana [J]. Plant & Cell Physiology, 2009, 50(12):2034-2046. doi:10.1093/pcp/pcp152.
[25] Lee S B, Suh M C. Advances in the understanding of cuticular waxes in Arabidopsis thaliana and crop species [J]. Plant Cell Reports, 2015, 34(4):557-572.doi:10.1007/s00299-015-1772-2.