大豆单株荚数是影响大豆产量的一个重要农艺性状,同时也是育种中的一个重要指标。在大豆产量构成因素(百粒质量、单株荚数、单株粒数等)中,单株荚数与产量相关性较高[1]。同时研究还发现,单株荚数与百粒质量、每荚粒数、主茎节数等重要性状关联度和相关系数较高[2],单株荚粒数的提高对百粒质量等农艺性状不会有较大的负面影响[3],说明通过选育较高单株荚数的材料来提高大豆产量是可行的。因此,可以通过选择较多单株荚数的材料进行聚合育种,从而更有效地提高大豆产量。
单株荚数是一个非常复杂的性状,它是由多因素调控的,其中最关键的3个过程分别为花芽分化数、胚珠成功受精比率和成功受精后的胚胎正常发育为荚果的比率[4]。花芽的分化和发育决定子房胚珠数和可育胚珠比率。成功受精胚珠比率是由受精过程所决定的,其中包括花粉育性、花粉与柱头的接触量、花粉粒的萌发、受精条件以及花粉管的导入等过程。受精后的胚珠最终是否能发育为种子由结合子发育过程决定。然而其中任何一个生物学过程都是相当复杂的,因此,将通过研究每一个过程的分子机理和功能,才能最终将单株荚数这个复杂的性状解析开来。
单株荚数是一个典型的数量性状,受多基因控制,同时也易受环境的影响。目前,就国内外已有的关于大豆荚数QTL定位研究的报道可知[5-16],定位了约50多个QTLs位点,这些位点几乎分布于大豆20条染色体上(除B2和H);解释表型变异平均值为13.4%,约有69%的QTLs解释表型变异在20%之下,主效荚数QTL也较少。
本研究利用多荚材料C025和少荚材料JD18为亲本,然后利用双亲构建的F2群体定位大豆单株荚数QTL,通过定位的大豆荚数QTL,阐明大豆单株荚数的分子机制;同时整合前人对大豆荚数定位的QTL,解析大豆单株荚数QTL在全基因组的分布以及联系,旨在为选育大豆较高单株荚数提供材料基础并为后续研究大豆单株荚数分子机制奠定基础。
1 材料和方法
1.1 试验材料
本研究使用的亲本材料C025(多荚)和JD18(少荚)来源于山西省农业科学院农作物品种资源研究所,双亲杂交得到182个单株F2群体。2016年5-10月在太原山西省农业科学院东阳基地完成杂交试验得到F1杂交种,2017年5-10月和2018年5-10月(太原2017年、太原2018年)连续2 a在太原山西省农业科学院东阳基地完成亲本和F2群体田间种植和表型鉴定。
1.2 试验方法
田间试验按照完全随机区组设计,3次重复,每个小区2行,行距(株距为50.0 cm×13.5 cm。大豆成熟时,每个小区选择10个代表单株人工统计荚数。具体调查标准参照《中国大豆品种志》[17]进行。
1.3 QTL定位分析
利用在公共数据库中已发表的大豆遗传连锁图谱(https://soybase.org/),结合荚数表型鉴定进行QTL定位。QTL扫描采用WinQTL Cartographer 2.5软件(http://statgen.ncsu.edu/qtlcart/WQTLCart.htm)中的复合区间作图法[18]。LOD阈值显著性采用1 000次排布测验分析[19],基本参数步长、窗口大小、扫描间距和运行速度分别设置为1,10,5,1 cM,P=0.05显著水平检测并确认QTL。采用QTL-元分析方法整合前人研究的不同环境不同群体检测的大豆荚数QTL[20]。
1.4 数据分析
利用SAS V8软件中的PROC ANOVA程序对不同环境单株荚数表型进行方差分析,从而估算遗传力。群体单株荚数频率分布和亲本间单株荚数的显著性分析采用Excel中的函数进行。
2 结果与分析
2.1 父母本及F2群体单株荚数表型变异和遗传力分析
亲本C025和JD18在2个不同环境中的单株荚数均表现出极显著差异(P<0.01),C025单株总荚数明显高于JD18,是JD18的近2倍,并且在2 a表型考察中较稳定(表1)。F2分离群体单株荚数在不同环境中都表现出广泛的变异(2~150),并且有些株系有明显的超亲分离,表明控制大豆荚数的基因在2个亲本中都存在。F2分离群体单株荚数在2个环境中都呈现近似正态分布,可以说明单株荚数符合数量性状特征,适合利用QTL定位分析(图1)。通过方差分析评估大豆荚数的遗传力为65%,说明大豆荚数遗传力较大,有利于进行后期的遗传改良。
表1 双亲和F2群体在2个不同环境下单株荚数表型分析
Tab.1 Phenotypic variation of pod number per plant for parents and F2 population in two investigated environments
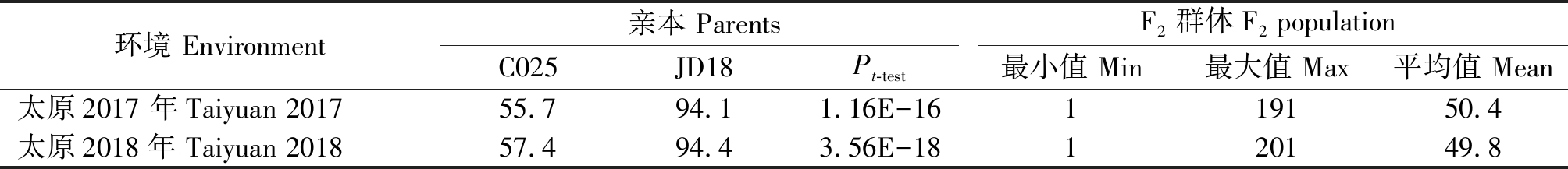
环境 Environment亲本 ParentsF2群体F2 populationC025JD18Pt-test最小值 Min最大值 Max平均值 Mean太原2017 年Taiyuan 201755.794.11.16E-16119150.4太原2018年 Taiyuan 201857.494.43.56E-18120149.8
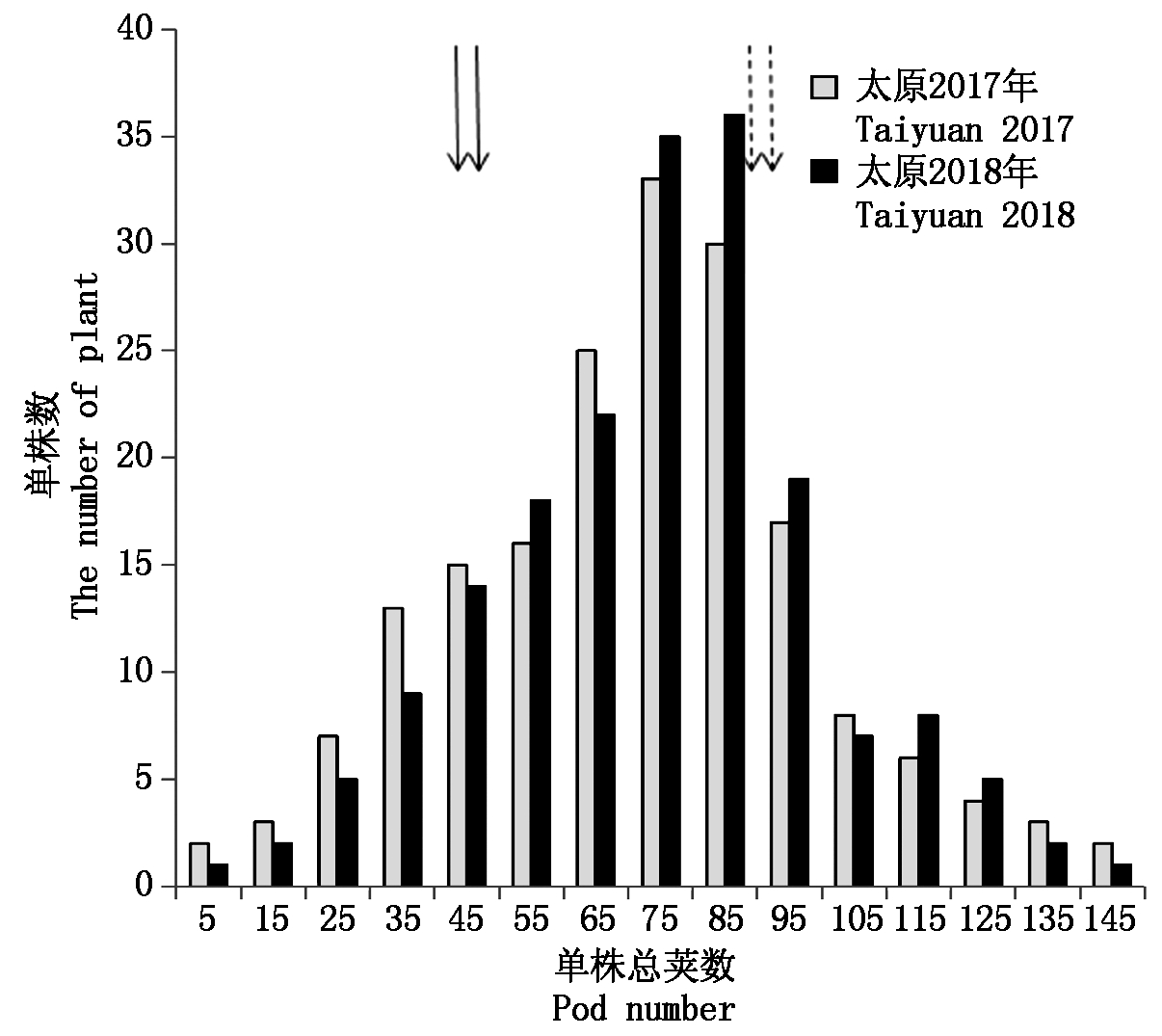
实心箭头是JD18;虚线箭头是C025。 Solid arrows JD18; Dashed arrows C025.
图1 F2群体2 a 2点单株荚数频率分布
Fig.1 Frequency distribution of branching for F2
population at two locations in two years
2.2 大豆单株荚数QTL定位分析
利用1张包括1 015个SSR标记的大豆遗传图谱(https://soybase.org/),其覆盖大豆基因组2 282.92 cM,长度为 71.31~165.73 cM,相邻标记间的平均距离为2.44 cM,这些SSR标记在 20 条染色体上分布较均匀。利用软件WinQTL Cartographer 2.5对2个环境的单株荚数分别进行全基因组QTL扫描,结果显示,单环境下共检测出33个QTLs,分别位于D1a、N、C1、C2、M、A2、K、O、B1、F、B2、E、J、D2、G、L和I 共17个染色体上,LOD值在2.8~15.1,贡献率在0.2%~56.4%,平均值为14.9%。经过元分析整合后,共定位23个QTLs,分别位于D1a、N、C1、C2、M、A2、K、O、B1、F、B2、E、J、D2、G、L和I 共17个染色体(表2)。其中,有10个QTLs在2个环境中被重复检测到,分别定位于A2、D2、C2、I、B1、C1和J染色体上,LOD值在2.8~15.1,平均值为9.4,解释的平均表型变异0.2%~56.4%,平均值为25.2%,其中有6个QTLs的贡献率高于20%。qPN.C2-4、qPN.C1、qPN.J、qPN.D2-1和qPN.A2-1的贡献率分别为29.5%,35.4%,38.9%,53.9%和56.4%,均高于前人定位的大豆荚数QTLs。通过比较发现,qPN.C2-4和qPN.C1的置信区间与前人的研究重叠,并且本研究的效应值高于前人,同时QTL-qPN.A2-1的贡献率为56.4%,是目前检测到的效应值最大的控制大豆单株荚数的QTL。因此,这3个QTLs可看作控制大豆单株荚数的主效QTL,并对其进一步深入解析。
表2 多环境检测到的大豆单株荚数QTLs
Tab.2 Detected QTLs for pod number per plant of soybean from different environment
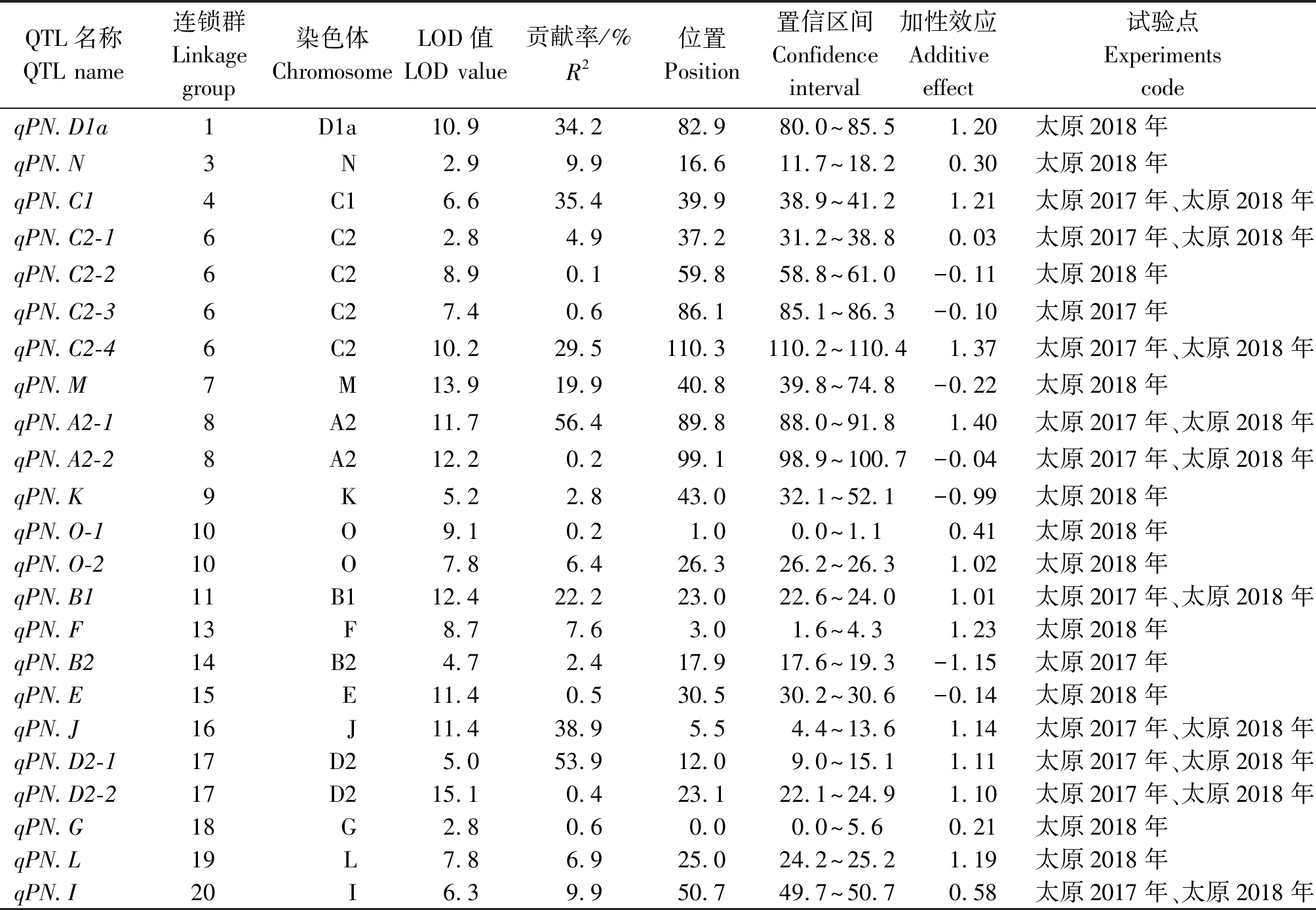
QTL名称QTL name连锁群Linkagegroup染色体ChromosomeLOD值LOD value贡献率/%R2位置Position置信区间Confidenceinterval加性效应Additiveeffect试验点ExperimentscodeqPN.D1a1D1a10.934.282.980.0~85.51.20太原2018年qPN.N3N2.99.916.611.7~18.20.30太原2018年qPN.C14C16.635.439.938.9~41.21.21太原2017年、太原2018年qPN.C2-16C22.84.937.231.2~38.80.03太原2017年、太原2018年qPN.C2-26C28.90.159.858.8~61.0-0.11太原2018年qPN.C2-36C27.40.686.185.1~86.3-0.10太原2017年qPN.C2-46C210.229.5110.3110.2~110.41.37太原2017年、太原2018年qPN.M7M13.919.940.839.8~74.8-0.22太原2018年qPN.A2-18A211.756.489.888.0~91.81.40太原2017年、太原2018年qPN.A2-28A212.20.299.198.9~100.7-0.04太原2017年、太原2018年qPN.K9K5.22.843.032.1~52.1-0.99太原2018年qPN.O-110O9.10.21.00.0~1.10.41太原2018年qPN.O-210O7.86.426.326.2~26.31.02太原2018年qPN.B111B112.4 22.223.0 22.6~24.01.01 太原2017年、太原2018年qPN.F13F8.7 7.6 3.0 1.6~4.31.23 太原2018年qPN.B214B24.7 2.4 17.9 17.6~19.3-1.15 太原2017年qPN.E15E11.4 0.5 30.5 30.2~30.6-0.14 太原2018年qPN.J16J11.4 38.9 5.5 4.4~13.61.14 太原2017年、太原2018年qPN.D2-117D25.0 53.9 12.0 9.0~15.11.11 太原2017年、太原2018年qPN.D2-217D215.1 0.4 23.1 22.1~24.91.10 太原2017年、太原2018年qPN.G18G2.8 0.60.0 0.0~5.60.21 太原2018年qPN.L19L7.8 6.9 25.0 24.2~25.21.19 太原2018年qPN.I20I6.3 9.9 50.7 49.7~50.70.58 太原2017年、太原2018年
2.3 大豆荚数QTL整合分析
通过查阅已报道的与大豆荚数定位相关的文献,结果显示,共定位了51个QTLs,在大豆18个染色体上均有分布(在B2和H中没有检测到荚数QTL),其中,在C2染色体上并且在一个区段内分布较为集中。经过元分析整合前人定位的大豆荚数QTLs,最终确定39个QTLs,LOD值为2.80~6.83,贡献率为6.54%~27.01%,平均值为13.4%(表3),约有69%的QTLs的贡献率在20%之下,约有5个QTLs可看作主效QTLs。整合的39个QTLs,其中有6个重复检测到的QTLs被整合,分别分布在B1、C1、C2、D1a和L染色体上。
与前人研究的结果相比,本研究新定位了18个大豆单株荚数QTLs,并且首次在B2染色体上定位了1个控制大豆单株荚数的QTL(qPN.B2),充分补充了大豆单株荚数分子机制研究。同时还发现,本研究定位的23个QTLs中有5个QTLs与前人的研究重叠,分别为qPN.C2-3、qPN.I、qPN.C2-4、qPN.C1和qPN.L,其中,qPN.C2-4和qPN.C1是本研究发现的主效QTL,说明qPN.C2-4和qPN.C1是可多环境多材料重复检测的大豆单株荚数主效QTLs,这可为后续大豆单株荚数分子研究奠定基础。
表3 整合不同研究中检测到的大豆荚数QTLs
Tab.3 Pod number QTLs of soybean in different studies
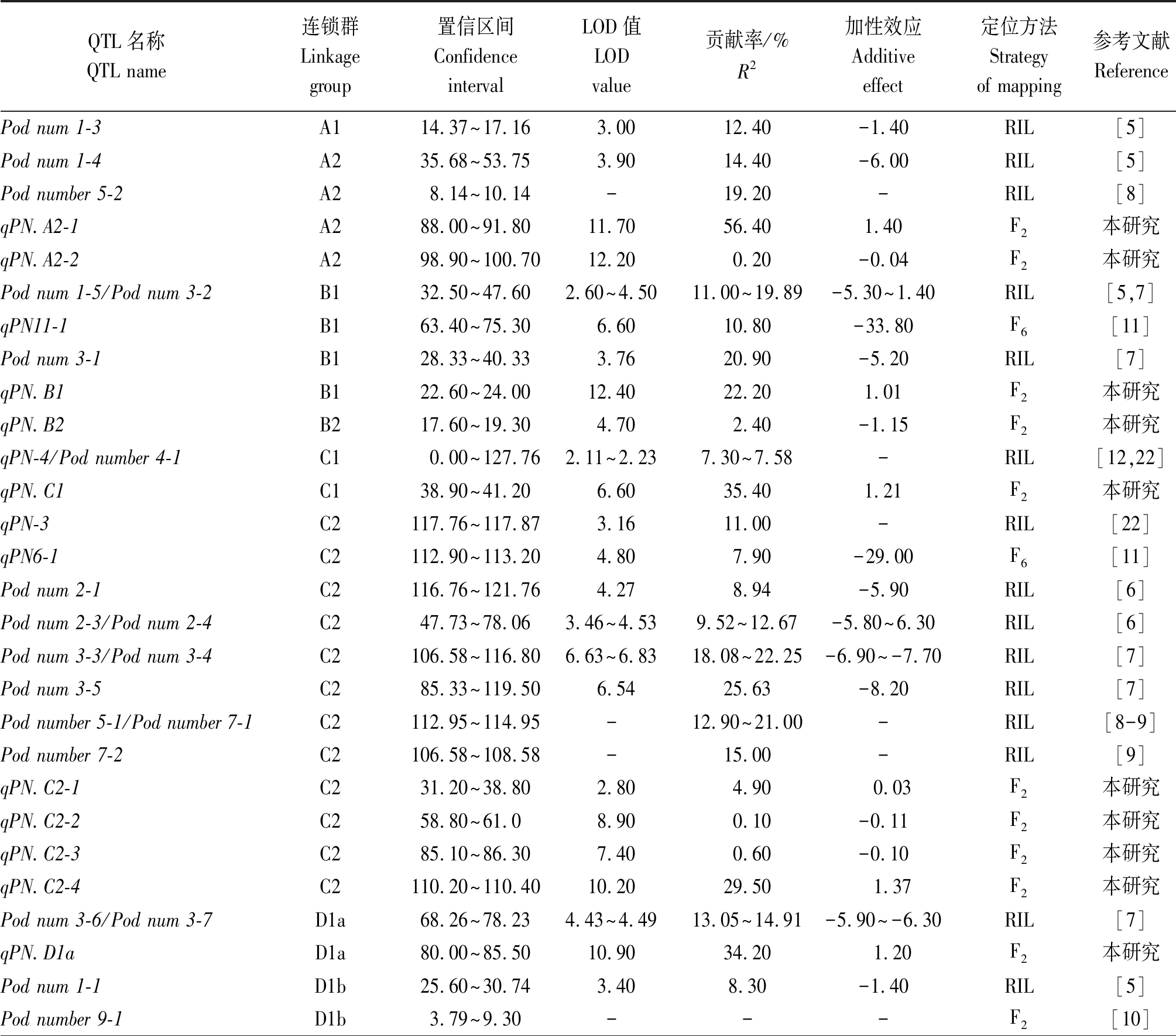
QTL名称QTL name连锁群Linkagegroup置信区间 Confidence intervalLOD值LODvalue贡献率/%R2加性效应Additive effect定位方法 Strategy of mapping参考文献ReferencePod num 1-3A114.37~17.163.00 12.40 -1.40 RIL[5]Pod num 1-4A235.68~53.753.90 14.40 -6.00 RIL[5]Pod number 5-2A28.14~10.14-19.20 -RIL[8]qPN.A2-1A288.00~91.8011.70 56.40 1.40F2本研究qPN.A2-2A298.90~100.7012.20 0.20 -0.04 F2本研究Pod num 1-5/Pod num 3-2B132.50~47.602.60~4.5011.00~19.89-5.30~1.40RIL[5,7]qPN11-1B163.40~75.306.60 10.80 -33.80 F6[11]Pod num 3-1B128.33~40.333.76 20.90 -5.20 RIL[7]qPN.B1B122.60~24.0012.40 22.20 1.01F2本研究qPN.B2B217.60~19.304.70 2.40 -1.15F2本研究qPN-4/Pod number 4-1C10.00~127.762.11~2.237.30~7.58-RIL[12,22]qPN.C1C138.90~41.206.60 35.40 1.21F2本研究qPN-3C2117.76~117.873.16 11.00 -RIL[22]qPN6-1C2112.90~113.204.80 7.90 -29.00 F6[11]Pod num 2-1C2116.76~121.764.27 8.94 -5.90 RIL[6]Pod num 2-3/Pod num 2-4C247.73~78.063.46~4.539.52~12.67-5.80~6.30RIL[6]Pod num 3-3/Pod num 3-4C2106.58~116.806.63~6.8318.08~22.25-6.90~-7.70RIL[7]Pod num 3-5C285.33~119.506.54 25.63 -8.20 RIL[7]Pod number 5-1/Pod number 7-1C2112.95~114.95-12.90~21.00-RIL[8-9]Pod number 7-2C2106.58~108.58-15.00 -RIL[9]qPN.C2-1C231.20~38.802.80 4.90 0.03 F2本研究qPN.C2-2C258.80~61.08.90 0.10 -0.11 F2本研究qPN.C2-3C285.10~86.307.40 0.60 -0.10 F2本研究qPN.C2-4C2110.20~110.4010.20 29.50 1.37 F2本研究Pod num 3-6/Pod num 3-7D1a68.26~78.234.43~4.4913.05~14.91-5.90~-6.30RIL[7]qPN.D1aD1a80.00~85.5010.90 34.20 1.20 F2本研究Pod num 1-1D1b25.60~30.743.40 8.30 -1.40 RIL[5]Pod number 9-1D1b3.79~9.30---F2[10]
表3(续)
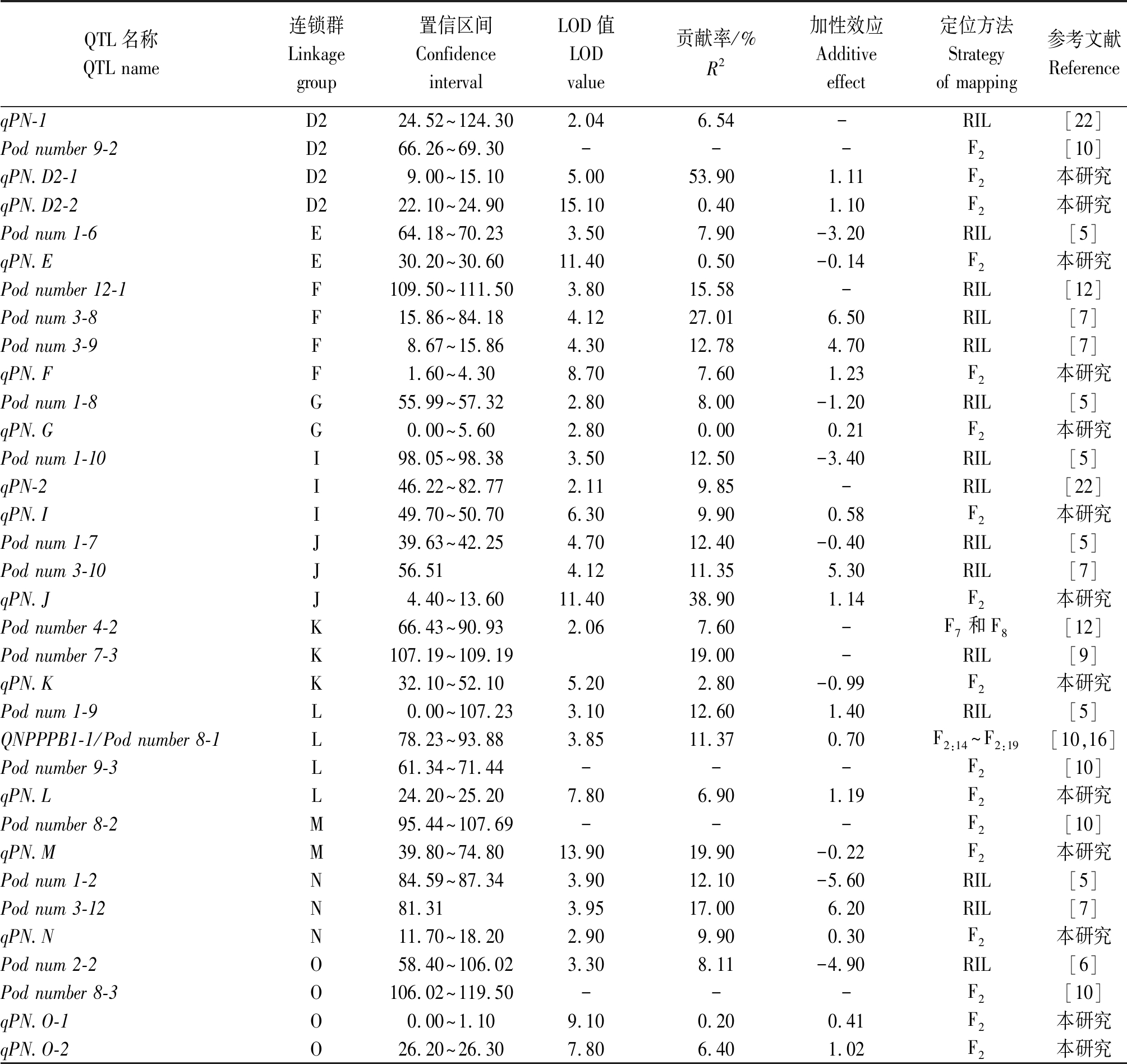
QTL名称QTL name连锁群Linkagegroup置信区间 Confidence intervalLOD值LODvalue贡献率/%R2加性效应Additive effect定位方法 Strategy of mapping参考文献ReferenceqPN-1D224.52~124.302.04 6.54 - RIL[22]Pod number 9-2D266.26~69.30---F2[10]qPN.D2-1D29.00~15.105.00 53.90 1.11 F2本研究qPN.D2-2D222.10~24.9015.10 0.40 1.10 F2本研究Pod num 1-6E64.18~70.233.50 7.90 -3.20 RIL[5]qPN.EE30.20~30.6011.40 0.50 -0.14 F2本研究Pod number 12-1F109.50~111.503.80 15.58 -RIL[12]Pod num 3-8F15.86~84.184.12 27.01 6.50 RIL[7]Pod num 3-9F8.67~15.864.30 12.78 4.70 RIL[7]qPN.FF1.60~4.308.70 7.60 1.23 F2本研究Pod num 1-8G55.99~57.322.80 8.00 -1.20 RIL[5]qPN.GG0.00~5.602.80 0.00 0.21 F2本研究Pod num 1-10I98.05~98.383.50 12.50 -3.40 RIL[5]qPN-2I46.22~82.772.11 9.85 -RIL[22]qPN.II49.70~50.706.30 9.90 0.58 F2本研究Pod num 1-7J39.63~42.254.70 12.40 -0.40 RIL[5]Pod num 3-10J56.514.12 11.35 5.30 RIL[7]qPN.JJ4.40~13.6011.40 38.90 1.14 F2本研究Pod number 4-2K66.43~90.932.06 7.60 -F7和F8[12]Pod number 7-3K107.19~109.1919.00 -RIL[9]qPN.KK32.10~52.105.20 2.80 -0.99 F2本研究Pod num 1-9L0.00~107.233.10 12.60 1.40 RIL[5]QNPPPB1-1/Pod number 8-1L78.23~93.883.85 11.37 0.70 F2:14~F2:19[10,16]Pod number 9-3L61.34~71.44---F2[10]qPN.LL24.20~25.207.80 6.90 1.19 F2本研究Pod number 8-2M95.44~107.69---F2[10]qPN.MM39.80~74.8013.90 19.90 -0.22 F2本研究Pod num 1-2N84.59~87.343.90 12.10 -5.60 RIL[5]Pod num 3-12N81.313.95 17.00 6.20 RIL[7]qPN.NN11.70~18.202.90 9.90 0.30 F2本研究Pod num 2-2O58.40~106.023.30 8.11 -4.90 RIL[6]Pod number 8-3O106.02~119.50---F2[10]qPN.O-1O0.00~1.109.10 0.20 0.41 F2本研究qPN.O-2O26.20~26.307.80 6.40 1.02 F2本研究
3 结论与讨论
由于前人对大豆荚数QTL定位研究所用的遗传图谱和标记不一致,以至于目前还未得到一个完整的大豆荚数分子遗传图谱。本研究基于已有研究的大豆遗传图谱,将大豆荚数QTL整合在同一张遗传图谱上。这些QTLs几乎分布于大豆20条染色体上(除H),其绝大部分的贡献率在20%以下,只有几个达到20%以上[7],这几个QTLs可为后续大豆单株荚数QTL精细定位和基因克隆奠定基础。
本研究共定位了23个大豆单株荚数QTLs,其中有5个与已报道的QTL重叠,另外18个代表新的大豆单株荚数QTL定位,填补了在B2染色体上没有检测到大豆单株荚数QTL的空白(本研究定位了qPN.B2)。qPN.C2-4和qPN.C1虽然与前人研究重叠[5, 7],但是本研究的主效QTL贡献率高于前人报道,说明QTL-qPN.C2-4和QTL-qPN.C1是控制大豆单株荚数的主效QTLs位点,可被不同材料和不同环境重复检测,可作为后续精细定位和基因克隆的首选QTLs。同时本研究多环境检测到qPN.A2-1的贡献率为56.4%,是目前检测到的效应值最大的QTL,并且与前人研究没有重叠。因此可以看作是一个新的控制大豆单株荚数的QTL。
整合前人对大豆单株荚数的研究发现,目前仅处于一个初步研究阶段,还未阐明大豆单株荚数分子机理。Wang等[21]在大豆中克隆GmCYP78A10基因,通过分析发现,GmCYP78A10基因的表达影响大豆单株荚数。目前,还未有其他有关基因在大豆中报道,这可能与大豆单株荚数性状复杂并且受环境条件影响较大难以精准鉴定有关。
总之,本研究定位的3个主效QTLs贡献率均较大,完全具备进一步精细定位和基因克隆的基础,从而可为大豆单株荚数遗传改良打下坚实的理论基础和技术服务。
[1] 王秋玲,郭凌云,刘艳,张桂花,刘凤洲. 夏大豆单株产量与主要农艺性状的灰色关联度分析 [J]. 大豆通报,2002(6): 5,12.doi:10.3969/j.issn.1674-3547.2002.06.005.
Wang Q L, Guo L Y, Liu Y, Zhang G H, Liu F Z.Analysis of grey related degree between individual plant yield and main agriculture character of summer soybean [J]. Soybean Bulletin, 2002(6):5,12.
[2] 王芳,李传仁. 大豆单株荚数与主要农艺性状关系的分析 [J]. 种业导刊,2008(11): 24-25,27.doi: 10.3969/j.issn.1003-4749.2008.11.006.
Wang F, Li C R. The analysis of the relationship between pod number per plant and the main agronomic traits in soybean [J]. Journal of Seed Industry Guide, 2008(11): 24-25,27.
[3] 戴瓯和. 夏大豆荚粒性状遗传变异、遗传相关和选择指数研究的初报 [J]. 安徽农业科学, 1982(2): 46-50.doi: 10.13989/j.cnki.0517-6611.1982.02.010.
Dai O H. The report of genetic variation, genetic correlation and selection index research of seed and pod in summer soybean [J]. Journal of Anhui Agricultural Sciences, 1982(2): 46-50.
[4] Qu B, Zhang W, Chen X H, Li N, Cui N, Li T L. Research progress of flower bud differentiation mechanism of plant [J]. Chinese Agricultural Science Bulletin, 2010, 26(24): 109-114.
[5] Zhang D,Cheng H,Wang H,Zhang H Y,Liu C Y,Yu D Y. Identification of genomic regions determining flower and pod numbers development in soybean(Glycine max L.)[J]. Journal of Genetics and Genomics, 2010, 37(8): 545-556. doi:10.1016/S1673-8527(09)60074-6.
[6] Chen Q S, Zhang Z C, Liu C Y, Xin D W, Qiu H M, Shan D P,Shan C Y, Hu G H. QTL analysis of major agronomic traits in soybean [J]. Agricultural Sciences in China, 2007, 6(4): 399-405. doi:10.1016/S1671-2927(07)60062-5.
[7] Sun D S, Li W B, Zhang Z C, Chen Q S, Ning H L, Qiu L J, Sun G L. Quantitative trait loci analysis for the developmental behavior of Soybean(Glycinemax L. Merr.)[J]. Theoretical and Applied Genetics, 2006, 112(4): 665-673.doi:10.1007/s00122-005-0169-y.
[8] Liu W X, Moon Young Kim, Van K, Lee Y H, Li H L, Liu X H, Lee S H. QTL identification of yield-related traits and their association with flowering and maturity in soybean [J]. Journal of Crop Science and Biotechnology, 2011, 14(1): 65-70. doi:10.1007/s12892-010-0115-7.
[9] Palomeque L, Liu L J, Li W B, Hedges B, Cober E R,Rajcan I. QTL in mega-environments:Ⅱ. agronomic trait QTL co-localized with seed yield QTL detected in a population derived from a cross of high-yielding adapted×high-yielding exotic soybean lines[J]. Theoretical and Applied Genetics, 2009, 119(3):429-436. doi:10.1007/s00122-009-1048-8.
[10] Yosuke Kuroda, Akito Kaga, Norihiko Tomooka, Hiroshi Yano, Yoshitake Takada,Shin Kato, Duncan Vaughan. QTL affecting fitness of hybrids between wild and cultivated soybeans in experimental fields [J]. Ecology and Evolution, 2013, 3(7): 2150-2168. doi:10.1002/ece3.606.
[11] Sangrea Shim, Moon Young Kim, Jungmin Ha, Yeong-Ho Lee, Suk-Ha Lee. Identification of QTLs for branching in soybean(Glycine max(L.)Merrill)[J]. Euphytica, 2017, 213(9): 225. doi:10.1007/s10681-017-2016-z.
[12] Bobby R, Bazzelle R, Clark W, Kantartzi S, Meksem K, Akond M, Kassem M A. Genetic analysis of yield components and seed isoflavones contents in the PI 438489B by "Hamilton" recombinant inbred line(RIL)population of soybean(Glycine max(L.)Merr)[J]. Journal of Agricultural Science, 2012, 4(9): 697-702. doi:10.5539/jas.v4n9p98.
[13] 李莹莹,李瑞超,程春光,赵圆圆,刘春燕,齐照明,李灿东,王囡囡,蒋洪蔚,陈庆山. 大豆荚粒数相关QTL的Meta和Overview分析及其候选基因预测 [J]. 农业生物技术学报,2018,26(11): 1821-1833. doi: 10.3969/j.issn.1674-7968.2018.11.001.
Li Y Y, Li R C, Cheng C G, Zhao Y Y, Liu C Y, Qi Z M, Li C D, Wang N N, Jiang H W, Chen Q S. Meta and overview analysis of QTL associated with pod and seed traits and candidate gene mining in Soybean(Glycine max)[J]. Journal of Agricultural Biotechnology, 2018, 26(11): 1821-1833.
[14] 杨胜先,牛远,李梦,魏世平,刘晓芬,吕海燕,章元明. 栽培大豆农艺性状的关联分析及优异等位变异挖掘[J].中国农业科学,2014,47(20):3941-3952. doi:10.3864/j.issn. 0578-1752.2014.20.002.
Yang S X, Niu Y, Li M, Wei S P, Liu X F, L H Y, Zhang Y M. Association mapping of agronomic traits in soybean(Glycine max L. Merr.)and mining of novel alleles[J]. Scientia Agricultura Sinica, 2014, 47(20):3941-3952.
[15] 徐琰,孙晓环,孙霞,王燕平,宗春美,齐玉鑫,白艳凤,任海洋,潘相文,杜维广,孔凡江,刘宝辉.大豆花荚脱落及单株荚数的QTL定位 [J]. 土壤与作物,2015,4(2): 71-76. doi:10.11689/j.issn.2095-2961.2015.02.004.
Xu Y, Sun X H, Sun X, Wang Y P, Zong C M, Qi Y X, Bai Y F, Ren H Y, Pan X W, Du W G, Kong F J, Liu B H. QTL mapping of flower and pod abscission and pod number per plant in soybean [J]. Soil and Crop, 2015, 4(2): 71-76.
[16] Orf J H, Chaseb K, Jarvikb T, Mansurc L M, Cregand P B, Adlerb F R, Lark K G. Genetics of soybean agronomic traits: I. Comparison of three related recombinant inbred populations [J]. Crop Science, 1999, 39(6): 1642-1651.doi: 10.2135/cropsci1999.3961642x.
[17] 吉林省农业科学院大豆研究所. 中国大豆品种志(1978-1992)[M]. 北京: 农业出版社,1993.
Soybean Institute of Jilin Agricultural Sciences. Chinese soybean cultivar catologue(1978-1992)[M]. Beijing: Agriculture Press, 1993.
[18] Zeng Z B. Precision mapping of quantitative trait loci [J]. Genetics, 1994, 136(4): 1457-1468.
[19] Churchill G A,Doerge R W.Empirical threshold values for quantitative trait mapping[J]. Genetics, 1994, 138(3): 963-971.
[20] Goffinet B, Gerber S. Quantitative trait loci: a meta-analysis [J]. Genetics, 2000, 155(1): 463-473.
[21] Wang X B, Li Y H, Zhang H W, Sun G L, Zhang W M, Qiu L J. Evolution and association analysis of GmCYP78A10 gene with seed size/weight and pod number in soybean [J]. Molecular Biology Reports, 2015, 42(2): 489-496. doi:10.1007/s11033-014-3792-3.