黄瓜(Cucumis sativus)在蔬菜生产和供应中占有极其重要的地位,但在农业生产特别是南方多雨季节或北方冬春季设施栽培中,很容易受到环境因素的影响而授粉受精不良,不能正常膨大,最终导致果实“化瓜”,严重影响了黄瓜的产量、品质和栽培效益。黄瓜果实膨大是一个复杂的生物学过程,迄今已从生理生化和分子水平进行了探索,但是有关黄瓜果实膨大的分子机制仍不清楚。
植物中的miRNA是一类重要的调控分子,通过剪切靶mRNA调节基因的表达,进而调控植物生长发育、胁迫应答和新陈代谢等多种功能[1]。研究表明,miRNA参与果实发育,在果实形状、大小、着色、成熟等方面发挥了重要作用[2]。Daniel等[3]发现,miRNA参与甜瓜果实发育调控。Zhang等[4]鉴定了哈密瓜果实发育中的miRNA及其靶基因,预测miR159、miR164、miR319、miR393和miR396及其靶基因参与了哈密瓜果实早期发育。黄瓜生长发育调控课题组在前期构建果实膨大small RNA文库和qRT-PCR分析的基础上,发现miR399b参与黄瓜果实膨大调控。已有研究表明,miR399在植物激素响应[5]、花期调控[6-7]和品质调控[8]等方面发挥重要作用。
本课题组在前期研究中还发现,黄瓜miR399基因家族的靶基因之一为泛素结合酶24(Ubiquitin conjugating enzyme E2 24, UBC24)。UBC是泛素-蛋白酶体途径(UPP)的关键作用酶之一[9],在种子膨大[10]、果实成熟调控[11-13]、光周期调控[14]、维管分化调控[15]、DNA修复[14,16-17]和逆境胁迫响应[18-20]等方面发挥着重要功能。目前,有关UBC参与黄瓜果实膨大的研究还未见报道。
本研究分析了黄瓜CsamiR399b的序列特征及其表达特性,并对靶基因CsUBC24进行了生物信息学分析和功能预测,为深入研究CsamiR399b及其靶基因CsUBC24在黄瓜果实膨大中的功能及其作用机制提供理论依据。
1 材料和方法
1.1 植物材料
以河南科技学院园艺园林学院试验基地蔬菜大棚非单性结实的高代自交黄瓜全雌系Cs0401为试材,于2018年7月17日分别采取黄瓜开花当天子房、授粉后5 d果实(膨大)和未授粉5 d子房(未膨大)样品,用无菌水清洗干净,切碎后液氮速冻,置于-80 ℃超低温冰箱冻存备用。
1.2 CsamiR399b序列及表达分析
从先前建立的small RNA文库和miBase数据库(http://www.mirbase.org/)获得CsamiR399b前体序列及成熟体序列,利用RNA Folding Form(http://unafold.rna.albany.edu/?q=mfold/RNA-Folding-Form)在线预测CsamiR399b前体序列的茎环结构。
分别提取黄瓜开花当天子房、授粉后5 d果实和未授粉5 d子房样品的总RNA,使用 Aidlab 公司反转录试剂盒(TUREscript 1st Stand cDNA SYNTHESIS Kit)合成cDNA,以U6为内参基因,用TaKaRa公司的实时荧光定量 PCR 试剂盒在ABI PRISM 7500 实时荧光定量PCR系统中进行扩增,检测CsamiR399b的表达特性,引物见表1。
表1 引物信息
Tab.1 Primer information
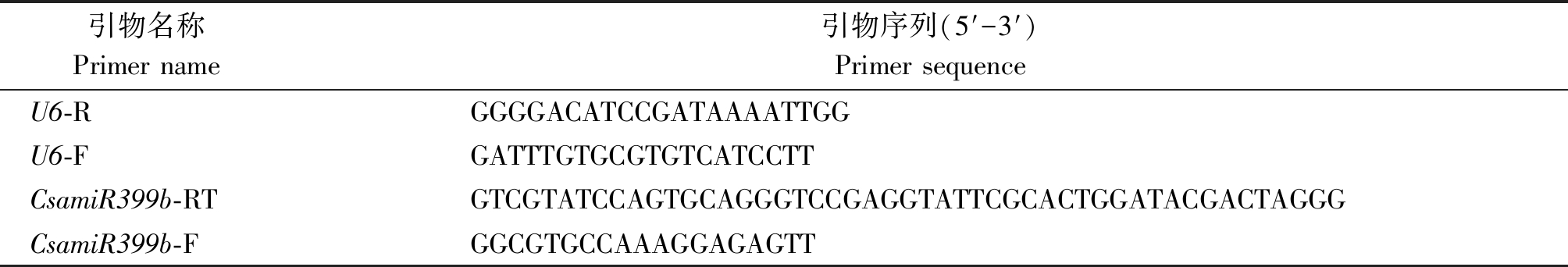
引物名称Primer name引物序列(5′-3′)Primer sequenceU6-RGGGGACATCCGATAAAATTGGU6-FGATTTGTGCGTGTCATCCTTCsamiR399b-RTGTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACTAGGGCsamiR399b-FGGCGTGCCAAAGGAGAGTT
1.3 CsUBC24的生物信息学分析
前期通过降解组测序发现,CsamiR399b靶基因为CsUBC24。从NCBI数据库(https://www.ncbi.nlm.nih.gov/)获得靶基因CsUBC24的蛋白质序列。使用NPS@:SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl? page=npsa_sopma.html)预测蛋白质的二级结构;使用SignalP 5.0(http://www.cbs.dtu.dk/services/SignalP/)进行信号肽分析;通过TMHMM Server v2.0(http://www.cbs.dtu.dk/services/TMHMM/)进行蛋白质跨膜性预测;利用WoLFPSORT(https://www.genscript.com/wolf-psort.html)、TargetP 1.1 Server(http://www.cbs.dtu.dk/services/TargetP/)和Plant-mPLoc(http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)对CsUBC24蛋白进行亚细胞定位分析;利用ProtScale(https://web.expasy.org/protscale/)进行亲疏水性分析;使用DNAMAN软件进行多序列比对;使用 MEGA 5.0 软件构建系统进化树;利用GSDS 2.0(http://gsds.cbi.pku.edu.cn/index.php)分析基因序列结构;利用SMART(http://smart.embl-heidelberg.de/)和NCBI进行保守结构域分析。
2 结果与分析
2.1 CsamiR399b序列分析
从small RNA文库中获取了CsamiR399b成熟体序列,长度为21 nt,从miBase数据库获得其前体序列。经RNA Folding Form预测发现,CsamiR399b基因前体序列具有稳定的二级发夹结构和二级茎环结构,其中成熟序列位于基因前体序列的3′端臂上(图1)。
2.2 CsamiR399b的表达分析
荧光定量PCR分析结果显示,与开花当天子房和未授粉5 d子房相比,CsamiR399b在授粉后5 d果实中表达量最高,为开花当天子房的2.5倍,为未授粉5 d子房的4倍(图2)。这说明CsamiR399b的表达与黄瓜果实膨大呈正相关,证实其参与黄瓜果实膨大调控。
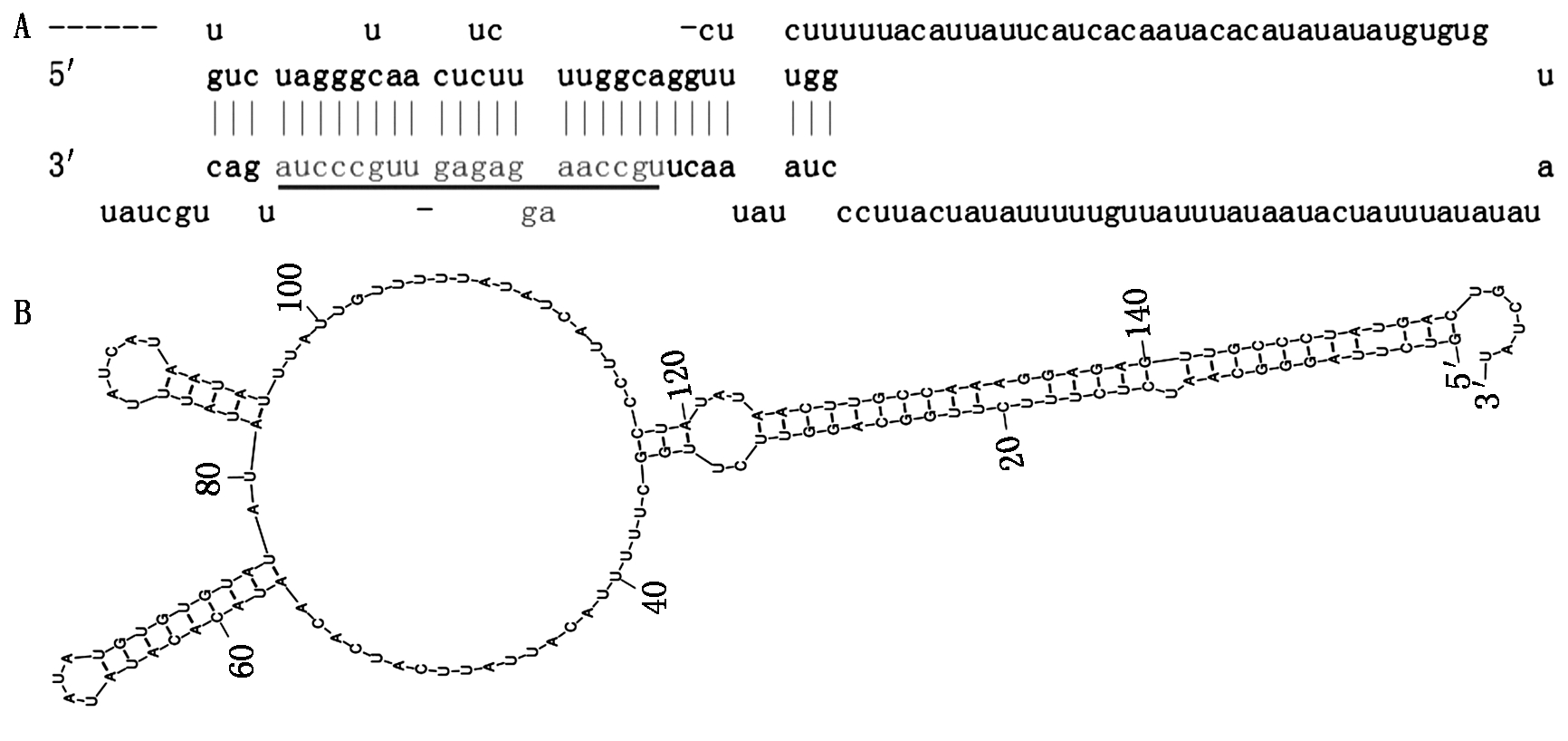
划线部分表示成熟序列。
The underlined part is the mature sequences.
图1 CsamiR399b二级发夹结构(A)和茎环结构(B)
Fig.1 Secondary hairpin structure(A)and stem-loop(B)of CsamiR399b
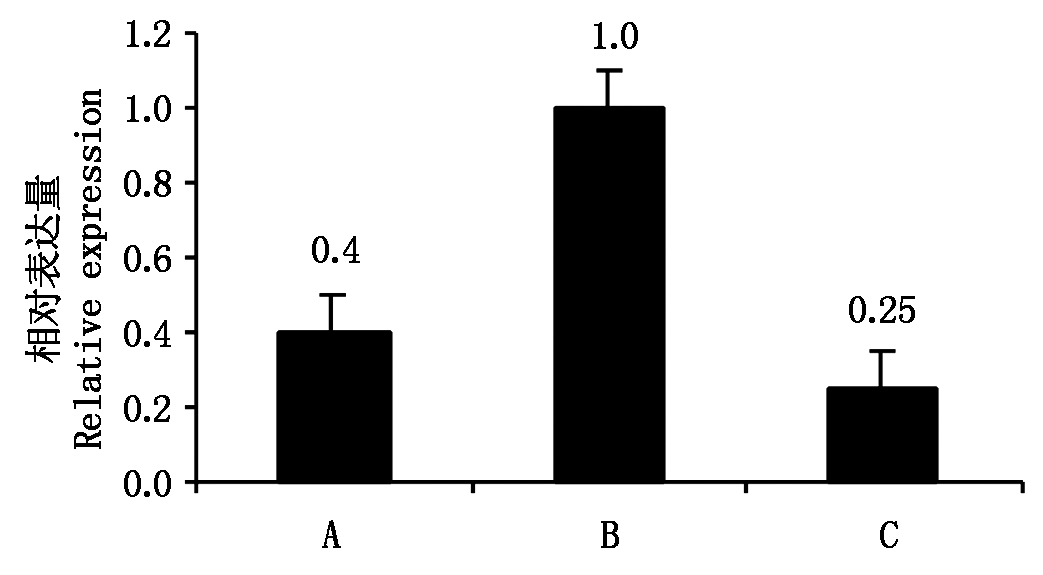
A.开花当天子房; B. 授粉后5 d果实(膨大); C. 未授粉5 d子房(未膨大)。
A. Ovary on the day of anthesis without pollination; B. 5 d fruit after pollination(expansion); C. 5 d ovary without pollination(unexpansion).
图2 CsamiR399b的表达特性
Fig.2 Expression characteristics of CsamiR399b
2.3 CsUBC24蛋白性状分析
通过NPS@:SOPMA分析了黄瓜CsUBC24蛋白序列,预测其二级结构,发现黄瓜CsUBC24蛋白主要由4种构象组成(表2),其中无规则卷曲(Cc)含有424个氨基酸,占整体的46.59%,比例最高;其次为α-螺旋(Hh)和延伸链(Ee),分别由246,176个氨基酸构成,占整体的27.03%和19.34%;β-转角(Tt)比例较少,有64个氨基酸,占整体的7.03%。蛋白质的N 端是否带有信号肽可判别该蛋白质是否为分泌蛋白,一个标准的信号肽,其C值和Y值应约等于+1,在剪切位点前后S值应该呈先上升后下降趋势。使用SignalP 5.0对黄瓜CsUBC24蛋白进行信号肽分析,得到C、Y、S值分别为0.111,0.105,0.121(表2)。可以判断,CsUBC24蛋白没有信号肽,不是分泌蛋白。
表2 CsUBC24蛋白二级结构和信号肽预测
Tab.2 Secondary structure and signal peptide prediction of CsUBC24 protein

蛋白质Protein二级结构组成Secondary structure composition信号肽分值Signal peptide scoreCc/%Hh/%Ee/%Tt/%CYSCsUBC2446.5927.0319.347.030.1110.1050.121
分析跨膜蛋白不仅可以了解特定生物膜的功能,而且有助于进一步确定蛋白质的定位与功能。经蛋白质跨膜性预测分析,CsUBC24蛋白没有跨膜结构,全部在膜外(图3)。细胞中蛋白质的位置决定了该蛋白质的功能。通过在线软件TargetP 1.1 Server预测黄瓜CsUBC24蛋白的亚细胞定位,发现CsUBC24蛋白位于叶绿体、线粒体或分泌通路的可能性较小(表3)。利用WoLF PSORT Prediction预测黄瓜CsUBC24蛋白的分布位置可知,该蛋白质位于细胞核。通过在线软件Plant-mPLoc再次验证,确定CsUBC24蛋白定位于细胞核上。CsUBC24蛋白的亲水氨基酸所占比例高达62.75%,而疏水性氨基酸为36.37%(图4)。由于亲水性区域所占比例远大于疏水性区域,因此,可以确定CsUBC24蛋白为可溶性蛋白。其中,亲水性最强的是第836位的丙氨酸,其值为-2.856。
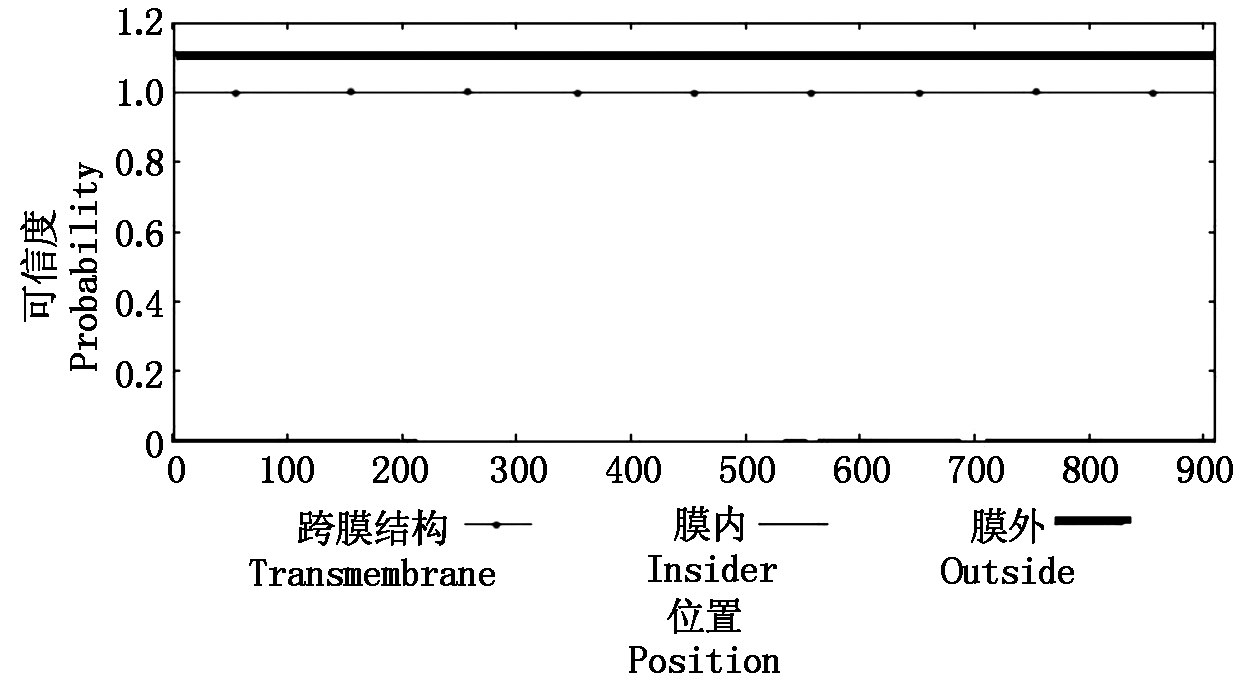
图3 CsUBC24蛋白的跨膜结构预测
Fig.3 Prediction of transmembrane structure of CsUBC24 protein
表3 CsUBC24蛋白的亚细胞定位预测
Tab.3 Prediction of subcellular location of CsUBC24 protein
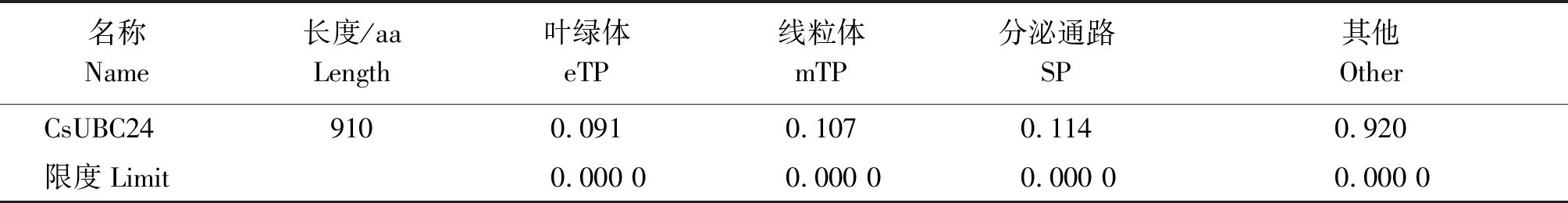
名称Name长度/aaLength叶绿体eTP线粒体mTP分泌通路SP其他OtherCsUBC24 9100.0910.1070.1140.920限度Limit 0.000 00.000 00.000 00.000 0
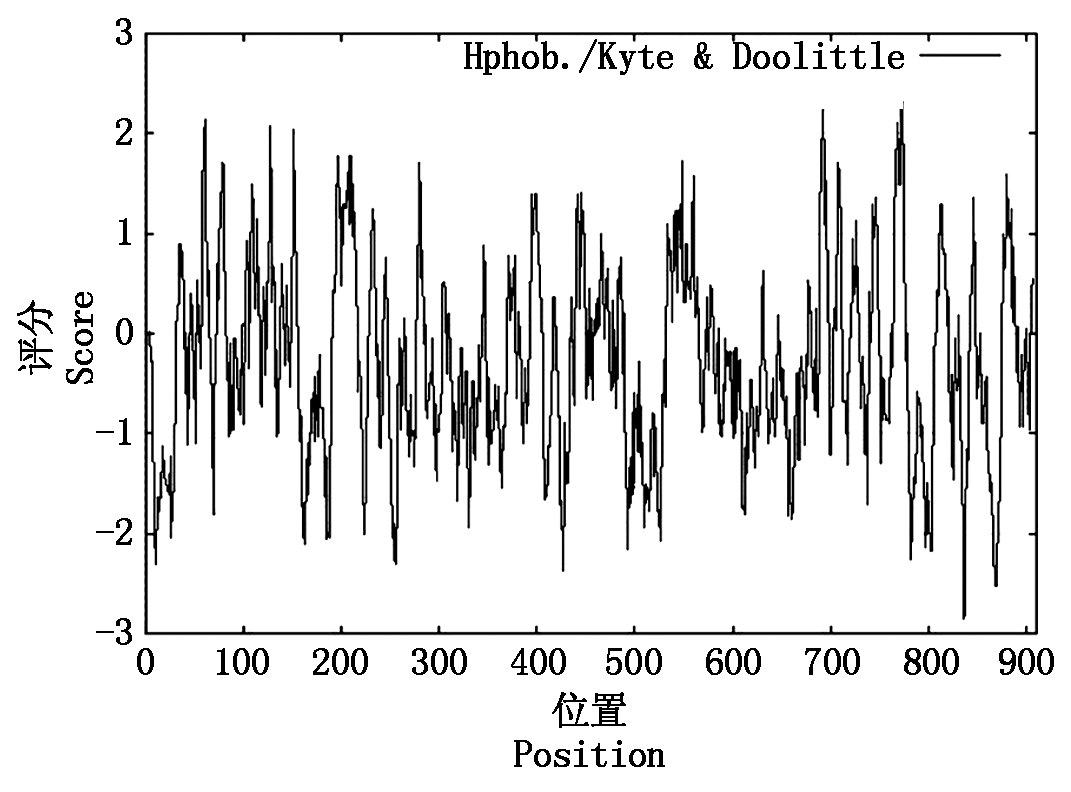
图4 CsUBC24蛋白的亲疏水性预测
Fig.4 The hydrophobicity prediction of CsUBC24 protein
2.4 CsUBC24序列分析
对CsUBC24蛋白进行Blast同源序列比对,用DNAMAN对黄瓜CsUBC24以及其同源物种的UBC24氨基酸序列进行多序列比对,用MEGA 5.0构建系统进化树,分析CsUBC24蛋白的进化关系。结果表明,黄瓜CsUBC24蛋白与甜瓜(Cucumis melo)UBC24蛋白进化关系最近,其次为南瓜(Cucurbita moschata)和苦瓜(Momordica charantia)UBC24蛋白(图5)。
使用SMART分析黄瓜CsUBC24蛋白的功能结构域,发现CsUBC24蛋白序列660-817位氨基酸为泛素结合酶E2结构域(UBCc),并且含有2个低成分复杂区域(Low complexity region)。同时使用NCBI在线预测CsUBC24的功能结构域,结果与SMART分析结果基本一致(图6),该蛋白质同样属于UBCc超级家族,结构域位于660-812位氨基酸。进一步对黄瓜、甜瓜、南瓜和苦瓜UBC24蛋白的功能结构域分析发现,4个物种均含有UBCc保守域,且由数量及位置大致相同的氨基酸构成,甜瓜、南瓜和苦瓜UBC24蛋白同样属于UBCc超级家族。这说明UBC24蛋白在各物种间功能保守。对黄瓜、甜瓜、南瓜和苦瓜UBC24基因核苷酸序列分析发现,黄瓜CsUBC24基因与甜瓜、南瓜和苦瓜UBC24基因结构相似,编码区长度基本相同,因此认为该基因的外显子区相对保守。氨基酸序列比对发现,UBC24蛋白家族氨基酸序列在整体上基本相同,具有较强的保守性,几乎含有共同的保守区域(图7)。
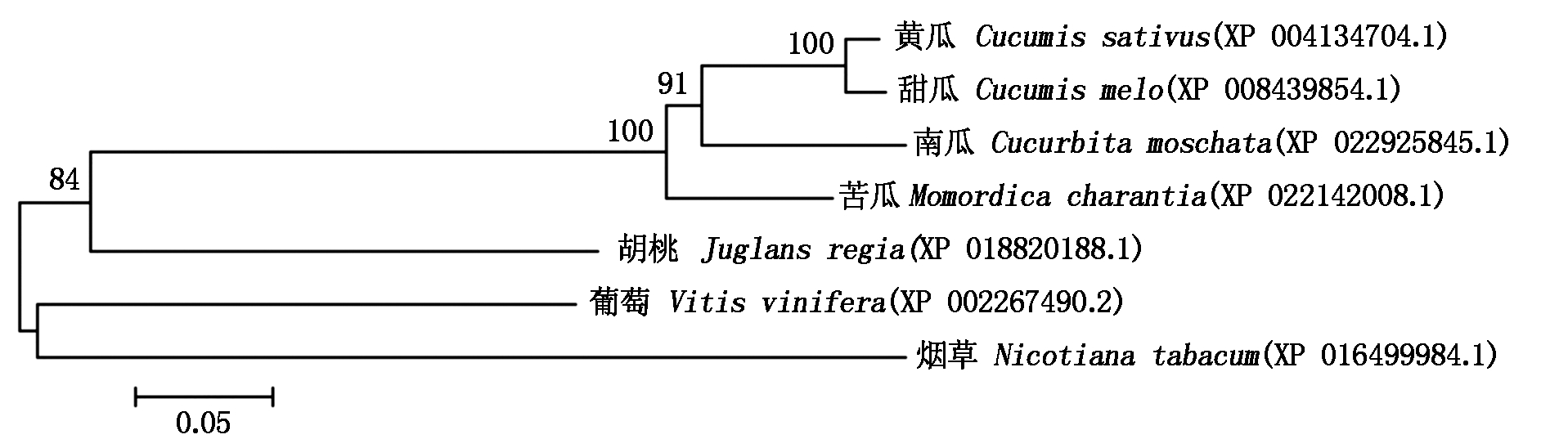
图5 UBC24家族系统进化树
Fig.5 Phylogenetic tree of UBC24 family

图6 CsUBC24蛋白的保守结构域预测
Fig.6 The conservative domain prediction of CsUBC24 protein

划线部分为功能结构域。
The underlined part is the functional domain.
图7 UBC24家族氨基酸序列比对
Fig.7 Amino acid sequence alignment of UBC24 family
3 结论与讨论
miR399家族是一类与植物耐低磷胁迫密切相关的miRNA家族[21-22],参与调节磷营养的吸收和转运或响应低磷胁迫诱导[23-26]。此外还发现,在野生型草莓(Fragaria × ananassa Duch.)果实中miR399表达水平与糖含量呈负相关性,其中果实发育期间miR399表达差异显著[27],因此其可能参与果实发育。本试验采用荧光定量PCR技术对黄瓜果实膨大期CsamiR399b的表达量进行分析,发现CsamiR399b在膨大的黄瓜果实中明显上调,证实CsamiR399b参与黄瓜果实膨大调控。本研究丰富了miR399在植物中的功能,为深入研究miR399与果实膨大的关系提供了理论基础。
生物信息学分析发现,黄瓜CsamiR399b的靶基因CsUBC24编码蛋白具有910个氨基酸。该蛋白质与小白菜BCUBCE2[28]、龙葵SorUBC[29]和菜心BclUBE2[30]蛋白质性状基本相同,其主要是由无规则卷曲与α-螺旋构成二级结构,没有信号肽及跨膜结构,定位于细胞核,为可溶性蛋白。黄瓜CsUBC24具有UBC蛋白共有的UBCc结构域,是UBCc超级家族的成员。黄瓜CsUBC24与甜瓜CmUBC24的同源性最高,其次为南瓜和苦瓜。多序列比对结果表明,不同种类植物的UBC24具有较高的相似性和序列保守性,该结果与Baloglu等[31]的研究结果相一致。这说明UBC家族是一个高保守基因家族。同一家族中基因的结构较为相似,并有相似的保守元件,那么该家族基因可能具有相似的功能[32]。高保守基因家族在不同植物中通常具有相类似的功能[33]。王园等[11]研究发现,香蕉果实跃变成熟后期MaUCE1表达增强,推测MaUCE1在果实成熟与衰老过程中发挥作用。高营营[13]通过对葡萄果实成熟过程中VvUBCs的表达模式分析发现,大多数VvUBCs在果实成熟过程中显著上调或下调,说明VvUBCs在葡萄果实成熟中发挥了重要作用。据此推测,UBC参与调控植物果实发育,且CsamiR399b可能通过调控CsUBC24基因表达参与了黄瓜果实膨大,本研究为揭示黄瓜果实膨大机理提供了理论基础。要进一步确定CsamiR399b及其靶基因CsUBC24在黄瓜果实膨大中的作用,还需通过基因克隆和遗传转化进行功能验证。
[1] 董淼,黄越,陈文铎,徐涛,郎秋蕾. 降解组测序技术在植物miRNA研究中的应用[J].植物学报,2013,48(3): 344-353.doi:10.3724/SP.J.1259.2013.00344.
Dong M,Huang Y,Chen W D,Xu T,Lang Q L. Use of degradome sequencing in study of plant microRNAs[J]. Chinese Bulletin of Botany,2013,48(3): 344-353.
[2] Chen C C,Zeng Z H,Liu Z R,Xia R. Small RNAs,emerging regulators critical for the development of horticultural traits[J]. Horticulture Research,2018,5(1): 63.
[3] Daniel G I,José B,Livia D,Montserrat S,Albert M C,Ana C D,Jordi G M,Cesar L,Miguel A A. Analysis of the melon(Cucumis melo)small RNAome by high-throughput pyrosequencing[J]. BMC Genomics,2011,12(1): 393. doi: 10.1186/1471-2164-12-393.
[4] Zhang H,Yin L,Wang H,Wang G,Ma X,Li M,Wu H,Fu Q,Zhang Y,Yi H. Genome-wide identification of Hami melon miRNAs with putative roles during fruit development[J]. PLoS One,2017,12(7): e0180600. doi:10.1371/journal.pone.0180600.
[5] 陈晓婷,刘军,孙翠霞,林桂芳. 草酸胁迫下拟南芥三个差异表达microRNA的分析[J]. 农业生物技术学报,2014,22(4): 432-439. doi: 10.3969/j.issn.1674-7968.2014.04.005.
Chen X T,Liu J,Sun C X,Lin G F. Three differentially expressed microRNAs in Arabidopsis thaliana under the stress of oxalic acid[J]. Journal of Agricultural Biotechnology,2014,22(4): 432-439.
[6] Kim W H,Ahn H J,Chiou T J,Ahn J H. The role of the miR399-PHO2 module in the regulation of flowering time in response to different ambient temperatures in Arabidopsis thaliana[J]. Molecules and Cells,2011,32(1): 83-88. doi: 10.1007/s10059-011-1043-1.
[7] 陶平. miR399调控大豆磷平衡及开花的初步研究[D]. 广州: 华南农业大学,2016.
Tao P. Studies on the roles of miR399 in phosphorus homeostasis and flowering time in soybean[D]. Guangzhou: South China Agricultural University,2016.
[8] Wang Y,Zhang J X,Cui W X,Guan C Y,Mao W J,Zhang Z H. Improvement of fruit quality by overexpressing miR399 in woodland strawberry[J]. Journal of Agricultural and Food Chemistry,2017,65(34): 7361-7370. doi: 10.1021/acs.jafc.7b01687.
[9] 蒋忠荣,申飞,刘志,郭瑞,李桥,闫海芳. 津田芜菁泛素结合酶Brubc11基因的克隆及表达分析[J]. 华北农学报,2015,30(1): 97-102. doi: 10.7668/hbnxb.2015.01.016.
Jiang Z R,Shen F,Liu Z,Guo R,Li Q,Yan H F. Cloning and expression analysis of ubiquitin-conjugating enzyme 11(BrUBC11)in Brassica rapa L. subsp. Rapa Tsuda[J]. Acta Agriculturae Boreali-Sinica,2015,30(1): 97-102.
[10] 林萍,姚小华,曹永庆,龙伟,王开良,腾建华. 普通油茶泛素结合酶UBE2-J2的cDNA序列及蛋白质结构分析[J]. 林业科学研究,2013,26(6): 744-751. doi: 10.3969/j.issn.1001-1498.2013.06.012.
Lin P,Yao X H,Cao Y Q,Long W,Wang K L,Teng J H. Characterization of a novel ubiquitin-conjugating enzyme from Camellia oleifera[J]. Forest Research,2013,26(6): 744-751.
[11] 王园,王甲水,谢学立,雷晓明,金志强. 香蕉泛素结合酶基因与果实成熟关系的研究[J]. 园艺学报,2010,37(5): 705-712.
Wang Y,Wang J S,Xie X L,Lei X M,Jin Z Q. Studies of the relationship between MaUCE1 and banana fruit ripening[J]. Acta Horticulturae Sinica,2010,37(5): 705-712.
[12] Wang Y Y,Wang W H,Cai J H,Zhang Y R,Qin G Z,Tian S P. Tomato nuclear proteome reveals the involvement of specific E2 ubiquitin-conjugating enzymes in fruit ripening[J]. Genome Biology,2014,15(12): 548. doi: 10.1186/s13059-014-0548-2.
[13] 高营营. 葡萄泛素结合酶E2基因家族全基因组鉴定及功能结构分析[D]. 北京: 中国科学院大学,2018.
Gao Y Y. Genome-wide identification and function-structure analysis of ubiquitin-conjugating E2 genes family in grape[D]. Beijing: University of Chinese Academy of Sciences,2018.
[14] Hoege C,Pfander B,Moldovan G L,Pyrowolakis G,Jentsch S. Rad6-dependent DNA repair is linked to modification of PCNA by ubiquitin and SUMO[J]. Nature,2002,419(6903): 135-141. doi: 10.1038/nature00991.
[15] Lau O S,Deng X W. Effect of Arabidopsis COP10 ubiquitin E2 enhancement activity across E2 families and functional conservation among its canonical homologues[J]. Biochemical Journal,2009,418(3): 683-690. doi: 10.1042/bj20081943.
[16] Wang B,Elledge S.Ubc13/Rnf8 ubiquitin ligases control foci formation of the Rap80/Abraxas/Brca1/Brcc36 complex in response to DNA damage[J]. Proceedings of the National Academy of Sciences,2007,104(52): 20759-20763. doi: 10.1073/pnas.0710061104.
[17] Huen M S Y,Huang J,Yuan J,Yamamoto M,Akira S,Ashley C,Xiao W,Chen J. Noncanonical E2 variant-independent function of UBC13 in promoting checkpoint protein assembly[J]. Molecular and Cellular Biology,2008,28(19): 6104-6112. doi: 10.1128/MCB.00987-08.
[18] Feussner K,Feussner I,Leopold I,Wasternack C. Isolation of a cDNA coding for an ubiquitin-conjugating enzyme UBC1 of tomato-The first stress-induced UBC of higher plants [J]. Febs Letters,1997,409(2): 211-215. doi: 10.1016/S0014-5793(97)00509-7.
[19] Zhou G A,Chang R Z,Qiu L J. Overexpression of soybean ubiquitin-conjugating enzyme gene GmUBC2 confers enhanced drought and salt tolerance through modulating abiotic stress-responsive gene expression in Arabidopsis[J]. Plant Molecular Biology,2010,72(4/5): 357-367. doi: 10.1007/s11103-009-9575-x.
[20] 徐东北,于月华,韩巧玲,马亚男,高世庆,田野,徐兆师,李连城,曲延英,马有志,李明,李耀锋. 大豆(Glycine max)GmDREB5互作蛋白GmUBC13的特性及功能[J]. 中国农业科学,2014,47(18): 3534-3544. doi: 10.3864/j.issn.0578-1752.2014.18.002.
Xu D B,Yu Y H,Han Q L,Ma Y N,Gao S Q,Tian Y,Xu Z S,Li L C,Qu Y Y,Ma Y Z,Li M,Li Y F. Characteristics and function of a GmDREB5-interacting protein GmUBC13 in soybean[J]. Scientia Agricultura Sinica,2014,47(18): 3534-3544.
[21] Sunkar R,Chinnusamy V,Zhu J,Zhu J K. Small RNAs as big players in plant abiotic stress responses and nutrient deprivation[J]. Trends in Plant Science,2007,12(7): 301-309. doi: 10.1016/j.tplants.2007.05.001.
[22] Li H,Mao W,Liu W,Dai H,Liu Y,Ma Y, Zhang Z. Deep sequencing discovery of novel and conserved microRNAs in wild type and a white-flesh mutant strawberry[J]. Planta,2010,238(4): 695-713. doi: 10.1007/s00425-013-1917-x.
[23] Wang C,Ying S,Huang H,Li K,Shou H. Involvement of OsSPX1 in phosphate homeostasis in rice[J]. The Plant Journal,2008,57(5):895-904. doi: 10.1111/j.1365-313X.2008.03734.x.
[24] Wang Z,Hu H,Huang H J,Duan K,Wu Z C,Wu P. Regulation of OsSPX1 and OsSPX3 on expression of OsSPX domain genes and Pi-starvation signaling in rice[J]. Journal of Integrative Plant Biology,2009,51(7): 663-674. doi:10.1111/j.1744-7909.2009.00834.x.
[25] 孙佃臣,沙爱华,单志慧,陈李淼,周新安. 拟南芥pre-miR399b植物表达载体的构建[J]. 华北农学报,2010,25(6): 30-33. doi: 10.7668/hbnxb.2010.06.006.
Sun D C,Sha A H,Shan Z H,Chen L M,Zhou X A. Construction of a plant expression vector targeting the Arabidopsis thaliana pre-miR399b gene[J]. Acta Agriculturae Boreali-Sinica,2010,25(6): 30-33.
[26] Hackenberg M,Shi B J,Gustafson P,Langridge P. Characterization of phosphorus-regulated miR399 and miR827 and their isomirs in barley under phosphorus-sufficient and phosphorus-deficient conditions[J]. BMC Plant Biology,2013,13(1): 214-231. doi: 10.1186/1471-2229-13-214.
[27] Lin S I,Chiang S F,Lin W Y,Chen J W,Tseng C Y,Chiou W T J. Regulatory network of microRNA399 and PHO2 by systemic signaling[J]. Plant Physiology,2008,147(2): 732-746. doi:10.1104/pp.108.116269.
[28] 赵瑞丽,钟凤林,林义章,高世超,林俊芳,杨碧云,胡海非. 小白菜BcUBCE2基因的克隆及表达分析[J]. 西北植物学报,2014,34(1): 60-65. doi: 10.7606/j.issn.1000-4025.2014.01.0060.
Zhao R L,Zhong F L,Lin Y Z,Gao S C,Lin J F,Yang B Y,Hu H F. Clone and expression analysis of BcUBCE2 gene from Brassica chinensis L.[J]. Acta Botanica Boreali-Occidentalia Sinica,2014,34(1): 60-65.
[29] 蔡佳文,金晓霞,于丽杰,崔柏杨,魏讯,董延龙. 龙葵E2泛素结合酶基因SorUBC克隆及表达特性分析[J]. 东北农业大学学报,2016,47(11): 26-36. doi: 10.3969/j.issn.1005-9369.2016.11.004.
Cai J W,Jin X X,Yu L J,Cui B Y,Wei X,Dong Y L. Clone and expression pattern analysis of E2 ubiquitin-binding enzyme gene SorUBC in Solanum nigrum L.[J]. Journal of Northeast Agricultural University,2016,47(11): 26-36.
[30] 曾小玲,赵瑞丽,钟开勤,朱朝辉,陈敏氡. 菜心BclUBE2基因的克隆与表达分析[J]. 热带作物学报,2018,39(9): 1772-1777. doi: 10.3969/j.issn.1000-2561.2018.09.014.
Zeng X L,Zhao R L,Zhong K Q,Zhu C H,Chen M D. Cloning and expression analysis of ubiquitin-conjugating enzyme BclUBE2 gene in Brassica campestris L. ssp. chinensis var. utilis Tsen et Lee[J]. Chinese Journal of Tropical Crops,2018,39(9): 1772-1777.
[31] Baloglu M C,Patir M G. Molecular characterization, 3D model analysis, and expression pattern of the CmUBC gene encoding the melon ubiquitin-conjugating enzyme under drought and salt stress conditions[J]. Biochemical Genetics,2014,52(1/2): 90-105. doi: 10.1007/s10528-013-9630-9.
[32] 赵雪惠,王庆杰,李晨,陈修德,肖伟,高东升,付喜玲. 桃ERF转录因子家族生物信息学分析及芽萌发相关基因筛选[J]. 植物学报,2018,53(5): 612-624. doi: 10.11983/CBB17117.
Zhao X H,Wang Q J,Li C,Chen X D,Xiao W,Gao D S,Fu X L. Genome-wide identification of ethylene responsive factor(ERF)family genes in peach and screening of genes related to germination[J]. Chinese Bulletin of Botany,2018,53(5): 612-624.
[33] 莫远亮,邓艳,王继文. miR-181a-5p靶基因预测及其在鹅卵泡颗粒层中的表达分析[J]. 华北农学报,2018,33(6): 95-102. doi: 10.7668/hbnxb.2018.06.013.
Mo Y L,Deng Y,Wang J W. Prediction of miR-181a-5p target gene and its expression analysis in goose follicle granular layer[J]. Acta Agriculturae Boreali-Sinica,2018,33(6): 95-102.