细胞分裂素是一类重要的植物激素,在细胞分裂、调控顶端优势、促进芽的分化、抑制主根伸长、叶绿素合成、延缓叶片衰老、种子发育、打破种子休眠、抵御盐和干旱等非生物胁迫及病原菌抵抗等过程中发挥了重要作用[1-8]。
植物细胞分裂素受体属于组氨酸蛋白激酶家族,拟南芥中有3个细胞分裂素受体,分别为AHK2、AHK3 和CRE1(又名AHK4 或WOL1)[9]。在拟南芥原生质体中瞬时过表达这3个受体中的任何一个都能够增加对细胞分裂素的敏感性[10]。细胞分裂素3个受体基因的单突变体对外源细胞分裂素的敏感性有不同程度的降低,双突变体对外源细胞分裂素的敏感性进一步降低,但除ahk2 ahk3外,其余单突变体和双突变体在正常生长条件下均无明显表型[11-12]。然而,ahk2 ahk3 ahk4三重突变体对外源细胞分裂素不敏感且存在严重的发育缺陷,如地上部分和根部的严重缩小、侧根数目减少、维管组织分化缺陷、叶片衰老加速、种子增大等[11, 13],暗示细胞分裂素受体参与了植株正常生长发育的许多方面,并且存在一定的功能叠加。此外,AHK2、AHK3和AHK4受干旱诱导表达,ahk2和ahk3单突变体以及ahk2 ahk3双突变体对干旱的抗性增加;表达谱分析显示,ahk2 ahk3双突变体中,大量胁迫和ABA诱导的基因表达量升高,表明AHK2和AHK3是干旱信号转导途径中的负调节因子。另外,ahk2、ahk3和cre1单突变体对外源ABA超敏感,表明AHK2、AHK3和CRE1同样是ABA信号转导途径的负调节因子[14]。
本研究以拟南芥细胞分裂素受体基因AHK4氨基酸序列为参考序列,同源克隆小麦细胞分裂素受体基因TaHK1,并利用生物信息学软件分析预测该基因的结构及其编码蛋白的理化性质、定位、功能结构域和表达规律等,为今后克隆该基因并开展功能研究奠定理论基础。
1 材料和方法
1.1 试验材料
利用拟南芥细胞分裂素受体基因AHK4(At2g01830)氨基酸序列比对EnsemblPlants小麦数据库(http://plants.ensembl.org),得到与其相似度最高的基因序列,以及该基因的基本信息(包括基因长度和外显子、内含子数目等)。
1.2 试验方法
1.2.1 TaHK1的生物信息学分析 目的基因的生物信息学分析主要参照高嵩等[15]的方法。具体如下:目的基因编码蛋白的理化性质通过ExPASy-ProtParam(https://web.expasy.org/protparam/)在线预测;目的基因编码蛋白的疏水性通过软件ExPASy-ProtScale(https://web.expasy.org/protscale/)在线分析,目的基因编码蛋白的二级结构通过NPS@:SOPM(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)在线预测;目的基因编码蛋白的亚细胞定位通过TargetP 1.1 Server(http://www.cbs.dtu.dk/services/TargetP/)、WoLF PSORT(https://wolfpsort.hgc.jp/)和iPSORT(http://ipsort.hgc.jp/)在线预测;通过SignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP/)预测目的基因编码蛋白的信号肽;通过TMHMM Server, v. 2.0(http://www.cbs.dtu.dk/services/TMHMM/)预测目的基因编码蛋白的跨膜域;蛋白的保守结构域通过SMART(http://smart.embl-heidelberg.de/)在线预测;蛋白的磷酸化修饰位点通过NetPhos 3.1(http://www.cbs.dtu.dk/services/NetPhos/)在线预测;目的基因的表达量通过Wheat Expression Browser(http://www.wheat-expression.com/)在线分析。
1.2.2 植物总RNA的提取及cDAN合成 利用TRNzol Reagent(TIANGEN)提取植物总RNA。利用PrimeScriptTM RT reagent Kit with gDNA Eraser(TaKaRa)反转录合成cDNA。
1.2.3 荧光实时定量PCR 利用Primer Express软件对目标基因进行qRT-PCR引物设计。上游引物TaHK1-QF:5′-AATGGCACTTCGCTAACCAG-3′,下游引物TaHK1-QR:5′-TTCACACCCGTTCTGTTTCA-3′。以小麦TaActin为内参,用SYBR®Premix ExTaq Ⅱ(TaKaRa)试剂盒进行实时荧光定量PCR分析,TaActin引物序列、扩增体系和程序参照孙丽静等[16]的方法。
2 结果与分析
2.1 小麦TaHK1序列的获得
以拟南芥AHK4(At2g01830)为参考序列,在EnsemblPlants小麦数据库中进行同源序列比对,得到同源性最高的基因TRIAE_CS42_4DS_TGACv1_362760 _AA1181940(EnsemblPlants),位于小麦4D染色体上,命名为TaHK1。TaHK1 基因组DNA序列全长7 190 bp,包含12个外显子和11个内含子(图1)。TaHK1 cDNA全长3 747 bp,包含一个2 991 bp的开放阅读框,编码996个氨基酸。

矩形.外显子;折线.内含子。
Rectangle.Exon; Broken line. Intron.
图1 TaHK1基因结构预测
Fig.1 Prediction of the TaHK1 gene structure
2.2 蛋白理化性质分析
通过ExPASy-ProtParam[17]预测TaHK1基因编码蛋白的理化性质。该蛋白的分子质量为109.70 ku,理论等电点为5.71;-0.148的亲水性平均系数表明该蛋白为亲水性蛋白(表1)。通过ExPASy网站提供的ProtScale工具进一步分析TaHK1基因编码蛋白的疏水性,峰值分布在0以下的氨基酸(56.48%)比分布在0以上的氨基酸(43.52%)多,同样表明该蛋白为亲水性蛋白(图2)。氨基酸组成及含量分析显示,TaHK1中含量最高的为Leu,占整体的10.2%(图3)。
表1 TaHK1蛋白理化性质分析
Tab.1 Physical and chemical property analysis of TaHK1
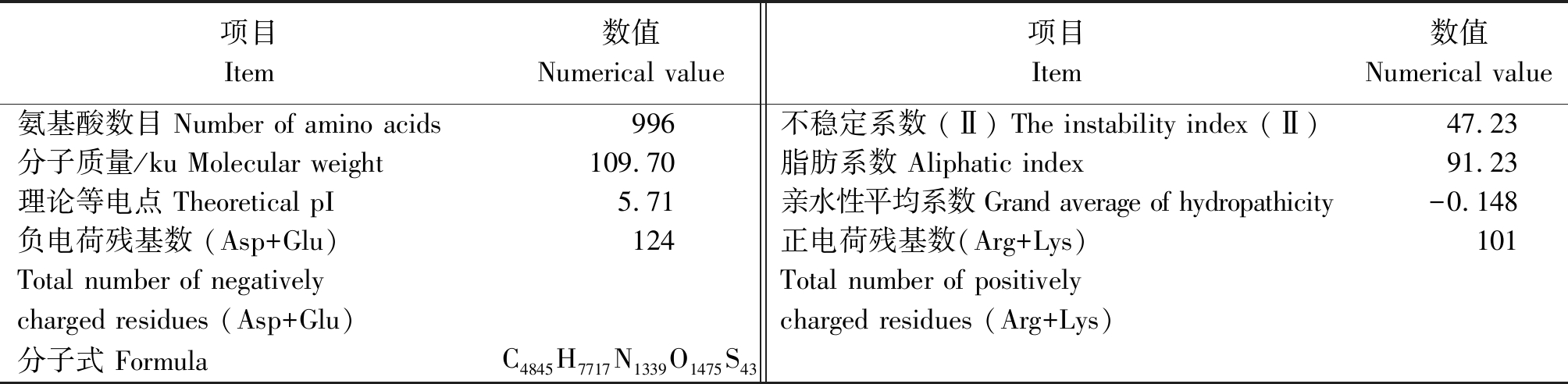
项目Item数值Numerical value项目Item数值Numerical value氨基酸数目 Number of amino acids996不稳定系数 (Ⅱ) The instability index (Ⅱ) 47.23分子质量/ku Molecular weight 109.70脂肪系数 Aliphatic index 91.23理论等电点 Theoretical pI 5.71亲水性平均系数 Grand average of hydropathicity -0.148负电荷残基数 (Asp+Glu) 124正电荷残基数(Arg+Lys) 101Total number of negativelyTotal number of positivelycharged residues (Asp+Glu)charged residues (Arg+Lys)分子式 FormulaC4845H7717N1339O1475S43
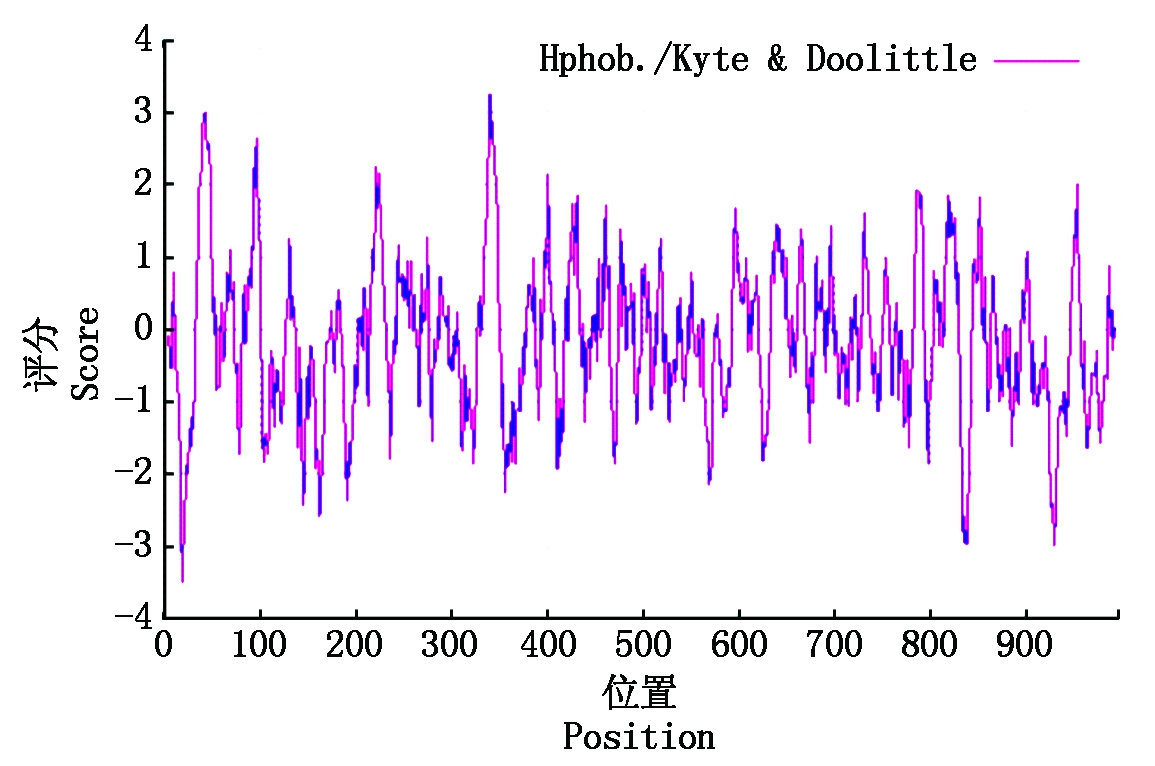
图2 TaHK1蛋白疏水性预测
Fig.2 Prediction of the TaHK1 protein hydrophobicity
2.3 蛋白二级结构预测
通过NPS@:SOPMA[18]分析TaHK1蛋白的二级结构,发现TaHK1蛋白中α-螺旋(Hh)比例最高,占比47.89%;之后依次为无规则卷曲(Cc)、延伸链(Ee)和β-转角(Tt),占比分别为35.04%,12.95%和4.12%(图4)。
2.4 蛋白质亚细胞定位预测
了解基因编码蛋白的亚细胞定位有助于了解该基因的功能。通过TargetP[19] 预测TaHK1蛋白的分布位置,发现其位于叶绿体、线粒体和分泌到细胞外的可能性很小,位于其他细胞器的可能性较大(表2)。
通过WoLF PSORT在线预测TaHK1蛋白的亚细胞定位,发现其细胞膜定位系数为8(plas:8),内质网定位系数为3(E.R.:3),细胞核定位系数为1(nucl:1),细胞质定位系数为1(cyto:1),液泡定位系数为1(vacu:1)。综上推测,TaHK1定位于细胞膜的可能性较大,这与其作为细胞分裂素受体接受来自细胞外的信号并将其转导到细胞内的功能是一致的。
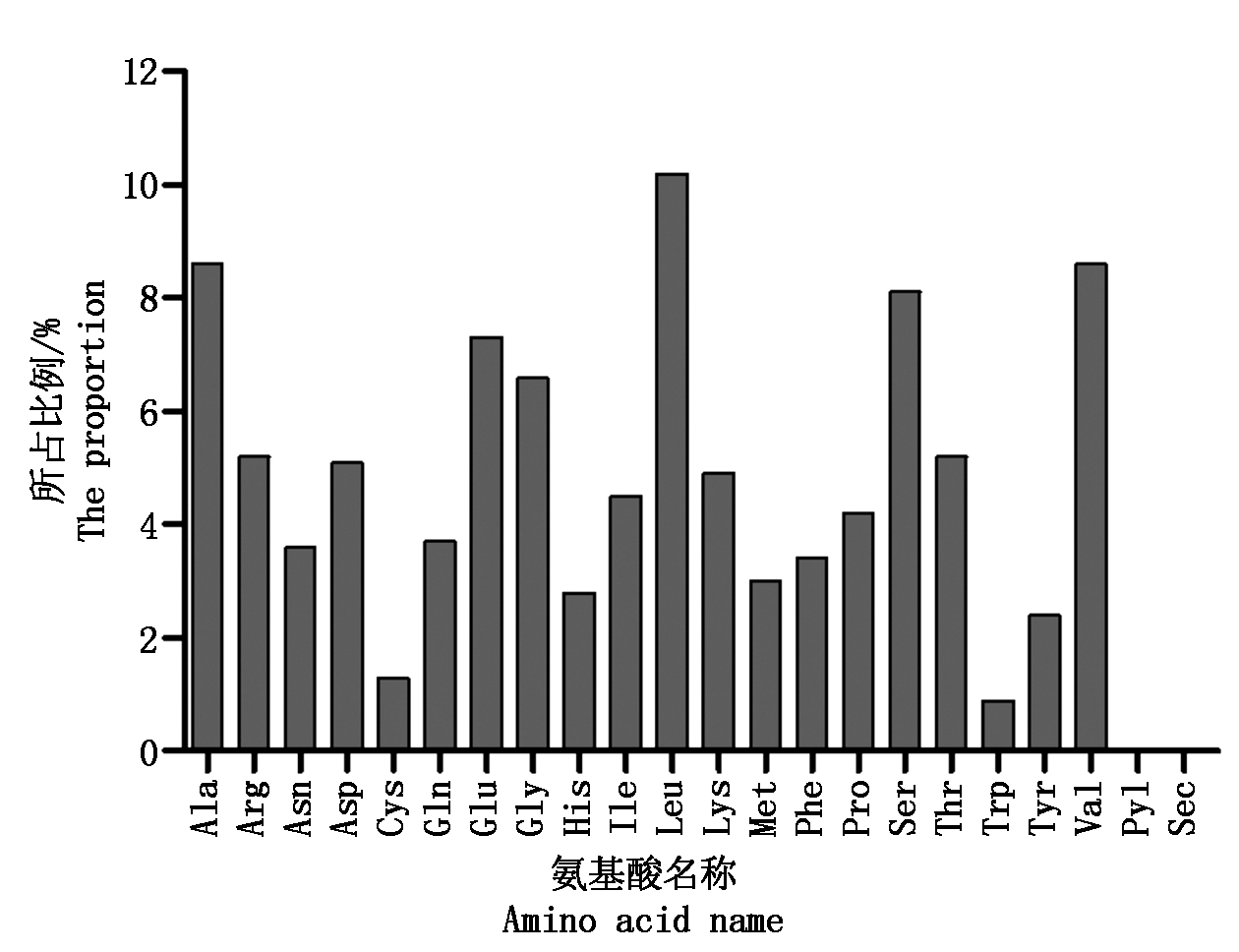
图3 TaHK1氨基酸组成及含量分析
Fig.3 Amino acid composition and content in TaHK1

图4 TaHK1二级结构预测
Fig.4 Secondary structure prediction of TaHK1
表2 TaHK1蛋白亚细胞定位预测
Tab.2 Subcellular location prediction of TaHK1
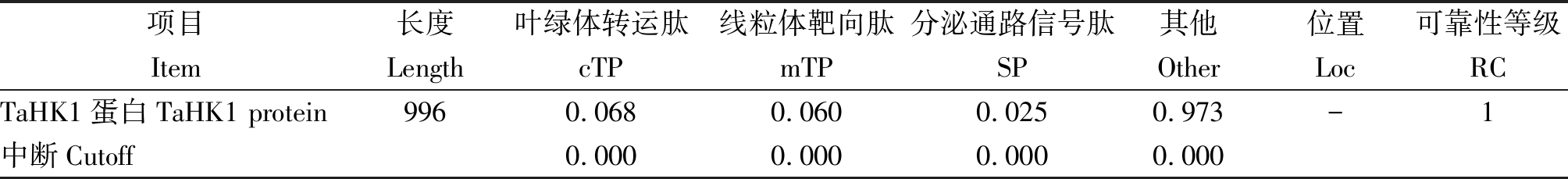
项目长度叶绿体转运肽线粒体靶向肽分泌通路信号肽其他位置可靠性等级ItemLengthcTPmTPSPOtherLocRCTaHK1蛋白TaHK1 protein9960.0680.0600.0250.973-1中断Cutoff0.0000.0000.0000.000
2.5 蛋白质信号肽预测
通过SignalP[20]对TaHK1蛋白进行信号肽预测,发现TaHK1蛋白没有信号肽(图5)。iPSORT[21]预测结果同样表明,TaHK1蛋白没有信号肽,并且也不含线粒体转运肽和叶绿体转运肽,与SignalP预测结果一致。
2.6 蛋白质跨膜结构预测
通过TMHMM[22]对TaHK1蛋白进行跨膜结构域预测,与拟南芥AHK4相同,TaHK1也有2个跨膜域,分别位于第34-53位氨基酸和326-348位氨基酸(图6),与WoLF PSORT预测的其细胞膜定位结果一致。
图5 TaHK1信号肽预测
Fig.5 Signal peptide prediction of TaHK1
A.小麦TaHK1;B.拟南芥AHK4.
A. TaHK1 of wheat;B. AHK4 of Arabidopsis.
图6 TaHK1跨膜结构预测
Fig.6 Transmembrane structure prediction of TaHK1
2.7 蛋白质保守结构域预测
通过SMART在线软件[23]分析TaHK1蛋白的保守结构域。结果显示,TaHK1第34-53位氨基酸和第326-348位氨基酸为2个跨膜域(Transmembrane region),与TMHMM分析结果一致;第106-290位氨基酸为CHASE(Cyclaseand histidine kinase-associated sensing extracellular)结构域,负责受体与细胞分裂素的结合;第378-443位氨基酸为组氨酸激酶(His kinase A domain)结构域;第490-671位氨基酸为类组氨酸激酶ATP酶(Histidine kinase-like ATPases)结构域;第851-986位氨基酸为REC(cheY-homologous receiver domain)结构域,负责接受来自激酶域的磷酸基团,并向信号转导通路下游的组氨酸磷酸转运蛋白传递(图7)。
2.8 磷酸化修饰位点预测
蛋白质磷酸化是调节和控制蛋白质活力和功能的最基本、最普遍,也是最重要的机制,在细胞信号转导的过程中起重要作用。通过NetPhos[24]对TaHK1蛋白进行磷酸化位点预测,发现TaHK1共有87个磷酸化位点,其中丝氨酸(Ser)磷酸化位点最多,为56个;其次为苏氨酸(Thr)和酪氨酸(Tyr)磷酸化位点,分别为25,6个(图8)。
图7 TaHK1保守结构域预测
Fig.7 Conserved domain prediction of TaHK1
图8 TaHK1磷酸化位点预测
Fig.8 The phosphorylation sites prediction of TaHK1
2.9 TaHK1基因的表达特性分析
为了更好的了解TaHK1的功能,通过Wheat Expression Browser在线分析TaHK1基因在不同组织的表达模式[25]。结果显示,TaHK1在根、茎、叶、雄蕊和小穗中的表达量较高,而在籽粒的糊粉层、淀粉性胚乳和种皮等组织的表达量较低(图9),暗示该基因可能在小麦生长发育的很多阶段和组织均发挥着一定作用。
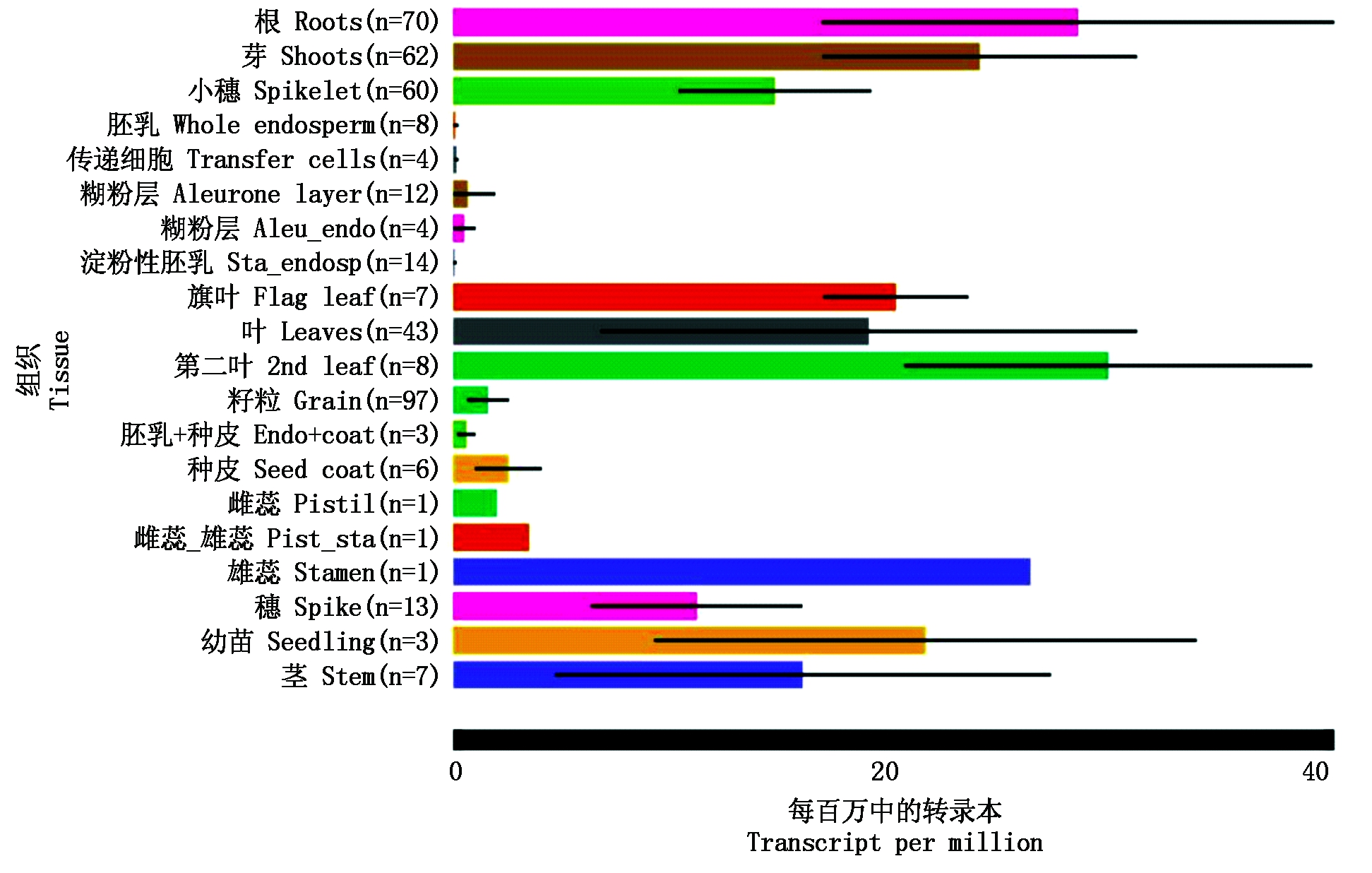
图9 TaHK1在小麦不同组织中的表达分析
Fig.9 Relative expression of TaHK1 in different tissues of wheat
此外,分析结果显示,TaHK1基因对胁迫处理和病原菌接种也有一定的响应。TaHK1表达量在低磷胁迫、接种叶枯病菌4 d、接种条锈病菌48 h和72 h、接种白粉病菌72 h明显升高;而在干旱胁迫6 h,TaHK1表达量则显著降低(图10)。以上结果表明,TaHK1在小麦抵御病原菌侵染和干旱胁迫的过程中也发挥着重要作用。
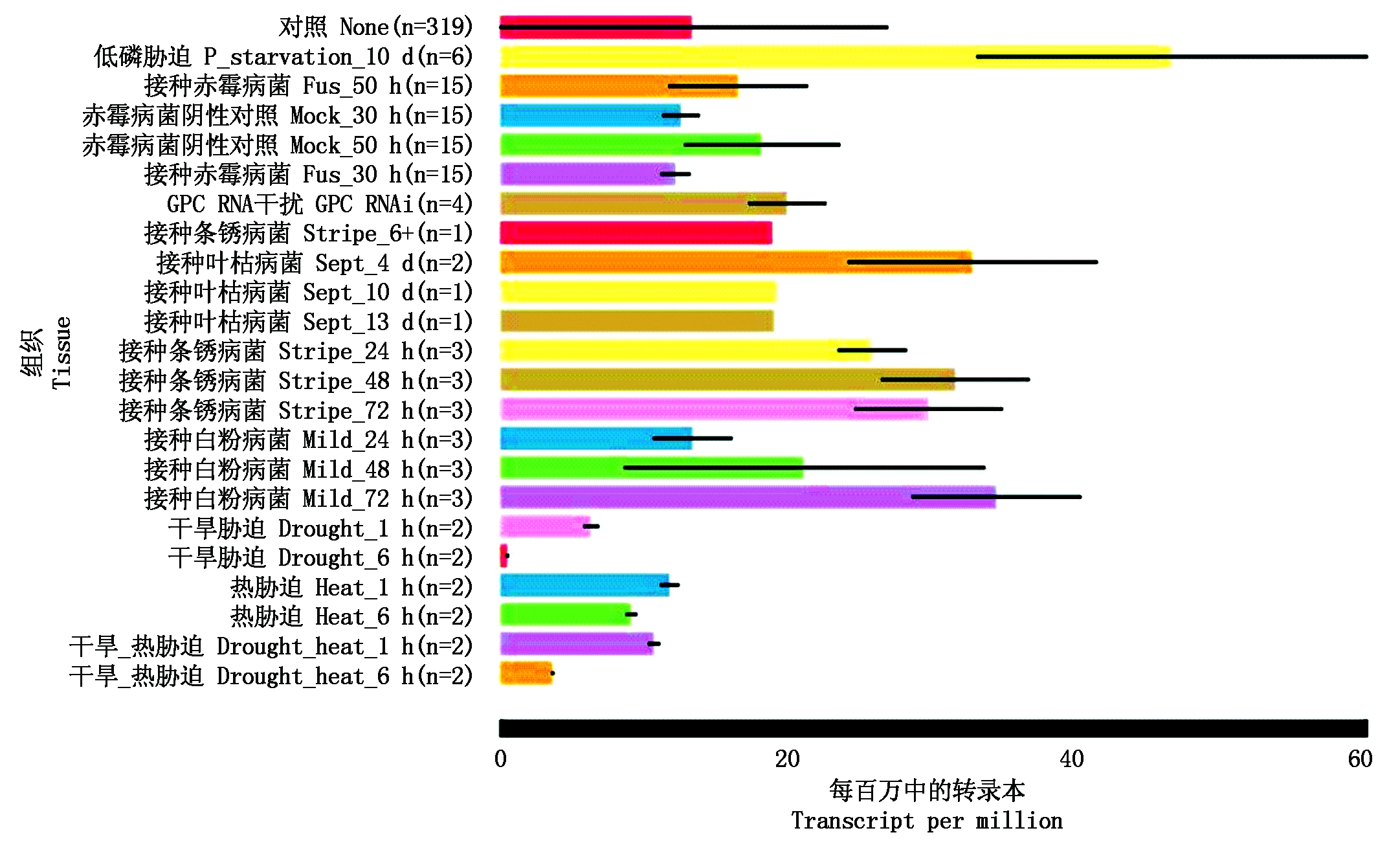
图10 TaHK1在胁迫处理和病原菌侵染下的相对表达量
Fig.10 Relative expression of TaHK1 under stress and disease treatments
2.10 实时荧光定量PCR分析TaHK1基因在小麦科农199不同组织的表达特性
为了进一步了解TaHK1基因在小麦科农199不同组织的表达特性,为其功能研究奠定基础,本研究提取科农199不同组织RNA,通过实时荧光定量PCR方法对TaHK1基因表达量进行了检测。结果显示,TaHK1在幼叶中表达量最高,其次是穗部,而幼根、旗叶等其他组织中表达量较低,在成熟根中的表达量最低(图11)。
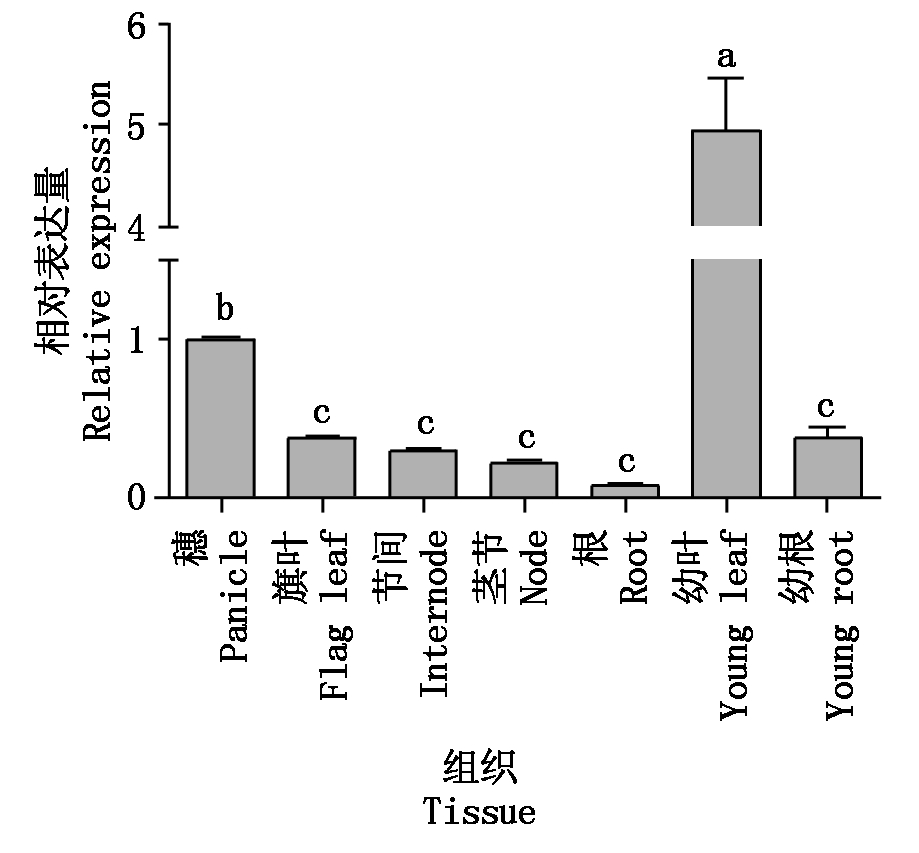
图柱上不同字母表示组织间在0.05水平上差异显著。
Values followed by different letters are significantly different at the 0.05 probability level.
图11 实时荧光定量PCR分析TaHK1在小麦科农199不同组织中的表达特性
Fig.11 Relative expression of TaHK1 in different tissues of wheat Kenong 199 by qRT-PCR
3 结论与讨论
TaHK1是小麦中的细胞分裂素受体基因,研究其编码蛋白的结构和功能将为小麦中细胞分裂素信号转导通路的研究奠定坚实的基础。本研究通过生物信息学分析发现,TaHK1蛋白由996个氨基酸组成,分子质量为109.70 ku,理论等电点为5.71;TaHK1蛋白二级结构由α-螺旋、无规则卷曲、延伸链和β-转角4种构象组成;通过亚细胞定位预测,TaHK1定位于细胞膜的可能性较大,并且跨膜结构域预测发现,其具有2个跨膜域;蛋白保守结构域分析发现,TaHK1具有典型的细胞分裂素受体的完整结构域,包括结合细胞分裂素的CHASE结构域、跨膜域、组氨酸激酶结构域和负责磷酸传递的REC结构域,并且含有87个磷酸化位点,推测其在细胞分裂素细胞信号转导过程中起重要作用。
细胞分裂素在植物发育[26-28]和应对盐渍、干旱等非生物胁迫[29-31] 的过程中均扮演着重要的角色。拟南芥中AHK2、AHK3和AHK4在根、茎、叶等各个组织均有广泛表达,能够很好地解释其三重突变体地上部分和根部的严重缩小、侧根数目减少、叶片衰老加速、种子增大等生长发育缺陷表型[12]。水稻中OsHK6(OsCKT1)也在根、茎、叶、幼穗等组织广泛表达[32]。与拟南芥和水稻中的同源基因类似,TaHK1在根、茎、叶、雄蕊和小穗等各个组织也有广泛表达,推测其在小麦生长发育的很多阶段和组织均发挥着一定作用。但是实时荧光定量PCR分析结果与Wheat Expression Browser在线分析TaHK1基因在不同组织的表达模式有一定的差异,推测其原因为通过网站在线分析为综合多个小麦品种多个时期的试验结果,而实时荧光定量PCR分析结果为特定小麦品种科农199中TaHK1基因的组织表达模式。另外,拟南芥中AHK2、AHK3和AHK4表达量受干旱诱导迅速上调,表型分析显示,ahk2和ahk3单突变体及ahk2 ahk3双突变体对干旱胁迫抗性增强[14]。相反,TaHK1表达量则受干旱胁迫抑制下调,推测其在小麦干旱胁迫中的功能可能与拟南芥中的同源基因不同。此外,TaHK1表达量还受低磷胁迫和多种病原菌侵染诱导,暗示其在小麦营养吸收和抵御病原菌侵染过程中可能也发挥着重要作用。
为了提高预测的准确性,本研究中的大量数据使用了多种软件进行分析,并且预测结果基本一致,具有较高的可信度。但是,生物信息学是基于已知信息进行的预测,所以具有一定的局限性。因此,TaHK1编码蛋白的性质和生物学功能还需要分子生物学和生物化学等试验的进一步验证。本研究为TaHK1的克隆提供了一定的理论依据,也为后续基因功能研究奠定了基础。
[1] Schaller G E, Bishopp A, Kieber J J. The yin-yang of hormones: cytokinin and auxin interactions in plant development[J]. Plant Cell, 2015, 27(1):44-63. doi: 10.1105/tpc.114.133595.
[2] Bielach A, Hrtyan M, Tognetti V B. Plants under stress: involvement of auxin and cytokinin[J]. Int J Mol Sci, 2017,18(7):1427. doi: 10.3390/ijms18071427.
[3] Xu Y, Burgess P, Huang B. Transcriptional regulation of hormone-synthesis and signaling pathways by overexpressing cytokinin-synthesis contributes to improved drought tolerance in creeping bentgrass[J]. Physiol Plant, 2017, 161(2):235-256. doi: 10.1111/ppl.12588.
[4] Li W, Herrera-Estrella L, Tran L P. The Yin-yang of cytokinin homeostasis and drought acclimation/adaptation[J]. Trends Plant Sci, 2016, 21(7):548-550. doi: 10.1016/j.tplants.2016.05.006.
[5] Zheng C F, Zhu Y J, Wang C Y, Guo T C. Wheat grain yield increase in response to pre-anthesis foliar application of 6-Benzylaminopurine is dependent on floret development[J]. PLoS One, 2016, 11(6):e0156627. doi: 10.1371/journal.pone.0156627.
[6] Mok D W, Mok M C. Cytokinin metabolism and action[J]. Annu Rev Plant Physiol Plant Mol Biol, 2001, 52:89-118. doi: 10.1146/annurev.arplant.52.1.89.
[7] Kundu A, DasGupta M. Silencing of putative cytokinin receptor histidine Kinase1 inhibits both inception and differentiation of root nodules in Arachis hypogaea[J]. Mol Plant Microbe Interact, 2018, 31(2):187-199. doi: 10.1094/MPMI-06-17-0144-R.
[8] Wu C, Cui K H, Wang W C, Li Q, Fahad S, Hu Q H, Huang J L, Nie L X, Mohapatra P K, Peng S B. Heat-induced cytokinin transportation and degradation are associated with reduced panicle cytokinin expression and fewer spikelets per panicle in rice[J]. Front Plant Sci, 2017, 8:371. doi: 10.3389/fpls.2017.00371.
[9] Heyl A, Schmülling T. Cytokinin signal perception and transduction[J]. Curr Opin Plant Biol, 2003, 6(5):480-488. doi: 10.1016/S1369-5266(03)00087-6.
[10] Hwang I, Sheen J. Two-component circuitry in Arabidopsis cytokinin signal transduction[J]. Nature, 2001, 413(6854):383-389. doi: 10.1038/35096500.
[11] Higuchi M, Pischke M S, Mähönen A P, Miyawaki K, Hashimoto Y, Seki M, Kobayashi M, Shinozaki K, Kato T, Tabata S, Helariutta Y, Sussman M R, Kakimoto T. In planta functions of the Arabidopsis cytokinin receptor family[J]. Proc Natl Acad Sci USA, 2004, 101(23):8821-8826. doi: 10.1073/pnas.0402887101.
[12] Nishimura C, Ohashi Y, Sato S, Kato T, Tabata S, Ueguchi C. Histidine kinase homologs that act as cytokinin receptors possess overlapping functions in the regulation of shoot and root growth in Arabidopsis[J]. Plant Cell, 2004, 16(6):1365-1377. doi: 10.1105/tpc.021477.
[13] Riefler M, Novak O, Strnad M, Schmülling T. Arabidopsis cytokinin receptor mutants reveal functions in shoot growth, leaf senescence, seed size, germination, root development, and cytokinin metabolism[J]. Plant Cell, 2006, 18(1):40-54. doi: 10.1105/tpc.105.037796.
[14] Tran L S P, Urao T, Qin F, Maruyama K, Kakimoto T, Shinozaki K, Yamaguchi-Shinozaki K. Functional analysis of AHK1/ATHK1 and cytokinin receptor histidine kinases in response to abscisic acid, drought, and salt stress in Arabidopsis[J]. Proc Natl Acad Sci USA, 2007, 104(51): 20623-20628. doi: 10.1073/pnas.0706547105.
[15] 高嵩, 吕庆雪, 何欢, 张建新, 张志军, 宋广树, 刘伟. 玉米中绒毡层发育调控基因ZmUdt1克隆的生物信息学分析[J]. 华北农学报, 2018, 33(1): 52-59.doi:10.7668/hbnxb.2018.01.009.
Gao S, Lü Q X, He H, Zhang J X, Zhang Z J, Song G S, Liu W. Bioinformatics analysis of ZmUdt1 gene in maize tapetum development[J]. Acta Agriculturae Boreali-sinica, 2018, 33(1): 52-59.
[16] 孙丽静, 张哲, 刘茜, 胡梦芸, 张颖君, 吕亮杰, 李辉. 小麦组氨酸磷酸转运蛋白TaHP4基因的克隆和表达分析[J]. 华北农学报, 2018, 33(2): 14-19. doi: 10.7668/hbnxb.2018.02.003.
Sun L J, Zhang Z, Liu Q, Hu M Y, Zhang Y J, Lü L J, Li H. Cloning and expression analysis of TaHP4 in wheat[J]. Acta Agriculturae Boreali-sinica, 2018, 33(2): 14-19.
[17] Wilkins M R, Gasteiger E, Bairoch A, Sanchez J C, Williams K L, Appel R D, Hochstrasser D F. Protein identification and analysis tools on the ExPASy server[J]. Methods Mol Biol, 1999, 112: 531-552. doi: 10.1385/1-59259-584-7:531.
[18] Geourjon C, Deléage G. SOPMA: significant improvements in protein secondary structure prediction by consensus prediction from multiple alignments[J]. Comput Appl Biosci, 1995, 11(6):681-684. doi: 10.1093/bioinformatics/11.6.681.
[19] Emanuelsson O, Nielsen H, Brunak S, von Heijne G. Predicting subcellular localization of proteins based on their N-terminal amino acid sequence[J]. Journal of Molecular Biology, 2000, 300(4):1005-1016. doi:10.1006/jmbi.2000.3903.
[20] Nielsen H. Predicting secretory proteins with SignalP[M]//Kihara D.Protein function prediction. New York:Humana Press, 2017:59-73. doi:10.1007/978-1-4939-7015-5_6.
[21] Bannai H, Tamada Y, Maruyama O, Nakai K, Miyano S. Extensive feature detection of N-terminal protein sorting signals[J].Bioinformatics,2002,18(2):298-305.doi: 10.1093/bioinformatics/18.2.298.
[22] Krogh A, Larsson B, von Heijne G, Sonnhammer E L L. Predicting transmembrane protein topology with a hidden Markov model: application to complete genomes[J]. J Mol Biol, 2001, 305(3):567-580. doi: 10.1006/jmbi.2000.4315.
[23] Letunic I, Bork P. 20 years of the SMART protein domain annotation resource[J]. Nucleic Acids Research, 2018, 46(D1): 493-496. doi: 10.1093/nar/gkx922.
[24] Blom N, Gammeltoft S, Brunak S. Sequence and structure-based prediction of eukaryotic protein phosphorylation sites[J]. J Mol Biol,1999,294(5):1351-1362.doi: 10.1006/jmbi.1999.3310.
[25] Borrill P, Ramirez-Gonzalez R, Uauy C. expVIP: a customisable RNA-seq data analysis and visualisation platform[J]. Plant Physiology, 2016,170(4):2172-2186. doi: 10.1104/pp.15.01667.
[26] Talla S K, Panigrahy M, Kappara S, Nirosha P, Neelamraju S, Ramanan R. Cytokinin delays dark-induced senescence in rice by maintaining the chlorophyll cycle and photosynthetic complexes[J]. J Exp Bot, 2016, 67(6):1839-1851. doi: 10.1093/jxb/erv575.
[27] Raines T, Shanks C, Cheng C Y, McPherson D, Argueso C T, Kim H J, Franco-Zorrilla J M, López-Vidriero I, Solano R, ![]() R, Schaller G E, Kieber J J. The cytokinin response factors modulate root and shoot growth and promote leaf senescence in Arabidopsis[J]. Plant J, 2016, 85(1):134-147. doi: 10.1111/tpj.13097.
R, Schaller G E, Kieber J J. The cytokinin response factors modulate root and shoot growth and promote leaf senescence in Arabidopsis[J]. Plant J, 2016, 85(1):134-147. doi: 10.1111/tpj.13097.
[28] Sakakibara H. Cytokinin biosynthesis and regulation[J]. Vitam Horm, 2005, 72:271-287. doi: 10.1016/S0083-6729(05)72008-2.
[29] Albacete A. Quantification of cytokinin levels and responses in abiotic stresses[M]//Dandekar T, Naseem M. Auxins and cytokinins in plant biology. New York: Humana Press, 2017:101-111. doi: 10.1007/978-1-4939-6831-2_8.
[30] Verslues P E. ABA and cytokinins: challenge and opportunity for plant stress research[J]. Plant Mol Biol, 2016, 91(6):629-640. doi: 10.1007/s11103-016-0458-7.
[31] Ha S, Vankova R, Yamaguchi-Shinozaki K, Shinozaki K, Tran L S. Cytokinins: metabolism and function in plant adaptation to environmental stresses[J]. Trends Plant Sci, 2012, 17(3): 172-179. doi: 10.1016/j.tplants.2011.12.005.
[32] Ding W, Tong H S, Zheng W J, Ye J, Pan Z C, Zhang B T, Zhu S H. Isolation, characterization and transcriptome analysis of a cytokinin receptor mutant Osckt1 in rice[J]. Front Plant Sci, 2017, 8:88. doi: 10.3389/fpls.2017.00088.