茄子是我国重要的蔬菜作物之一,近年来茄子播种面积不断加大,2016年我国茄子播种面积约93.33万hm2,位居蔬菜播种面积的第8位。对产量高、品质优的品种需求进一步增加,而在茄子育种改良中,萼片光滑无刺是一个重要的品质性状,因此,对茄子萼片刺性状形成机理进行研究具有重要的意义。
植物的刺是植物表皮上的表皮毛,表皮毛(Trichomes)是许多植物表面着生的附属物,由表皮细胞发育形成。表皮毛分为单细胞或多细胞,有腺体或无腺体。植物表皮毛具有一系列重要的功能:一般有腺体的表皮毛可以分泌一些次生代谢物质来抵抗病虫害,同时也能吸引动物或堆积盐分;无腺体的表皮毛一般具有散热、增强对逆境的抗性的作用[1]。关于植物表皮毛的研究主要集中在模式植物拟南芥上,目前,拟南芥表皮毛的形成已经成为研究植物细胞形态建成的模式系统。拟南芥的表皮细胞的发育涉及一个正调控和一个负调控,其中正调控是由3个转录因子(GL1、TTG1、GL3)组成的复合物来完成的,负调控是由一个抑制因子来完成的[2-3]。在蔬菜作物中,对黄瓜的刺瘤等表皮毛研究较为深入,与黄瓜果皮刺瘤相关的基因共有10个,主要包括果实黑刺基因(B)、无刺基因(gl)、多刺基因(ns)、果刺频率基因(s)和小刺基因(ss)[4]。茄科植物番茄的表皮毛的形成机理较为复杂,目前控制番茄腺毛的Wo基因已经被克隆出来,该基因编码的转录因子,通过调控B型细胞周期蛋白基因SlCycB2的表达来控制番茄型Ⅰ腺毛的形成[5]。hl基因导致番茄所有类型的腺毛扭曲,od-2基因导致番茄Ⅰ型和IV型腺毛呈圆柱状,腺体消失,VI型腺毛密度降低,腺体变小[6-7]。
利用转录组测序分析可以通过所有转录本的变化来揭示植物性状、抗性等表型变化,是分析、定位植物表型、抗性等重要农艺性状相关基因的重要手段之一。Guo等[8]通过比较栽培型和野生型西瓜的转录组差异,鉴定出了西瓜糖代谢、类胡萝卜素合成及代谢、果肉品质变化及乙烯合成等相关关键基因。Zhang等[9]通过比较抗根肿病甘蓝野生种及感病青花菜的转录组差异,鉴定出了调控根肿病抗性的相关基因及代谢途径。Wang等[10]通过比较耐热和耐高温不结球白菜的转录组差异,鉴定出了白菜耐热相关表达基因。在茄子遗传育种中,转录组应用也较为广泛,魏明明等[11]利用茄子转录组数据开发大量的可以应用的SSR分子标记。李笑[12]通过分析野生茄水茄接种黄萎菌后的转录组差异,筛选出一个与茄子黄萎病相关的WRKY转录因子基因StWRKY-1。王世界[13]通过分析紫色茄皮和白色茄皮的转录组差异,筛选到与茄子花青素生物合成相关的MYB转录因子3个和结构基因8个。
本研究采用高通量测序方法,在转录组水平上对茄子萼片有刺和无刺性状的差异基因表达量进行了分析。通过GO和KEGG富集和功能注释,期望筛选到影响茄子萼片刺的有无的转录因子,进而明确茄子萼片刺有无的分子机制,为茄子萼片无刺品种的选育奠定基础。
1 材料和方法
1.1 试验材料
试验材料均由北京市农林科学院蔬菜研究中心茄子课题组提供。自交系1607(紫黑长茄、绿萼、萼片无刺)和1608(紫黑长茄、绿萼、萼片有刺)均为纯合的高代自交系。2015年冬在海南三亚以1607为母本,1608为父本获得杂交组合,2016年春构建F2群体,同年9月定植于北京海淀区四季青试验温室。
1.2 田间表型调查及取样
调查每株茄子门茄刺的有无,有刺记为1,无刺记为0,为保证试验数据的准确性,分别在门茄蕾期、花期和果期进行调查。选双亲,30株萼片有刺和30株萼片无刺植株,分别取植株上成熟茄子的萼片,提取RNA,分别采用Nanodrop、Qubit 2.0、Aglient 2100方法检测RNA样品的纯度、浓度和完整性等,不合格样品重新取样提取,取等量RNA分别构建有刺和无刺RNA混池。
1.3 转录组测序
样品检测合格后,进行文库构建,分别使用Qubit 2.0和Agilent 2100对文库的浓度和插入片段大小(Insert Size)进行检测,使用Q-PCR 方法对文库的有效浓度进行准确定量,以保证文库质量。用HiSeqX Series进行高通量测序,测序读长为PE150。
1.4 转录组数据分析
1.4.1 数据处理 对测序获得的Raw Data进行数据过滤,去除其中的接头序列及低质量Reads,获得高质量的Clean Data。获得Clean Reads后,将其与参考基因组(Solanum melongena L.,ftp://ftp.solgenomics.net/genomes/Solanum_melongena/)进行序列比对,获取在参考基因组或基因上的位置信息,以及测序样品特有的序列特征信息。
1.4.2 基因表达量分析 使用Cufflinks软件计算每个基因的FPKM(Fragments Per Kilobase of transcript per Million fragments mapped)[14]。利用EBSeq[15]进行差异表达分析,获得2个样品之间的差异表达基因集。在差异表达基因检测过程中,以Fold Change≥2且FDR<0.01作为筛选标准。
1.4.3 差异基因Go分类和KEGG分析 利用Blast 2 Go程序获得茄子每个基因的Go注释。使用BlastP将差异基因序列比对到Swiss-Prot和TrEMBL数据库,从而得到差异基因的Go功能注释信息,利用WEGO在线软件进行Go分类和Go图的制作[16]。使用BBH(bi-directional best hit)对差异基因的KEGG(Kyoto Encyclopedia of Genes and Genomes)代谢途径进行注释。
2 结果与分析
2.1 RNA-Seq测序数据统计分析
基于边合成边测序(Sequencing By Synthesis, SBS)技术,利用IlluminaHiSeq4000高通量测序平台对双亲、混池的4个cDNA文库进行测序,共获得60.77 Gb的Clean Data,各样品Q30碱基百分比在88.9%以上(表1)。经过原始数据处理,4个文库共获得409 363 358个Clean Reads,利用Bowtie 2软件将Clean reads与参考基因组进行比对,共有311 756 616个比对到基因组,比对效率均大于75.00%。有刺混池和无刺混池分别有104 202 238个和109 191 303个Clean reads比对到参考基因组,其中比对到唯一的位置的Uniq Mapped Reads分别为72.93%和74.14%。以上比对结果表明,从建库到测序等均达到试验要求。
对样品的测序随机性进行了评估,分析测序数据在参考基因上的随机分布情况。萼片有刺混池和无刺混池的随机性图呈均匀分布(图1),表明测序reads在参考基因上的分布相对较为均匀,测序随机性较好。
表1 RNA-Seq数据的比对统计
Tab.1 Summary of RNA-Seq data
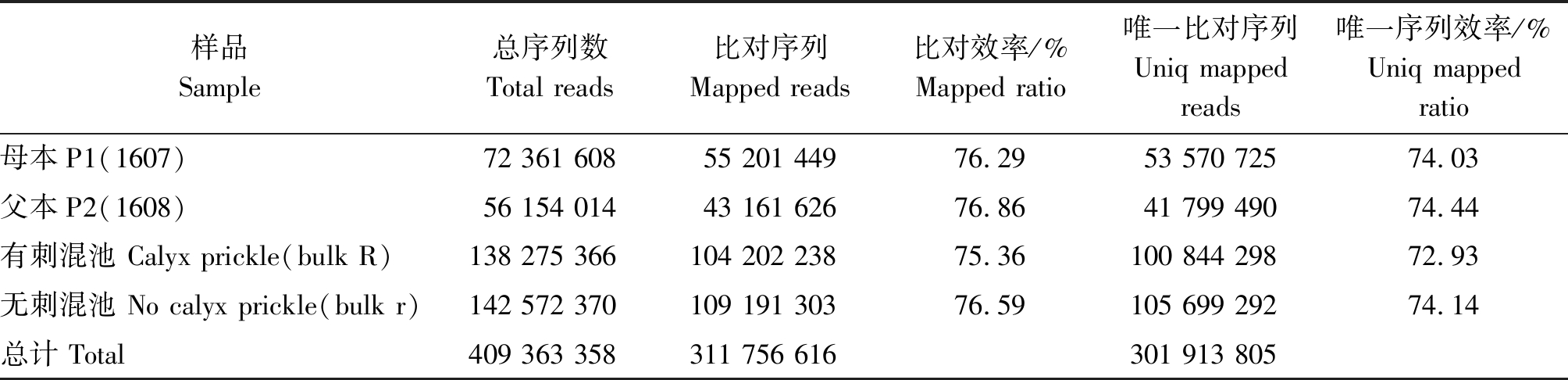
样品Sample总序列数Total reads比对序列Mapped reads比对效率/%Mapped ratio唯一比对序列Uniq mappedreads唯一序列效率/%Uniq mappedratio母本P1(1607)72 361 60855 201 44976.2953 570 72574.03父本P2(1608)56 154 01443 161 62676.8641 799 49074.44有刺混池 Calyx prickle(bulk R)138 275 366104 202 23875.36100 844 29872.93无刺混池 No calyx prickle(bulk r)142 572 370109 191 30376.59105 699 29274.14总计 Total409 363 358311 756 616301 913 805
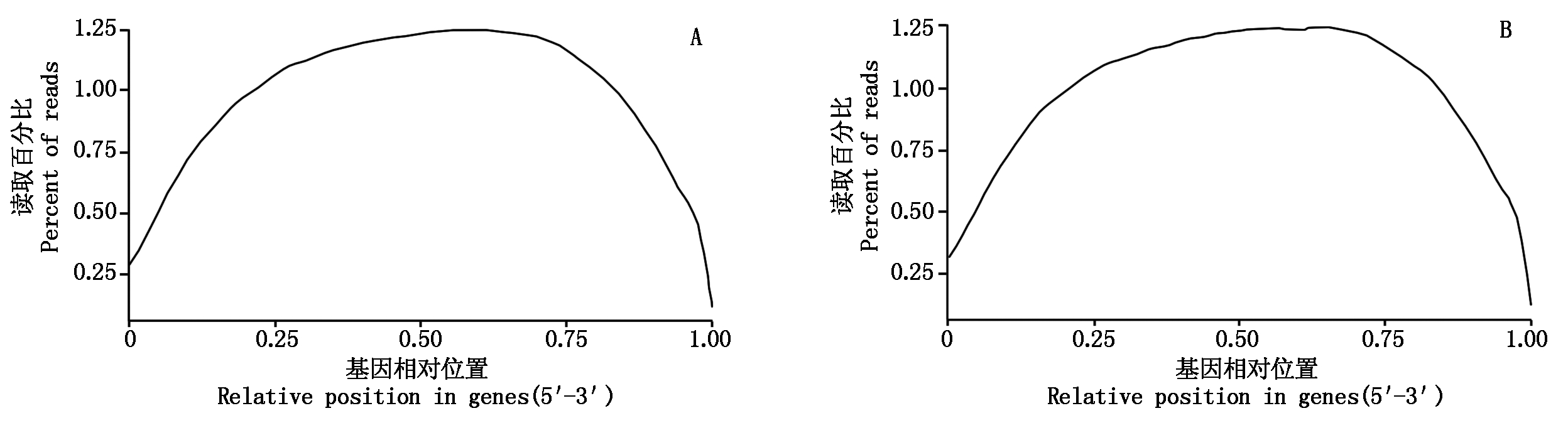
A.有刺混池; B.无刺混池。
A.Bulk R; B.Bulk r.
图1 测序随机性评估
Fig.1 Sequencing randomization assessment
2.2 新基因的发掘及功能注释
基于茄子参考基因组的序列,使用Cufflinks软件对Mapped Reads进行拼接,并与原基因组注释信息进行比较,找出未被注释的转录区域,获得新的转录本和基因,并对其进行注释。过滤掉少于50个氨基酸残基和只包含单个外显子的序列,共发掘24 310个新基因。
对获得的全部新基因与NR、Swiss-Prot、GO、COG和KEGG数据库进行比对,获得新基因的注释信息(表2)。获得注释信息的基因总数为1 540个,占新基因总数的6.33%,其中注释到GO和KEGG数据库的新基因数分别为771,550个。
表2 新基因功能注释结果统计
Tab.2 Summary of new gene function annotation
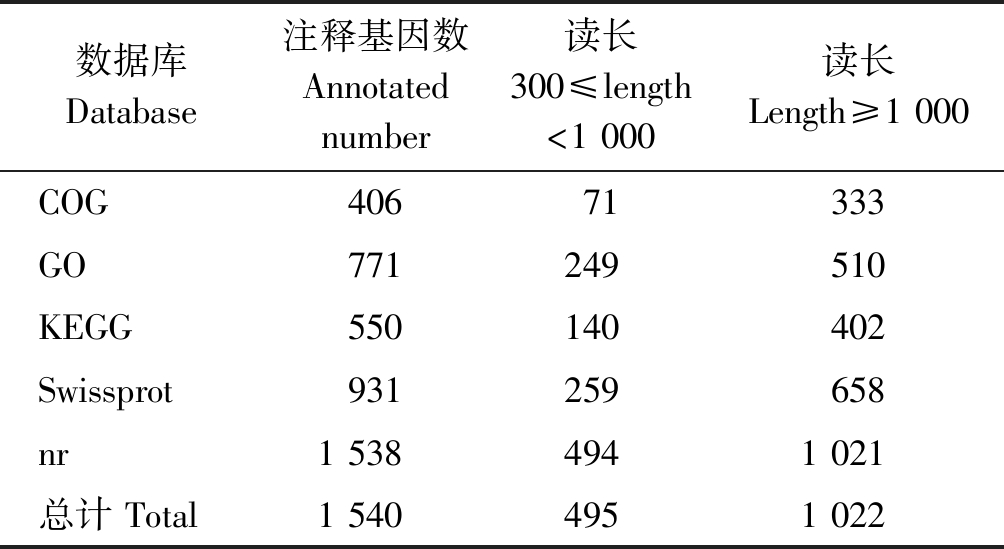
数据库Database注释基因数Annotated number读长300≤length<1 000读长Length≥1 000COG 40671333GO771249510KEGG550140402Swissprot931259658nr1 5384941 021总计 Total1 5404951 022
2.3 差异表达基因统计及Go和KEGG分析
2.3.1 差异表达基因统计 双亲和2个混池通过差异基因表达分析,得到2 574个差异表达基因,其中共同含有的差异基因97个(图2)。双亲间共有2 438个差异表达基因,其中839个基因在父本(萼片有刺)中的表达量显著高于母本(萼片无刺),1 599个基因在父本(萼片有刺)中的表达量显著低于母本(萼片无刺)。有刺和无刺混池间共有233个差异表达基因,其中127个基因在萼片有刺混池中的表达量显著高于无刺混池,106个基因在萼片有刺混池中的表达量显著低于无刺混池。
2.3.2 差异表达基因Go分析 对双亲和有刺无刺2个混池的差异表达基因进行了Go显著性富集分析,明确其在各Go项中的分布情况(图3)。Go注释分析显示在双亲和混池间分别有1 237,121个差异基因具有Go注释信息。双亲和混池差异基因在分子功能和生物过程中富集的Go项是一致的,均为在分子功能中催化活性和结合亚类呈极显著富集,生物过程中的代谢过程、细胞过程和单一生物过程等3个亚类呈极显著富集。双亲和混池在所处的细胞位置的富集有所差异,其中双亲分布最多的3个亚类分别为细胞、膜和细胞组分,混池分布最多的3个亚类分别为细胞、细胞器和细胞组分。
同时对双亲和有刺无刺2个混池的共有的97个差异表达基因进行Go功能注释,共计有50个基因具有Go注释信息。其中在分子功能中催化活性和结合2个亚类极显著富集,在生物过程中代谢过程,细胞过程和单一生物过程,生物调节和对刺激的反应等5个亚类呈极显著富集,细胞过程中细胞,细胞器和细胞组分极显著富集。
在茄子双亲和混池的共有差异基因的Go富集分析结果表明,富集基因最多的为生物过程中的代谢过程,共包含35个基因,表明茄子萼片刺基因的有无可能与代谢物质的合成与调节有关。
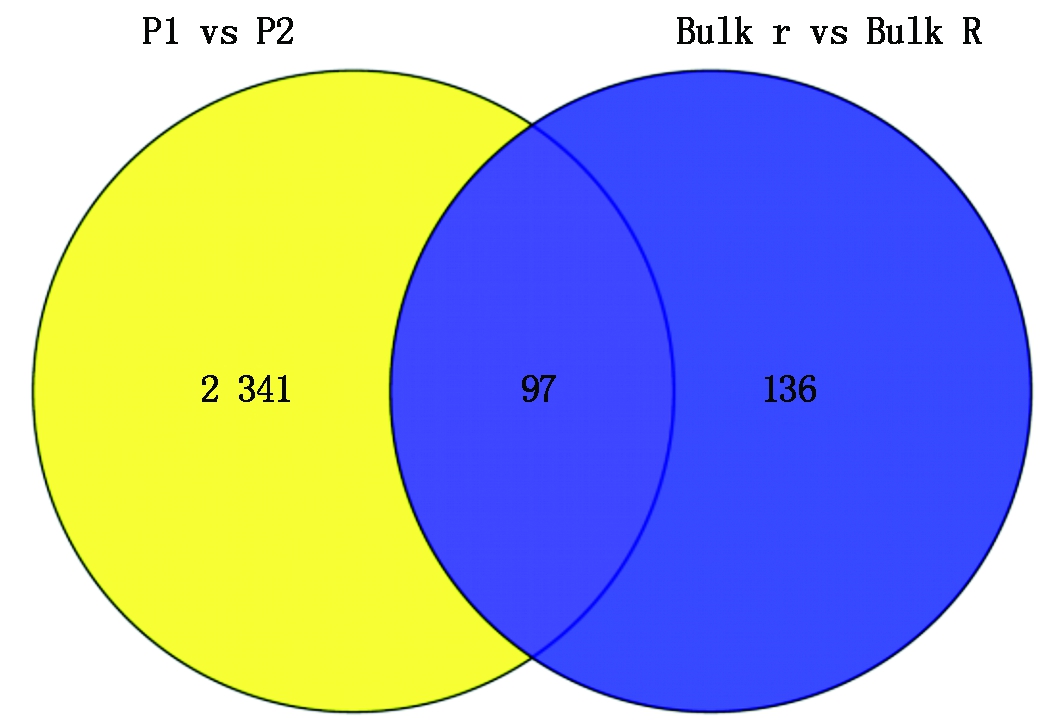
图2 差异表达基因数目统计
Fig.2 Summary of the differentially expressed genes
拟南芥中调控表皮毛的重要调控基因TTG1和GL3等都属于生物过程中的重要基因,且在茄子双亲和混池的共有差异基因中富集基因最多的为生物过程。在生物过程中通过Go功能注释筛选出茄子萼片刺合成相关的代谢过程:细胞分裂素分解代谢过程、细胞分裂素代谢过程、对生长素的反应、代谢过程、细胞过程等共筛选了7个基因(表3)。这7个代谢相关基因中有2个基因(Eggplant_newGene_1802、Sme2.5_09750.1_g00002)在双亲及混池间无刺相对于有刺是共同上调表达的,可能是参与了茄子萼片刺合成的关键基因。
2.3.3 差异表达基因KEGG分析 通过KEGG分析,分别对双亲和混池间的差异基因主要富集的代谢通路进行了分析(图4)。双亲中差异表达基因主要涉及的特异代谢通路为植物激素信号转导、植物-病原体相互作用和苯丙烷类生物合成。两混池中差异表达基因主要涉及的特异代谢通路为角质、小檗碱和蜡生物合成、淀粉和蔗糖代谢和戊糖和葡萄糖醛酸。对双亲和有刺无刺2个混池共有的97个差异表达基因进行KEGG分析,共计有17个基因涉及26个代谢通路,代谢通路较为分散,没有呈现显著富集。
在KEGG富集分析中角质、小檗碱和蜡生物合成代谢通路是形成植物角质、蜡质等表皮部分重要的途径。茄子萼片刺为茄子表皮毛细胞的变异,为茄子表皮的重要组成部分,因此进一步对角质、小檗碱和蜡生物合成代谢通路进行了统计分析(表4)。只有1个新基因Eggplant_newGene_4241在双亲和两混池之间同时被富集到,并且无刺相对于有刺该基因显著上调。有2个基因(Sme2.5_00096.1_g00020、Sme2.5_02057.1_g00007)仅在双亲间被富集到,有5个基因仅在两混池间被富集到,但无刺相对于有刺这7个基因都显著下调。
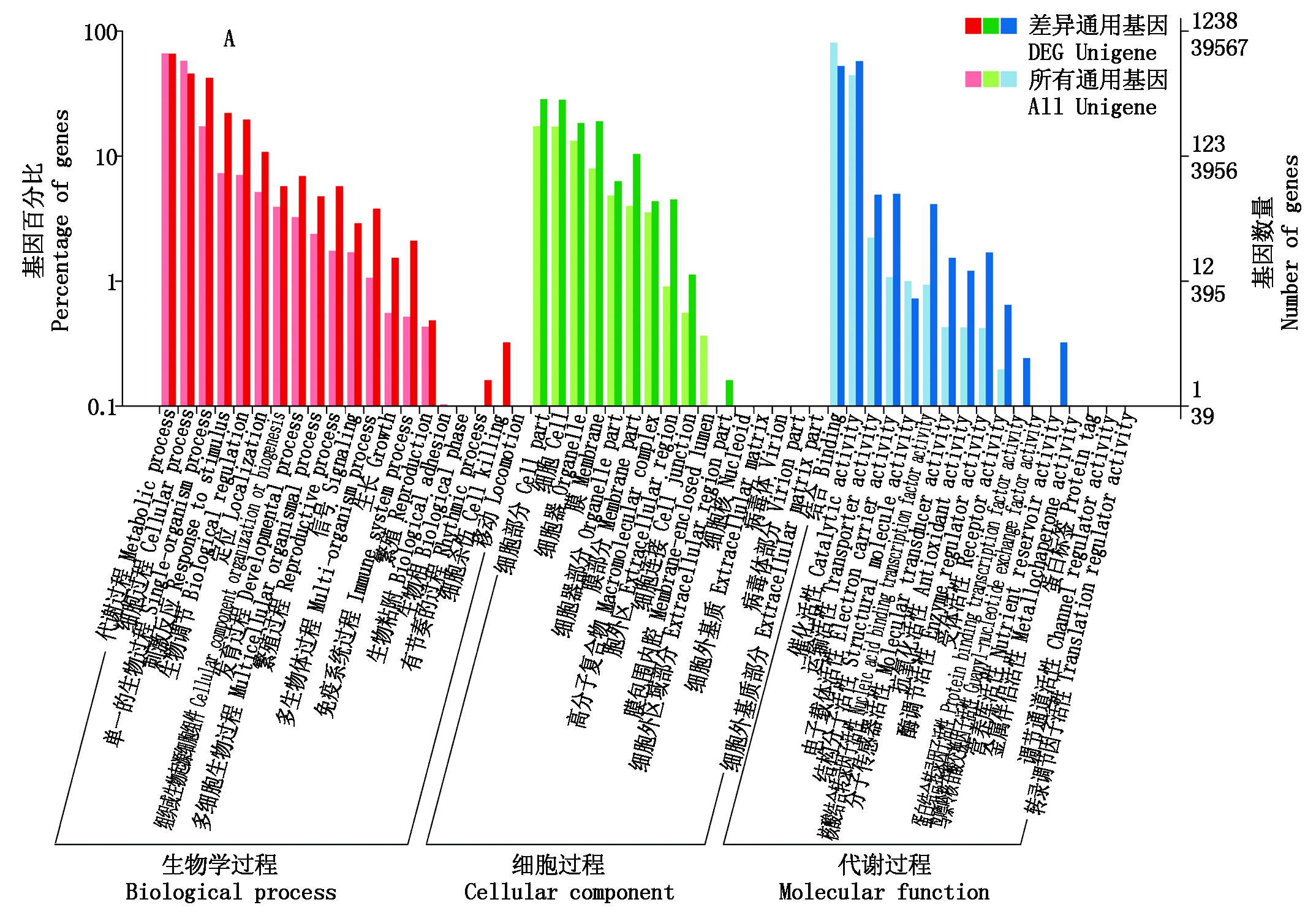

A,B图分别为双亲和两混池差异基因。
A. The GO categories of the parents; B. The GO categories of the mixed pools.
图3 差异表达基因的Go分类
Fig.3 Distribution of the differentially expressed genes(DEGS)with GO categories
表3 双亲及混池在生物过程中的差异表达基因分析
Tab.3 Differentially expressed genes in the the parents and the mixed pools

基因名称Genes name功能Function表达量(log2Fold-change value)P1 vs P2Bulk R vs Bulk rEggplant_newGene_1802细胞分裂素分解代谢过程1.551.42Sme2.5_00411.1_g00011细胞分裂素分解代谢过程-1.451.08Sme2.5_00083.1_g00005对生长素反应过程-7.291.62Sme2.5_33421.1_g00001代谢过程3.84-2.43Sme2.5_09750.1_g00002代谢过程1.221.37Sme2.5_00172.1_g00023代谢过程4.87-2.13Sme2.5_02682.1_g00003细胞过程6.35-2.87

A、B图分别为双亲和两混池中差异表达基因的KEGG分类图。
A.The KEGG pathway of the parents; B.The KEGG pathway of the mixed pools.
图4 差异表达基因的KEGG分类图
Fig.4 Distribution of the differentially expressed genes(DEGS)with KEGG pathway
表4 双亲及混池在“角质,小檗碱和蜡生物合成”KEGG途径中的差异表达基因
Tab.4 Differentially expressed genes in the KEGG pathway "Cutin, suberine and wax biosynthesis"

基因名称Genes功能Function表达量(log2Fold-change value)P1 vs P2Bulk R vs Bulk rEggplant_newGene_4241脂肪酰基辅酶A还原酶1.641.11Sme2.5_00096.1_g00020脂肪酸ω-羟化酶-4.71无Sme2.5_02057.1_g00007脂肪酸ω-羟化酶-1.78无Sme2.5_00021.1_g00010长链脂肪酸ω-单加氧酶无-2.46Sme2.5_02174.1_g00001脂肪酸ω-羟化酶无-2.46Sme2.5_02440.1_g00001ω-羟基棕榈酸O-阿魏酰转移酶无-2.36Sme2.5_03548.1_g00005脂肪酰基辅酶A还原酶无-1.88Sme2.5_11159.1_g00002醛脱羰酶无-3.34
2.4 相关候选基因Blast比对分析
将双亲和2个混池的差异基因通过Go和KEGG分析富集筛选的3个相关基因(Eggplant_newGene_1802、Sme2.5_09750.1_g00002、Eggplant_newGene_4241)和拟南芥、番茄的基因组进行同源Blast比对分析(表5)。和番茄基因组(Tomato Genome CDS(ITAG release 3.20))比对,Eggplant_newGene_4241和Sme2.5_09750.1_g00002在番茄的同源基因分别为Solyc11g067180.2和Solyc01g079300.3,它们在番茄中分别负责编码脂肪酰基辅酶A还原酶和水苏糖合成酶的合成,Eggplant_newGene_1802比对到2个同源基因,为Solyc04g080820.2和Solyc04g080810.3,它们在番茄中分别负责编码细胞分裂素氧化酶和细胞分裂素氧化酶/类脱氢酶的合成。和拟南芥基因组比对,Eggplant_newGene_4241未能找到与拟南芥同源基因,Eggplant_newGene_1802在拟南芥的同源基因为AT1G75450.1(AtCKX6),它编码催化类细胞分裂素降解的酶(细胞分裂素氧化酶/脱氢酶),Sme2.5_09750.1_g00002在拟南芥的同源基因为AT4G01970.1(AtRS4),它编码棉子糖和高亲和力的水苏糖合成酶以及水苏糖和Gol特异性半乳糖水解酶(Sta和Gol特异性半乳糖水解酶)。
表5 相关基因同源比对分析
Tab.5 The orthologous of the related genes

基因Gene ID番茄的同源比对 Orthologous gene in tomato拟南芥的同源比对 Orthologous gene in ArabidopsisID功能FunctionE值E-value(BlastX)ID功能FunctionE值E-value(BlastN)Eggplant_newGene_4241Solyc11g067180.2脂肪酰辅酶A还原酶9.00E-73无Eggplant_newGene_1802Solyc04g080820.2细胞分裂素氧化酶0AT1G75450.1类细胞分裂素氧化/脱氢酶4.00E-26Solyc04g080810.3类细胞分裂素氧化/脱氢酶 0Sme2.5_09750.1_g00002Solyc01g079300.3水苏糖合成酶0AT4G01970.1水苏糖合成酶/Gol特异性糖基水解酶1.00E-15
3 讨论与结论
茄子萼片是茄子果实的重要组成部分,育种工作中比较关心的萼片相关性主要包括萼片的颜色、大小、刺[17]。茄子萼片刺是重要的品质性状,萼片刺的有无、数量、颜色和大小都是影响茄子商品性的重要因素。本研究以茄萼片刺的有无为研究目标,利用转录组分析了影响萼片刺形成的调控因子。
3.1 茄子萼片刺与植物细胞分裂素分解代谢
细胞分裂素(Cytokinin, CK)是在植物体内存在的重要植物激素,它主要是促进细胞分裂,对调节根、芽的分化,维管束的形成等植物生长发育具有重要调节作用[18]。CK的合成和代谢是一个综合调控的过程,它的合成主要是异戊烯基转移酶(IPT)催化,天然异戊烯类CK及其核苷主要是以细胞分裂素氧化酶/脱氢酶(Cytokinin oxidase/dehydrogenase, CKX, EC: 1.5.99.12)来催化发生不可逆降解[19]。CKX在植物体内普遍存在,最早是由paces在烟草匀浆细胞中发现的,之后相继在拟南芥、水稻、大麦和小麦中被发现[20-22]。大多数植物含有多个CKX基因,拟南芥含有7个CKX基因(AtCKX1~AtCKX7),其中AtCKX7与其他CKX基因差异较大[23]。水稻至少含有11个CKX基因(OsCKX1~OsCKX11)[24],玉米至少含有13个CKX基因(ZmCKX1~ZmCKX13)[25]。
通过对差异基因的GO注释分析,在双亲及混池中富集到了一个与茄子的细胞分裂素氧化酶/脱氢酶相关的基因:Eggplant_newGene_1802(SmCKX),这个基因为茄子转录组获得的新基因,该基因与番茄的Solyc04g080820.2和Solyc04g080810.3同源性较高,和拟南芥中的AtCKX6具有同源性。在拟南芥中,AtCKX6能够显著增强CKX活性,降低植物细胞分裂素浓度并引起表型的变化[23]。参考拟南芥同源基因,以上结果表明,SmCKX可能是茄子中存在的CKX基因,该基因可能通过调控细胞分裂素的变化来控制茄子表型的变化。茄子萼片有刺该基因的表达量明显减少,说明SmCKX对茄子萼片的刺可能是一个负调控的过程,茄子萼片有刺的植株体内细胞分裂素可能受到抑制。
3.2 茄子萼片刺与糖代谢过程
水苏糖是在植物中普遍积累的一种棉子糖系列寡糖(Raffinose family oligosaccharieds, RFOs),RFOs在植物中的分布仅次于蔗糖,是植物韧皮部运输的重要物质,对植物在逆境中的抗性、种子耐脱水性等具有重要的作用[26-27]。水苏糖合成是很多酶参与的一个过程,水苏糖合成酶(Stachyose synthase,STS, EC 2.4.1.67)是水苏糖合成中最后一步反应的关键酶[28]。研究者先后从赤豆、豌豆、心叶假面花中克隆出了水苏糖合成酶相关基因,并研究了其在植物中的调节的相关生物学过程[29-30]。
参与茄子水苏糖合成的相关基因及其调控过程是未知的。通过萼片有刺和无刺的双亲及混池差异基因的GO分析,富集到了一个与水苏糖合成酶相关的基因: Sme2.5_09750.1_g00002(SmSTS),该基因属于糖苷水解酶家族(Glycoside hydrolase, super family),与番茄的Solyc01g079300.3基因和拟南芥的AT4G01970.1基因同源性较高。萼片无刺相对于有刺该基因显著上调。表明茄子萼片刺的形成与茄子植物内水苏糖的合成有一定的关联,茄子萼片刺的形成,茄子萼片上的水苏糖的合成可能会受到抑制,是一个负调控的过程。植物表皮刺的形成与植物的抗虫性等防御机制有一定的关系,这和水苏糖调节植物抗性有一定的类似作用。试验结果证明了2个途径在茄子中可能存在关联,但是如何通过茄子水苏糖合成酶(SmRS)来调节两者之间的关联表达还需要进一步研究。
3.3 茄子萼片刺与植物角质,小檗碱和蜡生物合成途径
陆地高等植物大部分的表皮细胞是由角质膜覆盖的,它从内到外分成角化层、角质层和蜡质层3层,其中角质层是角化膜最重要的部分,占整个角质膜的50%以上,角质层是植物适应外界环境的重要物质[31-32]。根据疏水层是否溶解于氯仿或正己烷等有机溶剂,可将植物角质层分成角质层蜡质和角质[33]。植物表皮的蜡质直接与外界环境接触,因此,其对植物蒸腾作用的调节、抗病、抗虫具有重要的意义[34-36]。表皮蜡质的生物合成最先始于脂肪酸的从头合成,它是细胞质中的C16和C18的生物合成[37]。C16/C18再经过酶的催化延长,通过酰基还原和脱羰基2个途径合成蜡质[38]。这2种途径中脂肪酰辅酶A还原酶(Fatty acyl-CoA reductase, FAR)都是催化形成脂肪醛的关键还原酶[39]。植物中的脂肪酞辅酶A还原酶大约由490~500个氨基酸构成,在拟南芥、水稻和小麦中均被克隆出来,其中拟南芥中含有3个类型的8个FAR基因[40]。
通过对差异基因的KEGG注释分析,在茄子萼片有刺和无刺的双亲及混池中均富集到了一个脂肪酰辅酶A还原酶相关基因:Eggplant_newGene_4241(SmFAR),该基因是一个新基因,与同属茄科的番茄Solyc11g067180.2基因(FARx相关基因)同源性较高,但是在拟南芥中没有找到相关同源基因。在番茄中,FARx相关基因的增加能够调控番茄表皮蜡质晶体的合成,增加番茄的保水能力。参考番茄同源基因,茄子萼片刺的合成可能与表皮的蜡质合成具有相同的合成代谢通路,需要通过脂肪酰辅酶A还原酶来调控,萼片有刺茄子可能通过该酶的下调,减少合成表面蜡质来合成茄子萼片等部位的刺。
[1] Werker E. Trichome diversity and development[J]. Advances in Botanical Research, 2000,31:1-35. doi: 10.1016/S0065-2296(00)31005-9.
[2] Payne C T, Zhang F, Lloyd A M. GL3 Encodes a bHLH protein that regulates trichome development in Arabidopsis through interaction with GL1 and TTG1[J]. Genetics, 2000, 156(3): 1349-1362.
[3] Larkin J C, Brown M L, Schiefelbein J. How do cells know what they want to be when they grow up? Lessons from epidermal patterning in Arabidopsis[J]. Annual Reviews of Plant Biology, 2003, 54: 403-430. doi: 10.1146/annurev.arplant.54.031902.134823.
[4] Pierce L K, Wehner T C. Review of genes and linkage groups in cucumber[J]. Hort Science, 1990, 25(6): 605-615.
[5] Yang C X, Li H X, Zhang J H, Luo Z D, Gong P J, Zhang C J, Li J H, Wang T T, Zhang Y Y, Lu Y E, Ye Z B.A regulatory gene induces trichome formation and embryo lethality in tomato[J]. Proceedings of the National Academy of Sciences, 2011, 108(29):11836-11841. doi: 10.1073/pnas.1100532108.
[6] Kang J H, Liu G G, Shi F, Jones A D, Beaudry R M, Howe G A. The tomato odorless-2 mutant is defective in trichome-based production of diverse specialized metabolites and broad-spectrum resistance to insect herbivores[J]. Plant Physiology, 2010, 154(1): 262-272. doi: 10.1104/pp.110.160192.
[7] Kang J H, Shi F, Jones A D, Marks M D, Howe G A. Distortion of trichome morphology by the hairless mutation of tomato affects leaf surface chemistry[J]. Journal of Experimental Botany 2010, 61(4): 1053-1064. doi: 10.1093/jxb/erp370.
[8] Guo S G, Sun H H, Zhang H Y, Liu J G, Ren Y, Gong G Y, Jiao C, Zheng Y, Yang W C, Fei Z J, Xu Y. Comparative transcriptome analysis of cultivated and wild watermelon during fruit development[J]. PLoS One, 2015, 10(6): e0130267. doi: 10.1371/journal.pone.0130267.
[9] Zhang H, Feng J, Hwang S F, Strelkov S E, Falak I, Huang X, Sun R. Mapping of clubroot(Plasmodiophora brassicae)resistance in canola(Brassica napes)[J]. Plant Pathology, 2016, 65(3):435-440. doi: 10.1111/ppa.12422.
[10] Wang A H, Hu J H, Huang X X, Li X, Zhou G L, Yan Z X. Comparative transcriptome analysis reveals Heat-Responsive genes in Chinese cabbage(Brassica rapa ssp. chinensis)[J]. Frontiers in Plant Science, 2016,7: 939. doi: 10.3389/fpls.2016.00939.
[11] 魏明明,陈钰辉,刘富中,张映,连勇. 基于转录组测序的茄子SSR标记开发[J]. 植物遗传资源学报,2016,17(6):1082-1091.doi: 10.13430/j.cnki.jpgr.2016.06.017.
Wei M M, Chen Y H, Liu F Z, Zhang Y, Lian Y. Evelopment of SSR markers for eggplant with transcriptome [J]. Sequencing Data Journal of Plant Genetic Resources, 2016,17(6):1082-1091.
[12] 李笑. 水茄抗黄萎病相关StWRKY-1基因的克隆与功能分析[D]. 扬州:扬州大学,2018.
Li X. Cloning and functional analysis of the StWRKY-1 gene from Solanum torvum[D]. Yangzhou: Yangzhou University, 2018.
[13] 王世界.茄子花青素生物合成关键MYB转录因子的筛选及克隆[D].泰安:山东农业大学,2016.
Wang S J. Screening and cloniing of key MYB transcription factor of athncyanin biosynthesis in eggplant(Solanum melongena L.)[D]. Taian: Shandong Agricultural University, 2016.
[14] Trapnell C, Williams B A, Pertea G, Mortazavi A, Kwan G, van Baren M J, Salzberg S L, Wold B J, Pachter L. Transcript assembly and quantification by RNA Seq reveals unannotated transcripts and isoform switching during cell differentiation[J]. Nature Biotechnology, 2010, 28(5):511-515. doi: 10.1038/nbt.1621.
[15] Leng N, Dawson J A, Thomson J A, Ruotti V, Rissman A I, Smits B M G, Haag J D, Gould M N, Stewart R M, Kendziorski C. EBSeq: An empirical Bayes hierarchical model for inference in RNA-seq experiments[J]. Bioinformatics, 2013, 29(8): 1035-1043. doi: 10.1093/bioinformatics/btt087.
[16] Ye J, Fang L, Zheng H K, Zhang Y, Chen J, Zhang Z J, Wang J, Li S T, Li R Q, Bolund L, Wang J.WEGO: A web tool for plotting GO annotations[J]. Nucleic Acids Research,2006,34(1):293-297. doi: 10.1093/nar/gkl031.
[17] 李锡香,朱德蔚. 茄子种质资源描述规范和数据标准[M]. 北京: 中国农业出版社,2006:5-6.
Li X X, Zhu W D. Description criterion and data standard for eggplant[M]. Beijing: China Agriculture Press, 2006: 5-6.
[18] Sakakibara H. Cytokinins: Activity, biosynthesis, and translocation[J]. Aanual Review of Plant Biology,2006,57:431-449. doi: 10.1146/annurev.arplant.57.032905.105231.
[19] Kowalska M, Galuszka P, Frébortová J,  ebela M, Bě-res T, Hluska T, Smehilova M, Bilyeu K D, Frěbort I. Vacuolar and cytosolic cytokinin dehydrogenases of Arabidopsis thaliana: heterologous expression, purification and properties[J]. Phytochemistry,2010,71(17-18):1970-1978. doi: 10.1016/j.phytochem.2010.08.013.
ebela M, Bě-res T, Hluska T, Smehilova M, Bilyeu K D, Frěbort I. Vacuolar and cytosolic cytokinin dehydrogenases of Arabidopsis thaliana: heterologous expression, purification and properties[J]. Phytochemistry,2010,71(17-18):1970-1978. doi: 10.1016/j.phytochem.2010.08.013.
[20] Werner T, Motyka V, Laucou V, Smets R, Van Onckelen H, Schmülling T. Cytokinin-deficieat transgenic Arabidopsis plants show multiple developmental alterations indicating opposite functions of cytokinias in the regulation of shoot and root meristem activity[J]. Plant Cell, 2003,15(11):2532-2550. doi: 10.1105/tpc.014928.
[21] Ashikari M, Sakakibara H, Lin S Y, Yamamoto T, Takashi T, Nishimura A, Angeles E R, Qian Q, Kitano H, Matsuoka M. Cytokinin oxidase regulates rice grain production[J].Science, 2005, 309(5735):741-745. doi: 10.1126/science.1113373.
[22] Galuszka P, Frébortová J, Werner T, Yamada M, Strnad M, Schmülling T, Frébort I. Cytokinin oxidase/dehydrogenase genes in barley and wheat: cloning and heterologous expression[J]. European Journal of Biochemistry, 2004, 271(20): 3990-4002. doi: 10.1111/j.1432-1033.2004.04334.x.
[23] Schmülling T, Werner T, Riefler M, Krupkov E, Bartrina y M I. Structure and function of cytokinin oxidase/dehydrogenase genes of maize, rice, Arabidopsis and other species[J]. Journal of Plant Research, 2003,116(3):241-252. doi: 10.1007/s10265-003-0096-4.
E, Bartrina y M I. Structure and function of cytokinin oxidase/dehydrogenase genes of maize, rice, Arabidopsis and other species[J]. Journal of Plant Research, 2003,116(3):241-252. doi: 10.1007/s10265-003-0096-4.
[24] Werner T, Köllmer I, Bartrina I, Holst K, Schmüllin T. New insights into the biology of cytokinin degradation[J]. Plant Biology, 2006,8(3):371-381. doi: 10.1055/s-2006-923928.
[25] Vyroubalová  ,Václavíková
,Václavíková ![]() á V,Novák O,
á V,Novák O, mehilová M,Hluska T, Ohnoutková L,Frébort I, Galuszka P. Characterization of new maize genes putatively involved in CK metabolism and their expression during osmotic stress in relation with cytokinin levels[J]. Plant Physiology, 2009, 151(1):433-447. doi: 10.1104/pp.109.142489.
mehilová M,Hluska T, Ohnoutková L,Frébort I, Galuszka P. Characterization of new maize genes putatively involved in CK metabolism and their expression during osmotic stress in relation with cytokinin levels[J]. Plant Physiology, 2009, 151(1):433-447. doi: 10.1104/pp.109.142489.
[26] Sengupta S, Mukherjee S, Basak P, Majumder A L. Significance of galactinol and raffinose family oligosaccharide synthesis in plants[J]. Frontiers in Plant Science,2015,6: 656. doi: 10.3389/fpls.2015.00656.
[27] Taji T, Ohsumi C, Iuchi S, Seki M, Kasuga M, Kobayashi M, Yamaguchi-Shinozaki K, Shinozaki K. Important roles of drought-and cold-inducible genes for galactinol synthase in stress tolerance in Arabidopsis thaliana[J]. Plant Journal, 2002, 29(4): 417-426. doi: 10.1046/j.0960-7412.2001.01227.x.
[28] Pharr D M, Huber S C, Sox H N. Leaf carbohydrate status and enzymes of translocate synthesis in fruiting and vegetative plant of Cucunais sativus L.[J]. Plant Physiolology, 1985,77(1):104-108. doi: 10.2307/4269086.
[29] Peterbauer T, Mach L, Mucha J, Richter A. Functional expression of a cDNA encoding pea(Pisum sativum L.)raffinose synthase, partial purification of the enzyme from seeds, and steady-state kinetic analysis of raffinose synthesis[J]. Planta, 2002, 215(5):839-846. doi: 10.1007/s00425-002-0804-7.
[30] Voitsekhovskaja O V, Rudashevskaya E L, Demchenko K N, Pakhomova M V, Batashev D R, Gamalei Y V, Lohaus G, Pawlowski K. Evidence for functional heterogeneity of sieve element-companion cell complexes in minor vein phloem of Alonsoa meridionalis[J]. Journal of Experimental Botany,2009,60(6):1873-1883. doi: 10.1093/jxb/erp074.
[31] Buschhaus C, Jetter R. Composition differences between epicuticular and intracuticular wax substructures: how do plants seal their epidermal surfaces? [J]. Journal of Experimental Botany,2010,62(3): 841-853. doi: 10.1093/jxb/erq366.
[32] Lee S B, Suh M C. Advances in the understanding of cuticular waxes in Arabidopsis thaliana and crop species[J]. Plant Cell Reports, 2015,34(4):557-572. doi: 10.1007/s00299-015-1772-2.
[33] Kunst L. New insights into biosynthesis of cuticular wax[J]. The FASEB Journal, 2015, 29(1):363-366.
[34] Mohammadian M A, Wading J R, Hill R S. The impact of epicuticular wax on gas-exchange and photoinhibition in Leucadendron lanigeruw(Proteaceae)[J]. Acta Oecologica, 2007,31(1):93-101. doi: 10.1016/j.actao.2006.10.005.
[35] Gniwotta F, Vogg G, Gartmann V, Carver T L, Riederer M, Jetter R. What do microbes encounter at the plant surface? Chemical composition of pea leaf cuticular waxes[J]. Plant Physiology, 2005,139(1):519-530. doi: 10.1104/pp.104.053579.
[36] Gaume L, Perret P, Gorb E, Gorb S, Labat J J, Rowe N. How do plant waxes cause flies to slide? Experimental tests of wax-based trapping mechanisms in three pitfall carnivorous plants[J]. Arthropod Structure & Development, 2004,33(1):103-111. doi: 10.1016/j.asd.2003.11.005.
[37] Cui W, Liang Y, Tian W X, Ji M J, Ma X F. Regulating effect of β-ketoacyl synthase domain of fatty acid synthase on fatty acyl chain length in de novo fatty acid synthesis[J]. Biochim Biophys Acta, 2016,1861(3):149-155. doi: 10.1016/j.bbalip.2015.12.002.
[38] Dusty P B. Biochemistry and molecular biology of wax production by plants[J]. Annual Review of Plant Physiology and Plant Molecular Biology, 1996,47: 405-430. doi: 10.1146/annurev.arplant.47.1.405.
[39] Kolattukudy P E. Enzymatic synthesis of fatty alcohols in Brassica oleracea[J]. Archives of Biochemistry & Biophysics,1971, 142(2): 701-709. doi: 10.1016/0003-9861(71)90536-4.
[40] Kunst L, Samuels A L. Biosynthesis and secretion of plant cuticular wax[J]. Progress in Lipid Research, 2003, 42(1): 51-80. doi: 10.1016/S0163-7827(02)00045-0.