草莓(Fragaria ananassa Duch.)因味道鲜美,富含大量的维生素而广受欢迎。近年来我国草莓种植面积不断增长,栽植草莓具有可观的经济效益,然而草莓畸形果的形成直接影响草莓商品价值。果实发育受到生长素(Auxin)、脱落酸(ABA)、乙烯(Ethylene)、赤霉素(GA)等多种植物激素的共同调控,草莓作为呼吸非跃变型果实的典型代表,其发育及成熟过程主要受到生长素和脱落酸的调控[1],激素之间的互作调控过程直接影响果实的品质,激素调控的失衡是形成草莓畸形的重要原因之一[2-3]。因此,了解生长素与脱落酸及两者互作对草莓果实发育的调控机制,筛选重要的调控因子,对开展草莓分子生物学育种及优良草莓品种培养具有重要意义。
生长素信号的转导受3个主要基因家族Aux/IAAs、SAURs 和GH3s 的调控[4]。生长素浓度较低时,Aux/IAAs与ARF(Auxin response factor)结合,抑制生长素响应基因的表达;生长素浓度较高时,生长素受体(TIR1/AFBs)与生长素结合后导致Aux/IAAs泛素化进而被26S蛋白酶体降解,使ARF从ARF-Aux/IAA聚合物中游离下来,从而激活生长素响应信号[5-7]。
近年来对Aux/IAAs 家族基因在植物发育过程中的功能研究越来越受到重视,其家族基因的功能不断被发掘[8-11]。Aux/IAA家族基因典型的4个保守结构域,分别是Domain Ⅰ、Ⅱ、Ⅲ、Ⅳ。Domain Ⅰ有转录抑制功能,具有一个LxLxLx典型基序;Domain Ⅱ对Aux/IAA 蛋白的稳定性起作用,核心序列为GWPPV;Domain Ⅲ和Ⅳ负责蛋白的二聚化,可与生长素响应因子ARF家族基因形成二聚体[12]。在拟南芥中的研究表明,大多数Aux/IAAs 家族成员大多与根的生长发育有关,如AtIAA28[13]、AtIAA19[14]、AtIAA14[15]等。水稻OsIAA9、 OsIAA11对水稻侧根的发育有重要调控作用[16-17];马铃薯StIAA2 下调表达使植株高度增加、叶柄表现出偏向下性、并降低了顶端叶原基生长的弯曲程度等[18]。目前,Aux/IAA家族基因已经在马铃薯[19]、大豆[20]、白菜[21]、木瓜[22]、油菜[23]等多种果蔬中被分离和鉴定。
关于Aux/IAA家族基因在果实发育过程中的功能研究主要以番茄为模式植物进行,抑制表达番茄SlIAA3蛋白使番茄植株对生长素的敏感性下降,从而表现出顶端优势、叶片形态及乙烯可诱导表型的改变等[24],抑制表达SlIAA9基因使植株表现出多种表型变化,如形成单叶,坐果过程提前发生[25]; 抑制表达Sl-IAA27后导致番茄果实变小[26];而抑制表达Sl-IAA17,通过影响细胞的膨大过程使番茄果实增大[27]。而关于Aux/IAA家族基因在草莓发育过程中的功能研究进展缓慢。
鉴于此,本研究以森林草莓(Fragaria vesca)为试验材料,克隆得到森林FvIAA17基因,通过DNAMAN、ProtParam、TargetP、MEGA 5等生物信息学工具对其序列特征进行分析,同时利用实时荧光定量PCR对其表达特性进行了初步的探究,以期为深入研究该基因的功能奠定基础。
1 材料和方法
1.1 试材与处理
选用森林草莓为试材,材料种苗来源于西安市农业技术推广中心,移栽种植于西安文理学院温室大棚中,培养条件为温度。采集成株盛期的根、茎、叶、花组织,果实取3个时期,即绿果(花后12 d)、1/2红果(花后20~21 d)、红熟果(花后25~26 d),将植物材料用液氮冷冻,保存于-70 ℃冰箱备用。
取长势良好的草莓幼苗,待其第5~6片叶子展开后,取地上部分分别置于蒸馏水、生长素(IAA,50 mg/L)、脱落酸(ABA,100 μmol/L)、IAA+ABA(终质量浓度分别为50 mg/L和100 μmol/L)溶液中,分别于处理后0,1,6,12 h取材。每个样品3次生物学重复。试材液氮冷冻后存于-70 ℃冰箱。
1.2 试验方法
1.2.1 RNA提取及cDNA合成 RNA提取试剂盒购于TIANGEN(北京),按照说明书进行RNA提取,利用1%的琼脂糖凝胶进行RNA完整性检测;反转录试剂盒购自TaKaRa宝生物工程有限公司(大连),根据试剂盒说明书合成cDNA。
1.2.2 FvIAA17基因的克隆 以草莓叶片cDNA为模板,用Primer 6.0设计IAA17基因特异性引物(表1)进行PCR扩增。PCR 反应条件为94 ℃预变性5 min; 94 ℃变性30 s, 58 ℃退火30 s, 72 ℃延伸40 s, 35个循环; 最后72 ℃延伸7 min。PCR 产物经回收、连接、转化后,挑取阳性克隆子送往上海生物工程有限公司进行DNA测序。
1.2.3 FvIAA17基因生物信息学分析 方法同文献[28]。
1.2.4 FvIAA17基因表达分析 利用Primer 6.0设计FvIAA17基因qPCR特异性引物(表1),内参为草莓Fv18S,引物由上海生工合成。qPCR采用罗氏FastStart Universal SYBR Green Master试剂盒。方法步骤参照说明书进行。每个样品3次重复。试验结果用2-ΔΔCT法对数据进行定量分析,同文献[28]。
表1 引物序列
Tab.1 Primer sequences
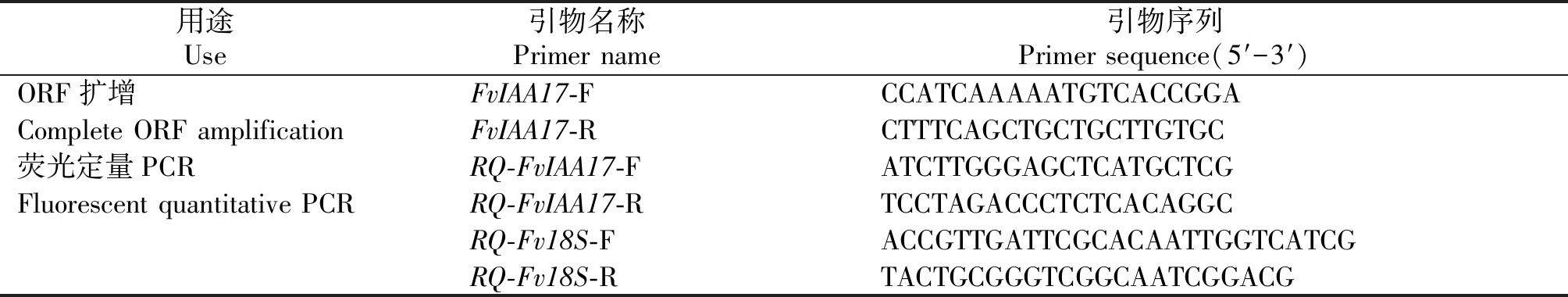
用途Use引物名称 Primer name引物序列Primer sequence(5′-3′)ORF扩增FvIAA17-FCCATCAAAAATGTCACCGGAComplete ORF amplificationFvIAA17-RCTTTCAGCTGCTGCTTGTGC荧光定量PCRRQ-FvIAA17-FATCTTGGGAGCTCATGCTCGFluorescent quantitative PCRRQ-FvIAA17-RTCCTAGACCCTCTCACAGGC RQ-Fv18S-FACCGTTGATTCGCACAATTGGTCATCGRQ-Fv18S-RTACTGCGGGTCGGCAATCGGACG
2 结果与分析
2.1 FvIAA17的克隆和序列分析
以草莓叶片cDNA为模板,利用特异性引物进行PCR 扩增(图1)。测序后得到约594 bp序列,分析后表明目标基因序列一致。FvIAA17基因编码197个氨基酸。预测其分子质量为21.85 ku,等电点为7.56。
2.2 FvIAA17蛋白生物信息学分析
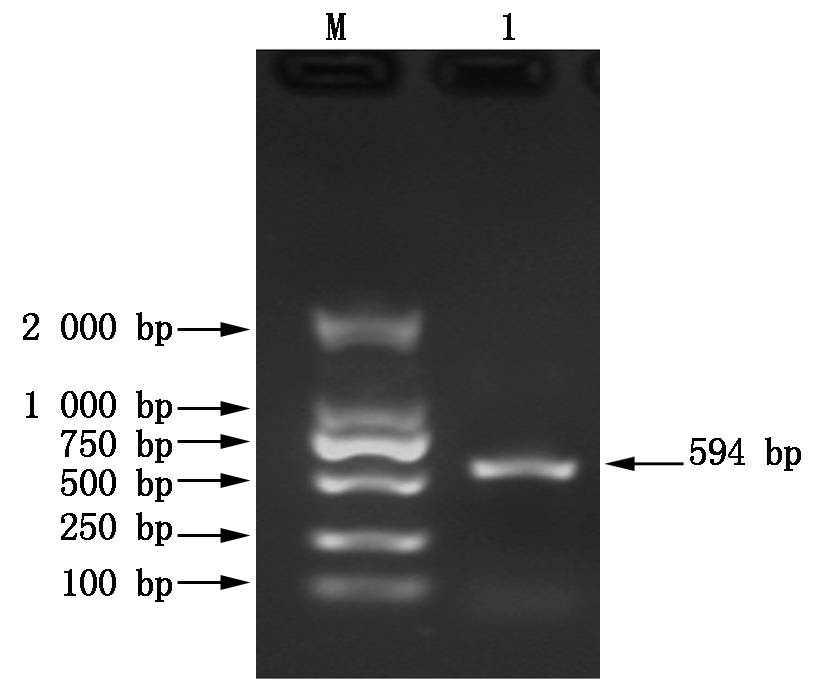
1.FvIAA17 基因克隆; M.M 2 000 bp Marker。
1.Amplification of the FvIAA17 gene; M.M 2 000 bp Marker.
图1 FvIAA17基因的PCR扩增
Fig.1 Agarose gel electrophoresis analysis of FvIAA17 gene fragments
2.2.1 FvIAA17基因启动子序列分析 分析表明(表2),FvIAA17启动子区含有TATA框、CAAT框等调控真核基因转录的基本元件,同时还具有多种植物激素及逆境胁迫响应元件,如ABA、MeJA茉莉酸甲酯诱导响应、低温、低氧胁迫元件等。
表2 FvIAA17启动子顺式作用元件分析
Tab.2 The cis-elements in the promoter of FvIAA17

应答元件类型The type of cis-elements数量Number脱落酸诱导 The abscisic acid responsive element6茉莉酸甲酯诱导 The MeJA responsiveness12光响应 The light responsiveness16低氧胁迫 The anaerobic responsiveness3低温胁迫 The low-temperature stress responsiveness1
2.2.2 FvIAA17基因同源序列比对及进化分析 利用DNAman软件将FvIAA17与其他物种中同源性较高的氨基酸序列进行比对。结果显示,FvIAA17的蛋白序列具有生长素诱导基因Aux/IAA家族典型的4个保守结构域(图2)。据编码FvIAA17的氨基酸序列并结合其他植物同源氨基酸序列,利用MEGA 5.0进行系统进化树分析(图3)。结果显示FvIAA17基因与苹果MdIAA1基因的亲缘关系最近。


NaIAA17-L.烟草(XP_019229757.1); PaIAA1.甜樱桃(XP_021817959.1); AtIAA17.拟南芥(NP_171921.1); CcIAA17.黄辣椒(PHU24404.1);RcIAA1.蓖麻(XP_002517529.1); StIAA17-L. 马铃薯(XP_006343103.2);VaIAA17.赤豆(XP_017425536.1);CsIAA1.甜橙(XP_006465436.1);PpIAA1.桃树(XP_007215971.1);FvIAA17.草莓(XP_004302956.1);SiIAA14.芝麻(XP_011081682.1);PbIAA1.白梨(XP_009339009.1);GaIAA16.树棉(XP_017605091.1);QsIAA17.栓皮栎(POF06625.1);JrIAA17-L.胡桃(XP_018847560.1); MdIAA1. 苹果(XP_008377585.1);MnIAA17. 桑树(XP_010110305.1)。黑色下划线为FvIAA17 4个保守结构域。
NaIAA17-L. Nicotiana attenuate(XP_019229757.1); PaIAA1. Prunus avium(XP_021817959.1); AtIAA17. Arabidopsis thaliana (NP_171921.1); CcIAA17.Capsicum chinense(PHU24404.1); RcIAA1. Ricinus communis(XP_002517529.1); StIAA17-L. Solanum tuberosum (XP_006343103.2); VaIAA17. Vigna angularis(XP_017425536.1); CsIAA1. Citrus sinensis (XP_006465436.1); PpIAA1. Prunus persica (XP_007215971.1); FvIAA17. Fragaria vesca (XP_004302956.1); SiIAA14. Sesamum indicum(XP_011081682.1); PbIAA1. Pyrus× bretschneideri(XP_009339009.1); GaIAA16. Gossypium arboretum(XP_017605091.1); QsIAA17. Quercus suber (POF06625.1); JrIAA17-L. Juglans regia(XP_018847560.1); MdIAA1. Malus domestica(XP_008377585.1); MnIAA17. Morus notabilis (XP_010110305.1).The conserved domainⅠ,Ⅱ,Ⅲ,Ⅳ of FvIAA17 was marked by single black line.
图2 草莓FvIAA17氨基酸序列和所选物种Aux/IAA家族基因氨基酸序列比对
Fig.2 Analysis of the FvIAA17 amino acid sequence and these selected Aux/IAA from other plant species

图3 FvIAA17基因与其他植物Aux/IAAs基因进化关系分析
Fig.3 Phylogenetic relationship based on FvIAA17 amino acid sequence and other plant Aux/IAAs
2.2.3 FvIAA17亚细胞定位、信号肽、跨膜结构分析及磷酸化位点预测 利用Cello对FvIAA17蛋白进行亚细胞定位预测(表3),表中数值越大表明结果可信度越高。因此,FvIAA17蛋白定位于周质间(表3),具体定位情况仍需试验进行验证确定。FvIAA17蛋白无信号肽,无跨膜结构;磷酸化位点进行预测结果表明,FvIAA17有18个Ser、15个Thr和2个Tyr。
表3 FvIAA17亚细胞定位预测
Tab.3 The information in predicting FvIAA17 subcellular localization

定位 Localization 可信度 Reliability周质 Periplasmic2.029∗细胞质Cytoplasmic1.225线粒体外膜Outer membrane0.971胞外Extracellular0.688线粒体内膜Inner membrane0.088
2.3 FvIAA17基因在草莓不同组织中的表达分析
以草莓Fv18S为内参基因,通过RT-qPCR检测发现,草莓中FvIAA17在草莓根、茎、叶、花、果实中均有表达,但相对表达量差异明显,以目的基因在根中表达量为对照,其在绿果、茎和1/2红果中表达量显著增加,特别是在绿果和茎中表达量较高,分别比其在根中表达量高4.0,2.3倍(图4),可能与该基因在不同组织中的功能差异相关。
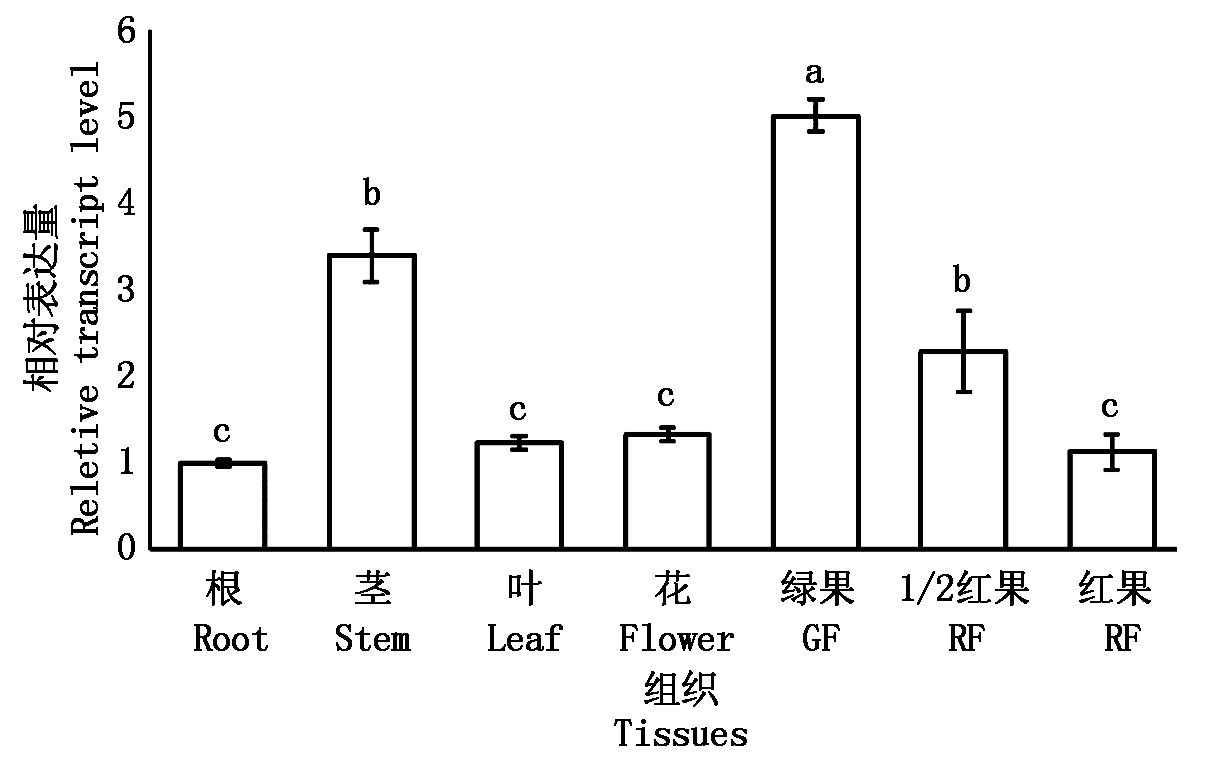
不同小写字母表示在0.05水平差异显著。图5同。
Different small letters show significantly different at 0.05.The same as Fig.5.
图4 FvIAA17基因在草莓不同组织中的表达情况
Fig.4 Relative expression of FvIAA17 gene in different tissues of strawberry
2.4 外源激素处理对FvIAA17基因表达水平的影响
RT-qPCR检测发现(图5),与无处理对照组相比,FvIAA17在IAA处理早期1,6 h受到强烈诱导呈上调表达,在处理后6 h达到最大表达量,而后随着处理时间增长渐恢复正常水平;ABA处理草莓植株后,与对照组相比,FvIAA17的表达水平在处理1,6 h均呈显著性下调趋势,12 h后恢复正常水平;IAA+ABA处理后,FvIAA17基因表达模式与IAA处理趋势相同,表现为先升高而后恢复,但上升的幅度有所下降,表明IAA和ABA联合作用时,IAA的调控作用处于主导地位,IAA与ABA之间可能存在互作调控关系。
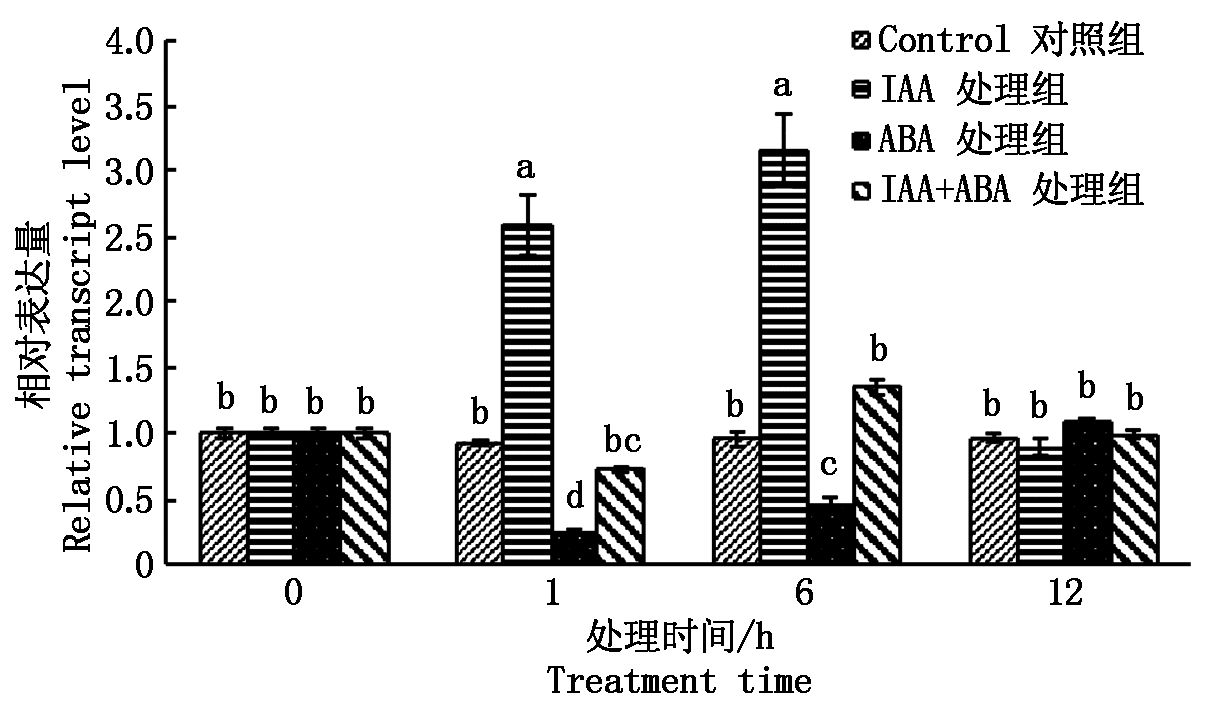
图5 不同激素处理下FvIAA17相对表达量
Fig.5 Relative expression of FvIAA17 gene under different hormone treatment.
3 结论与讨论
草莓属于蔷薇科草莓属,成熟过程中不形成乙烯合成高峰,属于呼吸非跃变型果实。与呼吸跃变型果实相比,植物激素调控呼吸非跃变型果实成熟机理的研究发展缓慢。生长素是第一个被鉴定的植物激素,研究表明,生长素调控果实生长发育过程,特别是果实的膨大期,但对于果实的成熟有一定的抑制作用[29]。对草莓和葡萄成熟机制研究表明,生长素与ABA可能互作调控果实的成熟过程[30-31],但二者互作的机制及关键的调控因子尚不清楚。生长素对草莓的花诱导控制及其授粉后果实的生长发育过程有重要调控作用,其分泌过多将导致草莓花芽分化异常、草莓畸形果等[32]。因此,生长素及其互作激素对草莓果实发育的调控机制研究,特别是解析关键调控因子的功能对通过分子育种手段培育优良草莓品种具有重要的指导意义。
本研究以森林草莓为材料,克隆得到FvIAA17基因全长,该基因CDS序列为594 bp,编码197个氨基酸,其分子质量和等电点分别为21.85 ku和7.56。生物信息学分析可知,FvIAA17基因具有典型的Aux/IAA家族的4个保守区,进化树分析表明,FvIAA17与苹果的MdIAA1基因亲缘关系较近。荧光定量PCR研究表明,FvIAA17基因在草莓的不同组织中均有表达,在绿果期果实中表达量最高,推测FvIAA17基因可能参与调控草莓果实早期的发育过程。外源激素IAA处理发现,FvIAA17基因在处理早期1,6 h受到强烈诱导呈上调表达,并在处理6 h后达到顶峰,而后渐恢复正常水平;ABA处理草莓植株,处理后1,6 h FvIAA17呈显著性下调表达,而后渐恢复正常水平;IAA与ABA联合处理草莓时,FvIAA17基因表达模式与IAA单独处理时表达趋势有相似性,但强度明显下降,表明2种激素同时存在,对草莓FvIAA17基因的调控作用IAA处于主导地位,表明在草莓发育过程中,生长素和ABA对FvIAA17基因有一定的联合调控作用,推测FvIAA17基因可能在响应生长素和脱落酸互作调控草莓发育过程中发挥一定的作用。
Aux/IAA家族基因在生长素信号转导过程的重要成员之一,本研究结果可初步推测FvIAA17基因可能在草莓发育有一定的功能作用,并且可能参与生长素与脱落酸互作调控草莓果实发育过程,但对于FvIAA17基因对草莓的生物学功能和作用机制需通过遗传转化手段进行进一步的验证,进而在分子水平上揭示FvIAA17基因调控草莓果实发育的机制,为将来草莓优良品种的培育提供理论基础。
[1] Symons G M, Chua Y J, Ross J J, Quittenden L J, Davies N W, Reid J B. Hormonal changes during non-climacteric ripening in strawberry[J]. Journal of Experimental Botany, 2012, 63(13): 4741-4750. doi: 10.1093/jxb/ers147.
[2] 张运涛, 王桂霞, 董静,王萍. 草莓畸形果形成的原因分析[J]. 落叶果树, 2004, 36(6): 10-13. doi: 10.3969/j.issn.1002-2910.2004.06.004.
Zhang Y T, Wang G Y, Dong J, Wang P. Reasons for occurrence of deformed berries in strawberry[J]. Deciduous Fruits, 2004, 36(6): 10-13.
[3] 程然, 生吉萍. 草莓果实成熟衰老影响因子及其调控机制研究进展[J]. 食品科学, 2015, 36(9): 242-247. doi: 10.7506/spkx1002-6630-201509045.
Cheng R, Sheng J P. Recent progress in research on influencing factors and regulation mechanisms of strawberry fruit ripening and senescence[J]. Food Science, 2015, 36(9): 242-247.
[4] Ma Q, Grones P, Robert S. Auxin signaling: a big question to be addressed by small molecules[J]. Journal of Experimental Botany, 2018, 69(2): 313-328. doi: 10.1093/jxb/erx375.
[5] Leyser O. Auxin signaling[J]. Plant Physiology, 2018, 176(1): 465-479. doi:10.1104/pp.17.00765.
[6] Roosjen M, Paque S, Weijers D. Auxin response factors: output control in auxin biology[J]. Journal of Experimental Botany,2018,69(2):179-188. doi: 10.1093/jxb/erx237.
[7] Luo J, Zhou J J, Zhang J Z. Aux/IAA gene family in plants: molecular structure, regulation, and function[J]. International Journal of Molecular Sciences, 2018, 19(1): 259. doi: 10.3390/ijms19010259.
[8] Xie R J, Pang S P, Ma Y Y, Deng L, He S L, Yi S L, Lü Q, Zheng Y Q. The ARF, AUX/IAA, and GH3, gene families in citrus: genome-wide identification and expression analysis during fruitlet drop from abscission zone A[J]. Molecular Genetics and Genomics, 2015, 290(6): 2089-2105. doi: 10.1007/s00438-015-1063-1.
[9] Filiz E, Koc I. Genome-wide identification and comparative analysis of EPSPS(aroA)genes in different plant species[J]. Journal of Plant Biochemistry and Biotechnology, 2016, 25(1): 21-29. doi: 10.1007/s13562-015-0303-0.
[10] Liu M C, Chen Y, Chen Y, Shin J H, Mila I, Audran C, Zouine M, Pirrello J, Bouzayen M. The tomato ethylene response factor Sl-ERF.B3 integrates ethylene and auxin signaling via direct regulation of Sl-Aux/IAA27[J]. New Phytologist, 2018,219(2):631-640. doi: 10.1111/nph.15165.
[11] Calderón Villalobos L I A, Lee S, De O C, Ivetac A, Brandt W, Armitage L, Sheard L B, Tan X, Parry G, Mao H B, Zheng N, Napier R, Kepinski S, Estelle M. A combinatorial TIR1/AFB-Aux/IAA co-receptor system for differential sensing of auxin[J]. Nature Chemical Biology, 2016, 8(5): 477-485. doi: 10.1038/nchembio.926.
[12] 李俊男, 燕晓杰, 李枢航, 张荣沭. 植物AUX/IAA基因家族研究进展[J]. 中国农学通报, 2018, 34(15): 89-92.
Li J N, Yan X J, Li S H, Zhang R M. Plants AUX/IAA gene family: research progress[J]. Chinese Agricultural Science Bulletin, 2018, 34(15): 89-92.
[13] Rogg L E, Lasswell J, Bartel B. A gain-of-function mutation in IAA28 suppresses lateral root development[J]. Plant Cell, 2013, 13(3): 465-480. doi: 10.2307/3871400.
[14] Tatematsu K, Kumagai S, Muto H, Sato A, Watahiki M K, Harper R M, Liscum E, Yamamotao K T. MASSUGU2 Encodes Aux/IAA19, an Auxin-Regulated protein that functions together with the transcriptional activator NPH4/ARF7 to regulate differential growth responses of hypocotyl and formation of lateral roots in Arabidopsis thaliana[J]. The Plant Cell Online, 2004, 16(2): 379-393.doi:10.1105/tpc.018630.
[15] Fukaki H, Tameda S, Masuda H, Tasaka M. Lateral root formation is blocked by a gain-of-function mutation in the SOLITARY-ROOT/IAA14 gene of Arabidopsis[J]. Plant Journal, 2010, 29(2): 153-168. doi: 10.1046/j.0960-7412.2001.0201.x.
[16] Sha L, Li Q Q, Liu S D, Pinos N M, Tian H N, Wang S C. Constitutive expression of OsIAA9 affects starch granules accumulation and root gravitropic response in Arabidopsis[J]. Frontiers in Plant Science, 2015, 6(295):01156.doi:10.1046/j.0960-7412.2001.01201.X.
[17] Chen Y, Yang Q F, Sang S H, Wei Z Y, Wang P. Rice inositol polyphosphate kinase(OsIPK2)directly interacts with OsIAA11 to regulate lateral root formation[J]. Plant & Cell Physiology, 2017, 58(11): 1891-1900. doi: 10.1093/pcp/pcx125.
[18] Kloosterman B, Visser R G F, Bachem C W B. Isolation and characterization of a novel potato auxin/indole-3-acetic acid family member(StIAA2)that is involved in petiole hyponasty and shoot morphogenesis[J]. Plant Physiology & Biochemistry, 2006, 44(11-12): 766-775.doi: 10.1016/j.plaphy.2006.10.026.
[19] Gao J P, Cao X L, Shi S D, Ma Y L, Wang K, Liu S J, Chen D, Chen Q, Ma H L. Genome-wide survey of Aux/IAA, gene family members in potato(Solanum tuberosum): identification, expression analysis, and evaluation of their roles in tuber development[J]. Biochemical and Biophysical Research Communications, 2016, 471(2): 320-327. doi: 10.1016/j.bbrc.2016.02.013.
[20] Singh V K, Jain M. Genome-wide survey and comprehensive expression profiling of Aux/IAA gene family in chickpea and soybean[J]. Frontiers in Plant Science, 2015, 6:918. doi: 10.3389/fpls.2015.00918.
[21] Paul P, Dhandapani V, Rameneni J J, Li X N, Sivanandhan G, Choi S R, Pang W X, Im S, Lim Y P. Genome-wide analysis and characterization of Aux/IAA family genes in Brassica rapa[J]. PLoS One, 2016, 11(4): e0151522. doi: 10.1371/journal.pone.0151522.
[22] Liu K D, Yuan C C, Feng S X, Zhong S T, Li H L, Zhong J D, Shen C J, Liu J X. Genome-wide analysis and characterization of Aux/IAA, family genes related to fruit ripening in papaya(Carica papaya, L.)[J]. Bmc Genomics, 2017, 18(1): 351. doi: 10.1186/s12864-017-3722-6.
[23] Li H, Wang B, Zhang Q, Wang J, King G J, Liu K. Genome-wide analysis of the auxin/indoleacetic acid(Aux/IAA)gene family in allotetraploid rapeseed(Brassica napus L.)[J]. BMC Plant Biology, 2017, 17(1): 204. doi: 10.1186/s12870-017-1165-5.
[24] Salma C, Alain L, Claude P J, Mondher B. Tomato Aux/IAA3 and HOOKLESS are important actors of the interplay between auxin and ethylene during apical hook formation[J]. Plant Signaling & Behavior, 2009, 4(4): 559-560. doi: 10.4161/Psb.4.6.8748.
[25] Wang H, Jones B, Li Z G, Frasse P, Delalande C, Regad F, Chaabouni S, Latché A, Pech J C, Bouzayen M. The tomato Aux/IAA transcription factor IAA9 is involved in fruit development and leaf morphogenesis[J]. Plant Cell, 2005, 17(10): 2676-2692. doi: 10.1105/tpc.105.033415.
[26] Bassa C, Mila I, Bouzayen M, Audran-Delalande C. Phenotypes associated with down-regulation of Sl-IAA27 support functional diversity among Aux/IAA family members in tomato[J]. Plant & Cell Physiology, 2012, 53(9): 1583-1595. doi: 10.1093/pcp/pcs101.
[27] Su L Y, Bassa C, Audran C, Mila I, Cheniclet C, Chevalier C, Bouzayen M, Roustan J P, Chervin C. The auxin Sl-IAA17 transcriptional repressor controls fruit size via the regulation of endoreduplication-related cell expansion[J]. Plant & Cell Physiology, 2014, 55(11): 1969-1976. doi: 10.1093/pcp/pcu124.
[28] 苏丽艳. 草莓生长素响应因子FvARF10和FvARF18基因的克隆及表达分析[J]. 西北植物学报, 2018, 38(7): 1203-1211. doi: 10.7606/j.issn.1000-4025.2018.07.1203.
Su L Y. Cloning and characterization of auxin response factor gene FvARF10 and FvARF18 in Fragaria vesca[J]. Acta Botanica Boreali-Occidentalia Sinica, 2018, 38(7): 1203-1211.
[29] Pattison R J, Csukasi F, Catalá C. Mechanisms regulating auxin action during fruit development[J]. Physiologia Plantarum, 2014, 151(1):62-72. doi: 10.1111/ppl.12142.
[30] Chen J X, Mao L C, Lu W J, Yin T J, Luo Z S. Transcriptome profiling of postharvest strawberry fruit in response to exogenous auxin and abscisic acid[J]. Planta, 2016, 243(1):183-197. doi: 10.1007/s00425-015-2402-5.
[31] Fortes A M, Teixeira R T, Agudelo-Romero P. Complex interplay of hormonal signals during grape berry ripening[J]. Molecules, 2015, 20(5): 9326-9343. doi: 10.3390/molecules20059326.
[32] 贾海锋, 赵密珍, 王庆莲,房经贵,赵鹏程,刘众杰,张成,纠松涛. 生长素和脱落酸在草莓果实发育过程中的作用[J]. 江苏农业科学, 2016, 44(11): 173-176. doi: 10.15889/j.issn.1002-1302.2016.11.051.
Jia H F, Zhao M Z, Wang Q L, Fang J G, Zhao P C, Liu Z J, Zhang C, Jiu S T. Effects of auxin and abscisic acid on the development of strawberry fruit[J]. Jiangsu Agricultural Sciences, 2016, 44(11): 173-176.