开花时间是作物最重要的农艺性状之一[1]。开花对植物授粉、种子发育和形态、籽粒产量、易驯化程度以及环境适应性等方面会产生重要影响。对植物开花时间的调控是植物繁殖的核心问题[2]。在特定的时间开花是植物由营养生长转变为生殖生长的关键步骤,是植物生殖产生后代的重要节点[3]。植物开花受多种内源和外源因素的调节[4]。近些年,通过对模式植物拟南芥突变体的研究,在调控植物开花的途径中已经发现了很多个重要的基因[5-10]。其中,光周期途径(Photoperiod pathway)是调节植物开花的四大重要途径之一[11]。拟南芥光周期开花调节途径中,光受体接收光照时间长度、强度等光信号,并将信号传递给生物节律钟[12]。根据光受体吸收光谱成分的不同可将其分为三类,即光敏色素(Phytochromes)、隐花色素(Cryptochromes)和向光蛋白(Phototropin)[12]。光敏色素包含五类,即phyA(phytochrome A)、phyB(phytochrome B)、phyC(phytochrome C)、phyD(phytochrome D)和phyE(phytochrome E)。光敏色素主要吸收红光(Red light,R)和远红光(Far-Red light,FR),前者抑制拟南芥花朵的开放,而远红光则作用相反[13]。在远红光条件下,phyA会抑制开花抑制因子的生成进而促使植物开花[14]。然而,相同条件下phyb突变体则提早开花,这说明phyB抑制开花,但也有研究表明phyB过量表达会促进植物开花[15]。另外,phyC的功能实现也与phyB的活化相关[16]。隐花色素有两类,Cry1(Cryptochromes 1)和Cry2(Cryptochromes 2),主要感知可见光中的蓝光[17]。Cry1与Cry12和phyA介导蓝光条件下植物的去黄化,促进开花[18]。向光蛋白包含Phot1(Phototropin 1)和Phot2(Phototropin 2),主要表现在影响植物对蓝光依赖的向光性反应[19],会对叶绿体的运动和植物气孔的开闭产生影响[20],另外,向光蛋白自身并不直接参与植物开花过程的调控,对开花时间不会产生影响。
光周期途径中,当植物体感受到光刺激时会产生规律的自我调节,而这一内在节律性被称为生物钟,又叫生理钟、昼夜节律钟。大多数真核生物的生理过程都是受生物钟的调控[21]。与此同时,由生物钟控制的许多输出路径会对生物钟产生负反馈,导致时钟变为复杂监管网络中的中心枢纽。植物的一些生理现象,例如叶片运动、气孔开闭、CO2的固定等均受到生物钟的调控,因此能够观察到植物的昼夜规律[22]。生物钟通常被分为3个部分[23-24]:①输入途径:使生物钟机制与昼夜、温度循环同步,能够向中心振荡子传送光信号或者温度信号,使中央振荡子与光暗循环保持相同的机制;②中心振荡子:是生物钟的核心组成部分,控制24 h的昼夜节律;③输出系统:受中心振荡子控制,由许多生化途径和发育途径组成。光信号将节律信号通过中央振荡器的波动传输给下游调节基因,参与下游成花诱导与花发育基因的表达调控。GIGANTEA(GI)是节律输出的通道基因,GI转录物的含量受节律钟的调控,在清晨时转录水平处于最低点,之后持续上升,在大约9 h后达到最高点。GI是控制植物开花的重要基因CO的上游基因,其与ELF3和FKF1共同作用,可以调控CO和FT的转录翻译进而促进开花[25-26]。通过植物本身对生物钟和光信号的处理,将这2种信号结合起来,进而保证CO(CONSTANS)基因的转录及翻译的稳定性[27]。CO(CONSTANS)是在拟南芥中发现的第一个由节律调控并且会对拟南芥开花产生影响的基因。中央振荡器是生物钟的最关键的部分,输入生物节律钟的光信号在中央振荡器中产生节律并向下游通路传递昼夜节律信号。中央振荡器主要由2个MYB类蛋白基因LHY(Late elongated hypocotyl)、CCA1(Circadian clock associated1)组成的具有自主调控机制的负反馈环和一种拟南芥伪应答调节蛋白基因家族APRRs(Arabidopsis pseudo-response regulators)组成[28]。
TOC1(Timing of CAB expression 1)又叫APRR1或PRR1,是PRR家族主要成员之一[29]。APRRs小家族有5个成员,包括APRR1/TOC1、APRR3、APRR5、APRR7、APRR9,并且其余4个基因的转录水平和翻译水平与TOC1类似,都表现出昼夜节律变化。与PRR家族其他成员结构类似,TOC1的N端存在PR结构域(Pseudoreceiver domain),C端存在CCT结构域(CONSTANS(CO), CO-like, TOC1), 但与 PRR5、PRR7 和 PRR9 不同的是,TOC1基因不存在IR区域(Intermediate region)[29]。TOC1主要负责中央振荡器中的调节。中央振荡器核心的负反馈环由Alabadí等[30]在2001年发现。Alabadí等[30]针对cca1、lhy以及TOC1过表达的拟南芥植株证明,在夜晚TOC1基因表达量升高,进而刺激CCA1/LHY表达量的升高,紧接着CCA1/LHY的积累会反馈抑制TOC1基因的表达,这就是中央振荡器中核心的负反馈环。
随后,Nakamichi等[31]发现了中央振荡器在早晨也会形成一个由CCA1/LHY与PRR9/PRR7构成的负反馈环。2006年,Locke等[32]依据试验构建出了一个完整的中央振荡器的反馈模型。TOC1在早晨的表达量达到最高值,紧接着CCA1/LHY与TOC1启动子区域的夜晚元件EE(EE-element,AAATATCT)相结合,使TOC1的转录被抑制。当CCA1/LHY表达量最大时, CCA1/LHY启动子区域的顺式作用元件(TOC1 morning element,T1ME)会与TOC1的CCT结构域结合进而抑制CCA1/LHY表达[33]。
生物钟与开花调节的信号网络紧密相连,这也就使得植物开花调节机制变得更为复杂。转录因子TOC1(Timing of CAB expression 1)是植物生物钟的基本组成成分,之前在拟南芥的研究中表明,AtTOC1会对LHY/CCA1的表达有促进作用,也与ZTL、FKF 1、ABA等基因产生互作。可是,在玉米中对TOC1的研究并不深入,并且TOC1在玉米中存在2个同源基因,因此,研究TOC1在玉米中的作用也更为必要。本研究通过拟南芥AtTOC1基因的氨基酸序列得到2个在玉米内的同源基因ZmTOC1a及ZmTOC1b,克隆获得了这2个基因的开放阅读框的序列,并对目的基因编码蛋白的特性和表达进行了分析,初步了解了ZmTOC1a和ZmTOC1b的表达模式。对植物光周期的调控因子的研究和进一步深入挖掘,不仅有利于加深对植物花发育过程生物学机理的了解,而且有利于利用热带、亚热带玉米种质资源。
1 材料和方法
1.1 试验材料
试验所需玉米材料B73由四川农业大学玉米研究所提供。DNA Marker、大肠杆菌感受态购自北京全式金生物技术有限公司;KOD 购自 TaKaRa 公司;限制性内切酶KpnⅠ、Xba Ⅰ购买于NEB公司;Plasmid Extraction Kit、Gel Extraction Kit 购自 Omega 公司;SuperScript®Ⅱ Reverse Transcriptase Kit 购自 Invitrogen 公司;RNase Free 的枪头购自 Axygen 公司;卡那霉素、氨苄青霉素、利福霉素、乙酰丁香酮等购自 Biotopped 公司。
1.2 试验方法
1.2.1 RNA提取及cDNA合成 将玉米种子放在有充足蒸馏水的滤纸上28 ℃催芽3 d,种植在营养土中培养至三叶一心时,挑选长势正常的植株。取幼嫩的玉米叶片 2 g 剪碎放入液氮预冷的研钵,加入适量液氮,迅速研磨直至叶片变为粉末后,将样本转入1.5 mL离心管中。按照RNA试剂盒提取说明书,提取总 RNA。使用浓度为1.5%的琼脂糖对样品进行条带完整性检测,并结合核酸蛋白仪检测RNA样品浓度和纯度。将电泳图中条带清晰、仪器检测浓度合适的样品,使用SuperScript®Ⅱ Reverse Transcriptase Kit 反转录为cDNA。
1.2.2 候选基因同源克隆 采用GenBank数据库中拟南芥AtTOC1(AT5G61380)的氨基酸序列,在玉米数据库MaizeGDB中Blast同源搜索到玉米TOC1基因。在UTR区域设计特异性引物TOC1a-F/R、TOC1b-F/R(表1),以基因组cDNA作为模板,使用KOD高保真酶进行基因CDS全长序列的扩增。扩增完成后,连接pEASY-BLUNT载体,进行测序。将测序正确的菌液提取质粒并保存于-80 ℃。
表1 ZmTOC1a和ZmTOC1b基因扩增引物信息
Tab.1 Information list of ZmTOC1a/ZmTOC1b amplification primer
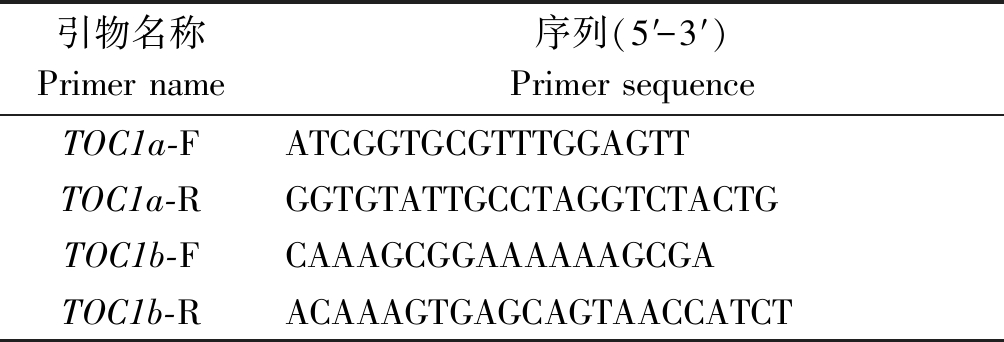
引物名称Primer name序列(5′-3′)Primer sequenceTOC1a-FATCGGTGCGTTTGGAGTTTOC1a-RGGTGTATTGCCTAGGTCTACTGTOC1b-FCAAAGCGGAAAAAAGCGATOC1b-RACAAAGTGAGCAGTAACCATCT
1.2.3 亚细胞定位分析
1.2.3.1 构建表达载体 根据ZmTOC1a/ZmTOC1b在Maize GDB上的序列信息,设计去除终止密码子的CDS区域的扩增引物,并在引物两端分别加上合适的同源臂序列。以保存的质粒为模板,使用带有同源臂的引物扩增基因完整的开放阅读框。将表达载体pCAMBIA2300-35S-eGFP使用限制性内切酶KpnⅠ及XbaⅠ线性化。而后使用ClonExpress®MultiS One Step Cloning Kit(南京,诺唯赞)将线性化载体与PCR产物连接并转化大肠杆菌感受态。挑取单克隆,进行PCR检测后,将阳性菌落进行质粒提取及测序。在测序结果比对正确后,将阳性单克隆的质粒转化农杆菌GV3101,从而构建目的基因与eGFP融合的过量表达载体: pCAMBIA2300-35S:ZmTOC1a-eGFP,pCAMBIA2300-35S:ZmTOC1b-eGFP。
1.2.3.2 农杆菌注射烟草 将含重组质粒的农杆菌100 μL加入至含有卡那霉素以及利福平(质量浓度均为50 mg/L)的YEB液体培养基中,28 ℃过夜振荡培养至生长对数期(OD600约等于0.6),4 000 r/min离心8 min收集菌体倒掉上清液。而后,使用无菌水(含有终浓度为 10 mmol/L MgCl2,100 mmol/L MES(pH值5.7)和100 μmol/L乙酰丁香酮)重悬菌体,使菌液浓度达到OD600≈1.0;选取叶龄在5周左右的本氏烟草,用1 mL注射器(去掉针头)在叶片背部注射菌液直至叶片组织空隙被液体填满;而后将注射后的烟草放置于25 ℃条件下培养,48 h后用共聚焦显微镜观察注射叶片。
1.2.4 蛋白特性分析及基因进化树构建 使用DNAMAN将目的基因的序列进行翻译;使用Prot PARAM对基因表达蛋白进行理化性质分析;使用NetPhos 3.1 Server对蛋白质磷酸化位点预测;使用MEGA 5.0 构建基因进化树, 进化树构建方法采用MEGA 软件的NJ 法(Neighbor Joining Method), Bootstrap 值由 1 000 次重复得到。
1.2.5 基因组织表达分析 分别取玉米的胚芽鞘、胚根、花药、雄穗、穗轴、花丝、苞叶、穗位叶、茎、气生根及根系,使用液氮速冻,并磨碎为粉末状。后使用RNA提取试剂盒将这11个部位叶片提取RNA,并反转录为cDNA(参考1.2.1),稀释5倍至工作浓度备用。根据基因序列设计引物,在11份不同组织的cDNA中扩增待测基因和内参基因,本次组织定量试验内参选择为18S。PCR 程序为 95 ℃ 30 s;95 ℃ 3 s,58 ℃ 5 s,40 个循环,每个样本3个重复。实时荧光定量PCR体系参照表2。进行相对定量 qRT-PCR 分析,使用2-ΔΔ Ct 法(Ct 表示循环数)对结果进行计算并使用 Excel 软件对最终结果进行统计学分析并绘制最终的表达量图表。
表2 实时荧光定量PCR体系
Tab.2 PCR system of qRT-PCR

名称Name体积/μLVolume双蒸水ddH2O 10.5正向引物/(10 μmol/L)Forward primer 0.5反向引物/(10 μmol/L)Reverse primer 0.5 染料法实时荧光定量试剂12.52×TransStart TM Green qPCR Super Mix 互补脱氧核糖核酸cDNA1.0
2 结果与分析
2.1 玉米ZmTOC1a、ZmTOC1b基因克隆
使用引物TOC1a-F/R、TOC1b-F/R, 进行PCR扩增,得到ZmTOC1a的开放阅读框的总长度为1 236 bp, 共编码411个氨基酸;ZmTOC1b的开放阅读框的全长为1 554 bp,共编码 517 个氨基酸(图1)。
2.2 玉米ZmTOC1a、ZmTOC1b蛋白特性
使用ProtParam在线预测ZmTOC1a、ZmTOC1b表达蛋白的信息。结果表明,ZmTOC1a蛋白分子式为C1982H3103N585O647S17,分子质量为46.02 ku;ZmTOC1b蛋白分子式为C2468H3909N735O797S29,分子质量57.56 ku。理论上,ZmTOC1a蛋白亲水性平均系数(GRAVY)为-0.818,脂肪系数(Aliphatic index)是56.47。ZmTOC1b蛋白亲水性平均系数(GRAVY)为-0.600,脂肪系数(Aliphatic index)是 66.94。ZmTOC1a、ZmTOC1b均为不稳定蛋白(稳定系数大于40),其中ZmTOC1a不稳定指数为58.88,ZmTOC1b不稳定指数为51.08 。另外,ZmTOC1a、ZmTOC1b在酵母(Yeast)内半衰期均大于20 h,在大肠杆菌(Escherichia coli)内半衰期均大于10 h。对ZmTOC1a、ZmTOC1b编码的氨基酸序列进行分析,结果表明,ZmTOC1a蛋白肽链带负电荷的残基总数(Asp+Glu)54,带正电荷的残基总数(Arg+Lys)49,预测等电点(pI)为6.05(<7),所以推测ZmTOC1a为酸性蛋白。ZmTOC1b蛋白肽链带负电荷的残基总数(Asp+Glu)65,带正电荷的残基总数(Arg+Lys)60,预测等电点(pI)为6.30(<7),所以推测ZmTOC1b为酸性蛋白。
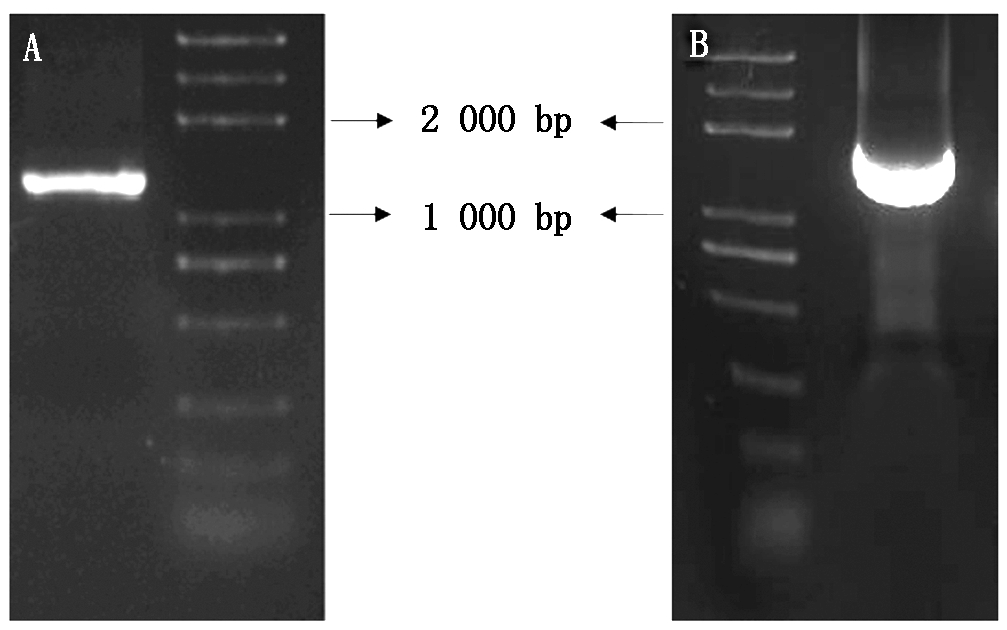
A.ZmTOC1a;B.ZmTOC1b。
图1 ZmTOC1基因克隆的琼脂糖凝胶电泳
Fig.1 Amplification of ZmTOC1 gene and agarose gel electrophoresis
NetPhos 3.1 Server预测结果表明(图2),ZmTOC1a共存在46个磷酸化位点,其中丝氨酸36个,苏氨酸8个,酪氨酸2个;ZmTOC1b共存在53个磷酸化位点, 38个丝氨酸位点,12个苏氨酸位点,3个酪氨酸位点。据此推测ZmTOC1a、ZmTOC1b翻译后的磷酸化修饰可能对蛋白功能产生重要的作用。
2.3 玉米ZmTOC1a、ZmTOC1b系统进化树分析
将玉米TOC1基因序列放在NCBI中Blast得到了小麦、大麦等几种主要农业作物及其他植物中的TOC1基因。几个主要物种中TOC1蛋白编号分别为XP_006647523.1(短穗野生稻,Oryza brachyantha)、BAD38854.1(水稻,Oryza sativa Japonica Group)、XP_002452462.1(高粱,Sorghum bicolor)、XP_004953120.1(谷子,Setaria italica)、AMK48976.1(小麦,Triticum aestivum)、AEW48242.1(大麦,Hordeum vulgare subsp.vulgare)。玉米中ZmTOC1a的编号为GRMZM2G020081, ZmTOC1b的编号为GRMZM2G148453。为了确认玉米ZmTOC1a及ZmTOC1b的系统进化位置,利用Mega 5.0软件制作了TOC1同源基因的系统进化树(图3),并进行了分析。结果发现,高粱中的TOC1基因与玉米TOC1基因亲缘关系最近。
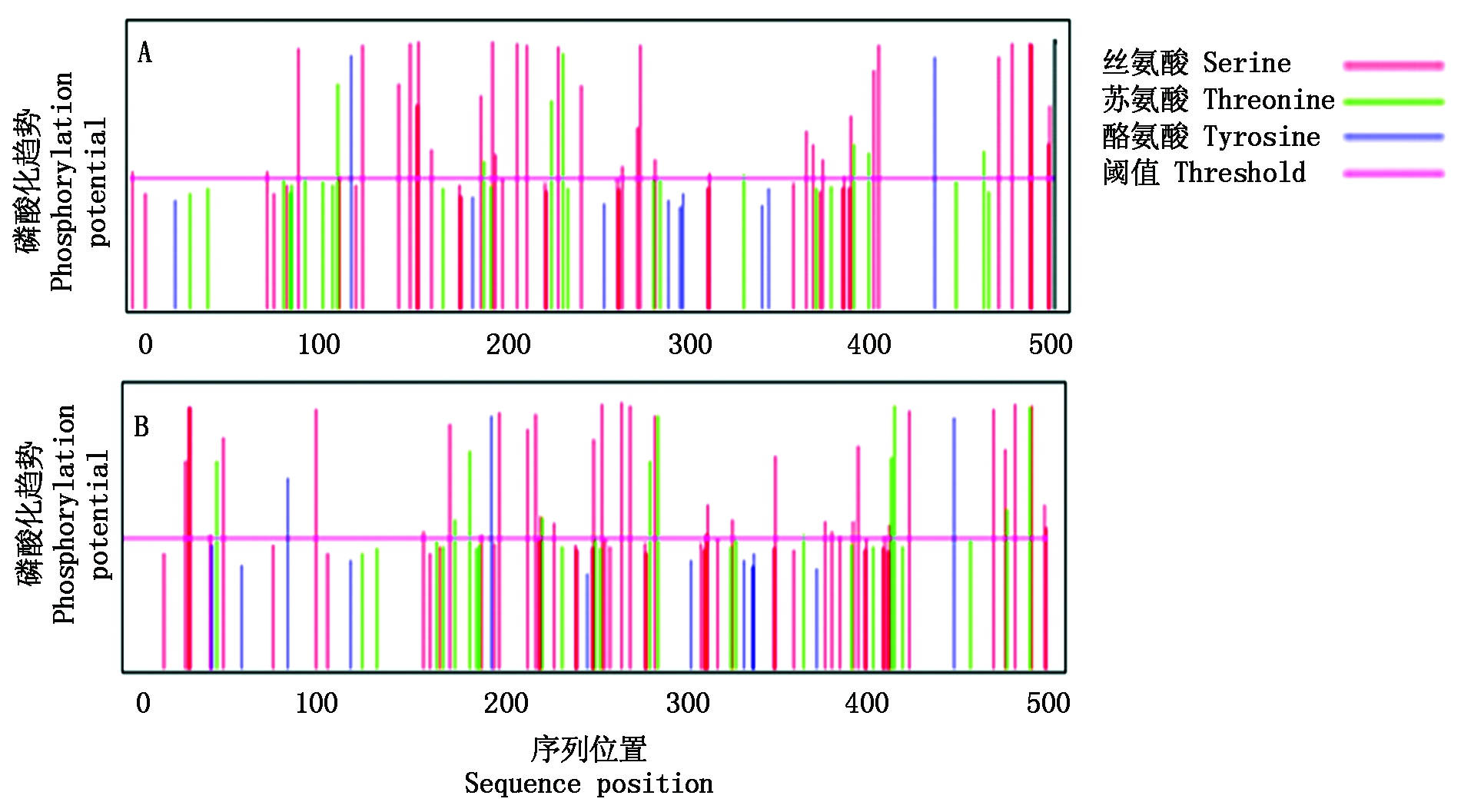
A.ZmTOC1a; B.ZmTOC1b。
图2 ZmTOC1磷酸化位点示意图
Fig.2 Phosphorylation prediction of ZmTOC1
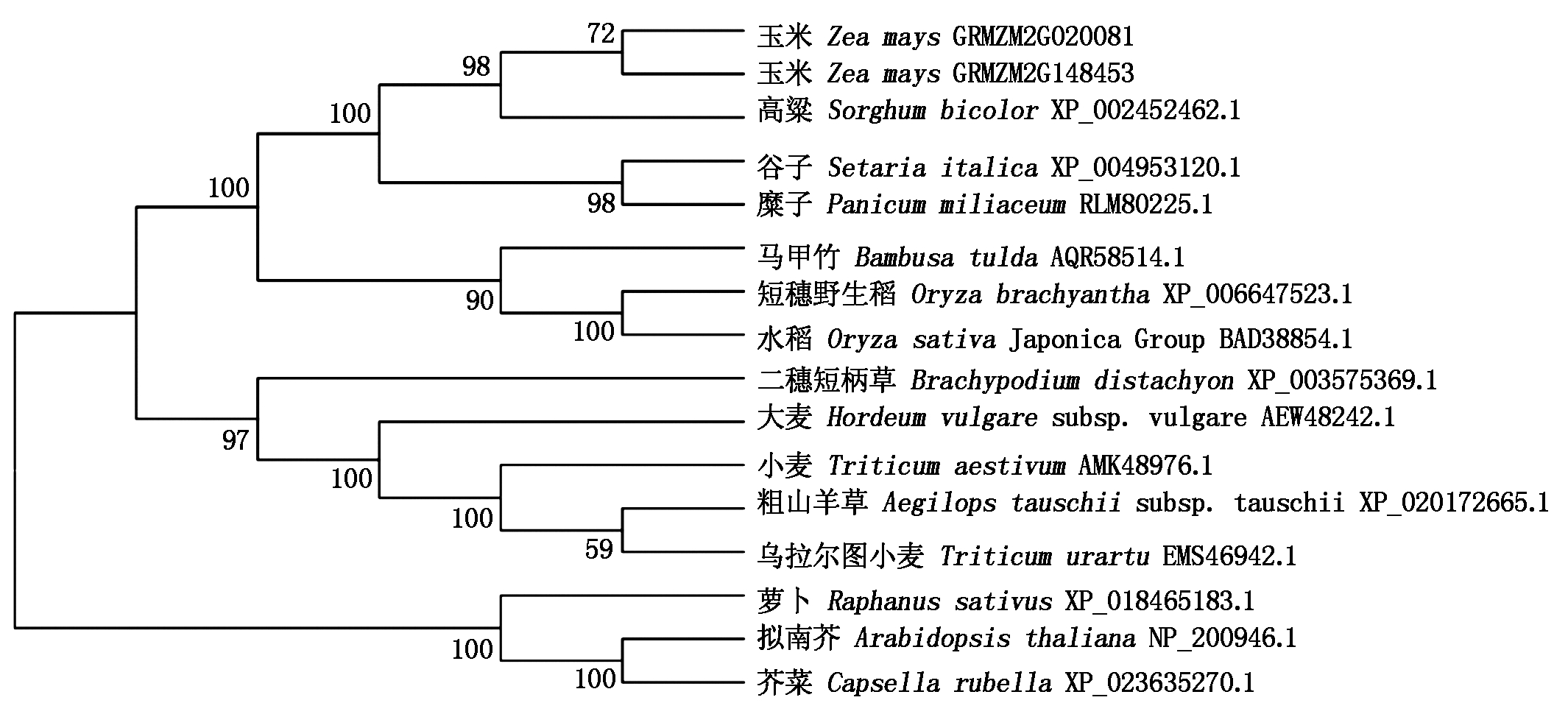
图3 玉米TOC1基因及同源基因系统发育树
Fig.3 Evolutionary tree of ZmTOC1 and other homologous gene
2.4 玉米ZmTOC1a、ZmTOC1b亚细胞定位结果
将测序正确的载体pCAMBIA2300-35S:ZmTOC1a-eGFP,pCAMBIA2300-35S:ZmTOC1b-eGFP以及空载体pCAMBIA2300-35S:eGFP转化农杆菌GV3101并进行菌液检测,将检测为阳性的农杆菌在含有抗性的YEB液体培养基中扩大培养,并注射烟草叶片。pCAMBIA2300-35S:eGFP作为阳性对照,48 h后进行观察。亚细胞定位结果发现,试验对照pCAMBIA2300-35S:eGFP空载体的绿色荧光分布在烟草叶片的细胞核、细胞质、质体以及细胞膜中;而在注射目的载体的烟草叶片的细胞核及细胞质内检测到了绿色荧光蛋白,且细胞核内的绿色荧光信号与细胞核定位蛋白荧光信号相重合,因此,可以初步确定ZmTOC1a、ZmTOC1b主要定位在细胞核以及少量细胞质中(图4)。
2.5 玉米ZmTOC1a、ZmTOC1b在不同组织中表达量分析
为了探究ZmTOC1a、ZmTOC1b的表达模式,使用qRT-PCR检测基因在玉米11个不同组织中的表达量。结果显示,ZmTOC1a及ZmTOC1b在多个组织中表达,但是在不同组织中表达量差异很大。其中2个基因分别在胚根以及胚芽鞘表达量最高。在穗位叶、胚芽鞘、胚根、茎、花丝中高表达,也在雄穗、气生根、根中有表达,但是在花粉和苞叶中无表达(图5)。

图4 ZmTOC1a、ZmTOC1b在烟草叶片中的亚细胞定位
Fig.4 Subcellular localization of ZmTOC1a,ZmTOC1b in tobacco
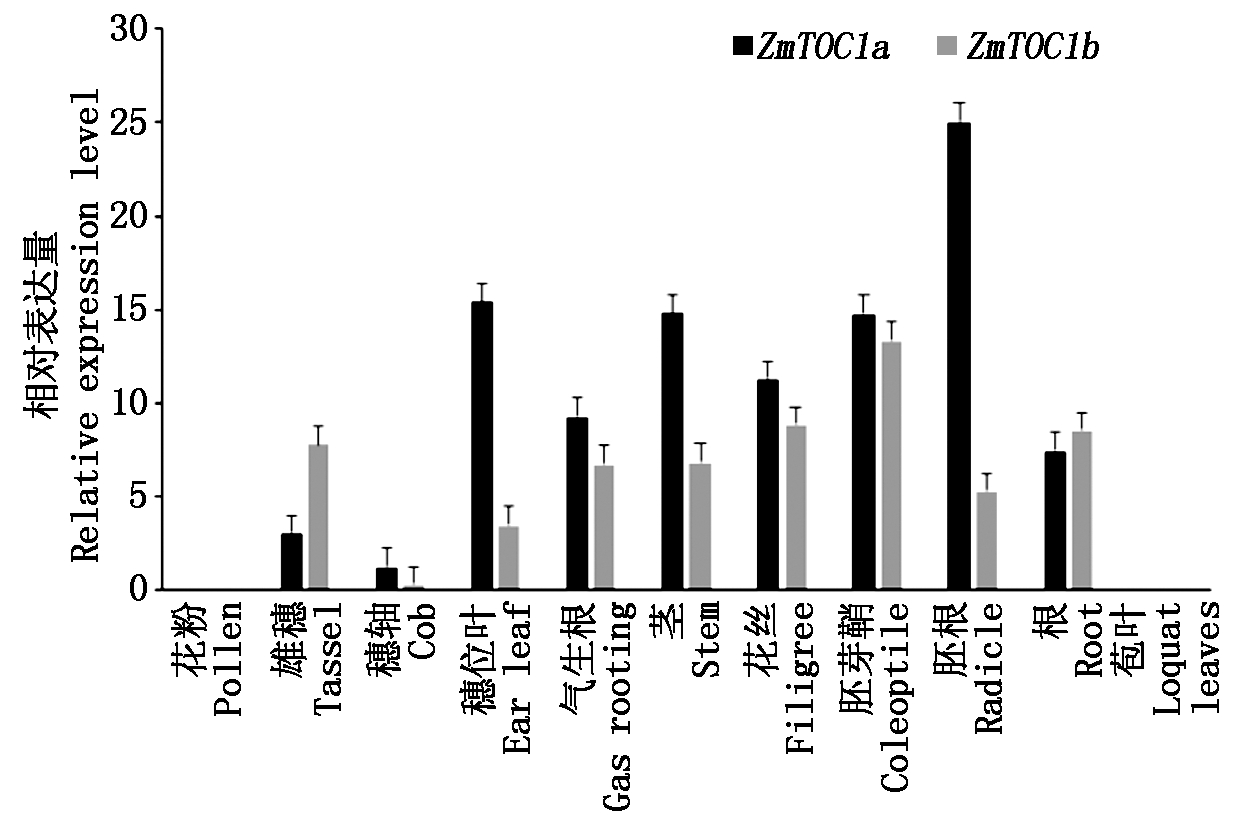
图5 玉米B73 11个不同组织中ZmTOC1a及ZmTOC1b的表达量分析
Fig.5 The expression of ZmTOC1a and ZmTOC1b in eleven different tissues of maize B73
3 结论与讨论
生物钟与开花调节的信号网络紧密相连,这也就使得植物开花调节机制变得更为复杂。拟南芥中央振荡器的主要组成部分AtTOC1会对LHY/CCA1的表达有促进作用,也与ZTL、FKF 1、ABA等基因产生互作进而会对拟南芥的生物钟产生影响。可是TOC1在玉米中的研究并不深入,并且笔者在MAIZE GDB中Blast得到了玉米中的2个同源基因ZmTOC1a、ZmTOC1b,因此研究TOC1在玉米中的作用也就更为必要。
使用PCR扩增得到了这2个基因的CDS序列,而后在线预测了ZmTOC1a、ZmTOC1b表达蛋白的信息。结果发现2个基因表达蛋白均为酸性。并且使用NetPhos 3.1 Server预测的结果表明,ZmTOC1a存在46个潜在磷酸化位点,ZmTOC1b存在53个潜在磷酸化位点。磷酸化是一种常见的可以调控蛋白功能和稳定性的修饰方式,据统计在人体中有500多个磷酸化激酶[34]。在目前已知的所有的翻译后的修饰中磷酸化的种类和数目最多。推测ZmTOC1a、ZmTOC1b翻译后的磷酸化修饰可能对蛋白功能产生重要的作用。而且信号蛋白的磷酸化会造成它所调控下游通路中的其他蛋白磷酸化,形成磷酸化级联反应。另外,系统发育树分析结果表明,ZmTOC1a、 ZmTOC1b在同一个分支中,且与之亲缘关系最近的是高粱中的TOC1基因。推测他们可能含有相类似的功能。有文献表明AtTOC1为转录因子及转录抑制剂[35],而大多数转录因子也是定位在细胞核中的。亚细胞定位结果显示,ZmTOC1a及ZmTOC1b表达的蛋白主要定位在细胞核中,与预期结果一致。基因组织表达结果表明,ZmTOC1a及ZmTOC1b在胚芽鞘、穗位叶、花丝等组织中高表达。这与该基因感受、传递光信号,参与调控开花的功能相关,对ZmTOC1a及ZmTOC1b基因功能的进一步研究有重要的意义。
TOC1除了在光周期通路中发挥作用,也参与到了脱落酸反馈通路中[36]。脱落酸参与调控植物的发育和抗逆过程。Pokhilko等[37]对拟南芥野生型植株进行不同浓度ABA处理,发现高浓度ABA时,AtTOC1表达量上调,生物节律周期延长。Legnaioli等[38]将TOC1-ox、WT和TOC1的RNAi转基因植株进行干旱处理,结果发现,TOC1过量表达的植株对干旱不耐受。TOC1基因在拟南芥中的其他功能有助于加强对ZmTOC1a及ZmTOC1b基因功能的理解,以及后续的功能探究。发掘与光周期敏感相关调控因子,揭示光周期调控途径的分子机制,对于热带以及亚热带玉米优异种质资源的利用,植物花发育过程生物学机制的深入研究以及玉米分子育种均具有重要的理论和实践意义。
[1] 李莹莹,熊炜,宋婷,曹振华,栾维江. 水稻开花时间基因OsDTH10的过表达分析[J]. 中国水稻科学, 2016,30(2):121-126. doi:10.16819/j.1001-7216.2016.5140.
Li Y Y, Xiong W, Song T, Cao Z H, Luan W J. Overexpressing analysis of the flowering time gene OsDTH10 in rice[J]. Chinese Journal of Rice Science,2016,30(2):121-126.
[2] Postma F M, Lundemo S, Agren J.Seed dormancy cycling and mortality differ between two locally adapted populations of Arabidopsis thaliana[J].Ann Bot,2016,117(2):249-56.doi: 10.1093/aob/mcv171.
[3] 许淑娟,种康. “先驱”转录因子LEC1在早期胚胎重置春化状态的机制[J]. 植物学报, 2018,53(1):1-4.doi: 10.11983/CBB17234.
Xu S J,Zhong K. Mechanism of the "Pioneer" transcription factor LEC1 in resetting vernalized state in early embryos[J]. Chinese Bulletin of Botany,2018,53(1):1-4.
[4] Song Y H,Ito S,Imaizumi T.Flowering time regulation: photoperiod and temperature-sensing in leaves[J]. Trends in Plant Sci, 2013,18(10): 575-583.doi: 10.1016/j.tplants.2013.05.003.
[5] Hohenstatt M L,Mikulski P,Komarynets O,Klose C,Kycia I,Jeltsch A,Farrona S, Schubert D.PWWP-DOMAIN INTERACTOR OF POLYCOMBS1 interacts with polycomb-group proteins and histones and regulates arabidopsis flowering and development[J]. Plant Cell,2018,30(1): 117-133. doi: 10.1105/tpc.17.00117.
[6] Xue Y Q,Xing J J,Wan Y L,Lü X Q,Fan L S,Zhang Y D,Song K,Wang L, Wang X H,Deng X,Baluska F,Christie J M, Lin J X.Arabidopsis blue light receptor phototropin 1 undergoes blue light-induced activation in membrane microdomains[J]. Mol Plant, 2018,11(6): 846-859.doi: 10.1016/j.molp.2018.04.003.
[7] Tan J J, Jin M N, Wang J C, Wu F Q, Sheng P K, Cheng Z J, Wang J L, Zheng X M, Chen L P, Wang M, Zhu S S, Guo X P, Zhang X, Liu X M, Wang C M, Wang H Y, Wu C Y, Wan J M. OsCOL10, a CONSTANS-like gene, functions as a flowering time repressor downstream of Ghd7 in rice[J]. Plant Cell Physiology, 2016,57(4): 798-812.doi: 10.1093/pcp/pcw025.
[8] Artlip T S,Wisniewski M E,Arora R,Norelli J L. An apple rootstock overexpressing a peach CBF gene alters growth and flowering in the scion but does not impact cold hardiness or dormancy[J]. Hortic Res,2016,3: 16006.doi: 10.1038/hortres.2016.6.
[9] 李玉舒, 杨炜茹, 程堂仁,王佳,张启翔. 梅花PmSOC1-like基因的克隆与表达分析[J]. 华北农学报, 2016,31(5):78-85. doi: 10.7668/hbnxb.2016.05.012.
Li Y S,Yang W R,Cheng T R, Wang J,Zhang Q X.Molecular cloning and expression analysis of flowering-regulating transcription factor PmSOC1-like gene in Prunus mume[J]. Acta Agriculturae Boreali-Sinica, 2016,31(5):78-85.
[10] Chen W W, Qin Q P, Zheng Y P, Wang C,Wang S,Zhou M B,Zhang C,Cui Y Y. Overexpression of Doritaenopsis hybrid EARLY FLOWERING 4-like4 gene, DhEFL4, postpones flowering in transgenic Arabidopsis[J]. Plant Molecular Biology Reporter, 2016,34(1): 103-117.doi: 10.1007/s11105-015-0899-1.
[11] Pan R, Xu L, Wei Q, Wu C, Tang W L, Oelmüller R, Zhang W Y.Piriformospora indica promotes early flowering in Arabidopsis through regulation of the photoperiod and gibberellin pathways[J]. PLoS One,2017,12(12): e0189791.doi: 10.1371/journal.pone.0189791.
[12] Pelaz S, Ditta G S, Baumann E, Wisman E, Yanofsky M F.B and C floral organ identity functions require SEPALLATA MADS-box genes[J]. Nature, 2000,405(6783): 200-203.doi: 10.1038/35012103.
[13] Kreslavski V D,Kosobryukhov A A,Schmitt F J,Semenova G A,Shirshikova G N, Khudyakova A Y,Allakhverdiev S I.Photochemical activity and the structure of chloroplasts in Arabidopsis thaliana L. mutants deficient in phytochrome A and B[J]. Protoplasma, 2017,254(3): 1283-1293.doi: 10.1007/s00709-016-1020-9.
[14] 杨宗举,闫蕾,宋梅芳,苏亮,孟凡华,李红丹,白建荣,郭林,杨建平.玉米光敏色素A1与A2在各种光处理下的转录表达特性[J]. 作物学报, 2016,42(10):1462-1470. doi: 10.3724/SP.J.1006.2016.01462.
Yang Z J,Yan L,Song M F,Su L,Meng F H,Li H D,Bai J R,Guo L,Yang J P.Transcription characteristics of ZmPHYA1 and ZmPHYA2 under different light treatments in maize[J]. Acta Agronomica Sinica,2016,42(10):1462-1470.
[15] Endo M,Nakamura S,Araki T,Mochizuki N,Nagatani A. Phytochrome B in the mesophyll delays flowering by suppressing FLOWERING LOCUS T expression in Arabidopsis vascular bundles[J]. Plant Cell,2005,17(7): 1941-1952.doi: 10.1105/tpc.105.032342.
[16] Monte E,Alonso J M,Ecker J R,Zhang Y L,Li X,Young J,Austin-Phillips S,Quail P H.Isolation and characterization of phyC mutants in Arabidopsis reveals complex crosstalk between phytochrome signaling pathways[J]. Plant Cell, 2003,15(9): 1962-1980. doi:10.1105/tpc.012971.
[17] Valverde F, Mouradov A, Soppe W, Ravenscroft D, Samach A, Coupland G. Photoreceptor regulation of CONSTANS protein in photoperiodic flowering[J]. Science, 2004,303(5660): 1003-1006.doi: 10.1126/science.1091761.
[18] Jeong S, Clark S E.Photoperiod regulates flower meristem development in Arabidopsis thaliana[J]. Genetics, 2005,169(2): 907-915.doi: 10.1534/genetics.104.033357.
[19] Okajima K. Molecular mechanism of phototropin light signaling[J]. Journal of Plant Research,2016,129(2): 149-157.doi: 10.1007/s10265-016-0783-6.
[20] Lin C T, Yang H Y, Guo H W, Mockler T, Chen J, Cashmore A R.Enhancement of blue-light sensitivity of Arabidopsis seedlings by a blue light receptor cryptochrome 2[J]. Proc Natl Acad Sci USA,1998,95(5): 2686-2690.doi: 10.1073/pnas.95.5.2686.
[21] 杨丽婷,刘森. 生物钟翻译后核心振荡机制[J]. 生命的化学, 2016,36(3):390-396. doi:10.13488/j.smhx.20160321.
Yang L T, Liu S.The research progresses in post-translational oscillation of circadian clock systems[J]. Chemistry of Life,2016,36(3):390-396.
[22] Greenham K,Mcclung C R . Integrating circadian dynamics with physiological processes in plants[J]. Nat Rev Genet,2015,16(10): 598-610. doi:10.1038/nrg3976.
[23] Dunlap J C.Molecular bases for circadian clocks[J]. Cell,1999,96(2): 271-290.doi: 10.1016/S0092-8674(00)80566-8.
[24] 魏琳子, 吴越, 陈强, 刘森. 蓝藻生物钟信号输出途径分子机制的研究进展[J]. 生命科学, 2017,29(8):785-789. doi: 10.13376/j.cbls/2017107.
Wei L Z, Wu Y,Chen Q,Liu S.Progress in the molecular mechanism of the output pathways of the cyanobacterial circadian clock[J]. Chinese Bulletin of Life Sciences,2017,29(8):785-789.
[25] Mizoguchi T, Wright L, Fujiwara S, Cremer F, Lee K, Onouchi H, Mouradov A, Fowler S, Kamada H, Putterill J, Coupland G. Distinct roles of GIGANTEA in promoting flowering and regulating circadian rhythms in Arabidopsis[J]. Plant Cell, 2005,17(8): 2255-2270. doi:10.1105/tpc.105.033464.
[26] Imaizumi T,Schultz T F,Harmon F G,Ho L A,Kay S A. FKF1 F-box protein mediates cyclic degradation of a repressor of CONSTANS in Arabidopsis[J]. Science,2005,309(5732): 293-297.doi: 10.1126/science.1110586.
[27] Hayama R, Coupland G.The molecular basis of diversity in the photoperiodic flowering responses of Arabidopsis and rice[J]. Plant Physiol,2004,135(2):677-684.doi: 10.1104/pp.104.042614.
[28] Matsushika A, Makino S, Kojima M, Mizuno T. Circadian waves of expression of the APRR1/TOC1 family of pseudo-response regulators in Arabidopsis thaliana: insight into the plant circadian clock[J]. Plant Cell Physiol, 2000,41(9): 1002-1012. doi:10.1093/pcp/pcd043.
[29] Wenkel S, Turck F, Singer K, Gissot L, Le Gourrierec J, Samach A, Coupland G. CONSTANS and the CCAAT Box binding complex share a functionally important domain and interact to regulate flowering of Arabidopsis[J]. The Plant Cell Online,2006,18(11): 2971-2984.doi: 10.1105/tpc.106.043299.
[30] Alabadí D, Oyama T, Yanovsky M J, Harmon F G, Más P, Kay S A. Reciprocal regulation between TOC1 and LHY/CCA1 within the Arabidopsis circadian clock[J].Science, 2001, 293(5531):880-883.doi: 10.1126/science.1061320.
[31] Nakamichi N, Kita M, Ito S, Sato E, Yamashino T, Mizuno T.The Arabidopsis pseudo-response regulators, PRR5 and PRR7, coordinately play essential roles for circadian clock function[J]. Plant Cell Physiol, 2005,46(4): 609-619. doi: 10.1093/pcp/pci061.
[32] Locke J C W, Kozma-Bognár L, Gould P D, Fehér B, Kevei É, Nagy F, Turner M S, Hall A, Millar A J. Experimental validation of a predicted feedback loop in the multi-oscillator clock of Arabidopsis thaliana[J]. Molecular Systems Biology,2006,2: 59.doi: 10.1038/msb4100102.
[33] Huang W, Pérez-García P, Pokhilko A, Millar A J, Antoshechkin I, Riechmann J L, Mas P. Mapping the core of the Arabidopsis circadian clock defines the network structure of the oscillator [J].Science,2012,336(6077):75-79. doi: 10.1126/science.1219075.
[34] Miller C J,Turk B E. Homing in: mechanisms of substrate targeting by protein kinases [J]. Trends Biochem Sci, 2018,43(5): 380-394.doi: 10.1016/j.tibs.2018.02.009.
[35] Para A,Farré E M,Imaizumi T,Pruneda-Paz J L,Harmon F G,Kay S A. PRR3 is a vascular regulator of TOC1 stability in the Arabidopsis circadian clock[J]. Plant Cell, 2007, 19(11):3462-3473.doi: 10.1105/tpc.107.054775.
[36] Fung-Uceda J, Lee K, Seo P J, Polyn S, De Veylder L, Mas P. The Circadian clock sets the time of DNA replication licensing to regulate growth in Arabidopsis[J]. Dev Cell, 2018, 45(1): 101-113.e4.doi: 10.1016/j.devcel.2018.02.022.
[37] Pokhilko A,Mas P,Millar A J.Modelling the widespread effects of TOC1 signalling on the plant circadian clock and its outputs [J]. BMC Systems Biology, 2013,7(1): 23.doi: 10.1186/1752-0509-7-23.
[38] Legnaioli T,Cuevas J,Mas P. TOC1 functions as a molecular switch connecting the circadian clock with plant responses to drought [J]. The EMBO Journal,2009,28(23): 3745-3757.doi: 10.1038/emboj.2009.297.