株高是农作物的重要性状,对作物的抗倒伏性、产量有重要影响。著名的“绿色革命”就是将半矮秆性状引入水稻育种,选育出矮化耐密植的新品种,使产量得到提升[1]。明确株高的分子遗传基础有利于利用分子育种手段选育理想株型的新品种,使作物的生态适应能力、水分利用效率、机械收割能力、产量潜能等获得综合改善。
株高受遗传网络调控,目前发现的矮秆表型多是由赤霉素、油菜素生物合成或者信号识别途径产生缺陷造成的[2-4]。此外,一些和细胞分裂、伸长相关的基因突变也会导致植株矮化,如水稻中编码F-box域基因D3发生突变会导致矮化表型,编码微管相关蛋白的dgl1基因缺陷也会导致植株矮化[5-6]。
谷子(Setaria italica)作为我国的传统作物,营养组成均衡,其较小的基因组(约510 Mb)、严格自花授粉、高光合等特点可以作为一个理想C4作物以及能源作物的模型[7-8]。株高与谷子倒数第一、第二、第三节倒伏指数存在一定负相关,而与穗长、穗粗、穗粒质量、穗码数4个产量相关性状呈显著正相关[9]。近10年基于双亲杂交分离群体的数量性状位点(QTL)定位方法已经用于谷子株高遗传基础的解析[10-13]。虽然通过上述研究定位了一些株高相关QTL位点,但是由于双亲的等位变异有限,检测到的QTL位点也存在一定局限性,而且构建分离群体也较为耗时耗力。
全基因组关联分析以自然群体为研究对象,利用分布于整个基因组的高密度DNA标记来发掘与目标性状关联的遗传位点,相比传统的基于双亲杂交分离群体的定位方法省去了构建分离群体的过程,而且可以检测到更多的目标性状关联位点。目前全基因组关联分析已经广泛应用于植物数量性状定位研究[14-17]。谷子基因组测序已经完成[18-19],但是有关全基因组关联分析的研究较少。Gupta等[20]调查了184份谷子材料的产量相关性状,用50对简单序列重复(SSR)标记进行了性状与标记的关联分析,发现有2个SSR标记与旗叶宽、籽粒产量有较强关联,且位于5号染色体。Jia等[21]对916种不同的谷子资源进行重测序,鉴定了258万个单核苷酸多态性(SNP)位点,并使用80万个高质量的SNP标记来构建谷子基因组的单倍型图谱,与47个性状进行了关联分析,获得了多个地理环境稳定表达的QTL位点,这是首次报道的基于重测序的谷子全基因组关联分析方面的研究。虽然目前已有较多关于谷子株高QTL定位的报道,但是针对不同光周期条件下谷子株高定位方面的研究还未开展。本研究在海南种植区短日照、洛阳种植区中日照、吉林种植区长日照3个不同光周期条件下,调查了98份来自国内以及国外的不同生态区谷子材料株高,并对这98份谷子材料进行重测序,利用测序数据开发海量SNP位点,开展位点与株高的全基因组关联分析,发掘与株高性状紧密连锁的分子标记,并且对关联位点区域存在的候选基因进行预测,以期为进一步克隆控制株高的基因奠定基础。
1 材料和方法
1.1 试验材料及表型鉴定
98份谷子材料包括了89份国内材料,来自河南、河北、山东、山西、陕西、甘肃、宁夏、内蒙古、吉林、辽宁、黑龙江、新疆、湖南、四川等地区;9份国外材料,来自美国、法国、日本、朝鲜、南非及国际半干旱研究所,材料信息见表1。
2015年5月中旬至10月中旬将98份谷子材料分别种植于河南科技大学开元校区试验田和吉林市农业科学院试验田,2015年11月初至2016年2月末将98份谷子材料种植于海南省乐东县九所镇河北农林科学院谷子研究所试验田。每份谷子材料种植2行,行长2 m、行距50 cm、株距3~5 cm。按当地常规管理,每份材料于成熟期选取行中部谷子(共10株)测量株高,取平均值作为最终测量值。用SPSS 19.0软件绘制直方图及计算株高的广义遗传力。
1.2 谷子材料重测序
98份谷子材料长到三叶期时采集幼嫩叶片,利用十六烷基三甲基溴化铵(CTAB)法提取基因组DNA,提取的DNA满足OD260/280=1.8~2.0,且没有明显降解,送上海美吉生物技术公司进行重测序。
表1 98份谷子材料信息
Tab.1 Information of 98 foxtail millet varieties

编号Number品种Variety 来源地Origin region编号Number品种Variety来源地Origin region编号Number品种Variety 来源地Origin region16-1郑谷2河南16-57衡谷9号河北16-126小苗谷宁夏16-3豫谷6河南16-60毛谷2号河北16-127大青苗鱼刺甘肃16-4郑06-6河南16-62南育3号河北16-128尕红谷甘肃16-6豫谷3河南16-66矮88河北16-133蒜皮白内蒙古16-7郑05-2河南16-68小青谷河北16-136金香玉内蒙古16-8郑州12河南16-69黄毛谷河北16-137朝鲜谷子朝鲜16-9郑315河南16-70齐头白河北16-139ISE-430美国16-10郑8041河南16-71老绳头河北16-140大王国日本16-11安5424 河南16-72龙爪谷河北16-144法谷28-81法国16-13豫谷15河南16-73小白苗河北16-146ISE770国际半干旱研究所16-14豫谷11河南16-74黑色腰河北16-148岛原日本16-16豫谷16河南16-84钱串子山东16-149六十日日本16-18豫谷13号河南16-87早谷 山东16-152Red manna南非16-19豫谷18号河南16-88晋汾13山西16-154Ise-455美国16-20安04-5014河南16-89晋谷35号山西16-1568322-14未知16-21豫谷2号河南16-90汾选5号山西16-162芝麻粟未知16-23毛毛亮河南16-97大同黄山西16-163大头糯湖南16-25冀谷27河北16-99白谷9号吉林16-164乐山白糯四川16-28金谷1号河北16-100公谷66号吉林16-17215HN-206河北16-29冀特5号河北16-101公谷69号吉林16-176六十天还家吉林16-30冀谷24河北16-102公矮3号吉林16-179小明谷子甘肃16-34冀谷31河北16-104白秆白沙吉林16-182茄谷甘肃16-37冀谷15河北16-105糟皮一把奇黑龙江16-183然谷陕西16-39冀谷26号河北16-108安丰黑龙江16-184铁谷4号辽宁16-41谷丰1号河北16-109老来变 黑龙江16-186叩根吉林16-42532未知16-11106-766北京16-188铁7924辽宁16-43张农8河北16-112小早谷北京16-189小金苗吉林16-44坝谷6河北16-115延谷11号 陕西16-190嫩选十号黑龙江16-45坝谷81河北16-120谷子新疆16-192辽谷1号辽宁16-46坝谷210河北16-121谷子新疆16-196黑谷子河北16-48坝谷139河北16-122谷上谷 甘肃16-198赤谷6号内蒙古16-51毛毛谷河北16-123陇谷11号甘肃16-200二不黄山西16-53大九根齐河北16-124塞外香谷子宁夏
1.3 SNP标记的开发
采用GATK软件进行SNP、InDel(插入、缺失)信息的鉴定,并用Samtools提供的vcfutils工具对检测到的SNP、InDel位点进行过滤,再利用ANNOVAR软件以及参考基因组的基因组特征描述(gff)信息对SNP位点注释。
1.4 群体结构分析
采用主成分分析法确定98份谷子材料的群体结构,选择均匀分布谷子9条染色体的3 600个SNP位点,用GCTA软件进行主成分分析,得到样品的群体聚类情况。
1.5 连锁不平衡(LD)分析
用连锁不平衡系数(r2)衡量染色体上SNP位点间的连锁不平衡水平,r2=0时则说明该群体处于连锁平衡或者无LD,r2越大,SNP位点间距离越接近;一般r2大于或等于0.8,则视为两位点属于连锁不平衡。用r2衰退到一半时对应的位点间的距离作为连锁不平衡衰减距离,用VCFtools软件来计算连锁不平衡的衰减的数值,并以非线性回归模型绘制出r2随着SNP位点间物理距离的增加而衰减的趋势图。
1.6 株高与SNP标记的关联分析及候选基因的筛选
以群体结构作为固定效应,个体亲缘关系作为随机效应,校正群体结构和个体间亲缘关系的影响,采用Tassel Version 5软件中的混合线性模型(Mixed Linear Model, MLM)进行性状与标记间的关联分析,当SNP位点的P<0.000 1时视该SNP位点与性状关联。把与株高关联的SNP标记定位到谷子的基因组上,并确定相对应的置信区域,在关联SNP标记连锁不平衡衰减距离的区域范围内,搜寻与性状相关的候选基因。对筛选出的基因与基因本体联合会(GO)、非冗余(NR)、京都基因与基因组百科全书(KEGG)等功能数据库比对,实现对候选基因的注释。
2 结果与分析
2.1 株高性状的表型分析
在3个不同光周期条件下,谷子株高的表型变异和频率分布分别见表2和图1。谷子株高性状均表现较大的变异范围,其中海南种植区短日照条件下株高变异在58.5~152.4 cm,平均株高为108.1 cm;洛阳种植区中日照条件下株高变异在70.7~155.4 cm,平均株高为110.5 cm;吉林种植区长日照条件下变异在70.7~169.3 cm,平均株高127.6 cm。利用约束最大似然法估算出株高的广义遗传力为0.501。随着日照时间的增长,谷子的株高也呈现增高趋势,海南、洛阳2个种植区光周期间株高差异不显著,但是海南、洛阳和吉林3个种植区光周期间株高存在显著差异(P<0.01)。
表2 不同种植区光周期条件下株高的表型变异
Tab.2 Phenotypic variation of plant height under photoperiod conditions of different cultivation regions
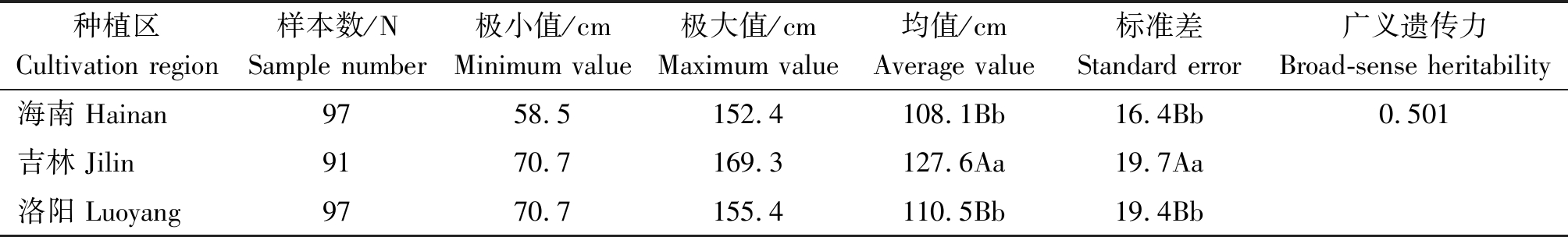
种植区Cultivation region 样本数/NSample number极小值/cmMinimum value极大值/cmMaximum value均值/cmAverage value标准差Standard error广义遗传力Broad-sense heritability海南 Hainan9758.5152.4108.1Bb16.4Bb0.501吉林 Jilin9170.7169.3127.6Aa19.7Aa洛阳 Luoyang9770.7155.4110.5Bb19.4Bb
注:不同大写字母代表极显著(P<0.01),不同小写字母代表显著(P<0.05),相同小写字母代表不显著(P>0.05)。
Note: The different capital letters represent extremely significant (P<0.01), the different lower-case letters represent significant (P<0.05), the same lower-case letter represents no significant (P>0.05).
从图1可以看出,海南种植区短日照条件下株高集中分布在100~120 cm,这个区间往两侧移动呈现递减趋势,基本符合正态分布规律;洛阳种植区中日照条件下株高同样集中分布在100~120 cm,这个区间往两侧移动也表现出一定的递减趋势,总体上接近正态分布规律;吉林种植区长日照条件下株高分布表现一定双峰模式,在120 cm附近有一个峰,另一个峰在140 cm附近。株高符合多基因控制的数量性状遗传模式。
2.2 98份谷子材料基因组重测序结果
98份谷子材料重测序获得序列与豫谷1号参照基因组比对,基因组覆盖率在86.10%~97.55%,测序深度在6.407 9~13.327 5倍,平均测序深度为6.8倍,表明基因组被覆盖地较均匀,测序的随机性较好。利用基因组重测序检测到900多万SNP位点,经过滤获得4 482 208个高质量的SNP标记,这些SNP标记有4 037 579个在基因间隔区,占所有标记总数的90%。剩余的SNP标记主要分布在内含子区域、转录起始位点的上游区域、基因下游区域,其相应数目为122 698,102 355,81 070。在外显子区域内的标记可以分为5种情况:非同义变异、终止密码子获得变异、终止密码子丢失变异、同义变异、未知变异,其数目分别为31 477,459,66,25 381,332个,共计57 715个。在非编码RNA区域内的标记也可根据位置可分为5种:非编码RNA外显子区域、非编码RNA内含子区域、非编码RNA 3′UTR、非编码RNA 5′UTR、非编码RNA剪接位点区域,其具体的SNP标记数目分别为14 371,28 855,39,21,101,共计为43 387(表3)。
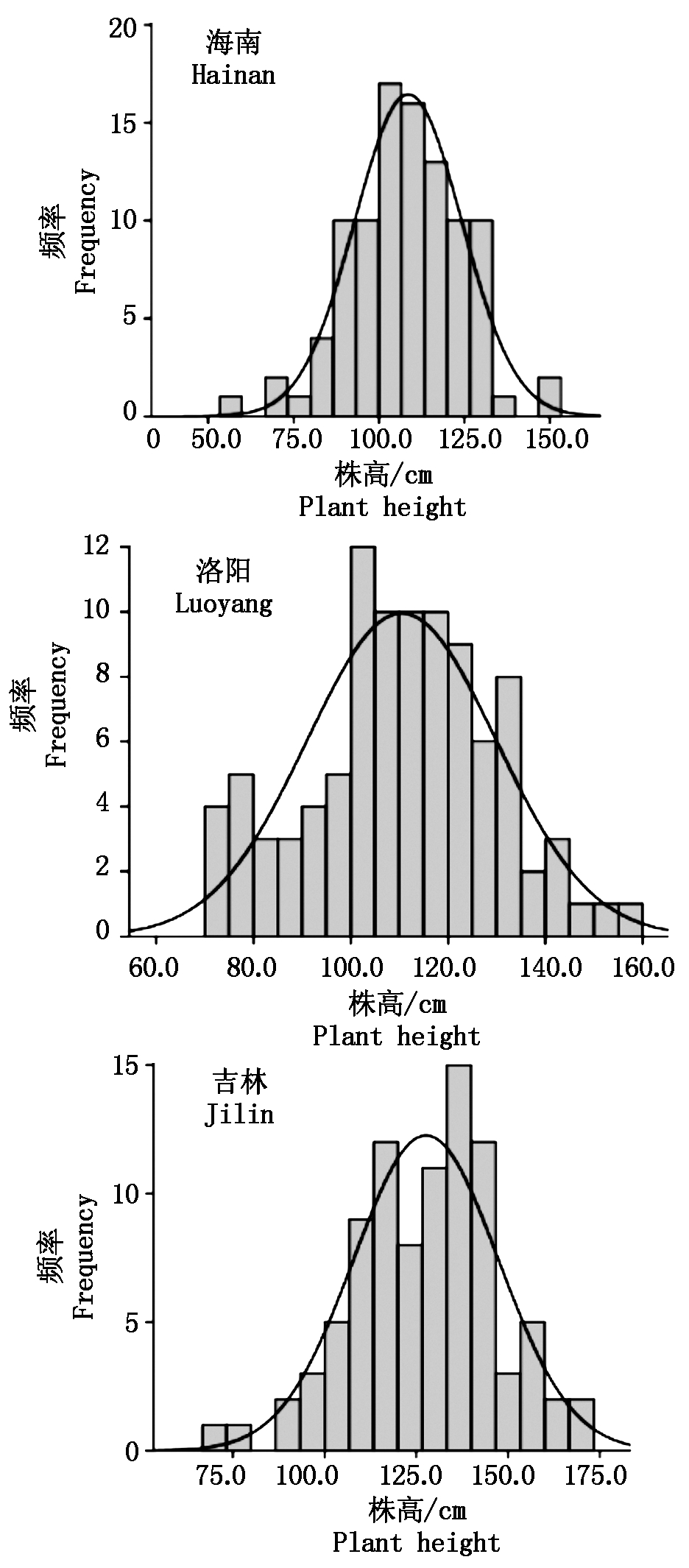
图1 株高在不同种植区光周期条件下的频率分布
Fig.1 Plant height frequency under photoperiod conditions of different cultivation regions
表3 SNP类型统计
Tab.3 SNP types statistics

类型Type SNP数目 SNP number转录起始位点上游区域 Upstream region of transcription initiation site102 355外显子 Exon 非同义变异 Nonsynonymous mutation 31 477终止密码子获得变异 Termination codon gain mutation459终止密码子丢失变异 Termination codon loss mutation66同义变异 Synonymous mutation25 381未知变异 Unknown mutation332内含子 Intron122 698剪接位点 Splicing site222基因下游 Downstream of gene81 070一个基因上游同时另一个基因下游 Upstream of one gene and downstream of another gene11 647基因间隔区 Intergenic region4 037 5793′非翻译区 3′UTR 14 9775′非翻译区 5′UTR10 540一个基因3′非翻译区同时另一个基因5′非翻译区 3′UTR of one gene and 5′UTR of another gene18非编码RNA外显子区域 Exonic region of no-coding RNA14 371非编码RNA内含子区域 Intronic region of no-coding RNA28 855非编码RNA的3′非翻译区 3′UTR of no-coding RNA39非编码RNA的剪接位点 Splicing site of no-coding RNA101非编码RNA的5′非翻译区 5′UTR of no-coding RNA21总数 Total4 482 208
2.3 群体结构分析
利用所开发的3 000个SNP标记,通过 GCTA 软件进行主成分分析,得到98个谷子材料的主要成分聚类情况。如图2所示,98个谷子材料聚为三维,pca1、pca2 、pca3 分别为3个主成分,说明最佳分群数为3个。
2.4 LD分析
从图3可以看出,当r2值降低到最高值的一半时,SNP标记间距离为47.5 kb,因此,本研究中谷子基因组LD衰减距离为47.5 kb。较小的LD衰退距离有利于缩小后续关联分析候选区域,提高关联结果的精确度。
2.5 谷子株高的全基因组关联分析
将重测序开发的SNP标记与3个不同光周期条件下的株高数据进行关联分析,共检测到10 703个SNP位点与株高性状显著关联(P<0.000 1),其中海南种植区短日照条件与株高关联的SNP位点数量最多,为10 608个,且多数集中在1号染色体上(10 458个),其次是洛阳种植区中日照条件,关联的SNP位点数量为78个,吉林种植区长日照条件关联的SNP位点数量最少,为17个(图4)。在海南种植区短日照条件下关联的SNP位点在谷子的9条染色体上均有分布,在洛阳种植区中日照条件下关联的SNP位点分布在除3号、8号以外的其他染色体上,在吉林种植区长日照条件下关联的SNP位点分布在除2号、9号以外的其他染色体上。其中1号染色体上的3个标记(SNP13861443、SNP14872616、SNP18601830)在海南、洛阳2个种植区光周期条件下同时检测到,而在吉林种植区长日照条件下未检测到,这3个株高关联SNP位点分别位于1号染色体的13 861 443,14 872 616,18 601 830 bp处。其余SNP标记不能在2个或者3个光周期条件下同时检测到。因此,推定1号染色体可能存在中、短日照条件表达的控制株高的QTL位点,而在吉林种植区长日照条件下也在1号染色体检测到3个与株高关联的SNP位点(SNP8797106、SNP8797129、SNP9880380)(表4),说明谷子1号染色体可能存在对光周期具有不同反应的控制株高的QTL位点。
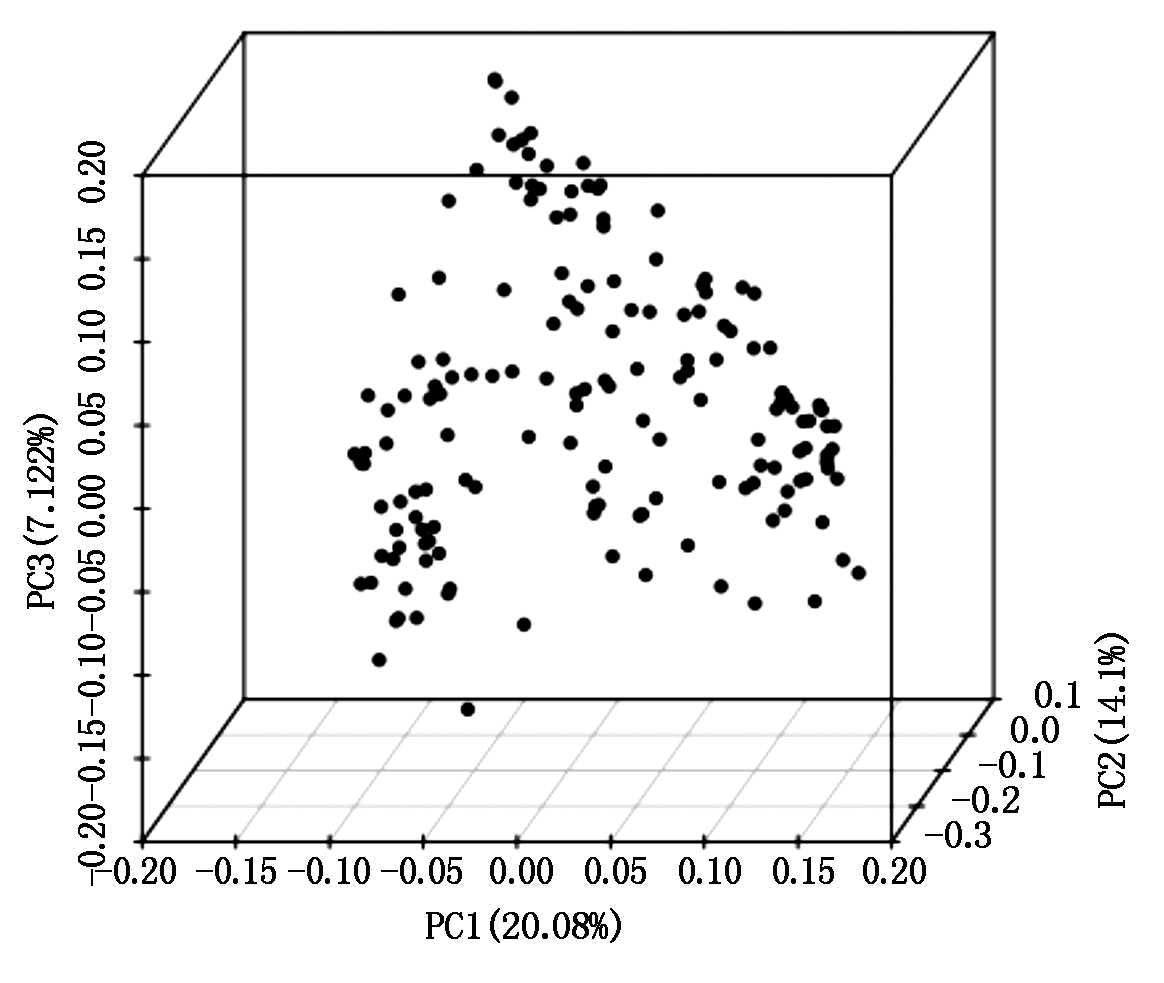
图2 98个谷子材料PCA聚类
Fig.2 PCA cluster diagram of 98 foxtail millet varieties
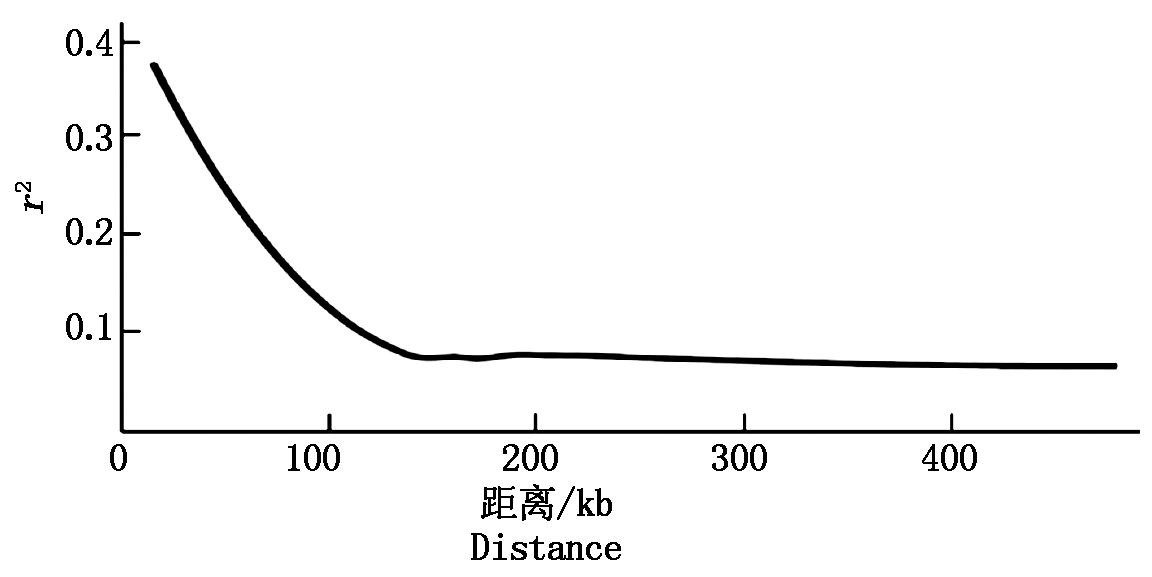
图3 LD衰减曲线
Fig.3 The attenuation curve of LD
在1号染色体与株高关联的SNP位点两侧100 kb范围内搜索候选基因,在海南种植区搜索到10个候选基因,这些基因功能主要包括维生素E生物合成、细胞膜组成、代谢过程、DNA结合以及一些功能未知基因。在洛阳种植区区搜索到4个候选基因,其中2个基因功能为代谢、DNA结合,还有2个功能未知基因。在吉林种植区搜索到1个候选基因,功能为线粒体组成。在海南、洛阳种植区共同搜索到的候选基因有3个,包括1个功能未知基因、1个成蛋白基因(DRF)、一个DNA结合蛋白基因(ESCAROLA),只有成蛋白基因外显子区检测到SNP位点(SNP14876527),其余2个候选基因未发现SNP位点(表5)。因此,推测成蛋白基因突变可能导致了海南、洛阳种植区谷子株高的变异,该基因为株高主要候选基因。
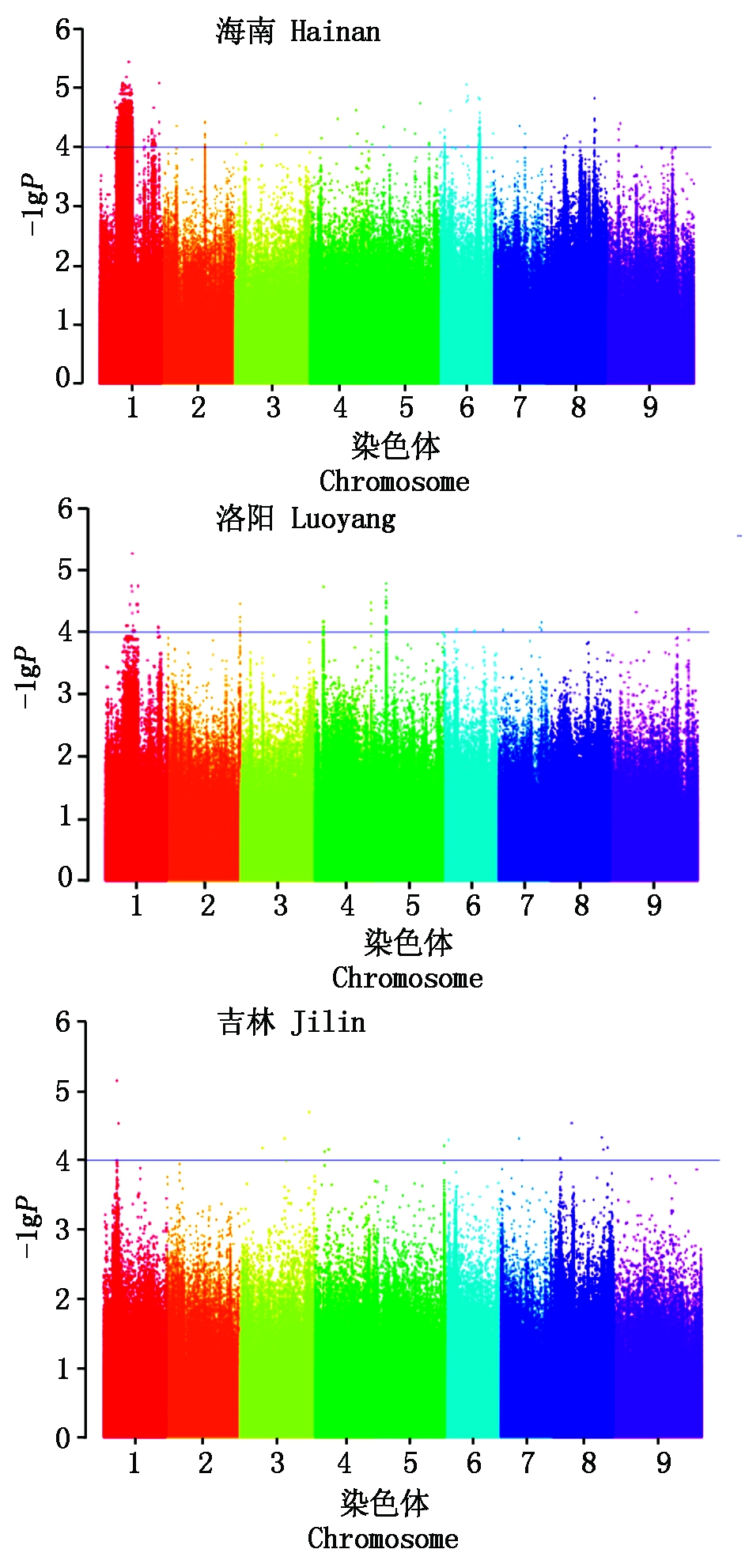
图4 海南、洛阳、吉林3个种植区株高的曼哈顿散点图
Fig.4 Manhattan plot of plant height in Hainan, Luoyang and Jilin cultivation regions
表4 谷子材料1号染色体与株高关联的SNPs位点
Tab.4 The SNPs associated with plant height on Chr.1 of foxtail millet
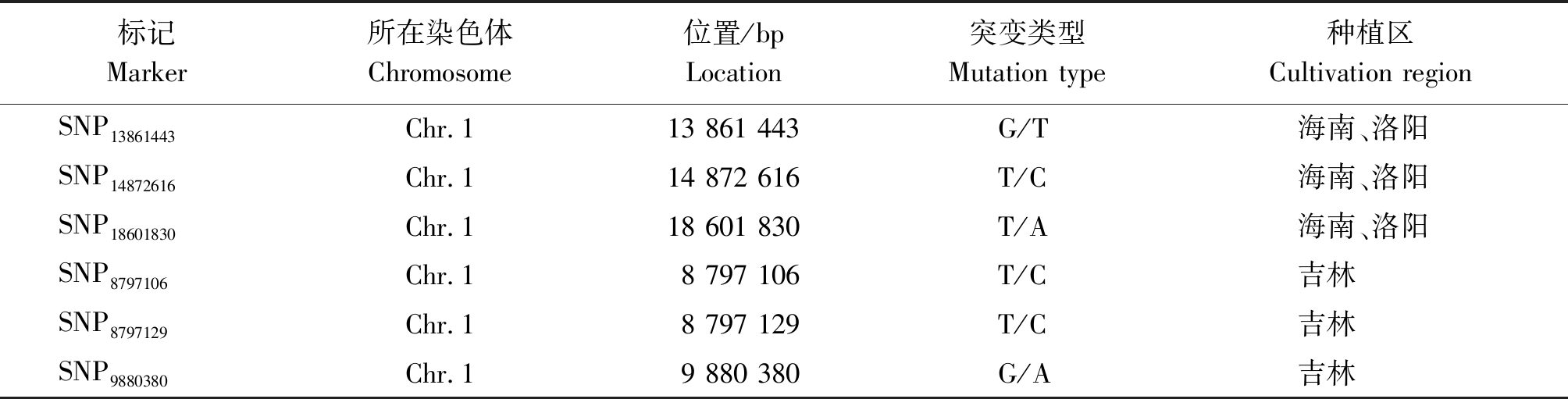
标记Marker所在染色体Chromosome位置/bpLocation突变类型Mutation type种植区Cultivation regionSNP13861443Chr.113 861 443G/T海南、洛阳SNP14872616Chr.114 872 616T/C海南、洛阳SNP18601830Chr.118 601 830T/A海南、洛阳SNP8797106Chr.18 797 106T/C吉林SNP8797129Chr.18 797 129T/C吉林SNP9880380Chr.19 880 380G/A吉林
表5 海南、洛阳种植区共同检测到的株高关联候选基因
Tab.5 The candidate genes associated with plant height in Hainan and Luoyang cultivation regions

基因名称Gene nameSNP标记SNP markerSNP位置/bpSNP positionSNP突变类型SNP mutation type同源基因名称Homologous gene name推定功能Putative functionLOC101771936功能未知基因未知LOC101783280SNP1487652714 876 527C/TDRF形态发生、细胞极性LOC101777891ESCAROLADNA结合
3 结论与讨论
本研究在海南种植区短日照、洛阳种植区中日照2个光周期条件下获得3个与谷子株高关联的SNP位点,这3个位点都位于1号染色体上。王晓宇等[10]在谷子2号染色体定位到2个控制株高的QTL位点;赵美丞[11]利用图位克隆法在谷子9号染色体获得一个半显性矮秆基因SiDw1;Wang等[12]、Zhang等[13]利用高密度的SNP标记将谷子株高控制位点定位于1、4、5、6、7、9号染色体上。前人在1号染色体上将株高QTL位点定位在184.84,144.32 cM 2个峰值点附近区域[12-13],本研究在1号染色体获得的3个株高关联SNP位点位于13 861 443,14 872 616,18 601 830 bp,尽管本研究关联位点位置使用的是物理距离,而前人报道的关联位点使用的是遗传距离,Wang等[12]报道的控制株高的QTL位点位于1号染色体的末端,本研究3个关联位点位于1号染色体的中部靠前,初步判断本研究获得的QTL位点与前人报道的不在同一个区间内。Zhang等[13]在宣化种植区长日照条件下检测到1号染色体144.32 cM位置存在株高QTL位点,而在海南三亚种植区短日照条件下并未在1号染色体检测到控制株高的QTL位点,且长日照检测到的QTL位点位于1号染色体的后部,据此推断本研究在1号染色体关联到的株高控制位点应该为在短日照、中日照条件特定表达的新的QTL位点。
本研究在海南、洛阳种植区2个光周期条件关联到3个候选基因,但是2个候选基因在基因内部未检测到SNP位点,只有一个成蛋白类基因(LOC101783280)在外显子区检测到一个SNP位点(SNP14876527),推测该基因突变可能和株高相关。目前,在水稻等作物报道的株高相关基因主要包括参与赤霉素生物合成及信号传递、油菜内酯生物合成、独角金内酯生物合成3类基因[22]。成蛋白或者形成素(Formin)是一类多结构域蛋白,具有保守的串联FH1、FH2结构域,对微丝的聚合有重要作用。植物中的成蛋白分为2个亚家族,第一亚家族在氨基端具有跨膜域和信号肽,第二亚家族具有PTEN结构域。植物成蛋白基因也具有微丝成核、成束、聚合等作用,在细胞分裂、极性生长中具有重要作用[23-24]。已有研究表明,水稻成蛋白基因OsFH5突变会导致植株矮化、顶部节尖弯曲等表型[25],本研究在海南、洛阳2个种植区光周期条件下都检测到与株高关联的成蛋白类基因,该基因突变是否是导致谷子植株矮化的主要原因仍需进一步做基因表达分析及转基因验证。
在海南、洛阳、吉林3个种植区不同光周期条件下调查了98份谷子材料的株高性状,3个种植区株高变异幅度在58.5~169.3 cm,广义遗传力为0.501。对98份谷子材料进行重测序开展SNP标记与株高的全基因组关联分析,获得10 703个与株高关联的SNP位点,其中只有1号染色体的3个SNP(SNP13861443、SNP14872616、SNP18601830)在海南、洛阳2个种植区光周期条件下能被稳定检测到,在3个关联SNP候选区域检测到3个候选基因,只有推定的成蛋白基因(LOC101783280)在外显子区检测到一个SNP位点(SNP14876527),因此,该成蛋白基因可能是海南短日照、洛阳中日照光周期条件下导致谷子株高变异的主要候选基因。
[1] Jiang H, Guo L B, Qian Q. Recent progress on rice genetics in China[J]. Journal of Integrative Plant Biology, 2007, 49(6): 776-790. doi: 10.1111/j.1744-7909.2007.00492.x.
[2] Plackett A R G, Powers S J,Fernandez-Garcia N, Urbanova T, Takebayashi Y, Jikumaru Y, Benlloch R, Nilsson O, Ruiz-Rivero O, Phillips A L, Wilson Z A, Thomas S G, Hedden P. Analysis of the developmental roles of the Arabidopsis gibberellin 20-oxidases demonstrates that GA20ox1,-2, and-3 are the dominant paralogs[J]. The Plant Cell,2012,24(3):941-960. doi: 10.1105/tpc.111.095109.
[3] Teng F, Zhai L H, Liu R X, Bai W, Wang L Q, Huo D G, Tao Y S, Zheng Y L, Zhang Z X. ZmGA3ox2, a candidate gene for a major QTL, qPH3.1, for plant height in maize[J]. Plant Journal, 2013, 73(3): 405-416. doi: 10.1111/tpj.12038.
[4] Yang C J, Zhang C, Lu Y N, Jin J Q, Wang X L. The mechanisms of brassinosteroids′ action:From signal transduction to plant development[J]. Molecular Plant, 2011, 4(4): 588-600. doi: 10.1093/mp/ssr020.
[5] Ishikawa S, Maekawa M, Arite T, Onishi K, Takamure I, Kyozuka J. Suppression of tiller bud activity in tillering dwarf mutants of rice[J]. Plant & cell physiology,2005,46(1):79-86. doi: 10.1093/pcp/pci022.
[6] Komorisono M, Ueguchi-Tanaka M, Aichi I, Hasegawa Y, Ashikari M, Kitano H, Matsuoka Makoto, Sazuka T. Analysis of the rice mutant dwarf and gladius leaf 1. aberrant katanin-mediated microtubule organization causes up-regulation of gibberellin biosynthetic genes independently of gibberellin signaling[J].Plant Physiology, 2005,138(4):1982-1993. doi: 10.1104/pp.105.062968.
[7] Li P, Brutnell T P. Setaria viridis and Setaria italica, model genetic systems for the Panicoid grasses[J]. Journal of Experimental Botany, 2011, 62(9): 3031-3037. doi: 10.1093/jxb/err096.
[8] Lata C, Gupta S, Prasad M. Foxtail millet: A model crop for genetic and genomic studies in bioenergy grasses[J]. Critical Reviews in Biotechnology, 2012, 33(3): 328-343. doi: 10.3109/07388551.2012.716809.
[9] 贾小平,董普辉,张红晓,全建章,董志平. 谷子抗倒伏性和株高、穗部性状的相关性研究[J]. 植物遗传资源学报,2015,16(6):1188-1193. doi:10.13430/j.cnki.jpg2.2015.06.008
Jia X P, Dong P H, Zhang H X, Quan J Z, Dong Z P. Correlation study of lodging resistance and plant height, panicle traits in foxtail millet[J]. Journal of Plant Genetic Resources,2015,16(6):1188-1193.
[10] 王晓宇,刁现民,王节之,王春芳,王根全,郝晓芬,梁增浩,王雪梅,赵芳芳. 谷子SSR分子图谱构建及主要农艺性状QTL定位[J]. 植物遗传资源学报,2013,14(5):871-878.doi:10.134301/j.cnki.jpgr.2013.05.016.
Wang X Y, Diao X M, Wang J Z, Wang C F, Wang G Q, Hao X F, Liang Z H, Wang X M, Zhao F F. Construction of genetic map and QTL analysis of some main agronomic traits in millet[J]. Journal of Plant Genetic Resources,2013,14(5):871-878.
[11] 赵美丞. 谷子半显性矮秆基因SiDw1的图位克隆及形成机制分析[D]. 北京:中国农业科学院,2013.
Zhao M C. Map cloning and formation mechanism of semi-dominant dwarf gene SiDw1 in millet[D]. Beijing: Chinese Academy of Agricultural Sciences, 2013.
[12] Wang J, Wang Z L, Du X F, Yang H Q, Han F, Han Y H, Yuan F, Zhang L Y, Peng S Z, Guo E H. A high-density genetic map and QTL analysis of agronomic traits in foxtail millet (Setaria italica(L.)P. Beauv.)Using RAD-seq[J]. PLoS One, 2017, 12(6): e0179717. doi:10.1371/journal.pone.0179717.
[13] Zhang K, Fan G Y, Zhang X X, Zhao F, Wei W, Du G H, Feng X L, Wang X M, Wang F, Song G L, Zou H F, Zhang X L, Li S D, Ni X M, Zhang G Y, Zhao Z H. Identification of QTLs for 14 agronomically important traits in Setaria italica based on SNPs generated from high-throughput sequencing[J]. G3, 2017, 7(5): 1587-1594.doi: 10.1534/g3.117.041517.
[14] 陈广凤,陈建省,田纪春. 小麦株高相关性状与SNP标记全基因组关联分析[J]. 作物学报,2015,41(10):1500-1509. doi: 10.3724/SP.J.1006.2015.01500.
Chen G F, Chen J S, Tian J C. Genome-wide association analysis between SNP markers and plant height related traits in wheat[J].Acta Agronomica Sinica,2015,41(10):1500-1509.
[15] 黄静,程明星,唐敏强,张凤启,张园园,童超波,刘越英,程晓晖,董彩华,黄军艳,刘胜毅. 甘蓝型油菜RIL群体株高性状的全基因组关联分析[J]. 中国油料作物学报, 2016, 38(5): 543-548. doi: 10. 7505/j. issn. 1007-9084. 2016. 05. 001.
Huang J, Cheng M X, Tang M Q, Zhang F Q, Zhang Y Y, Tong C B, Liu Y Y, Cheng X H, Huang J Y, Liu S Y, Dong C H. Genome-wide association study of plant height in rapeseed RIL population[J]. Chinese Journal of Oil Crop Sciences, 2016, 38(5): 543-548.
[16] 刘凯,邓志英,张莹,王芳芳,刘佟佟,李青芳,邵文,赵宾,田纪春,陈建省. 小麦茎秆断裂强度相关性状QTL的连锁和关联分析[J]. 作物学报,2017,43(4):483-495. doi: 10.3724/SP.J.1006.2017.00483.
Liu K, Deng Z Y, Zhang Y, Wang F F, Liu T T, Li Q F, Shao W, Zhao B, Tian J C, Chen J S. Linkage analysis and genome-wide association study of QTLs controlling stem-breaking-strength-related traits in wheat[J]. Acta Agronomica Sinica,2017,43(4):483-495.
[17] 高宝祯,刘博,李石开,梁建丽,程锋,王晓武,武剑. 白菜类作物开花时间的全基因组关联分析[J]. 中国农业科学,2017,50(17):3375-3385. doi: 10.3864/j.issn.0578-1752.2017.17.012.
Gao B Z, Liu B, Li S K, Liang J L, Cheng F, Wang X W, Wu J. Genome-wide association studies for flowering time in Brassica rapa[J]. Scientia Agricultura Sinica, 2017, 50(17):3375-3385.
[18] Bennetzen J L, Schmutz J, Wang H, Percifield R, Hawkins J, Pontaroli A C, Estep M, Feng L, Vaughn J N, Grimwood J, Jenkins J, Barry K, Lindquist E, Hellsten U, Deshpande S, Wang X W, Wu X M, Mitros T, Triplett J, Yang X H, Ye C Y, Mauro-Herrera M, Wang L, Li P H, Sharma M, Sharma R, Ronald P C, Panaud O, Kellogg E A, Brutnell T P, Doust A N, Tuskan G A, Rokhsar D, Devos K M. Reference genome sequence of the model plant Setaria[J]. Nature Biotechnology,2012,30(6):555-561. doi: 10.1038/nbt.2196.
[19] Zhang G Y, Liu X, Quan Z W, Cheng S F, Xu X, Pan S K, Xie M, Zeng P, Yue Z, Wang W L, Tao Y, Bian C, Han C L, Xia Q J, Peng X H, Gao R, Yang X H, Zhan D L, Hu J C, Zhang Y X, Li H N, Li H, Li N, Wang J Y, Wang C C, Wang R Y, Guo T, Cai Y J, Liu C Z, Xiang H T, Shi Q X, Huang P, Chen Q C, Li Y R, Wang J, Zhao Z H, Wang J. Genome sequence of foxtail millet(Setaria italica)provides insights into grass evolution and biofuel potential[J]. Nature Biotechnology,2012,30(6):549-554. doi: 10.1038/nbt.2195.
[20] Gupta S,Kumari K, Muthamilarasan M, Parida S K, Prasad M. Population structure and association mapping of yield contributing agronomic traits in foxtail millet[J]. Plant Cell Reports,2014,33(6):881-893. doi: 10.1007/s00299-014-1564-0.
[21] Jia G Q, Huang X H, Zhi H, Zhao Y, Zhao Q, Li W J, Chai Y, Yang L F, Liu K Y, Lu H Y, Zhu C R, Lu Y Q, Zhou C C, Fan D L, Weng Q J, Guo Y L, Huang T, Zhang L, Lu T T, Feng Q, Hao H F, Liu H K, Lu P, Zhang N, Li Y H, Guo E H, Wang S J, Wang S Y, Liu J R, Zhang W F, Chen G Q, Zhang B J, Li W, Wang Y F, Li H Q, Zhao B H, Li J Y, Diao X M, Han B. A haplotype map of genomic variations and genome-wide association studies of agronomic traits in foxtail millet(Setaria italica)[J]. Nature Genetics, 2013,45(8):957-961. doi: 10.1038/ng.2673.
[22] 林泽川,曹立勇. 水稻株型相关基因的定位与克隆研究进展[J]. 中国稻米,2014,20(1):17-22,27.doi: 10.3969/j.issn.1006-8082.2014.01.004.
Lin Z C, Cao L Y. Progress on mapping and cloning of genes related to rice plant type[J]. China Rice,2014,20(1):17-22,27.
[23] Li Y H, Shen Y, Cai C, Zhong C C, Zhu L, Yuan M, Ren H Y. The type Ⅱ Arabidopsis formin14 interacts with microtubules and microfilaments to regulate cell division[J]. The Plant Cell, 2010,22(8):2710-2726. doi: 10.4161/psb.6.5.14979.
[24] Vidali L, van Gisbergen P A C, Guérin C, Franco P, Li M, Burkart G M, Augustine R C, Blanchoin L, Bezanilla M. Rapid formin-mediated actin-filament elongation is essential for polarized plant cell growth[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(32): 13341-13346. doi:10.1073/pnas.0901170106.
[25] Zhang Z, Zhang Y, Tan H X, Wang Y, Li G, Liang W Q, Yuan Z, Hu J P, Ren H Y, Zhang D B. Rice morphlogy determinant encodes the type Ⅱ formin FH5 and regulates rice morphogenesis[J]. Plant Cell, 2011, 23(2): 681-700. doi: 10.1105/tpc.110.081349.