组蛋白去乙酰化酶(Histone deacetylase, HDAC)通过去乙酰化作用移除染色质组蛋白上的乙酰基,导致染色质浓缩并抑制基因表达[1]。目前,植物中的HDAC可分为4个家族,即RPD3家族、HDA1家族、SIR2家族和HD2家族,其中HD2蛋白是植物特有的HDAC成员[2-3]。近年来,HD2家族成员的功能得到了深入研究。研究表明,拟南芥中有4个成员,分别是HD2A(HDT1)、HD2B(HDT2)、HD2C(HDT3)和HD2D(HDT4)。AtHD2A、AtHD2B和AtHD2C能够调控转录抑制,其中,在拟南芥中,下调表达AtHD2A导致种子产量降低[4-5];AtHD2C与脱落酸(ABA)、盐、干旱等胁迫反应相关[6-7]。Fu等[8]在水稻中发现HD2基因家族至少有2个成员,分别是OsHDT701和OsHDT702。下调表达OsHDT702水稻植株的叶片严重变窄,表明OsHDT702可能参与细胞分裂或增殖[7]。OsHDT701通过降低组蛋白H4乙酰化和GA合成相关基因的表达,影响水稻种子萌发,过表达OsHDT701可以提高植株对高盐和干旱的耐受性[9]。Ding等[10]研究发现,OsHDT701通过调节PRR和防卫相关蛋白的组氨酸H4乙酰化,负向调控水稻的天然免疫。这就表明植物HDACs广泛参与植物的生长发育及逆境响应过程,对水稻的HD2家族基因深入研究有重要的意义[11]。
选择性剪接又称为可变剪接(Alternative splicing,AS),是指一个基因的转录产物在不同的生长发育阶段、特异的组织器官和外界环境影响下,通过不同的剪接方式,可以得到不同的mRNA和蛋白质产物,这种现象是导致基因功能多样性的重要原因之一[12]。可变剪接事件广泛存在于真核生物基因表达过程中,调节植物的70%基因的外显子[13-14]。一些研究表明,植物通过可变剪接来调节其生理和新陈代谢,从而在正常和压力条件下保持体内稳态,包括寒冷、干旱、高温、高盐度和病原体感染[15-18]。可变剪接可以通过调节细胞内基因的表达,在植物的生长发育、生理代谢、生物和非生物胁迫等过程中发挥调控作用[19-20]。例如,拟南芥开花自主途径中FCA(Flowering time control protein)基因通过可变剪接产生4种转录本,并通过对转录本的表达量调控来完成自身的负调控表达,最终影响开花时间[21]。
在Phytozome网站(https://phytozome.jgi.doe.gov/pz/portal.html)获得了水稻HD2基因家族的另一成员OsHDT703(LOC_Os01g68160),该基因的克隆、表达特性和生物学功能分析等尚未见报道,有待进一步研究。本研究中,依据Phytozome网站数据库资源,首先利用生物信息学分析了该基因的结构特点和蛋白进化关系,并发现OsHDT703基因可能存在多种不同的可变剪接表达产物,进而以粳稻中花11为材料,设计各种可变剪接体的特异引物,通过RT-PCR对不同剪接体进行扩增,经TA克隆后进行测序分析,以确定OsHDT703真实存在的可变剪切体,为后续深入研究该基因在水稻生长发育、生理代谢、生物和非生物胁迫等过程中可能具有的生物学功能奠定基础。
1 材料和方法
1.1 植物材料
试验材料为粳稻中花11(ZH11)的叶片和幼穗。
1.2 质粒和菌株
用于TA克隆的pMD18-T(TaKaRa)载体,大肠杆菌菌株DH5α。
1.3 试验方法
1.3.1 总RNA提取以及cDNA第一链合成 本试验参照吴蔚蔚等[22]的方法,采用液氮对水稻叶片和幼穗进行研磨,用TRIzol法抽提水稻叶片和幼穗的总RNA,并按照TIANGEN的TIANscriptⅡRT kit cDNA第一链合成试剂盒说明书合成cDNA第一链。
1.3.2 OsHDT703不同剪接体分析和引物设计 Phytozome网站(https://phytozome.jgi.doe.gov/pz/portal.html)的数据库资源表明,OsHDT703可能存在5种不同的剪接体(表1),分别编号为1~5号可变剪接体,根据这些可变剪接体的序列特征,设计了可变剪接体的RT-PCR特异引物(表2)。其中LOC_Os01g68160.3和LOC_Os01g68160.4的CDS序列一样,其蛋白序列也就相同,故未设计区分这2个可变剪接体的特异引物。不同引物所在的位置如图1所示。
表1 水稻OsHDT703基因可能存在的5种可变剪接体
Tab.1 Five putative splice variants of rice OsHDT703 gene
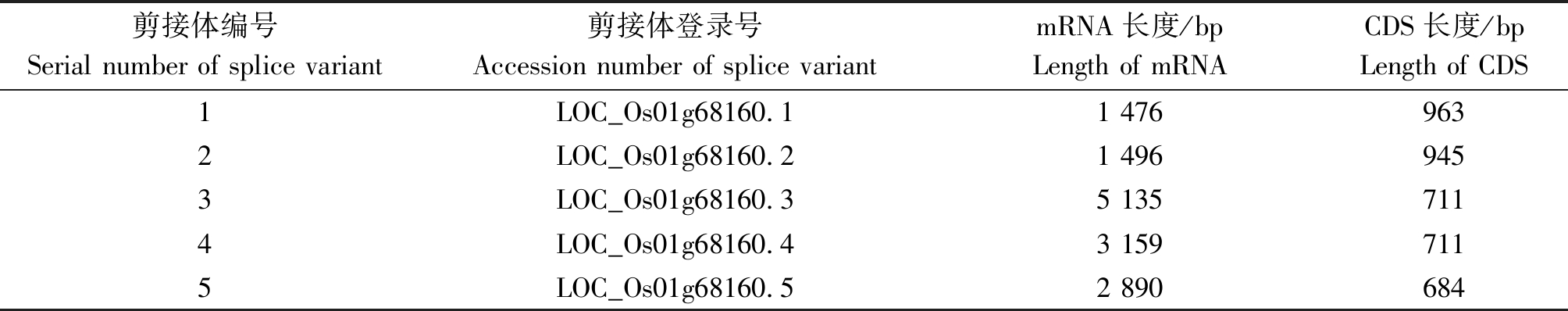
剪接体编号Serial number of splice variant剪接体登录号Accession number of splice variantmRNA长度/bpLength of mRNA CDS长度/bpLength of CDS 1LOC_Os01g68160.11 4769632LOC_Os01g68160.21 4969453LOC_Os01g68160.35 1357114LOC_Os01g68160.43 1597115LOC_Os01g68160.52 890684
1.3.3 RT-PCR扩增和目的片段割胶纯化回收 以叶片/幼穗cDNA为模板,利用TransTaq HiFi DNA Polymerase酶和表2中的引物对进行OsHDT703基因的可变剪接体RT-PCR扩增,电泳后割胶纯化回收获得预期大小的PCR产物,用于后续的TA克隆试验。
1.3.4 TA克隆 利用TaKaRa公司的pMD18-T Vector Cloning kit 试剂盒,将纯化的目的片段连接到pMD18-T载体,连接产物转化大肠杆菌感受态DH5α,挑选阳性克隆后做菌液PCR鉴定,随后进行测序检测和序列比对分析。
表2 引物与PCR扩增产物信息
Tab.2 Information of primers and PCR product

引物对名称Name of primer pairs引物对序列(5′-3′)Sequence of primer pairs (5′-3′)片段长度/bpPCR product 可能的PCR产物Putative PCR productsmFCGTCATTTTTCCTTTTTCTTCCTCCTC1 3221号剪接体m12RGAAGAGACCATCGTCTCGAATCCTAATA1 3422号剪接体mFCGTCATTTTTCCTTTTTCTTCCTCCTC1 1312号剪接体m2RGTCCCTGAGCAAGTTAGCAAGTGCDSFATGGACGTCGATTTTTGGGGAGT7173/4号剪接体CDS345RCCCAAATCAATCTACACATACCTTTGC6905号剪接体CDSFATGGACGTCGATTTTTGGGGAGT3765号剪接体CDS5RGGCATCTAACTTGGGGTATAGACGAATAC
1.3.5 OsHDT703蛋白进化分析 基于上述获得的OsHDT703的可变剪接体对应的蛋白序列以及NCBI数据库(https://www.ncbi.nlm.nih.gov)中拟南芥、水稻、大豆、玉米、小麦、大麦、茄属植物等的一些HD2家族蛋白序列,利用ClustalX软件,参考Wang等[23]的方法,分析水稻OsHDT703蛋白的进化关系。
2 结果与分析
2.1 OsHDT703基因的可变剪接体及其RT-PCR特异引物位置
Phytozome网站(https://phytozome.jgi.doe.gov/pz/portal.html)上的数据库资源显示,OsHDT703可能存在5种不同的剪接体(图1),1~5号可变剪接体分别代表了LOC_Os01g68160.1、LOC_Os01g68160.2、LOC_Os01g68160.3、LOC_Os01g68160.4和LOC_Os01g68160.5,其中2号剪切体的mRNA比1号剪切体多了20 bp序列(即CACTTGCTAACTT GCTCAGG),3和4号剪接体的CDS序列完全相同,比5号剪接体多了27 bp序列(即GTATTCGTCTAT ACCCCAAGTTAGATG)。根据这些可变剪接体序列,设计了其RT-PCR扩增的特异引物,在剪接体上的位置见图1。
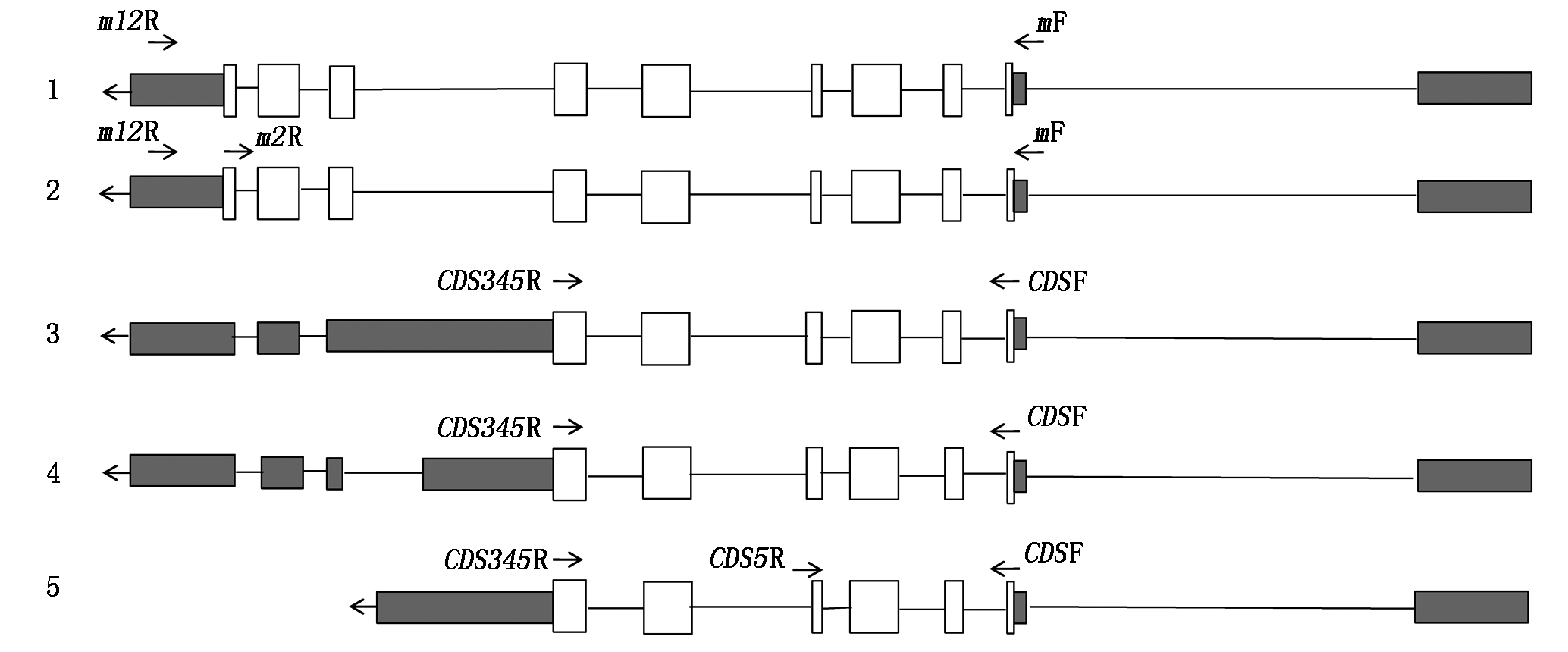
1~5分别为剪接体LOC_Os01g68160.1、LOC_Os01g68160.2、LOC_Os01g68160.3、LOC_Os01g68160.4和LOC_Os01g68160.5。
1-5 represent splice variants LOC_Os01g68160.1, LOC_Os01g68160.2, LOC_Os01g68160.3, LOC_Os01g68160.4 and LOC_Os01g68160.5.
图1 OsHDT703可能存在的5种剪接体及其RT-PCR特异引物在剪接体上的位置
Fig.1 Five putative splice variants of OsHDT703 and their specific RT-PCR primer locations
2.2 OsHDT703基因可变剪接体的克隆
首先,以ZH11的cDNA为模板,采用引物mF/m12R和CDSF/CDS345R分别做PCR扩增,得到预期的条带(图2-A),分别约为1 322/1 342 bp和717/690 bp,初步说明图2-A中泳道1的条带可能包含1和/或2号剪切体,泳道2的条带可能包含3/4和/或5号剪接体。然后分别以上述mF/m12R和CDSF/CDS345R的PCR产物为模板,利用mF/m2R和CDSF/CDS5R引物对进行RT-PCR扩增,获得了预期的PCR产物(图2-B),图2-B中泳道1为引物mF/m2R的扩增条带,与预期的1 131 bp大小基本一致,初步说明2号剪接体的存在,泳道2为CDSF/CDS5R的扩增条带,与预期的376 bp大小接近,初步说明5号剪接体的存在。
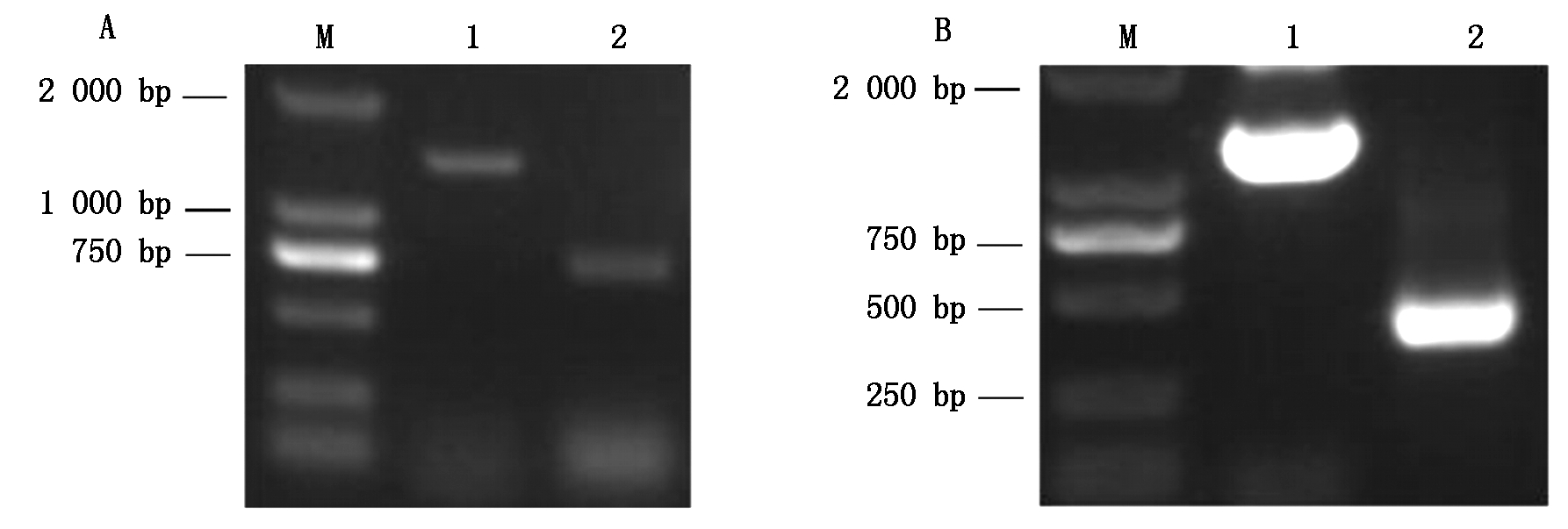
A: M.Marker;1.引物mF/m12R扩增条带; 2.引物CDSF/CDS345R扩增条带。
B:M.Marker;1.引物mF/m2R扩增条带; 2.引物CDSF/CDS5R扩增条带。
A:M.Marker; 1.mF/m12R; 2.CDSF/CDS345R. B:M.Marker; 1.mF/m2R; 2.CDSF/CDS5R.
图2 OsHDT703不同剪接体PCR扩增结果
Fig.2 PCR amplification of different splice variants of OsHDT703
为了进一步确定OsHDT703剪接体的真实存在,将上述引物对mF/m12R和CDSF/CDS345R的PCR扩增电泳条带进行割胶,按照琼脂糖凝胶DNA回收试剂盒说明书进行PCR产物的纯化回收,将纯化回收得到的产物连接到pMD18-T载体,将连接产物转化大肠杆菌DH5α感受态,再用基因特异性引物进行菌落PCR鉴定阳性单菌落,部分菌落PCR结果如图3所示。
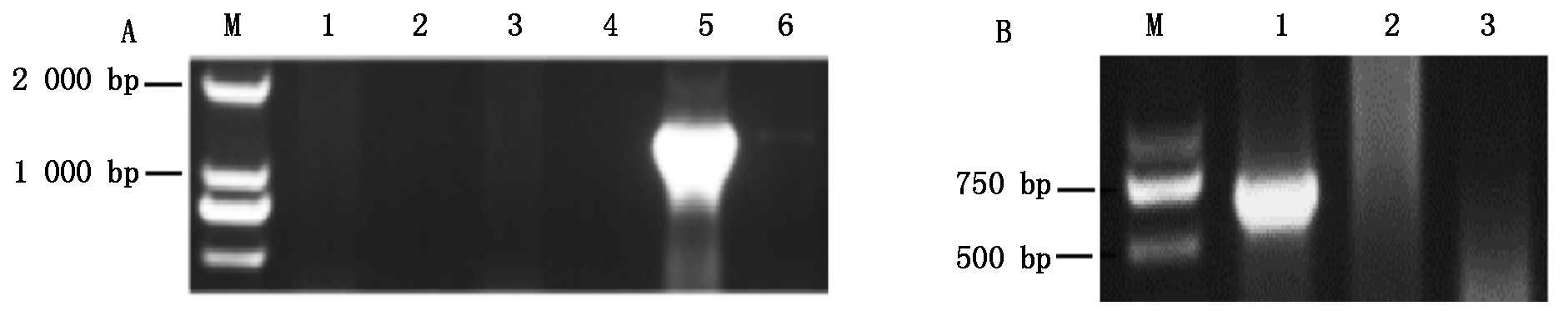
A: 1号剪接体菌落PCR鉴定结果;M.Marker;1-6.单菌落;B:3/4号剪接体菌落PCR鉴定结果;M.Marker;1-3.单菌落。
A: Results of colony PCR for No.1 splice variant; M.Marker;1-6.Single colonies;B:Results of colony PCR for No.3/4 splice variant; M.Marker;1-3.Single colonies.
图3 OsHDT703基因部分剪接体的菌落PCR鉴定结果
Fig.3 Partial results of colony PCR for splice variants of OsHDT703

A.1号剪接体序列比对;B.2号剪接体序列比对;C.3/4号剪接体序列比对;D.5号剪接体序列比对。
A.Sequence alignment of No.1 splice variant; B.Sequence alignment of No. 2 splice variant; C. Sequence alignment of No.3/4 splice variant; D.Sequence alignment of No.5 splice variant.
图4 OsHDT703可变剪接体测序结果的比对分析
Fig.4 Alignments of sequencing results of OsHDT703 splice variants
2.3 OsHDT703基因不同剪接体的测序结果分析
挑选上述若干单克隆阳性菌落摇菌,提取质粒送北京擎科新业生物科技公司进行测序,测序结果在NCBI上进行Blast分析,比对结果如图4。其中,图4-A-B显示引物对mF/m12R扩增到的产物中分别有1和2号剪切体的存在;图4-C-D显示引物对CDSF/CDS345R扩增到的产物中分别有3、4和5号剪切体的存在。3号剪切体和4号剪接体CDS序列完全相同,未能区分,可能存在一种或者二者均存在。至此,结果显示,OsHDT703基因预测的5种可变剪接体中,至少存在4种不同的可变剪接体。
2.4 OsHDT703蛋白的结构与进化分析
从Phytozome数据库中下载OsHDT701、OsHDT702和OsHDT703蛋白序列,对蛋白序列进行序列分析发现,蛋白N端均具有NPL保守结构域(图5),说明鉴定到的OsHDT703蛋白为水稻HD2家族成员。

图5 OsHDT703蛋白的结构分析
Fig.5 Protein sequence analysis of OsHDT703
通过Phytozome(https://phytozome.jgi.doe.gov/pz/portal.html)网站获知,OsHDT703基因主要编码1个由320个氨基酸组成的蛋白质,借助NCBI数据库(https://www.ncbi.nlm.nih.gov)中的蛋白质序列分析,选取来源于拟南芥、水稻、大豆、玉米、小麦、大麦、茄属植物等共16个蛋白,用于分析OsHDT703蛋白的进化关系,采用ClustalX(Version 2.0)软件和MEGA 7进行序列比对,构建了无根进化树(图6)。结果显示, 水稻OsHDT703蛋白与水稻另一个HD2家族蛋白OsHDT702以及大麦中的一个HD2家族蛋白处于同一分支,亲缘关系较近;但是水稻OsHDT701蛋白与玉米中的HD2蛋白、小麦和大麦中的HD2蛋白关系较近,说明水稻中HD2蛋白的功能可能存在一定的分化。
3 讨论
Fu等[8]在水稻中已报道了2个HD2基因家族成员,分别是OsHDT701和OsHDT702,其编码的蛋白质具有NPL结构域。基于Phytozome网站数据库资源,本研究利用RT-PCR、TA克隆和测序等方法克隆了尚未见功能报道的OsHDT703基因的可变剪接体,其编码的蛋白序列N端含有NPL结构域,说明OsHDT703属于HD2蛋白家族成员。
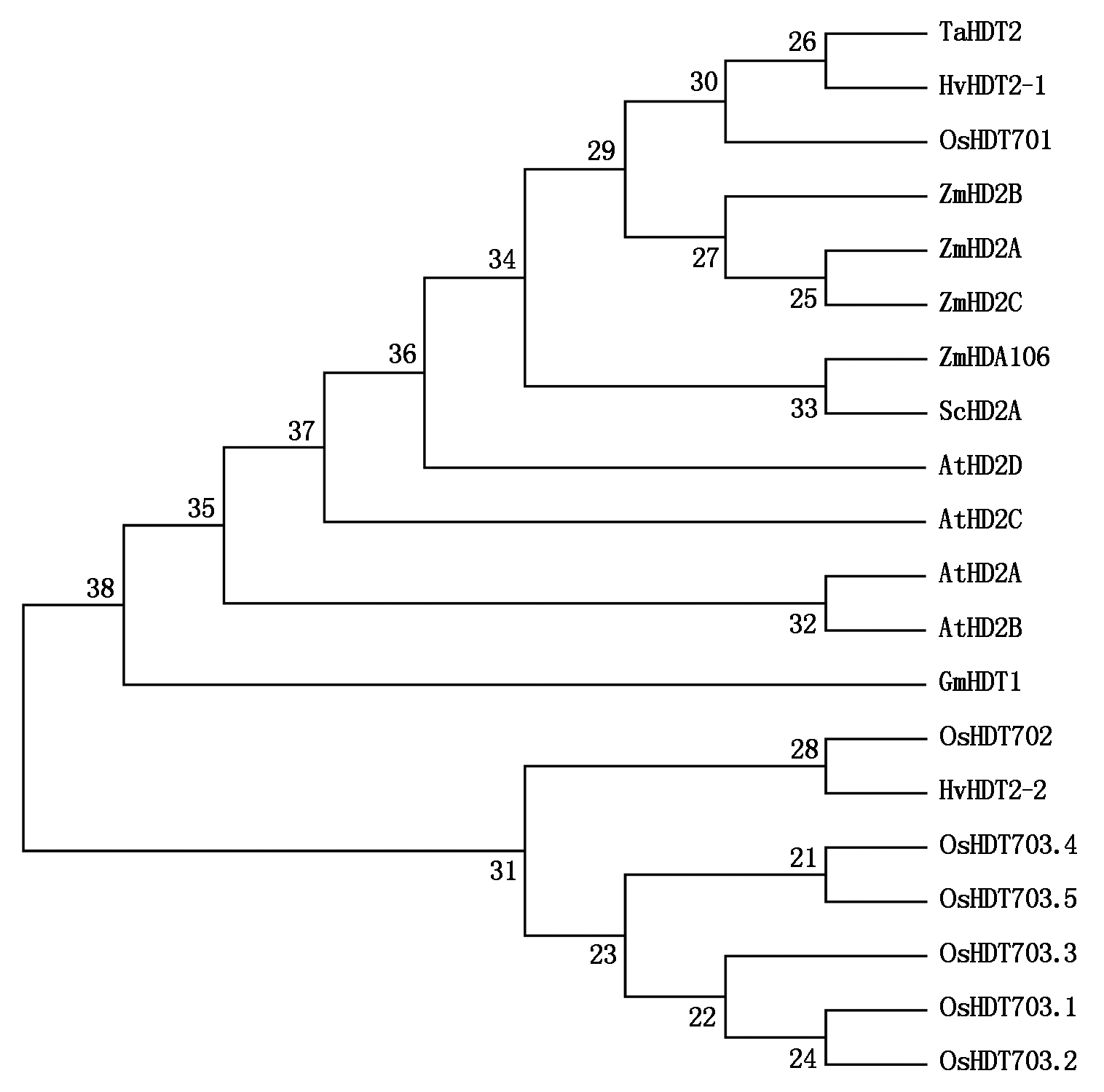
选择来源于7个物种的16个蛋白构建Neighbor joining树,At.拟南芥; Os.水稻; Hv.大麦; Zm.玉米; Ta.小麦;Gm.大豆;Sc.茄属植物。
There were 16 genes chosen from different common species like At. Arabidopsis thaliana; Os.Oryza sativa; Hv.Hordeum vulgare; Zm.Zea mays; Ta.Triticum aestivum; Gm.Glycine max; Sc.Solanum chacoense.
图6 OsHDT703的进化分析
Fig.6 Neighbor joining tree of OsHDT703 in different species
同一家族基因常常具有类似功能,也存在一定的分化。在拟南芥中,下调表达AtHD2A导致种子产量降低[5];AtHD2C与脱落酸(ABA)、盐、干旱等胁迫反应相关[6-7],说明HD2家族成员的功能具有一定的分化。通过蛋白进化分析结果显示,水稻OsHDT703与水稻中另一个HD2家族基因OsHDT702以及大麦中的一个HD2家族基因较近;而水稻OsHDT701蛋白与玉米中的另外3个蛋白、大麦和小麦中的HD2蛋白关系较近,说明水稻中HD2蛋白的功能可能存在一定的分化。研究表明,OsHDT702可能通过参与细胞分裂或增殖来调控水稻植株叶片的宽度[7]。水稻OsHDT703蛋白与水稻中另一个HD2家族蛋白OsHDT702处于同一分支,可能具有类似功能,尚需进一步验证。
可变剪接事件广泛存在于真核生物基因表达过程中[13],发生于参与表达调控过程的基因,如信号传导通路[13]及程序性死亡[15]相关基因。在拟南芥中,通过转录组测序发现了数万种AS,可能参与到非生物胁迫、器官发育和细胞分化等过程[24-25]。拟南芥开花自主途径中FCA(Flowering time control protein)基因通过可变剪接产生4种转录本,并通过对转录本的表达量调控来完成自身的负调控表达,最终影响开花时间[19]。水稻NAL1基因的可变剪切调控水稻旗叶宽主效QTL(qFLW4),进而影响叶绿素含量和剑叶大小[26]。Phytozome网站数据显示OsHDT703存在5种可变剪接,本研究确认了OsHDT703基因至少有4种不同可变剪接体。前人研究结果已经表明,拟南芥、水稻等植物中HD2家族基因广泛参与植物的生物和非生物胁迫的调控[7-9]。OsHDT703基因可能通过不同可变剪接体参与植物的生长发育及对逆境的响应过程中。水稻OsHDT703基因可变剪接体的验证结果为进一步研究水稻OsHDT703基因的功能奠定基础,为后续的试验研究提供了理论依据。
[1] Farhi J, Tian G, Fang H, Maxwell D, Xing T, Tian L N.Histone deacetylase HD2D is involved in regulating plant development and flowering time in Arabidopsis [J]. Plant Signaling & Behavior, 2017, 12(18):e1300742. doi: 10.1080/15592324.2017.1300742.
[2] Pandey R, Müller A, Napoli C A, Selinger D A, Pikaard C S, Richards E J, Bender J, Mount D W, Jorgensen R A.Analysis of histone acetyltransferase and histone deacetylase families of Arabidopsis thaliana suggests functional diversification of chromatin modification among multicellular eukaryotes [J]. Nucleic Acids Research, 2002, 30(23):5036-5055. doi:10.1093/ nar/gkf660.
[3] Yang C, Shen W J, Chen H F, Chu L T, Xu Y C, Zhou X C, Liu C L, Chen C M, Zeng J H, Liu J, Li Q F, Gao C J, Charron J B, Luo M.Characterization and subcellular localization of histone deacetylases and their roles in response to abiotic stresses in soybean [J]. BMC Plant Biology, 2018, 18:226. doi: 10.1186/s12870-018-1454-7.
[4] Wu K Q, Tian L N, Malik K, Brown D, Miki B.Functional analysis of HD2 histone deacetylase homologues in Arabidopsis thaliana [J]. The Plant Journal, 2010, 22(1):19-27. doi: 10.1046/j.1365-313x.2000.00711.x.
[5] Sridha S, Wu K. Identification of AtHD2C as a novel regulator of abscisic acid responses in Arabidopsis [J]. The Plant Journal, 2006, 46(1):124-133. doi: 10.111/j.1365-313x.2006.02678.x.
[6] Luo M, Wang Y Y, Liu X C, Yang S G, Lu Q, Cui Y H, Wu K Q.HD2C interacts with HDA6 and is involved in ABA and salt stress response in Arabidopsis [J]. Journal of Experimental Botany, 2012, 63(8):3297-3306. doi:10.1093/jxb/ers059.
[7] Hu Y F, Qin F J, Huang L M,Sun Q W, Li C, Zhao Y, Zhou D X. Rice histone deacetylase genes display specific expression patterns and developmental functions [J]. Biochemical and Biophysical Research Communications, 2009, 388(2):266-271. doi:10.1016/j.bbrc.2009.07.162.
[8] Fu W, Wu K Q, Duan J. Sequence and expression analysis of histone deacetylases in rice[J]. Biochemical and Biophysical Research Communications, 2007, 356(4):843-850. doi:10.1016/j. bbrc.2007.03.010.
[9] Zhao J H, Zhang J X, Zhang W, Wu K L, Zheng F, Tian L N, Liu X C, Duan J. Expression and functional analysis of the plant-specific histone deacetylase HDT701 in rice [J]. Frontiers in Plant Science, 2015, 5:764. doi:10.3389/fpls.2014.00764.
[10] Ding B, Bellizzi M R, Ning Y, Meyers B C, Wang G L. HDT701, a histone H4 deacetylase, negatively regulates plant innate immunity by modulating histone h4 acetylation of defense-related genes in rice [J]. The Plant Cell, 2012, 24(9):3783-3794. doi:10.1105/tpc. 112.101972.
[11] Luo M, Liu X C, Singh P, Cui Y H, Zimmerli L, Wu K Q. Chromatin modifications and remodeling in plant abiotic stress responses [J]. Biochim Biophys Acta, 2012, 1819(2):129-136. doi:10.1016/j.bbagrm.2011.06.008.
[12] Roberts G C, Smith C W. Alternative splicing: combinatorial output from the genome [J]. Current Opinion in Chemical Biology, 2002, 6(3):375-383. doi:10.1016/S1367-5931(02)00320-4.
[13] Zhang R X, Calixto C, Marquez Y, Venhuizen P, Tzioutziou N A, Guo W B, Spensley M, Entizne J C, Lewandowska D, Ten H S, Frei Dit F N, Hirt H, James A B, Nimmo H G, Barta A, Kalyna M, Brown J W S. A high quality Arabidopsis transcriptome for accurate transcript-level analysis of alternative splicing [J]. Nucleic Acids Research, 2017, 45(9):5061-5073. doi: 10.1093/nar/gkx267.
[14] Chomala S, Feng G Q, Chavarro C, Barbazuk W B. Genome-wide identification of evolutionarily conserved alternative splicing events in flowering plants [J]. Frontiers in Bioengineering and Biotechnology, 2015, 3:33. doi: 10.3389/fbioe.2015.00033.
[15] Filichkin S A, Hamilton M, Dharmawardhana P D, Singh S K, Sullivan C, Ben-Hur A, Reddy A S N, Taiswal P.Abiotic stresses modulate landscape of poplar transcriptome via alternative splicing, differential intron retention, and isoform ratio switching [J]. Frontiers in Plant Science, 2018, 9:5. doi: 10.3389/fpls.2018.00005.
[16] Jiang J F, Liu X N, Liu C H, Liu G T, Li S H, Wang L J. Integrating omics and alternative splicing reveals insights into grape response to high temperature[J]. Plant Physiology, 2017, 173(2):1502-1518. doi: 10.1104/pp.16.01305.
[17] Hartmann L, Drewe-Boβ P, Wieβner T, Wagner G, Geue S, Lee H C, Obermüller D M, Kahles A, Behr J, Sinz F H, Rätsch G, Wachter A. Alternative splicing substantially diversifies the transcriptome during early photomorphogenesis and correlates with the energy availability in Arabidopsis [J]. The Plant Cell, 2016, 28(11): 2715-2734. doi: 10.1105/tpc.16.00508.
[18] Filichkin S A, Cumbie J S, Dharmawardhana P, Jaiswal P, Chang J H, Palusa S G, Reddy A S, Megraw M, Mockler T C. Environmental stresses modulate abundance and timing of alternatively spliced circadian transcripts in Arabidopsis [J]. Molecular Plant, 2015, 8(2):207-227. doi:10.1016/j.molp.2014.10.011.
[19] Leviatan N, Alkan N, Leshkowitz D, Fluhr R. Genome-wide survey of cold stress regulated alternative splicing in Arabidopsis thaliana with tiling microarray [J]. PLoS One, 2013, 8(6): e66511. doi:10.1371/journal.pone.0066511.
[20] Staiger D, Brown J W. Alternative splicing at the intersection of biological timing, development, and stress responses [J]. The Plant Cell, 2013, 25(10): 3640-3656. doi: 10.1105/tpc. 113.113803.
[21] Quesada V, Macknight R, Dean C, Simpson G G.Autoregulation of FCA pre-mRNA processing controls Arabidopsis flowering time [J]. EMBO Journal, 2003, 22(12):3142-3152. doi:10.1093/emboj/cdg305.
[22] 吴蔚蔚,童普国,王鑫,阎新,李绍波,欧阳解秀. 水稻OsGPRP家族基因克隆及其对非生物胁迫的响应 [J]. 华北农学报, 2018, 33(1):39-44. doi:10.7668/hbnxb.2018.01.007.
Wu W W, Tong P G, Wang X, Yan X, Li S B, Ouyang J X. Cloning of OsGPRP family genes and their responses to abiotic stresses in rice [J]. Acta Agriculturae Boreali-sinica, 2018, 33(1):39-44.
[23] Wang X, Zhang H, Shao L Y, Yan X, Peng H, Ouyang J X, Li S B. Expression and function analysis of a rice OsHSP40 gene under salt stress [J]. Genes & Genomics, 2019, 41(2):175-182. doi: 10.1007/s13258-018-0749-2.
[24] Shikata H, Hanada K, Ushijima T, Nakashima M, Suzuki Y, Matsushita T. Phytochrome controls alternative splicing to mediate light responses in Arabidopsis [J]. Proceedings of the National Academy of Sciences of the United States of America,2014,111(52):18781-18786. doi:10.1073/pnas.1407147112.
[25] Li S, Yamada M, Han X, Ohler U, Benfey P N. High resolution expression map of the Arabidopsis root reveals alternative splicing and lincRNA regulation[J]. Developmental Cell,2016, 39(4): 508-522. doi: 10.1016/j.devcel.2016.10.012.
[26] Chen M L, Luo J, Shao G N, Wei X J, Tang S Q, Sheng Z H, Song J, Hu P S.Fine mapping of a major QTL for flag leaf width in rice, qFLW4, which might be caused by alternative splicing of NAL1[J]. Plant Cell Reports, 2012, 31(5):863-872. doi: 10.1007/s00299-011-1207-7.