水稻是全球最主要的粮食作物之一,全世界约一半的人口以大米作为主食,伴随着人们生活水平的不断提高,人们对水稻的需求不仅停留在量上,也在寻求质的提高。香味是衡量水稻品质的标准之一,而且香米也具有很高的营养价值,富含多种维生素、氨基酸[1],因此香味性状越来越受到市场的青睐和人们的关注,同时也成为育种家们研究的热点之一[2]。黑龙江省作为我国重要的水稻生产基地,近年来在香味性状育种上也取得了显著的成绩[3],但香稻遗传背景较为单一,香稻品种大都是由五优稻4号和绥粳4号为基础衍生而来,导致黑龙江省优质香稻数量有限且遗传基础较为狭窄。通过传统的育种方法解决这一问题,具有周期长、突变方向不确定等缺点,所以急需利用新的遗传改良手段进行粳稻的香味改良。
水稻香味基因由第8号染色体上的隐性基因Badh2控制,该基因由15个外显子组成[4],Badh2编码1个甜菜碱醛脱氢酶,具有醛脱氢酶活性,催化甜菜碱醛、4-氨基丁醛(AB-ald)和3-氨基丙醛的氧化。Buttery等[5]研究表明,水稻中的香味物质主要是2-乙酰-1-吡咯啉(2-AP),在非香型水稻品种中,Badh2蛋白催化4-氨基丁醛的氧化,而4-氨基丁醛是2-AP的合成前体,当4-氨基丁醛被Badh2氧化后,抑制了2-AP的合成,因此,稻米丧失了香味。当Badh2基因发生功能突变时,导致Badh2蛋白丧失功能,不能催化4-氨基丁醛的氧化,造成4-氨基丁醛的积累,从而促进了2-AP的合成,使稻米产生香味[6]。Badh2只有全转录本未发生突变时才具有甜菜碱脱氢酶活性,如果部分碱基发生缺失或其他突变会导致该基因的翻译提前终止[7-9]。
基因组编辑技术可以定向修饰和改造基因,且周期短、效率高,能有效地弥补传统育种的不足。基因组编辑技术目前包含锌指核酸酶技术(ZFNs)[10]、类转录激活因子效应物核酸酶技术(TALENs)[11]以及CRIPSR/Cas9技术[12]等,这些编辑技术在基因组特定位置引发双链断裂(DSB),引起细胞中的自我修复机制即非同源末端连接(NHEJ),进而造成修复的位置发生小片段的碱基缺失或插入,导致目标基因的移码突变[13]。但前两项技术操作繁琐,成本较高,而CRIPSR/Cas9系统因具有效率高、成本低、操作简单等优势,近年来应用的较为广泛[14],不仅成功地应用于小鼠[15]、斑马鱼[16]、人[17]等多个物种中,而且在拟南芥、水稻、小麦、玉米、杨树等植物中也成功实现了定点编辑[18-21]。水稻作为单子叶模式植物,CRIPSR/Cas9系统不仅应用于水稻的基因功能研究[22-24],而且在性状遗传改良方面也被广泛地研究[25-27],表明CRIPSR/Cas9系统对水稻的基因编辑可以稳定地遗传至下一代,该技术对水稻育种具有重大的意义。
本研究以高产、优质、多抗的非香型粳稻品种东农425为试验材料,利用CRIPSR/Cas9技术,针对Badh2的第2和第3外显子进行定点编辑,以期获得香味显著提高、无转基因成分且主要农艺性状无显著变化的纯合突变材料,为加快香型粳稻品种的培育提供理论和技术支持。
1 材料和方法
1.1 试验材料
以东北农业大学选育的非香型粳稻品种东农425为转基因受体材料,该品种具有耐寒性强、产量高、米质优良等特点。本研究所用U3-gRNA、U6a-gRNA载体和pYLCRISPR/Cas9Pubi-H载体均由华南农业大学刘耀光院士惠赠。
1.2 gRNA靶点接头引物设计
通过NCBI获得Badh2基因(Os08g0424500)的CDS序列。利用CRISPR-GE网站(http://skl.scau.edu.cn/)[28],针对Badh2的第2外显子和第3外显子分别设计了2对gRNA靶点接头引物,B1-U3-T1-F/R、B1-U6a-T2-F/R(表1)以及B2-U3-T1-F/R、B2-U6a-T1-F/R(表1),并且分析靶点的特异性,排除潜在脱靶序列。
表1 本研究所使用的引物
Tab.1 Primers used in this study

引物名称Primer name引物序列(5′-3′)SequenceB1-U3-T1-FGGCACAAGTACCTCCGCGCAATCGB1-U3-T1-RAAACCGATTGCGCGGAGGTACTTGB1-U6a-T2-FGCCGAATCGAGAGGAAATCTGAGCB1-U6a-T2-RAAACGCTCAGATTTCCTCTCGATTB2-U3-T1-FGGCAGGCGAGATCCCGGCGGGCAB2-U3-T1-RAAACCCGTGCCCGCCGGGATCTCGB2-U6a-T2-FGCCGAGAGGAACCGGGGCCGCGACB2-U6a-T2-RAAACGTCGCGGCCCCGGTTCCTCTB1′TTCAGAGGTCTCTCTCGCACTGGAATCGGCAGCAAAGGB2AGCGTGGGTCTCGTCAGGGTCCATCCACTCCAAGCTCB2′TTCAGAGGTCTCTCTGACACTGGAATCGGCAGCAAAGGBLAGCGTGGGTCTCGACCGGGTCCATCCACTCCAAGCTCNOSFGCGGTGTCATCTATGTTACTAGM13F-47CGCCAGGGTTTTCCCAGTCACGACSeq(1)-T1-FTACCCTCCTCTTCACCCTCTCCSeq(1)-T1-RAACCTACTGGAGAGCCACGCSeq(1)-T2-FGCCTTGACGATGGGACTSeq(1)-T2-RTAGCTGCTAGGTACAATTTGTGAGACSeq(2)-T-FCTCTCCCACCACCACTCCACACSeq(2)-T-RACACAATCAGCCATGCTTCCAACas9-FCTGACGCTAACCTCGACAAGCas9-RCCGATCTAGTAACATAGATGACACC
1.3 CRISPR/Cas9表达载体的构建
参考Ma等[29]的方法进行2个载体构建。①制备靶点双链接头。将设计好的上下游靶位点接头引物混合,95 ℃处理30 s后移至室温退火。②酶切。利用BsaⅠ内切酶切割2对pYLgRNA-U3和pYLgRNA-U6a载体,再利用T4连接酶将上一步制备好的靶点接头连入gRNA表达盒中。③两轮巢式PCR。第1轮PCR:利用通用引物对连接好靶点的gRNA表达盒进行扩增。第2轮PCR:取上一轮产物稀释20倍作为扩增模板,分别利用引物B1′/B2(表1)和B2′/BL(表1)对其进行扩增。④产物纯化。电泳检测2对靶点的gRNA表达盒PCR产物,符合条带大小后切胶回收。⑤gRNA表达盒克隆到Cas9载体。此步骤基于“金门”克隆法,一边酶切混合好的2对gRNA表达盒和2个pYLCRISPR/Cas9Pubi-H载体,一边将2对靶点的gRNA表达盒连接到2个pYLCRISPR/Cas9Pubi-H载体上。⑥转化及检测。将2个连接好的载体通过热激法转入大肠杆菌Match1-T1中,菌落PCR检测,提取阳性菌落质粒。利用检测引物NOSF/M13F-47(表1)对载体质粒进行PCR,产物送华大公司测序,得到检测无误的质粒通过热激法转入农杆菌EHA105中。
1.4 T0转基因植株的获得和检测
利用农杆菌介导法分别将2个pYLCRISPR/Cas9-Badh2-gRNA载体分别转入东农425水稻愈伤组织,经过潮霉素筛选,分化出T0再生植株,经过炼苗,移栽至温室种植。在分蘖盛期,采用CTAB法提取T0植株叶片DNA,利用Cas9检测引物Cas9-F/R(表1)进行PCR检测,PCR产物大小为572 bp,电泳结果为572 bp的植株为阳性转基因植株。
1.5 Badh2基因靶点序列的测序分析
由于第一个载体的2个靶点相距较远(1 062 bp),因此,在2个靶点附近各设计了1对PCR引物,Seq(1)-T1-F/R和Seq(1)-T2-F/R(表1),PCR产物理论大小为314,1 027 bp,而第2个载体的2个靶点相距较近(48 bp),因此,针对第1个靶点的上游和第2个靶点的下游位置仅设计了1对PCR引物,Seq(2)-T-F/R(表1),PCR产物理论大小为823 bp,将PCR产物送华大公司进行测序,测序结果利用刘耀光课题组的DSDecode解码方法(http://dsdecode.scgene.com/)进行分析[30-31],挑选纯合植株留种继续种植。
1.6 无T-DNA元件Badh2突变体的筛选及其香味检测
T1纯合的突变体植株和东农425野生型植株在相同的条件下种植,采用CTAB法提取T1植株叶片DNA,利用Cas9检测引物(表1)进行PCR检测,不能扩增出目的条带的植株是无T-DNA元件的纯合突变植株。在成熟期,选取所有无T-DNA元件纯合株系的单株和野生型植株进行香味检测,本研究采用2种方法对突变体材料进行香味检测:①咀嚼法。5人小组进行鉴定,取少量成熟的籽粒先进行咀嚼随后吸气,判断籽料香味的浓郁程度[32];②氢氧化钾浸泡法。对成熟的籽粒进行去壳处理,研碎后称取2 g放入带盖的培养皿中,加入10 mL 1.7%的KOH溶液,盖上培养皿盖保持密封,室温浸泡15 min,打开培养皿盖5人小组鉴定其香味浓郁程度。5人鉴定小组按香味的浓郁程度进行评级,等级分为0~3(0:无香味,1:淡香,2:香,3:浓香),2种方法所得的香味等级取平均数。
1.7 T1植株农艺性状考察
在T1于成熟期对筛选出的香味突变植株和野生型植株的主要农艺性状进行考察,包括株高、穗数、穗粒数、结实率和千粒质量。
2 结果与分析
2.1 东农425野生型植株的Badh2基因检测及靶点设计
Badh2基因由15个外显子组成(图1),对受体材料东农425的Badh2基因进行测序分析,结果发现,其CDS序列与日本晴相同,未发生突变。
本研究设计的第1个载体的2个靶点分别在第2和第3外显子上,第2个载体的2个靶点均在第2外显子上(图2)。第1个载体的2个靶点相距1 062 bp,第2个载体的2个靶点相距48 bp。
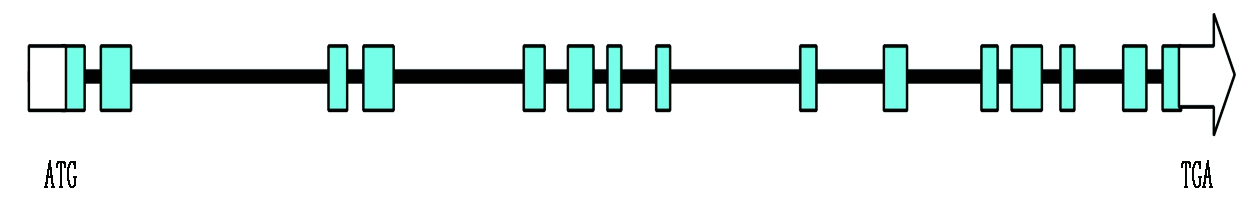
图1 Badh2基因结构
Fig.1 Badh2 structure
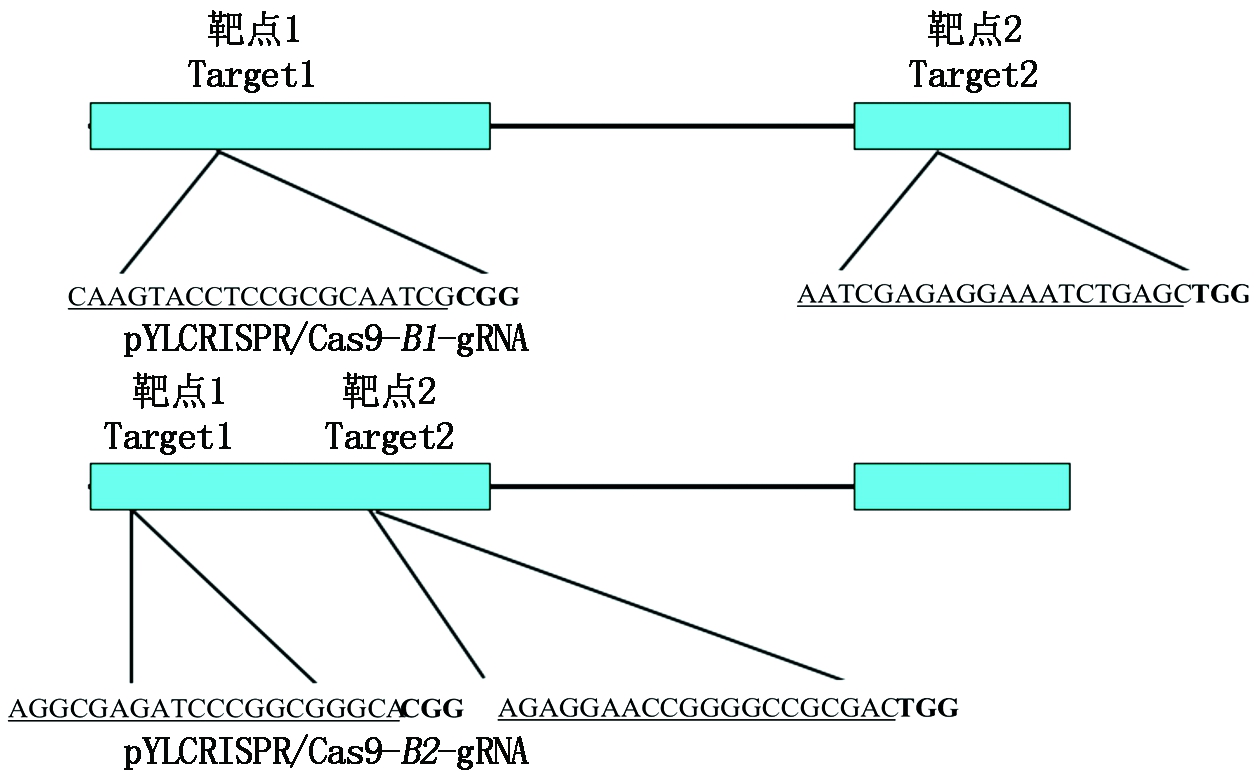
下划线序列为靶点序列,加粗序列为PAM序列。
The underlined sequence is the target sequence and the bold sequence is the PAM sequence.
图2 两个载体gRNA靶点信息
Fig.2 gRNA target information of two vectors
2.2 Badh2基因表达载体的构建
利用PCR和“金门”克隆法将带有靶点的gRNA连接到Cas9载体骨架上,构建好的载体即为pYLCRISPR/Cas9-B1-gRNA和pYLCRISPR/Cas9-B2-gRNA载体(图3),以下简称B1和B2载体。利用AscⅠ酶切鉴定载体质粒(图4),载体质粒被切出1.1 kb,2个载体的酶切结果均符合预期条带大小,表明2个靶点的gRNA表达盒顺利连接到pYLCRISPR/Cas9Pubi-H载体上。为了进一步确定各靶点序列的准确性,利用gRNA表达盒通用引物NOSF/M13F-47(表1)对2个载体上的靶点进行PCR测序分析。结果表明,测序的各靶点序列与设计的靶点序列一致(图5),所构建的2个载体可以进行下一步的水稻遗传转化试验。
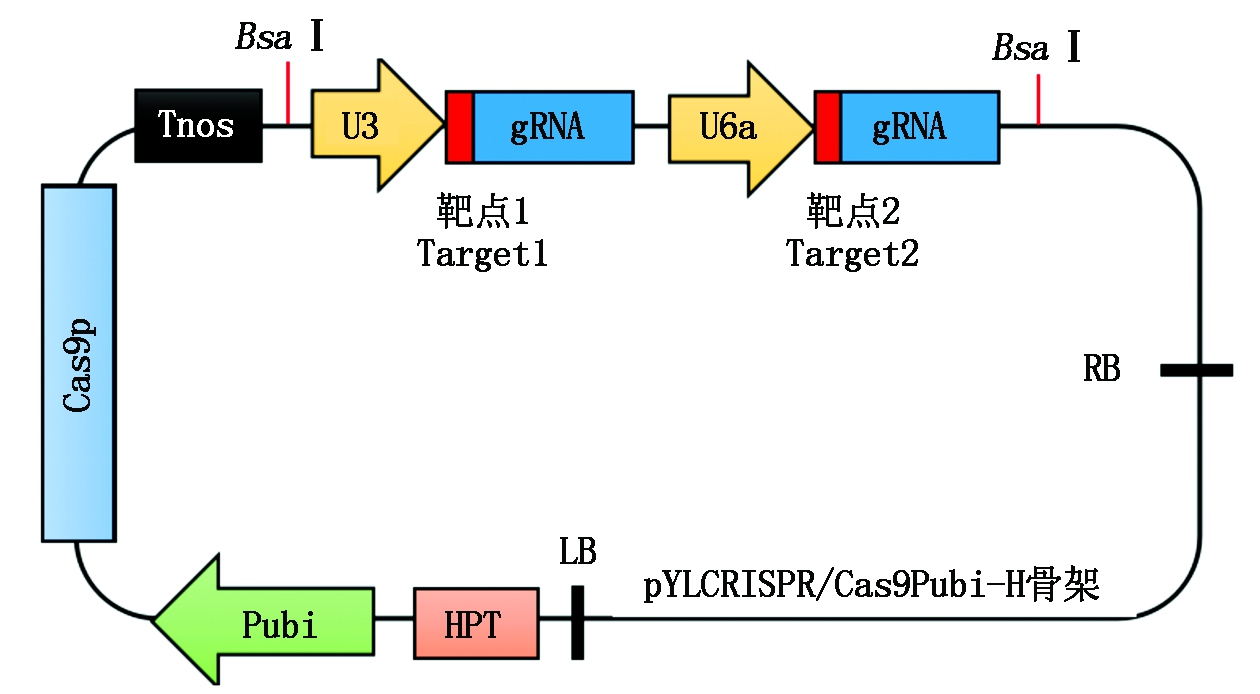
图3 两个pLYCRISPR/Cas9-Badh2-gRNA载体的组装图
Fig.3 The structure of two pLYCRISPR/Cas9-Badh2-gRNA vectors
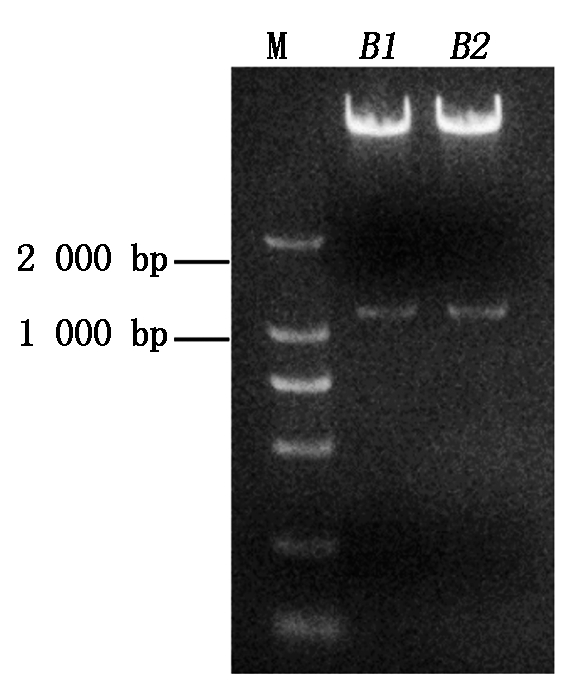
M.DM2000 DNA标记;B1、B2.构建好的
pLYCRISPR/Cas9-Badh2-gRNA载体。
M.DM2000 DNA Marker; B1,B2. pLYCRISPR/Cas9-Badh2-gRNA vectors.
图4 两个pYLCRISPR/Cas9-Badh2-gRNA载体的AscⅠ酶切检测
Fig.4 Identification of the two pYLCRISPR/Cas9-Badh2-gRNA plasmids digested with AscⅠ
2.3 T0转基因植株检测
分别用带有B1载体和B2载体的农杆菌侵染东农425,各获得18株和19株再生植株,移栽后,在分蘖盛期利用CTAB法提取这37株叶片DNA,用Cas9检测引物Cas9-F和Cas9-R鉴定Cas9载体元件是否整合到再生植株的基因组上,最终共筛选出32株转基因阳性植株,B1载体的阳性植株有15株,命名为B1-#,B2载体的阳性植株有17株,命名为B2-#。利用设计好的特异性引物Seq(1)-T1-F/R、Seq(1)-T2-F/R和Seq(2)-T-F/R对阳性植株进行测序分析,结果表明,B1载体纯合植株有5个,纯合率为33.33%,B2载体的纯合植株有9个,纯合率为52.94%。可以看出,B2载体的T0阳性植株纯合率更高。通过刘耀光课题组的DSDecode解码方法(http://dsdecode.scgene.com/),分析纯合突变体的具体碱基突变类型(图6),在T0共获得8种突变类型的植株,其中B1-2和B1-8、B1-9和B1-10的基因型完全一致,B2-12和B2-17、B2-6和B2-9以及B2-10和B2-11的基因型完全一致。B1载体的植株多为1 bp碱基的插入/缺失,而B2载体的植株出现了67,76 bp的大片段碱基缺失,详细情况见图6。
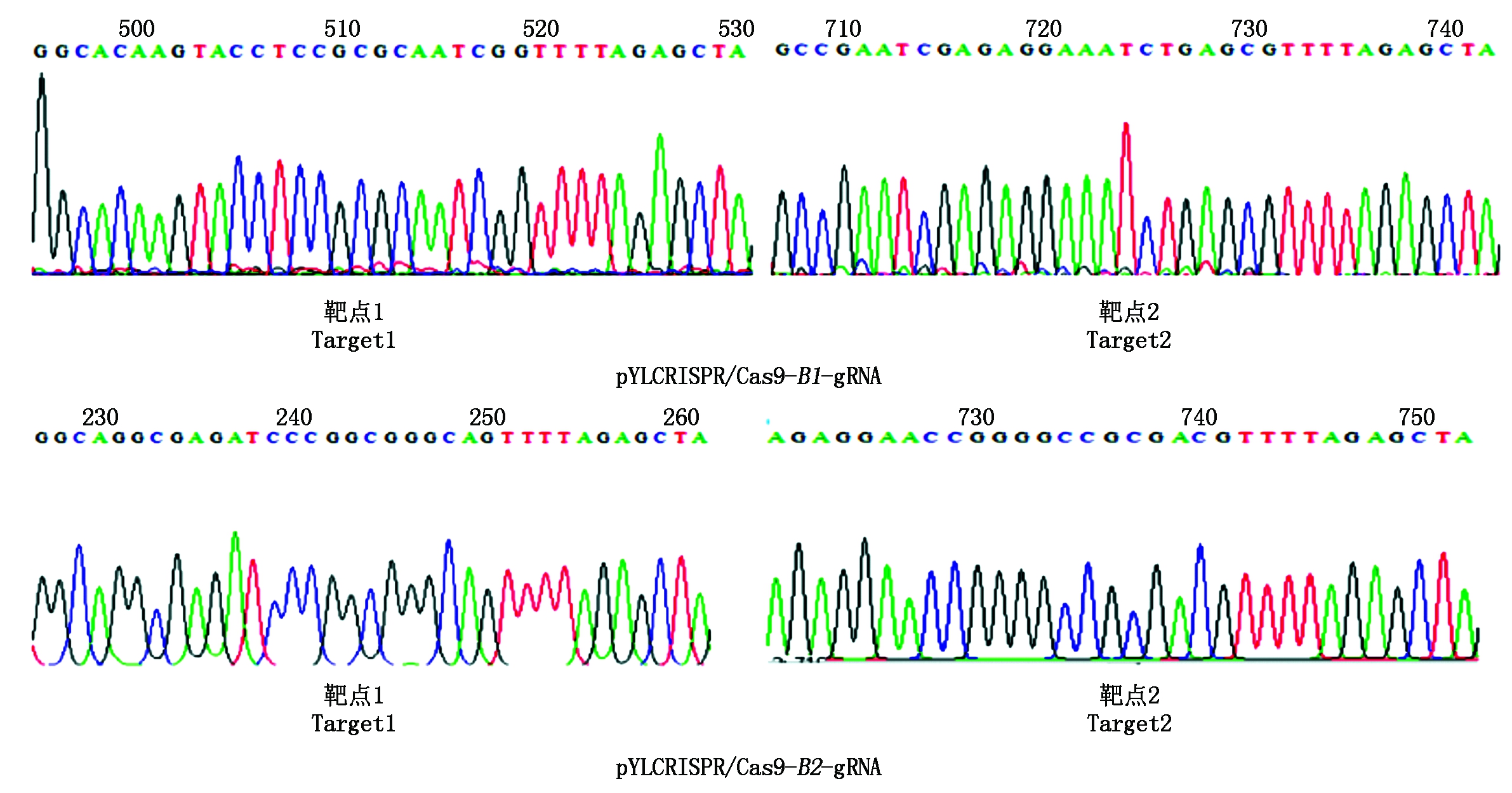
图5 两个pLYCRISPR/Cas9-Badh2-gRNA载体的测序结果
Fig.5 Sequencing results for the two pYLCRISPR/Cas9-Badh2-gRNA plasmids

下划线的碱基是靶点序列,省略的序列用连续小黑点表示,缺失的碱基用黑色连字符表示,插入的碱基用小写字母表示。WT.野生型。
The base with underline is the target sequence, the omitted sequences are shown by consecutive small black dots, the deleted bases are represented by black hyphens, the inserted bases are represented by lowercase letters. WT.Wild-type.
图6 T0纯合植株突变类型分析
Fig.6 Mutation types of homozygous mutants in T0 generation
2.4 无T-DNA元件的Badh2突变体的检测及香味鉴定
B1载体T0的纯合突变体有3个基因型不同的株系,即B1-2/8、B1-9/10和B1-12,将其重新命名为B1-M1、B1-M2和B1-M3。B2载体T0的纯合突变体有5个基因型不同的株系,即B2-1、B2-2/12/17、B2-3、B2-6/9和B2-10/11,将其重新命名为B2-M1、B2-M2、B2-M3、B2-M4和B2-M5。2个载体的8个T0纯合株系和野生型株系种植在相同条件下,获得T1株系,在分蘖盛期每个株系各选取30个T1单株提取叶片DNA,用Cas9-F/R引物进行PCR检测,图7为部分电泳结果图,没有扩增出目的条带的植株为不含有转基因元件的突变植株。B1载体的3个株系共筛选出26株无T-DNA元件的纯合植株,B2载体的5个株系共筛选出39株无T-DNA元件的纯合植株,载体的分离基本符合孟德尔遗传规律。
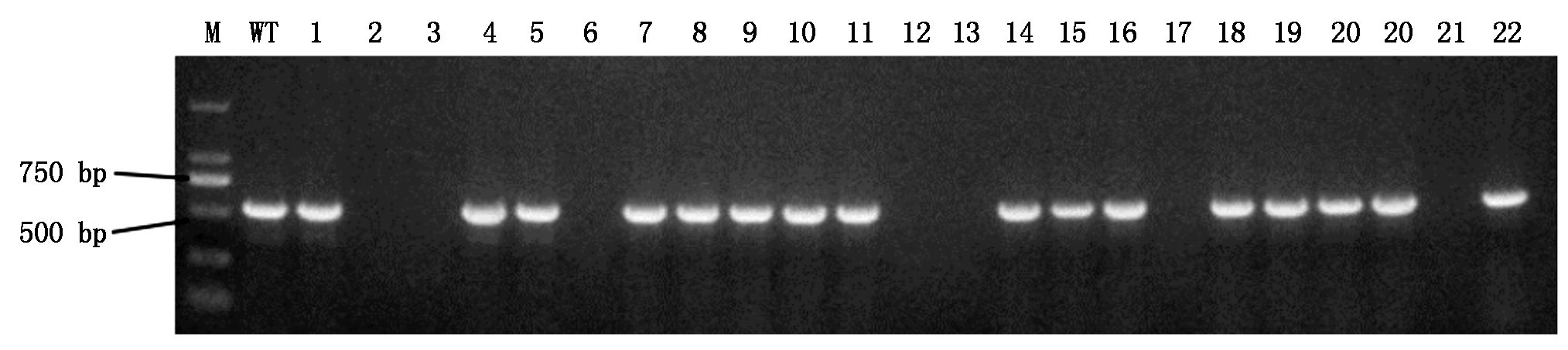
M.DM2000 DNA标记;WT.野生型;1-22.T1转基因植株。
M.DM2000 DNA Marker; WT.Wild-type; 1-22.T1 transgenic plants.
图7 部分Badh2突变体的T-DNA元件PCR检测
Fig.7 PCR identification for T-DNA of parts of Badh2 mutants
在成熟期,采用咀嚼法和氢氧化钾浸泡法对8个无T-DNA元件的纯合株系进行香味检测,同时对香味进行评级分析,2种方法所得等级取平均数(表2)。结果表明,所有纯合突变株系均有不同程度的香味,有5个株系的香味等级大于2.5,表现为浓香;有2个株系表现为香;有1个株系的香味等级小于1.5,为淡香。其中B1载体的3个株系均为浓香,B2载体的B2-M1和B2-M4株系为浓香,B2-M2和B2-M3株系为香,B2-M5株系为淡香。总体上,B1载体株系的香味比B2载体株系的香味更为浓郁。
2.5 香味突变材料的主要农艺性状考察
在T1进一步对8个香味突变株系和野生型材料的株高、穗数、穗粒数、结实率和千粒质量进行了考察(图8)。结果表明,Badh2基因成功编辑的后代除了株高与东农425有显著差异外,其余性状均无明显差异。本研究创建的Badh2纯合突变体的主要农艺性状并未因Badh2基因的编辑而受到影响。
表2 东农425野生型和纯合突变植株的香味分析
Tab.2 Fragrance analysis of Dongnong 425 wild-type and homozygous mutant plants
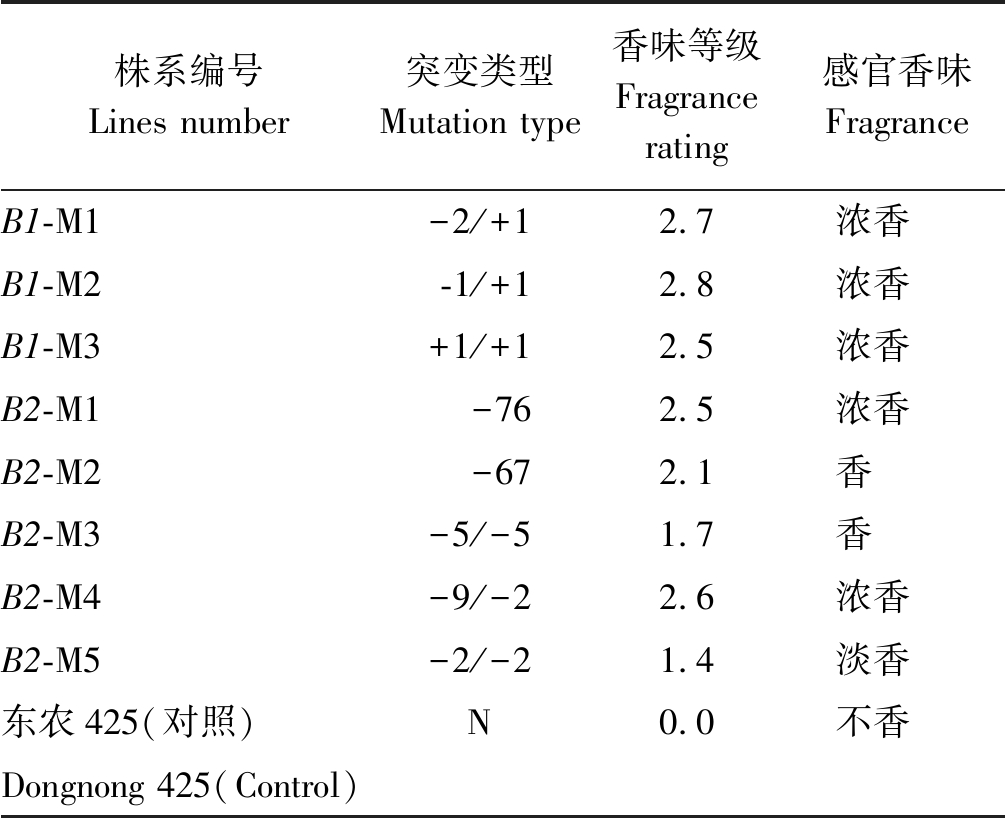
株系编号Lines number突变类型Mutation type香味等级Fragrancerating感官香味FragranceB1-M1-2/+12.7浓香B1-M2-1/+12.8浓香B1-M3+1/+12.5浓香B2-M1-762.5浓香B2-M2-672.1香B2-M3-5/-51.7香B2-M4-9/-22.6浓香B2-M5-2/-21.4淡香东农425(对照)N0.0不香Dongnong 425(Control)
注:+.插入碱基;-.缺失碱基;N.未发生突变。
Note:+. A inserted base;-. A deleted base;N. No mutation.
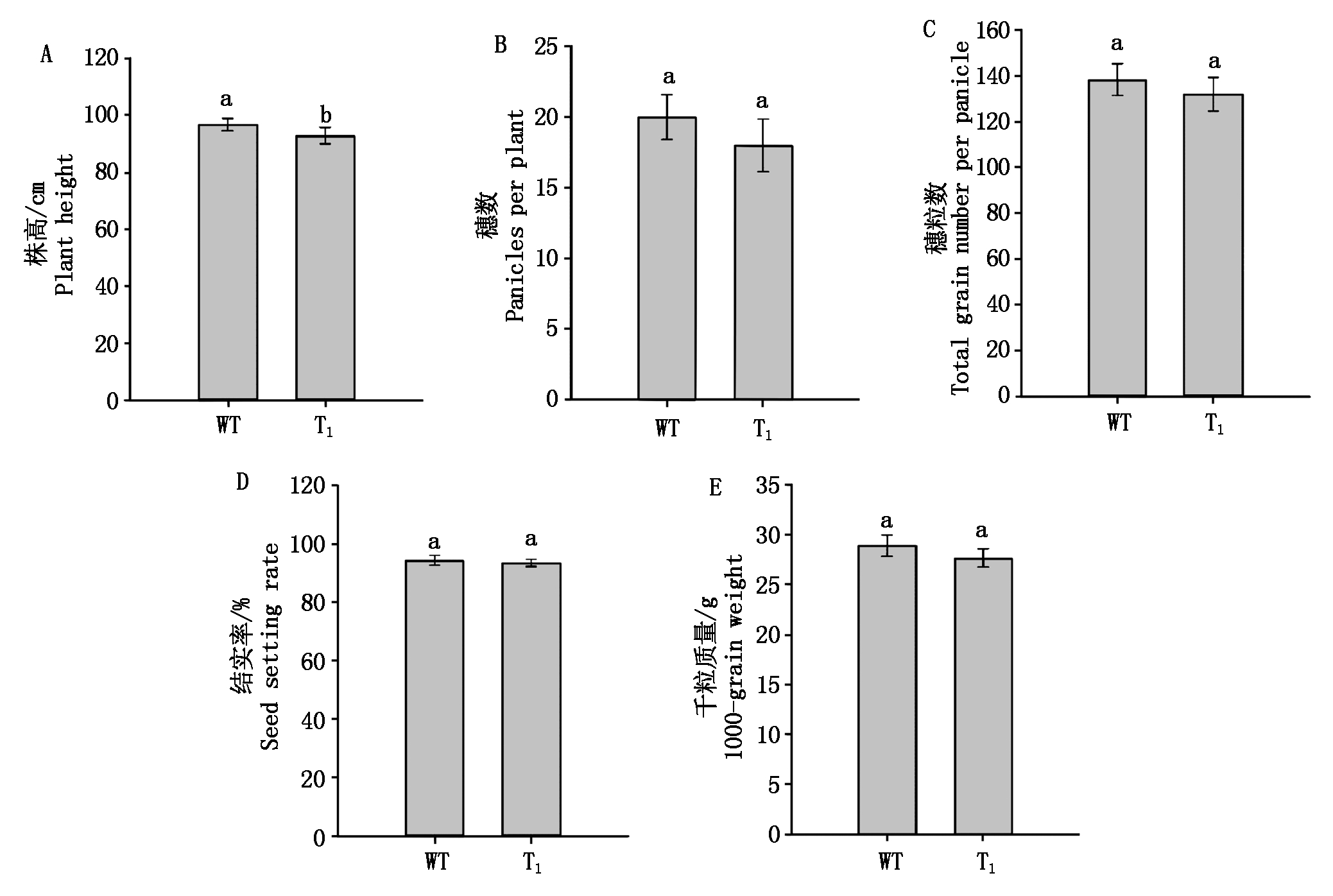
WT.野生型;T1.T1无T-DNA元件植株;数值用平均数±标准差表示,不同的小写字母表示在0.05水平上差异显著(t检验)。
WT.Wild-type; T1.Plants without T-DNA component in T1 generation; Values are shown as mean±SD.
Different lowercase letters present significant difference at P<0.05 level by t-test.
图8 水稻产量在野生型及突变体材料中的表现
Fig.8 Performance of rice yield in Dongnong 425 and mutants
3 结论与讨论
香稻不仅有独特的味道和良好的口感,而且具有很高的商业价值,市场占有率很高,一般价格是非香型大米的2~3倍,所以近年来成为育种家们的研究热点。传统的香稻育种大多是通过杂交和回交选育香稻品种,但因其后代分离不稳定、试验周期漫长、抗逆性较弱、产量较低等因素,所以培育出稳定遗传的优良香稻品种较为困难,难以打破原有遗传背景的束缚[33]。CRISPR/Cas9基因编辑技术,与传统育种技术相比,更加快捷、高效、稳定,有效地弥补了传统育种技术的不足。
虽然CRISPR/Cas9系统存在脱靶效应,但靶点越多脱靶可能性才越大[34],另外,Tang等[35]的研究表明,通过设计高特异性的靶序列就可以有效避免脱靶效应,本研究所构建的2个载体,分别具有2个靶点,且选择的均是高特异性的靶点,因此脱靶效应可以忽略不计。
在T0成功获得了各种类型的Badh2突变体,突变类型以纯合突变和双等位突变为主,2个载体的T0植株纯合率分别为33.33%,52.94%,B2载体2个靶点相距仅为48 bp,所以突变体的基因型出现了大片段缺失的情况,笔者推测正是由于碱基大片段缺失的出现,导致了植株纯合概率的增加。杂合突变和双等位突变的后代会发生功能减弱和无义突变的分离,影响了后代遗传的稳定性,而纯合突变体的后代不会分离,所以在T0只选择了纯合突变体继续播种。在T1对纯合突变体的T-DNA元件进行PCR检测,结果表明,T-DNA元件的后代分离符合孟德尔定律,因此,通过T0纯合突变植株的自交,就可以在T1获得不含转基因成分的纯合植株。这些纯合植株并未携带T-DNA元件,可直接应用于水稻育种研究,培育新的香型水稻材料,加速了香型粳稻品种的选育进程。
香味的测定有多种方式,评价起来比较主观,只用一种方法可能不是很准确,所以本研究应用了2种方便快捷的香味检测方式,对野生型和无T-DNA元件的纯合突变体进行香味分析,Badh2的功能丧失型突变体具有不同程度的香味,达到了对非香型粳稻东农425改良的目的,其中B1载体的突变体香味普遍要比B2载体的突变体更浓郁。B1载体同时对Badh2的2个外显子进行了编辑,所以笔者推测,对Badh2基因多个外显子的同时编辑,可能会造成更显著的香味表型。为了了解转基因植株后代的其他表型是否会受到影响,本研究对无转基因元件突变体的主要农艺性状进行了考察,Badh2基因成功编辑的后代除了株高与东农425有显著差异外,其余性状均无明显差异,在编辑香味基因的同时,并没有影响除株高以外的其他性状。最终,本研究成功地利用CRISPR/Cas9技术对Badh2基因进行了定点编辑,并且获得了不含有转基因成分的香型水稻材料,为粳稻香味的研究提供了丰富的材料,且为加快香型粳稻的培育开辟了新途径。
[1] 俞桂林. 香米的遗传特点及营养品质[J]. 浙江农业科学, 1987(3): 113-115. doi:10.16178/j.issn.0528-9017.1987.03.004.
Yu G L. Genetic characteristics and nutritional quality of fragrant rice[J]. Journal of Zhejiang Agricultural Sciences, 1987(3): 113-115.
[2] 吕艳梅, 谭伟平, 肖层林. 稻米香味研究综述[J]. 湖南农业科学, 2010(11): 19-20, 23. doi: 10.3969/j.issn.1006-060X.2010.11.007.
Lü Y M, Tang W P, Xiao C L. Summarization of the scent of scented rice[J]. Hunan Agricultural Sciences, 2010(11): 19-20, 23.
[3] 黄晓群, 张淑华, 赵海新, 刘传雪, 张兰民, 王瑞英, 关世武, 王翠. 黑龙江省水稻品种现状分析及研发对策[J]. 黑龙江农业科学, 2009(6): 40-43. doi: 10.3969/j.issn.1002-2767.2009.06.014.
Huang X Q, Zhang S H, Zhao H X, Liu C X, Zhang L M, Wang R Y, Guan S W, Wang C. Analysis conditions of rice varieties and tactics of study in Heilongjiang Province[J]. Heilongjiang Agricultural Sciences, 2009(6): 40-43.
[4] Chen S H, Yang Y, Shi W W, Ji Q, He F, Zhang Z D, Cheng Z K, Liu X N, Xu M L. Badh2, encoding betaine aldehyde dehydrogenase, inhibits the biosynthesis of 2-acetyl-1-pyrroline, a major component in rice fragrance[J]. Plant Cell, 2008, 20(7): 1850-1861. doi: 10.1105/tpc.108.058917.
[5] Buttery R G, Ling L C, Juliano B O, Turnbaugh J G. Cooked rice aroma and 2-acetyl-1-pyrroline[J]. Journal of Agricultural and Food Chemistry, 1983, 31(4): 823-826. doi: 10.1021/jf00118a036.
[6] Vanavichit A, Yoshihashi T. Chapter 2-Molecular aspects of fragrance and aroma in rice[J]. Advances in Botanical Research, 2010, 56: 49-73. doi: 10.1016/B978-0-12-381518-7.00002-9.
[7] Shao G N, Tang A, Tang S Q, Luo J, Jiao G A, Wu J L, Hu P S. A new deletion mutation of fragrant gene and the development of three molecular markers for fragrance in rice[J]. Plant Breeding, 2011, 130(2): 172-176. doi: 10.1111/j.1439-0523.2009.01764.x.
[8] Shao G N, Tang S Q, Chen M L, Wei X J, He J W, Luo J, Jiao G A, Hu Y C, Xie L H, Hu P S. Haplotype variation at Badh2, the gene determining fragrance in rice[J]. Genomics, 2013, 101(2): 157-162. doi: 10.1016/j.ygeno.2012.11.010.
[9] Shan Q W, Zhang Y, Chen K L, Zhang K, Gao C X. Creation of fragrant rice by targeted knockout of the OsBADH2 gene using TALEN technology[J]. Plant Biotechnology Journal, 2015, 13(6): 791-800. doi: 10.1111/pbi.12312.
[10] Bibikova M, Beumer K, Trautman J K, Carroll D. Enhancing gene targeting with designed zinc finger nucleases[J]. Science, 2003, 300(5620): 764. doi: 10.1126/science.1079512.
[11] Bedell V M, Wang Y, Campbell J M, Poshusta T L, Starker C G, Krug R G, Tan W F, Penheiter S G, Ma A C, Leung A Y H, Fahrenkrug S C, Carlson D F, Voytas D F ,Clark K J, Essner J J, Ekker S C. In vivo genome editing using a high-efficiency TALEN system[J]. Nature, 2012, 491(7422): 114-118. doi: 10.1038/nature11537.
[12] Doudna J A, Charpentier E. The new frontier of genome engineering with CRISPR-Cas9[J]. Science, 2014, 346(6213): 1258096. doi: 10.1126/science.1258096.
[13] Smih F, Rouet P, Romanienko P J, Jasin M. Double-strand breaks at the target locus stimulate gene targeting in embryonic stem cells[J]. Nucleic Acids Research, 1995, 23(24): 5012-5019. doi: 10.1093/nar/23.24.5012.
[14] Gaj T, Gersbach C A, Barbas C F. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering[J]. Trends in Biotechnology, 2013, 31(7): 397-405. doi: 10.1016/j.tibtech.2013.04.004.
[15] Wang H Y, Yang H, Shivalila C S, Dawlaty M M, Cheng A W, Zhang F, Jaenisch R. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering[J]. Cell, 2013, 153(4): 910-918. doi: 10.1016/j.cell.2013.04.025.
[16] Auer T O, Del Bene F. CRISPR/Cas9 and TALEN-mediated knock-in approaches in zebrafish[J]. Methods, 2014, 69(2): 142-150. doi: 10.1016/j.ymeth.2014.03.027.
[17] Mali P, Yang L H, Esvelt K M, Aach J, Guell M, DiCarlo J E, Norville J E, Church G M. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339(6121): 823-826. doi: 10.1126/science.1232033.
[18] Feng Z Y, Zhang B T, Ding W N, Liu X D, Yang D L, Wei P L, Cao F Q, Zhu S H, Zhang F, Mao Y F, Zhu J K. Efficient genome editing in plants using a CRISPR/Cas system[J]. Cell Research, 2013, 23(10): 1229-1232. doi: 10.1038/cr.2013.114.
[19] Wang Y P, Cheng X, Shan Q W, Zhang Y, Liu J X, Gao C X, Qiu J L. Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew[J]. Nature Biotechnology, 2014, 32(9): 947-951. doi: 10.1038/nbt.2969.
[20] Liang Z, Zhang K, Chen K L, Gao C X. Targeted mutagenesis in Zea mays using TALENs and the CRISPR/Cas system [J]. Journal of Genetics and Genomics, 2014, 41(2): 63. doi: 10.1016/j.jgg.2013.12.001.
[21] Fan D, Liu T T, Li C F, Jiao B, Li S, Hou Y S, Luo K M. Efficient CRISPR/Cas9-mediated targeted mutagenesis in populus in the first generation[J]. Scientific Reports, 2015, 5: 12217. doi: 10.1038/srep12217.
[22] Miao J, Guo D S, Zhang J Z, Huang Q P, Qin G J, Zhang X, Wan J M, Gu H Y, Qu L J. Targeted mutagenesis in rice using CRISPR-Cas system[J]. Cell Research, 2013, 23(10): 1233-1236. doi: 10.1038/cr.2013.123.
[23] Feng Z Y, Zhang B T, Ding W N, Liu X D, Yang D L, Wei P L, Cao F Q, Zhu S H, Zhang F, Mao Y F, Zhu J K. Efficient genome editing in plants using a CRISPR/Cas system[J]. Cell Research, 2013, 23(10): 1229-1232. doi: 10.1038/cr.2013.114.
[24] Zhang H, Zhang J S, Wei P L, Zhang B T, Gou F, Feng Z Y, Mao Y F, Yang L, Zhang H, Xu N F. The CRISPR/Cas9 system produces specific and homozygous targeted gene editing in rice in one generation[J]. Plant Biotechnology Journal, 2014, 12(6): 797-807. doi: 10.1111/pbi.12200.
[25] Shan Q W, Wang Y P, Li J, Zhang Y, Chen K L, Liang Z, Zhang K, Liu J X, Xi J J, Qiu J L, Cao C X. Targeted genome modification of crop plants using a CRISPR-Cas system[J]. Nature Biotechnology, 2013, 31(8): 686-688. doi: 10.1038/nbt.2650.
[26] Li M R, Li X X, Zhou Z J, Wu P Z, Fang M C, Pan X P, Lin Q P, Luo W B, Wu G J, Li H Q. Reassessment of the four yield-related genes Gn1a, DEP1, GS3, and IPA1 in rice using a CRISPR/Cas9 system[J]. Frontiers in Plant Science, 2016, 7(12217): 377. doi: 10.3389/fpls.2016.00377.
[27] 王加峰, 郑才敏, 刘维, 罗文龙, 王慧, 陈志强, 郭涛. 基于CRISPR/Cas9技术的水稻千粒质量基因tgw6突变体的创建[J]. 作物学报, 2016, 42(8): 1160-1167. doi: 10.3724/SP.J.1006.2016.01160.
Wang J F, Zheng C M, Liu W, Luo W L, Wang H, Cheng Z Q, Guo T. Construction of tgw6 mutants in rice based on CRISPR/Cas9 technology[J]. Acta Agronomica Sinica, 2016, 42(8): 1160-1167.
[28] Zeng D C, Ma X L, Xie X R, Zhu Q L, Liu Y G. A protocol for CRISPR/Cas9-based multi-gene editing and sequence decoding of mutant sites in plants[J]. Scientia Sinica, 2018,48(7):783-794. doi: 10.1360/N052018-00069.
[29] Ma X L, Zhang Q Y, Zhu Q L, Liu W, Chen Y, Qiu R, Wang B, Yang Z F, Li H Y, Lin Y R, Xie Y Y, Shen R X, Chen S F, Wang Z, Chen Y L, Guo J X, Chen L T, Zhao X C, Dong Z C, Liu Y G. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants[J]. Molecular Plant, 2015, 8(8): 1274-1284. doi: 10.1016/j.molp.2015.04.007.
[30] Xie X R, Ma X L, Zhu Q L, Zeng D C, Li G S, Liu Y G. CRISPR-GE: A convenient software toolkit for CRISPR-Based genome editing[J]. Molecular Plant, 2017, 10(9): 1246-1249. doi: 10.1016/j.molp.2017.06.004.
[31] Liu W Z, Xie X R, Ma X L, Li J, Chen J H, Liu Y G. DSDecode: A web-based tool for decoding of sequencing chromatograms for genotyping of targeted mutations[J]. Molecular Plant, 2015, 8(9): 1431-1433. doi: 10.1016/j.molp.2015.05.009.
[32] Dhulappanavar C V. Inheritance of scent in rice[J]. Euphytica, 1976, 25(1): 659-662. doi: 10.1007/BF00041603.
[33] 朱立宏. 关于我国水稻高产育种的我见[J]. 南京农业大学学报, 2007, 30(1): 129-135. doi: 10.3321/j.issn:1000-2030.2007.01.026.
Zhu L H. Some critical considerations on rice high-yielding breeding in China[J]. Journal of Nanjing Agricultural University, 2007, 30(1): 129-135.
[34] Endo M, Mikami M, Toki S. Multigene knockout utilizing off-target mutations of the CRISPR/Cas9 system in rice[J]. Plant and Cell Physiology, 2015, 56(1): 41-47.
[35] Tang X, Liu G Q, Zhou J P, Ren Q R, You Q, Tian L, Xin X H, Zhong Z H, Liu B L, Zheng X L, Zhang D W, Malzahn A, Gong Z Y, Qi Y P, Zhang T, Zhang Y. A large-scale whole-genome sequencing analysis reveals highly specific genome editing by both Cas9 and Cpf1 (Cas12a) nucleases in rice[J]. Genome Biology, 2018, 19(1): 84. doi: 10.1186/s13059-018-1458-5.