大白菜(Brassica rapa L. ssp. pekinensis)是十字花科芸薹属中的常见蔬菜,种植和分布面积广,是我国菜篮子中的重要蔬菜。近年来,由于芸薹根肿菌(Plasmodiophora brassicae Woron.)的侵染,大白菜根肿病频发,且在世界范围内的危害面积日益扩大,导致大白菜的品质和产量受到了极大影响,严重时甚至导致绝收,该病现已成为大白菜育种过程中亟须解决的问题[1]。由于根肿菌在土壤中的存活时间长,携带休眠孢子的土壤很容易导致病害的持续发生[2],而且利用农业、化学和生物等防治手段不能从根本上解决根肿病的防治问题,所以,培育抗根肿病的大白菜新品种是防治该病的最佳方法。
传统育种方法周期长,选择准确性差,极易受到外界环境的影响,造成病菌的传播。而利用与目标基因紧密连锁的分子标记对抗病性进行选择,不受植株生长发育时期和环境条件的限制,降低了误选目的基因的可能性,显著提高了目标性状选择的准确性和抗病育种工作的效率[3-7]。因此,本研究对抗根肿病基因进行初定位,筛选与抗根肿病基因连锁的分子标记,为分子标记辅助选择培育抗根肿病大白菜新品种提供技术参考。
1 材料和方法
1.1 试验材料
供试材料:感病亲本为津白56的高代自交系G4,抗病亲本为药膳春的高代自交系G6,G4和G6杂交获得的F1,F1自交构建的F2分离群体。所有试材均由天津科润蔬菜研究所大白菜课题组提供。
人工接种所用根肿病菌株采自湖北省恩施市火烧坪乡的大白菜根肿病发病肿根。
供试分子标记引物:共计680个,包括文献中已发表的与大白菜抗根肿病基因连锁的引物标记29个[8],王艳[9]构建的大白菜遗传图谱中的415个InDel和92个SSR标记引物,大白菜基因组数据库Brassica Database(http://brassicadb.org)中的144个SSR标记引物。以上引物均由华大基因合成。
1.2 试验方法
1.2.1 人工接种根肿菌与育苗培养 采用菌土法[10]进行人工接种,将带有根肿菌的肿根放入组织搅拌机内,再加入适量蒸馏水打成匀浆制成孢子悬浮液,然后和灭菌土混合,使菌土孢子含量达到108个/g[11-12],pH值调至6.5左右,含水量控制在60%[13]。
2016年3月,所有供试材料均播种于天津科润蔬菜研究所的温室内。抗、感亲本及F1各播种25株,F2群体播种500株。播种后苗盘置于温室通风阴凉处,温度控制在20~25 ℃,避免阳光长时间直晒,苗盘下保持1 cm水层,正常管理。
1.2.2 根肿病抗性鉴定与病情调查 2016年4月,在接菌培养30 d后,对亲本、F1和F2群体进行人工接种的抗病性鉴定。将每个单株从育苗盘里取出,用清水洗净根部后观察记录发病情况,并采集以上所有单株新鲜叶片2~3片,编号放入对应的自封袋中,-80 ℃保存,用于基因组DNA的提取。
大白菜苗期根肿病病情调查方法和分级标准参考王彤彤[14]和Piao等[15]的描述,按照0~7级标准调查单株发病情况(图1),0,1 级记为抗病,3,5,7级记为感病。根据以上鉴定结果,对其分离情况进行χ2检验,分析供试大白菜材料根肿病抗性遗传规律。

图1 人工接种抗性鉴定分级标准
Fig.1 Artificial inoculation identification classification standard
1.2.3 大白菜基因组DNA提取和质量检测 使用CTAB法[16]提取亲本、F1和F2大白菜植株叶片的基因组DNA,利用1%的琼脂糖凝胶电泳(电泳缓冲液为1×TAE)检测提取大白菜基因组DNA的质量,最后用蒸馏水将DNA原液稀释到50 ng/μL,-20 ℃保存备用。
1.2.4 PCR反应体系与抗根肿病基因连锁标记的筛选 本试验采用10 μL的PCR反应体系:基因组DNA(50 ng/μL)1 μL,正向引物(10 μmol/L)0.4 μL,反向引物(10 μmol/L)0.4 μL,PCR Mix(10 μg/mL) 5 μL,ddH2O 3.2 μL,总体积10 μL。
取基因组DNA样品1 μL进行PCR扩增。PCR扩增程序为94 ℃预变性5 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,35个循环;72 ℃延伸10 min。最后将扩增产物4 ℃保存。PCR扩增产物进行8%非变性聚丙烯酰胺凝胶电泳,快速银染法染色检测、照胶保存,统计多态性条带。
1.2.5 遗传图谱的构建与抗病基因的定位 整理统计所有分子标记在亲本、F1和F2单株中的扩增条带类型,筛选出连锁标记。根据单株抗病性表型和单株分子标记的基因型鉴定结果,利用JoinMap 4.0软件分析目的基因和分子标记的连锁关系,进行基因定位,设置LOD值为3.0,构建连锁图谱。
1.2.6 新定位抗根肿基因位点与Crr1的对比 由于新定位抗根肿基因位点与Crr1均位于A08染色体上,为验证二者是否为同一位点,同时在已有研究基础上,开发Crr1基因的多态性分子标记,笔者根据文献报道中Crr1基因的定位区间,对供试大白菜抗、感亲本材料Crr1基因定位区间内序列进行测序,比对测序结果(图2),同时结合大白菜数据库(http://brassicadb.org/brad/)基因组信息,在序列差异区域使用Primer 5.0软件设计了6对新的InDel分子标记(表1),并在F2群体中进行验证,绘制连锁图谱,筛选新的多态性标记,并分析新标记与新定位位点间的位置关系。

图2 抗、感亲本材料定位区域部分序列比对结果
Fig.2 Resistance and susceptible parental material localization region partial sequence alignment result
表1 新设计的InDel分子标记引物
Tab.1 New design InDel molecular marker primers

引物名称 Primer name上游引物序列Upstream primer base sequence下游引物序列Downstream primer base sequenceBrID10378TGTGGGAAGGAAATAAAGTAAGTACATCCATCCACTTGAGATTTCTAATCBrID10379GAGCTCCTAATGGAAATAAAAATGACATTAAGAAAAAGACGATCCTCAGCBrID10380CTCAAGCTGTGCTCAAGCCTTATGGCTTGAGATTGGTCATATTTCCAATABrID10381TTCTCAAGATTAGTCATATTACTAAGGATGTTCAAGTCTTATGGAGCTTCBrID10382ACTTGTAATCAATGGATGCACAAAGAACTTTGTCCTGTAAAAAGAGCABrID10383CGATTCAGTTTGAGGCAGTTAACAAACTTGTAATCAATGGATGCACAAAG
2 结果与分析
2.1 根肿病抗性鉴定结果和遗传学分析
根据根肿病抗性分级标准,对抗、感亲本、F1 以及160株F2分离群体进行抗病性鉴定(图3-4),G4全表现为感病,G6全表现为抗病,F1全表现为抗病,160株F2群体单株抗病性状发生分离,其中抗病植株118株,感病植株42株。经χ2检测,![]() 符合孟德尔遗传规律3∶1的性状分离比,说明该材料对根肿病的抗性受显性单基因控制。
符合孟德尔遗传规律3∶1的性状分离比,说明该材料对根肿病的抗性受显性单基因控制。

图3 感病、抗病亲本及F1抗病性鉴定结果
Fig.3 The parents,F1 generation of disease resistance identification result

图4 F2群体部分单株抗病性鉴定结果
Fig.4 Part of the plants in F2 populations result
2.2 分子标记筛选与抗根肿病基因定位
本研究先利用抗、感亲本和F1对文献中已经发表的与大白菜抗根肿病基因紧密连锁的29对分子标记,王艳[9]报道的均匀分布在大白菜10条染色体上的415对InDel标记和92对SSR标记,以及大白菜基因组数据库中的144对SSR引物标记进行多态性筛选,得到分布在10条染色体上的83对存在多态性的分子标记(图5)。
在F2群体中挑选24株极抗和24株极感单株组成48株极端表型群体,将83对多态性标记在48株极端表型群体中进行验证,对得到的连锁标记再进一步扩大F2群体进行验证,结果得到2个与抗病基因连锁的InDel分子标记(图6),分别为BrID10727和BrID10427,这2个分子标记都位于A08染色体上,初步判定试验材料所含抗根肿病基因位于大白菜A08染色体上。
查阅文献,寻找A08染色体上与抗病基因连锁的分子标记,经过在双亲和F1间进行多态性筛选和在F2群体中的进一步验证,又得到了3个与抗根肿基因连锁的InDel标记,分别为BrID10867、BrID11507和BrID11233,利用JoinMap 4.0软件构建遗传连锁图谱(图7)。
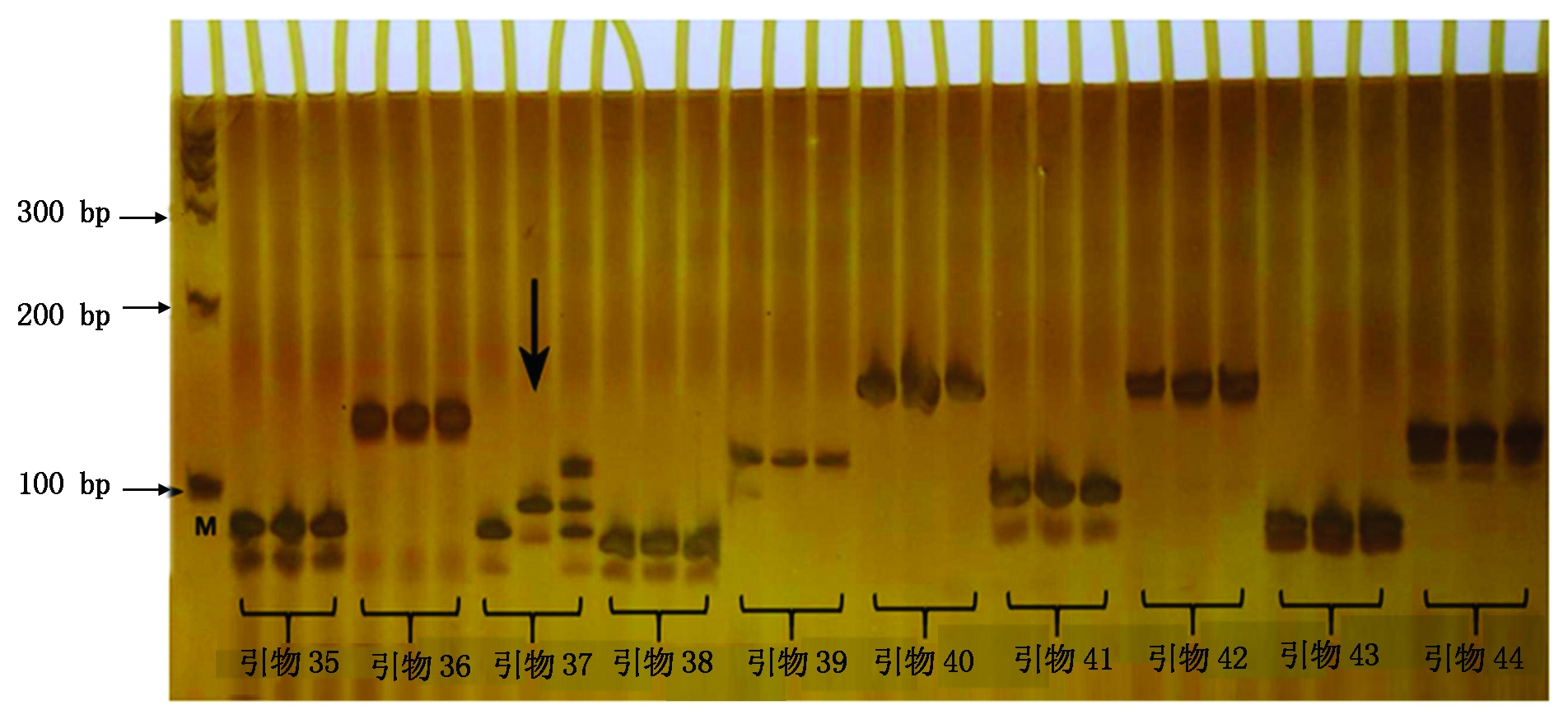
箭头所指为多态性分子标记的引物扩增条带。
The arrow indicates the polymorphism marker amplified bands.
图5 部分分子标记在亲本、F1间的多态性筛选试验结果
Fig.5 Screening of polymorphic markers between the partial parents and F1
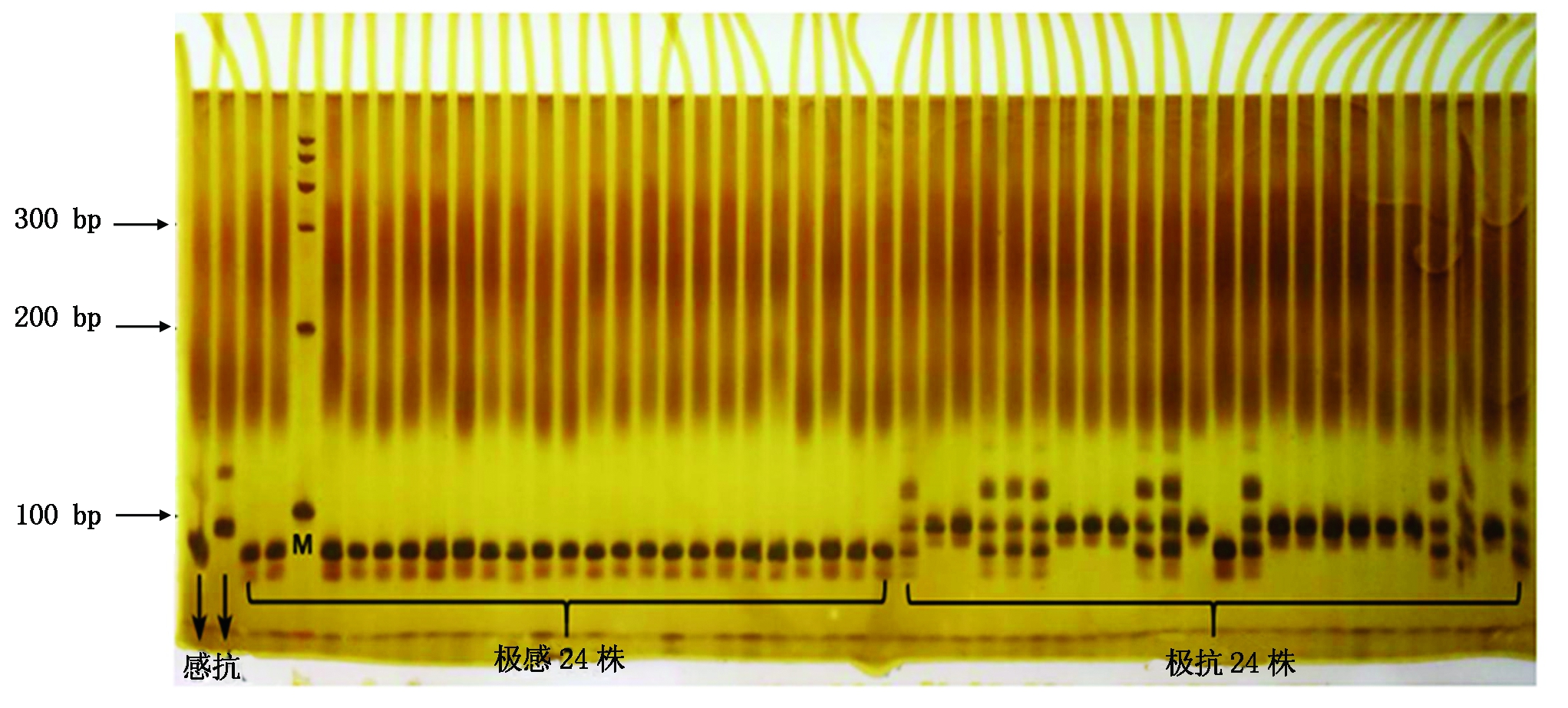
图6 多态性分子标记BrID10427在48株极端表型群体间的检测结果
Fig.6 Polymorphism markers BrID10427 between 48 extreme phenotypic groups test result
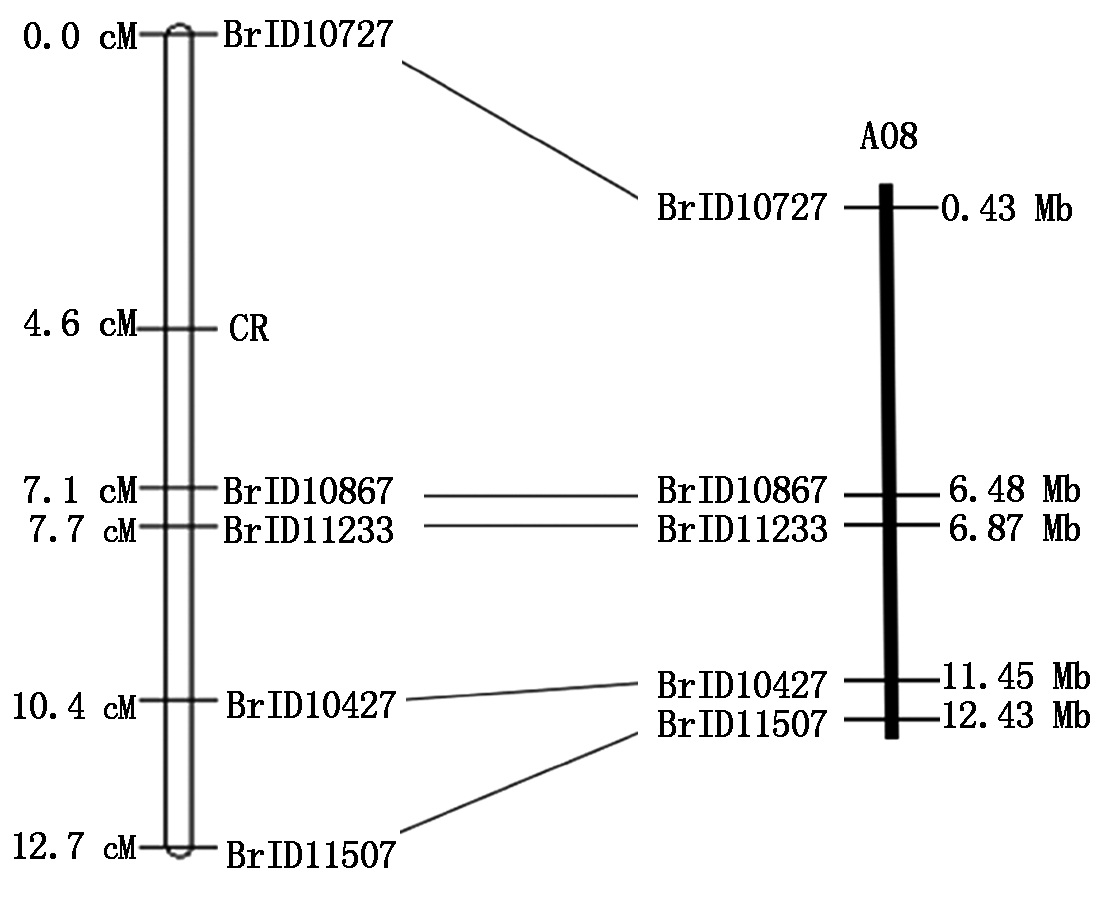
图7 多态性分子标记在遗传图谱和物理图谱中的位置
Fig.7 The location of polymorphic molecular markers in genetic maps and physical maps
结果显示,抗病基因被定位在BrID10727-BrID10867,与抗病基因的遗传距离分别为4.6,2.5 cM,通过将这2个标记的上下游引物序列与大白菜基因组序列信息进行比对后发现,这2个分子标记之间的物理区间为0.43~6.48 Mb。其余3个分子标记BrID11233、BrID10427和BrID11507与CR基因的遗传距离分别为3.1,5.8,8.1 cM,且都和BrID10867位于一侧。
2.3 新定位抗根肿基因位点与Crr1的对比
重复2.2中的步骤,将基于Crr1基因序列新设计的6个InDel标记在F2群体中进行筛选验证,获得1个与抗病基因连锁的多态性分子标记,即BrID10381(图8)。利用JoinMap 4.0分析数据并绘制遗传连锁图谱(图9)。结果显示,基于Crr1基因序列开发的InDel标记BrID10381与新定位抗根肿病基因位点的遗传距离为6.5 cM,该标记在物理图中的位置为11.61 Mb,在新定位抗根肿基因初定位区间的外侧,因此,可以推断新定位抗根肿基因位点与Crr1并非同一位点。
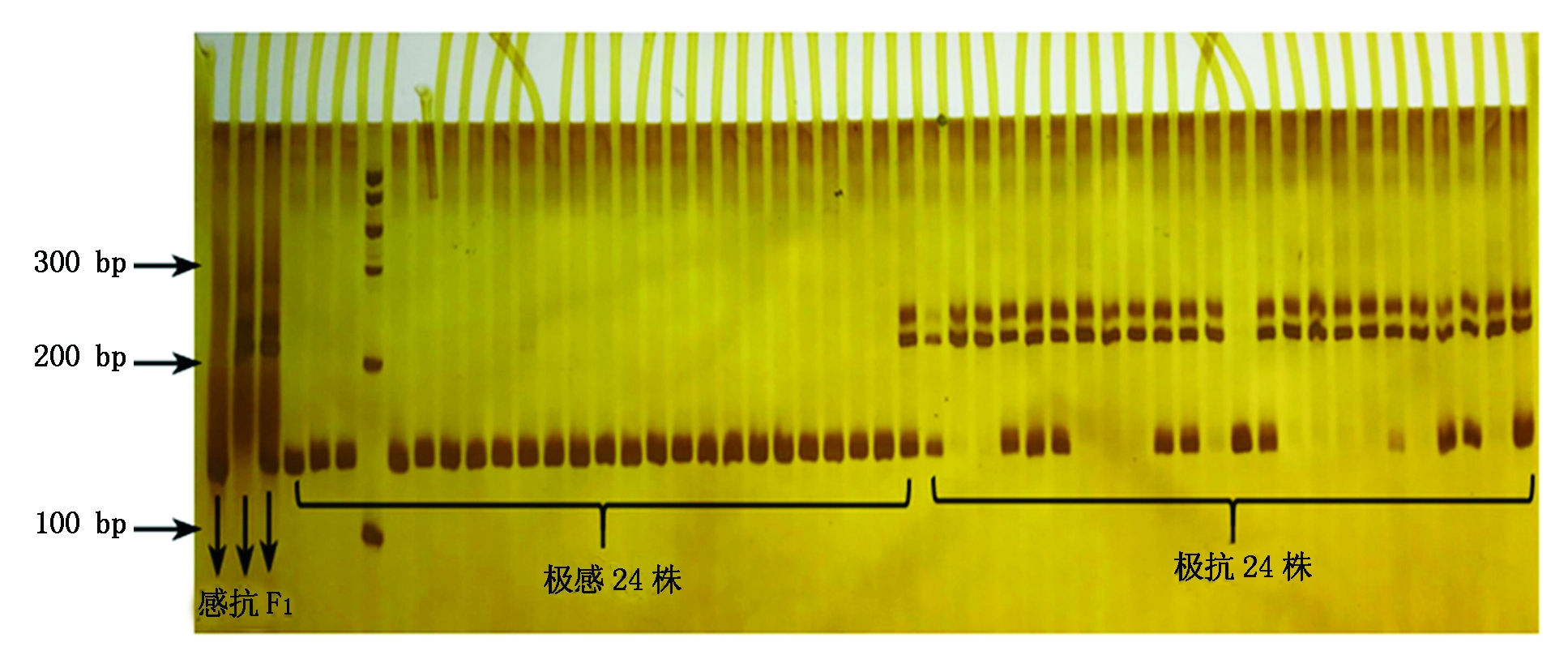
图8 新设计标记BrID10381在48株极端表型群体间的检测结果
Fig.8 New design marker BrID10381 between 48 extreme phenotypic groups test result
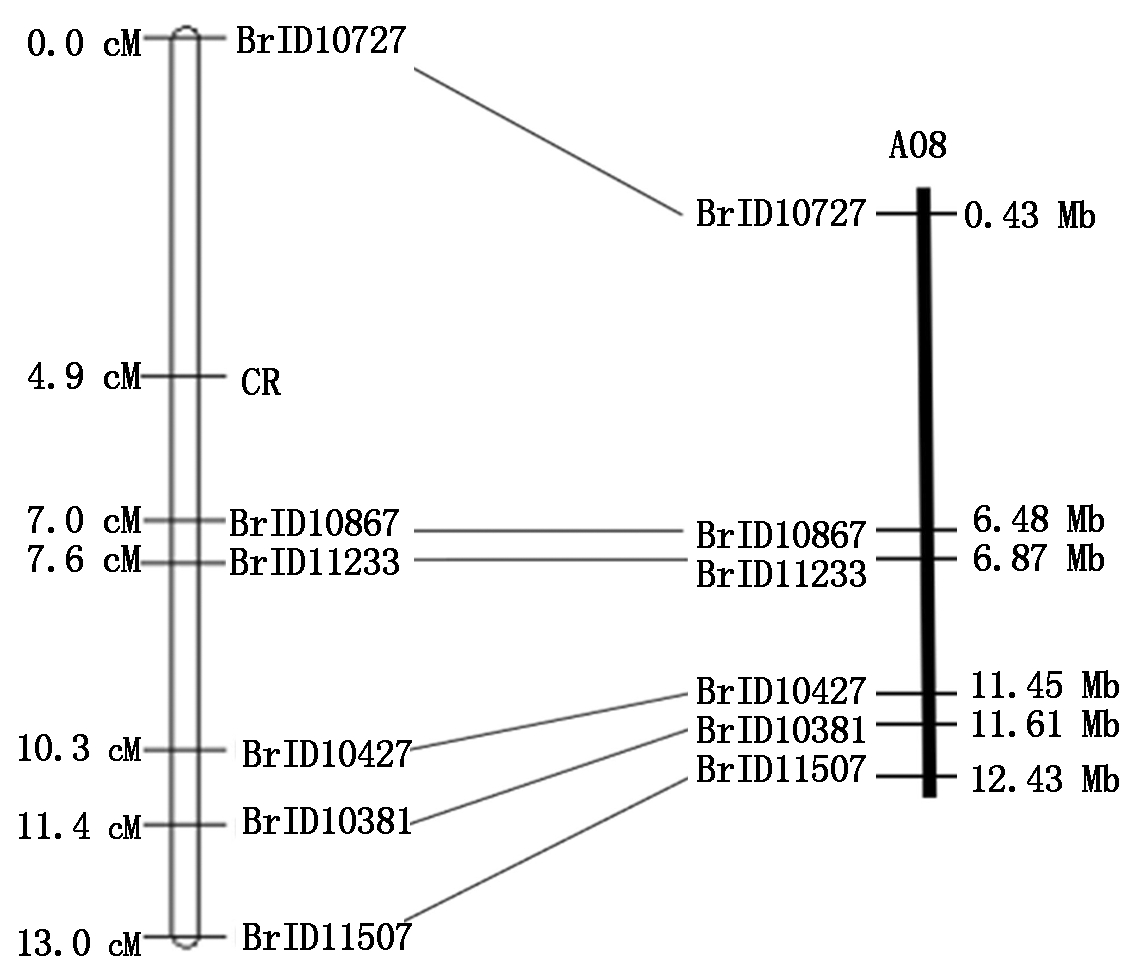
图9 标记BrID10381在遗传图谱和物理图谱中的位置
Fig.9 The location of BrID10381 in genetic maps and physical maps
3 结论与讨论
本研究采用人工接种鉴定的方法对供试抗、感亲本材料、F1和F2群体进行了根肿病抗性鉴定,经过分析发现,本试验材料中的根肿病抗性受显性单基因控制,与前人研究结果一致,Suwabe等[17]研究发现,大多用于研究的大白菜抗病材料中,根肿病抗性均是受单个显性主效基因控制,只有G004这一抗病材料中根肿病抗性是数量遗传,受多基因控制。
本研究将抗根肿病基因初步定位在了大白菜A08染色体上6.05 Mb范围内,2个侧翼分子标记的物理位置分别为0.43,6.48 Mb。已报道的8个抗根肿病基因分别被定位在5条染色体上,其中位于A08上的基因有一个,为Crr1[18-23]。虽然本研究中的抗根肿病基因与Crr1同样位于A08上,但与Crr1紧密连锁的SSR分子标记BRMS-088[24-25]在本研究供试群体中验证后不具有多态性,且经验证发现,基于Crr1基因序列新开发的标记BrID10381在物理图中的位置为11.61 Mb,不在新定位抗根肿基因初定位区间内,因此,可以推断新定位抗根肿基因位点与Crr1并非同一位点,同时基于Crr1基因序列新开发的标记BrID10381具有多态性,与BRMS-088相比可以用于Crr1基因的分子标记辅助选择,在大白菜抗根肿病育种实践中具有很大的应用价值。
本研究结果为下一步该抗病基因的精细定位和基因克隆以及分子标记辅助选择(MAS)育种奠定了基础,有助于未来大白菜抗根肿病新品种的选育。
[1] 张凡,李林梅,崔海芳,尹俊龙,郭英琪,岳艳玲. 十字花科植物抗根肿病研究进展[J]. 湖南生态科学学报,2017,4(3):57-62. doi:10.3969/j.issn.2095-7300.2017.03-057.
Zhang F,Li L H,Cui H F,Yin J L,Guo Y Q,Yue Y Y. Current research of crucifer clubroot resistance[J]. Journal of Hunan Environment Biological Polytechnic,2017,4(3):57-62.
[2] Kageyama K,Asano T. Life cycle of Plasmodiophora brassicae[J]. Journal of Plant Growth Regulation,2009,28(3):203. doi:10.1007/s00344-009-9101-z.
[3] Zhang T,Zhao Z,Zhang C,Pang W X,Choi S R,Lin Y P,Piao Z Y. Fine genetic and physical mapping of the CRb gene conferring resistance to clubroot disease in Brassica rapa[J]. Molecular Breeding,2014,34(3):1173-1183. doi:10.1007/s11032-014-0108-1.
[4] 董森森. 白菜类作物抗根病种质资源遗传多样性分析及分子标记辅助选择[D]. 郑州:郑州大学,2014.
Dong S S. Analysis of genetic diversity of the clubroot resistance germplasms in Brassica rapa and molecular marker-assisted selection[D]. Zhengzhou:Zhengzhou University,2014.
[5] Nguyen M L,Monakhos G F,Komakhin R A,Monakhos S G. The new clubroot resistance locus is located on Chromosome A05 in Chinese cabbage(Brassica rapa L.)[J]. Russian Journal of Genetics,2018,54(3):296-304. doi:10.1134/S1022795418030080.
[6] 胡靖锋,杨红丽,徐学忠,和江明,陈龙正. 微型大白菜“娃娃菜”根肿病抗性遗传规律及分子标记辅助选择研究[J]. 福建农业学报,2017,32(2):185-188. doi:10.19303/j.issn.1008-0384.2017.02.016.
Hu J F,Yang H L,Xu X Z,He J M,Chen L Z. Application of molecular markers-assisted selection for studying inheritance of clubroot-resistance in miniature chinese Baby cabbage [J]. Fujian Journal of Agricultural Sciences,2017,32(2):185-188.
[7] 薛银鸽.大白菜根肿病抗性基因分子标记与定位[D]. 郑州:郑州大学,2014.
Xue Y G. Molecular marker analysis and mapping of clubroot resistance gene in Chinese cabbage[D]. Zhengzhou:Zhengzhou University,2014.
[8] 王彤彤,张淑江,章时蕃,李菲,张慧,孙日飞.大白菜根肿病抗性基因的标记和定位[J]. 中国蔬菜,2012,1(14):31-35.
Wang T T,Zhang S J,Zhang S F,Li F,Zhang H,Sun R F. Mapping of clubroot resistance gene in Chinese cabbage[J]. China Vegetables,2012,1(14):31-35.
[9] 王艳. 白菜参考遗传图谱的构建[D].北京:中国农业科学院,2011.
Wang Y. A sequence-based genetic linkage map as a reference for Brassica rapa L. ssp. pekinensis[D]. Beijing:Chinese Academy of Agricultural Sciences,2011.
[10] 柴阿丽,朱发娣,王惟萍,石延霞,谢学文,李宝聚.十字花科根肿病接种技术及发病条件研究[J]. 华北农学报,2015,30(S1):266-271. doi:10.7668/hbnxb.2015.S1.048.
Chai A L,Zhu F D,Wang W P,Shi Y X,Xie X W,Li B J. Inoculation methods and incidence conditions of clubroot of Cruciferous[J]. Acta Agriculturae Boreali-Sinica,2015,30(S1):266-271.
[11] Pang W X,Liang S,Li X N,Li P P,Yu S,Lin Y P,Piao Z Y. Genetic detection of clubroot resistance loci in a new population of Brassica rapa[J]. Horticulture,Environment,and Biotechnology,2014,55(6):540-547. doi:10.1007/s13580-014-0079-5.
[12] 董正中,陈超权,刘子萌,郑柳青,吴薇,谢涵冰,葛柳群,刘盛兰嘉,余小林. 普通白菜形态学性状及其根肿病抗性鉴定[J]. 浙江农业科学,2017(11):140-143. doi:10.16178/j. issn. 0528-9017. 20171142.
Dong Z Z,Chen C Q,Liu Z M,Zheng L Q,Wu W,Xie H B,Ge L Q,Liu S L J,Yu X L. The Morphological characters and identification of clubroot resistance of Brassica campestris(syn.B.rapa)L.ssp.chinensis(L)Makino[J]. Zhejiang Agricultural Sciences,2017(11):140-143.
[13] 张红,张斌,王超楠,李梅,黄志银,刘俊峰. 青麻叶大白菜根肿病人工接种方法及条件研究[J]. 华北农学报,2016,31(1):182-185. doi:10.7668/hbnxb.2016.01.029.
Zhang H,Zhang B,Wang C N,Li M,Huang Z Y,Liu J F. Study on inoculation methods and condition of clubroot in Qingmaye Chinese cabbage[J]. Acta Agriculturae Boreali-Sinica,2016,31(1):182-185.
[14] 王彤彤. 大白菜根肿病抗性基因的分子标记及定位[D]. 北京:中国农业科学院,2012.
Wang T T. Molecular marker analysis and mapping of clubroot resistance genes in Brassica rapa L. ssp. pekinensis[D].Beijing:Chinese Academy of Agricultural Sciences,2012.
[15] Piao Z Y,Deng Y Q,Choi S R,Park Y J,Lim Y P. SCAR and CAPS mapping of CRb,a gene conferring resistance to Plasmodiophora brassicae,in Chinese cabbage(Brassica rapa ssp. pekinensis)[J]. Theoretical and Applied Genetics,2004,108(8):1458-65.
[16] Narzary D,Verma S,Mahar K S,Rana T S. A rapid and effective method for isolation of genomic DNA from small amount of silica-dried leaf tissues[J]. National Academy Science Letters,2015,38(5):441-444. doi:10.1007/s40009-015-0357-5.
[17] Suwabe K,Tsukazaki H,Iketani H,Hatakeyama K,Kondo M,Fujimura M,Nunome T,Fukuoka H,Hirai M,Matsumoto S. Simple sequence repeat-based comparative genomics between Brassica rapa and Arabidopsis thaliana:the genetic origin of clubroot resistance[J]. Genetics,2006,173(1):309-319. doi:10.1534/genetics.104.038968.
[18] Matsumoto E,Yasui C,Ohi M,Tsukada M. Linkage analysis of RFLP markers for clubroot resistance and pigmentation in Chinese cabbage[J]. Euphytica,1998,104:79-86.doi:10.1023/a:1018370418201.
[19] Hayashida N,Takabatake Y,Nakazawa N,Aruga D,Nakanishi H,Taguchi G,Sakamoto K,Matsumoto E. Construction of a practical SCAR marker linked to clubroot resistance in Chinese cabbage with intensive analysis of HC352b genes[J]. Japan Soc Hort Sci,2008,77(2):150-154.
[20] Hirai M,Harada T,Kubo N,Tsukada M,Suwabe K,Matsumoto S. A novel locus for clubroot resistance in,Brassica rapa,and its linkage markers[J]. Theoretiscal and Applied Genetics,2004,108(4):639-643. doi:10.1007/s00122-003-1475-x.
[21] Sakamoto K,Saito A,Hayashida N,Taguchi G,Matsumoto E. Mapping of isolate-specific QTLs for clubroot resistance in Chinese cabbage(Brassica rapa L. ssp. pekinensis)[J].Theoretical and Applied Genetics,2008,117(5):759-767.doi:10.1007/s00122-008-0817-0.
[22] 任平平,马安峰,程斐,孙朝辉,高建伟. 大白菜抗根肿病基因CRb的分子标记验证与种质资源筛选[J]. 山东农业科学,2016,48(6):7-10.doi:10.14083/j.issn,1001-4942.2016. 06.002.
Ren P P,Ma A F,Chen F,Sun C H,Gao J W. Identification of molecular markers for clubroot disease resistant gene cRb and screening of germplasm resources of Chinese cabbage[J]. Shandong Agricultural Sciences,2016,48(6):7-10.
[23] Hatakeyama K,Niwa T,Kato T,Ohara T,Kakizaki T,Matsumoto S. The tandem repeated organization of NB-LRR genes in the clubroot-resistant CRb locus in Brassica rapa L. [J]. Molecular Genetics and Genomics,2017,292(2):397-405.doi:10.1007/s00438-016-1281-1.
[24] Suwabe K,Tsukazaki H,Iketani H,Hatakeyama K,Fujimura M,Nunome T,Ukuoka H,Matsumoto S,Hirai M. Identification of two loci for resistance to clubroot(Plasmodiophora brassicae Woron.)in Brassica rapa L.[J]. Theoretical and Applied Genetics,2003,107(6):997-1002. doi:10.1007/s00122-003-1309-x.
[25] Hatakeyama K,Suwabe K,Tomita R N,Kato T,Nunome T,Fukuoka H,Matsumoto S. Identification and characterization of crr1a,a gene for resistance to clubroot disease(Plasmodiophora brassicae Woron.)in Brassica rapa L.[J]. PLoS One,2013,8(1):268-277. doi:10.1371/journal.pone.0054745.