辣椒疫病(Phytophthora blight)是由辣椒疫霉菌(Phytophthora capsici Leonian)引起的一种土传病害[1],而辣椒疫霉菌是一种重要的植物病原卵菌,为害茄科、葫芦科和豆类等多种农作物以及可可树等梧桐科植物。辣椒疫病是世界范围内最具破坏性的疾病之一[2],在适宜条件下,其会导致辣椒产量急剧降低,轻者减产20%~30%,重者减产90%,甚至绝收。目前,山西辣椒种植面积约5万hm2,由疫病引起的辣椒产量减少、品质下降、效益下滑的问题已直接威胁到辣椒产业的健康发展。因此,开展辣椒抗疫病转录组的研究对育种工作者针对抗性材料的筛选和改良具有必要性和紧迫性。
辣椒疫病在辣椒整个生育期均可发生,叶片受到辣椒疫霉菌侵染时,病斑呈现圆形或近圆形,然后逐渐扩展到叶片边缘,导致叶片软腐并呈现黄绿色;花受侵染后变褐、脱落;受到其侵染时果蒂最先开始发病,最终霉烂[3]。辣椒疫病的发生以及流行主要与辣椒疫霉菌生长的环境条件密切相关,主要包括温湿度、栽培土壤的类型以及栽培方式等。辣椒疫霉菌主要以菌丝、厚垣孢子等形式在寄主植物的病株残体、土壤、种子或其他寄主上越冬并存活下来,成为第2年的初侵染源。等到第2年条件适宜时,辣椒疫霉菌借助雨水或风力等外界传播媒介侵入寄主植物体内的各个部位,从而引起病害发生[4-5]。近年来,国内外关于辣椒疫病抗性育种工作研究不断深入,结果表明,抗辣椒疫病是由多基因控制。因此,只有通过聚合高抗辣椒疫病品种优良基因,才能选育出较好的抗疫病辣椒品种。目前,辣椒基因组测序的完成以及分子标记技术和高通量转录组测序(RNA-seq)技术的不断进步[6],为辣椒抗疫病育种打下扎实的分子基础,特别是新一代转录组测序技术的发展对辣椒育种研究起到了巨大的推动作用,该技术可以在特定组织或细胞的某个时期来研究基因功能及结构。前人已经利用转录组测序方法研究了辣椒的一些性状,如对辣椒雄性不育转录组测序分析[7],辣椒果实不同形态(铃形和牛角形)转录组测序分析[8]。其中,Wang等[9]对辣椒抗疫病材料PI201234转录组进行测序分析,结果筛选到204个抗病基因,其表达模式包括细胞壁修饰、植保素合成、激素信号和转录调控4个方面。因此得出,利用RNA-seq技术可以快速全面发现特定组织、特定细胞内起重要功能的基因,从而为育种工作奠定基础。
目前,辣椒疫病危害巨大,其已被全球辣椒育种工作者高度重视。虽然,对辣椒疫病的抗性机制研究已有数十年,但到目前为止,抗病基因还未被全部克隆,相关抗病机理依然模棱两可。
本研究利用RNA-seq分子生物学技术,从114份辣椒资源中筛选出对辣椒疫病产生抗性的相关基因,旨在今后的研究中可用来快速分辨辣椒资源的抗性,加快抗病育种进度和辣椒抗疫病新品种的更新换代,其对促进辣椒产业的发展和农民增收具有重要的意义。
1 材料和方法
1.1 试验材料
研究所用的辣椒材料均由山西省农业科学院蔬菜研究所搜集并提供。在播种前选择籽粒饱满、颜色和大小一致的辣椒种子,首先在55 ℃温水中浸种20 min,然后将种子均匀铺在装有湿润滤纸的培养皿中,于温度为28 ℃、相对湿度为60%的全黑暗人工气候箱中进行催芽。大约在80%的种子露白后,就可以将其播在装有基质(腐殖质∶珍珠岩=3∶1)的穴盘中,于山西省农业科学院东阳基地大棚中进行培养。每个材料设置 3个重复,每个重复种植10株。
辣椒疫霉菌菌株由山西省农业科学院植物保护研究所保存并提供。首先将保存的病原菌进行复壮,在超净工作台上将保存于石蜡油中的菌丝接种到PDA固体培养基(300 g马铃薯削皮切片,加入18 g琼脂、20 g葡糖糖,置于微波炉中煮30 min(每10 min搅拌一次),然后用纱布过滤去除残渣,最后蒸馏水定容至1 000 mL)上进行活化,28 ℃黑暗条件下培养7 d[10]。待白色菌落形成后置于光下进行孢子囊的诱导,大约2 d后,将产生的孢子囊刮到无菌水中,4 ℃下1 h,而后28 ℃下30 min促使游动孢子的释放。用高温灭菌的纱布过滤,收集下层的滤液,然后在血球计数板上计数,用无菌水调整成最终浓度为 1×104个/mL的孢子悬浮液,备用。
待辣椒苗长出4片真叶后,移入整理好的苗圃内,并做好标号。长出6~8片真叶时,采用灌根法对其进行疫霉菌接种。本试验采用的接种方法为灌根接种法[11]。接种15 d后,按农业部1999年颁布的分级标准调查辣椒发病级别(表1)。
表1 辣椒疫病分级标准
Tab.1 The standard classification of pepper Phytophthora blight

病级 Disease stage病症描述 Description of disease0级 Zero stage 无症状 1级 One stage 幼苗茎基部稍有变黑,但植株健康不萎蔫 2级 Two stage 幼苗茎基部变黑达1~2 cm,叶片不可恢复性萎蔫,下部叶片偶有脱落 3级 Three stage 幼苗茎基部变黑达2 cm以上,叶片明显萎蔫或明显脱落 4级 Four stage 幼苗茎基部变黑缢缩,除生长点外全部落叶或者整株萎蔫 5级 Five stage 植株全部枯死
1.2 试验方法
1.2.1 田间取样和RNA提取 灌根15 d后调查植株发病情况,发现L1、L2和L4这3个材料属于高感辣椒疫病材料,病级属于4~5级;L3属于高抗辣椒疫病材料,完全不发病,病级为0级。每个材料分别取10株混合样,迅速置于液氮中冷冻,然后放在-80 ℃的超低温冰箱中保存备用。
利用TRIzol(Invitrogen,USA)试剂提取辣椒L1、L2、L3和L4整株混样总RNA,然后利用RNase-free DNase Ⅰ(Thermo Scientific,USA)除去总RNA中残余的DNA。利用NanoDrop 2000(Thermo Scientific,USA)分光光度计检测提取辣椒总RNA的浓度和纯度。
1.2.2 转录组文库构建及高通量测序 将提取的4个辣椒样品L1、L2、L3和L4的总RNA,参考刘峰等[8]的方法构建特异性文库。文库构建完成后,采用PCR扩增进行文库片段富集,文库大小一般在300~400 bp。然后参考Chen等[7]的方法进行Agilen及文库有效浓度检测,样品经过RNA抽提、纯化、建库之后,送往生工生物工程(上海)有限公司,采用Illumina RNA-seq测序平台,对文库进行双末端测序。样品经测序后生成FASTQ格式的原始数据,然后经过数据过滤,要求平均质量分数不低于Q20。最后采用FastQC对数据进行单碱基质量、Base Content分布、GC Content分布、Sequence Base Quality 4个指标的质控分析。
1.2.3 转录组测序数据分析 将测序过滤后数据与参考基因组Pepper_Zunla_1_Ref_v1.0(NCBI)进行比对,根据基因组注释信息,可得到序列的来源基因以及表达产物的结构。然后基于比对到基因的序列数目,用统计学方法计算表达量,并进一步比较基因、转录本和外显子在不同样品和分组之间的表达差异[8]。采用 RPKM(Reads Per Kilo bases per Million reads)对基因的表达量进行标准化,并在此基础上以FDR≤0.001和|log2 Ratio|≥1为条件筛选出2组样本间差异表达基因。目的是为了能够在样品内以及样品间比较基因的表达量。其计算公式如下:
①
1.2.4 Real time-PCR 为了鉴定Illumina测序后筛选出的差异基因的不同表达模式,随机选择20个差异基因,然后利用qRT-PCR进行验证。首先使用 Primer 5.0 设计引物,尽可能跨内含子/外显子边界,以避免在实时定量RT-PCR时扩增基因组DNA。每个样品取3.0 μg 总RNA,利用TaKaRa公司 PrimeScript @ Reverse Transcriptase 试剂盒(商品编号:D2680A)方法(参考说明书)进行反转录。用大豆β-actin 基因设立阳性对照,根据 TaKaRa公司SYBR@ Premix Ex Taq TM(Tli RNaseh Plus)(商品编号为 DRR420S)说明书进行荧光定量PCR反应(ABI 7 300),每个反应重复 3 次,最后利用2-ΔΔCT方法分析结果。
2 结果与分析
2.1 四份辣椒材料在苗期的抗病性鉴定
将山西省农业科学院蔬菜研究所辣椒课题组收集的114份辣椒材料/品种,在幼苗长至6~8片叶时,接种辣椒疫霉菌。接种7 d后,按照农业部1999年颁布的0~5级6个抗病分级标准进行病级统计。结果显示,辣椒疫病6个病级,每级材料份数分别为3,7,11,25,36,32(图1)。由此可以看出,抗辣椒疫病的材料较少,感疫病材料较多。本研究从0级抗病的3份材料中选择1份抗病材料L3,从5级感病的32份材料中选择L1、L2、L4这3份感病材料进行后期深入研究。
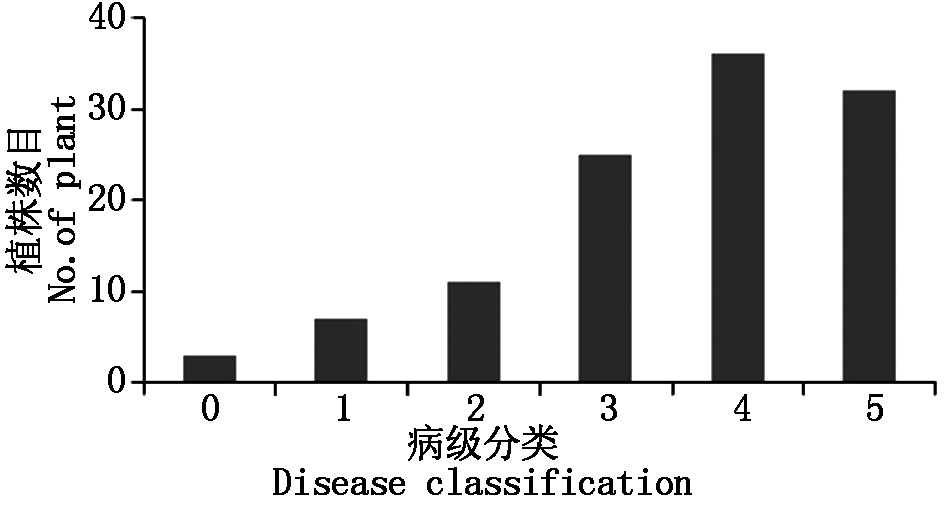
图1 辣椒田间病级统计结果
Fig.1 The statistics result of disease pepper in field
2.2 转录组测序数据质量检测
4份辣椒材料苗期转录组测序数据统计和质量检测如表2,3所示。由表2可知,本次测序试验共获得177 238 390条的 reads,总碱基数为26.59 Gb,每个样品均获得6.2 Gb以上的碱基。其中,Q20在97%以上,Q30在93%以上,GC含量均占43%。说明试验转录组测序质量具有较高的可信度,可用于后续数据分析。经过对原始总数据质量过滤后,L1、L2、L3和L4这4份样本的Clean数据与表2中的数据量相比均有所下降,筛选后的有效数据量均在98%以上(表3),平均质量分数不低于Q20。
利用Tophat/Tophat2(基于Bowtie/Bowtie2)软件成功地将82.66%~89.99%的Clean数据比对到辣椒参考基因组(http://www.ensembl.org/),并且有78.95%~85.11%的Clean数据比对到唯一的基因组位点(表4)。说明4个样品的Clean数据与参考基因组的比对效率较高。
表2 测序数据统计
Tab.2 The statistical results of sequencing data
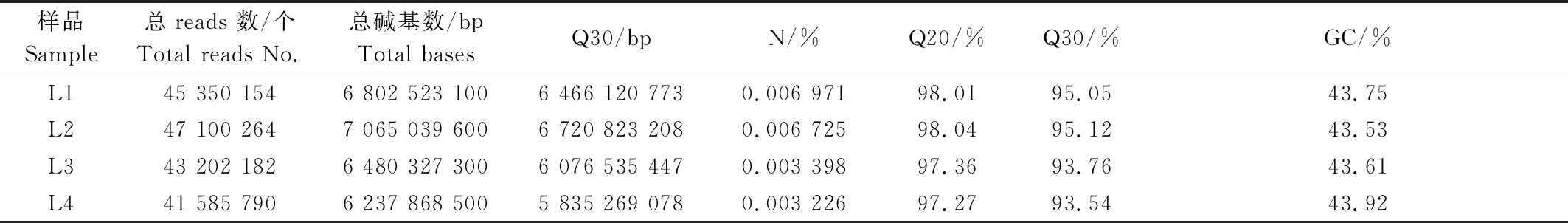
样品Sample总reads 数/个Total reads No.总碱基数/bp Total basesQ30/bpN/%Q20/%Q30/%GC/%L145 350 1546 802 523 1006 466 120 7730.006 97198.0195.0543.75L247 100 2647 065 039 6006 720 823 2080.006 72598.0495.1243.53L343 202 1826 480 327 3006 076 535 4470.003 39897.3693.7643.61L441 585 7906 237 868 5005 835 269 0780.003 22697.2793.5443.92
注:Qx 指质量值大于或等于x的碱基所占的百分比。
Note:Qx means the percentage of quality value greater or equal to x base.
表3 转录组测序数据质量检测
Tab.3 The result of transcriptome sequencing data
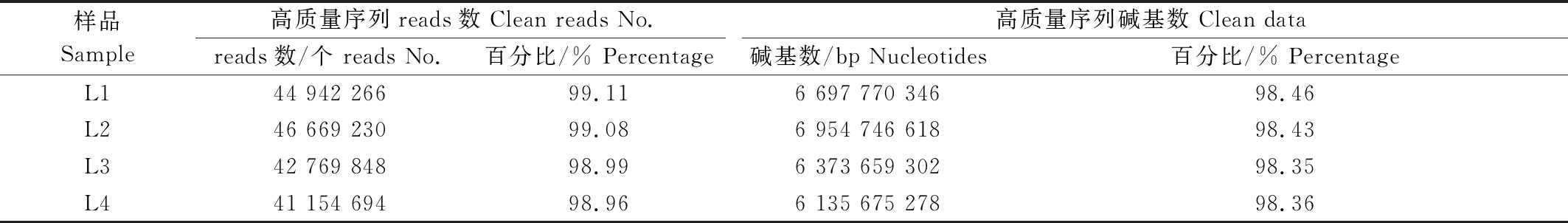
样品Sample高质量序列reads数 Clean reads No.高质量序列碱基数 Clean data reads数/个 reads No.百分比/% Percentage碱基数/bp Nucleotides 百分比/% Percentage L144 942 26699.116 697 770 34698.46L246 669 23099.086 954 746 61898.43L342 769 84898.996 373 659 30298.35L441 154 69498.966 135 675 27898.36
表4 四份辣椒材料RNA转录组测序结果与辣椒参考基因组比对统计
Tab.4 Statistics of RNA-seq for pepper phytophthora blight of four pepper genotypes referring to pepper genome
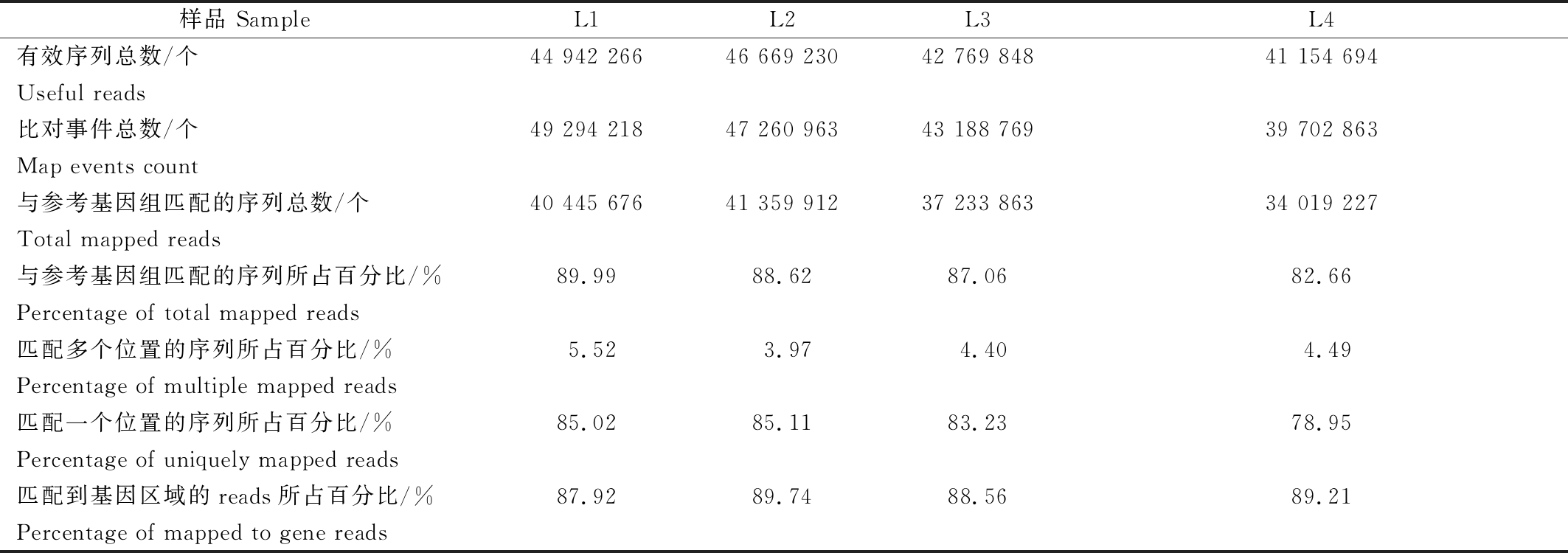
样品 SampleL1L2L3L4有效序列总数/个 44 942 26646 669 23042 769 84841 154 694Useful reads比对事件总数/个49 294 21847 260 96343 188 76939 702 863Map events count与参考基因组匹配的序列总数/个40 445 67641 359 91237 233 86334 019 227Total mapped reads与参考基因组匹配的序列所占百分比/%89.9988.6287.0682.66Percentage of total mapped reads 匹配多个位置的序列所占百分比/%5.523.974.404.49Percentage of multiple mapped reads 匹配一个位置的序列所占百分比/%85.0285.1183.2378.95Percentage of uniquely mapped reads 匹配到基因区域的reads所占百分比/%87.9289.7488.5689.21Percentage of mapped to gene reads
2.3 辣椒抗疫病和感疫病2组材料差异表达基因分析
依据4份材料间的差异基因表达水平进行PCA主成分分析,将相似材料进行聚类,结果显示,L3的值远高于在同一组的L1、L2和L4这3份材料(图2-A),表明前期在材料筛选中足够精确,试验设计也合理。采用 DESeq(Version 1.18.0)方法对辣椒抗疫病和感疫病2组材料间基因表达进行差异分析,通过成对比较可以消除基因型特异性转录组的背景干扰,从而获得与抗病和感病相关的更多数据。首先筛选抗病和感病2个材料间的差异表达基因条件为:表达倍数差异 |log2 fold-change|>1,显著性P-value<0.05。依据这2个标准,将感病组的L1、L2和L4 这3个材料分别与抗病材料L3相比,结果显示,分别得到1 127,1 165,1 062个显著差异表达基因,其中,上调基因分别为364,375,713个,下调基因分别为763,790,349个(表5),有331个基因在 3组比对结果中均表现差异,包括68个上调基因和263个下调基因(图2-B)。
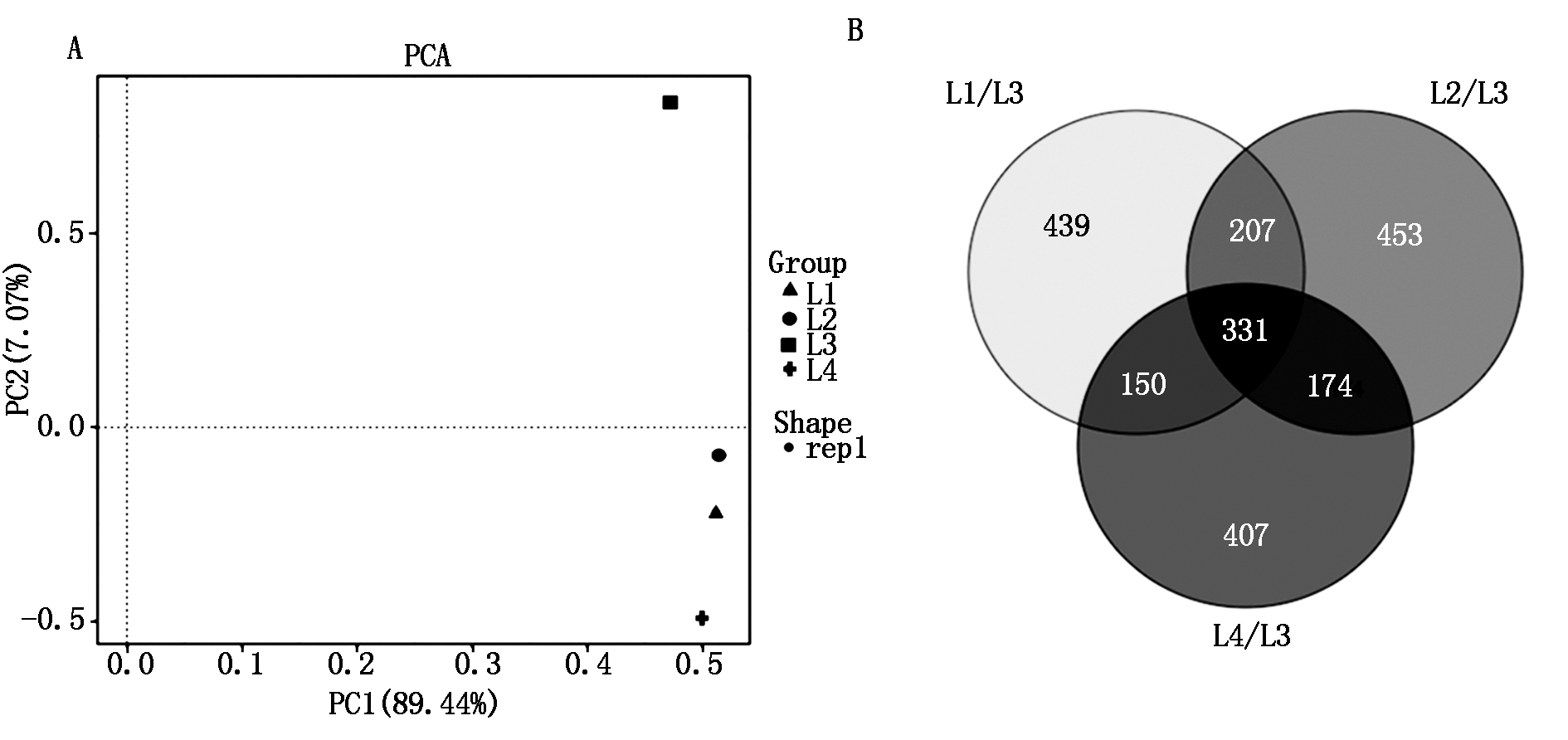
图2 四个辣椒材料PCA主成分分析图(A)和差异基因表达维恩图(B)
Fig.2 PCA diagram of four pepper materials(A)and the Venn diagram showing the DEGs(B)
表5 表达差异分析结果统计
Tab.5 The statistical results of DEGs
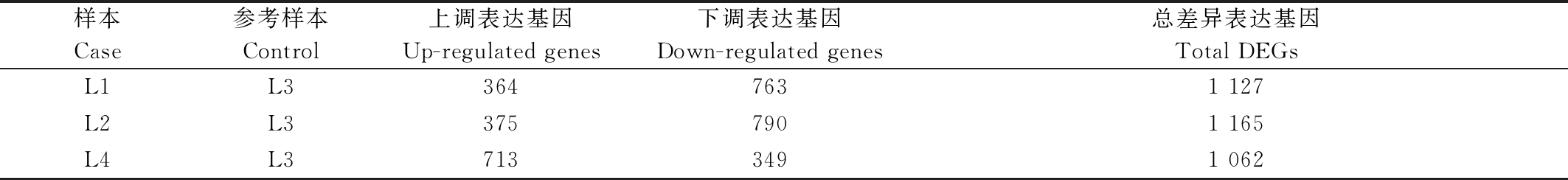
样本Case参考样本 Control上调表达基因Up-regulated genes下调表达基因Down-regulated genes总差异表达基因Total DEGsL1L33647631 127L2L33757901 165L4L37133491 062
2.4 利用qRT-PCR 荧光定量验证差异表达基因
为了验证转录组测序分析的差异基因的可靠性,随机从2组材料筛选出的差异表达基因中挑选10个差异基因(包括5个上调基因和5个下调基因)(表6),采用荧光定量qRT-PCR技术进行表达验证,结果显示,荧光定量qRT-PCR检测的随机选择的10个差异基因在4份辣椒材料中的表达模式与RNA-seq分析结果高度相似,进而证实,转录组测序8个基因的表达水平与RNA-seq结果一致,其中,107861905基因在L2中几乎不表达,107862280基因在L3中的表达量反而高于其他 3份感病材料(图3)。因此,可以证明RNA-seq分析结果可靠。
表6 RNA-seq分析的10个表达差异基因
Tab.6 10 DEGs in the RNA-seq analysis of four pepper materials
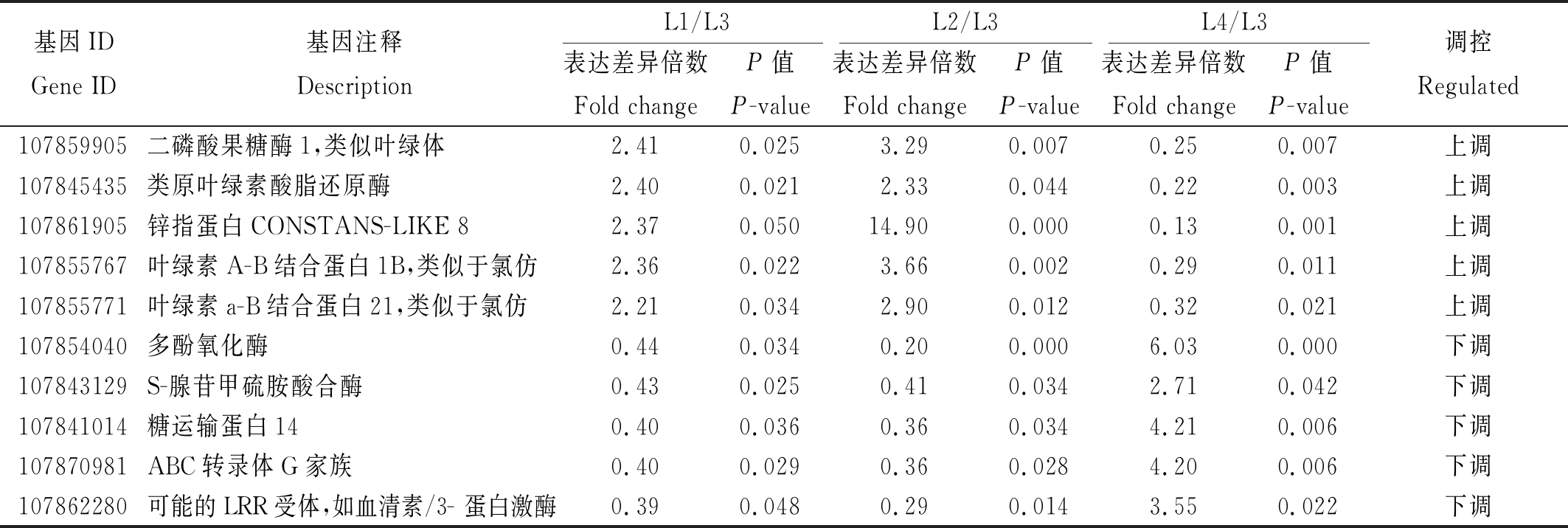
基因IDGene ID基因注释DescriptionL1/L3L2/L3L4/L3表达差异倍数Fold changeP值P-value表达差异倍数Fold changeP值P-value表达差异倍数Fold changeP值P-value调控Regulated107859905二磷酸果糖酶1,类似叶绿体 2.410.0253.290.0070.250.007上调107845435类原叶绿素酸脂还原酶 2.400.0212.330.0440.220.003上调107861905锌指蛋白CONSTANS-LIKE 8 2.370.05014.900.0000.130.001上调107855767叶绿素A-B结合蛋白1B,类似于氯仿 2.360.0223.660.0020.290.011上调107855771叶绿素a-B结合蛋白21,类似于氯仿 2.210.0342.900.0120.320.021上调107854040多酚氧化酶 0.440.0340.200.0006.030.000下调107843129S-腺苷甲硫胺酸合酶0.430.0250.410.0342.710.042下调107841014糖运输蛋白14 0.400.0360.360.0344.210.006下调107870981ABC转录体G家族 0.400.0290.360.0284.200.006下调107862280可能的LRR受体,如血清素/3- 蛋白激酶0.390.0480.290.0143.550.022下调
2.5 差异基因功能显著性富集分析
Gene Ontology(GO)富集分析能够全面描绘差异基因和基因产物的功能属性,因此将其用于评价辣椒抗疫病和感疫病相关表达具有显著差异基因的功能。根据 P<0.05 的阈值来选择,结果显示,14 624个差异表达基因被富集,占全基因组的46.58%,将预测的和已知的转录本分成 3组,包括分子功能转录本、细胞组成转录本和生化过程转录本。本研究具体分析在 3组比对中都表现出差异的331个差异基因,其中,有223个基因被GO富集、41个上调基因、182个下调基因。通过排列前10的 GO分类可以看出(表7),上调差异基因和下调差异基因主要功能是分子功能和生化过程,而细胞组成分类很少。其中,分子功能主要子类有氧化还原酶活性、碳氧裂解酶活性、过氧化物酶活性等,而生化功能子类主要是代谢过程和逆境响应过程。以上结果可以说明,差异基因编码不同的酶类活性、代谢过程和逆境响应,这些可能与辣椒抗疫病和感疫病差异相关的基因有关。
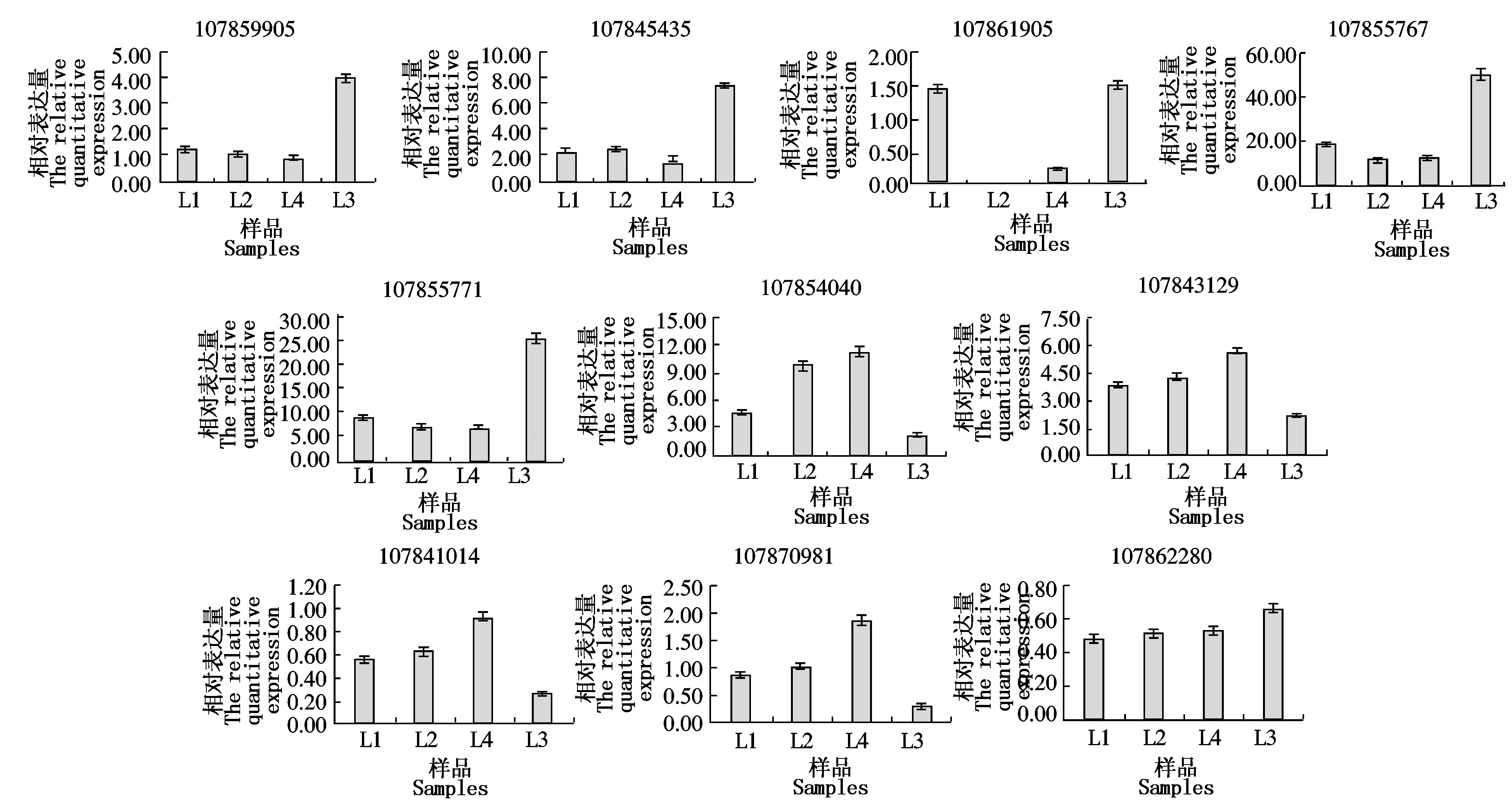
图3 实时定量PCR验证10个随机选择的差异表达基因
Fig.3 Validation of 10 randomly chosen DEGs by qRT-PCR
与整个转录组背景相比,共有117个差异基因归入KEGG通路,其中,包括25个上调差异基因,92个下调差异基因。在这些通路中,RNA退化、泛素介导的蛋白水解作用、氧化磷酸化、内质网蛋白处理和氨基酸的生物合成是上调差异基因对应的前5个通路(表7)。而下调差异基因对应的前5个通路,分别为植物激素信号转导、磷脂酶D信号通路、磷脂酰肌醇信号系统、黏液、氯烷烃和氯烯烃降解。因此,从整体观察发现,辣椒抗疫病和感疫病差异基因主要集中于植物激素调节和蛋白调节等通路。将GO富集和KEGG富集结合起来分析发现,辣椒抗疫病和感疫病差异主要与酶类活性功能、逆境响应过程相关,其次为结合蛋白代谢途径、植物激素调节等过程。
表7 转录组测序分析中差异基因的GO子类详细分析和KEGG通路分析
Tab.7 The detailed GO subcategory analysis and KEGG pathways for DEGs in the RNA-seq analysis
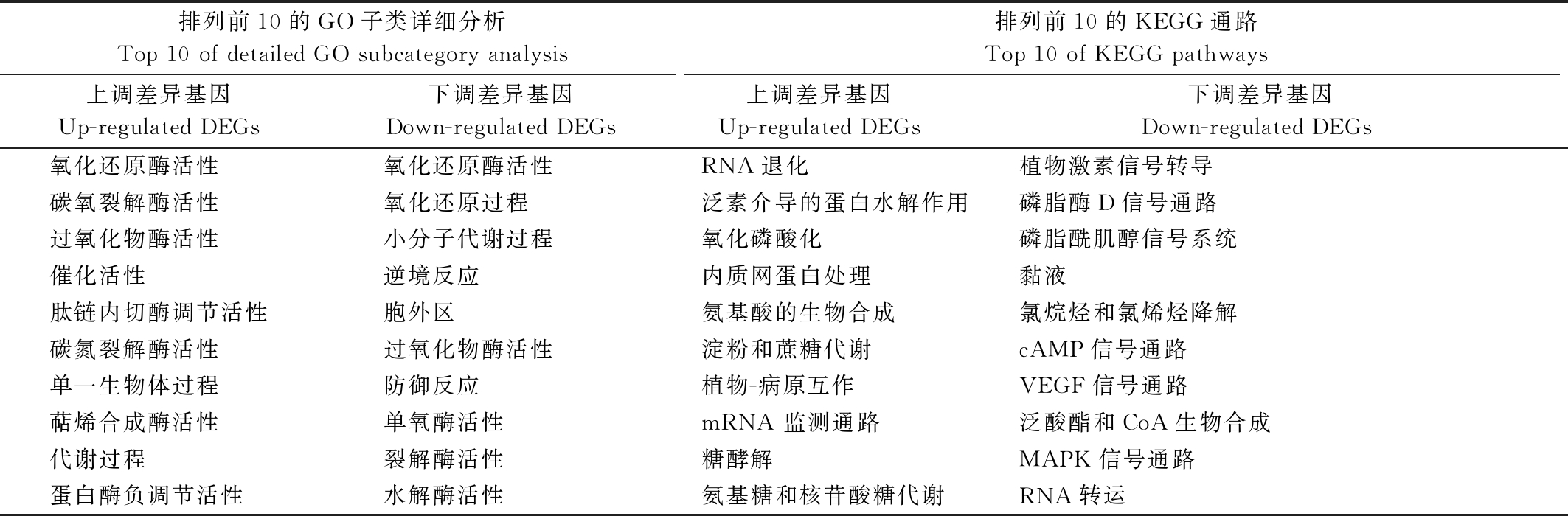
排列前10的GO子类详细分析 Top 10 of detailed GO subcategory analysis排列前10的KEGG通路 Top 10 of KEGG pathways上调差异基因 Up-regulated DEGs 下调差异基因 Down-regulated DEGs 上调差异基因 Up-regulated DEGs 下调差异基因 Down-regulated DEGs 氧化还原酶活性 氧化还原酶活性 RNA退化植物激素信号转导碳氧裂解酶活性 氧化还原过程 泛素介导的蛋白水解作用 磷脂酶D信号通路 过氧化物酶活性 小分子代谢过程 氧化磷酸化 磷脂酰肌醇信号系统 催化活性 逆境反应 内质网蛋白处理 黏液 肽链内切酶调节活性 胞外区 氨基酸的生物合成 氯烷烃和氯烯烃降解 碳氮裂解酶活性过氧化物酶活性 淀粉和蔗糖代谢 cAMP信号通路单一生物体过程 防御反应 植物-病原互作 VEGF信号通路萜烯合成酶活性 单氧酶活性 mRNA 监测通路泛酸酯和CoA生物合成 代谢过程 裂解酶活性 糖酵解 MAPK信号通路蛋白酶负调节活性 水解酶活性 氨基糖和核苷酸糖代谢 RNA转运
3 结论与讨论
本研究利用114份辣椒自然群体通过人工灌根法接种辣椒疫病,通过对病情的调查发现,高抗材料较少,大多数材料都会感病,只是发病程度不一致。灌根法接种辣椒疫病这种方法理论上更符合大田病害发生实际侵染模式,基本上可模拟田间病害发生过程。
从筛选出的抗病和感病材料中选择1份抗疫病材料L3和 3份感病材料L1、L2和L4进行RNA-seq高通量测序分析。Illumina 高通量测序结果数据量大,并且试验效率高,成本低,适合于辣椒疫病转录组测序研究。本研究共获得了177 238 390条的reads,总碱基数为26.59 Gb,每个样品均获得6.2 Gb以上的碱基。有82.66%~89.99%的Clean数据比对到辣椒参考基因组上,并且有78.95%~85.11%的Clean数据比对到唯一的基因组位点,这一结果显示,本研究高通量测序结果可靠,并且可以大量挖掘辣椒抗疫病过程中表达的重要基因。通过对辣椒抗病和感病的转录组比较分析,经GO和KEGG差异基因功能显著性富集分析,结果与前人研究辣椒疫病[12-15]、马铃薯疫病[16-19]等结果较一致。与辣椒疫病相关的基因功能主要表现为酶类、植物激素和泛素化等。通过抗病和感病成对比较L3/L1、L3/L2、L3/L4,结果显示,3组样本共同的差异基因331个,其中,68个上调基因,主要包含氧化还原酶活性、碳氧裂解酶活性、过氧化物酶活性、催化活性、代谢过程、蛋白酶负调节活性等相关基因;263个下调基因,主要包含氧化还原酶活性、氧化还原过程、逆境反应、胞外区、防御反应以及一些蛋白酶活性等相关基因。
过氧化物酶可以通过抑制真菌生长、清除过氧化氢,从而达到保护植物组织免受细胞损伤的作用。Alc zar等[20] 发现,接种辣椒疫病后,辣椒体内过氧化物酶活性均升高,尤其是高抗病品种中增加的幅度最大;同时也发现,只在抗病品种细胞间检测出过氧化物酶,说明在抗病品种中沿菌丝生长方向的过氧化物酶阻止了菌丝的迅速蔓延。同时Mozzetti等[21]研究也指出,过氧化物酶活性与辣椒疫病病情严重度相关。与辣椒疫病相关的酶类还有很多,如多酚氧化酶,将酚类物质氧化为醌类物质,并且聚合其为单宁类物质,这样就可以迅速杀死疫病侵入点周围的细胞,从而达到抑制病原发生的目的。蛋白酶类通过磷酸化和去磷酸化可逆过程作为细胞信号,参与调节各种细胞反应,从而调节植物生长发育以及应答对环境的胁迫。
zar等[20] 发现,接种辣椒疫病后,辣椒体内过氧化物酶活性均升高,尤其是高抗病品种中增加的幅度最大;同时也发现,只在抗病品种细胞间检测出过氧化物酶,说明在抗病品种中沿菌丝生长方向的过氧化物酶阻止了菌丝的迅速蔓延。同时Mozzetti等[21]研究也指出,过氧化物酶活性与辣椒疫病病情严重度相关。与辣椒疫病相关的酶类还有很多,如多酚氧化酶,将酚类物质氧化为醌类物质,并且聚合其为单宁类物质,这样就可以迅速杀死疫病侵入点周围的细胞,从而达到抑制病原发生的目的。蛋白酶类通过磷酸化和去磷酸化可逆过程作为细胞信号,参与调节各种细胞反应,从而调节植物生长发育以及应答对环境的胁迫。
植物激素对辣椒疫病也有防御调控,主要有茉莉酸(JA)、乙烯(ET)和水杨酸(SA)等[22-24]。其中,JA在植物抗病反应中是重要的信号分子,可诱导多酚氧化酶、苯丙烯酰苯合成酶、过氧化氨酶等防御蛋白的合成[25-27]。JA不仅是一种植物激素,同时也是植物抗性诱导剂,喷施JA等激素可以诱导植物产生抗病性,同时也能增加富含羟辅氨酸的糖蛋白的含量。也有人研究发现,JA能激活许多与植物防御相关的基因,如编码抗真菌蛋白的植物防卫素、硫素蛋白等合成的基因[28]。SA是植物免疫反应中一个重要的信号分子,植物在受到病原菌侵染后,体内SA含量显著提高,可产生系统获得性抗性。ET途径与JA途径都可以调控拟南芥中抗病防卫基因PDF1.2的表达,具有协同作用,同时二者还可抑制SA的合成,均与SA途径存在拮抗作用。
泛素化也是植物参与抗病反应的途径之一,由泛素激活酶、泛素结合酶和泛素连接酶共同完成,其中,泛素连接酶能特异性识别病原物效应蛋白并使其降解,是泛素化在植物抗病研究中的热门和关注对象[29-31]。根据结构域和组成的差异,泛素连接酶可分为HETC型、RING/U-BOX型、Cullin-RING型和APC/C型,其中,Cullin-RING型和RING/U-BOX型泛素连接酶在植物抗病中具有重要作用[32]。Cullin-RING型泛素连接酶可通过特异性降解水杨酸(SA)途径中调控蛋白NPR1、TGA2、NIMIN1等来介导SA抗病信号途径[33-34]。Cullin-RING型泛素连接酶亚基F-box蛋白COI1可以介导植物的茉莉酸(JA)抗病信号途径的传导,对死体营养型病原物产生抗性[35]。编码RING/U-BOX型泛素连接酶ATL基因在拟南芥、番茄和水稻中均发现,在免疫反应中调控了对病原物的抗性[31]。一些具有泛素连接酶活性的U-BOX蛋白也已被证明具有调控植物细胞死亡、过氧化氢和茉莉酸含量,进而影响植物免疫反应的功能[36]。
辣椒对疫霉的免疫反应是一个极其复杂、多途径协同的过程,其中主要包括酶类活性调节、植物激素抗病信号途径和泛素化。这些途径在调控辣椒对病原物的抗性过程中除了行使其单一功能外,还互相影响参加其他途径。说明辣椒对疫病的抗性并不是由个别基因所控制,而是一个系统性调节机制,需要大量的功能基因参与其中,这与本研究的转录组分析结果相契合。
研究通过高通量转录组测序,对辣椒抗疫病和感疫病2组材料进行了比较深入的转录组学研究,其结果可为在转录水平上全面了解辣椒抗疫病分子基础提供有价值的参考。通过研究发现,辣椒抗疫病是一个高度复杂的过程,它由多个交叉通路调节,包括新陈代谢过程、酶活性调节、结合蛋白代谢、防御反应及激素调节等,其结果为后期更深入研究辣椒抗疫病分子机制奠定了基础。
[1] Zhang Y L,Jia Q L,Li D W,Wang J E,Yin Y X,Gong Z H. Characteristic of the pepper CaRGA2 gene in defense responses against Phytophthora capsici Leonian[J]. International Journal of Molecular Sciences,2013,14:8985-9004. doi:10.3390/ijms14058985.
[2] Naegele R P,Ashrafi H,Hill T A,Chinwo S R,Van Deynze A E, Hausbeck M K. QTL mapping of fruit rot resistance to the plant pathogen Phytophthora capsici in a recombinant inbred line Capsicum annuum population[J]. Phytopathology,2014,104(5):479-83.doi:10.1094/PHYTO-05-13-0143-R.
[3] Ristiano J B,Johnston S A. Ecologically based approaches to management of Phytophthora blight on bell pepper[J]. Plant Disease,1999,83:1080-1089.doi:10.1094/PDIS.1999.83.12.1080.
[4] 杨学辉,肖崇刚,袁洁. 贵州辣椒疫病病原鉴定及生物学特性研究[J]. 西南大学学报(自然科学版),2004,26(4):413-416. doi:10.3969/j.issn.1673-9868.2004.04.010.
Yang X H,Xiao C G,Yuan J. Studies on identification of Phytophthora capsici in guizhou province and on its biological characteristics[J]. Journal of Southwest Agricultural University(Natural Science),2004,26(4):413-416.
[5] 冯宝珍. 辣椒疫霉(Phytophthora capsici)诱导坏死蛋白基因克隆及功能研究[D]. 泰安:山东农业大学,2011. doi:10.7666/d.d143924.
Feng B Z. Cloning and functional analysis of NPP(Necrosis Phytophythora Protein)genes from Phytophthora capsici[D]. Taian:Shandong Agricultural University,2011
[6] Lamour K H,Mudge J,Gobena D,Hurtadogonzales O P,Schmutz J,Kuo A,Miller N A,Rice B J,Raffaele S,Cano L M. Genome sequencing and mapping reveal loss of heterozygosity as a mechanism for rapid adaptation in the vegetable pathogen Phytophthora capsici [J]. Molecular Plant-microbe Interactions,2012,25:1350. doi:10.1094/MPMI-02-12-0028-R.
[7] Chen L,Ning M,Wang P Y,Nan F,Shen H L. Transcriptome sequencing and De Novo analysis of a cytoplasmic male sterile line and its Near-isogenic restorer line in Chili Pepper(Capsicum annuum L.)[J]. PLoS One,2013,8:e65209. doi:10.1371/journal.pone.0065209.
[8] 刘峰,谢玲玲,弭宝彬,欧阳娴,茆振川,谢丙炎. 辣椒转录组SNP挖掘及多态性分析[J]. 园艺学报,2014,41(2):343-348. doi:10.3969/j.issn.0513-353X.2014.02.016.
Liu F,Xie L L,Mi B B,Ouyang X,Mao Z C,Xie B Y. SNP Mining in pepper transeriptome and the polymorphism analysis[J]. Acta Horticulturae Sinica,2014,41(2):343-348.
[9] Wang P,Wang L,Guo J,Yang W,Shen H. Molecular mapping of a gene conferring resistance to Phytophthora capsici Leonian race 2 in pepper line PI201234(Capsicum annuum L.)[J]. Molecular Breeding,2016,36(6):66. doi:10.1007/s11032-016-0464-0.
[10] Kim H J,Nahm S H,Lee H R,Yoon G B,Kim K T,Kang B C,Choi D,Kweon O Y,Cho M C,Kwon J K. BAC-derived markers converted from RFLP linked to Phytophthora capsici resistance in pepper(Capsicum annuum L.)[J]. Theoretical and Applied Genetics,2008,118:15. doi:10.1007/s00122-008-0873-5.
[11] Sy O,Steiner R,Bosland P W. Recombinant inbred line differential identifies race-specific resistance to phytophthora root rot in Capsicum annuum[J]. Phytopathology,2008,98:867-870. doi:10.1094/PHYTO-98-8-0867.
[12] Lee Y K,Hippe-Sanwald S,Jung H W,Hong J K,Hause B,Hwang B K. In situ localization of chitinase mRNA and protein in compatible and incompatible interactions of pepper stems with Phytophthora capsic[J]. Protoplasma,2000,211:64-75. doi:10.1007/BF01279900.
[13] Cheng C M,Palloix A,Lefebvre V. Isolation,mapping and characterization of allelic polymorphism of Chi3-P1,a class Ⅲ chitinase of Capsicum annuum L[J]. Plant Science,2002,163:481-489. doi:10.1016/S0168-9452(02)00151-6.
[14] Yoo T H,Park C J,Ham B K,Kim K J,Paek K H. Ornithine decarboxylase gene(CaODC1)is specifically induced during TMV-mediated but salicylate-independent resistant response in hot pepper[J]. Plant and Cell Physiology,2004,45:1537-1542. doi:10.1093/pcp/pch176.
[15] Wang J E,Li D W,Gong Z H,Zhang Y L. Optimization of virus-induced gene silencing in pepper(Capsicum annuum L.)[J]. Genetics and Molecular Research,2013,12:2492-2506. doi:10.4238/2013.July.24.4.
[16] Or owska E,Fiil A,Kirk H G,Llorente B,Cvitanich C. Differential gene induction in resistant and susceptible potato cultivars at early stages of infection by Phytophthora infestans[J]. Plant Cell Reports,2012,31(1):187-203. doi:10.1007/s00299-011-1155-2.
owska E,Fiil A,Kirk H G,Llorente B,Cvitanich C. Differential gene induction in resistant and susceptible potato cultivars at early stages of infection by Phytophthora infestans[J]. Plant Cell Reports,2012,31(1):187-203. doi:10.1007/s00299-011-1155-2.
[17] Yogendra K N,Kumar A,Sarkar K,Li Y,Pushpa D,Mosa K A,Duggavathi R,Kushalappa A C. Transcription factor StWRKY1 regulates phenylpropanoid metabolites conferring late blight resistance in potato[J]. Journal of Experimental Botany,2015,66:7377-7389. doi:10.1093/jxb/erv434.
[18] Studham M E,Macintosh G C. Phytohormone signaling pathway analysis method for comparing hormone responses in plant-pest interactions[J]. BMC Research Notes,2012,5:1-7. doi:10. 1186/1756-0500-5-392.
[19] Wang B,Liu J,Tian Z,Song B,Xie C. Monitoring the expression patterns of potato genes associated with quantitative resistance to late blight during Phytophthora infestans infection using cDNA microarrays[J]. Plant Science,2005,169:1155-1167. doi:10.1016/j.plantsci.2005.07.020.
[20] Alcázar M D,Egea C,Espín A,Candela M E. Peroxidase isoenzymes in the defense response of Capsicum annuum to Phytophthora capsici[J]. Physiologia Plantarum,2010,94:736-742. doi:10.1111/j.1399-3054.1995.tb00992.x.
[21] Mozzetti C,Ferraris L,Tamietti G,Matta A. Variation in enzyme activities in leaves and cell suspension as markers of incompatibility in different Phytophthora-pepper interactions[J]. Physiological and Molecular Plant Pathology,1995,46:95-107. doi:10.1051/litt/201113003.
[22] Nú![]() R,Arcos-Ortega G F,Souza-Perera R A,Sánchez-Borges C A,Nakazawa-Ueji Y E,Guzmán-Antonio A A,Zú
R,Arcos-Ortega G F,Souza-Perera R A,Sánchez-Borges C A,Nakazawa-Ueji Y E,Guzmán-Antonio A A,Zú iga-Aguilar J J. Ethylene,but not salicylic acid or methyl jasmonate,induces a resistance response against Phytophthora capsici in Habanero pepper[J]. European Journal of Plant Pathology,2011,131:669-683. doi:10.1007/s10658-011-9841-z.
iga-Aguilar J J. Ethylene,but not salicylic acid or methyl jasmonate,induces a resistance response against Phytophthora capsici in Habanero pepper[J]. European Journal of Plant Pathology,2011,131:669-683. doi:10.1007/s10658-011-9841-z.
[23] Kang D S, Min K J, Kwak A M, Lee S Y, Kang H W. Defense response and suppression of Phytophthora blight disease of pepper by water extract from spent mushroom substrate of Lentinula edodes[J]. The Plant Pathology Journal, 2017, 33(3):264-275. doi:10.5423/ppj.oa.02.2017.0030.
[24] Rookes J E,Wright M L,Cahill D M. Elucidation of defence responses and signalling pathways induced in Arabidopsis thaliana following challenge with Phytophthora cinnamomi[J]. Physiological and Molecular Plant Pathology,2008,72:151-161.doi:10.1016/j.pmpp.2008.08.005.
[25] Ueeda M,Kubota M,Nishi K. Contribution of jasmonic acid to resistance against Phytophthora blight in Capsicum annuum cv. SCM334[J]. Physiological and Molecular Plant Pathology,2005,67:149-154. doi:10.1016/j.pmpp.2005.12.002.
[26] Maksimov I V,Sorokan′ A V,Chereoanova E A,Surina O B,Troshina N B,Yarullina L G. Effects of salicylic and jasmonic acids on the components of pro/antioxidant system in potato plants infected with late blight[J]. Russian Journal of Plant Physiology,2011,58(2):299-306. doi:10.1134/s1021443711010109.
[27] Boevink P C,Wang X,Hazel M L,He Q,Shaista N,Armstrong M R,Zhang W,Ingo H,Gilroy E M,Tian Z. A Phytophthora infestans RXLR effector targets plant PP1c isoforms that promote late blight disease[J]. Nature Communications,2016,7:10311. doi:10.1038/ncomms10311.
[28] 王妮妍,蒋德安. 茉莉酸及其甲酯与植物诱导抗病性[J]. 植物生理学报,2002,38:279-284. doi:10.13592/j .cnki .ppj .2002.03.033.
Wang N Y,Jiang D A. The role of Jasmonic acid and methyl jasmonate in plant induced disease resistance[J]. Plant Physiology Journal,2002,38:279-284.
[29] Birch P R,Armstrong M,Bos J,Boevink P,Gilroy E M,Taylor R M,Wawra S,Pritchard L,Conti L,Ewan R. Towards understanding the virulence functions of RXLR effectors of the oomycete plant pathogen Phytophthora infestans[J]. Journal of Experimental Botany,2009,60:1133. doi:10.1093/jxb/ern353.
[30] Balaji V,Sessa G,Smart C D. Silencing of host basal defense response-related gene expression increases susceptibility of Nicotiana benthamiana to Clavibacter michiganensis subsp.michiganensis[J]. Phytopathology,2011,101:349-357. doi:10.1094/PHYTO-05-10-0132.
[31] 胡婷丽,李魏,刘雄伦,戴良英. 泛素化在植物抗病中的作用[J]. 微生物学通报,2014,41:1175-1179. doi:10.13344/j.microbiol.china.140148.
Hu T L,Li W,Liu Xi L,Dai L Y. The role of ubiquitination in plant disease resistance[J]. Microbiology China,2014,41:1175-1179.
[32] Vierstra R D. The ubiquitin-26S proteasome system at the nexus of plant biology[J]. Nature Reviews Molecular Cell Biology,2009,10(6):385-397. doi:10.1038/nrm2688.
[33] Spoel S H,Mou Z,Tada Y. Proteasome-mediated turnover of the transcription coactivator NPR1 plays dual roles in regulating plant immunity[J]. Cell,2009,137(5):860-872. doi:10.1016/j.cell.2009.03.038.
[34] Backer R,Mahomed W,Reeksting B J,Engelbrecht J,Ibarralaclette E,Berg N V D. Phylogenetic and expression analysis of the NPR1-like gene family from Persea americana(Mill.)[J]. Frontiers in Plant Science,2015,6:300. doi:10.3389/fpls.2015.00300.
[35] Liu X Y,Wang J Y,Fan B L,Shang Y T,Sun Y F,Dang C,Xie C J,Wang Z Y,Peng Y K. A coi1 gene in wheat contributes to the early defence response against wheat powdery mildew[J]. Journal of Phytopathology,2018,166(2):116-122. doi:10.1111/jph.12667.
[36] Zhou J,Lu D,Xu G,Finlayson S A,He P,Shan L. The dominant negative arm domain uncovers multiple functions of pub13 in arabidopsis immunity,flowering,and senescence[J]. Journal of Experimental Botany,2015,66(11):3353. doi:10.1093/jxb/erv148.