植物在生长和发育的不同阶段,会遇到各种生物及非生物胁迫,例如昆虫、机械损伤、干旱、冷、热、盐等。在胁迫条件下,细胞常常受到不同程度的损伤,然而,植物在漫长的进化过程中产生了多种适应或抵抗这些逆境条件的机制[1-3],其中,转录激活调控是胁迫应答调控机制之一。转录因子,例如NAC、MYB、WRKY等调控下游基因转录[4-5]。
NAC转录因子(Transcription factor,TF)是植物特有的最大转录因子家族之一,参与调控植物生命活动的各阶段[6]。NAC转录因子由3类基因组成:牵牛花中发现的NAM(No apical meristem)、拟南芥中的ATAF(Arabidopsis transcription activation factor)和CUC(Cup-shaped cotyledon)[7]。NAC转录因子家族基因具有保守的NAC结构域,该结构域由大约150个氨基酸构成,位于氨基酸链的N末端,高度保守,作为DNA结合结构域;C端相对可变,作为转录激活结构域[8]。NAC转录因子参与调控植物生长发育及逆境胁迫应答,在不同物种中,已有许多NAC基因的功能被报道:Mahmood等[9-10]研究表明,ANAC032促进拟南芥衰老的同时,在蔗糖、盐、氧化等胁迫条件下,也抑制拟南芥花青素的合成;拟南芥ANAC046能促进拟南芥叶绿素降解和叶片衰老[11];Chen等[12]研究表明,小麦中过表达TaRNAC1基因促进小麦根系生长、增强抗旱性;中间锦鸡儿NAC转录因子分析表明,过表达CiNAC1和CiNAC4促进拟南芥主根的伸长及侧根数目的增加[13],CiNAC3和CiNAC4提高转基因拟南芥对盐胁迫的耐受性[14]。
本研究以抗干旱、耐贫瘠、耐寒、耐酷热的豆科锦鸡儿属灌木中间锦鸡儿(Caragana intermedia)为材料,克隆NAC家族基因CiATAF1,利用生物信息学方法对其序列及结构进行分析。经荧光定量PCR检测了CiATAF1基因的组织表达特异性及其在干旱、盐、冷、ABA条件下的表达模式,为后续研究该基因的功能提供依据。
1 材料和方法
1.1 试验材料
本试验所用材料中间锦鸡儿采自内蒙古自治区乌兰察布市四子王旗;大肠杆菌感受态、克隆载体pEASY-Blunt Simple购自北京全式金生物技术有限公司;拟南芥野生型(Col-0)、表达载体pCAMBIA1302保存于内蒙古植物逆境生理与分子生物学重点实验室;限制性内切酶、T4 DNA连接酶购自Thermo公司;氯化钠(分析纯)购自天津市风船化学试剂科技有限公司;总RNA试剂盒购自天根生化科技(北京)有限公司;荧光定量分析试剂购自Roche公司;所用到引物由Primer Premier 5设计,见表1,并在上海生工生物工程股份有限公司合成。
表1 引物序列
Tab.1 Primer sequence

引物用途Major application引物名称Primer name引物序列(5'-3')Primer sequence (5'-3')CiATAF1基因3'RACERACE-ATAF1-senseCAGAGCGAGCCAAAGTGGAACGAGTGCiATAF1 gene 3'RACECiATAF1基因5'RACERACE-ATAF1-antiCCGTTCGGGTATTTCCGGTCTCGCCiATAF1 gene 5'RACECiATAF1基因特异引物HA-ATAF1-senseGCGTCGACAACATGGCATCTGAGCTTCAACiATAF1 specific primersHA-ATAF1-antiGCACTAGTTCAGAAGGGCTTGGGCAACiATAF1基因特异引物(GFP)GFP-ATAF1-senseGCCCATGGACATGGCATCTGAGCTTCAATTCiATAF1 specific primers(GFP)GFP-ATAF1-antiGCACTAGTTCAGAAGGGCTTGGGCAACiATAF1基因qRT-PCR引物G-ATAF1-senseTAAGGCTGGATGACTGGGTGCCiATAF1 gene qRT-PCR prim-ersG-ATAF1-antiCACTCGTTCCACTTTGGCTCGCiATAF1启动子克隆ATAF1-SP1TTCGGGTGACCAATGGGCTTATCCiATAF1 promotor clonedATAF1-SP2CCTCGAACCGTTCGGGTATTTCATAF1-SP3CCCAAGGATCGTATTTGTAGAGGTCA
1.2 材料处理
选取颗粒饱满的中间锦鸡儿种子种植于蛭石与营养土比例为3∶1的钵子中,在25 ℃长日照(18 h光照/6 h黑暗)培养,21 d后取长势较好的植株作为试验材料。
组织表达特异性:从钵子中选取长势较好的中间锦鸡儿3株,蒸馏水去除根部泥土,将根、茎、叶分离,分别在液氮条件下冷冻,保存于-80 ℃冰箱。另取3株中间锦鸡儿整株同上条件保存。
干旱处理:中间锦鸡儿最后一次浇水3 d后开始取样,一直持续停止浇水,每隔3 d取一次样品,每次样品取3株。干旱12 d后开始复水,复水3 d后最后一次取样。取样时间为0,3,6,9,12 d和复水3 d。
NaCl处理:中间锦鸡儿最后一次浇水3 d后,取对照0 h样品,立即浇3 L的200 mmol/L NaCl,取样时间点为0,0.5,1,3,6,12,24,48 h。
冷处理:处理前取对照0 h样品,立即将中间锦鸡儿置于4 ℃培养箱,然后根据NaCl处理的取样时间点取处理后样品。
ABA处理:处理前取对照0 h样品,立即向中间锦鸡儿叶片喷浓度为100 μmol/L的ABA(加0.05%的Tween)后取样,然后根据NaCl处理的取样时间点取处理后样品。以水(加0.05%的Tween)为对照。
1.3 中间锦鸡儿总RNA提取与反转录
中间锦鸡儿样品在液氮条件下低温研磨,取60~70 mg按照总RNA试剂盒步骤提取。琼脂糖凝胶电泳检测RNA的纯度和是否降解,同时,使用超微量紫外分光光度计对RNA浓度进行测量。选取2 μg浓度较高且纯度较好的RNA,反转录生成cDNA,作为基因克隆的模板。
1.4 CiATAF1基因克隆
在PrimeSTAR酶的催化下,PCR扩增条件:98 ℃预变性1 min,98 ℃变性10 s,60 ℃退火10 s,72 ℃延伸1 min,72 ℃补充延伸10 min,30个循环。
1.5 CiATAF1基因亚细胞定位
取5~7个21 d左右野生型拟南芥叶片,经纤维素酶和离析酶酶解,获得野生型拟南芥原生质体。稀释原生质体至浓度为每毫升200 000个,取100 μL原生质体同20 μg CiATAF1-GFP重组质粒混匀,在PEG介导下CiATAF1基因瞬时表达。黑暗条件下,25 ℃培养16 h。在荧光显微镜下,观察CiATAF1-GFP重组质粒亚细胞定位情况。
1.6 中间锦鸡儿CiATAF1基因生物信息学分析
CiATAF1基因序列在NCBI网站分析(https://blast.ncbi.nlm.nih.gov/Blast.cgi);通过PlantCARE在线数据库(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)分析CiATAF1启动子顺式作用元件[15];通过ExPASY在线数据库(https://www.expasy.org/resources)中ProtParam预测分析CiATAF1蛋白质理化性质;通过SOMPA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl? page=npsa_sopma .html)在线分析CiATAF1蛋白二级结构[16]。
2 结果与分析
2.1 CiATAF1基因克隆
从内蒙古植物逆境生理与分子生物学重点实验室的干旱转录组数据库中获得CiATAF1基因序列,经比对分析,该序列没有完整开放阅读框(Open reading frame,ORF),且两端均不完整。设计3′-RACE引物(RACE-ATAF1-sense)和5′-RACE引物(RACE-ATAF1-anti),以中间锦鸡儿cDNA为模板,按照RACE体系进行PCR扩增。产物于1%琼脂糖凝胶电泳,测序分析后,将2次RACE序列同已有基因序列拼接,获得CiATAF1基因全长,共1 805 bp,包括完整ORF及非编码区。
在已获得基因序列基础之上,设计CiATAF1基因全长特异性引物GFP-ATAF1-sense和GFP-ATAF1-anti。以中间锦鸡儿cDNA为模板,PCR扩增获得CiATAF1基因ORF全长序列(图1),共873 bp。同时,以中间锦鸡儿gDNA为模板,增加延伸时间至2 min,其他条件不变,获得CiATAF1基因gDNA全长(图1),共1 445 bp。
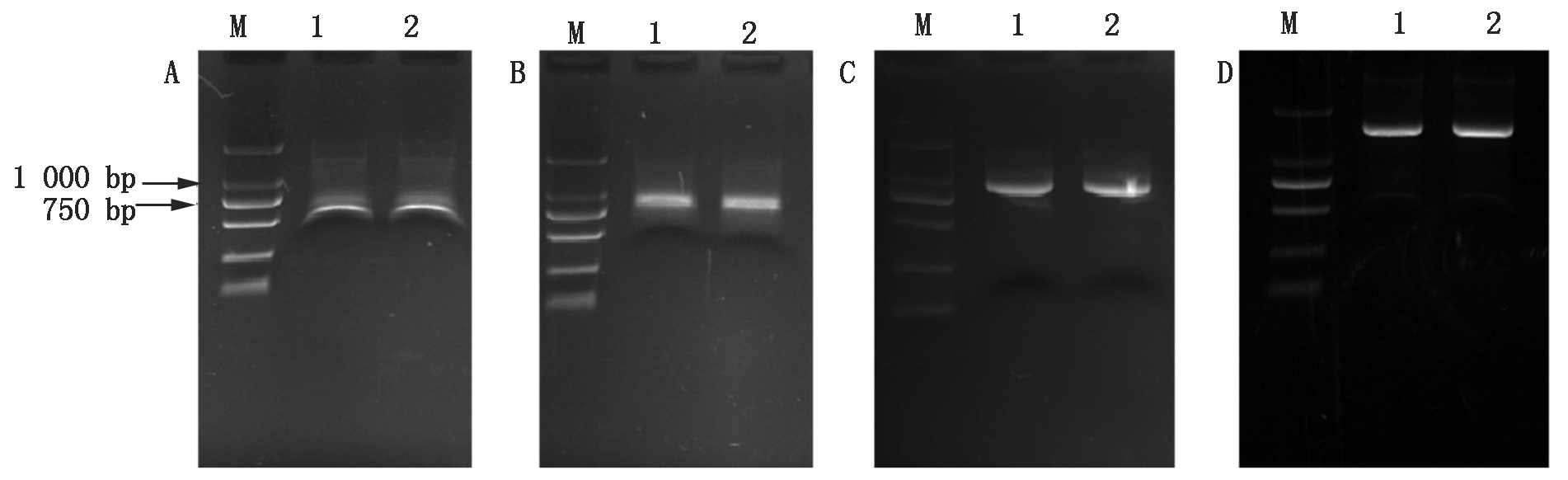
A.5′-RACE;B.3′-RACE;C.cDNA全长;D.gDNA全长; M.DL2000; 1-2.相同的PCR产物。
A.5′-RACE; B.3′-RACE; C.Full length cDNA;D.Full length gDNA; M.DL2000 DNA Marker; 1-2.The same PCR product.
图1 CiATAF1基因克隆电泳
Fig.1 Agarose gel electrophoresis of the PCR products of the CiATAF1 gene cloned
2.2 CiATAF1基因序列分析
CiATAF1基因的核苷酸序列及其编码氨基酸序列分析(图2),CiATAF1基因组DNA序列全长为1 445 bp,由2个内含子和3个外显子构成。ORF全长为873 bp,编码291个氨基酸,起始密码子为ATG,终止密码子为TGA,图中大写字母表示编码区序列,小写字母表示非编码区序列。NCBI网站Blast分析表明,CiATAF1基因具有完整的NAC结构域,位于28-105个氨基酸,是典型的NAC转录因子家族基因。

图2 CiATAF1基因的全长序列及推导的氨基酸序列
Fig.2 Full-length and deduced amino acid sequences of CiATAF1
2.3 CiATAF1基因启动子克隆及顺式作用元件分析
设计并获得CiATAF1基因启动子克隆引物ATAF1-SP1、ATAF1-SP2、ATAF1-SP3,以中间锦鸡儿gDNA为模板,通过染色体步移法,克隆CiATAF1基因侧翼启动子序列(图3)。经过3轮PCR扩增,获得特异性条带,长度在750~1 000 bp,回收该片段,并经测序分析,该片段为CiATAF1基因的启动子。
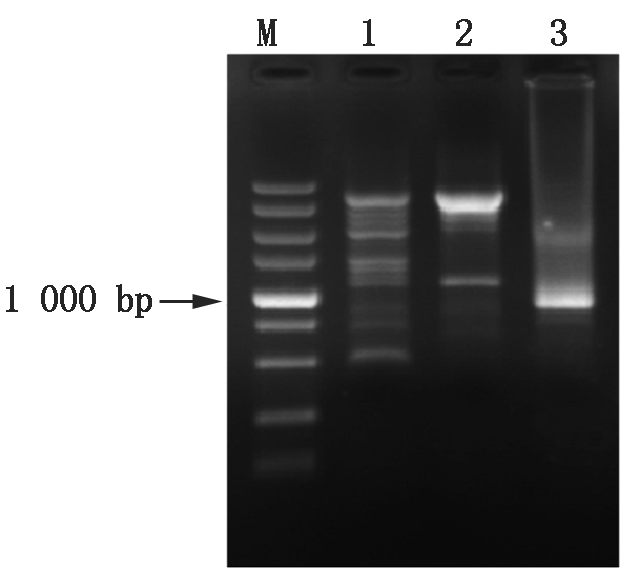
M.DL5000; 1.第1轮PCR; 2.第2轮PCR; 3.第3轮PCR。
M.DL5000 DNA Marker; 1.The first round of PCR; 2.The second round of PCR; 3.The third round of PCR.
图3 CiATAF1基因启动子克隆电泳
Fig.3 Agarose gel electrophoresis of CiATAF1 gene promoter cloned
扩增获得CiATAF1基因启动子序列为826 bp。经PlantCARE在线分析(表2),该启动子序列除真核生物普遍具有的TATA-box和CAAT-box之外,还包括许多光响应元件(G-Box、GT1-motif、Sp1等)、植物激素脱落酸响应元件(ABRE)、厌氧诱导相关元件(ARE)以及MYB结合位点(MBS)。
表2 CiATAF1基因启动子顺式作用元件分析
Tab.2 CiATAF1 promotor cis-acting elements analysis

元件名称Regulatory elements序列Sequence可能的功能Possible function数量QuantityCAAT-boxCAAT、CAAAT启动子中常见的顺式作用元件7TATA-box TATATA、TATA核心启动子元件11ABREGGACACGTGGC 脱落酸应答5ARETGGTTT厌氧诱导1AT1-motifATTAATTTTACA光响应1Box 4ATTAAT1G-BoxCACGTG10ACACGTGTCACCACACGTGTGT1-motifGGTTAA1GATA-motifGATAGGA1Sp1CC(G/A)CCC2Box ⅡTGGTAATAA1MBSCAACTG干旱诱导MYB结合位点1
2.4 CiATAF1基因亚细胞定位
经NcoⅠ和SpeⅠ内切酶催化,将CiATAF1基因片段从克隆载体pEASY-Blunt Simple酶切并回收,连接线性化pCAMBIA302-GFP载体。连接成功后通过NcoⅠ和SpeⅠ双酶切验证,如图4,产生约为1 000 bp长的片段,表明重组质粒构建成功。
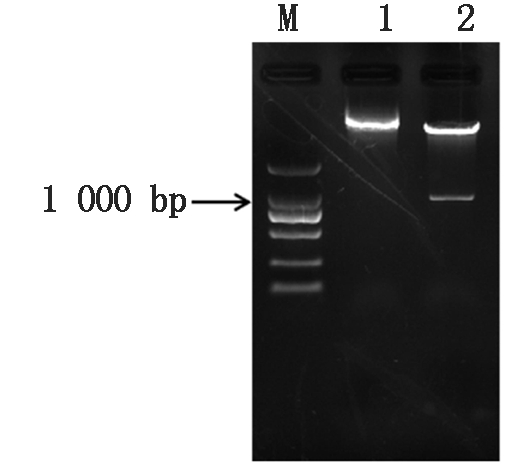
M.DL5000; 1.CiATAF1-GFP融合表达载体对照; 2.双酶切CiATAF1-GFP融合表达载体。
M.DL5000 DNA Marker;1.CiATAF1-GFP fusion expression vector control; 2.Double enzymes digestion of CiATAF1-GFP fusion expression vector.
图4 CiATAF1基因与GFP融合的双元表达载体酶切鉴定
Fig.4 Identification of the binary vector containing CiATAF1 fused GFP
PEG法分别转化35S∶ GFP空载体与CiATAF1-GFP融合表达载体至拟南芥原生质体,观察亚细胞定位情况。在荧光显微镜下,如图5,35S∶ GFP空载体的绿色荧光蛋白分布于整个细胞,而CiATAF1-GFP融合表达载体的绿色荧光蛋白集中分布于细胞核中。该试验证明CiATAF1基因同一般其他转录因子类似,在细胞核中行使其转录调控功能。
2.5 CiATAF1基因组织特异性表达
以中间锦鸡儿根、茎、叶及整株的cDNA为模板,荧光定量PCR检测CiATAF1基因组织表达特异性。结果表明,CiATAF1基因在中间锦鸡儿叶片中表达量最高,在根中表达量最少(图6-A)。
2.6 CiATAF1基因在不同胁迫条件下表达分析
以干旱、NaCl、冷、ABA处理后中间锦鸡儿cDNA为模板,检测CiATAF1基因在胁迫处理下表达情况。在干旱处理条件下,CiATAF1基因从第3天开始到第12天,表达量持续增加,复水3 d后,基因表达量恢复到处理前水平(图6-B);在NaCl处理条件下,CiATAF1基因从0.5 h开始表达量增加,在48 h时,表达量开始下降,但处理过程总体基因表达量变化不大(图6-C);冷处理后,CiATAF1基因在0.5~1 h没有显著变化,从3 h开始,该基因表达量增加,直到冷处理后48 h(图6-D);以水作为处理对照(图6-E),经ABA处理后,CiATAF1基因除了在0.5 h表达量略高外,整体上在0~12 h内表达量呈现上升趋势,并在12~24 h表达量达到最高,约为处理前的6倍(图6-F)。试验表明,CiATAF1基因在干旱、盐、冷以及植物激素ABA处理下表达上调,预测该基因可能参与植物非生物胁迫应答机制的调控。
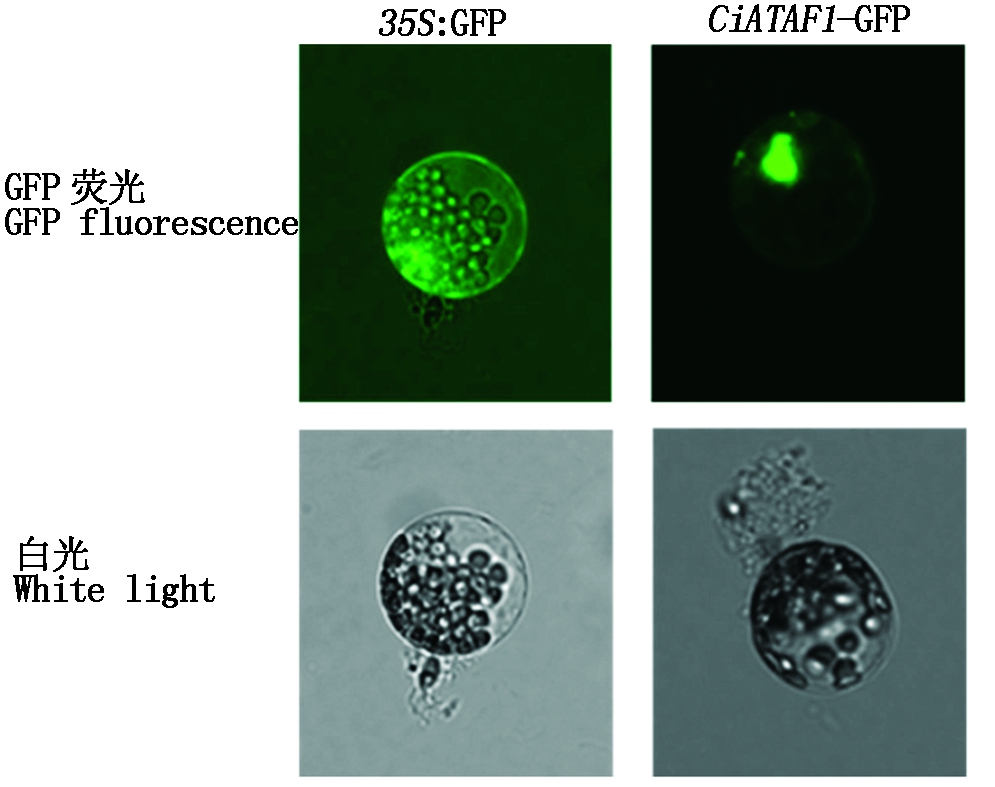
图5 CiATAF1基因在拟南芥叶肉原生质体亚细胞定位
Fig.5 Subcellular localization of CiATAF1 in Arabidopsis mesophyll protoplast

图6 CiATAF1基因表达模式分析
Fig.6 Analysis of CiATAF1 gene expression patterns
2.7 CiATAF1多重序列比对
CiATAF1蛋白序列同模式植物拟南芥(Arabidopsis thaliana)和豆科其他植物鹰嘴豆(Cicer arietinum)、红车轴草(Trifolium pratense)、蒺藜苜蓿(Medicago truncatula)、大豆(Glycine max)、赤豆(Vigna angularis)、菜豆(Phaseolus vulgaris)、绿豆(Vignaradiata var. radiata)、落花生(Arachis hypogaea)以及狭叶羽扇豆(Lupinus angustifolius)的同源基因进行多序列比对分析,如图7。序列一致性为81.14%,CiATAF1在各植物中保守性较强,主要集中在氨基末端结构域。

图7 CiATAF1多重序列比对分析
Fig.7 CiATAF1 multiple sequence alignment analysis
2.8 CiATAF1蛋白生物信息学分析
通过ExPASY在线数据库中ProtParam预测分析,CiATAF1蛋白分子质量为33.194 ku,等电点(Isoelectric point,pI)为7.64,不稳定指数(Instability index,Ⅱ)为59.74,平均亲水性为(Grand average of hydropathicity,GRAVY)-0.751,属不稳定的亲水蛋白。
通过SOMPA在线分析CiATAF1蛋白二级结构,该蛋白的二级结构主要由α-螺旋、β折叠、无规则卷曲构成。其中,α-螺旋共40个氨基酸,占氨基酸总数的13.75%;β折叠共60个氨基酸,占氨基酸总数的20.62%;无规则卷曲共191个氨基酸,占氨基酸总数的65.64%。
3 结论与讨论
本研究以中间锦鸡儿为材料,克隆并分析CiATAF1基因。CiATAF1基因具有完整的NAC结构域,是典型的NAC转录因子家族基因。对CiATAF1基因做了不同组织部位和不同非生物胁迫下的定量分析,研究表明CiATAF1基因的表达具有组织特异性,在中间锦鸡儿叶片中表达量最高,在根中表达量最低;同时,CiATAF1基因受到干旱、盐、冷以及植物激素ABA的诱导表达,推测CiATAF1基因可能参与植物非生物胁迫的应答调控。经染色体步移法克隆了CiATAF1启动子,长度为826 bp。CiATAF1启动子序列中包括多种光响应元件、植物激素响应元件等。构建CiATAF1-GFP融合表达载体,PEG诱导转化拟南芥叶片原生质体瞬时表达分析表明,CiATAF1基因定位于细胞核中,这与Lu等[17]研究表明拟南芥ATAF1基因定位于细胞核中的结果一致。
本研究尚未开展CiATAF1基因的功能验证试验,但ATAF1是拟南芥NAC转录因子家族中发现较早的基因之一,其结构和功能已经有较为完整的分析。已经有研究表明,拟南芥ATAF1基因通过调控胁迫相关基因表达,参与植物对干旱、高盐、ABA等非生物胁迫应答[17-19];另外,在拟南芥生长生理方面,ATAF1基因作为上游衰老激活因子,促进植物衰老[20]。也有研究表明, ATAF1基因促进ABA的合成[21];He等[22]在棉花中发现GhATAF1基因促进GhAVP1、GhRD22、GhDREB2A、GhLEA3和GhLEA6等胁迫相关基因表达,且增强植物对病原菌侵染的敏感性。综上所述,ATAF1基因确实参与到植物生长发育的各个阶段,且参与调控生物及非生物胁迫应答中。在此基础上,将进一步探究及分析中间锦鸡儿CiATAF1基因的功能。
[1] Shim J S,Oh N,Chung P J,Kim Y S,Choi Y D,Kim J K.Overexpression of OsNAC14 improves drought tolerance in rice[J]. Frontiers in Plant Science,2018(9):310-324.doi:10.3389/fpls.2018.00310.
[2] 张红梅,刘晓庆,陈华涛,袁星星,崔晓艳,张智民,黄中文,陈新. 大豆转录因子GmWRKY58亚细胞定位及在非生物胁迫下的表达分析[J]. 华北农学报,2018,33(2):49-57.doi:10.7668/hbnxb.2018.02.008.
Zhang H M,Liu X Q,Chen H T,Yuan X X,Cui X Y,Zhang Z M,Huang Z W,Chen X. Subcellular localization and expression analysis of a soybean transcription factor GmWRKY58 in response to abiotic stresses[J].Acta Agriculturae Boreali-Sinica,2018,33(2):49-57.
[3] 朱长保,刘志,徐辰峰,刘仁梅,林中正,成立瑞. 全基因组分析盐胁迫下水稻转录因子的表达[J]. 北方农业学报,2018,46(6):38-43.doi:10.3969/j.issn.2096-1197.2018.06.08.
Zhu C B,Liu Z,Xu C F,Liu R M,Lin Z Z,Cheng L R. Global genome expression analysis of transcription factors in rice under salt stress[J].Journal of Northern Agriculture,2018,46(6):38-43.
[4] 田李,孙景宽,刘京涛. 植物转录因子家族在耐盐抗旱调控网络中的作用[J]. 生命科学,2015,27(2):217-227.doi:10.13376/j.cbls/2015031.
Tian L,Sun J K,Liu J T. Role of different transcription factor families in the regulatory networks ofdrought and salinity tolerance in plants[J].Chinese Bulletin of Life Sciences,2015,27(2):217-227.
[5] Soutourina J. Transcription regulation by the Mediator complex[J]. Nat Rev Mol Cell Biol,2018,19(4):262-274.doi:10.1038/nrm.2017.115.
[6] Wu D,Sun Y,Wang H,Shi H,Su M,Shan H,Li T,Li Q. The SlNAC8 gene of the halophyte Suaeda liaotungensis enhances drought and salt stress tolerance in transgenic Arabidopsis thaliana[J]. Gene,2018,662:10-20.doi:10.1016/j.gene.2018.04.012.
[7] Kamiuchi Y,Yamamoto K,Furutani M,Tasaka M,Aida M. The CUC1 and CUC2 genes promote carpel margin meristem formation during Arabidopsis gynoecium development[J]. Frontiers in Plant Science,2014(5):1-9.doi:10.3389/fpls.2014.00165.
[8] Olsen A N,Ernst H A,Leggio L L,Skriver,K. NAC transcription factors:structurally distinct,functionally diverse[J]. Trends in Plant Science,2005,10(2):79-87.doi:10.1016/j.tplants.2004.12.010.
[9] Mahmood K,El-Kereamy A,Kim S H,Nambara E,Rothstein S J. ANAC032 positively regulates age-dependent and stress-induced senescence in Arabidopsis thaliana[J]. Plant and Cell Physiology,2016,57(10):2029-2046.doi:10.1093/pcp/pcw120.
[10] Mahmood K,Xu Z H,El-Kereamy A,Casaretto J A,Rothstein S J. The Arabidopsis transcription factor ANAC032 represses anthocyanin biosynthesis in response to high sucrose and oxidative and abiotic stresses[J]. Frontiers in Plant Science,2016(7):1-15.doi:10.3389/Fpls.2016.01548.
[11] Oda-Yamamizo C,Mitsuda N,Sakamoto S,Ogawa D,Ohme-Takagi M,Ohmiya A. The NAC transcription factor ANAC046 is a positive regulator of chlorophyll degradation and senescence in Arabidopsis leaves[J]. Sci Rep,2016(6):1-12.doi:10.1038/srep23609.
[12] Chen D,Chai S,McIntyre C L,Xue G P. Overexpression of a predominantly root-expressed NAC transcription factor in wheat roots enhances root length,biomass and drought tolerance[J]. Plant Cell Reports,2018,37(2):225-237.doi:10.1007/s00299-017-2224-y.
[13] 岳俊燕,岳文冉,杨杞,于秀敏,杨飞芸,王光霞,李国婧,王瑞刚,丛靖宇. 中间锦鸡儿转录因子基因CiNAC1的克隆及功能分析[J]. 西北植物学报,2016,36(7):1285-1293.doi:10.7606/j.issn.1000-4025.2016.07.1285.
Yue J Y,Yue W R,Yang Q,Yu X M,Yang F Y,Wang G X,Li G J,Wang R G,Cong J Y. Cloning and functional analysis of CiNAC1 from Caragana intermedia[J].Acta Botanica Boreali-Occidentalia Sinica,2016,36(7):1285-1293.
[14] Han X,Feng Z,Xing D,Yang Q,Wang R,Qi L,Li G. Two NAC transcription factors from Caragana intermedia altered salt tolerance of the transgenic Arabidopsis[J]. Bmc Plant Biology,2015,15(1):208-211.doi:10.1186/s12870-015-0591-5.
[15] Lescot M,Dehais P,Thijs G,Marchal K,Moreau Y,Van de Peer Y,Rouze P,Rombauts S. PlantCARE,a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences[J]. Nucleic Acids Research,2002,30(1):325-327.doi:10.1093/nar/30.1.325.
[16] Artimo P,Jonnalagedda M,Arnold K,Baratin D,Csardi G,de Castro E,Duvaud S,Flegel V,Fortier A,Gasteiger E,Grosdidier A,Hernandez C,Ioannidis V,Kuznetsov D,Liechti R,Moretti S,Mostaguir K,Redaschi N,Rossier G,Xenarios I,Stockinger H.ExPASy:SIB bioinformatics resource portal[J]. Nucleic Acids Research,2012,40(W1):W597-W603.doi:10.1093/nar/gks400.
[17] Lu P L,Chen N Z,An R,Su Z,Qi B S,Ren F,Chen J,Wang X C. A novel drought-inducible gene,ATAF1,encodes a NAC family protein that negatively regulates the expression of stress-responsive genes in Arabidopsis[J]. Plant Molecular Biology,2007,63(2):289-305.doi:10.1007/s11103-006-9089-8.
[18] Wu Y R,Deng Z Y,Lai J B,Zhang Y Y,Yang C P,Yin B J,Zhao Q Z,Zhang L,Li Y,Yang C W,Xie Q. Dual function of Arabidopsis ATAF1 in abiotic and biotic stress responses[J]. Cell Research,2009,19(11):1279-1290.doi:10.1038/cr.2009.108.
[19] Liu Y C,Sun J,and Wu Y R. Arabidopsis ATAF1 enhances the tolerance to salt stress and ABA in transgenic rice[J]. Journal of Plant Research,2016,129(5):955-962.doi:10.1007/s10265-016-0833-0.
[20] Garapati P,Xue G P,Munne-Bosch S,Balazadeh S. Transcription Factor ATAF1 in Arabidopsis promotes senescence by direct regulation of key chloroplast maintenance and senescence transcriptional cascades[J]. Plant Physiology,2015,168(3):1122-1139.doi:10.1104/pp.15.00567.
[21] Jensen M K,Lindemose S,de Masi F,Reimer J J,Nielsen M,Perera V,Workman C T,Turck F,Grant M R,Mundy J,Petersen M,Skriver K. ATAF1 transcription factor directly regulates abscisic acid biosynthetic gene NCED3 in Arabidopsis thaliana[J]. Febs Open Bio,2013(3):321-327.doi:10.1016/j.fob.2013.07.006.
[22] He X,Zhu L,Xu L,Guo W F,Zhang X L. GhATAF1,a NAC transcription factor,confers abiotic and biotic stress responses by regulating phytohormonal signaling networks[J]. Plant Cell Reports,2016,35(10):2167-2179.doi:10.1007/s00299-016-2027-6.