草莓(Fragaria vesca)属于蔷薇科草莓属,果实富含多种维生素、矿物质等营养成分,植株易栽培、繁殖快、成本低及收益高等特点成为我国果业和设施农业的新兴产业。草莓果实是由子房进一步膨大而形成[1]。植物激素分泌异常对于果实的生长发育有重要的影响,如生长素分泌过多则引起草莓花芽分化异常,并最终导致草莓畸形果的形成[2-3]。因此,了解生长素在草莓果实发育的功能对于通过分子育种手段培养优良的草莓品种具有重要的指导意义。
生长素响应因子(Auxin response factor,ARF)是生长素信号途径的重要组成部分之一,能够与生长素响应基因的启动子区结合,从而特异性地调节下游基因的表达,在生长素信号转导途径中有着重要作用[4-5]。ARF家族基因存在于大多数植物体内,目前已经在拟南芥[6]、甜橙[7]、葡萄[8]、香蕉[9]、番茄[10]等多种植物中得到鉴定和分离,ARF家族基因对植物的多个生长发育过程均有调控作用[11-15]。
ARF5是ARF家族中的一员,研究表明,拟南芥ARF5/MONOPTEROS(MP)功能缺失可以导致其胚胎和幼苗发育缺陷,拟南芥ARF5/MP基因功能丧失的突变体植株因无法形成根尖而导致胚胎死亡[16-18],近期研究表明,ARF5/MP通过调控靶基因细胞分裂素响应因子2(Cytokinin response factor 2,CRF2)和茎端分生组织特异性调控因子(Shoot meristemless,STM)进而调控拟南芥芽组织的形成[19],且拟南芥ARF5基因可通过影响轴向极性调控叶片和茎的发育[20]。番茄中,抑制表达SlARF5基因导致番茄单性结实,并通过调控生长素/赤霉素信号通路中的关键因子调控细胞的分裂及膨大过程,进而影响番茄果实的大小,ARF5可能是生长素-赤霉素互作调控果实发育网络中的一个关键调控因子[21]。然而,草莓中ARF家族基因功能研究较少,草莓ARF5基因的功能尚不清楚。
本研究根据草莓基因组数据库信息中草莓ARF5的ORF序列设计特异性引物,克隆了FvARF5基因编码区全长。利用生物信息学软件分析基因的结构域及序列特征,并利用MEGA 5.0软件将草莓FvARF5与其他物种的ARF家族基因氨基酸序列进行了聚类分析。同时,采用荧光定量PCR技术分析了FvARF5在草莓不同组织中表达水平及其对外源激素处理的应答情况,为进一步分析FvARF5蛋白在草莓生长发育过程中的功能奠定基础,为将来利用分子手段进行草莓品种改良提供基因储备。
1 材料和方法
1.1 材料与试剂
供试草莓材料为丰香,由西安市农业中心草莓研究所提供。取旺盛生长期植株的不同组织,包括根、茎、叶、当天开放的花朵、绿果、1/2红果及全红果,液氮冷冻后保存于-80 ℃超低温冰箱。
本研究中所用的分子试剂,高保真聚合酶、FastStart Universal SYBR Green Master(ROX)试剂盒购自罗氏公司;去多糖多酚RNA提取试剂盒、DNA凝胶回收试剂、DNA分子量标记购自北京天根生化科技有限公司;载体购自TaKaRa; 反转录试剂盒购自Fermentas; 其他试剂购自上海生工技术服务有限公司。
1.2 外源激素处理草莓方法
草莓幼苗长至第5~6片叶完全展开后,取地上部分分别置于蒸馏水、IAA(50 mg/L)、GA(10 mg/L)、ABA(100 μmol/L)溶液中,于25 ℃、光照强度200 μmol/(m2·s)条件下培养。分别于处理后0,1,6,12,24 h取材。3个生物学重复。试材液氮冷冻后保存于-80 ℃超低温冰箱。
1.3 基因序列分析
利用DNAMAN对不同植物基因的氨基酸序列进行同源性比对;利用在线软件ProtParam进行蛋白质分子质量和等电点分析;利用Cello(http://cello.life.nctu.edu.tw/)进行FvARF5蛋白的亚细胞定位预测;利用plantCARE进行启动子序列分析;利用MEGA 5.0软件进行系统进化树分析,并生成报告图形。
1.4 果实RNA提取和FvARF5基因克隆
利用去多糖多酚RNA提取试剂盒(北京天根)和逆转录试剂盒(Fermentas)提取草莓不同组织样品RNA,并合成第一链cDNA,具体方法参照盒内说明书。利用Primer 5.0设计草莓FvARF5基因ORF序列特异性扩增引物(表1)。目标基因FvARF5的扩增体系为20 μL,包括5×Buffer 4 μL,2 μL dNTP(2.5 mmol/L),上下游引物各0.5 μL(10 mmol/L),1 μL cDNA。程序为:94 ℃预变性5 min;94 ℃ 高温变性 30 s,58 ℃ 低温退火 30 s,72 ℃中温延伸2 min,35次循环; 72 ℃ 延伸10 min。PCR产物经1%琼脂糖凝胶电泳检测,对目标片段回收纯化后,连接T载体转化大肠杆菌,筛选阳性克隆子后送上海生工测序。
1.5 FvARF5基因的表达分析
以上述cDNA为模板,以18S rRNA为内参,采用RT-PCR方法对其进行表达分析。操作步骤按照罗氏荧光定量试剂盒(FastStart Universal SYBR Green Master)说明书进行。反应体系为:总体积20 μL,包括10 μL SYBR premix,1 μL cDNA(质量浓度 30 ng/μL),上下游引物(序列见表2)各1 μL(浓度10 μmol/L),ddH2O 7 μL。qPCR试验程序为:95 ℃ 5 min;95 ℃ 15 s,60 ℃ 30 s,72 ℃ 30 s,共40循环;cDNA样品均3次生物样本重复。溶解曲线及试验结果分析参照苏丽艳等[22]。
表1 引物序列
Tab.1 The sequences of the primers

用途Use引物名称Primer name序列(5'-3')SequenceORF扩增FvARF5-FATGGAGGAGAAGATGAGAGComplete ORF amplificationFvARF5-RTTACGACACAGTGGTATG荧光定量PCRRQ-FvARF5-FACGCTACACGCAGACAAAGAFluorescent quantitative PCRRQ-FvARF5-RGCTTGCTAGGCTTCAGTCCARQ-Fv18S-FACCGTTGATTCGCACAATTGGTCATCGRQ-Fv18S-RTACTGCGGGTCGGCAATCGGACG
2 结果与分析
2.1 FvARF5基因的克隆
以提取的草莓叶片RNA为模板,反转录成cDNA后,利用PCR对草莓的FvARF5基因全长序列进行克隆,琼脂糖凝胶电泳检测扩增结果。获得约为2 700 bp的片段(图1),将目标带回收纯化后连接pMD-19T载体后提取质粒测序检测,结果与GenBank上公布的草莓ARF5基因序列(XP_004291385.1)一致。该基因开放阅读框为2 745 bp,编码914个氨基酸,命名为FvARF5。
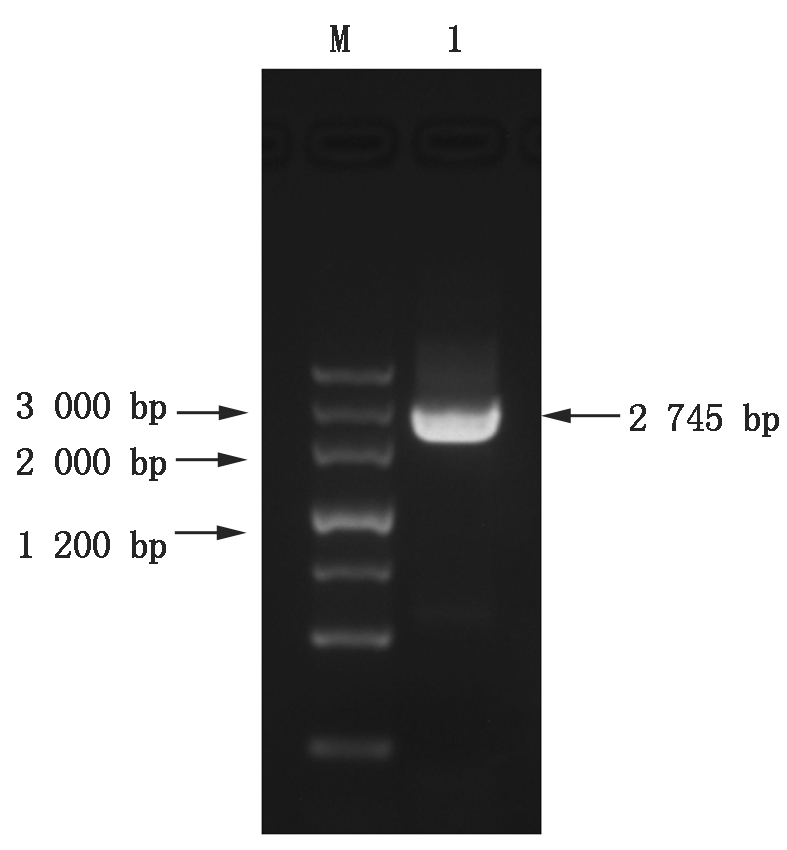
1.FvARF5基因 PCR扩增结果;M.分子量标准。
1.PCR products of FvARF5 gene; M.The molecular weight standard.
图1 FvARF5基因的PCR扩增结果
Fig.1 Agarosegel electrophoresis analysis of FvARF5 gene fragments
2.2 FvARF5蛋白生物信息学分析
2.2.1 FvARF5蛋白理化性质分析 ProtParam分析得知,FvARF5由914个氨基酸组成,分子式为C4395H6887N1217O1400S46,分子质量为100.65 ku,理论等电点为5.25,不稳定系数为58.40,系不稳定蛋白。该基因编码的多肽中苏氨酸(Ser)含量最高,约为12%,其次是赖氨酸(Leu),占总氨基酸的10.2%。其总疏水平均系数(GRAVY)为-0.397,亲水性氨基酸比例较高,与ProtScale分析结果具有一致性,可知FvARF5蛋白为亲水性蛋白(图2)。
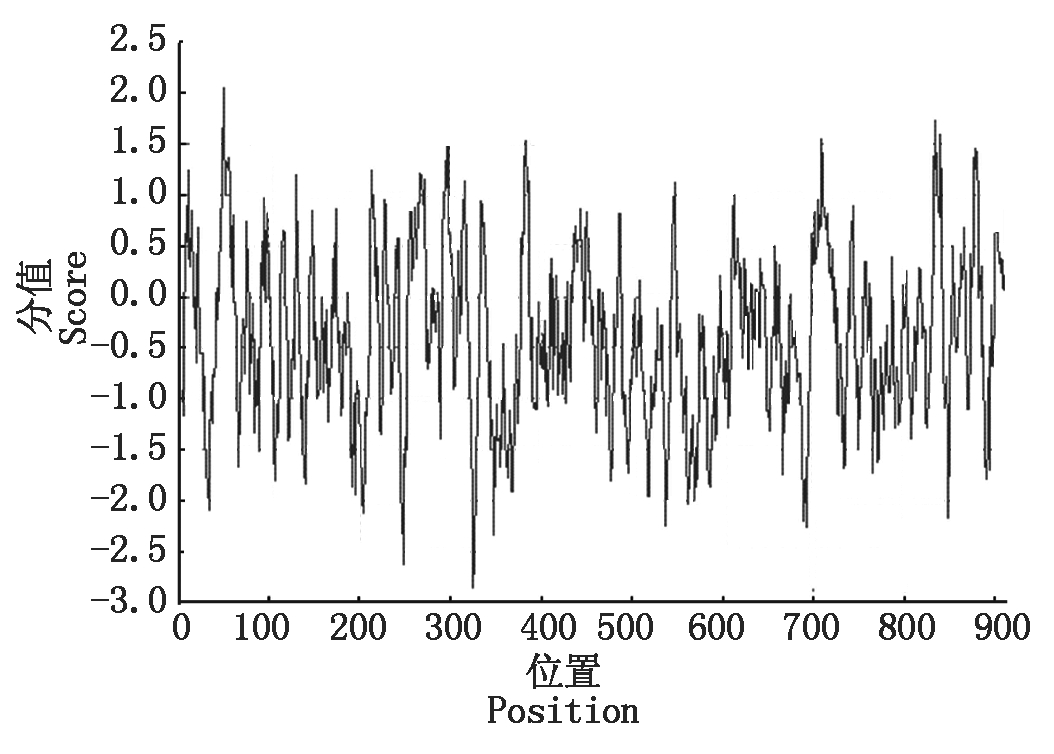
图2 FvARF5基因编码蛋白的亲疏水性预测
Fig.2 Hydrophilicity and hydrophobicity prediction of FvARF5 gene coding protein
2.2.2 FvARF5蛋白亚细胞定位预测及启动子序列分析 Cello分析结果表明,FvARF5蛋白定位于细胞核(表2),这与作为转录因子发挥作用一致。启动子序列分析表明(表3),FvARF5含有多种激素及胁迫响应功能元件,如生长素、赤霉素、脱落酸、茉莉酸、水杨酸诱导响应、热胁迫、低温、干旱胁迫、低氧、光响应、真菌胁迫响应元件等。
表2 FvARF5蛋白的亚细胞定位分析
Tab.2 Subcellular localization analysis of FvARF5 protein
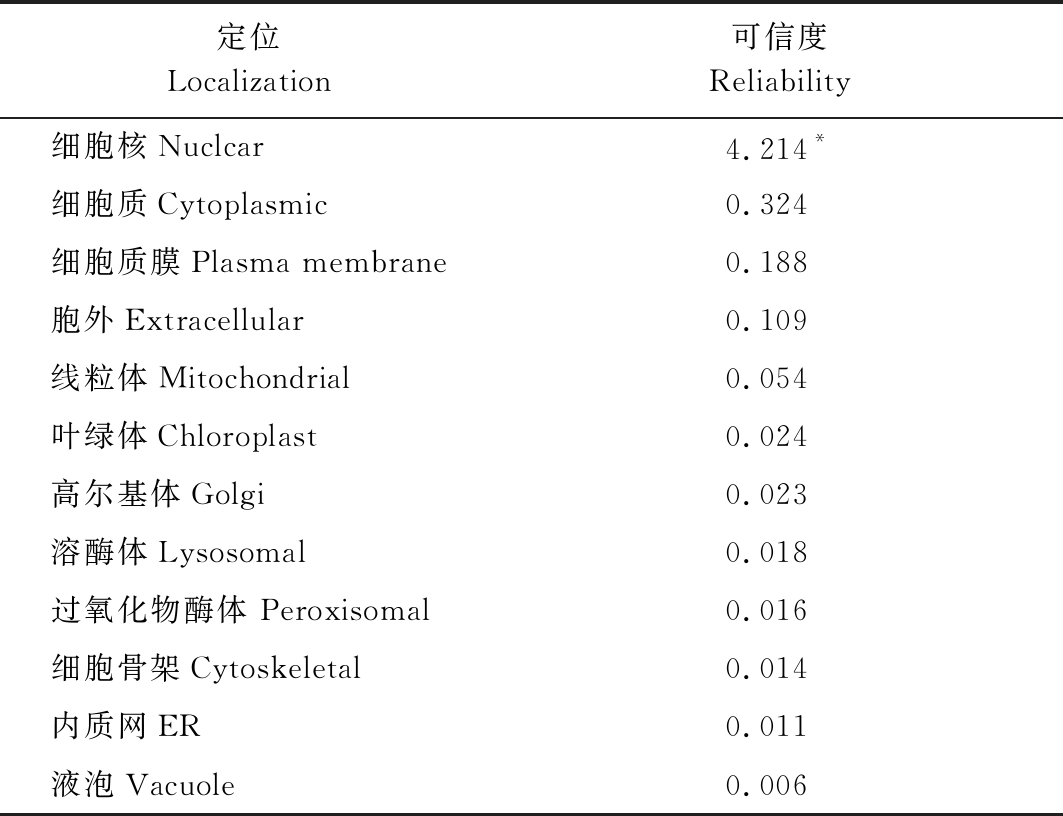
定位Localization 可信度 Reliability细胞核Nuclcar4.214*细胞质Cytoplasmic0.324细胞质膜Plasma membrane0.188胞外Extracellular0.109线粒体Mitochondrial0.054叶绿体Chloroplast0.024高尔基体Golgi0.023溶酶体Lysosomal0.018过氧化物酶体 Peroxisomal0.016细胞骨架Cytoskeletal0.014内质网ER0.011液泡Vacuole0.006
表3 FvARF5启动子顺式作用元件分析
Tab.3 The cis-elements in the promoter of FvARF5
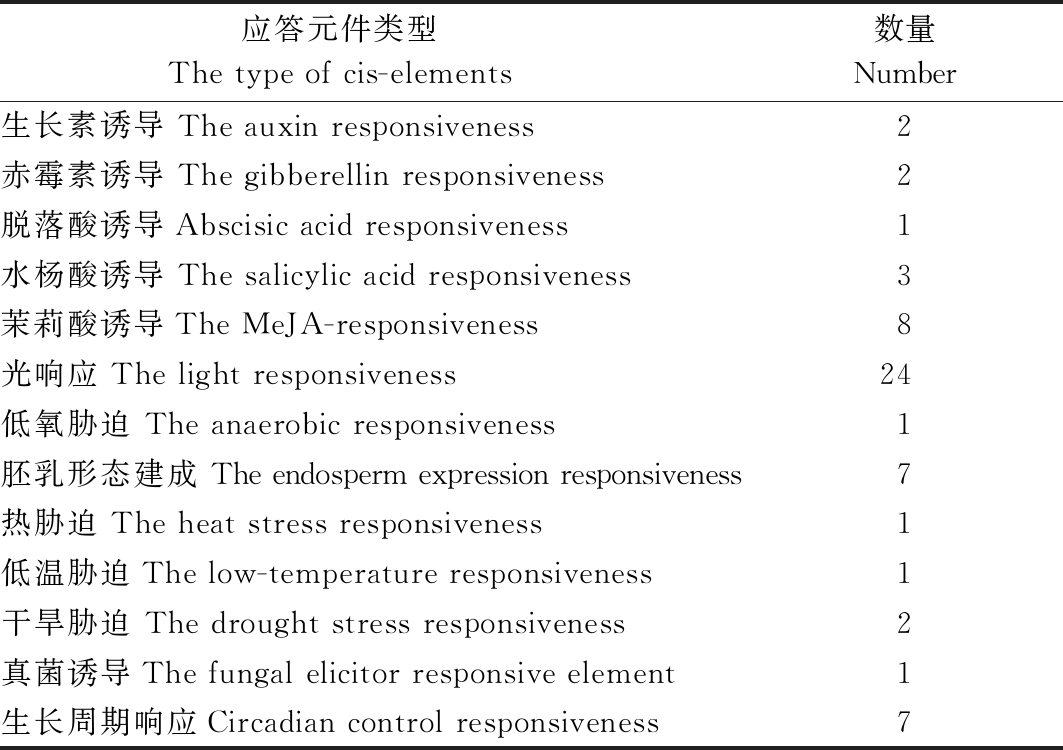
应答元件类型 The type of cis-elements 数量Number生长素诱导 The auxin responsiveness2赤霉素诱导 The gibberellin responsiveness2脱落酸诱导Abscisic acid responsiveness1水杨酸诱导 The salicylic acid responsiveness3茉莉酸诱导The MeJA-responsiveness8光响应 The light responsiveness24低氧胁迫 The anaerobic responsiveness1胚乳形态建成 The endosperm expression responsiveness7热胁迫 The heat stress responsiveness1低温胁迫The low-temperature responsiveness1干旱胁迫 The drought stress responsiveness2真菌诱导The fungal elicitor responsive element1生长周期响应Circadian control responsiveness7
2.2.3 FvARF5氨基酸序列及聚类分析 通过NCBI的CDD在线预测表明,草莓FvARF5具有生长素响应因子特有的保守结构域,即B3 DNA结合结构域和生长素响应因子保守结构域,分别位于开放阅读框的145-247和277-354。利用DNAman软件将草莓FvARF5与川桑(XP_010095167.1)、番茄(NP_001234545.1)、核桃(XP_018807020.1)、黄瓜(XP_004147836.1)、可可(XP_017981253.1)、梨(XP_018498243.1)、榴莲(XP_022769143.1)、麻风树(XP_012073833.1)、木豆(XP_020233090.1)、拟南芥(NP_173414.1)、苹果(XP_008387956.1)、葡萄(XP_003634382.2)、栓皮栎(XP_023900074.1)、桃(XP_020414821.1)、锦葵(XP_021285434.1)、杏(AVD68943.1)、樱桃(XP_021831015.1)、月季(XP_024166617.1)、枣(XP_015884813.1)进行氨基酸序列比对分析。结果表明,不同物种间的ARF5结构域具有高度相似性(图3)。家族系统进化关系分析表明,草莓FvARF5基因与蔷薇科月季ARF5基因的进化关系最近,相似度达95%(图4)。


保守结构域黑色线标记。单行线标记为B3 DNA 结合结构域,双行线为生长素响应因子保守结构域。
The conserved domains were marked by black lines. Single line was B3 DNA binding domain and double line was auxin response factor conserved domain.
图3 草莓FvARF5氨基酸序列和所选物种ARF5氨基酸序列比对
Fig.3 Analysis of the FvARF5 amino acid sequence and these selected ARF5 from other plant species

图4 草莓FvARF5系统进化关系分析
Fig.4 The phylogenetic relationships of FvARF5 from various organism
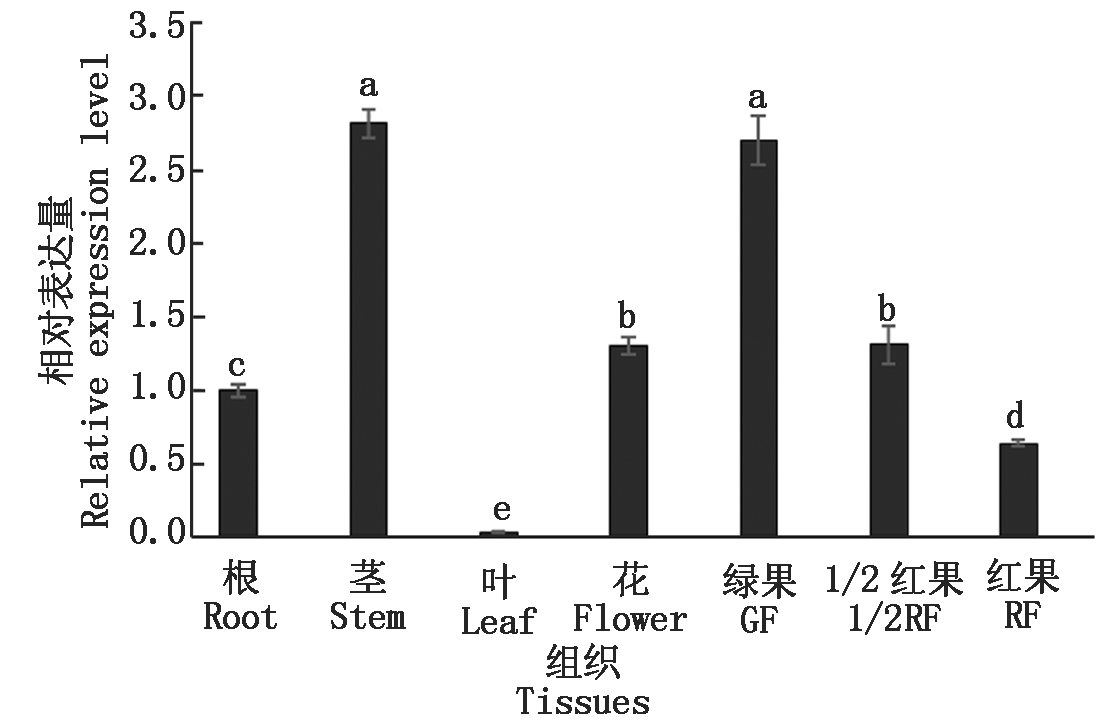
2.3 FvARF5基因在草莓不同组织中的表达分析
通过RT-qPCR检测发现,FvARF5在草莓各组织中均有表达,但相对表达量存在显著性差异,其在茎中表达量最高,其次是绿果期(图5)。推测 FvARF5基因在草莓发育不同时期及不同组织中有不同的调控功能。
2.4 FvARF5基因对外源激素应答分析
利用荧光定量PCR分析FvARF5在IAA、GA和ABA诱导下的表达模式。与对照组相比,FvARF5在IAA处理1 h后,表达量迅速提高并达到顶峰,而后恢复正常水平(图6-A);GA处理草莓植株1~6 h后,FvARF5的表达水平明显增强,而后其表达量恢复对照水平(图6-B);FvARF5基因对ABA处理应答较弱(图6-C)。
不同小写字母表示在0.05水平差异显著。图6同。
Different small letters show significantly different at 0.05. The same as Fig.6.
图5 FvARF5基因在草莓不同组织中的表达情况
Fig.5 Relative expression of FvARF5 gene in different tissues of strawberry
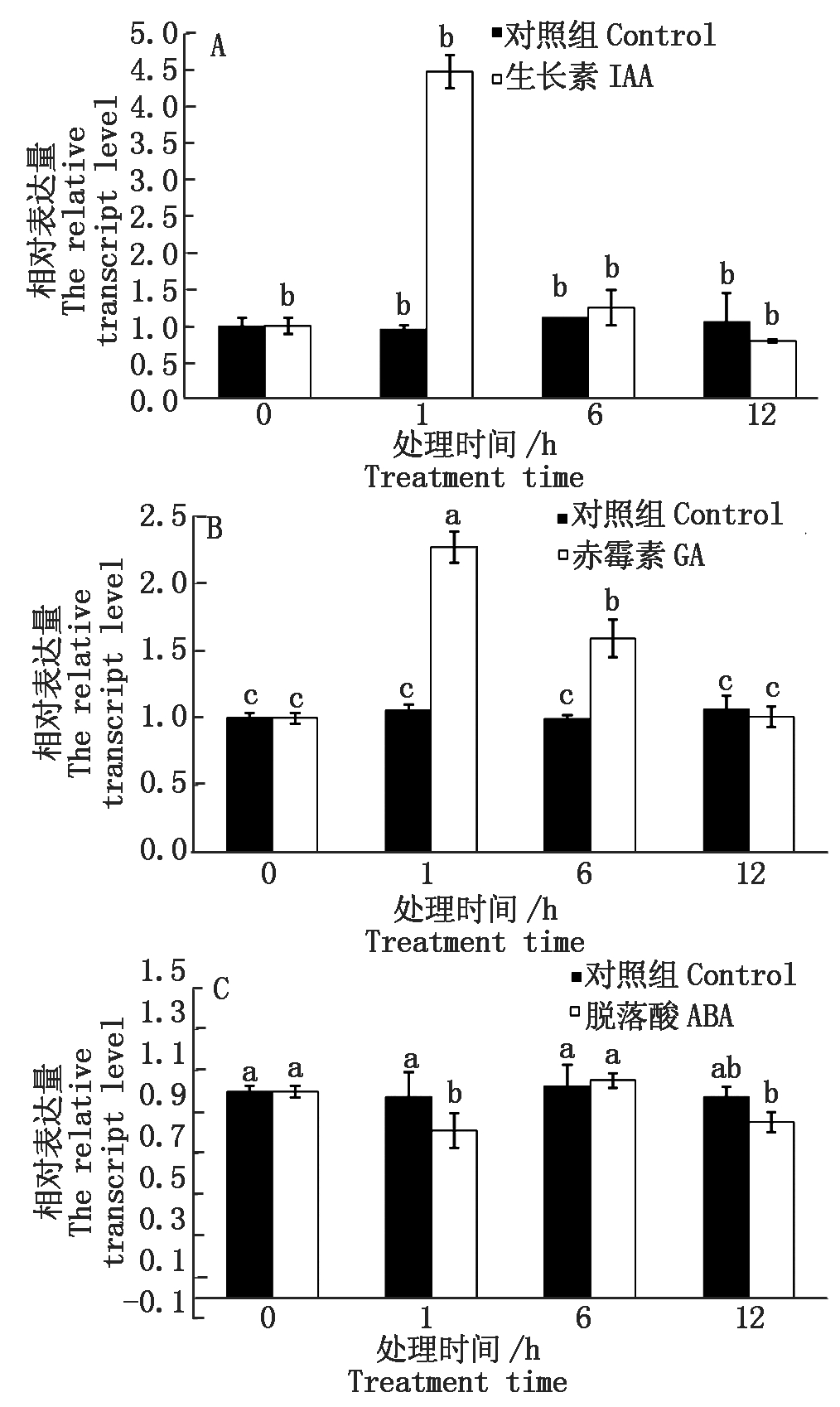
图6 不同植物激素处理后FvARF5相对表达量分析
Fig.6 Relative expression of FvARF5 gene under different phytohormones treatment
3 结论与讨论
本研究对草莓FvARF5基因进行克隆,得到2 745 bp、编码914个氨基酸的基因序列。对FvARF5基因编码的氨基酸序列分析表明,草莓FvARF5与蔷薇科月季ARF5基因的进化关系最近,相似度达95%,且具有生长素响应因子典型的B3 DNA结合结构域和生长素响应因子保守结构域。对FvARF5基因编码的蛋白理化性质分析表明,该蛋白为不稳定亲水性蛋白,分子质量为100.65 ku,理论等电点为5.25。亚细胞定位预测FvARF5蛋白定位于细胞核,启动子序列分析发现FvARF5具有生长素、赤霉素、脱落酸、茉莉酸甲酯、水杨酸等多种激素及胁迫响应功能元件。
ARF家族基因为多基因家族,各成员之间的功能不同[23-26]。因此,研究FvARF5在不同组织中的表达情况对于其功能的挖掘有重要意义。组织特异性表达分析显示,FvARF5在草莓不同组织中均有表达但表达量存在显著差异,其在茎和绿果中表达较高,预示着FvARF5基因可能对草莓茎和果实的发育有重要调控作用。如前所述,在拟南芥和番茄中的研究表明,ARF5(MP)基因对于新生茎芽形成、果实的发育及坐果具有重要的调控作用,而本研究中,草莓FvARF5基因对于植物激素生长素及赤霉素的诱导有显著性应答,说明FvARF5基因可能通过参与生长素和赤霉素的信号转导过程参与草莓的生长发育调控过程,这与前人研究中番茄SlARF5基因通过介导生长素和赤霉素信号转导调控番茄的果实坐果及果实发育过程结果具有一致性[21]。番茄属于呼吸跃变型果实,而草莓属于呼吸非跃变型果实,因此,关于FvARF5在草莓生长发育过程中的功能还有待于进一步分析验证。
ARF家族基因对于果实的生长发育有重要的调控过程,本研究为进一步揭示FvARF5的功能及其网络调控机制奠定基础。下一步的工作中,将通过转基因直接验证草莓FvARF5基因调控草莓果实发育的相关功能,为将来在分子水平上解析生长素调控草莓果实发育及其与其他植物激素互作调控果实发育的网络机制提供理论基础。
[1] 程然,生吉萍. 草莓果实成熟衰老影响因子及其调控机制研究进展[J].食品科学,2015,36(9):242-247. doi:10.7506/spkx1002-6630-201509045.
Cheng R,Sheng J P. Recent progress in research on influencing factors and regulation mechanisms of strawberry fruit ripening and senescence[J]. Food Science,2015,36(9):234-234.
[2] 张运涛,王桂霞,董静,王萍. 草莓畸形果形成的原因分析[J]. 落叶果树,2004,36(6):10-13. doi:10.3969/j.issn.1002-2910.2004.06.004.
Zhang Y T,Wang G X,Dong J,Wang P. Analysis of the reason of the formation of malformed fruits in strawberry[J]. Deciduous Fruits,2004,36(6):10-13.
[3] Estrada-johnson E,Csukasi F,Pizarro C M,Vallarino J G,Kiryakova Y,Vioque A,Brumos J,Medina-Escobar N,Botella1 M A,Alonso J M,Fernie A R,S nchez-Sevilla J F,Osorio S,Valpuesta V. Transcriptomic analysis in strawberry fruits reveals active auxin biosynthesis and signaling in the ripe receptacle[J]. Frontiers in Plant Science,2017,8:889. doi:10.3389/fpls.2017.00889.
nchez-Sevilla J F,Osorio S,Valpuesta V. Transcriptomic analysis in strawberry fruits reveals active auxin biosynthesis and signaling in the ripe receptacle[J]. Frontiers in Plant Science,2017,8:889. doi:10.3389/fpls.2017.00889.
[4] Guilfoyle T J,Hagen G. Auxin response factors[J]. Plant Growth Regulation,2007,10(5):453. doi:10.1016/j.pbi.2007.08.014.
[5] Li S B,Xie Z Z,Hu C G,Zhang J Z. A review of auxin response factors(ARFs)in plants[J]. Frontiers in Plant Science,2016,7(742):47. doi:10.3389/fpls.2016.00047.
[6] Okushima Y,Overvoorde P J,Arima K,Alonso J M,Chan A,Chang C,Ecker J R,Hughes B,Lui A,Nguyen D,Onodera C,Quach H,Smith A,Yu G,Theologis A. Functional genomic analysis of the AUXIN RESPONSE FACTOR gene family members in arabidopsis thaliana:unique and overlapping functions of ARF 7 and ARF 19[J]. Plant Cell,2005,17(2):444-463. doi:10.1105/tpc.104.028316.
[7] Li S B,Ouyang W Z,Hou X J,Xie L L,Hu C G,Zhang J Z. Genome-wide identification,isolation and expression analysis of auxin response factor(ARF)gene family in sweet orange(Citrus sinensis)[J]. Frontiers in Plant Science,2015,6:119. doi:10.3389/fpls.2015.00119.
[8] Wan S,Li W,Zhu Y,Yang S H,Song Y J,Wang J H. Genome-wide identification,characterization and expression analysis of the auxin response factor gene family in Vitis vinifera[J]. Plant Cell Reports,2014,33(8):1365-1375. doi:10.1007/s00299-014-1622-7.
[9] Wei H,Jiao Z,Hou X W,Yan Y,Wei Y X,Liu J H,Li M Y,Xu B Y,Jin Z Q. The auxin response factor gene family in banana:genome-wide identification and expression analyses during development,ripening,and abiotic stress[J]. Frontiers in Plant Science,2015,6(742):742. doi:10.3389/fpls.2015.00742.
[10] Zouine M,Fu Y,Chateigner-Boutin A L,Mila I,Frasse P,Wang H,Audran C,Roustan J P,Bouzayen M. Characterization of the tomato ARF gene family uncovers a multi-levels post-transcriptional regulation including alternative splicing[J]. PLoS One,2014,9(1):e84203. doi:10.1371/journal.pone.0084203.
[11] Ellis C M,Nagpal P,Young J C,Hagen G,Guilfoyle T J,Reed J W. AUXIN RESPONSE FACTOR 1 and AUXIN RESPONSE FACTOR 2 regulate senescence and floral organ abscission in Arabidopsis thaliana[J]. Development,2005,132(20):4563-4574. doi:10.1242/dev.02012.
[12] Goetz M,Vivian-Smith A,Johnson S D,Koltunow A M. AUXIN RESPONSE FACTOR 8 is a negative regulator of fruit initiation in Arabidopsis[J]. Plant Cell,2006,18(8):1873-1886. doi:10.1105/tpc.105.037192.
[13] Wilmoth J C,Wang S,Tiwari S B,Joshi A D,Hagen G,Guilfoyle T J,Alonso J M,Ecker J R,Reed J W. NPH4/ARF7 and ARF19 promote leaf expansion and auxin-induced lateral root for mation[J]. Plant Journal,2005,43(1):118-130. doi:10.1111/j.1365-313X.2005.02432.x.
[14] Okushima Y,Fukaki H,Onoda M,Theologis A,Tasaka M. ARF7 and ARF19 regulate lateral root formation via direct activation of LBD/ASL genes in Arabidopsis[J]. Plant Cell,2007,19(1):118-130. doi:10.1105/tpc.106.047761.
[15] Sagar M,Chervin C,Mila I,Hao Y W,Roustan J P,Benichou M,Gibon Y,Biais B,Maury P,Latche A,Pech J C,Bouzayen M,Zouine M. Sl-ARF4,an auxin response factor involved in the control of sugar metabolism during tomato fruit development[J]. Plant Physiology,2013,161(3):1362-1374. doi:10.1104/pp.113.213843.
[16] Aida M,Vernoux T,Furutani M,Traas J,Tasaka M. Roles of PIN-FORMED1 and MONOPTEROS in pattern formation of the apical region of the Arabidopsis embryo[J]. Development,2002,129(17):3965. doi:10.1101/gad.242202.
[17] Wenzel C L,Schuetz M,Yu Q,Mattsson J. Dynamics of MONOPTEROS and PIN‐FORMED1 expression during leaf vein pattern formation in Arabidopsis thaliana[J]. Plant Journal,2010,49(3):387-398. doi:10.1111/j.1365-313X.2006.02977.x.
[18] 李青青,南文斌,张汉马. ARF5/MONOPTEROS(MP)调控作用研究进展[J]. 西北植物学报,2016,36(1):197-203. doi:10.7606/j.issn.1000-4025.2016.01.0197.
Li Q Q,Nan W B,Zhang H M. Progress on the functions of ARF5/MONOPTEROS(MP)[J]. Acta Botanica Boreali-Occidentalia Sinica,2016,36(1):197-203.
[19] Ckurshumova W,Smirnova T,Marcos D,Zayed Y,Berleth T. Irrepressible MONOPTEROS/ARF5 promotes de novo shoot formation[J]. New Phytologist,2015,204(3):556-566. doi:10.1111/nph.13014.
[20] Krogan N T,Berleth T. A dominant mutation reveals asymmetry in MP/ARF5 function along the adaxial-abaxial axis of shoot lateral organs[J]. Plant Signaling & Behavior,2012,7(8):940-943. doi:10.1111/nph.13014.
[21] Liu S,Zhang Y,Feng Q,Qin L,Pan C,Lamin-Samu A T,Lu G. ONSE FACTOR 5 regulates fruit set and development via the Tomato AUXIN RESP mediation of auxin and gibberellin signaling[J]. Scientific Reports,2018,8(1):2971. doi:10.1038/s41598-018-21315-y.
[22] 苏丽艳,田爱梅,陶贵荣,李蒙. 苹果MdCaM的克隆及其对果实釆后非生物胁迫的响应[J]. 华北农学报,2017,32(1):47-52.doi:10.7668/hbnxb.2017.01.008.
Su L Y,Tian A M,Tao G R,Li M. Cloning of MdCaM and its expression responses under abiotic stresses during Malus domestica postharvest storage[J]. Acta Agriculturae Boreali-Sinica,2017,32(1):47-52.
[23] Kumar R,Tyagi A K,Sharma A K. Genome-wide analysis of auxin response factor(ARF)gene family from tomato and analysis of their role in flower and fruit development[J]. Molecular Genetics & Genomics,2011,285(3):245-260. doi:10.1007/s00438-011-0602-7.
[24] Shen C,Yue R,Sun T,Zhang L,Xu L Q,Tie S G,Wang H Z,Yang Y J. Genome-wide identification and expression analysis of auxin response factor gene family in Medicago truncatula[J]. Frontiers in Plant Science,2015,6(11):73. doi:10.3389/fpls.2015.00073.
[25] De J M,Woltersarts M,Garcíamartínez J L,Mariani C,Vriezen W H. The Solanum lycopersicum AUXIN RESPONSE FACTOR 7(SlARF7)mediates cross-talk between auxin and gibberellin signalling during tomato fruit set and development[J]. Journal of Experimental Botany,2011,62(2):617. doi:10.1093/jxb/erq293.
[26] Liu N,Wu S,Van J H,Wang Y,Ding B,Fei Z,Clarke T H,Reed J W,van der Knaap E. Down-regulation of AUXIN RESPONSE FACTORS 6 and 8 by microRNA 167 leads to floral development defects and female sterility in tomato[J]. Journal of Experimental Botany,2014,65(9):2507-2520. doi:10.1093/jxb/eru141.