番茄褪绿病毒(Tomato chlorosis virus, ToCV)自20世纪90年代在美国发现以来,已在世界多地相继发生,并造成了严重的损失[1-6],天津地区的番茄于2014年开始受到危害[7]。ToCV属于长线形病毒科(Closteroviridae)毛形病毒属(Crinivirus),为正单链RNA病毒,感染该病毒的番茄植株,下部叶片首先发病,发病初期叶脉间变黄,叶脉颜色加深,叶片增厚,植株死亡,极大降低了番茄品质与产量。
番茄黄化曲叶病毒(Tomato yellow leaf curl virus, TYLCV)是早于ToCV在天津番茄产区发病的病毒,其可直接导致番茄减产,并严重影响番茄品质[8]。该病毒于1964年首次在爱尔兰岛发现,此后,在世界上众多的国家和地区均有发现[9-10]。在我国绝大多数省市均有发生的报道[11-13],且侵染寄主的范围扩大到茄子[14]、黄瓜[15]、木瓜[16]和早开堇菜[17],引起的主要症状为叶片黄脉、曲叶、曲茎、叶片皱缩、植株矮化等。
ToCV和TYLCV的主要传播介体均为烟粉虱,均以番茄为主要寄主,且二者在番茄上引起的病症都会表现出黄化,故田间ToCV会被TYLCV的症状所掩盖,从而被误诊。近年来,天津地区这2种病毒复合侵染已成为一种新的威胁番茄产业的病毒病害。
国外很多研究表明,ToCV可以与其他植物病毒发生复合侵染,如番茄灼烧病毒(Tomato torrado virus,TOTV)、番茄斑萎病毒(Tomato spotted wilt virus,YSWV)、凤果花叶病毒(Pepinomasaic virus,PMV)等[18-19]。在古巴也发现了TYLCV与ToCV复合侵染的情况[20]。在我国,山东最先报道了ToCV可与TYLCV发生复合侵染[21],随后在南京和云南采集的番茄病样检测结果也证实这2种病毒可以复合侵染番茄[22-23]。自2015年天津市农业生物技术研究中心对天津地区番茄病毒病进行调查,发现也存在ToCV和TYLCV复合侵染的现象且呈逐年上升的趋势。
目前,关于ToCV和TYLCV复合侵染的报道仅限于2种病毒在植物和介体中检测的研究,而有关二者复合侵染后病毒基因变异情况至今尚未见报道。本研究拟从复合侵染的样品中克隆ToCV的CP基因(Coat protein,CP)、HSP70基因(Heat shock protein 70,HSP70)及TYLCV全基因,进行序列分析并与单独感染的样品序列进行比对分析,同时对复合侵染和单独侵染植株体内的病毒积累量进行荧光定量PCR检测,为深入研究复合侵染样品中ToCV与TYLCV的互作及其感染机制奠定基础,也为制定正确的防病抗病策略提供参考。
1 材料和方法
1.1 试验材料
RNA提取试剂盒、酶、克隆载体及菌株、转录试剂盒PrimerScriptTM RT Master Mix和克隆载体购自大连宝生物公司(TaKaRa);Axygen DNA凝胶回收试剂盒购自北京博迈德生物技术有限公司。
样品:2017年秋季自天津市西青区第六阜温室大棚内采集番茄褪绿病毒和番茄黄化曲叶病毒复合侵染的番茄叶片,编号为I-1、I-2、I-3、I-4、I-5;番茄褪绿病毒单独侵染的番茄叶片,编号为A2、Q5;番茄黄化曲叶病毒单独侵染的番茄叶片,编号为X1、X2。所有供试叶片-70 ℃保存待用。
1.2 试验方法
1.2.1 核酸提取 取发病植株的叶片,CTAB法提取总DNA;采用大连宝生物公司(TaKaRa)RNA提取试剂盒提取RNA;NanoDrop ND1000(NanoDrop Technologies, Rockland, DE)进行核酸浓度检测。
1.2.2 引物的设计与合成 根据本研究组[7-8]登录的天津地区番茄黄化曲叶病毒的基因组序列及番茄褪绿病毒的CP和HSP70基因序列设计特异性引物(表1)。
1.2.3 ToCV HSP和CP基因扩增及产物克隆 利用随机引物对样本的总RNA进行反转录,得到cDNA后用CP-F/R引物扩增ToCV的CP基因,用HSP-F/R和HSP2-F/R引物扩增ToCV的HSP基因。对获得的目的片段进行回收和T/A克隆。体系参照产品说明书。自动测序由宝生物工程(大连)有限公司完成,测序时选取2个阳性克隆,双向测序。
1.2.4 TYLCV全长基因组的PCR扩增与克隆 PCR总体积为25 μL,其中包括10×反应缓冲液(含Mg2+)2.5 μL,dNTP混合物(2.5 mmol/L)0.5 μL,上游引物(10 μmol/L)2.5 μL,下游引物(10 μmol/L)2.5 μL,模板 DNA 0.5~2.0 μL,DNA聚合酶(5 U/μL)0.5 μL,其余用水补足。反应程序:94 ℃变性2 min;94 ℃ 30 s,54 ℃ 30 s,72 ℃ 1.5 min,35个循环;72 ℃延伸10 min。将PCR 产物分离纯化后,克隆到T-easy载体(按试剂盒说明操作)。自动测序由宝生物工程(大连)有限公司完成,测序时选取2个阳性克隆,双向测序。
表1 本研究中用到的PCR引物
Tab.1 The primers used in this study

病毒Viruses引物名称Names序列Sequences产物大小/bpSizeTYLCVShanghai F5′-GAGGGATGAGTACAGGCCATATA-3′575Shanghai R5′-GCCCATGTATAGGAAGCCCAG-3′TJ-F5′-TTGCATACACTGGATTAGAGGC-3′2 248TJ-R5′-GAGAGAGAACAATTGGGATATG-3′Forward Primer TYI F5′-GTCCAGTCTTATGAGCAA-3′76Reverse Primer TYI R5′-GATCCACGAGTAACATCA-3′ToCV CP-F5′-GAATCTTTTAGAAGCTTTGGTTTAAGG-3′840CP-R 5′-GATCCTCTTGATCCTCATAGATTTC-3′HSP-F5′-GCGGTCCAGTTAACTCAG-3′1 021HSP-R5′-CCACCGACAAGAGGCAGCG-3′HSP2-F5′-CAAGACAGTTAGTTCTCAGTACTAAAC-3′911HSP2-R5′-CATACAATATAATAAGTGATGATGGGA-3′Forward Primer ToCVI F5′-GTTGCAAACACTGGTGATA-3′121Reverse Primer ToCVI R5′-GCCGGAATAAGAGATGT-3′
1.2.5 序列分析 利用Blast(www.ncbi.nlm.nih.gov/BLAST/)进行序列比较,DNAStar进行序列拼接,DNAMAN Version 5.2.9 (Lynnon Biosoft,Quebe Canada) 进行序列比较。
1.2.6 荧光实时定量PCR检测 TYLCV和ToCV 2种病毒均采用染料法进行定量。反应体系为20 μL,其中SYBR Premix Ex TaqII(2×)10 μL,Forward Primer(10 μmol/L)1 μL,Reverse Primer(10 μmol/L)1 μL,ROX Reference Dye(50×)0.4 μL,模板2 μL,ddH2O 5.6 μL。设置荧光实时定量PCR仪反应参数为95 ℃,5 min ;95 ℃ 30 s,54 ℃ 30 s,72 ℃ 1.5 min,40个循环。
标准曲线制备:以TYLCV的DNA和ToCV反转录的cDNA为模板,分别以实时定量PCR引物TYIF/R扩增长约76 bp的TYLCV目的检测片段;以实时定量PCR引物ToCVIF/R扩增长约121 bp的ToCV目的检测片段。利用高保真酶PrimerSTARHS进行PCR扩增,然后回收扩增片段,进行克隆,得到质粒TYLCV-CP和ToCV-CP,使用Nano drop测定质粒的浓度,使用下列公式计算拷贝数,-20 ℃保存备用。
每微升质粒拷贝数=6.02×1023×质粒浓度×10-9/(DNA(碱基数)×660)
绝对定量检测:用鲑鱼精稀释质粒使质粒拷贝数分别为10-4,10-5,10-6,10-7,10-8,建立标准曲线。将样品编号,进行实时定量PCR检测,每个样品设置3个重复,2次平行试验,计算荧光信号基线的循环数平均值(Cycle threshold,Ct值),然后根据所测样品的Ct平均值计算样品中病毒的含量。
2 结果与分析
2.1 TYLCV DNA-A基因的检测
采集具有典型症状的TYLCV单独侵染和TYLCV、ToCV复合侵染的叶片,利用Shanghai F/R引物进行PCR扩增,获得1条预期大小约575 bp的特异片段(图1)。在对该特异片段进行克隆和序列测定的基础上,又设计出2条特异引物TJ-R和TJ-F。利用这2条引物进行PCR扩增。扩增出1条与预期大小一致的2 248 bp的特异片段,2个特异片段序列进行拼接后获得大小为2 781 bp的DNA-A完整序列。
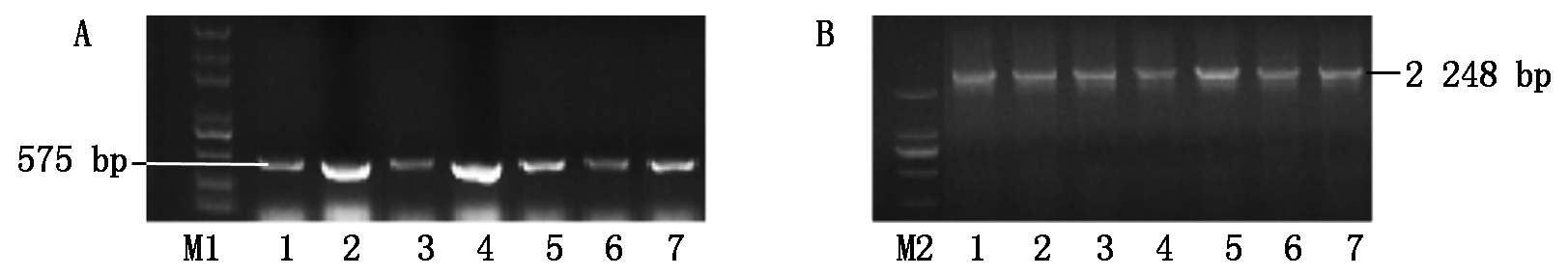
A.Shanghai F/R引物的扩增;B.特异引物TJ-R/F的扩增。1-7.I-1、I-2、I-3、I-4、I-5、X1、X2;M1.DL5000;M2.DL2000。
A.The PCR result by primers Shanghai F/R;B.The PCR result by primers TJ-R/F.1-7.I-1,I-2,I-3,I-4,I-5,X1,X2;M1.DL5000;M2.DL2000.
图1 检测TYLCV PCR电泳结果
Fig.1 The results of TYLCV gene amplified with PCR
2.2 ToCV编码CP和HSP70基因的检测
采集具有典型症状的ToCV单独侵染和TYLCV、ToCV复合侵染的叶片,进行RNA提取和RT-PCR检测。利用CP-F/R扩增出了840 bp大小的条带(图2)。
利用HSP-F/R和HSP2-F/R 2对引物进行PCR扩增,分别获得了大小约1 021,911 bp的特异片段(图3)。2个特异片段序列进行拼接后获得大小为1 665 bp的HSP70基因的完整序列。
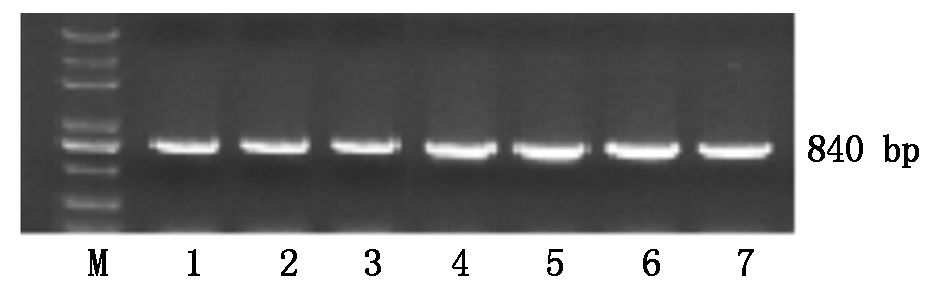
1-7.I-1、I-2、I-3、I-4、I-5、A2、Q5; M.DL5000。
图2 CP-F/R引物PCR扩增
Fig.2 The PCR result by primers CP-F/R
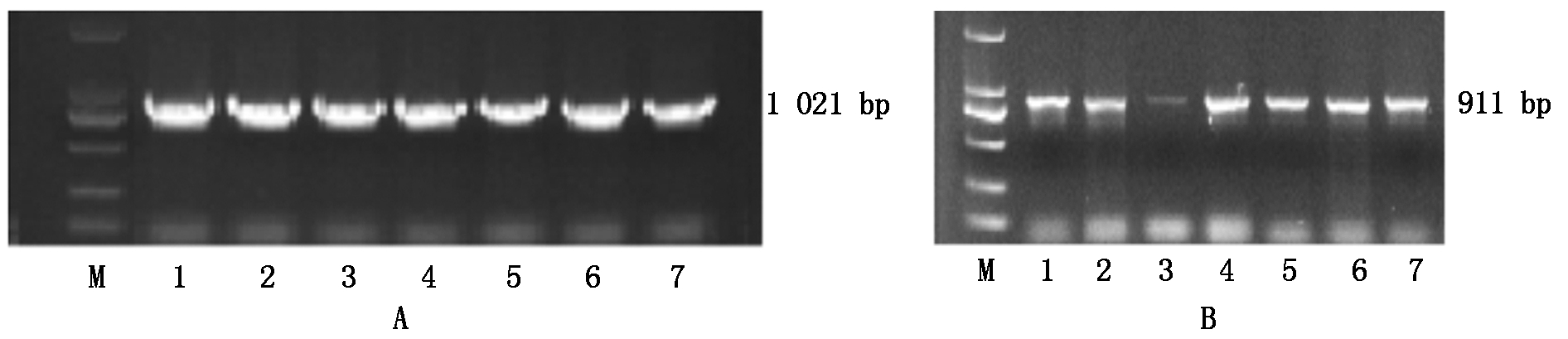
A.HSP-F/R扩增产物;B. HSP2-F/R扩增产物。1-7.I-1、I-2、I-3、I-4、I-5、A2、Q5;M. DL2000。
A.The PCR result by primers HSP-F/R;B.The PCR result by primers HSP2-F/R.1-7.I-1,I-2,I-3,I-4,I-5,A2,Q5;M.DL2000.
图3 引物HSP-F/R和HSP2-F/R的扩增产物
Fig.3 The PCR products using primers HSP-F/R and HSP2-F/R
2.3 单独侵染和复合侵染的病毒基因变异情况
2.3.1 TYLCV基因组的变异 利用表1中所列引物扩增TYLCV的全基因组序列。将获得的基因片段进行拼接,得到这个病毒的全基因组信息。在所测的复合侵染和单独侵染的样品中,TYLCV全长均为2 781 bp。经NCBI Blast比对,与GenBank中已登录的TYLCV相似性均在99%以上,确定其为TYLCV侵染。利用DNAMAN基因分析软件将复合侵染的样品和单独侵染的样品进行序列对比。结果表明(图4),在全长2 781个碱基序列中,与单独侵染样品相比,复合侵染的样品共有6处碱基发生了变异,分别为73位A→C、92位A→G、347位C→T、1 209位C→T、1 618位A→G、2 107位A→T。除73位和92位的变异未在编码区,其余的变异碱基均在ORF(Open Reading Frame)框内。位于AC3蛋白编码区的1 209位为同义突变,其他均发生了错义突变。347位在AV1的编码区,氨基酸由A(丙氨酸)突变为V(缬氨酸);1 618位在AC2和AC1蛋白编码区,氨基酸分别由L(亮氨酸)突变为P(脯氨酸)和I(异亮氨酸)突变为T(苏氨酸);2 107位在AC1蛋白编码区,氨基酸由L(亮氨酸)变为F(苯丙氨酸)。
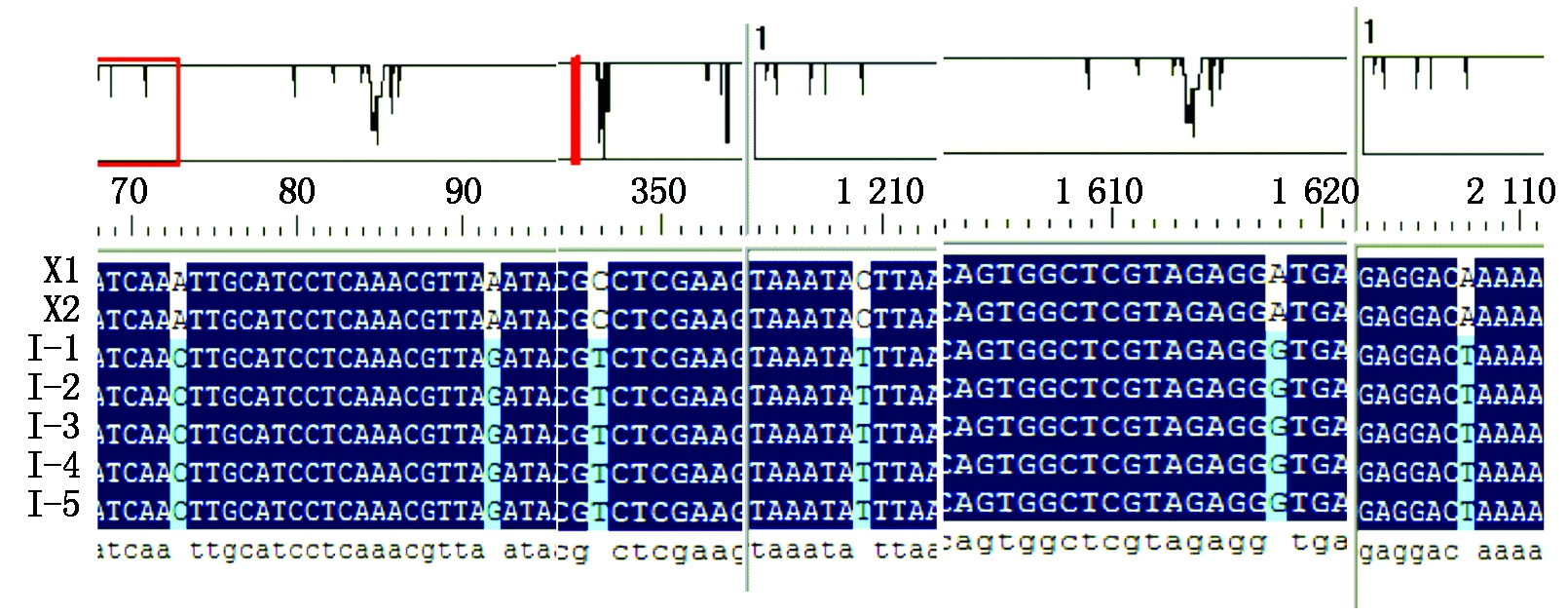
X1、X2为单独侵染样品;I-1、I-2、I-3、I-4、I-5为复合侵染样品。
X1, X2 are single infected samples;I-1,I-2,I-3,I-4,I-5 are mixed infected samples.
图4 TYLCV基因序列在单独侵染和复合侵染样品中的变异
Fig.4 Variation of TYLCV gene in single and mixed infected samples
2.3.2 ToCV的CP和HSP70基因的变异 利用表1中所列引物扩增ToCV的CP基因和HSP70基因并进行测序。ToCV的CP基因长度为775 bp,HSP70基因的长度为1 665 bp。经NCBI Blast比对,与GenBank中的ToCV病毒的CP和HSP70基因的相似性均在99%以上,确定其为ToCV。同样利用DNAMAN基因分析软件将复合侵染的样品和单独侵染的样品进行序列对比。结果表明,ToCV病毒的CP和HSP70序列均有变异,其中,在编码CP蛋白的基因中第15位的C突变为T,利用DNAMAN进行蛋白翻译,再进行序列比对表明此处为同义突变(图5);HSP70基因共发生了4处碱基变异(图6),其中826位由T→C和1 166位由G→A均为同义突变,而1 395位和1 630位均由A→G,相应的氨基酸由T(苏氨酸)突变为A(丙氨酸)和由N(天冬酰氨) 突变为S(丝氨酸)。

Q5、A2为单独侵染样品;I-1、I-2、I-3、I-4、I-5为复合侵染样品。图6同。
Q5,A2 are single infected samples;I-1,I-2,I-3,I-4,I-5 are mixed infected samples.The same as Fig.6.
图5 ToCV CP基因序列的变异
Fig.5 Variation of ToCV CP gene
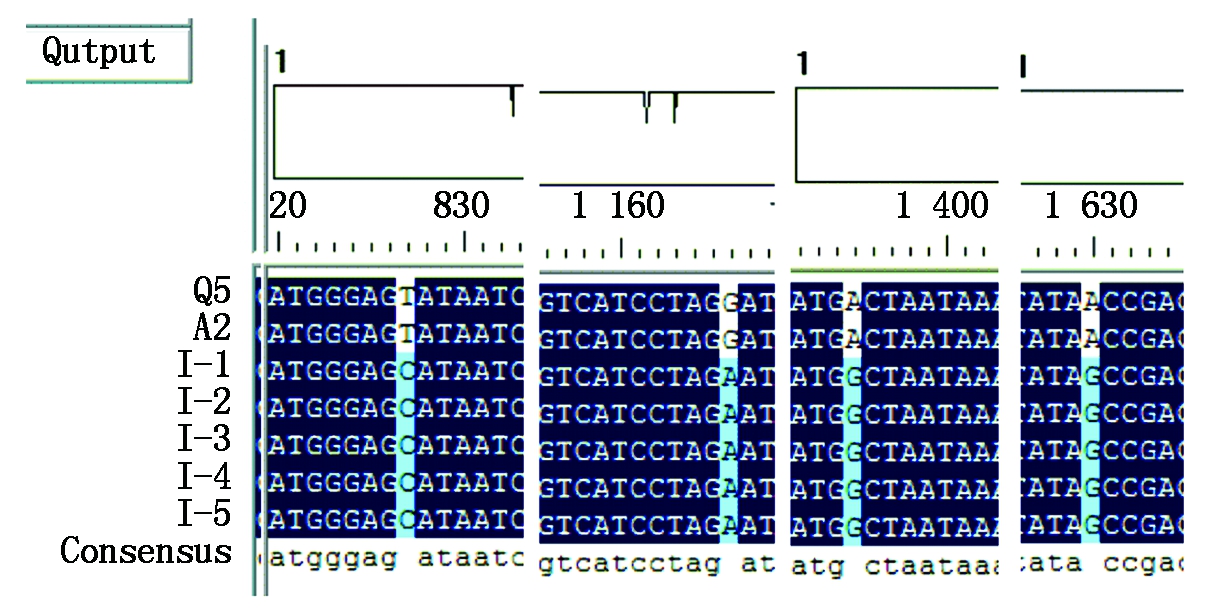
图6 ToCV HSP70基因序列的变异
Fig.6 Variation of ToCV HSP70 gene
2.4 单独侵染和复合侵染病毒的含量
2.4.1 复合感染样品中2种病毒的定量检测 利用含有ToCV CP和TYLCV CP基因片段的质粒建立标准曲线,以拷贝数为单位进行浓度梯度稀释,分别为9.4×102,9.4×103,9.4×104,9.4×105,9.4×106,9.4×107个拷贝,在扩增ToCV CP时,标准曲线的阈值为0.109,相关系数为0.998,扩增效率Eff=102.14%,Y=-3.46×lgX+40.58;在扩增TYLCV CP时,标准曲线的阈值为3.19,相关系数为0.998,扩增效率Eff=101.97%,Y=-3.27×lgX+34.486,其中X为模板拷贝数,Y为样品循环数。
2.4.2 单独侵染和复合侵染初始拷贝数计算 从图7中可以看出,实时定量PCR的扩增曲线呈S形,说明扩增效果良好。图8中的熔解曲线无杂峰,说明没有引物二聚体等非特异扩增,数据可靠。
对待测样品进行2次平行测定,分别得到复合侵染样品中2种病毒的Ct平均值,并计算得标准差及Ct值的变异系数(表2,3)。其中,Ct值的变异系数均小于5%,在误差允许范围内,说明建立的实时荧光定量PCR检测方法对样品检测结果均有很好的重复性。
通过在同一板上同时建立标准曲线,对待测样品进行定量,获得2种病毒在复合侵染番茄时的具体拷贝数(表2,3)。从表中可以看出,经方差分析,在田间单独侵染和复合侵染的样品中, TYLCV的拷贝数差异不显著,ToCV的拷贝数在单独侵染和复合侵染的样品中差异也不显著(P>0.05)。在复合侵染的样品中,TYLCV的拷贝数比ToCV提高了2个数量级,为262~436倍(表4)。
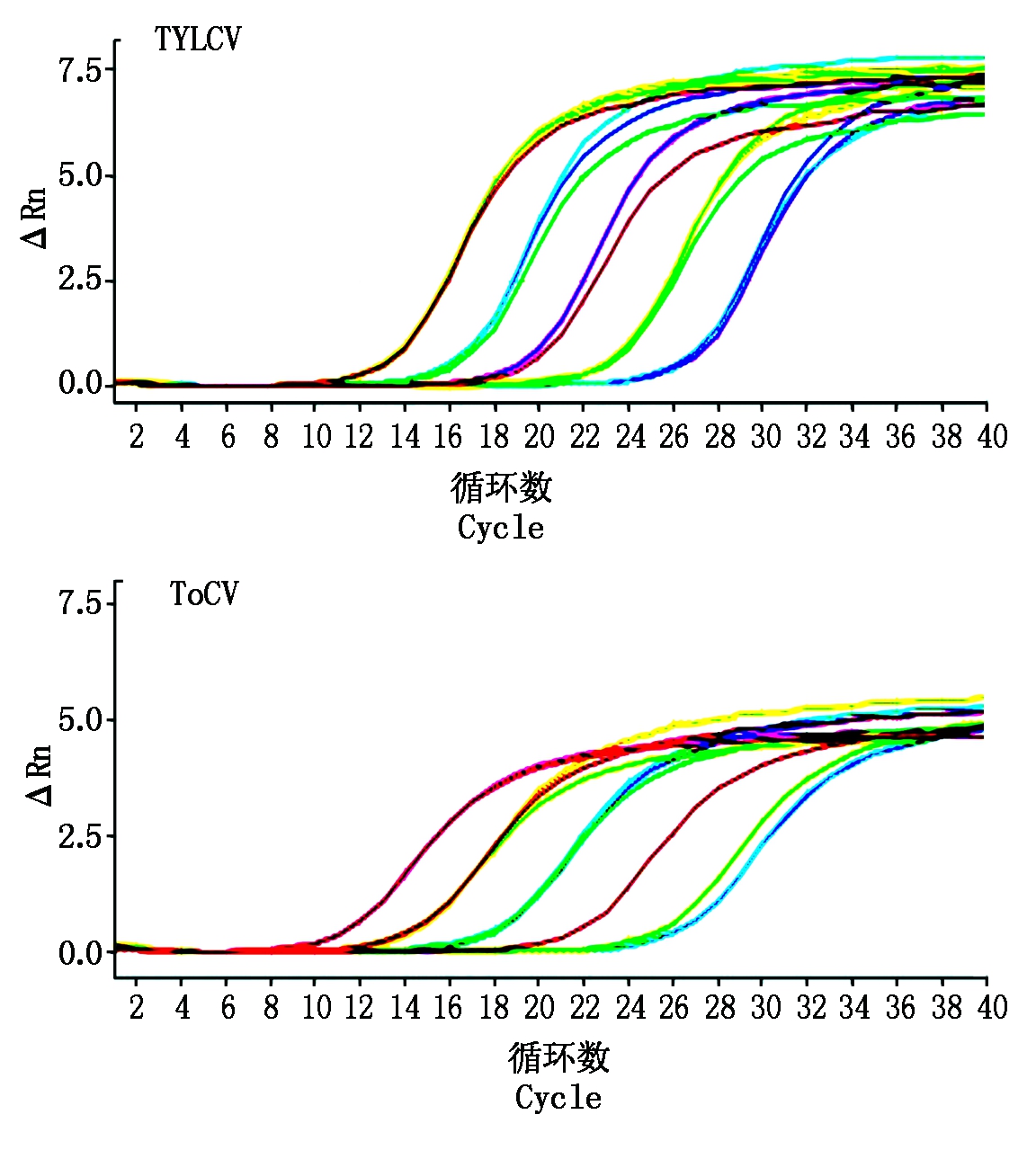
图7 实时定量PCR扩增曲线
Fig.7 Real-time quantitative PCR amplification curve
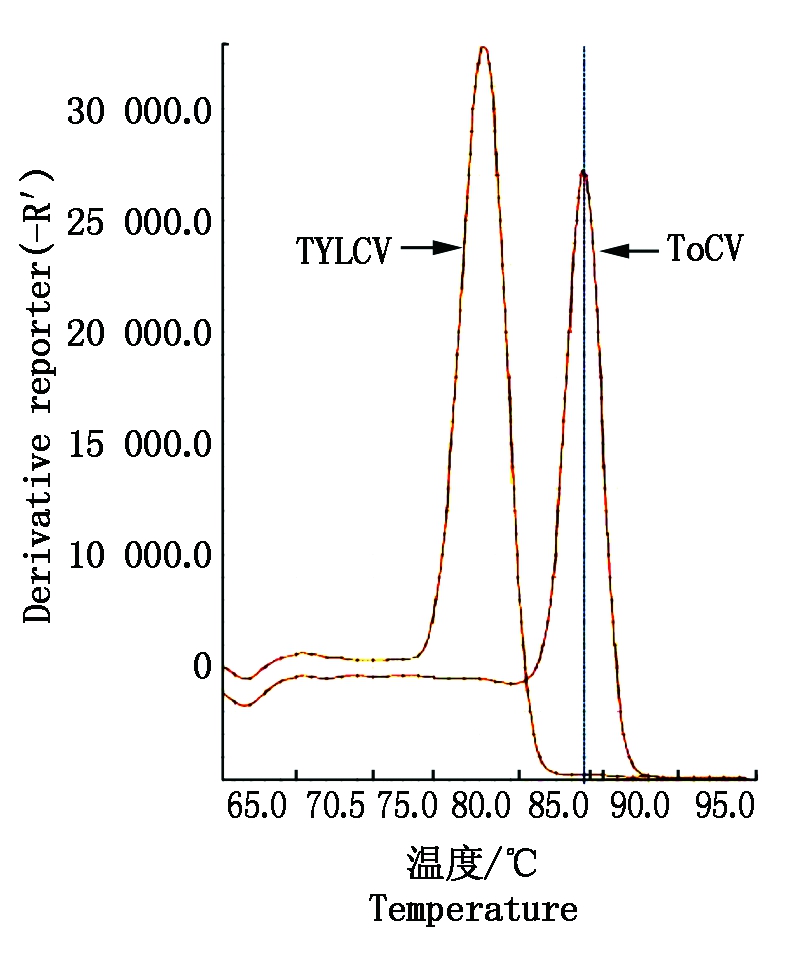
图8 实时定量PCR熔解曲线
Fig.8 The melting curve of Real-time quantitative PCR
3 结论与讨论
为了对TYLCV和ToCV复合侵染番茄的现象进行初步的研究,本研究将TYLCV的全基因组和ToCV的2个最为保守的CP基因和HSP70基因进行了序列分析。经一系列比对分析发现,与单独侵染样品相比,TYLCV和ToCV在复合侵染的样品中存在碱基变异,且除ToCV的CP外,其余基因大部分为错义突变。这很有可能是TYLCV和ToCV在共同侵染番茄后,2种病毒为了适应新的环境,协生共存,而从自身的碱基发生的改变。
表2 ToCV实时定量PCR重复试验结果
Tab.2 The reproducibility of ToCV Real-time quantitative PCR
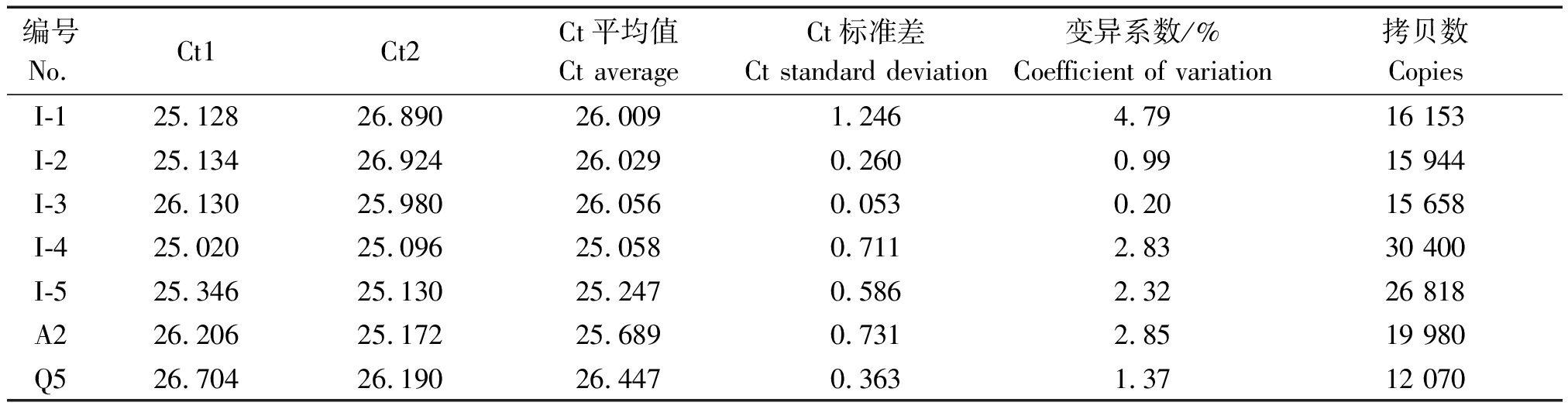
编号No.Ct1Ct2Ct平均值Ct averageCt标准差Ct standard deviation变异系数/%Coefficient of variation拷贝数CopiesI-125.12826.89026.0091.2464.7916 153I-225.13426.92426.0290.2600.9915 944I-326.13025.98026.0560.0530.2015 658I-425.02025.09625.0580.7112.8330 400I-525.34625.13025.2470.5862.3226 818A226.20625.17225.6890.7312.8519 980Q526.70426.19026.4470.3631.3712 070
表3 TYLCV实时定量PCR重复试验结果
Tab.3 The reproducibility of TYLCV Real-time quantitative PCR
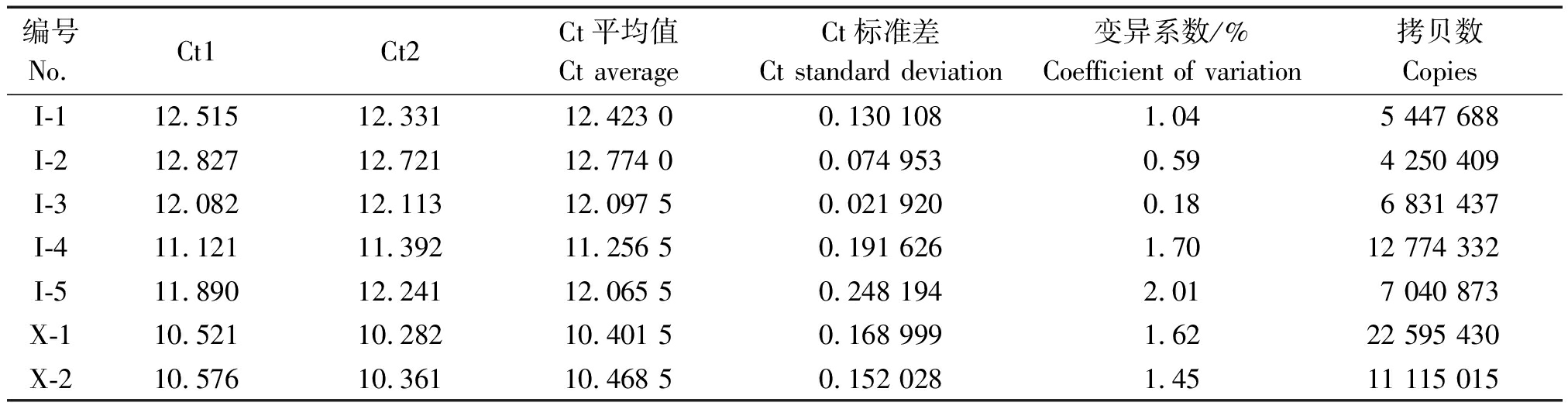
编号No.Ct1Ct2Ct平均值Ct averageCt标准差Ct standard deviation变异系数/%Coefficient of variation拷贝数CopiesI-112.51512.33112.423 00.130 1081.045 447 688I-212.82712.72112.774 00.074 9530.594 250 409I-312.08212.11312.097 50.021 9200.186 831 437I-411.12111.39211.256 50.191 6261.7012 774 332I-511.89012.24112.065 50.248 1942.017 040 873X-110.52110.28210.401 50.168 9991.6222 595 430X-210.57610.36110.468 50.152 0281.4511 115 015
表4 复合侵染样品Ct值及拷贝数
Tab.4 The sample Ct value and copy number

项目ItemsI-1I-2I-3I-4I-5TYLCV拷贝数 TYLCV copies5 447 6884 250 4096 831 43712 774 3327 040 873ToCV拷贝数 ToCV copies16 15315 94415 65830 40026 818TYLCV/ToCV337266436420262
在复合侵染的样品中,TYLCV的AV1基因(347位)、AC1基因(1 618,2 107位)、AC2基因(1 618位)均发生了错义突变。其中AV1基因编码外壳蛋白(CP),与病毒的包壳、介体传播及与寄主的互作相关[24];AC1基因编码复制相关蛋白(Replication-associated protein,Rep),参与病毒基因组DNA的复制起始[25];AC2基因编码转录激活蛋白(Transcriptional activator protein,TrAP),该蛋白能激活病毒链上的基因转录[26]。这2个蛋白是病毒侵染、复制、转录的重要蛋白,也许是这些基因的突变增强了病毒适应环境的能力。另外,虽然73位和92位的变异未在编码区,但它对病毒的表达可能起着调控的作用,也可能与其他蛋白的变异协同作用改变了病毒的适应能力。
自然界中植物病毒的复合侵染现象普遍存在。目前已有一些关于TYLCV和ToCV复合侵染番茄的报道[21-23],但仅限于这2种病毒复合侵染在植物和介体中的检测和发现的研究。
很多研究显示,多种病毒的复合侵染往往具有协生作用,有助于其突破寄主的抗性,导致新的病害发生和流行[27-28]。玉米褪绿斑驳病毒(Maize chlorotic mottle virus,MCMV)与甘蔗花叶病毒(Sugarcane mosaic virus,SCMV)复合侵染时产生协生作用,引起更严重的致死性坏死症状[29]。ToCV和TSWV(Tomato spotted wilt virus,TSWV)同时侵染含有抗病基因Sw-5的番茄时,ToCV的侵染会导致寄主番茄对TSWV抗性丧失[30]。PVY(Potato virus Y,PVY)和烟草花叶病毒(Tobacco mosaic virus,TMV)共同侵染马铃薯野生种时表现协生作用[31]。TYLCV与莴苣褪绿病毒(Lettuce chlorosis virus,LCV)和木瓜环斑病毒(Papaya ringspot virus,PRSV)复合侵染木瓜后表现出更严重的症状[32]。在本研究中,TYLCV和ToCV复合侵染的症状表现出更严重的矮化和黄化,但病毒的浓度在复合侵染和单独侵染的样品中差异不明显。也许2种病毒在复合侵染后,为了适应新的环境只是达到了一个暂时的平衡。植物病毒的复合侵染比单独侵染更复杂,涉及病毒、寄主、环境的相互作用,因而可能受到温度、品种、病毒的侵染次序等多种因素的影响[33-34]。为了深入了解TYLCV和ToCV复合侵染后2种病毒的作用,在后续试验中将对这几方面进行研究。另外,在复合侵染的样品中,TYLCV要比ToCV在量上占有优势。在本研究中采集的样品均为叶片,在其他的组织是否也如此尚待研究。
[1] Pasev G,Radeva V,Kostova D. First report of Tomato infectious chlorosis virus on tomato in Bulgaria[J]. Journal of Phytopathology,2012,160(3):160-162.doi:10.1111/j.1439-0434.2011.01874.
[2] Salem N M,Mansour A N,Abdeen A O,Araj S,Khrfan W I. First report of tomato chlorosis virus infecting tomato crops in Jordan[J]. Plant Disease,2015,99(9):1503. doi:10.1094/PDIS-01-15-0130-PDN.
[3] 周涛,杨普云,赵汝娜,师迎春,原锴,范在丰.警惕番茄褪绿病毒在我国的传播和危害[J].植物保护,2014,40(5):196-199. doi:10.3969/j.issn.0529-1542.2014.05.038.
Zhou T,Yang P Y,Zhao R N,Shi Y C,Yuan K,Fan Z F.Alarming the transmission and damage of Tomato chlorosis virus in China[J].Plant Protection,2014,40(5):196-199.
[4] 刘永光,魏家鹏,乔宁,李美芹,刘晓明,竺晓平.番茄褪绿病毒在山东暴发及其防治措施[J].中国蔬菜,2014(5):67-69.doi:10.3969/j.issn.1000-6346.2014.05.018.
Liu Y G,Wei J P,Qiao N,Li M Q,Liu X M,Zhu X P. Outbreak of Tomato chlorosis virus in Shandong and its control measures[J].China Vegetables,2014(5):67-69.
[5] 汤亚飞,何自福,佘小漫,蓝国兵.侵染广东番茄的番茄褪绿病毒分子鉴定[J].植物保护,2017,43(2):133-137.doi:10.3969/j.issn.0529-1542.2017.02.022.
Tang Y F,He Z F,She X M,Lan G B. Molecular identification of Tomato chlorosis virus infecting tomato in Guangdong province[J]. Plant Protection,2017,43(2):133-137.
[6] 王翠琳,冯佳,孙晓辉,王少立,赵静,刘金亮,竺晓平.北方四省区番茄褪绿病毒的分子鉴定[J].植物保护,2017,43(2):141-145.doi:10.3969/j.issn.0529-1542.2017.02.024.
Wang C L,Feng J,Sun X H,Wang S L,Zhao J,Liu J L,Zhu X P. Molecular identification of Tomato chlorosis virus from four provinces or autonomous region in northern China[J].Plant Protection,2017,43(2):141-145.
[7] 金凤媚,薛俊,宋建.天津地区番茄褪绿病毒的分子检测与基因组部分序列分析[J].华北农学报,2016,31(2):23-27. doi:10.7668/hbnxb.2016.02.005.
Jin F M,Xue J,Song J. Molecular detection and partial genome sequence analysis of tomato chlorosis virus in Tianjin[J]. Acta Agriculturae Boreali-Sinica,2016,31(2):23-27.
[8] 金凤媚,薛俊,郏艳红,周祥明,王建江,刘仲齐.天津地区番茄黄化曲叶病毒DNA-A的克隆和序列分析[J]. 华北农学报,2011,26(1):58-62.doi:10.7668/hbnxb.2011.01.013.
Jin F M,Xue J,Jia Y H,Zhou X M,Wang J J,Liu Z Q. Cloning and sequence analysis of TYLCV DNA-A in Tianjin[J].Acta Agriculturae Boreali-Sinica,2011,26(1):58-62.
[9] Chinnaraja C,Ramkissoon A,Ramsubhag A,Jayaraj J. First report of tomato yellow leaf curl virus Infecting tomatoes in Trinidad[J].Plant Disease,2016,100(9):1958.doi:10.1094/PDIS-04-16-0446-PDN.
[10] Verdin E,Desbiez C,Catherine W S,Gognalons P,Ahmed K P, Gronenborn B,Mirzayeva S,Sultanova N,Mammadov A,Huseynova I. First report of tomato yellow leaf curl virus infecting tomato in Azerbaijan[J]. Journal Plant Pathology,2018,100(2):335.doi:10.1007/s42161-018-0050-x.
[11] 张前荣,李大忠,朱海生,刘建汀,李永平,温庆放.番茄黄化曲叶病毒研究进展[J].分子植物育种,2017,15(9):3709-3716.
Zhang Q R,Li D Z,Zhu H S,Liu J T,Li Y P,Wen Q F.A review of tomato yellow leaf curl virus[J].Molecular Plant Breeding,2017,15(9):3709-3716.
[12] 宋晰,师迎春,张世晨,梁彦,王廿,陈善义,周涛.北京地区番茄黄化曲叶病病毒分离物测定及株系的初步鉴定[J].植物病理学报,2013,43(2):113-119.doi:10.3969/j.issn.0412-0914.2013.02.001.
Song X,Shi Y C,Zhang S C,Liang Y,Wang N,Chen S Y,Zhou T. Detection and preliminary strain identification of the pathogen induing tomato yellow leaf curl disease in Beijing[J].Acta Phytopathologica Sinica,2013,43(2):113-119.
[13] 曹骞,李晶,买热木古丽·克依木,王惠卿,李国志,马德英.新疆地区烟粉虱生物型的区域分布及其携带的番茄黄化曲叶病毒检测[J].昆虫学报,2013,56(6):652-664.
Cao Q,Li J,Mairemuguli K Y M,Wang H J,Li G Z,Ma D Y. Biotype distribution and infection status of tomato yellow leaf curl virus(TYLCV) in Bemisia tabaci(Hemiptera:Aleyrodidae) in Xinjiang, northwestern China[J]. Acta Entomalogica Sinica,2013,56(6):652-664.
[14] Rabie M,Ratti C,Aleem E A,Fattouh F. Detection and molecular characterization of tomato yellow leaf curl virus naturally infecting Lycopersicon esculentum in Egypt[J]. Actavirologica,2017,61(3):252-263. doi:10.4149/av_2017_303.
[15] Ebtisam A,Hanadi A,Ahmed B H,Aqeel H AI. First report of tomato yellow leaf curl virus infecting cucumber in Kuwait[J]. Plant Disease,2016,100(3):656.doi:10.1094/PDIS-07-15-0830-PDN.
[16] Alabi O J,Rwahnih A M.,Brown J K,Idris A M,Gregg L,Kmieciak E,Setamou M,Jifon J L. First report of papaya (Carica papaya) naturally infected with the introduced tomato yellow leaf curl virus-Israel[J]. Plant Disease,2016,100(9):1959.doi:10.1094/PDIS-04-16-0469-PDN.
[17] Zhou Y,Luo C,Zhao J,Wei S J,Chen Z,Yan J Y,Li X H. First report of tomato yellow leaf curl virus in viola prionantha in China[J]. Plant Disease,2016,100(1):231.doi:10.1094/PDIS-01-15-0104-PDN.
[18] Garcia-cano E,Resende R O,Fernandez-Munoz R,Moriones E. Synergistic interaction between Tomato chlorosis virus and Tomato spotted wilt virus results in breakdown of resistance in tomato[J].Phytopathology,2006,96(11):1263-1269.doi: 10.1094/phyto-96-1263.
[19] Alfaro-Fernández A,Medina V,Cordoba M C,Font M I,Jornet J,Cebrian M C,Jorda C. Ultrastructrual aspects of tomato leaves infected by Tomato torradovirus(ToTV)and co-infected by other viruses[J]. Plant Pathology,2010,59(2):231-239.doi:10.1111/j.1365-3059.2009.02215.
[20] Martínez-Zubiaur Y, FialloOlivé E,Tripp J C,Bustamante R R. First report of Tomato chlorosis virus infecting tomato in single and mixed infections with Tomato yellow leaf curl virus in Cuba[J]. Plant Disease,2008,92(5):836.doi:10.1094/PDIS-92-5-0836C.
[21] 赵黎明,李刚,刘永光,国家进,魏家鹏,竺晓平.山东设施番茄上番茄褪绿病毒与番茄黄化曲叶病毒复合侵染的分子鉴定[J].中国蔬菜,2014(12):15-20.doi:10.3969/j.issn.1000-6346.2014.12.006.
Zhao L M,Li G,Liu Y G,Guo J J,Wei J P,Zhu X P. Molecular Identification on Mixed Infections of Tomato chlorosis virus and Tomato yellow leaf curl virus[J].China Vegetables,2014(12):15-20.
[22] 吴淑华,李廷芳,赵文浩,程兆榜 ,郭青云, 赵统敏, 余文贵, 朱叶芹, 季英华.江苏省番茄黄化曲叶病毒和褪绿病毒复合侵染的分子检测[J].园艺学报,2016,43(1):89-99. doi:10.16420/j.issn.0513-353x.2015-0584.
Wu S H,Li T F,Zhao W H,Cheng Z B,Guo Q Y,Zhao T M,Yu W G,Zhu Y Q,Ji Y H. Molecular identification on mixed infection of tomato yellow leaf curl virus and tomato chlorosis virus on tomato in Jiangsu Province[J].Acta Horticulturae Sinica,2016,43(1):89-99.
[23] 刘微,史晓斌,唐鑫,张宇,张德咏,周序国,刘勇.云南番茄褪绿病毒和番茄黄化曲叶病毒复合侵染的分子鉴定[J].园艺学报,2018,45(3):552-560.doi:10.16420/j.issn.0513-353x.2017-0735.
Liu W,Shi X B,Tang X,Zhang Y,Zhang D Y,Zhou X G,Liu Y. Molecular Identification of Tomato chlorosis virus and Tomato yellow leaf curl virus in Yunnan Province[J].Acta Horticulturae Sinica,2018,45(3):552-560.
[24] Harrison B D,Swanson M M,Fargette D. Begomovirus coat protein: serology,variation and functions[J]. Physiological and Molecular Plant Pathology,2002,60:257-271.doi:10.1006/pmpp.2002.0404.
[25] Laufs J,Jupin I,David C,Schumacher S, Heyraud-Nitschke F, Gronenborn B. Geminivirus replication: Genetic and biochemical characterization of Rep protein function[J]. A Review Biochimie, 1995,77:765-773.doi:10.1177/14.12.884.
[26] Sunter G,Bisaro D M. Transactivation of Geminivirus AR1 and BR1 gene expression by the viral AL2 gene product occurs at the level of transcription[J]. Plant Cell,1992,4,1321-1331.doi:10.1105/tpc.4.10.1321.
[27] Harrison B D,Zhou X,Otim-Nape G W. Role of a novel type of double infection in Geminivirus-induced epidemic of severe cassava mosaic in Uganda[J]. Annals of Applied Biology,1997,131:437-448.doi:10.1111/j.1744-7348.1997.tb05171.
[28] Vance V B. Replication of potato virus×RNA is altered in coinfections with Potato virus Y[J].Virology,1991,182 (9):486-494.doi:10.1016/0042-6822(91)90589-4.
[29] Xia Z H,Zhao Z X, Chen ,Li M J,Zhou T,Deng C L,Zhou Q, Fan Z F. Synergistic infection of two viruses MCMV and SCMV increases the accumulations of both CMV and MCMV-derived siRNAs in maize[J]. Scientific Reports,2016,6:20520. doi:10.1038/srep20520.
[30] Garcia-cano E,Resende R O, Fernandez-Munoz R,Moriones E. Synergistic interacti on between Tomato chlorosis virus and Tomato spotted wilt virus results in breakdown of resistance in tomato[J].Phytopathology,2006,96(11):1263-1269.doi:10.1094/phyto-96-1263.
[31] Valkonen J. Accumulation of potato virus Y is enhanced in Solatiumbrevidens also infected with tobacco mosaic virus of potato spindle tuber viroid [J].Annals of Applied Biology,1992,121(2):321-32.doi:10.1111/j.1744-7348.1992.tb03445.
[32] Alabi O J,Rwahnih M A,Jifon J L,Sétamou M,Brown J K, Gregg L, Park J W.Mixed infection of Lettuce chlorosis virus, Papaya ringspot virus, and Tomato yellow leaf curl virus-IL detected in a Texas Papaya Orchard affected by a virus-Like disease outbreak[J]. Plant Disease,2017,101(7):1094-1102.
[33] 鲁瑞芳,李为民,彭学贤.植物病毒协生作用及其分子机理[J].中国病毒学,2001,16(3):195-201.
Lu R F,Li W M,Peng X X. Molecular mechanism of synergism of plant virus[J].Virological Sinica,2001,16(3):195-201.
[34] 郭兴启,冯炘,李向东,郭恒俊,李照会.PVY/PVX协生作用对病毒浓度及寄主细胞超微结构的影响[J].中国农业科学,2003,36(3):281-286.doi:10.3321/j.issn:0578-1752.2003.03.009.
Guo X Q,Feng X,Li X D,Guo H J,Li Z H.Effect of synergism between PVY and PVX on the viruses titer and the ultrastructure of infected cell of tobacco plant[J].Scientia Agricultura Sinica,2003,36(3):281-286.