Whirly基因家族的蛋白质成员在整个植物界中都存在,主要定位于线粒体和叶绿体中,具有和单链DNA结合的能力[1]。Desveaux等[2]在2000年从马铃薯中分离出的PBF-2转录因子是第一个Whirly基因家族成员,它和cDNA编码的蛋白质都以序列特异性的方式与诱导子响应元件 (Elicitor response element)单链形式结合。
Desveaux等[3]在2005年研究发现,拟南芥和马铃薯中的Whirly转录因子具有调节防御基因表达的作用,其可能在防御反应以外的过程中发挥作用,并可能在叶绿体和核中起作用。Whirly蛋白在细胞核内参与水杨酸(SA)依赖的抗病信号转导,Desveaux等[4]研究发现,拟南芥中StWhy1同源基因AtWHY1的DNA结合活性由SA诱导,并且是SA依赖的抗病性和SA诱导的系统获得性抗性(SAR)响应基因表达所需的。Whirly蛋白家族能够调节端粒结构稳定,拟南芥AtWHY1被鉴定为一个新的端粒末端结合蛋白,在调节拟南芥端粒的稳态中能够发挥作用,主要表现在:在AtWHY1缺陷型植株中端粒延长,端粒酶活性增加;在含有AtWHY1的转基因植物中端粒酶活性降低,端粒缩短[5]。Whirly蛋白在质体内能够维持质体基因组的稳定,调节质体基因的表达。Marechal等[6]发现,Whirly单链DNA结合蛋白在维持拟南芥质体基因组稳定方面起到重要的作用。Cappadocia等[7]的研究结果表明,Whirly蛋白与单链DNA的结合有助于准确修复断裂的双链DNA。
辣椒(Capsicum annuum L.)是茄科辣椒属的1年生或多年生草本植物,是世界上种植面积最广的香料作物,目前辣椒的全基因组测序和物理图谱已经完成[8]。目前国内外对Whirly基因家族的研究主要以拟南芥为对象,针对Whirly蛋白的结构和功能进行研究,而对于辣椒Whirly基因家族的研究还没有详细报道。为此,利用生物信息学的方法,对辣椒CM334基因组中Whirly基因家族的全部序列进行鉴定分析,并利用荧光定量PCR(qRT-PCR)检测了CM334中Whirly基因在逆境胁迫下的表达情况,为进一步研究Whirly基因家族在辣椒生长发育中的功能奠定基础。
1 材料和方法
1.1 植物材料
所用辣椒材料CM334由韩国首尔国立大学馈赠。辣椒培养条件参考文献[9-10]。当辣椒幼苗有6~8片真叶展开时按如下方法处理。
脱落酸(ABA)和茉莉酸甲酯(MeJA)处理:用脱落酸(100 μmol/L)和茉莉酸甲酯(200 μmol/L)分别喷洒辣椒叶片的正反面,以喷洒无菌水作为对照。
高温处理(Heat):对辣椒叶片进行高温处理,白天温度设置为(42±2)℃,晚上设置为(35±2)℃。对照组白天温度设置为28 ℃,晚上设置为26 ℃[11]。
低温处理(Cold):预先将光照培养箱的温度降至5 ℃(昼)/0 ℃(夜),随后将辣椒苗放入光照培养箱中培养。对照组培养温度与高温处理对照组相同。
疫病(YB)处理:将辣椒幼苗的根置于用Hoagland′s培养液配制的病原菌(来自河南农业大学毛庄实验基地)孢子悬浮液中,以无菌水作对照。28 ℃保湿24 h后,在温度28 ℃、相对湿度70%~90%条件下培养。
上述处理的辣椒植株在培养箱中培养的光照强度均为160 μmol/(m2·s),光周期均为12 h光/12 h暗。
1.2 辣椒Whirly基因家族成员的鉴定
将Whirly(pfam:PF08536)(http://pfam.xfam.org/)[12]的保守结构域在辣椒基因组数据库PGP(http://peppergenome.snu.ac.kr/,CM334蛋白)中进行Blast比对,获得的所有候选序列在NCBI(National Center for Biotechnology Information)中进行Blast 比对。利用在线分析工具ProtParam (http://web.sxpasy.org/protparam/)分析候选氨基酸序列的等电点及分子质量[13]。
1.3 基因结构和蛋白质基序分析
以候选Whirly家族成员的CDS序列和基因序列为参照,通过在线GSDS 2.0(Gene Structure Display Server,http://gsds.cbi.pku.edu.cn/)进行基因结构分析[14]。分析时将CDS序列和基因序列都转换成FASTA格式,还要保持同一基因2种序列的对应。用MEME Suite 4.12.0在线软件(http://meme-suite.org/tools/meme)对Whirly家族蛋白质基序进行鉴定。
1.4 系统进化树分析
Whirly家族蛋白质全长序列用ClustalW进行多序列比对,ClustalW比对参数如下。空位罚分:10;空位延伸罚分:0.2;蛋白质重量矩阵:gonnet;残留特定的罚分:开;亲水罚分:开;间隙分隔距离:4;末端间隙分割:关闭;负矩阵:关闭;延伸分歧截止:30%。比对后Whirly蛋白序列用MEGA 7.0软件,使用邻近法构建进化树,其中校验参数Bootstrap值设置为1 000次重复。
1.5 辣椒Whirly基因组织表达模式分析
在辣椒参考基因组(https://www.nature.com/articles/ng.2877)中获取CM334 RNA-seq数据,利用HemI绘制Whirly基因在辣椒不同组织和果实发育中的表达热图。
1.6 辣椒总RNA的提取及cDNA第1链的合成
总RNA提取采用TaKaRa MiniBeST Plant RNA Extraction Kit (TaKaRa, Inc., Dalian, China)试剂盒,按照说明书进行操作。cDNA第1链合成采用PrimeScriptTM 1st Strand cDNA Synthesis Kit(TaKaRa, Inc., Dalian, China)。
1.7 辣椒Whirly基因家族表达分析
采用qRT-PCR分析,引物见表1。反应体系为20.0 μL,其中cDNA为2.0 μL,上、下游引物均为1.0 μL,2×SYBR®Premix Ex TaqTM (TliRNaseH Plus) 10.0 μL以及ddH2O 6.0 μL。设置程序:95 ℃预变性30 s;95 ℃变性5 s,55 ℃退火30 s,72 ℃延伸60 s,循环40次[9]。采用2-ΔΔCT法计算基因相对表达量[15]。
表1 qRT-PCR引物序列
Tab.1 qRT-PCR primer sequences
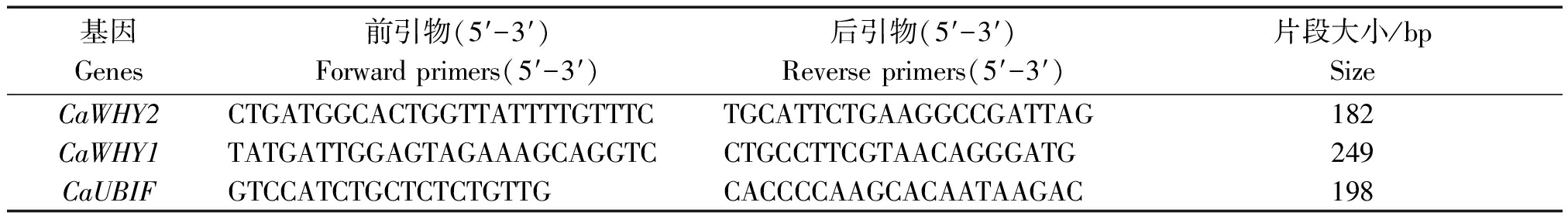
基因Genes前引物(5′-3′)Forward primers(5′-3′)后引物(5′-3′)Reverse primers(5′-3′)片段大小/bpSizeCaWHY2CTGATGGCACTGGTTATTTTGTTTCTGCATTCTGAAGGCCGATTAG182CaWHY1TATGATTGGAGTAGAAAGCAGGTCCTGCCTTCGTAACAGGGATG249CaUBIFGTCCATCTGCTCTCTGTTGCACCCCAAGCACAATAAGAC198
表2 Whirly基因家族成员鉴定结果
Tab.2 Whirly family member identification list

编号No.基因名称Gene name序列IDSequence ID开放阅读框/bpORF length氨基酸数/个Number of amino acids分子质量/kuMolecular weight等电点pI1AtWHY3AT2G0274080726829.03 9.542AtWHY1AT1G1441079226328.38 9.383AtWHY2AT1G7126071723825.68 9.654BdWHY1BRADI1G5104082527429.67 8.985BdWHY2BRADI3G0445069623124.79 9.526BoWHY1.bBo8g06603090630132.18 9.577BoWHY3Bo7g08056078326027.96 9.588BoWHY2Bo6g11284072324025.61 9.489BoWHY1.aBo7g0452501 07435738.97 9.7110CsWHY2Csa5M64729072924226.12 9.5611CsWHY1Csa6M40424083427730.01 9.0012CaWHY2CA10g0810071423725.71 9.7113CaWHY1CA11g0538048316017.35 6.8414GmWHY1.eGLYMA18G1620069022924.64 9.5415GmWHY1.dGLYMA08G4085081026929.25 9.2816GmWHY2.aGLYMA03G4127072924226.37 9.1017GmWHY1.cGLYMA02G0244138112614.27 5.5418GmWHY1.bGLYMA02G0239078926229.09 9.7119GmWHY1.aGLYMA01G0500079226329.04 9.5620GmWHY2.bGLYMA19G4388083727830.44 9.5821SiWHY1Si006951m93030933.91 9.9622SiWHY2Si018290m 66922224.08 9.2023SlWHY1Solyc05g00710080726828.84 9.1324SlWHY2Solyc11g04475071123625.83 9.9825VvWHY1VIT_01s0026g0172080726829.46 9.6826VvWHY2VIT_18s0089g0090069623125.18 9.40
2 结果与分析
2.1 辣椒Whirly基因家族成员的鉴定
以拟南芥的Whirly蛋白氨基酸序列为查询序列进行BlastP相似性检索,共鉴定了26个单子叶(短柄草、谷子)和双子叶(辣椒、拟南芥、甘蓝、黄瓜、大豆、番茄、葡萄)的Whirly基因家族成员(表2),其中辣椒CM334的Whirly基因有2个,分别为CaWHY1和CaWHY2。从表2可知,除了BdWHY1(pI=8.98)、CaWHY1(pI=6.84)、GmWHY1.c(pI=5.54)外,其他Whirly蛋白的等电点均在9.50左右。除CaWHY1和GmWHY1.c的分子质量分别为17.35,14.27 ku之外,其余Whirly蛋白的分子质量均在24.08~38.97 ku。
2.2 辣椒Whirly基因结构分析
通过GSDS基因结构分析可知,辣椒Whirly基因家族中CaWHY1有5个外显子、4个内含子,CaWHY2有7个外显子、6个内含子,CaWHY2和SlWHY2的基因结构完全一样(图1)。从图1还可以看出,除GmWHY1.c有2个内含子外,其他Whirly基因都有多个内含子(4~8个)。

图1 26个Whirly家族基因结构分析
Fig.1 Structure analysis of 26 Whirly family genes

图2 26个Whirly蛋白保守基序分布
Fig.2 Motif distribution of 26 Whirly proteins
2.3 辣椒Whirly蛋白保守基序分析
为了研究Whirly转录因子之间结构的多样性,利用MEME Suite 4.12.0 在线工具对26个不同物种的Whirly蛋白进行保守基序分析[16]。如图2所示,在Whirly家族中鉴定出3个保守基序。之前有研究报道表明,Whirly蛋白有3个结构域:N末端结构域、Whirly结构域和C末端多变区[17]。通过基序分析发现,Whirly基因家族在进化上结构具有保守性。
2.4 辣椒Whirly蛋白系统进化树分析
为更好地分析辣椒Whirly基因家族的进化,构建了辣椒、拟南芥、短柄草、甘蓝、黄瓜、大豆、谷子、番茄和葡萄的Whirly蛋白系统进化树(图3)。根据进化树的聚类结果,26个不同物种的Whirly蛋白明显分为3类:Class Ⅰ、 Class Ⅱ、Class Ⅲ。几乎所有的Whirly1属于Class Ⅰ,Whirly2属于Class Ⅱ,Whirly3属于Class Ⅲ。CaWHY1和SlWHY1聚在一起,CaWHY2和SlWHY2聚在一起,辣椒和番茄都属于茄科植物,其基因在功能上也有相似之处。同时,可以明显看出单子叶植物和双子叶植物分别聚在一起,表明单子叶和双子叶植物的Whirly基因在进化上有差异。
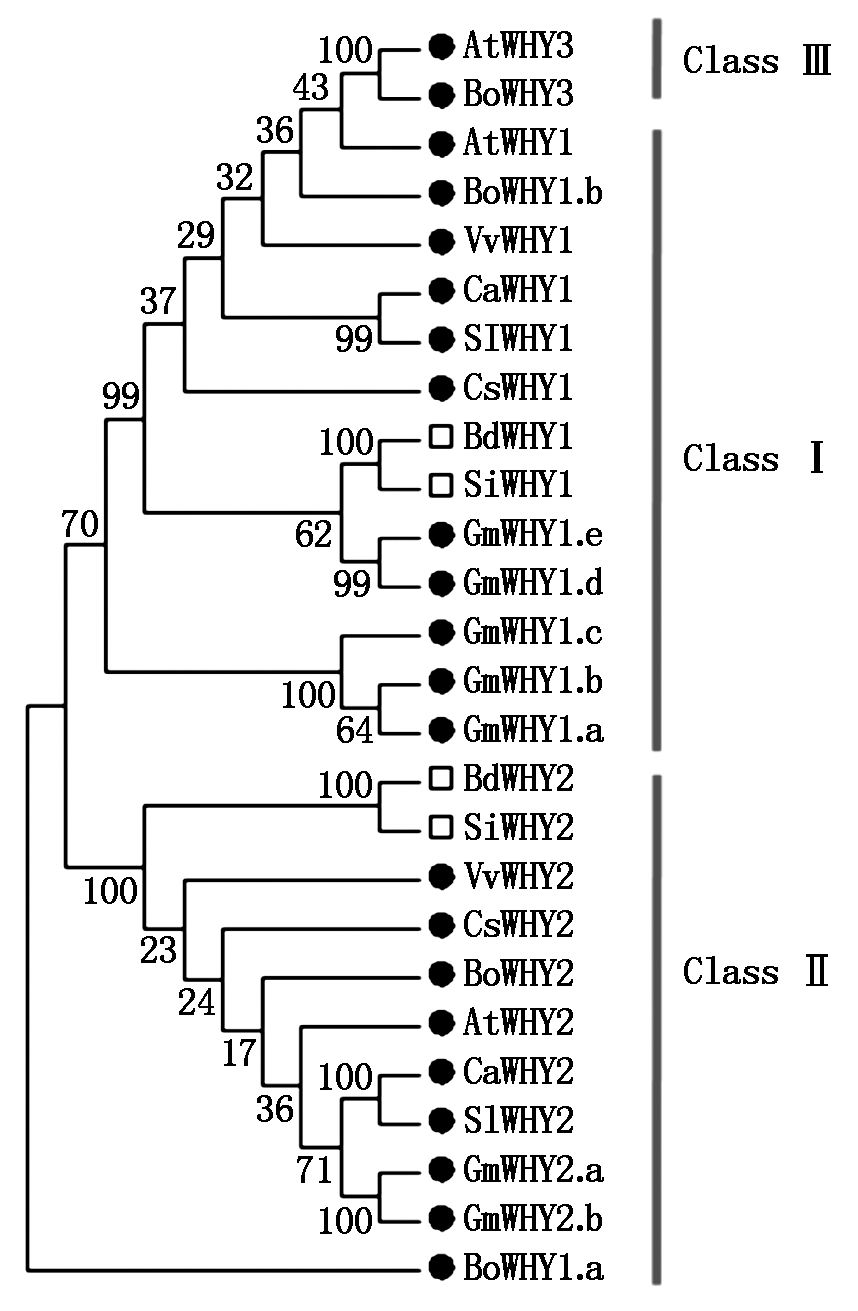
●.双子叶植物;□.单子叶植物。
●.Dicotyledon;□.Monocotyledon.
图3 26个Whirly蛋白的系统进化树
Fig.3 Phylogenetic tree constructed from 26 Whirly proteins
2.5 辣椒Whirly基因时空表达分析
为了探究Whirly基因在辣椒不同组织及果实发育中的作用,利用CM334的RNA-seq数据绘制了Whirly基因家族在4个组织(根、茎、叶和果实)以及7个果实发育时期的热图(图4)。结果显示,CaWHY1和CaWHY2在不同组织中的表达量有差异,CaWHY2在根、茎和叶中均表达,CaWHY1在根、茎、叶中表达量却很低。同时,在果实发育中CaWHY1表达量也很低。

图4 Whirly基因在辣椒不同组织和果实发育中的表达模式
Fig.4 Expression analysis of pepper Whirly genes in different tissues and fruit development
2.6 辣椒Whirly基因家族胁迫响应表达分析
利用qRT-PCR分析了辣椒CM334中的Whirly基因家族在逆境胁迫下的表达情况(图5)。辣椒的CaWHY1基因表达量在疫病胁迫下变化最为明显, 0~2 h急剧上升,2 h时达到最大值,为对照的4.5倍,随后表达量逐渐下降,24 h时下降到最小值,24~48 h又逐渐上升,48 h时其表达量为对照的1.8倍。CaWHY1在高温处理条件下,24 h时表达量达到最大值。在脱落酸、低温处理、茉莉酸甲酯胁迫下CaWHY1的表达量变化幅度相对不大。
CaWHY2在疫病和高温处理条件下的表达量变化最为明显,其中疫病胁迫下,在2 h 时其表达量达到最高值,为对照的2.3倍,随后逐渐降低,在12 h之后缓慢降低有趋于稳定之势,但最后表达量较对照有所下降。CaWHY2在高温处理条件下,2 h时其表达量上升至对照的1.7倍,6 h时又显著下降,之后逐渐上升,在24 h时表达量达到最大值,为对照的2.1倍,之后又逐渐下降,48 h时下降至对照的1.6倍。CaWHY2在脱落酸和低温处理条件下的表达模式相反,即当脱落酸胁迫下的表达量上升时,低温处理条件下的表达量下降,但脱落酸和低温处理条件下表达量的变化幅度不同。CaWHY2在茉莉酸甲酯处理条件下,0~2 h有不明显的上升,之后逐渐下降,24 h时降至最低值,约为对照的1/2,24~48 h逐渐上升,48 h时达到最大表达量,为对照的1.7倍。
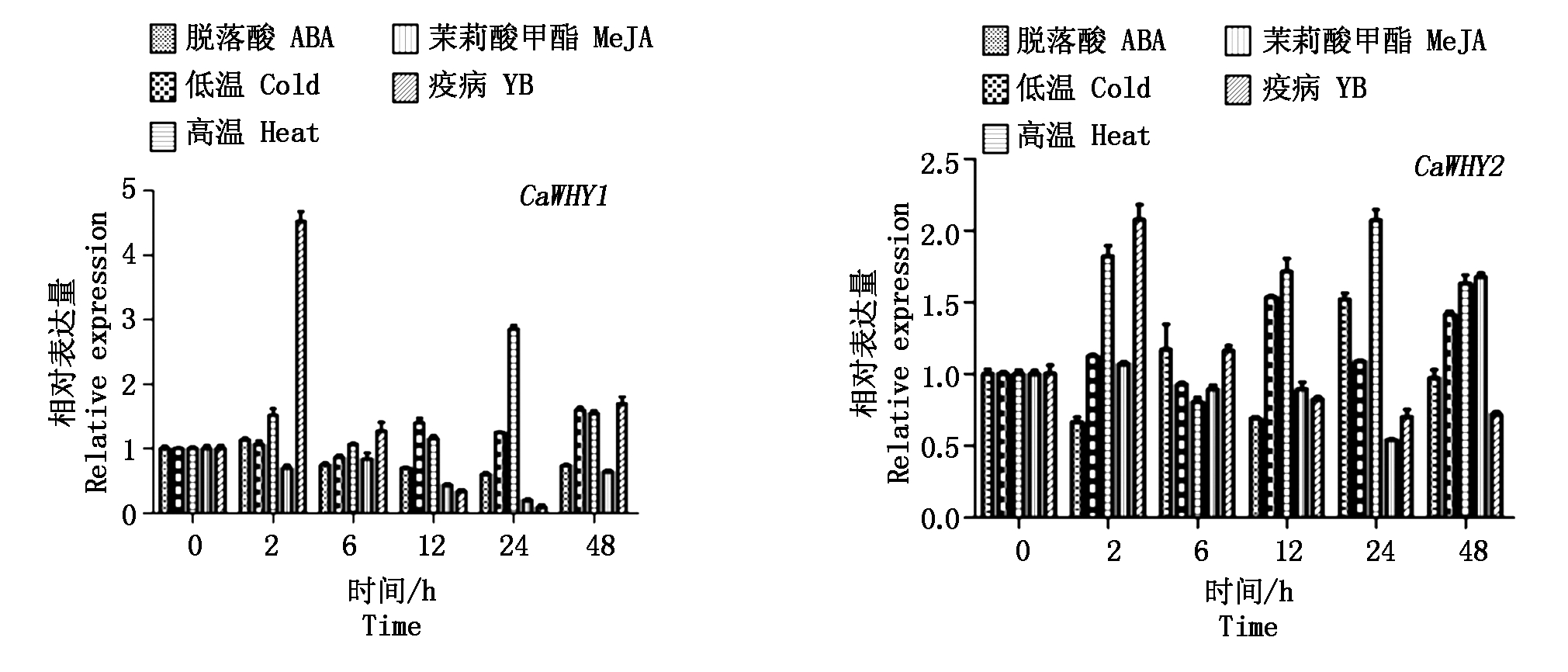
图5 辣椒Whirly基因家族响应胁迫的qRT-PCR分析
Fig.5 qRT-PCR analysis of pepper Whirly gene family reacting to stress
3 结论与讨论
Whirly基因家族在双子叶和单子叶植物中均有发现。本研究在辣椒CM334中鉴定了CaWHY1和CaWHY2 2个Whirly基因,其中CaWHY2和番茄SlWHY2的基因结构完全一样,均有7个外显子、6个内含子,在进化树中CaWHY2和SlWHY2也是聚在一起,这可能是由于辣椒和番茄均属于茄科,亲缘关系较密切[18]。对蛋白质基序分析发现,Whirly蛋白在植物中高度保守,含有3个保守的基序。Whirly基因表达模式分析表明,CaWHY1和CaWHY2在不同组织和果实发育时期的表达水平存在差异。在脱落酸、低温处理、高温处理、茉莉酸甲酯和疫病处理条件下,CM334的Whirly基因均有表达。在疫病处理下,CaWHY2和CaWHY1表达量均增加,其中CaWHY1基因的表达量较CaWHY2增加更明显。CaWHY2基因在脱落酸和低温处理条件下表达量的变化趋势相反。
Whirly转录因子在植物的生长发育过程中起到很重要的作用[19]。另有研究证明,Whirly转录因子在叶绿体和细胞核中也发挥一定作用[20]。本研究对辣椒Whirly基因表达模式分析表明,Whirly基因家族在辣椒的生长发育中起调控作用。植物需要适应不利的环境条件,包括生物和非生物胁迫[21-22]。杨洋等[23]研究发现,Whirly转录因子在植物应对低温胁迫中发挥作用。本研究中,Whirly基因在各种胁迫处理下的表达量变化再次证明,Whirly基因在多种生物和非生物抗性反应中起重要作用。
Whirly基因在植物界中广泛存在,在其他物种尤其是拟南芥中Whirly基因的研究较为详细,但是有关辣椒Whirly基因的报道却较少。本研究鉴定了辣椒CM334中的Whirly基因,对其结构、进化关系、保守基序和表达模式进行了初步分析,并对其在逆境胁迫下的表达量变化进行了分析,为进一步研究Whirly基因在辣椒中的功能奠定了基础。
[1] Krause K, Kilbienski I, Mulisch M, Rodiger A, Schafer A, Krupinska K. DNA-binding proteins of the Whirly family in Arabidopsis thaliana are targeted to the organelles[J]. FEBS Letters, 2005, 579(17): 3707-3712.doi:10.1016/j.febslet.2005.05.059.
[2] Desveaux D, Despres C, Joyeux A, Subramaniam R, Brisson N. PBF-2 is a novel single-stranded DNA binding factor implicated in PR-10a gene activation in potato[J]. The Plant Cell, 2000, 12(8): 1477-1489.doi:10.1105/tpc.12.8.1477.
[3] Desveaux D, Marechal A, Brisson N. Whirly transcription factors:Defense gene regulation and beyond[J]. Trends Plant Sci, 2005, 10(2): 95-102. doi:10.1016/j.tplants.2004.12.008.
[4] Desveaux D, Subramaniam R, Despres C, Mess J N, Levesque C, Fobert P R. A "Whirly" transcription factor is required for salicylic acid-dependent disease resistance in Arabidopsis[J]. Developmental Cell, 2004, 6(2): 229-240. doi:10.1016/s1534-5807(04)00028-0.
[5] Yoo H H, Kwon C, Lee M M, Chung I K. Single-stranded DNA binding factor AtWHY1 modulates telomere length homeostasis in Arabidopsis [J]. The Plant Journal, 2007, 49(3): 442-451. doi:10.1111/j.1365-313X.2006.02974.x.
[6] Marechal A, Parent J S, Veronneau-lafortune F, Joyeux A, Lang B F, Brisson N. Whirly proteins maintain plastid genome stability in Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(34): 14693-14698. doi:10.1073/pnas.0901710106.
[7] Cappadocia L, Marechal A, Parent J S, Lepage E, Sygusch J, Brisson N. Crystal structures of DNA-Whirly complexes and their role in Arabidopsis organelle genome repair[J]. The Plant Cell, 2010, 22(6): 1849-1867. doi:10.1105/tpc.109.071399.
[8] Kim S, Park M, Yeom S I, Kim Y M, Lee J M, Lee H A. Genome sequence of the hot pepper provides insights into the evolution of pungency in Capsicum species[J]. Nature Genetics, 2014, 46(3): 270-278. doi:10.1038/ng.2877.
[9] 魏小春, 姚秋菊, 原玉香, 赵艳艳, 王志勇, 姜俊, 蒋武生,张晓伟. 辣椒CaWRKY13基因克隆及非生物胁迫下表达分析[J].分子植物育种,2016,14(10):2582-2588. doi:10.13271/j.mpb.014.002582.
Wei X C Yao Q J, Yuan Y X, Zhao Y Y, Wang Z Y, Jiang J,Jiang W S, Zhang X W. Cloning and expression analysis of CaWRKY13 gene from Capsicum annuum L. under abiotic stress[J]. Molecular Plant Breeding, 2016, 14(10): 2582-2588.
[10] 魏小春, 李艳, 姚秋菊, 原玉香, 赵艳艳, 王志勇, 姜俊, 段俊枝, 蒋武生, 张晓伟. 辣椒CaCBF1A基因的克隆及非生物胁迫下表达分析[J]. 河南农业科学, 2016,45(12):110-115. doi:10.15933/j.cnki.1004-3268.2016.12.022.
Wei X C, Li Y, Yao Q J, Yuan Y X, Zhao Y Y, Wang Z Y, Jiang J, Duan J Z, Jiang W S, Zhang X W. Cloning and expression analysis of CaCBF1A gene from Capsicum annuum L.under abiotic stress[J]. Journal of Henan Agricultural Sciences, 2016,45(12):110-115.
[11] 闫秋艳, 董飞, 段增强, 李汛,王嫒华,汤英. 土壤温度对不同施肥方式下辣椒生长及土壤理化性质的影响[J]. 华北农学报, 2018,33(2):195-201. doi:10.7668/hbnxb.2018.02.027.
Yan Q Y, Dong F, Duan Z Q, Li X, Wang A H, Tang Y. Effect of soil temperature on soil physical-chemical properties and pepper growth in different fertilizer treatments[J]. Acta Agriculturae Boreali-Sinica,2018,33(2):195-201.
[12] Xu Q, Dunbrack R L. Assignment of protein sequences to existing domain and family classification systems: Pfam and the PDB[J]. Bioinformatics, 2012, 28(21): 2763-2772. doi:10.1093/bioinformatics/bts533.
[13] Artimo P, Jonnalagedda M, Arnold K, Baratin D, Csardi G, de Castro E. ExPASy: SIB bioinformatics resource portal[J]. Nucleic Acids Research, 2012, 40: 597-603. doi:10.1093/nar/gks400.
[14] Barve A, Joshi B K. A smart source code editor for C[J]. International Journal of Computer Science Engineering & Bioinformatics, 2014, 4(3): 23-28.
[15] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-delta delta C(T)) method[J]. Methods, 2001, 25(4): 402-408. doi:10.1006/meth.2001.1262.
[16] Bailey T L, Elkan C. Fitting a mixture model by expectation maximization to discover motifs in biopolymers[J]. Proceedings International Conference on Intelligent Systems for Molecular Biology, 1994, 2:28-36.
[17] 孔凡英, 邓永胜, 周斌, 孟庆伟. Whirly转录因子研究进展[J]. 植物生理学报, 2012, 48(7): 643-653. doi:10.13592/j.cnki.ppj.2012.07.014.
Kong F Y, Deng Y S, Zhou B, Meng Q W. Research advancement of Whirly transcription factors[J]. Plant Physiology Journal, 2012, 48(7): 643-653.
[18] 魏瑞敏, 谢玲玲, 欧阳娴, 张亚利, 戴雄泽, 刘峰. 辣椒ARF基因家族的鉴定与表达分析[J]. 西北植物学报, 2017, 37(6): 1047-1058. doi:10.7606/j.issn.1000-4025.2017.06.1047.
Wei R M, Xie L L, Ouyang X, Zhang Y L, Dai X Z, Liu F.Identification and expression analysis of ARF gene family in pepper[J]. Acta Botanica Boreali-Occidentalia Sinica, 2017, 37(6): 1047-1058.
[19] Grabowski E, Miao Y, Mulisch M, Krupinska K. Single stranded DNA-binding protein Whirly1 in barley leaves is located in plastids and the nucleus of the same cell[J]. Plant Physiol,2008,147:1800-1804. doi: 10.1104/pp.108.122796.
[20] Desveaux D, Marechal A, Brisson N. Whirly transcription factors:Defense gene regulation and beyond[J]. Trends in Plant Science, 2005, 10(2): 95-102. doi:10.1016/j.tplants.2004.12.008.
[21] Miller G, Suzuki N, Ciftci-yilmaz S, Mittler R. Reactive oxygen species homeostasis and signalling during drought and salinity stresses[J]. Plant, Cell & Environment, 2010, 33(4): 453-467. doi:10.1111/j.1365-3040.2009.02041.x.
[22] Chinnusamy V, Zhu J H, Zhu J K. Cold stress regulation of gene expression in plants[J]. Trends in Plant Science, 2007, 12(10): 444-451. doi:10.1016/j.tplants.2007.07.002.
[23] 杨洋, 赖恭梯, 赖钟雄. 三明野生蕉Whirly转录因子的克隆及其在低温胁迫下的定量表达分析[J]. 热带作物学报, 2014,35(8):1533-1538.doi:10.3969/j.issn.1000-2561.2014.08.015.
Yang Y, Lai G T, Lai Z X. Cloning and expression under low temperature conditions of Whirly transcription factor in a wild banana accession (Musa spp.) from Sanming city[J]. Chinese Journal of Tropical Crops,2014,35(8):1533-1538.