在真核生物中,反式作用因子(Trans-acting factor)能直接或间接识别、结合启动子及RNA聚合酶[1]。此类蛋白的不同功能区通过结合功能基因或调控基因的启动子顺式作用元件或与其他蛋白互作来调控基因的表达[2]。植物体内参与逆境胁迫响应的主要转录因子家族包括AP2/ERF、bZIP、WRKY、NAC、MYB等,而植物体内普遍存在且数量众多、功能多样的bZIP转录因子家族是抗逆性形成的主要参与者[3]。碱性亮氨酸拉链(Basic leucine zipper,bZIP)是广泛存在于真核生物中的保守转录因子,多序列比对分析显示,其核心bZIP结构域由60~80个氨基酸残基组成,包括碱性结构域(BR)与亮氨酸拉链区(LZ)[4]。BR位于bZIP蛋白的N端,由约16个氨基酸残基组成N-X7-R/K(X代表任意氨基酸残基)基序[3],是包括核定位信号以及可以结合顺式作用元件的碱性区域,而LZ区每隔7个氨基酸残基存在1个亮氨酸,通过形成双极性的α-螺旋结构与BR区紧密结合并调节bZIP蛋白与DNA结合之前的二聚体化过程[3,5]。bZIP 类转录因子识别基因启动子的核心序列为ACGT顺式作用元件如CACGTG(G盒)、GACGTC(C盒)、TACGTA(A盒)、AACGTT(T盒)以及TGA(G/C)TCA(GCN4)等,一些受光或脱落酸(ABA)诱导的基因的启动子区都含有这些元件,其中G盒元件普遍存在于受 ABA、生长素、茉莉酸、水杨酸诱导的基因中[3,6-7]。根据bZIP转录因子DNA的结合特异性及碱性区域的氨基酸排列顺序,Jakoby等[3]将bZIP基因家族分为A、B、C、D、E、F、G、H、I及S 10个亚族,其中,A亚族与S亚族研究较为广泛,而B亚族与F亚族则报道较少。文献报道,A、C、S亚族主要参与植物非生物胁迫响应过程及激素信号转导过程的调控,而D、G、H、I亚族在植物防御生物病害、种子成熟、光形态建成、氮素同化、维管系统发育等生理生化过程发挥重要作用[8]。
目前,多种植物的bZIP基因家族已经在全基因组水平进行了鉴定及分析,其中拟南芥75个[3]、水稻89个[4]、玉米125个[5]、大豆131个[9]、高粱92个[10]、葡萄55个[11]、番茄76个[12]、苹果120个[13]。
小桐子(Jatropha curcas L.)属大戟科(Euphorbiaceae)麻疯树属(Jatropha)落叶灌木或小乔木,作为新兴开发的木本油料植物,其种子含油量高,品质优良,成分接近石化柴油,兼具良好的经济效益与生态效应,具有极为广阔的开发前景。2011年,小桐子基因组由日本Kazusa DNA研究所首次测序完成[14],2014年,中国科学院对小桐子基因组进行了高通量重测序,为从基因组水平上分析小桐子bZIP基因家族提供了条件。本研究对小桐子bZIP基因家族进行了全基因组鉴定,并系统分析了其基因成员的理化性质、基因结构、功能结构域、进化关系、染色体定位、共线性关系及低温条件下的表达特性等,为筛选潜在的与小桐子抗冷相关bZIP基因并进行功能鉴定奠定了基础。
1 材料和方法
1.1 试验材料及处理
试验所用小桐子种子取自云南省楚雄州元谋县干热河谷地区(东经101°40′,北纬25°23′)。选取饱满的小桐子种子,用1.0% CuSO4消毒20 min,无菌水漂洗5次,置烧杯中水浸吸涨24 h。将吸涨的种子在无菌水中漂洗3次,播于垫有饱和无菌水海绵的托盘中,于温度26 ℃、相对湿度75%的恒温培养箱中萌发5 d。将发芽的种子播于消毒的培养土中并于温度26 ℃、相对湿度75%、光周期16 h/8 h的恒温培养箱中生长15 d至第2片真叶展开,每天用无菌水浇灌培养土。将生长15 d的小桐子幼苗置于温度12 ℃、相对湿度75%、16 h/8 h光周期的低温培养箱中进行低温处理,分别取低温处理12,24,48 h与对照(CK,正常培养)的第2片真叶与根,以及吸涨24 h的种子,液氮速冻后保存于-80 ℃冰箱中用于RNA的提取。
1.2 试验方法
1.2.1 小桐子bZIP基因家族的鉴定 从GenBank下载小桐子最新注释蛋白质数据库。通过Pfam(http://pfam.xfam.org/)下载bZIP结构域的隐马可夫HMM模型(PF00170、PF07716、CPF12498、PF03131),利用Hmmer 3.0软件的Hmmsearch程序对小桐子蛋白质数据库进行检索(阈值E<1e-10,序列相似性>50%),得到候选的小桐子bZIP蛋白质序列,利用Excel脚本去除重复序列。从TAIR数据库(https://www.arabidopsis.org/)下载75个已经鉴定的拟南芥bZIP基因的蛋白质序列[3],通过ClustalX进行多重序列比对,利用Hmmer 3.0软件的Hmmbuild程序将比对文件转换为HMM模型,同上条件对小桐子蛋白质数据库进行检索。将以上2种方法获得的小桐子bZIP蛋白质序列取并集,经过去除重复序列,将非冗余的候选序列利用Pfam与CDD在线工具分析bZIP蛋白结构域做进一步筛选,得到最终的小桐子bZIP家族蛋白质序列。
1.2.2 小桐子bZIP家族基因的基本参数分析 将鉴定到的小桐子bZIP蛋白质序列对小桐子基因组数据库进行tBlastN相似性检索,下载其对应的基因序列、ORF(Open Reading Frame)序列、mRNA序列以及CDS(Coding sequence)序列。利用ExPaSy提供的在线工具ProtParam(http://web.expasy.org/protparam/)对得到的小桐子bZIP蛋白质序列进行氨基酸数目、理论分子量(Mw)、等电点(pI)、氨基酸组成等基本参数的分析。亚细胞定位采用CELLO服务器V2.5(http://cello.life.nctu.edu.tw/)进行鉴定。另外,根据bZIP的CDS序列对小桐子基因组进行BlastN相似性检索得到各bZIP基因起始密码子ATG上游1 500 bp的调控序列,并通过PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)对其顺式作用元件进行鉴定。
1.2.3 系统进化树构建与蛋白质结构域分析 将鉴定到的小桐子bZIP蛋白序列利用ClustalX(Version 2.0)进行序列相似性比对,然后用MEGA 6.0软件通过邻接法(NJ)构建系统进化树,并采用自展法(Bootstrap)进行检验。利用软件GenBank的Spidey工具(https://www.ncbi.nlm.nih.gov/sutils/splign/splign.cgi)进行bZIP的CDS序列与基因序列比对以确定基因内含子与外显子的结构,并利用GSDS(Gene Structure Display Server,http://gsds.cbi.pku.edu.cn/)绘制基因结构图。利用MEME(http://meme-suite.org/tools/meme)在线分析工具对bZIP蛋白质序列进行motif分析(参数设置:最优氨基酸残基宽度6~200;任意数量的重复次数;最多预测10个motif)。利用GenDOC软件对ClustalX比对结果进行bZIP蛋白的bZIP保守结构域分析。
1.2.4 染色体定位及基因家族复制与共线性分析 染色体定位以Wu等[15]构建的小桐子遗传连锁图谱进行锚定,并通过MapChart(Version 2.1)绘制基因定位图。利用Mauve(Version 2.4)进行小桐子bZIP家族基因的复制共线性分析,其中,片段复制(Ancient segmental duplication)基因对及对应的Ka(The number of nonsynonymous substitutions per nonsynonymous site,非同义突变率)、Ks(The number of synonymous substitutions per synonymous site,同义突变率)值通过PAL2NAL在线软件的CODEMAL程序进行鉴定与计算[16],并通过Ka/Ks比值以确定基因在进化中受到的选择压力,分化时间采用Ks/2λ计算,其中λ取值为6.1×10-9,同时,利用Circos(http://www.circos.ca/)(Versioin0.69)绘制bZIP家族基因在小桐子染色体的定位及基因间的共线性关系。
1.2.5 小桐子bZIP家族基因的表达分析 从GenBank的SRA数据库下载小桐子不同器官的Illumina高通量测序数据(叶片SRR1639660、根SRR1639659、种子SRR1639661)。通过Bowtie2与Samtools工具将鉴定到的小桐子bZIP家族基因与测序数据进行比对,得到各bZIP基因的表达Reads数据,之后通过Cufflinks程序计算每个基因的表达量FPKM(Fragments Per Kilobase per Million)值,进行以2为底的对数转化,选择层次聚类法(Hierarchical clustering)对基因与不同器官分别进行聚类。另外,以笔者前期小桐子转录组[17]与数字基因表达谱(Digital gene expression)[18]数据为基础,提取对照与12 ℃低温处理12,24,48 h的bZIP基因的原始Clean Taq数据,通过TPM(Transcript per million clean tags)获得标准化的基因表达量[19-20],得到小桐子bZIP家族基因在低温处理下的差异表达数据。利用R软件(Version 3.4.1)的Gplots与Pheatmap函数进行聚类分析热图(Heatmap)的绘制。
利用TRIzol法(Invitrogen公司)提取小桐子叶片、根、种子及对照与不同低温处理下叶片与根的总RNA,并利用DNase Ⅰ(Thermo公司)消化RNA中的残余基因组DNA,得到纯化的总RNA。分别取3 μg总RNA,以Random primer为逆转录引物,利用RevertAid First Strand cDNA Synthesis Kit(Thermo公司)合成第一链cDNA。基于高通量差异表达数据,分别以β-Actin与GADPH为器官表达与低温表达内参,进行bZIP基因的器官(JcbZIP3、JcbZIP17、JcbZIP22、JcbZIP33、JcbZIP41)与低温处理(JcbZIP1、JcbZIP3、JcbZIP6、JcbZIP10、JcbZIP14)qRT-PCR(Quantitative real-time PCR)表达分析,所用仪器为Bio-Rad CFX Connect,试剂为Power SYBR Green PCR Master Mix(Thermo公司),所用引物序列见表1,20 μL反应体系,每个样品重复3次。扩增条件为:95 ℃预变性3 min;95 ℃变性10 s,55 ℃退火20 s,72 ℃延伸20 s,45个循环;之后增加溶解曲线程序:65~95 ℃连续检测信号5 s,0.5 ℃增量。采用2-ΔΔCt法进行相对表达量分析。器官差异表达分析以叶片的表达量为基准,低温差异表达分析以对照(CK)的表达量为基准。
表1 qRT-PCR表达分析引物序列
Tab.1 Primer sequences for qRT-PCR expression analysis

引物名称Primer name上游引物 (5′-3′)Forward primer (5′-3′)下游引物 (5′-3′)Reverse primer (5′-3′)扩增长度/bpAmplification lengthJcbZIP1GCAGACGATGGAGCAACAAGACCCACCACCATTTGCCATA113JcbZIP3TACCACAGCGACAAACTGCTGGTCCATGATGCGGATGGAT165JcbZIP6ACGGAAACAGGCTCACACAAGTGGTGGTCTTCGCTTCTCA158JcbZIP10TGGGAGACTACGAGCCTTGTTGTCACAGGATCACGACCAG166JcbZIP14ACAAGGACTCGGCTGCTTCCGGGGTCTTCATCGTCGTT114JcbZIP17CGGCATCAGATCAACGACAACATGGTAGGCTGCTGTGTTGTT130JcbZIP22CCGCCAGGCCGATAATTTGAGGACGAGATGCCCAAAGG145JcbZIP33CACTACTCAGACCTCAAGCCTGAGACGAACAACCTGAGACCA180JcbZIP41GCTACGGAATAGGGTCTCTGCTTTCGAAGCATGGCGTTTTCA153β-ActinGTCAGCCACACAGTCCCTATCCCTCACAATTTCCCGTTCAGC156GAPDHTGAAGGACTGGAGAGGTGGAATCAACAGTTGGAACACGGAA140

AA.氨基酸残基数量;Ip.等电点;EN.外显子数量。
The AA, Ip, and EN represent amino acid residue count, isoelectric point, and exon count, respectively.
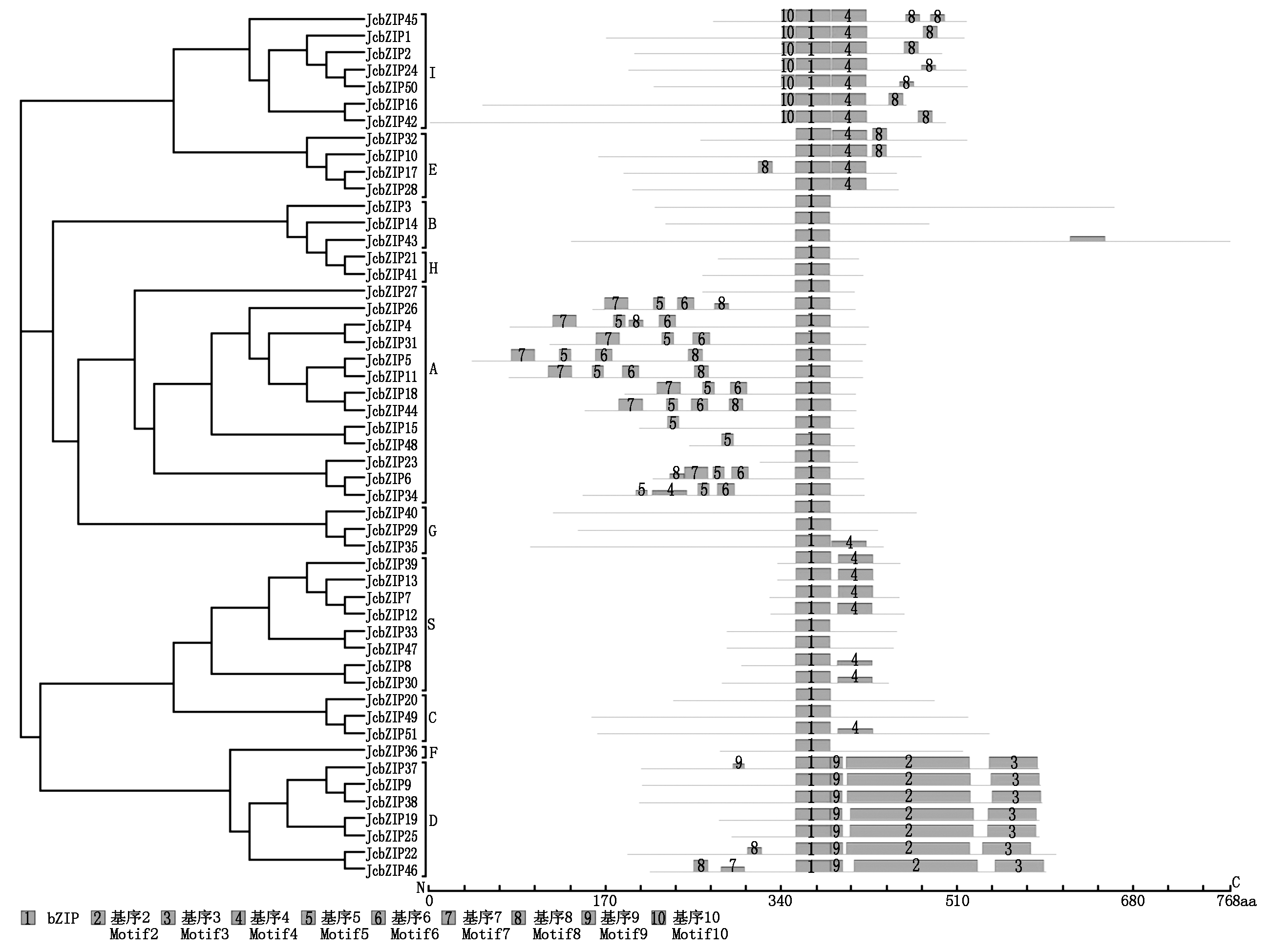
图1 小桐子bZIP基因家族的基因结构
Fig.1 Gene structure of J.curcas bZIP genes
2 结果与分析
2.1 小桐子bZIP基因家族的鉴定与序列特征
通过bZIP结构域的HMM模型检索,在小桐子全基因组共鉴定到51个bZIP基因家族成员,分别命名为JcbZIP1-JcbZIP51(表2、图1)。理化参数分析结果表明,小桐子51个bZIP基因的基因长度分布在647(JcbZIP23)~16 972(JcbZIP6)bp,蛋白质长度分布在113(JcbZIP13)~768(JcbZIP43)个氨基酸,理论等电点分布在4.70(JcbZIP14)~10.30(JcbZIP15)。根据CELLO的预测显示,除JcbZIP34定位在叶绿体之外,其他50个小桐子bZIP基因都定位在细胞核(表2)。
表2 小桐子bZIP基因家族基因参数
Tab.2 Gene parameters of bZIP genes in J.curcas

基因名称Gene names基因编号Gene ID基因长度/bpGene length蛋白质编号Protein ID氨基酸数目Amino acid等电点Ip分子量/kuMw亚细胞定位Subcellular localization染色体Chromosome外显子数目Exon number正负链(+/-)Plus/Minus strandJcbZIP11056284103 944XP_012065205.14185.9645.452 31细胞核24-JcbZIP21056291245 582XP_012066035.23598.7839.311 76细胞核14+JcbZIP31056294736 001XP_012066463.15368.4458.304 33细胞核66+JcbZIP41056294913 711XP_012066490.14197.7445.944 97细胞核66+JcbZIP51056298275 086XP_012066866.14569.5849.808 56细胞核104-JcbZIP610563006916 972XP_012067143.12476.8727.884 44细胞核24-JcbZIP71056301301 665XP_012067226.11528.1417.082 26细胞核22-JcbZIP8105630813921XP_012068179.11539.0917.647 88细胞核41-JcbZIP91056312438 223XP_012068684.14656.5451.360 42细胞核212-JcbZIP101056333197 285XP_012071280.13778.4542.491 71细胞核105-JcbZIP111056336384 424XP_012071654.14139.6044.863 34细胞核45-JcbZIP121056338421 551XP_012071923.11575.8917.903 97细胞核41+JcbZIP13105634632342XP_012072901.111310.1713.497 42细胞核91-JcbZIP141056351602 023NP_001292941.13084.7034.373 76细胞核43+JcbZIP151056353041 787XP_020535384.125110.3028.245 67细胞核44+JcbZIP161056353783 650XP_012073848.14946.7053.551 66细胞核44-JcbZIP171056357332 883XP_012074209.13195.7736.210 10细胞核14+JcbZIP181056361426 994XP_012074718.12708.7830.126 85细胞核83-JcbZIP191056365127 345XP_012075186.13747.7542.040 88细胞核79+JcbZIP201056374501 828XP_012076304.13056.1833.660 95细胞核45+JcbZIP211056376612 411XP_012076602.11659.6818.311 17细胞核44-JcbZIP221056386406 640XP_012077857.15006.9655.394 30细胞核213+JcbZIP23105638644647XP_012077862.11159.8613.718 85细胞核23+JcbZIP241056387442 810XP_012077996.13946.2142.917 47细胞核44-JcbZIP251056388596 634XP_012078125.13596.8240.560 68细胞核49+JcbZIP261056390261 744XP_012078352.13076.8834.645 72细胞核43-JcbZIP271056391261 448XP_012078465.11789.8820.220 66细胞核83-JcbZIP281056395623 384XP_012079033.13116.1634.816 70细胞核74+JcbZIP291056395875 955XP_012079074.13505.4837.253 85细胞核712-JcbZIP301056399981 437XP_012079592.11959.5522.638 22细胞核71+JcbZIP311056401543 207XP_012079779.13697.7541.352 14细胞核25+JcbZIP321056407242 818XP_012080512.13119.1335.657 62细胞核45+JcbZIP33105640790976XP_012080573.11995.9822.939 57细胞核51+JcbZIP341056409375 849XP_012080743.13299.3936.169 89叶绿体44+JcbZIP3510564095713 490XP_012080771.14126.1843.700 25细胞核313+JcbZIP361056417884 060XP_012081777.12845.9331.015 49细胞核23+JcbZIP371056419983 776XP_012082054.14646.0551.229 24细胞核511+JcbZIP381056423567 529XP_012082564.14686.7051.952 16细胞核611-JcbZIP391056426211 312XP_012082892.11445.5616.641 84细胞核81+JcbZIP401056428945 539XP_012083267.14248.4844.961 67细胞核813-JcbZIP411056436501 400XP_012084230.11889.4621.356 97细胞核14+JcbZIP421056438244 023XP_012084427.16016.1865.477 92细胞核25-JcbZIP431056438783 535XP_012084505.17686.4683.056 91细胞核62+JcbZIP441056442378 063XP_020538914.13178.6035.627 28细胞核104-JcbZIP451056447122 600XP_012085550.12964.8733.362 99细胞核74+JcbZIP461056464896 835XP_020539936.14626.4152.096 80细胞核611-JcbZIP471056467661 299XP_012088083.11955.5122.569 91细胞核91-JcbZIP481056471351 768XP_020540142.119410.1021.790 36细胞核93+JcbZIP491056476884 835XP_012089255.14396.2847.193 18细胞核66+JcbZIP501056491814 213XP_012091145.13665.7440.613 24细胞核114+JcbZIP511056499103 552XP_012092128.14576.8549.029 45细胞核36+
2.2 小桐子bZIP基因家族的基因结构与结构域分析
根据Jakoby等[3]拟南芥bZIP基因家族的分类标准,通过ClustalX构建小桐子bZIP基因家族进化树,依据聚类特点及bZIP结构域的氨基酸特性,分为A(13个)、B(3个)、C(3个)、D(7个)、E(4个)、F(1个)、G(3个)、H(2个)、I(7个)及S (8个)10个亚族(图1)。结果显示,聚类亚族与bZIP基因结构、氨基酸长度、等电点及外显子数量呈直接相关性。其中,D、E、G、H及S亚族的氨基酸长度分布较为集中,S亚族蛋白长度主要分布在100~200个氨基酸,E亚族蛋白分布在300~400个氨基酸,而C与G亚族蛋白分布在300~450个氨基酸。另外,C与D亚族成员的等电点多集中在6~7,而A与H亚族成员的等电点多偏碱性,大部分分布在7~10。同时,各亚族成员都存在显著的基因结构特异性,S亚族除JcbZIP7之外,7个基因都只有1个外显子,且基因长度较短,都存在5′-UTR与3′-UTR区域;E、H及I亚族基因存在4~5个外显子,且大部分基因的第一与最后一个外显子分别与5′-UTR和3′-UTR区域重合,而中间的2~3个外显子则较短且较为分散;A亚族基因存在3~6个外显子,且部分基因如JcbZIP4、JcbZIP11、JcbZIP31及JcbZIP44存在独立外显子的5′-UTR区域,而D(JcbZIP9、JcbZIP19、JcbZIP22及JcbZIP25)与G(JcbZIP29、JcbZIP35及JcbZIP40)亚族部分基因也存在类似现象;G亚族3个基因包含12~13个外显子,是小桐子bZIP基因家族中外显子最多的,且分布规律与D亚族基因类似(图1)。
通过MEME对小桐子51个bZIP基因的蛋白序列进行结构域检索,都鉴定到1个长度为60~80个氨基酸的保守bZIP结构域(图2)。其中, D与S亚族成员bZIP结构域主要分布在N端,亚族A、E、G及H主要分布在C端,而亚族B、C、F及I则主要集中在氨基酸序列的中部。进一步通过ClustalX对小桐子51个bZIP基因的bZIP结构域进行多序列比对,表明小桐子bZIP结构域N端都含有一个碱性区域N-X7-R/K,接着R/K 9个氨基酸之后,每隔7个氨基酸存在一个Leu,即亮氨酸拉链区域。其中,碱性区域R/K氨基酸残基(图3星号所示)之后的-RK-(图3箭头所示)绝对保守,所有小桐子bZIP成员都相同,亮氨酸拉链区的第2个Leu也较为保守,而第3个Leu则在不同的bZIP成员中差异较大(图3)。
1号表示bZIP结构域。
bZIP domains are indicated by No.1.
图2 小桐子bZIP家族蛋白的保守基序
Fig.2 Conserved motifs of J.curcas bZIP genes
2.3 小桐子bZIP家族基因顺式作用元件鉴定
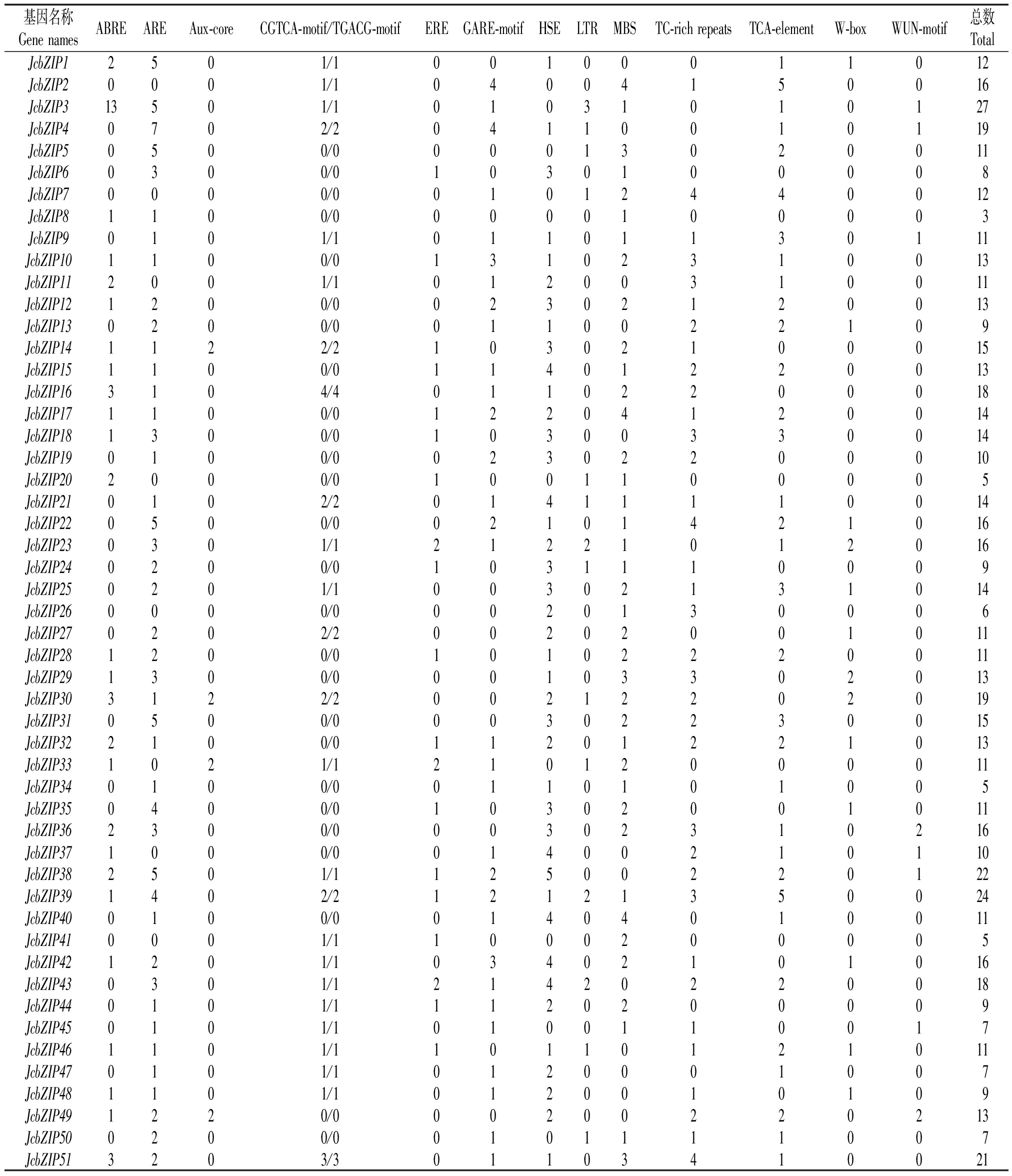
通过PlantCARE工具对小桐子51个bZIP基因起始密码子ATG上游1 500 bp启动子序列的13个顺式作用元件进行鉴定(表3),结果表明,JcbZIP3基因鉴定到最多27个元件,而JcbZIP8仅发现3个元件。其中,响应激素的元件中,包含赤霉素元件(GARE-motif)、脱落酸元件(ABRE)、乙烯元件(ERE)、生长素元件(Aux-core)的bZIP基因分别鉴定到31,26,19,4个,其中JcbZIP2与JcbZIP4最多鉴定到4个赤霉素响应元件、JcbZIP3最多鉴定到13个脱落酸响应元件。另外,分别有25,33个小桐子bZIP基因鉴定到响应茉莉酸甲酯(CGTCA-motif/TGACG-motif)与水杨酸(TCA-element)信号分子元件。同时有35个bZIP基因还发现了防御与胁迫响应相关元件(TC-rich repeats),占小桐子bZIP基因总数的68.7%,预示大部分bZIP基因参与小桐子逆境胁迫应答过程,其中,41个(80.4%)bZIP基因包含高温响应元件(HSE)、21个包含创伤响应元件(W-box、WUN-motif)、14个包含低温响应元件(LTR)。

实线表示碱性结构域(BR),虚线表示亮氨酸拉链区(LZ);星号表示N-X7-R/K基序的保守Arg(R)或Lys(K)残基;三角形表示保守的Leu残基;箭头表示保守的-RK-残基。
Basic region (BR) and Leucine zipper (LZ) are showed by solid and dotted lines, respectively; the asterisk represents the conserved Arg or Lys residues in N-X7-R/K motif; the triangles indicate the three conserved Leu residues in leucine zipper region; and the arrows mean the conserved-RK-residues.
图3 小桐子bZIP结构域序列比对
Fig.3 Sequence alignment of bZIP domain in J.curcas
表3 小桐子bZIP家族基因启动子中顺式作用元件的鉴定
Tab.3 Identification of cis-acting elements in promoter sequence of bZIP genes in J.curcas
基因名称Gene namesABREAREAux-coreCGTCA-motif/TGACG-motifEREGARE-motifHSELTRMBSTC-rich repeatsTCA-elementW-boxWUN-motif总数TotalJcbZIP12501/100100011012JcbZIP20001/104004150016JcbZIP313501/101031010127JcbZIP40702/204110010119JcbZIP50500/000013020011JcbZIP60300/01030100008JcbZIP70000/001012440012JcbZIP81100/00000100003JcbZIP90101/101101130111JcbZIP101100/013102310013JcbZIP112001/101200310011JcbZIP121200/002302120013JcbZIP130200/00110022109JcbZIP141122/210302100015JcbZIP151100/011401220013JcbZIP163104/401102200018JcbZIP171100/012204120014JcbZIP181300/010300330014JcbZIP190100/002302200010JcbZIP202000/01001100005JcbZIP210102/201411110014JcbZIP220500/002101421016JcbZIP230301/121221012016JcbZIP240200/01031110009JcbZIP250201/100302131014JcbZIP260000/00020130006JcbZIP270202/200202001011JcbZIP281200/010102220011JcbZIP291300/000103302013JcbZIP303122/200212202019JcbZIP310500/000302230015JcbZIP322100/011201221013JcbZIP331021/121012000011JcbZIP340100/00110101005JcbZIP350400/010302001011JcbZIP362300/000302310216JcbZIP371000/001400210110JcbZIP382501/112500220122JcbZIP391402/212121350024JcbZIP400100/001404010011JcbZIP410001/11000200005JcbZIP421201/103402101016JcbZIP430301/121420220018JcbZIP440101/11120200009JcbZIP450101/10100110017JcbZIP461101/110110121011JcbZIP470101/10120001007JcbZIP481101/10120010109JcbZIP491220/000200220213JcbZIP500200/00101111007JcbZIP513203/301103410021
注:ABRE (TACGTG).脱落酸响应元件;ARE (TGGTTT/AAACCA).厌氧响应元件; Aux-core (GGTCCAT).生长素响应元件;CGTCA-motif/TGACG-motif (CGTCA/TGACG).茉莉酸甲酯响应元件; ERE (ATTTCAAA).乙烯响应元件;GARE-motif (AAACAGA/TCTGTTG).赤霉素响应元件; HSE (GAAAATTCG/AAAAAATTTC/AAAAAATTTC/AAAAAATTTC).高温响应元件; LTR (CCGAAA).低温响应元件; MBS (CGGTCA).MYB结合元件;TC-rich repeats (ATTCTCTAAC/ATTTTCTTCA/GTTTTCTTAC/ATTTTCTCCA).防御响应元件;TCA-element (CAGAAAAGGA/TCAGAAGAGG/GAGAAGAATA/CCATCTTTTT).水杨酸响应元件;W-box (TTGACC).创伤与病原菌响应元件; WUN-motif (TCATTACGAA/AAATTTCCT).创伤响应元件。
Note:ABRE (TACGTG).Cis-acting element involved in abscisic acid responsiveness; ARE (TGGTTT/AAACCA). Cis-acting regulatory element essential for the anaerobic induction; Aux-core (GGTCCAT). Cis-acting regulatory element involved in auxin responsiveness; CGTCA-motif/TGACG-motif (CGTCA/TGACG). Cis-acting regulatory element involved in MeJA responsiveness; ERE (ATTTCAAA). Ethylene-responsive element; GARE-motif (AAACAGA/TCTGTTG). Gibberellin-responsive element; HSE (GAAAATTCG/AAAAAATTTC/AAAAAATTTC/AAAAAATTTC). Cis-acting element involved in heat stress responsiveness; LTR (CCGAAA). Cis-acting element involved in low-temperature responsiveness; MBS (CGGTCA). MYB binding site; TC-rich repeats (ATTCTCTAAC/ATTTTCTTCA/GTTTTCTTAC/ATTTTCTCCA). Cis-acting element involved in defense and stress responsiveness; TCA-element (CAGAAAAGGA/TCAGAAGAGG/GAGAAGAATA/CCATCTTTTT). Cis-acting element involved in salicylic acid responsiveness; W-box (TTGACC). Cis-acting regulatory element involved in wounding and pathogen responsiveness; WUN-motif (TCATTACGAA/AAATTTCCT). Wound-responsive element.
2.4 小桐子bZIP基因家族染色体定位及复制分析
依据Wu等[15]构建的小桐子高密度遗传连锁图谱,通过基因组Scaffold数据在染色体水平定位每个小桐子bZIP基因,并使用MapChart使信息可视化(图4),结果表明,51个小桐子bZIP基因不均匀地分布于11条染色体上,其中4号染色体上的基因数量最多13个,占小桐子bZIP基因总数的25.5%,11号染色体的基因数最少,仅有1个。基因组及基因片段复制与基因串联复制是基因家族扩张与蛋白质功能多样性演化的重要方式。在小桐子2号与4号染色体鉴定到bZIP基因家族的5对串联复制,如JcbZIP22/JcbZIP31、JcbZIP24/JcbZIP32、JcbZIP25/JcbZIP26、JcbZIP14/JcbZIP20、JcbZIP16/JcbZIP21,另外,小桐子bZIP基因家族在进化中存在11对片段复制现象(图4虚线所示),进一步对其进行进化选择压力分析表明(表4),所有片段复制基因对的Ka/Ks<1,表明这些基因都受纯化选择(Purifying selection),进化分歧时间在108.442 6百万~445.024 6百万年。以上结果暗示,小桐子bZIP基因家族在扩张进化与功能分化过程中片段复制起更主要的作用,与拟南芥[3]、葡萄[11]、番茄[12]中的研究结果一致。同时,共线性分析显示,大量bZIP基因对存在交叉共线性关系,表明小桐子bZIP基因在进化过程中,含有这些bZIP基因的Scaffold或染色体片段发生了基因组倍增(Paleopolyploidy)(图5)。
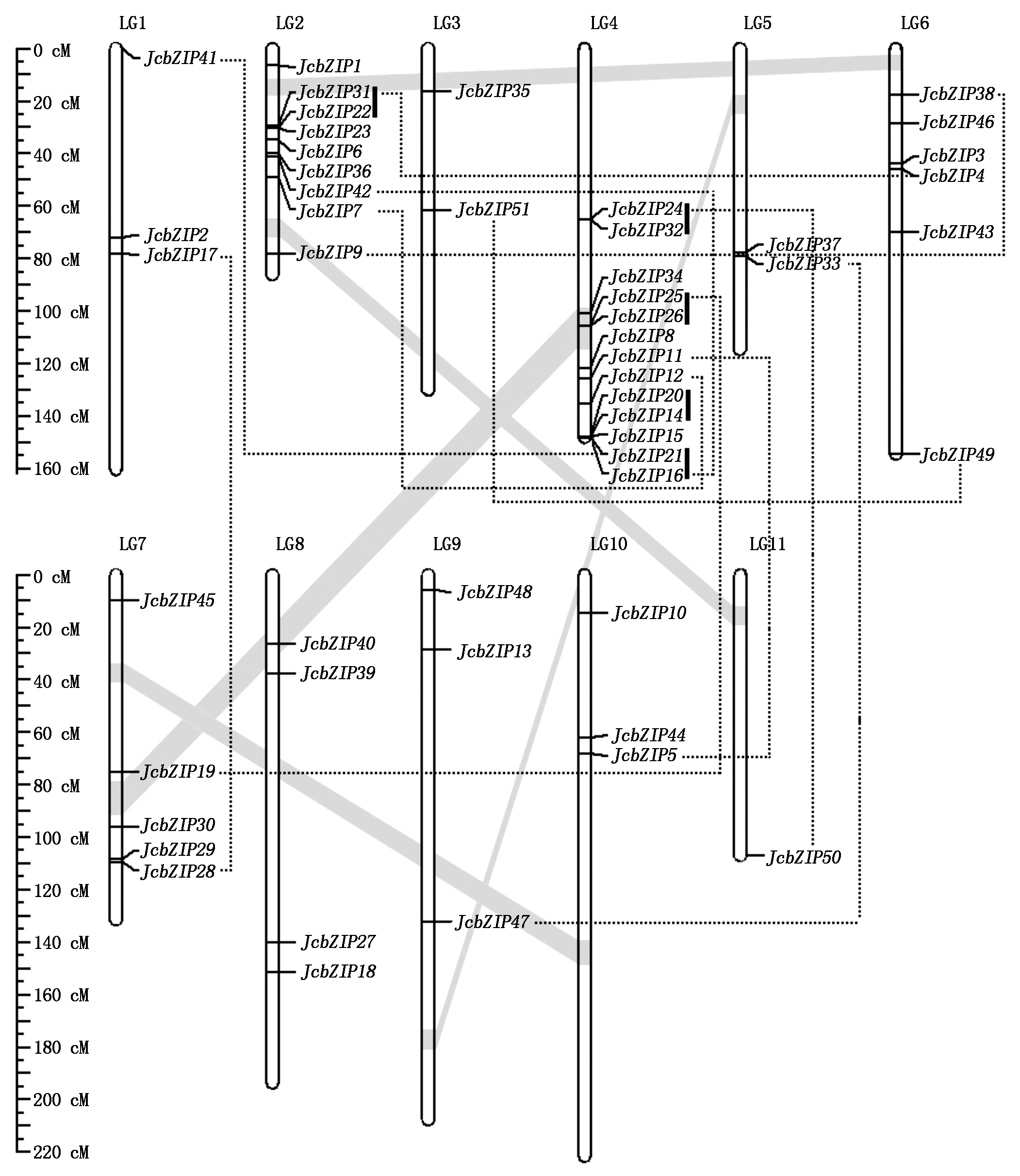
灰色区域表示基因组片段复制,虚线表示基因片段复制,竖线表示基因串联复制;刻度表示厘摩;LG表示染色体。
The gray areas represent genome-wide segmental duplication, the dotted lines indicate ancient segmental duplication, and the vertical black lines indicate tandem duplication based on genome synteny; The scale is in centiMorgans (cM); LG.Chromosome.
图4 小桐子bZIP家族基因的染色体定位
Fig.4 Genomic localization of J.curcas bZIP genes on chromosomes

图5 小桐子bZIP家族基因的共线性分析
Fig.5 Synteny analysis of J.curcas bZIP genes
表4 小桐子bZIP基因家族片段复制基因的进化选择分析
Tab.4 Evolutional selection analysis for ancient segmental duplication genes in J.curcas
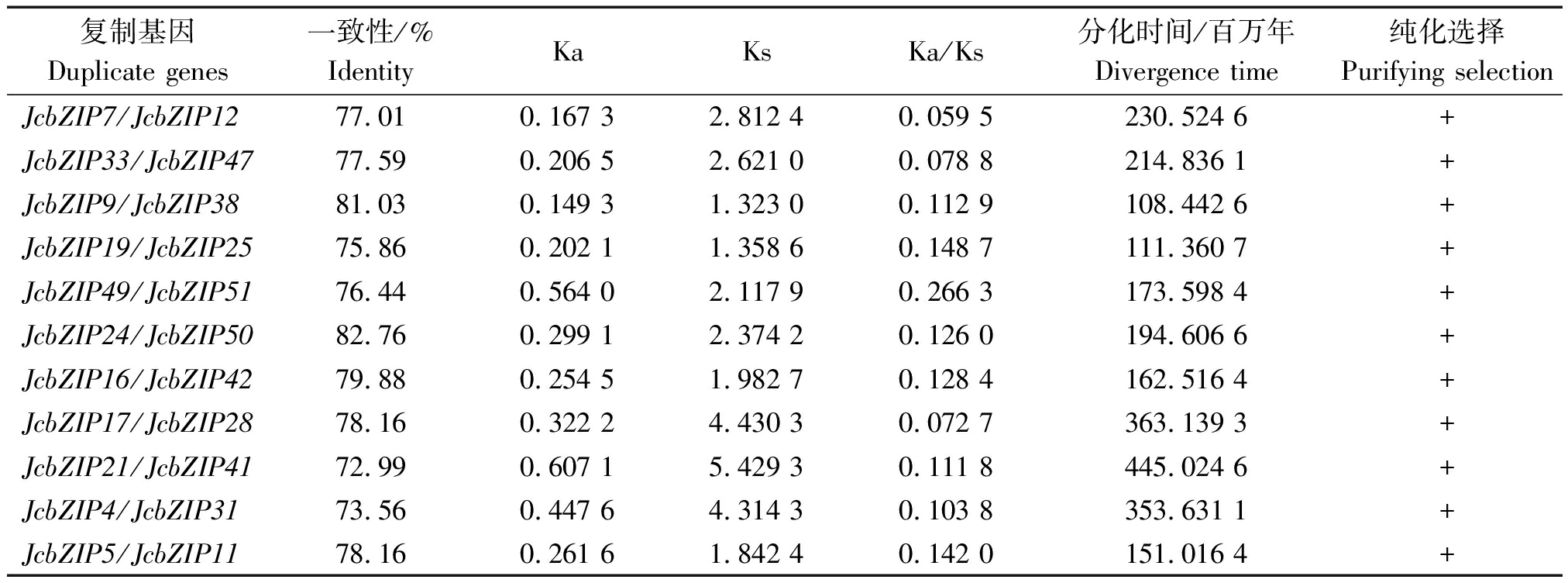
复制基因Duplicate genes一致性/%IdentityKaKsKa/Ks分化时间/百万年Divergence time纯化选择Purifying selectionJcbZIP7/JcbZIP1277.010.167 32.812 40.059 5230.524 6+JcbZIP33/JcbZIP4777.590.206 52.621 00.078 8214.836 1+JcbZIP9/JcbZIP3881.030.149 31.323 00.112 9108.442 6+JcbZIP19/JcbZIP2575.860.202 11.358 60.148 7111.360 7+JcbZIP49/JcbZIP5176.440.564 02.117 90.266 3173.598 4+JcbZIP24/JcbZIP5082.760.299 12.374 20.126 0194.606 6+JcbZIP16/JcbZIP4279.880.254 51.982 70.128 4162.516 4+JcbZIP17/JcbZIP2878.160.322 24.430 30.072 7363.139 3+JcbZIP21/JcbZIP4172.990.607 15.429 30.111 8445.024 6+JcbZIP4/JcbZIP3173.560.447 64.314 30.103 8353.631 1+JcbZIP5/JcbZIP1178.160.261 61.842 40.142 0151.016 4+
注:一致性表示两基因相互匹配部分的相似性;Ka、Ks分别表示非同义突变率与同义突变率;分化时间用Ks/2λ计算,其中λ取值为6.1×10-9 。
Note: Identity indicates the identity of alignment part in two genes; Ka and Ks indicate the number of nonsynonymous substitutions per nonsynonymous site and the number of synonymous substitutions per synonymous site, respectively; Divergence time was calculated by Ks/2λ (λ=6.1×10-9).
2.5 小桐子bZIP家族基因的表达分析
从GenBank的SRA数据库下载小桐子叶片、根及种子的高通量表达测序数据,以FPKM为参数,通过Cufflinks程序得到小桐子51个bZIP基因的表达量,利用R软件对表达数据进行聚类分析及热图绘制(图6-A)。结果表明,有26个bZIP基因在小桐子3种器官中都有表达,其中,JcbZIP7、JcbZIP12、JcbZIP14、JcbZIP39及JcbZIP43的表达量较高,而JcbZIP13、JcbZIP15、JcbZIP23、JcbZIP26、JcbZIP27及JcbZIP48在3种器官中都没有表达。在叶片、根及种子中分别鉴定到有30,42,40个bZIP基因表达,另外,有25个小桐子bZIP基因的表达存在器官特异性,如JcbZIP17、JcbZIP33及JcbZIP46仅在根中有表达;JcbZIP31、JcbZIP32及JcbZIP45仅在种子有表达;JcbZIP4、JcbZIP8、JcbZIP22、JcbZIP24、JcbZIP28、JcbZIP30、JcbZIP37及JcbZIP50在根与种子中表达量较高,而在叶片中表达量较低或不表达;JcbZIP41在叶片与根中表达,而在种子中不表达。通过qRT-PCR试验验证JcbZIP17与JcbZIP33在根中表达量较高,而JcbZIP41在叶片中表达量较高(图7),与测序数据的结果一致。
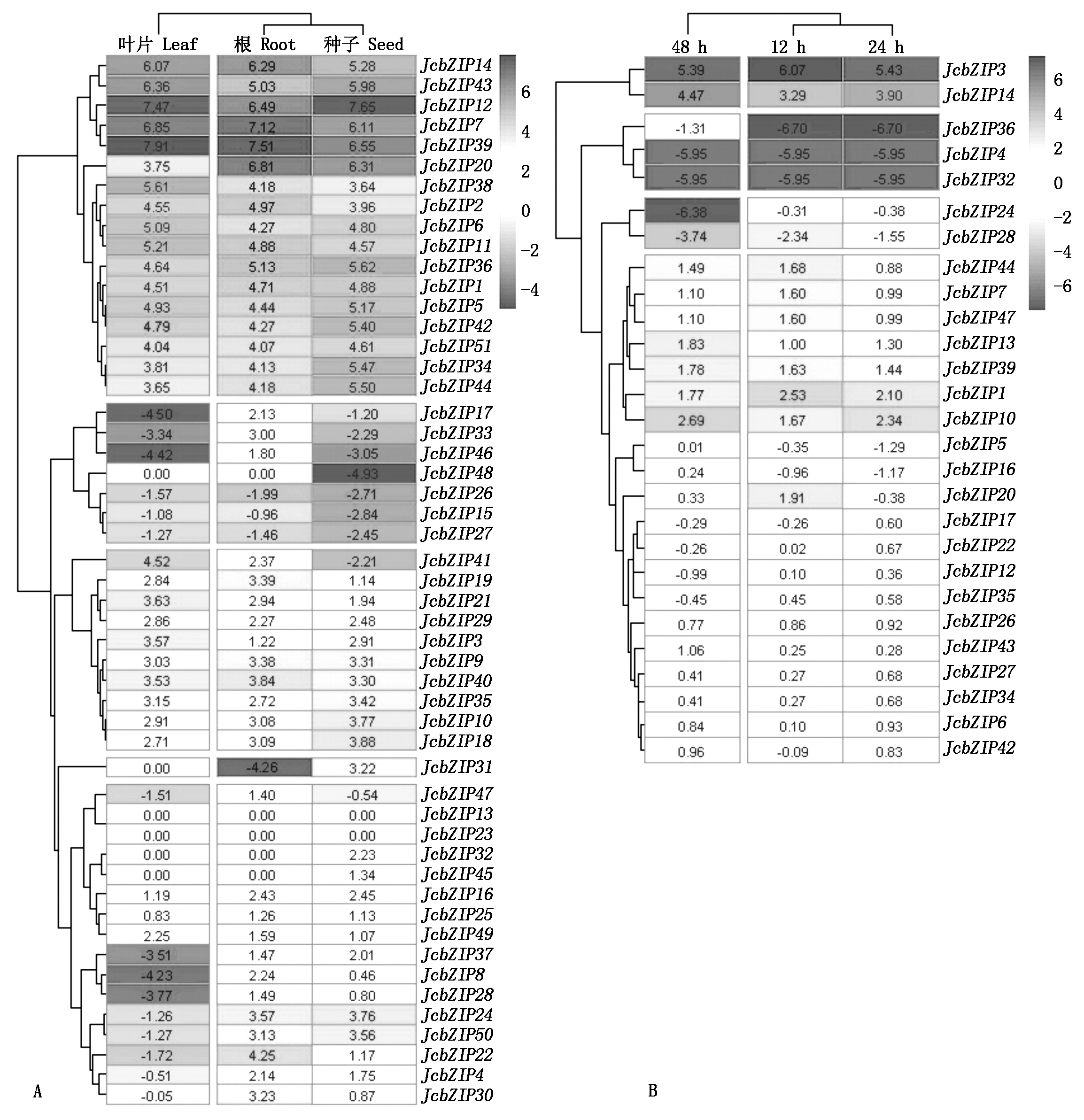
A.器官差异表达; B.低温处理差异表达。
A.Differential expression in different organs; B.Differential expression under chilling hardening.
图6 小桐子bZIP家族基因的表达分析
Fig.6 Differential expression analysis of J.curcas bZIP genes
通过数字基因表达谱数据分析得到27个小桐子bZIP家族基因低温处理下在叶片中的表达数据(图6-B),其中,鉴定到14个bZIP基因在低温条件下上调表达。与对照相比,JcbZIP1、JcbZIP3、JcbZIP7、JcbZIP13、JcbZIP14、JcbZIP39、JcbZIP44及JcbZIP47在低温条件下上调表达显著,而JcbZIP3与JcbZIP14达到了极显著水平(P<0.01),qRT-PCR试验数据也显示两者表达量在低温锻炼24 h时达到最高(图8),与小桐子抗冷性的形成及低温信号转导过程直接相关,也作为抗冷功能基因筛选的候选目标。同时,JcbZIP6、JcbZIP26、JcbZIP27、JcbZIP34及JcbZIP43在低温锻炼的不同时间段都有小量上调表达,但都未达到显著水平,其中,JcbZIP6基因的qRT-PCR试验验证与测序数据吻合(图8)。相反,JcbZIP4、JcbZIP24、JcbZIP28、JcbZIP32及JcbZIP36下调表达明显,其中,JcbZIP36在低温处理12,24 h时较对照都下调表达104.0倍(P<0.01)。另外,对5个bZIP基因在根中的低温表达进行qRT-PCR分析,结果表明,低温处理下,JcbZIP1、JcbZIP3、JcbZIP6及JcbZIP10在低温处理12 h时表达量都有一定上调,但都未达到显著水平,说明在根中其对低温处理不敏感,而JcbZIP14基因随着低温处理时间的延长,表达量持续上调,可能参与小桐子根中低温胁迫的响应过程(图9)。
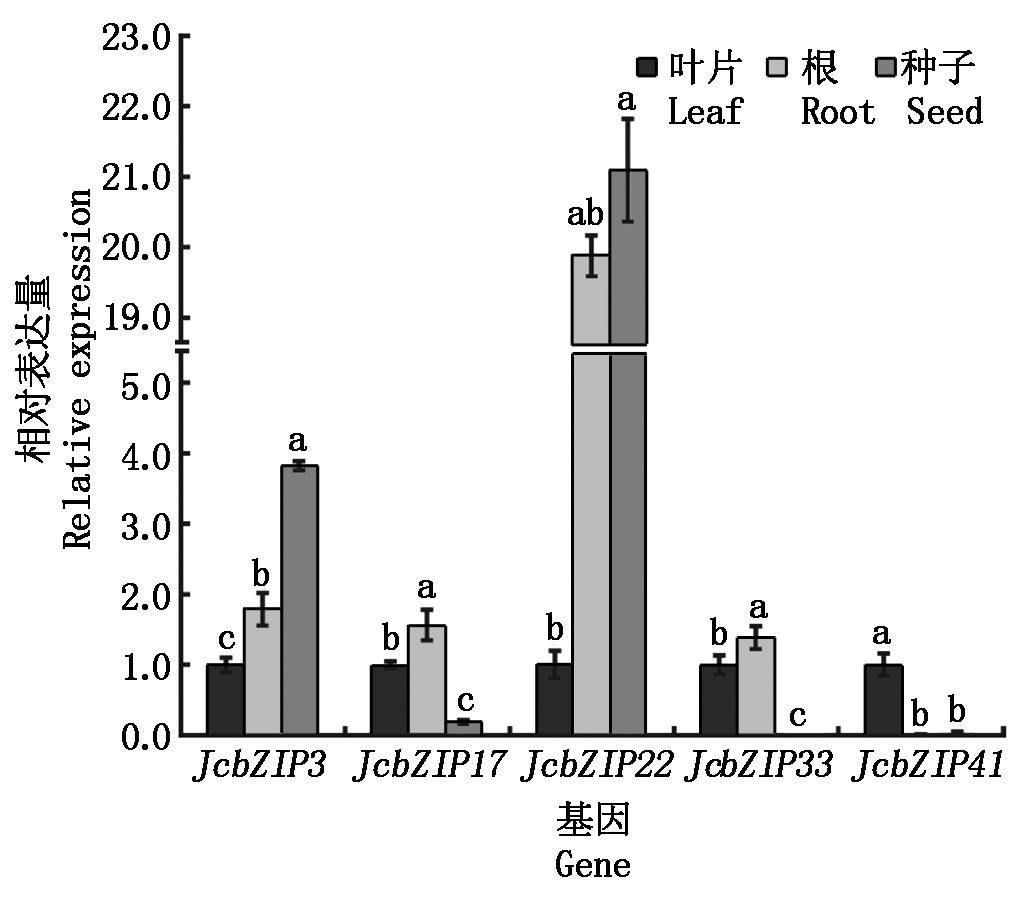
图7 小桐子不同器官中bZIP基因的qRT-PCR差异表达分析
Fig.7 Differential expression analysis of bZIP genes in different organs
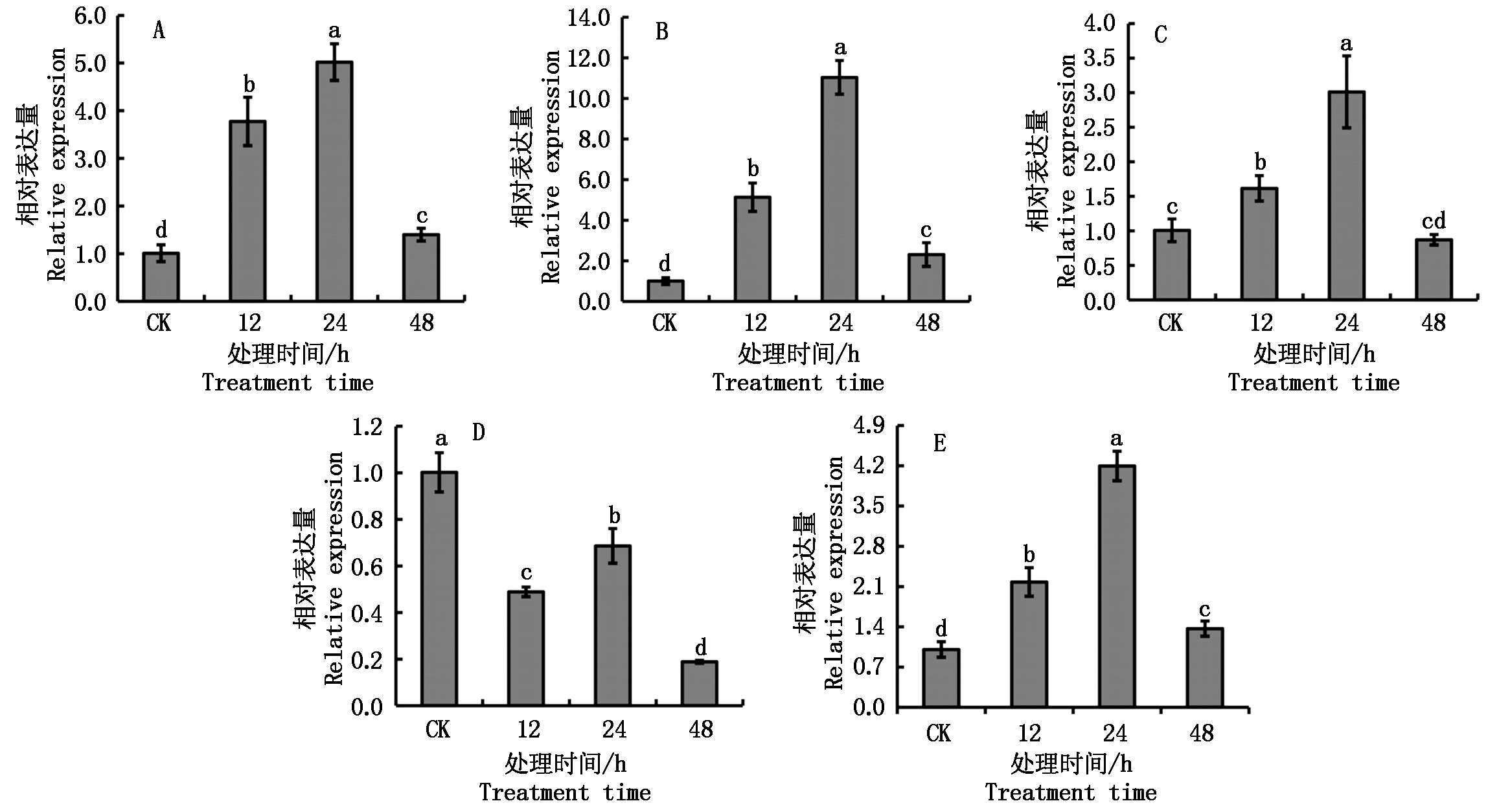
A.JcbZIP1; B.JcbZIP3; C.JcbZIP6; D.JcbZIP10; E.JcbZIP14。图9同。
A.JcbZIP1; B.JcbZIP3; C.JcbZIP6; D.JcbZIP10; E.JcbZIP14.The same as Fig.9.
图8 低温处理下小桐子bZIP基因在叶片中的qRT-PCR表达分析
Fig.8 qRT-PCR relative expression levels of J.curcas bZIP genes in leaves under chilling hardening
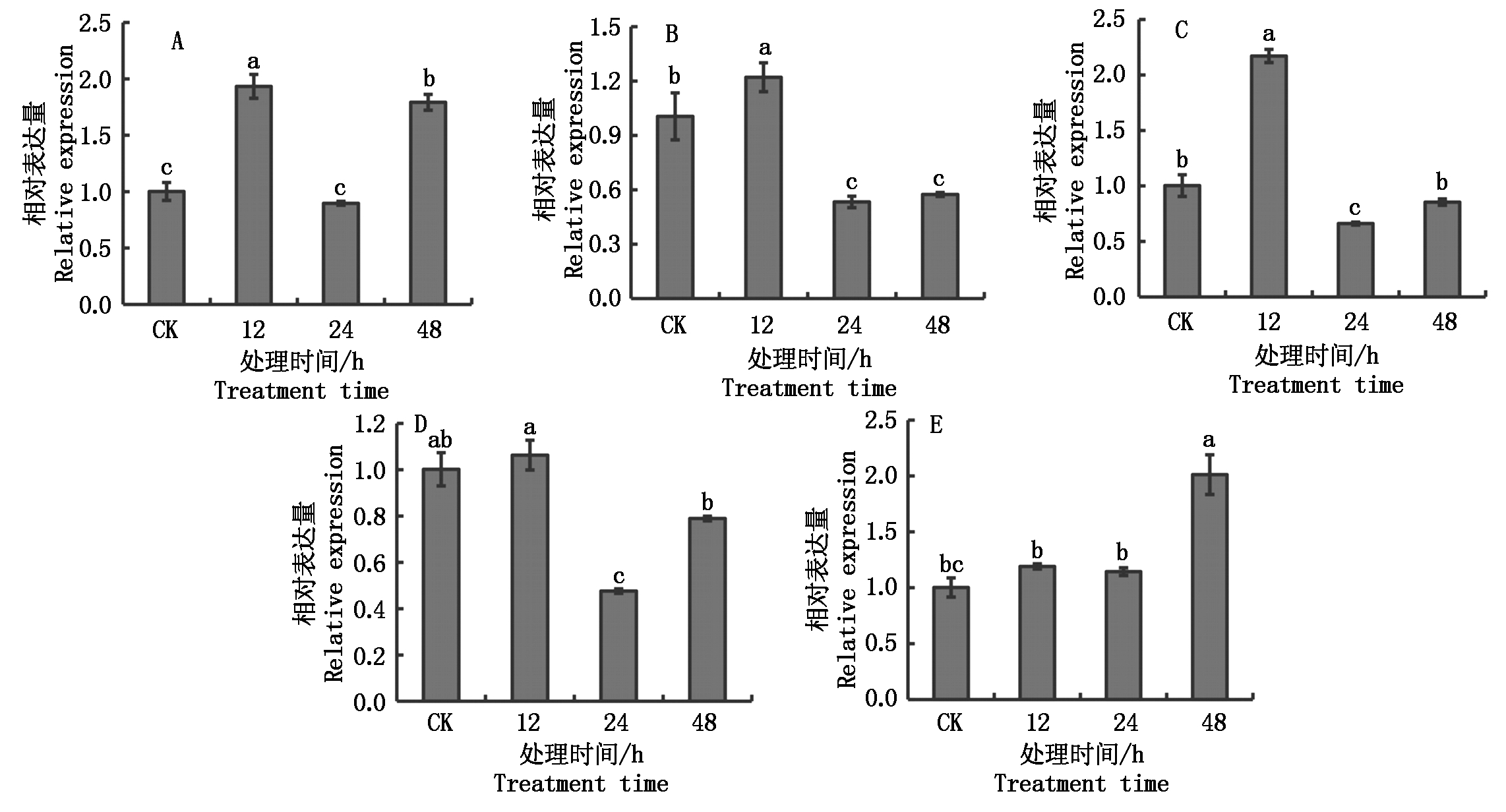
图9 低温处理下小桐子bZIP基因在根中的qRT-PCR表达分析
Fig.9 qRT-PCR relative expression levels of J.curcas bZIP genes in roots under chilling hardening
3 讨论
文献报道,A、B、H及S亚族bZIP基因主要参与植物非生物胁迫响应及激素信号转导过程。其中,A亚族bZIP基因的信号传递主要依赖于ABA信号途径,如拟南芥A亚族的AREB(ABA response element)/ABFs(ABA-responsive element binding proteins)类bZIP转录因子,可激活非生物逆境胁迫下的ABA依赖基因的表达[21]。其中,ABF1主要参与低温、ABA胁迫应答,ABF2与ABF4主要参与干旱、高盐、高温及氧化胁迫应答,而ABF3则受到ABA、低温胁迫的诱导[8,21-22]。在小桐子A亚族中鉴定到8个AREB/ABFs类bZIP转录因子,JcbZIP4、JcbZIP5、JcbZIP18、JcbZIP26、JcbZIP31、JcbZIP34、JcbZIP44及JcbZIP48,且在其bZIP结构域的碱性区域都发现M/KIK与QAY/Q特有基序[8],以上bZIP转录因子通过结合非生物胁迫响应基因启动子区域的ABA响应元件ABRE(ABA response element)从而激活基因的表达,参与小桐子一系列抗逆性响应过程。另外,本研究中鉴定的51个小桐子bZIP基因,有26个基因在其启动子区域发现了ABRE元件,其中JcbZIP3最多有13个ABRE元件,说明部分小桐子bZIP基因的表达也受到ABA信号分子的调控,与拟南芥报道的ABI5转录因子一致[23],综合分析发现,小桐子bZIP基因的JcbZIP18与JcbZIP48同时兼具ABRE元件受到ABA的表达调控,同时也可以结合其他基因的ABRE元件调控相关基因的表达。根据拟南芥bZIP基因的功能验证报道,B亚族bZIP转录因子如拟南芥AtbZIP17(At2g40950)[24]、AtbZIP28(At3g10800)[25-26]可以作为非生物逆境胁迫的感应器和传感器,通过C端锚定在内质网膜上,促进胁迫响应基因的表达,尤其在抗盐胁迫中发挥作用[26],而小桐子中与AtbZIP17及AtbZIP28的同源基因JcbZIP43在其启动子中鉴定到防御相关元件、高温及低温响应元件,同时氨基酸序列的C端二级结构预测以α-螺旋为主,且富含非极性氨基酸残基如Leu、Val,预示JcbZIP43可能通过C端α-螺旋定位在膜结构上,发挥调节胁迫相关基因的表达。另外,拟南芥H亚族的HY5(AtbZIP56、At5g11260)与HYH(AtbZIP64、At3g17609)主要在光形态建成中发挥作用[27],都含有CKII磷酸化位点与WD40作用结构域[3,28-29],另外还可以结合生长素(AUX/IAAs)、乙烯(ERFs)及赤霉素途径的靶基因从而响应激素应答信号[30]。小桐子H亚族也包含2个基因,顺式作用元件分析表明,分别在小桐子HY5同源基因JcbZIP21与HYH同源基因JcbZIP21的启动子序列中发现赤霉素(GARE)与乙烯(ERE)响应元件,说明小桐子H亚族2个基因同样也参与激素信号转导过程。S亚族bZIP转录因子一般成员较多,大部分基因广泛受胁迫处理的激活,如低温等[4-5,31]。进一步分析表明,小桐子51个bZIP基因中有14个在其启动子中鉴定到低温响应元件(LTR),其中,小桐子S亚族bZIP基因中,JcbZIP7、JcbZIP30、JcbZIP33及JcbZIP39包含LTR元件,可能参与小桐子抗冷性形成过程,其余10个小桐子低温响应bZIP基因分别属于A(JcbZIP4、JcbZIP5、JcbZIP23)、B(JcbZIP3、JcbZIP43)、C(JcbZIP20)、D(JcbZIP46)、H(JcbZIP21)及I(JcbZIP24、JcbZIP50)亚族,同时,数字基因表达谱数据也显示,JcbZIP3、JcbZIP7、JcbZIP20、JcbZIP39及JcbZIP43在低温处理下上调表达,与预测结果一致。
同源或异源的2个bZIP结构域形成二聚体,通过碱性区域(Basic region)特异结合下游基因启动子序列从而调控其表达,其核心元件序列为ACGT[32]。部分植物病原相关基因启动子上也存在G-box(CACGTG)或C-box(GACGTC)序列(下划线表示核心元件序列ACGT),功能研究表明,当植物遇到病原浸染时,拟南芥D亚族bZIP转录因子如TGA1(At5g65210)[33]、TGA2/AHBP-1b(At5g06950)[34]、TGA3(At1g22070)[35]、TGA4/OBF4(At5g10030)[36]、TGA5/OBF5(At5g06960)[34]、TGA6(At3g12250)[34]可以结合以上元件,从而诱导病原相关基因(PR)的通体表达,使植物获得系统抗病性(SAR),其中,除bZIP结构域外的DOG1基序对于D亚族bZIP转录因子功能的发挥也是必需的。本研究中7个小桐子D亚族bZIP转录因子的bZIP结构域之后也都鉴定到DOG1基序,包括保守的序列-D/ELRI/L-与-GGFRS/PSELLK/NLL-,另外,小桐子G亚族3个bZIP转录因子JcbZIP29、JcbZIP35、JcbZIP40的bZIP结构域之前都发现了MFMR结构域,该结构域长度150~200个氨基酸,其N端主要促进bZIP转录因子与G-box的结合从而调控光信号感应与传递相关基因的表达,而C端包含富Pro区域(Proline rich domain,PRD)与核定位信号序列,并且3个小桐子G亚族bZIP转录因子都鉴定到该结构域保守序列-P[HP]YMW-、-MMPP/SYGT/AP-及-YAHP-,与拟南芥G亚族同源转录因子GBF1(At4g36730)[37]、GBF2(At4g01120)[38]及GBF3(At2g46270)[39]的蛋白结构与功能验证相符。
[1] Nakashima K, Ito Y, Yamaguchi-Shinozaki K. Transcriptional regulatory networks in response to abiotic stresses in Arabidopsis and grasses [J]. Plant Physiology, 2009, 149(1): 88-95. doi: 10.1104/pp.108.129791.
[2] Amorim L L B, da Fonseca Dos Santos R, Neto J P B, Guida-Santos M, Crovella S, Benko-Iseppon A M. Transcription factors involved in plant resistance to pathogens [J]. Current Protein & Peptide Science, 2017, 18(4): 335-351. doi: 10.2174/1389203717666160619185308.
[3] Jakoby M, Weisshaar B, Dröge-Laser W, Vicente-Carbajosa J, Tiedemann J, Kroj T, Parcy F. bZIP transcription factors in Arabidopsis[J]. Trends in Plant Science, 2002, 7(3): 106-111. doi: 10.1016/S1360-1385(01)02223-3.
[4] Nijhawan A, Jain M, Tyagi A K, Khurana J P. Genomic survey and gene expression analysis of the basic leucine zipper transcription factor family in rice [J]. Plant Physiology, 2008, 146(2): 333-350. doi: 10.1104/pp.107.112821.
[5] Wei K F, Chen J, Wang Y M, Chen Y H, Chen S X, Lin Y N, Pan S, Zhong X J, Xie D X. Genome-wide analysis of bZIP-encoding genes in maize [J]. DNA Research: an International Journal for Rapid Publication of Reports on Genes and Genomes, 2012, 19(6): 463-476. doi: 10.1093/dnares/dss026.
[6] Banerjee A, Roychoudhury A. Abscisic-acid-dependent basic leucine zipper (bZIP) transcription factors in plant abiotic stress [J]. Protoplasma, 2017, 254(1): 3-16. doi: 10.1007/s00709-015-0920-4.
[7] E Z G, Zhang Y P, Zhou J H, Wang L. Mini review roles of the bZIP gene family in rice [J]. Genetics and Molecular Research, 2014, 13(2): 3025-3036. doi: 10.4238/2014.April.16.11.
[8] Noman A, Liu Z P, Aqeel M, Zainab M, Khan M I, Hussain A, Ashraf M F, Li X, Weng Y H, He S L. Basic leucine zipper domain transcription factors: the vanguards in plant immunity [J]. Biotechnology Letters, 2017, 39(12): 1779-1791. doi: 10.1007/s10529-017-2431-1.
[9] Zhang M, Liu Y H, Shi H, Guo M L, Chai M N, He Q, Yan M K, Cao D, Zhao L H, Cai H Y, Qin Y. Evolutionary and expression analyses of soybean basic leucine zipper transcription factor family [J]. BMC Genomics, 2018, 19(1): 159. doi: 10.1186/s12864-018-4511-6.
[10] Wang J Z, Zhou J X, Zhang B L, Vanitha J, Ramachandran S, Jiang S Y. Genome-wide expansion and expression divergence of the basic leucine zipper transcription factors in higher plants with an emphasis on sorghum [J]. Journal of Integrative Plant Biology, 2011, 53(3): 212-231. doi: 10.1111/j.1744-7909.2010.01017.x.
[11] Liu J Y, Chen N N, Chen F, Cai B, Dal Santo S, Tornielli G B, Pezzotti M, Cheng Z M. Genome-wide analysis and expression profile of the bZIP transcription factor gene family in grapevine (Vitis vinifera) [J]. BMC Genomics, 2014, 15(8): 281. doi: 10.1186/1471-2164-15-281.
[12] 张珍珠, 陈秀玲, 王沛文, 戚飞, 谢莹, 王傲雪. 番茄bZIP基因家族的系统进化分析 [J]. 东北农业大学学报, 2014, 45(9): 47-55. doi: 10.3969/j.issn.1005-9369.2014.09.007.s.
Zhang Z Z, Chen X L, Wang P W, Qi F, Xie Y, Wang A X. Phyletic evolution analysis of bZIP family in tomato [J]. Journal of Northeast Agricultural University, 2014, 45(9): 47-55.
[13] 孙明岳, 周君, 谭秋平, 付喜玲, 陈修德, 李玲, 高东升. 苹果bZIP转录因子家族生物信息学分析及其在休眠芽中的表达[J]. 中国农业科学, 2016, 49(7): 1325-1345. doi: 10.3864/j.issn.0578-1752. 2016.07.010.
Sun M Y, Zhou J, Tan Q P, Fu X L, Chen X D, Li L, Gao D S. Analysis of basic leucine zipper genes and their expression during bud dormancy in apple (Malus×domestica) [J]. Scientia Agricultural Sinica, 2016, 49(7): 1325-1345.
[14] Sato S, Hirakawa H, Isobe S, Fukai E, Watanabe A, Kato M, Kawashima K, Minami C, Muraki A, Nakazaki N, Takahashi C, Nakayama S, Kishida Y, Kohara M, Yamada M, Tsuruoka H, Sasamoto S, Tabata S, Aizu T, Toyoda A, Shini T, Minakuchi Y, Kohara Y, Fujiyama A, Tsuchimoto S, Kajiyama S, Makigano E, Ohmido N, Shibagaki N, Cartagena J A, Wada N, Kohinata T, Atefeh A, Yuasa S, Matsunaga S, Fukui K. Sequence analysis of the genome of an oil-bearing tree, Jatropha curcas L. [J]. DNA Research: an International Journal for Rapid Publication of Reports on Genes and Genomes, 2011, 18(1): 65-76. doi: 10.1093/dnares/dsq030.
[15] Wu P Z, Zhou C P, Cheng S F, Wu Z Y, Lu W J, Han J L, Chen Y B, Chen Y, Ni P X, Wang Y, Xu X, Huang Y, Song C, Wang Z W, Shi N, Zhang X D, Fang X H, Yang Q, Jiang H W, Chen Y P, Li M R, Wang Y, Chen F, Wang J, Wu G J. Integrated genome sequence and linkage map of physic nut (Jatropha curcas L.), a biodiesel plant [J]. The Plant Journal : for Cell and Molecular Biology, 2015, 81(5): 810-821. doi: 10.1111/tpj.12761.
[16] Suyama M, Torrents D, Bork P. PAL2NAL: robust conversion of protein sequence alignments into the corresponding codon alignments [J]. Nucleic Acids Research, 2006, 34(SI): W609-W612. doi: 10.1093/nar/gkl315.
[17] Wang H B, Zou Z R, Wang S S, Gong M. Deep sequencing-based transcriptome analysis of the oil-bearing plant Physic Nut (Jatropha curcas L.) under cold treatments [J]. Plant Omics, 2014, 7(3): 178-187.
[18] Wang H B, Zou Z R, Wang S S, Gong M. Global analysis of transcriptome responses and gene expression profiles to cold stress of Jatropha curcas L. [J]. PLoS One, 2013, 8(12): e82817. doi: 10.1371/journal.pone.0082817.
[19] tHoen P A, Ariyurek Y, Thygesen H H, Vreugdenhil E, Vossen R H, de Menezes R X, Boer J M, van Ommen G J, den Dunnen J T. Deep sequencing-based expression analysis shows major advances in robustness, resolution and inter-lab portability over five microarray platforms [J]. Nucleic Acids Research, 2008, 36(21): e141. doi: 10.1093/nar/gkn705.
[20] Morrissy A S, Morin R D, Delaney A, Zeng T, McDonald H, Jones S, Zhao Y, Hirst M, Marra M A. Next-generation tag sequencing for cancer gene expression profiling [J]. Genome Research, 2009, 19(10): 1825-1835. doi: 10.1101/gr.094482.109.
[21] Bai Y L, Zhu W B, Hu X C, Sun C C, Li Y L, Wang D D, Wang Q H, Pei G L, Zhang Y F, Guo A G, Zhao H X, Lu H B, Mu X Q, Hu J J, Zhou X N, Xie C G. Genome-wide analysis of the bZIP gene family identifies two ABI5-Like bZIP transcription factors, BrABI5a and BrABI5b, as positive modulators of ABA signalling in Chinese cabbage [J]. PLoS One, 2016, 11(7): e0158966. doi: 10.1371/journal.pone.0158966.
[22] Uno Y, Furihata T, Abe H, Yoshida R, Shinozaki K, Yamaguchi-Shinozaki K. Arabidopsis basic leucine zipper transcription factors involved in an abscisic acid-dependent signal transduction pathway under drought and high-salinity conditions [J]. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(21): 11632-11637. doi: 10.1073/pnas.190309197.
[23] Finkelstein R R, Lynch T J. The Arabidopsis abscisic acid response gene ABI5 encodes a basic leucine zipper transcription factor [J]. The Plant Cell, 2000, 12(4): 599-609. doi: 10.2307/3871072.
[24] Liu J X, Srivastava R, Howell S H. Stress-induced expression of an activated form of AtbZIP17 provides protection from salt stress in Arabidopsis[J]. Plant, Cell & Environment, 2008, 31(12): 1735-1743. doi: 10.1111/j.1365-3040.2008.01873.x.
[25] Srivastava R, Deng Y, Shah S, Rao A G, Howell S H. BINDING PROTEIN is a master regulator of the endoplasmic reticulum stress sensor/transducer bZIP28 in Arabidopsis[J]. Plant Cell, 2013, 25(4): 1416-1429. doi: 10.1105/tpc.113.110684.
[26] Liu J X, Srivastava R, Che P, Howell S H. An endoplasmic reticulum stress response in Arabidopsis is mediated by proteolytic processing and nuclear relocation of a membrane-associated transcription factor, bZIP28 [J]. Plant Cell, 2007, 19(12): 4111-4119. doi: 10.1105/tpc.106.050021.
[27] Lee J, He K, Stolc V, Lee H, Figueroa P, Gao Y, Tongprasit W, Zhao H Y, Lee I, Deng X W. Analysis of transcription factor HY5 genomic binding sites revealed its hierarchical role in light regulation of development [J]. Plant Cell, 2007, 19(3): 731-749. doi: 10.1105/tpc.106.047688.
[28] Zhang Y Q, Zheng S, Liu Z J, Wang L G, Bi Y R. Both HY5 and HYH are necessary regulators for low temperature-induced anthocyanin accumulation in Arabidopsis seedlings [J]. Journal of Plant Physiology, 2011, 168(4): 367-374. doi: 10.1016/j.jplph.2010.07.025.
[29] Singh A, Ram H, Abbas N, Chattopadhyay S. Molecular interactions of GBF1 with HY5 and HYH proteins during light-mediated seedling development in Arabidopsis thaliana[J]. The Journal of Biological Chemistry, 2012, 287(31): 25995-26009. doi: 10.1074/jbc.M111.333906.
[30] Gangappa S N, Botto J F. The multifaceted roles of HY5 in plant growth and development [J]. Molecular Plant, 2016, 9(10): 1353-1365. doi: 10.1016/j.molp.2016.07.002.
[31] Kusano T, Berberich T, Harada M, Suzuki N, Sugawara K. A maize DNA-binding factor with a bZIP motif is induced by low temperature [J]. Molecular & General Genetics, 1995, 248(5): 507-517. doi: 10.1007/bf02423445.
[32] Izawa T, Foster R, Chua N H. Plant bZIP protein DNA binding specificity [J]. Journal of Molecular Biology, 1993, 230(4): 1131-1144. doi: 10.1006/jmbi.1993.1230.
[33] Schindler U, Beckmann H, Cashmore A R. TGA1 and G-box binding factors: two distinct classes of Arabidopsis leucine zipper protiens compete for the G-box-like element TGACGTGG [J]. Plant Cell, 1992, 4(10): 1309-1319. doi: 10.2307/3869416.
[34] Zhang Y L, Tessaro M J, Lassner M, Li X. Knockout analysis of Arabidopsis transcription factors TGA2, TGA5, and TGA6 reveals their redundant and essential roles in systemic acquired resistance [J]. Plant Cell, 2003, 15(11): 2647-2653. doi: 10.1105/tpc.014894.
[35] Miao Z H, Liu X, Lam E. TGA3 is a distinct member of the TGA family of bZIP transcription factors in Arabidopsis thaliana[J]. Plant Molecular Biology, 1994, 25(1): 1-11. doi: 10.1007/bf00024193.
[36] Foley R C, Singh K B. TGA5 acts as a positive and TGA4 acts as a negative regulator of ocs element activity in Arabidopsis roots in response to defence signals [J]. FEBS Letters, 2004, 563(1/3): 141-145. doi: 10.1016/S0014-5793(04)00288-1.
[37] Mallappa C, Yadav V, Negi P, Chattopadhyay S. A basic leucine zipper transcription factor, G-box-binding factor 1, regulates blue light-mediated photomorphogenic growth in Arabidopsis[J]. The Journal of Biological Chemistry, 2006, 281(31): 22190-22199. doi: 10.1074/jbc.M601172200.
[38] Terzaghi W B, Bertekap R L Jr, Cashmore A R. Intracellular localization of GBF proteins and blue light-induced import of GBF2 fusion proteins into the nucleus of cultured Arabidopsis and soybean cells [J]. The Plant Journal: for Cell and Molecular Biology, 1997, 11(5): 967-982. doi: 10.1046/j.1365-313X. 1997.11050967.x.
[39] Menkens A E, Schindler U, Cashmore A R. The G-box: a ubiquitous regulatory DNA element in plants bound by the GBF family of bZIP proteins [J]. Trends in Biochemical Sciences, 1995, 20(12): 506-510. doi: 10.1016/S0968-0004(00)89118-5.