乙烯作为一种重要植物激素参与多个植物生长发育过程和应答胁迫反应[1-2]。研究发现,乙烯合成存在2种系统,系统Ⅰ和系统Ⅱ[3]。系统Ⅰ完成基础乙烯合成,主要存在于未成熟果实和营养生长的器官中,通过自身抑制的方式进行调控;而系统Ⅱ参与果实成熟和花衰老时乙烯的大量合成,通过自身催化的方式调控[4-6]。在番茄中,系统Ⅰ和系统Ⅱ由不同的ACO、ACS家族成员参与[4,7-8]。
乙烯合成关键酶ACO和ACS基因受到转录因子的调控。Lin等[9]发现1个亮氨酸拉链蛋白的同源蛋白LeHB1结合在番茄LeACO1基因启动子9 bp 的AATA(A)TATT或10 bp 的AATA(AA)TATT特定序列上,对该基因进行转录水平调节。通过病毒介导的基因沉默技术,当LeHB1基因下调后,LeACO1基因的表达受到了抑制,果实的成熟也被延迟。Ito等[10]通过染色质免疫沉淀PCR检测发现,番茄的1个MADS-box蛋白LeMADS-RIN(RIN)会特异性地结合到LeACS2启动子的CArG box区域。rin隐性突变的番茄果实成熟发育出现障碍,致使该植株既无法大量产生内源性乙烯,又无法对外源乙烯做出应答,导致果实无法成熟[11]。RIN被认为是乙烯生物合成与调控中重要的上游调节蛋白。
乙烯进入细胞后,通过信号转导途径来调节植物各种生理活动。乙烯受体位于内质网膜上,它首先感受乙烯信号,目前已经鉴定的拟南芥乙烯受体共5个,即ETR1、 ETR2、 ERS1、ERS2、EIN4[12]。乙烯受体的下游组分CTR1也位于内质网膜上,与乙烯受体相互作用[13]。CTR1的C末端具有Ser/Thr激酶活性,而N末端可以与自磷酸化的乙烯受体如ETR1的组氨酸激酶域形成复合物,从而起到负调控乙烯信号转导的作用[14]。
本研究在甜瓜全基因组范围内鉴定了乙烯生物合成及信号转导相关的4个基因家族HB(Homeo box)、RIN(Ripening inhibitor)、ETR(Ethylene receptor)和CTR(Constitutive triple reaction),并进行了生物信息学分析。以甜瓜品种河套蜜瓜原种和内蒙古大学牧草与特色作物生物技术教育部重点实验室培育的转反义CmACO1基因的河套蜜瓜M9品系[15]为试验材料,分析了上述基因在果实生长期和成熟期的表达特性,旨在为发掘与甜瓜果实成熟相关的基因。
1 材料和方法
1.1 植物材料
甜瓜品种河套蜜瓜原种由内蒙古大学牧草与特色作物生物技术教育部重点实验室保存,转反义CmACO1基因的河套蜜瓜M9品系(以下简称M9)由内蒙古大学牧草与特色作物生物技术教育部重点实验室培育[14]。将2种材料播种于日光温室,按常规方法进行栽培管理,盛花期人工自交授粉。采摘授粉后10 d(以下简称生长期)和30 d果实(以下简称成熟期)(图1),每个时间点每个材料采摘3颗果实,取中果皮组织,速冻于液氮,用于总RNA提取,进行转录组测序分析。
1.2 四个基因家族的全基因组鉴定
根据NCBI数据库中已确认功能的基因序列,找到番茄LeETR1、LeMADS-RIN1、LeHB1基因和拟南芥AtCTR1基因作为探针基因,并查找各自对应的蛋白质序列(表1),下载FASTA格式的序列文件。然后将该FASTA文件输入InterPro数据库进行分析,将探针基因中所有功能域所在区域的序列作为探针序列。再将探针序列分别输入甜瓜基因组数据库(MELONOMICS)的Blast工具中,获得具有与目标功能域序列高度相似(E<10-10)序列的蛋白质登录号并下载其FASTA格式文件。最后将甜瓜数据库中筛选到的序列输入InterPro数据库中验证,剔除功能域不完整的序列,获得相应的基因家族成员。

A.M9生长期果实;B.河套蜜瓜原种生长期果实;C.M9成熟期果实;D.河套蜜瓜原种成熟期果实。
A.M9 fruit at the growth stage; B.Hetao melon fruit at the growth stage; C.M9 fruit at the ripening stage; D.Hetao melon fruit at the ripening stage.
图1 M9和河套蜜瓜原种不同发育时期的果实
Fig.1 Fruits of M9 and Hetao melons at different development stages
表1 甜瓜基因家族鉴定探针基因信息
Tab.1 Information of probe genes for gene family identification in melon
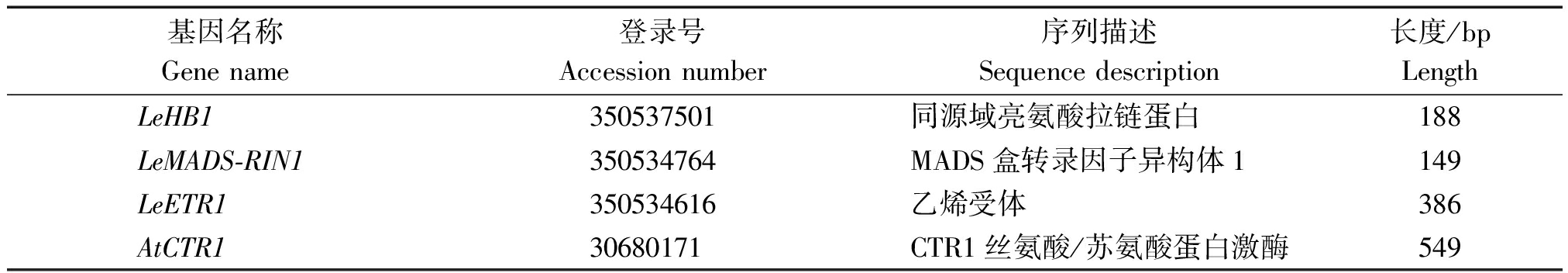
基因名称Gene name登录号Accession number序列描述Sequence description长度/bpLengthLeHB1350537501同源域亮氨酸拉链蛋白188LeMADS-RIN1350534764MADS盒转录因子异构体1149LeETR1350534616乙烯受体386AtCTR130680171CTR1丝氨酸/苏氨酸蛋白激酶549
1.3 基因编码蛋白的生物信息学分析
将鉴定获得的蛋白序列使用DNAMAN 8.0软件进行比对,获得保守序列。使用MEME(http://meme-suite.org/tools/meme)在线软件预测各基因家族蛋白序列的保守基序。
1.4 总RNA提取及转录组测序
使用康为世纪公司TRIzol总RNA提取试剂盒,按照说明书提取总RNA。对总RNA进行电泳检测和纯度检测后交由北京诺禾致源科技股份有限公司完成转录组测序。
1.5 基因的表达量分析
从转录组测序结果中提取4个基因家族成员的表达量FPKM值(Fragments per kilobase of transcript per milion fragment mapped),使用R 语言工具的 Pheatmap 拓展包绘制聚类热图(Heatmap)。筛选高表达基因,使用SPSS 22.0版本进行方差分析,获得基因差异表达显著性水平。
2 结果与分析
2.1 四个基因家族的全基因组鉴定
对4个基因在甜瓜中进行全基因组鉴定发现,CmETR家族有3个成员、CmCTR家族有20个成员,CmHB家族有17个成员,CmRIN家族有21个成员(表2)。
2.2 四个基因家族编码蛋白氨基酸序列分析
对4个家族成员的序列进行比对,发现不同家族的氨基酸序列均存在较多的保守位点,序列相似性较高,说明基因家族鉴定结果是可靠的。CmETR家族的3个成员序列相似度较高,保守序列位点分布的位置较广(图2-A),这暗示该家族可能存在多个保守基序。进一步对该家族保守基序分析发现,基序1,2,3,4的保守性较高(图3-A),这与序列比对结果具有一致性。蛋白质的低级结构决定高级结构,高级结构决定其功能。该家族成员具有较高的位点保守性,暗示其在甜瓜中的功能也可能非常相近。CmCTR家族保守序列集中在2个位置(图2-B),对其保守基序分析发现,基序1,2的保守性最高(图3-B),这2个保守基序可能是该家族基因共有的功能位点。CmHB和CmRIN两家族的保守序列相对集中(图2-C-D),分别对2个家族的保守基序分析发现,CmHB的基序5(图3-C)和CmRIN的基序6(图3-D)都是非常保守,是这2个家族成员的典型特征序列。
表2 四个基因家族成员鉴定结果
Tab.2 The identification of four gene families

基因家族名称Gene family name基因名称Gene name登录号Accession number基因家族名称Gene family name基因名称Gene name登录号Accession numberCmCTRCmCTR1MELO3C002125T1CmHB12MELO3C013923T1CmCTR2MELO3C002407T1CmHB13MELO3C014944T1CmCTR3MELO3C003518T1CmHB14MELO3C021534T1CmCTR4MELO3C003722T1CmHB15MELO3C021978T1CmCTR5MELO3C004081T1CmHB16MELO3C023514T1CmCTR6MELO3C006335T1CmHB17MELO3C024507T1CmCTR7MELO3C008367T1CmETRCmETR1MELO3C003906T1CmCTR8MELO3C009433T1CmETR2MELO3C006451T1CmCTR9MELO3C010263T1CmETR3MELO3C015961T1CmCTR10MELO3C013322T1CmRINCmRIN1MELO3C002049T1CmCTR11MELO3C013322T2CmRIN2MELO3C002050T1CmCTR12MELO3C013571T1CmRIN3MELO3C002691T1CmCTR13MELO3C013739T1CmRIN4MELO3C003502T1CmCTR14MELO3C013739T2CmRIN5MELO3C005617T2CmCTR15MELO3C015389T1CmRIN6MELO3C006159T4CmCTR16MELO3C016868T2CmRIN7MELO3C007181T1CmCTR17MELO3C019637T1CmRIN8MELO3C007181T2CmCTR18MELO3C021267T1CmRIN9MELO3C007181T3CmCTR19MELO3C021267T2CmRIN10MELO3C011093T1CmCTR20MELO3C024518T1CmRIN11MELO3C011093T3CmHBCmHB1MELO3C002209T1CmRIN12MELO3C011409T1CmHB2MELO3C002209T2CmRIN13MELO3C018049T1CmHB3MELO3C003554T1CmRIN14MELO3C022209T1CmHB4MELO3C005992T1CmRIN15MELO3C022316T1CmHB5MELO3C007736T1CmRIN16MELO3C022516T1CmHB6MELO3C007736T2CmRIN17MELO3C022516T2CmHB7MELO3C009420T1CmRIN18MELO3C024001T1CmHB8MELO3C009948T1CmRIN19MELO3C026300T1CmHB9MELO3C009948T2CmRIN20MELO3C026300T2CmHB10MELO3C012064T1CmRIN21MELO3C026300T3CmHB11MELO3C012079T1


A.CmETR家族氨基酸序列比对结果;B.CmCTR家族氨基酸序列比对结果;C.CmHB家族氨基酸序列比对结果;D.CmRIN家族氨基酸序列比对结果。
A.Sequence alignment of CmETR family; B.Sequence alignment of CmCTR family; C.Sequence alignment of CmHB family; D.Sequence alignment of CmRIN family.
图2 不同基因家族蛋白氨基酸序列比对结果
Fig.2 Sequence alignment of different gene families proteins

A.CmETR家族氨基酸序列比对分析结果;B.CmCTR家族氨基酸序列比对分析结果;C.CmHB家族氨基酸序列比对分析结果;D.CmRIN家族氨基酸序列比对分析结果。
A.Motifs analysis of CmETR family; B.Motifs analysis of CmCTR family; C.Motifs analysis of CmHB family; D.Motifs analysis of CmRIN family.
图3 不同基因家族基序分析结果
Fig.3 Motifs analysis of different gene families
2.3 四个基因家族在不同发育时期果实中的表达分析
对4个基因家族的表达量聚类结果(图4)分析发现,CmETR、CmCTR和CmHB 3个家族成员在2个材料(M9和河套蜜瓜原种)中的表达量聚为两类,即生长期的表达量聚为一类,成熟期的表达量聚为一类;而CmRIN家族的表达量分为3类,2个材料成熟期的表达量聚为一类,生长期的河套蜜瓜原种和生长期的M9的表达量各为一类。
CmETR家族3个成员中CmETR1基因在2个材料中的2个时期均有较高水平的表达,CmETR2和CmETR3则均表现出基因下调趋势。

A.CmETR家族成员表达量热图;B.CmCTR家族成员表达量热图;C.CmHB家族成员表达量热图;D.CmRIN家族成员表达量热图;DF_M_group_fpkm.M9生长期果实;DF_C_group_fpkm.河套蜜瓜原种生长期果实;RF_M_group_fpkm.M9成熟期果实;RF_C_group_fpkm.河套蜜瓜原种成熟期果实。
A.Heatmap of CmETR family expression; B.Heatmap of CmCTR family expression; C.Heatmap of CmHB family expression; D.Heatmap of CmRIN family expression; DF_M_group_fpkm.M9 fruit at the growth stage; DF_C_group_fpkm.Hetao melon fruit at the growth stage; RF_M_group_fpkm.M9 fruit at ripening stage; RF_C_group_fpkm.Hetao melon fruit at ripening stage.
图4 四个基因家族在不同发育时期果实中表达量的热图
Fig.4 Heatmap of the four gene families expression in different fruits
CmCTR的20个成员中CmCTR1、CmCTR12在原种和M9的生长期果实中的表达水平均略高于成熟期,CmCTR15则具有相反表达模式。CmCTR8和CmCTR20在2个材料的果实中均有表达,在生长期和成熟期的表达量没有明显变化。CmCTR2在M9的成熟期果实中的表达水平略高于河套蜜瓜原种。CmCTR17和CmCTR5在河套蜜瓜原种中下调,而在M9材料中表达上调,但需要进行统计分析。CmCTR7在河套蜜瓜原种与M9中2个时期的表达趋势变化相同,均呈现上调。
CmHB家族17个成员中CmHB4和CmHB9的表达在2个材料中均表现出上调,其中CmHB4在生长期几乎不表达,在成熟期果实中则大量表达。CmHB12和CmHB15则与CmHB4表达模式相反,其在2个材料中均表现下调。CmHB3在2个材料的成熟期果实中均有微量表达。CmHB5在2个材料的生长期果实中均有较高水平的表达。CmHB11在2个材料中均明显表现下调模式。CmHB13在河套蜜瓜原种的2个时期表达水平均很低。
CmRIN家族的21成员中CmRIN2在2个材料的表达量表现出一致的下调模式,但其表达水平较低。CmRIN3在不同发育时期果实中的表达水平整体较高且表现出上调。CmRIN14和CmRIN15在2个材料成熟期果实中的表达水平相近,但在不同材料中的表达模式相反。CmRIN18的表达水平整体较低。
2.4 四个家族基因表达差异分析
筛选FPKM值大于1的所有基因,对其不同发育时期果实中的表达量进行方差分析,由图5可看出,河套蜜瓜原种2个时期显著差异表达的基因有13个,除了CmHB5以外,其余12个基因的表达量在M9的2个时期也呈显著性差异。这12个基因分别是:CmETR2、CmETR3、CmCTR12、CmHB3、CmHB4、CmHB9、CmHB11、CmHB12、CmHB15、CmRIN2、CmRIN14、CmRIN15。进一步分析发现,在河套蜜瓜原种中CmHB3和CmHB11生长期的表达量是成熟期的42,9倍,而CmHB4成熟期的表达量是生长期的27倍,其表达量均呈极显著差异;在M9中CmHB3和CmHB11生长期的表达量是成熟期表达量的6,3倍,而CmHB4成熟期的表达量是生长期的41倍,其表达量均呈极显著差异。
另外发现,在生长期2个材料间有5个基因呈显著性差异表达,其中除了CmHB11基因外,其余3个基因的表达量在成熟期的2个材料间也呈显著性差异,这3个基因分别是CmHB3、CmRIN14、CmRIN15,其中CmHB3、CmHB4的表达量呈极显著差异。
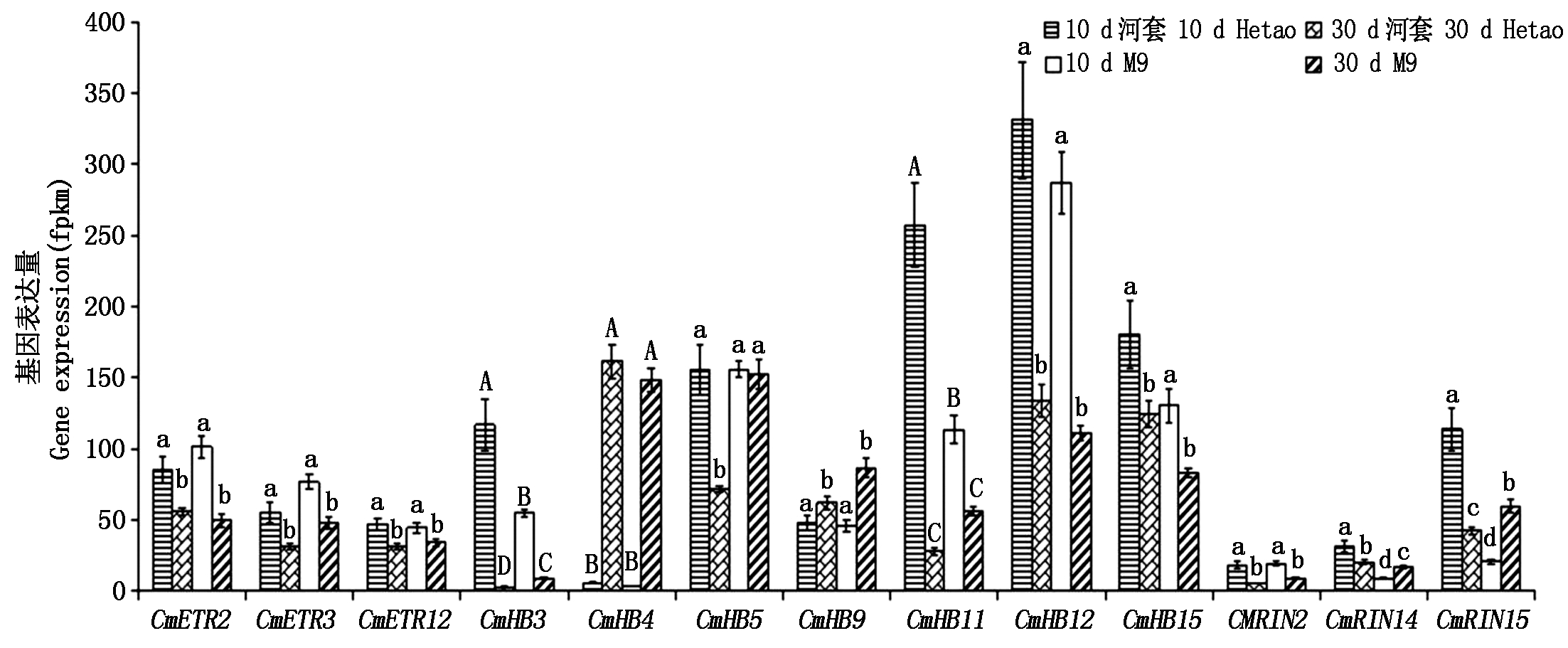
10 d河套.河套蜜瓜原种生长期的果实;30 d河套.河套蜜瓜原种成熟期的果实;10 d M9.M9生长期的果实;30 d M9.M9成熟期的果实;不同小写字母a,b,c,d代表存在显著性差异;不同大写字母A,B,C,D代表存在极显著差异;字母相同则无差异。
10 d Hetao.Hetao melon fruit at growth stage; 30 d Hetao.Hetao melon fruit at ripening stage; 10 d M9.M9 fruit at growth stage; 30 d M9.M9 fruit at ripening stage; There was significant difference between a, b, c, d; There was highly significant difference between A, B, C, D; The same letters means no difference.
图5 基因表达差异性分析
Fig.5 Difference analysis of gene expression
3 结论与讨论
果实成熟的众多调控因子中,乙烯被认为是最关键的影响因子[16]。果实成熟时包括细胞壁代谢、类胡萝卜素合成、叶绿素降解、芳香物质、糖和有机酸合成等多个代谢途径的相关基因都受乙烯调控[17]。乙烯通过信号转导来调控植物体内的生理活动[18]。利用转录组测序的方法对目标基因家族的表达量检测具有成本低、高通量的优势,是目前挖掘功能基因的主要手段之一[19]。本研究选取在果实发育和成熟中已报道的4个相关基因作为目标基因,在甜瓜基因组中鉴定获得其家族成员。对这些基因在河套蜜瓜原种和M9品系不同发育时期果实中的表达量分析,发现CmETR家族成员在成熟过程的表达水平在河套蜜瓜原种和M9中都呈下调趋势。Ciardi等[20]发现乙烯受体在乙烯信号通路中是通过负调控来传递信号的,ETR的表达水平降低可提升植株对乙烯的敏感性。此外,CmCTR12基因在果实发育过程中显著差异表达,且后期呈下调模式,这与姚远等[21]在甜瓜中克隆的Cm-CTR1基因具有相同的表达模式。
本研究中13个基因在河套蜜瓜原种的2个时期存在显著差异表达,12基因在M9的2个时期存在显著差异表达,其中CmHB3和CmHB11呈下调表达模式且不同时期的表达量存在极显著差异,推测其可能在生长期起作用;而CmHB4呈上调表达模式且不同时期的表达量存在极显著差异,推测其可能在成熟期发挥作用。在生长期的河套蜜瓜原种和M9中,5个基因的表达量存在显著差异,其中CmHB3和CmHB11的表达量在不同材料间存在极显著差异;而在成熟期的河套蜜瓜原种和M9中,4个基因的表达量在2个材料间存在显著差异,其中CmHB3和CmHB11的表达量在不同材料间存在极显著差异,推测在果实发育过程中CmACO1的表达影响CmHB3和CmHB11的表达水平。番茄中LeHB1基因表达的抑制导致LeACO1基因的表达也降低[9],而本研究中发现CmHB3和CmHB11 2个基因在转反义CmACO1基因的材料中的表达量显著低于河套蜜瓜原种,暗示甜瓜中CmACO1基因的表达也影响CmHB基因的表达水平。另外,CmRIN14和CmRIN15的表达量在河套蜜瓜原种和M9中存在截然相反的情况,表明其表达模式受CmACO1表达水平的影响。
[1] Abeles F B, Morgan P W, Saltveit J M E. Ethylene in plant biology[M]. Second Edition. London: Harcourt Brace Jovanovich, 2012: 120-181. doi:10.1016/B978-0-08-091628-6.50015-1.
[2] Kazan K. Diverse roles of jasmonates and ethylene in abiotic stress tolerance[J]. Trends in Plant Science, 2015, 20(4): 219-229. doi: 10.1016/j.tplants.2015.02.001.
[3] Mcmurchie E J, Mcglasson W B, Eaks I L. Treatment of fruit with propylene gives information about the biogenesis of ethylene[J]. Nature, 1972, 237(5352): 235-236. doi: 10.1038/237235a0.
[4] Lelièvre J M, Latchè A, Jones B, Bouzayen M. Ethylene and fruit ripening[J]. Physiologia Plantarum, 2007, 26(2): 143-159. doi: 10.1002/9781118223086.ch11.
[5] Nakatsuka A, Murachi S, Okunishi H, Shiomi S, Nakano R, Kubo Y, Inaba A. Differential expression and internal feedback regulation of 1-aminocyclopropane-1-Carboxylate synthase, 1-aminocyclopropane-1-carboxylate oxidase, and ethylene receptor genes in tomato fruit during development and ripening[J]. Plant Physiology, 1998, 118(4): 1295-1305. doi: 10.2307/4278560.
[6] Inaba A. Studies on the internal feedback regulation of ethylene biosynthesis and signal transduction during fruit ripening, and the improvement of fruit quality[J]. Journal of the Japanese Society for Horticultural Science, 2007, 76(1): 1-12. doi: 10.2503/jjshs.76.1.
[7] Barry C S, Lloptous M I, Grierson D. The regulation of 1-aminocyclopropane-1-carboxylic acid synthase gene expression during the transition from system-1 to system-2 ethylene synthesis in tomato[J]. Plant Physiology, 2000, 123(3): 979-986. doi: 10.2307/4279329.
[8] Naoki Y, Ryohei N, Shunsuke I, Nagata M, Inaba A, Kubo Y. Ripening-associated ethylene biosynthesis in tomato fruit is autocatalytically and developmentally regulated[J]. Journal of Experimental Botany, 2009, 60(12): 3433-3442. doi: 10.1093/jxb/erp185.
[9] Lin Z, Hong Y, Yin M, Zhang K, Grierson D. A tomato HD-Zip homeobox protein, LeHB-1, plays an important role in floral organogenesis and ripening[J]. Plant Journal, 2008, 55(2): 301-310. doi: 10.1111/j.1365-313X.2008.03505.x.
[10] Ito Y, Kitagawa M, Ihashi N, Yabe K, Kimbara J, Yasuda J, Ito H, Inakuma T, Hiroi S, Kasumi T. DNA-binding specificity, transcriptional activation potential, and the rin mutation effect for the tomato fruit-ripening regulator RIN[J]. Plant Journal, 2008, 55: 212-223. doi: 10.1111/j.1365-313X.2008. 03491.x.
[11] Vrebalov J, Ruezinsky D, Padmanabhan V, White R, Medrano D, Drake R, Schuch W, Giovannoni J. A MADS-box gene necessary for fruit ripening at the tomato ripening-inhibitor (Rin) locus[J]. Science, 2002, 296:343-346. doi: 10.1126/science.1068181.
[12] Lacey R F, Binder B M. How plants sense ethylene gas-the ethylene receptors[J]. Journal of Inorganic Biochemistry, 2014, 133(2): 58-62. doi: 10.1016/j.jinorgbio.2014.01.006.
[13] Gao Z, Chen Y F, Randlett M D, Zhao X C, Findell J L, Kieber J J, Schaller G E. Localization of the Raf-like kinase CTR1 to the endoplasmic reticulum of Arabidopsis through participation in ethylene receptor signaling complexes[J]. Journal of Biological Chemistry, 2003, 278(36): 34725-34732. doi: 10.1074/jbc.M305548200.
[14] Zhong S, Lin Z, Grierson D. Tomato ethylene receptor CTR interactions: visualization of NEVER-RIPE interactions with multiple CTRs at the endoplasmic reticulum[J]. Journal of Experimental Botany, 2008, 59(4): 965-972. doi: 10.1093/jxb/ern021.
[15] 玉庄. 转ACC氧化酶反义基因河套蜜瓜耐贮藏品系选育[D]. 呼和浩特: 内蒙古大学, 2008. doi: 10.7666/d.y1376451.
Yu Z. The selection of longer shelf-life strains of Cucumis melo L. cv. Hetao transformed with an antisense ACC oxidase gene[D]. Hohhot: Inner Mongolia University, 2008.
[16] 田世平. 果实成熟和衰老的分子调控机制[J]. 植物学报, 2013, 48(5): 481-488. doi: 10.3724/SP.J.1259.2013.00481.
Tian S P. Molecular regulation mechanism of fruit ripening and senescence[J]. Chinese Bulletin of Botany, 2013, 48(5): 481-488.
[17] Gapper N E, Mcquinn R P, Giovannoni J J. Molecular and genetic regulation of fruit ripening[J]. Plant Molecular Biology, 2013, 82(6): 575-591. doi: 10.1007/s11103-013-0050-3.
[18] Seymour G B,Φstergaard L, Chapman N H, Knapp S, Martin C. Fruit development and ripening[J]. Annual review of Plant Biology, 2013, 64(16): 219-241. doi: 10.1146/annurev-arplant-050312-120057.
[19] 张自强,白晨,张惠忠,李晓东,付增娟,赵尚敏,鄂圆圆,张辉,王良,张必周.转录组测序及其在甜菜功能基因挖掘中的应用[J].北方农业学报,2018,46(5):39-43. doi: 10.3969/j.issn.2096-1197.2018.05.06.
Zhang Z Q, Bai C, Zhang H Z, Li X D, Fu Z J, Zhao S M, E Y Y, Zhang H, Wang L, Zhang B Z. Transcriptome sequencing and its application in sugar beet functional gene resource discovery[J]. Journal of Northern Agriculture, 2018,46(5):39-43.
[20] Ciardi J A, Tieman D M, Jones J B. Reduced expression of the tomato ethylene receptor gene LeETR4 enhances the hypersensitive response to Xanthomonas campestris pv. vesicatoria[J]. Molecular plant-microbe interactions, 2001, 14(4): 487-495. doi:10.1094/MPMI.2001.14.4.487.
[21] 姚远, 高峰, 郝金凤, 哈斯阿古拉. 甜瓜乙烯信号转导途径关键因子基因CTR1的克隆及表达特性分析[J]. 生物技术通报, 2011(11): 83-87. doi: 10.13560/j.cnki.biotech.bull.1985.2011.11.004.
Yao Y, Gao F, Hao J F, Hasi A. Cloning and expression analysis of a key gene CTR1 in Ethylene Signal transduction in Cucumis melo[J]. Biotechnology Bulletin, 2011(11): 83-87.