高等植物在逆境胁迫下会感知并传导逆境胁迫信号,从而启动植物对逆境胁迫的响应机制,通过调控相关特定功能基因做出各种适应性反应[1-3]。类受体蛋白激酶 (Receptor-like protein kinase, RLK) 是在植物中大量存在的一类跨膜蛋白激酶,在双子叶模式植物拟南芥中有600个成员,在单子叶模式植物水稻中有1 200多个成员,通常包含胞外配体结合结构域、跨膜结构域和胞内激酶结构域三部分[4-11]。胞外配体结合结构域可感知一些外部逆境信号,进一步通过跨膜传导,激活胞内的丝氨酸/苏氨酸激酶结构域,通过磷酸化级联反应将信号传递给下游底物,使信号逐级放大,从而激活下游转录因子,诱导抗逆相关基因的表达,开启胁迫响应应答机制。
富含亮氨酸重复序列的RLK (LRR-RLK)家族是RLK家族中最大的一类亚家族,约占1/2[12-13]。该家族成员最典型的特点是胞外配体结合结构域含有多个LRR,LRR通常是一个由20~29个氨基酸组成的基序,可与配体结合,将胞外信号传递至下游,最终使胞内完成响应应答。不同LRR-RLK成员所含有的LRR基序的数目及分布间隔不同,重复次数一般介于3~27次。胞外LRR的多样性使得LRR-RLK能够特异地识别不同配体分子,从而正确传递不同的胞外信号。近年来,越来越多的证据表明,LRR-RLK成员在调控植物对抗病原菌及各种非生物胁迫中具有十分重要的作用。目前,在植物中已发现并被详细研究的LRR-RLK成员主要有拟南芥中的BRI1、BKI1、MIK2基因及水稻中的Xa21、LRRK1、DOCS1基因等[14-25],水稻中大部分LRR-RLK基因的功能仍未被揭示。
水稻是我国乃至世界上的主要粮食作物,其产量、品质对我国乃至世界均有重要影响。近年来,水稻生长发育过程中受到低磷胁迫的影响越来越严重,降低了水稻的产量和品质。然而,目前针对水稻耐低磷胁迫方面的LRR-RLK研究并不十分深入。因此,为筛选低磷胁迫响应的水稻LRR-RLK基因,河南省水稻生物学重点实验室对低磷处理的水稻基因芯片表达谱进行分析,发现1个候选LRR-RLK基因的转录水平明显受低磷胁迫诱导。由于该基因为10个候选低磷胁迫响应基因 (命名为LP1-LP10) 中的第7个,因此,将其暂命名为LP7 (LOC_Os05g24010),而且其相关研究暂无报道。为此,对LP7基因进行分子克隆、序列比对、亚细胞定位分析,并研究其组织表达模式及低磷胁迫下的表达特征,为深入研究该基因功能奠定理论基础。
1 材料和方法
1.1 试验材料及低磷胁迫处理
水稻品种日本晴(Nipponbare)种子及本氏烟草(Nicotiana benthamiana)种子均为河南省水稻生物学重点实验室保存;瞬时表达载体p35S-GFP由河南农业大学农学院陈锋课题组赵磊老师提供,由pBI121改造而成。
利用1/2 MS营养液培养水稻幼苗至两叶一心期,选取整齐度均匀一致的幼苗进行低磷 (10 μmol/L) 胁迫处理,以不进行低磷胁迫处理的材料为对照。处理12 h后,取整株幼苗作为试验样品,3次生物学重复。取样后迅速将样品置于液氮中冷冻,并保存于-80 ℃超低温冰箱中备用。
1.2 试验方法
1.2.1 水稻总RNA的提取及cDNA合成 按照TRNzol总RNA提取试剂盒(TIANGEN)说明书提取水稻不同组织及低磷胁迫处理样品的总RNA。然后取0.1 μg 总RNA作为模板,按照PrimeScriptTM RT reagent Kit with gDNA Eraser (TaKaRa)试剂盒的操作说明书反转录合成cDNA第一链。
1.2.2 LP7基因的克隆 根据Rice Genome Annotation Project (http://rice.plantbiology.msu.edu/) 网站上公布的水稻LP7基因 (LOC_Os05g24010) 的核苷酸序列,利用Primer Premier 5软件设计特异引物,上游引物为LP7-F: 5′-ATGGTGAAGAGGCGG CTGTT-3′,下游引物为LP7-R: 5′-CTACGTGCCCCT GTTCTTCA-3′。引物由生工生物工程(上海)股份有限公司合成。以反转录合成的cDNA第1链为模板进行PCR扩增,50 μL的PCR反应体系包含25 μL Premix PrimeSTAR HS (TaKaRa) DNA聚合酶,上、下游引物 (10 μmol/L) 各2.5 μL,cDNA模板1 μL,ddH2O 补齐至50 μL。PCR反应程序为:98 ℃预变性2 min;98 ℃变性10 s,60 ℃退火10 s,68 ℃延伸 60 s,30个循环;最后68 ℃延伸6 min。PCR产物经1%琼脂糖凝胶电泳分离,切胶纯化回收。回收产物连接至pMD18-T载体,采用热激法转化至大肠杆菌DH5α感受态细胞中,筛选阳性克隆送测序。
1.2.3 蛋白质结构及序列分析 利用SMART(http://smart.embl-heidelberg.de/)预测蛋白质结构;利用ExPASy (https://www.expasy.org/)预测蛋白质的分子质量和等电点;利用DNAman 软件进行多序列比对。
1.2.4 实时荧光定量PCR (qRT-PCR) 分析 利用Primer Premier 5软件设计qRT-PCR引物,上游引物为LP7-Q-F: 5′-GAGGTGATAATGCAGGACA-3′,下游引物为LP7-Q-R: 5′-TGCGTTGGAGTATGGGT-3′。以水稻OsACTIN基因为内参,其上游引物为:OsACTIN-Q-F: 5′-AACATCGTTCTCAGTGGTGGTA-3′,下游引物为OsACTIN-Q-R: 5′-GGAGGACGGCGATAA CAG-3′,引物均由生工生物工程(上海)股份有限公司合成。利用SYBR®Premix Ex TaqTM Ⅱ(TaKaRa)试剂盒在Bio-Rad CFX96TM Real-time PCR仪上进行qRT-PCR反应。反应体系为:2×SYBR®Premix Ex TaqTM Ⅱ 10 μL,Rox reference Dye Ⅱ 0.2 μL,上、下游引物各2 μL,cDNA模板1 μL (15 ng/μL),ddH2O 补齐至20 μL。反应程序为:95 ℃ 30 s;95 ℃ 10 s,60 ℃ 30 s,35个循环。每个样品设置3个技术性重复,3个生物学重复。相对表达量根据2-ΔΔCT方法计算[26]。
1.2.5 亚细胞定位 利用Primer Premier 5软件设计携带SmaⅠ酶切位点 (上游引物) 和SpeⅠ酶切位点 (下游引物) 的LP7基因的引物,上游引物为:LP7-GFP-F:5′-CCCGGGATGGTGAAGAGGCGGCTGT T-3′,下游引物为LP7-GFP-R:5′-ACTAGTCGTGCC CCTGTTCTTCAGG-3′。以测序正确的单克隆为模板,PCR扩增LP7基因序列,用SmaⅠ和SpeⅠ进行双酶切,同时用SmaⅠ和SpeⅠ双酶切瞬时表达载体p35S-GFP, 分别回收片段,用T4 DNA连接酶连接,获得35S-LP7-GFP融合表达载体,采用热激法转化至大肠杆菌DH5α感受态细胞中,用菌落PCR和酶切方法鉴定阳性克隆,并测序验证。
参照文献[27]的方法将获得的重组质粒35S-LP7-GFP转入农杆菌EHA105菌株中,然后挑取单克隆于5 mL LB液体培养基 (含50 mg/L卡那霉素、100 mg/L利福平)中,28 ℃,180 r/min过夜培养16 h,室温下4 000 r/min离心5 min,收集菌体。用5 mL注射培养液(10 mmol/L MgCl2、10 mmol/L MES(一水吗啉乙磺酸)、200 μmmol/L乙酰丁香酮)悬浮菌体,4 000 r/min离心10 min,然后弃上清,最后用注射培养液悬浮至OD600为0.1~0.2,静置1~2 h,用无针头的1 mL注射器将其注射至4周龄的本氏烟草叶片中。注射后,25 ℃弱光培养48 h,用激光共聚焦显微镜观察荧光。
2 结果与分析
2.1 LP7基因的克隆及其编码蛋白序列分析
利用LP7基因序列设计特异引物,以水稻品种日本晴cDNA为模板进行PCR扩增,扩增出1条单一的条带 (图1-A)。测序结果显示,其与Rice Genome Annotation Project (http://rice.plantbiology.msu.edu/) 网站上公布的LP7基因序列完全一致,目的片段长2 832 bp,具有1个完整的开放阅读框,表明成功克隆了水稻LP7基因。利用SMART软件分析表明,LP7是典型的LRR-RLK基因家族成员,包含3个典型的结构域:含有2个LRR基序的胞外配体结合结构域、跨膜结构域、胞内激酶结构域(图1-B)。
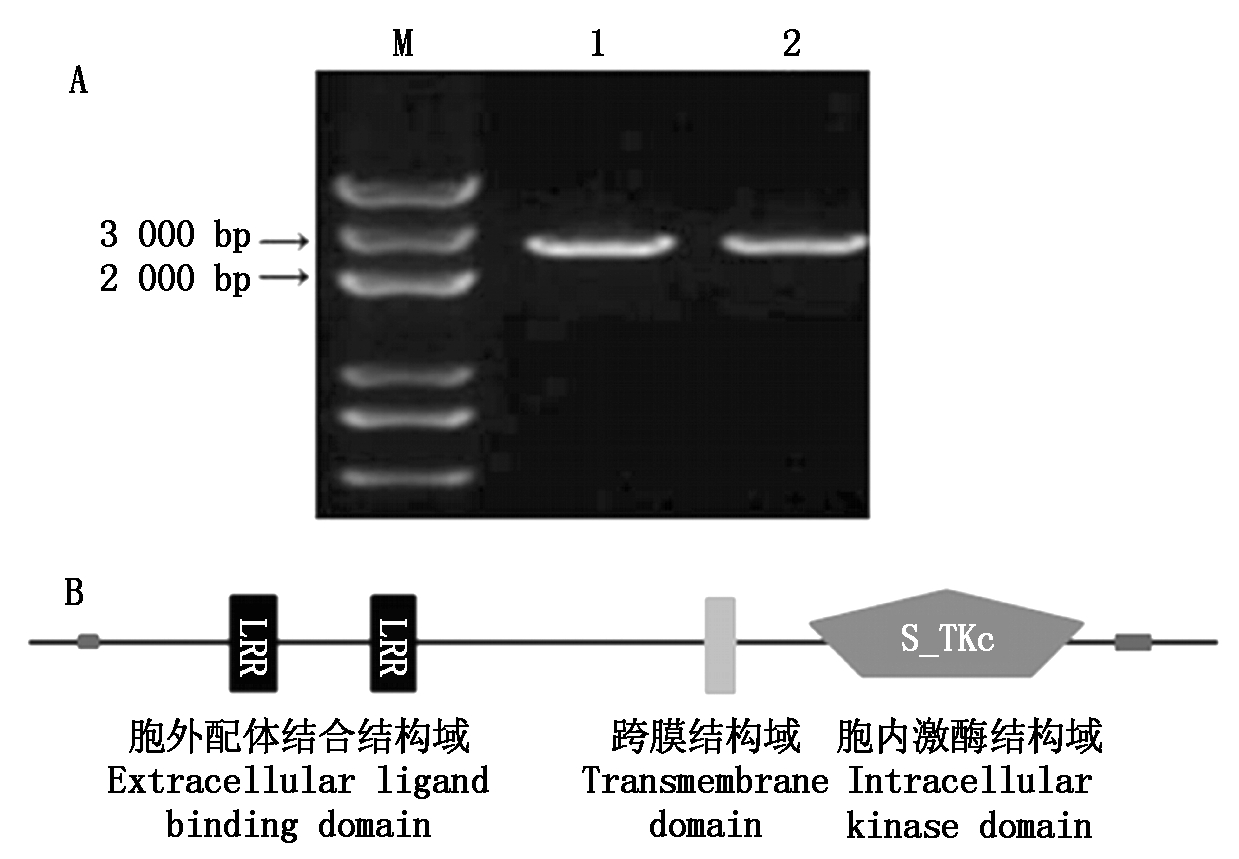
M.DL5000;1-2.LP7基因扩增产物。
M.DL5000; 1-2.LP7 gene amplification products.
图1 LP7基因的PCR扩增产物检测结果(A)及编码蛋白质序列(B)
Fig.1 Detection results of PCR amplification products of LP7 gene (A) and protein sequence encoded by LP7 gene (B)
2.2 LP7蛋白的氨基酸序列比对分析
经ExPASy预测发现,LP7基因编码蛋白含943个氨基酸,分子质量为103.67 ku,等电点为8.16。将预测的LP7蛋白的氨基酸序列与来自水稻、玉米中的另外2个同源蛋白序列 (EEE63180、NP_001131018) 进行比对 (图2)发现,LP7蛋白与水稻中的同源蛋白EEE63180的同源性达99%,仅在第75位氨基酸处存在差异,其余序列均完全一致,与玉米中的同源蛋白NP_001131018的同源性达77%,表明LP7基因在单子叶植物中相对比较保守。
2.3 LP7基因的组织表达模式
由图3可知,LP7基因在水稻根、茎、叶、穗等组织中均有一定量的表达,但在不同组织中的表达水平存在差异,在叶中表达量最高,其次是根,在穗中表达量最低,这可能与植物叶、根在感知逆境信号中发挥重要作用有关。
2.4 LP7蛋白的亚细胞定位分析
由图4可知,对于转35S-LP7-GFP载体的烟草叶片,绿色荧光蛋白分布在细胞膜上;而对于转35S-GFP载体的烟草叶片,绿色荧光蛋白分布在整个细胞膜、细胞质和细胞核中,说明LP7蛋白定位于细胞膜上,这也与SMART在线预测的LP7为一个LRR-RLK的结果一致,暗示LP7作为膜蛋白在接收胞内外信号方面发挥功能。

图2 LP7蛋白与其他同源蛋白质的氨基酸序列比对结果
Fig.2 Multiple amino acid sequences alignment of LP7 and other homologous protein
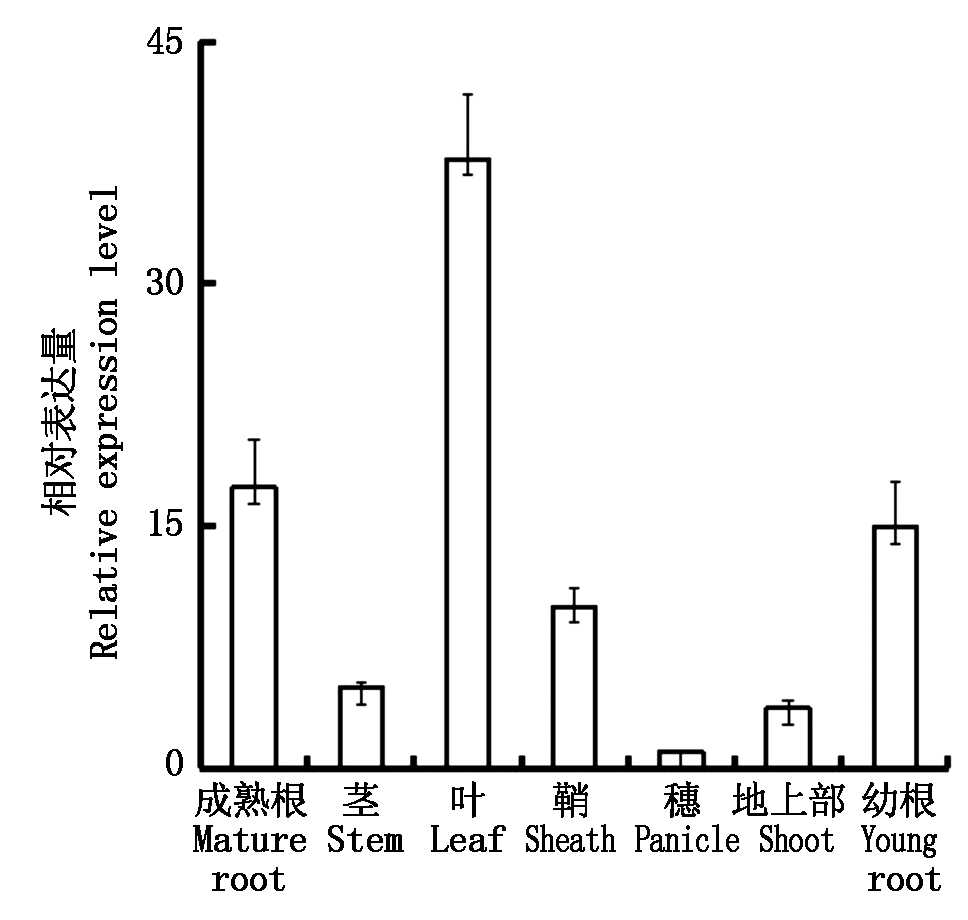
图3 LP7基因的组织表达模式分析
Fig.3 Expression profile of rice LP7 gene in tissues
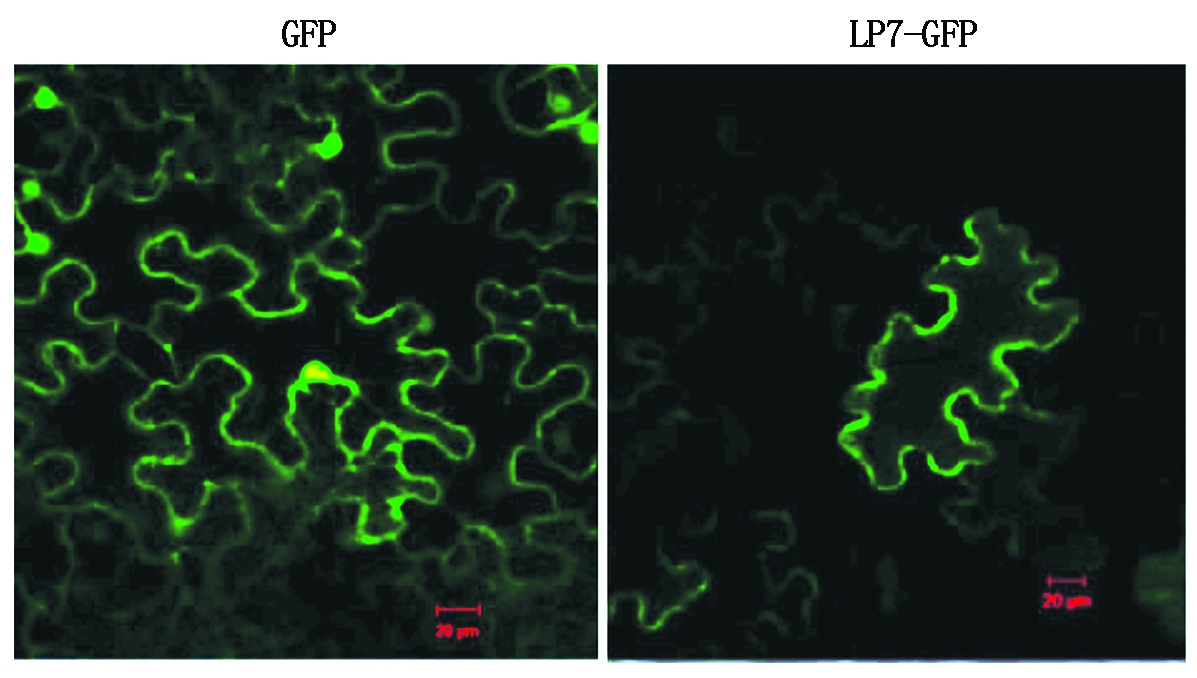
图4 LP7-GFP蛋白与GFP蛋白在本氏烟草叶片细胞中的定位
Fig.4 Subcellular localization of LP7-GFP and GFP proteins in leaves of Nicotiana benthamiana
2.5 LP7基因在低磷胁迫条件下的表达分析
由图5可知,低磷胁迫能明显诱导LP7基因表达,其表达量较对照增加15倍,表明LP7基因可能在水稻低磷胁迫响应过程中发挥重要功能。
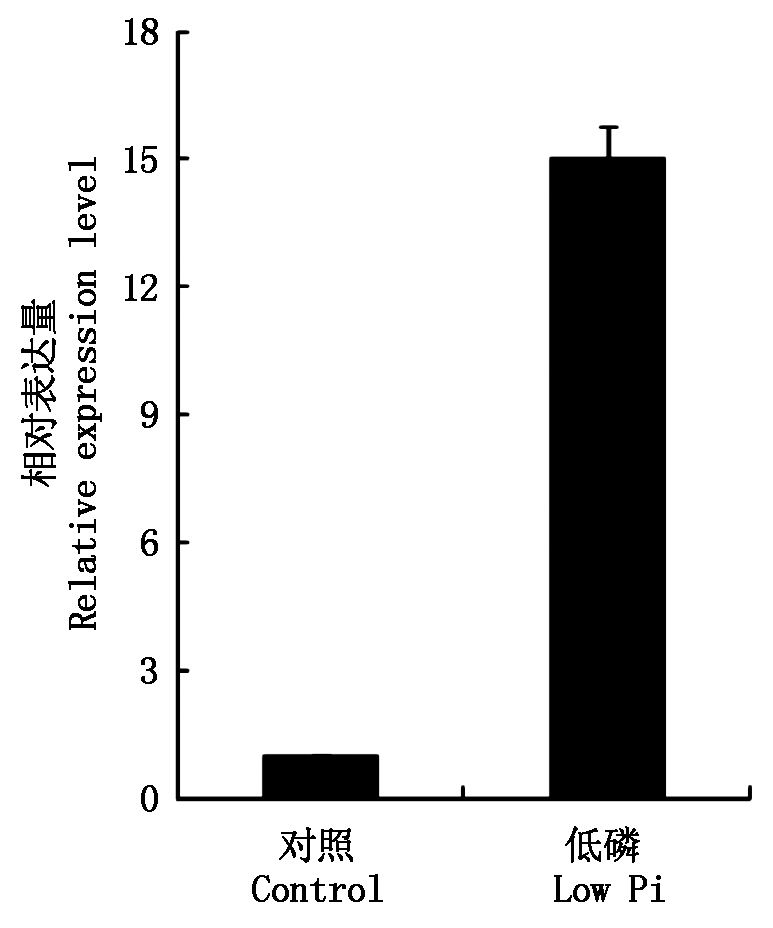
图5 LP7基因在低磷胁迫条件下的表达
Fig.5 Expression level of LP7 gene under low Pi stress
3 结论与讨论
RLK在调控植物生长发育过程中具有非常重要的作用。已有研究表明,LRR-RLK作为RLK家族中最大的一类亚家族,在调控植物体生长发育及抗逆胁迫等方面均发挥着十分重要的作用[28-31]。为研究参与低磷胁迫响应的LRR-RLK基因的功能特点,本研究从水稻品种日本晴中克隆到1个LRR-RLK成员LP7基因。氨基酸序列分析表明,LP7蛋白具有典型的LRR-RLK成员特征,为1个LRR-RLK新成员,目前其功能未知。
不同物种的氨基酸序列同源性分析表明,LP7与玉米中的同源蛋白NP_001131018同源性比较高,同源性高达77%,暗示LP7基因在单子叶植物中相对比较保守。
蛋白质在细胞中的定位决定了其行使功能的具体部位。烟草的瞬时表达试验结果表明,LP7定位在细胞膜上,该结果与已报道的LRR-RLK的亚细胞定位结果一致[28],同时也与LP7作为1个RLK参与细胞外信号接收、传递的功能相吻合。
基因组织表达模式分析对研究基因功能十分重要。本研究发现,LP7基因在各组织中均有一定量的表达,在叶中表达量最高,穗中表达量最低。推测可能与植物叶在感知逆境信号中发挥重要作用有关。低磷胁迫条件下的表达分析表明,LP7基因受低磷胁迫诱导表达,与前期芯片数据一致,暗示LP7基因可能在水稻响应低磷胁迫中发挥一定的功能。
[1] Baker B,Zambryski P,Staskawicz B, Dinesh-Kumar S P. Signaling in plant-microbe interactions[J]. Science, 1997, 276: 726-733. doi: 10.1126/science.276.5313.726.
[2] Torii K U. Receptor kinase activation and signal transduction in plants: An emerging picture[J]. Current Opinion in Plant Biology, 2000, 3(5): 361-367. doi: 10.1016/s1369-5266(00)00097-2.
[3] Mccarty D R, Chory J. Conservation and innovation in plant signaling pathways[J]. Cell, 2000, 103(2): 201-209. doi: 10.1016/S0092-8674(00)00113-6.
[4] Braun D M, Walker J C. Plant transmembrane receptors:New pieces in the signaling puzzle[J]. Trends in Biochemical Sciences, 1996, 21(2): 70. doi: 10.1016/S0968-0004(96)80185-X.
[5] Walker J C, Zhang R. Relationship of a putative receptor protein kinase from maize to the S-locus glycoproteins of Brassica[J]. Nature, 1990, 345(6277): 743-746. doi: 10.1038/345743a0.
[6] Shiu S H, Bleecker A B. Receptor-like kinases from Arabidopsis form a monophyletic gene family related to animal receptor kinases[J]. Proc Natl Acad Sci USA, 2001, 98(19): 10763-10768. doi: 10.1073/pnas.181141598.
[7] De S I, Voss U G, Jurgens G, Beeckman T. Receptor-like kinases shape the plant[J]. Nature Cell Biology, 2009, 11(10): 1166-1173. doi: 10.1038/ncb1009-1166.
[8] Antolín-Llovera M, Ried M K, Binder A, Parniske M. Receptor kinase signaling pathways in plant-microbe interactions[J]. Annual Review of Phytopathology, 2012, 50(1): 451-473. doi: 10.1146/annurev-phyto-081211-173002.
[9] 张蕾, 吕应堂. 植物受体蛋白激酶的研究进展[J]. 生命科学, 2002, 14(2): 95-98. doi: 10.3969/j.issn.1004-0374.2002.02.009.
Zhang L, Lu Y T. Research progress of plant receptor protein kinase[J]. Chinese Bulletin of Life Sciences, 2002, 14(2): 95-98.
[10] Walker J C. Structure and function of the receptor-like protein kinases of higher plants[J]. Plant Molecular Biology, 1994, 26(5): 1599-1609. doi: 10.1007/BF00016492.
[11] 朱巍巍, 马天意, 张梅娟, 沙伟. 类受体蛋白激酶在植物中的研究进展[J]. 基因组学与应用生物学, 2018, 37(1): 451-458. doi: 10.13417/j.gab.037.000451.
Zhu W W, Ma T Y, Zhang M J, Sha W. Advances in the study of receptor-like protein kinases in plants[J]. Genomics and Applied Biology, 2018, 37(1): 451-458.
[12] Dievart A, Clark S. LRR-containing receptors regulate plant development and defense[J]. Development, 2004,131(2): 251-261. doi: 10.1242/dev.00998.
[13] Torii K U, Mitsukawa N, Oosumi T, Matsuura Y, Yokoyama R, Whittier R,Komeda Y. The Arabidopsis ERECTA gene encodes a putative receptor protein kinase with extracellular leucine-rich repeats[J]. Plant Cell, 1996, 8(4): 735-746. doi: 10.2307/3870348.
[14] Clark S E, Williams R W, Meyerowitz E M. The CLAVATA1 gene encodes a putative receptor kinase that controls shoot and floral meristem size in Arabidopsis[J]. Cell, 1997, 89(4):575-85. doi: 10.1016/S0092-8674(00)80239-1.
[15] Jinn T L, Stone J M, Walker J C. HAESA, an Arabidopsis leucine-rich repeat receptor kinase, controls floral organ abscission[J]. Genes & Development, 2000, 14(1): 108-17. doi: 10.1101/gad.14.1.108.
[16] Clouse S D, Langford M, Mcmorris T C. A brassinosteroid-lnsensitive mutant in Arabidopsis thaliana exhibits multiple defects in growth and development[J]. Plant Physiology, 1996, 111(3): 671-678. doi: 10.1104/pp.111.3.671.
[17] Li J M, Chory J. A putative leucine-rich repeat receptor kinase involved in brassinosteroid signal transduction[J]. Cell, 1997, 90(5): 929-938. doi: 10.1016/S0092-8674(00)80357-8.
[18] Song W Y, Wang G L, Chen L L, Kim H S, Pi L Y, Holsten T, Gardner J, Wang B, Zhai W X, Zhu L H, Fauquet C, Ronald P. A receptor kinase-like protein encoded by the rice disease resistance gene, Xa21[J]. Science, 1995, 270(5243): 1804-1806. doi: 10.1126/science.270.5243.1804.
[19] Wang G L, Ruan D L, Song W Y, Sideris S, Chen L L, Pi L Y, Zhang S P, Zhang Z, Fauquet C, Gaut B, Whalen M, Ronald P. Xa21D encodes a receptor-like molecule with a leucine-rich repeat domain that determines race-specific recognition and is subject to adaptive evolution[J]. The Plant Cell, 1998, 10(5): 765-779. doi: 10.2307/3870663.
[20] 白辉,李莉云,刘国振. 水稻抗白叶枯病基因Xa21的研究进展[J]. 遗传,2006, 28(6): 745-753. doi: 10.3321/j.issn:0253-9772.2006.06.020.
Bai H, Li L Y, Liu G Z. Research progress of rice bacterial blight resistance gene Xa21[J]. Hereditas, 2006, 28(6): 745-753.
[21] Park C J, Lee S W, Chern M, Sharma R, Canlas P E, Song M Y, Jeon J S, Ronald P C. Ectopic expression of rice Xa21 overcomes developmentally controlled resistance to Xanthomonas oryzae pv. oryzae[J]. Plant Science An International Journal of Experimental Plant Biology, 2010, 179(5): 466-471. doi: 10.1016/j.plantsci.2010.07.008.
[22] Zhou Y B, Wang D, Wu T, Yang Y Z, Liu C, Yan L, Tang D Y, Zhao X Y, Zhu Y H, Lin J Z, Liu X M. LRRK1, a receptor-like cytoplasmic kinase, regulates leaf rolling through modulating bulliform cell development in rice[J]. Molecular Breeding, 2018, 38(5): 48. doi: 10.1007/s11032-018-0811-4.
[23] Bettembourg M, Dal-Soglio M, Bureau C, Vernet A, Dardoux A, Portefaix M, Bes M, Meynard D, Mieulet D, Cayrol B, Perin C, Courtois B, Ma J F, Dievart A. Root cone angle is enlarged in docs1 LRR-RLK mutants in rice[J]. Rice, 2017, 10(1): 50. doi: 10.1186/s12284-017-0190-1.
[24] Wang D X, Yang C J, Wang H J, Wu Z H, Jiang J, Liu J J, He Z N, Chang F, Ma H, Wang X L. BKI1 regulates plant architecture through coordinated inhibition of the brassinosteroid and ERECTA signaling pathways in Arabidopsis[J]. Molecular Plant, 2017, 10(2): 297-308. doi: 10.1016/j.molp.2016.11.014.
[25] Van der Does D, Boutrot F, Engelsdorf T, Rhodes J, McKenna J F, Vernhettes S, Koevoets I, Tintor N, Veerabagu M, Miedes E, Segonzac C, Roux M, Breda A S, Hardtke C S, Molina A, Rep M, Testerink C, Mouille G, Höfte H, Hamann T, Zipfel C. The Arabidopsis leucine-rich repeat receptor kinase MIK2/LRR-KISS connects cell wall integrity sensing, root growth and response to abiotic and biotic stresses[J]. PLoS Genetics, 2017, 13(6): e1006832. doi: 10.1371/journal.pgen.1006832.
[26] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2 method[J]. Methods, 2001, 25(4): 402-408. doi: 10.1006/meth.2001.1262.
[27] Sparkes I A, Runions J, Kearns A, Hawes C. Rapid, transient expression of fluorescent fusion proteins in tobacco plants and generation of stably transformed plants[J]. Nature Protocols, 2006, 1(4): 2019-2025. doi: 10.1038/nprot.2006.286.
[28] Wang J, Liu S H, Li C C, Wang T L, Zhang P Y, Chen K S. PnLRR-RLK27, a novel leucine-rich repeats receptor-like protein kinase from the Antarctic moss Pohlia nutans, positively pegulates salinity and oxidation-stress tolerance[J]. PLoS One, 2017, 12(2): e0172869. doi: 10.1371/journal.pone.0172869.
[29] Liu P L, Du L, Huang Y, Gao S M, Yu M. Origin and diversification of leucine-rich repeat receptor-like protein kinase (LRR-RLK) genes in plants[J]. BMC Evolutionary Biology, 2017, 17(1): 47. doi: 10.1186/s12862-017-0891-5.
[30] Wang J, Li C C, Yao X H, Liu S H, Zhang P Y, Chen K S. The Antarctic moss leucine-rich repeat receptor-like kinase (PnLRR-RLK2) functions in salinity and drought stress adaptation[J]. Polar Biology, 2018,41(2):353-364. doi: 10.1007/s00300-017-2195-z.
[31] Wang J, Liu S, Li C, Wang T, Zhang P, Chen K. PnLRR-RLK27, a novel leucine-rich repeats receptor-like protein kinase from the Antarctic moss Pohlia nutans, positively regulates salinity and oxidation-stress tolerance[J]. PLoS One, 2017, 12(2): e0172869. doi: 10.1371/journal.pone.0172869.