MicroRNA(miRNA)是一类约22个核苷酸的小分子非编码RNA,通过Argonaute(AGO)蛋白和RNA诱导沉默复合体(RNA-induced silencing complex,RISC)的形式,在转录或转录后水平抑制靶基因的表达[1],从而参与基因的表达调控[2]。miRNA的初级转录本pri-miRNA通过编码具有调节功能的多肽,促进miRNA积累,调节基因表达[3]。miRNA在调节植物基因表达、促进生长发育和抵御胁迫等方面发挥作用。miR399是miRNA的重要家族成员,在植物响应低磷胁迫过程中发挥重要作用[4]。miR399受低磷诱导上调表达,通过抑制PHO2基因表达[5],参与了植物体内磷酸盐的转运[6]。
目前,多个物种miR399家族成员已被鉴定,然而该家族成员在各植物中的数量分布、进化关系及其功能的比较研究较少。为全面了解植物miR399家族成员进化与功能特征,本研究以miRBase数据库中记录的植物miR399为研究对象,通过生物信息学方法,对物种分布、序列特征、启动子区特征和靶基因功能等进行研究,为该家族成员的进一步开发利用提供借鉴。
1 材料和方法
1.1 植物miR399数据的获取
所有植物miR399数据下载于miRBase数据库(http://www.mirbase.org/)和PMRD数据库(http://bioinformatics.cau.edu.cn/PMRD/)。
1.2 miR399序列比对分析
利用ClustalX进行序列比对,利用MEGA 5.0软件,采用邻接法(Neighbor-joining,NJ)构建系统发育树。
1.3 二级结构预测分析
采用Mfold在线软件(http://unafold.rna.albany.edu/?q=mfold)对pre-miR399进行二级结构预测,并对成熟体位置进行分析。
1.4 启动子特征分析
通过PMRD数据库(http://bioinformatics.cau.edu.cn/PMRD/)获取拟南芥和水稻per-miRNA转录起始位点上游1.5 kb序列,通过PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)数据库进行基因启动子区顺式作用元件分析。
1.5 miR399靶基因预测
从PMRD下载获得拟南芥、水稻miRNA靶基因,并在psRNATarget数据库(http://plantgrn.noble.org/psRNATarget/)中进行验证。利用AGRiGO数据库(http://bioinfo.cau.edu.cn/agriGO/analysis.php)对水稻miR399靶基因的基因本体(Gene ontology,GO)进行分析。
2 结果与分析
2.1 植物miR399家族成员统计分析
截至2018年2月1日,通过miRBase数据库共搜索到32个植物物种的206个pre-miR399和244个成熟miR399。miR399仅分布在被子植物中,其中25种为双子叶植物,pre-miR399和miR399的数量分别为166,192个,7种为单子叶植物,pre-miRNA和成熟miRNA的数量分别为40,52个(表1)。各物种间miR399家族成员数量不同,即使同科植物间也相差较大,pre-miR399和成熟miR399数量在豆科的蒺藜苜蓿分别为20,22个,而菜豆中均为1,在禾本科的玉米分别为10,20个,而小麦中均为1,表明植物miR399家族成员的数量与植物的进化程度关系不大。各物种pre-miR399和成熟miR399数量之间比例关系分为两类,一类二者接近1∶1,大多数植物属于此类,另一类是接近1∶2,仅有山羊草、玉米、马铃薯和琴叶拟南芥属于此类。
pre-miR399广泛分布在63~286 nt,呈钟型分布,平均长度为116 nt,数量最多的核苷酸数为64 nt,有10条序列,其次为94 nt的有8条,长度为122,123 nt的分别有7条,长度为101~140 nt的序列数量有113条,占全部数量的54.85%(图1)。成熟miR399序列长度在19~23 nt,其中21 nt的占93.85%,其次为22 nt的有12条,说明miR399在植物中的长度较稳定。
2.2 植物miR399家族的序列分析
植物前体和成熟miRNAs在植物进化过程中高度保守[7]。利用Weblogo在线软件对植物miR399家族前体序列进行分析(图2),发现5′和3′端均有较保守序列,其中3′端一致序列为UGC CAAAGGAGA*UUGCCC*G,该序列即为大部分miR399序列。
表1 植物miR399家族成员分布
Tab.1 Distribution of plant miR399 family members
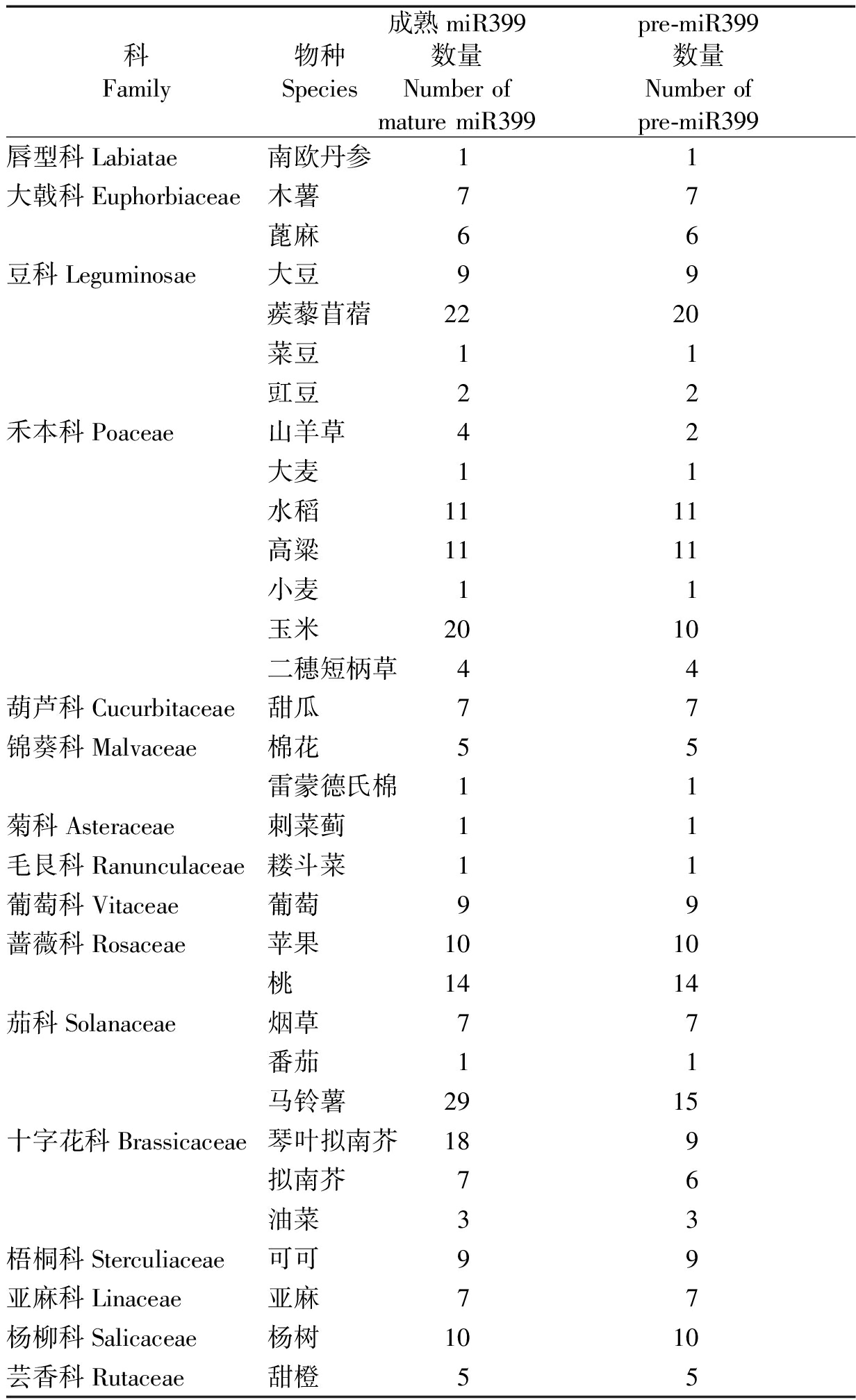
科Family物种Species成熟miR399数量Number of mature miR399pre-miR399数量Number of pre-miR399唇型科Labiatae南欧丹参11大戟科Euphorbiaceae木薯77蓖麻66豆科Leguminosae大豆99蒺藜苜蓿2220菜豆11豇豆22禾本科Poaceae山羊草42大麦11水稻1111高粱1111小麦11玉米2010二穗短柄草44葫芦科Cucurbitaceae甜瓜77锦葵科Malvaceae棉花55雷蒙德氏棉11菊科Asteraceae刺菜蓟11毛艮科Ranunculaceae耧斗菜11葡萄科Vitaceae葡萄99蔷薇科Rosaceae苹果1010桃1414茄科Solanaceae烟草77番茄11马铃薯2915十字花科Brassicaceae琴叶拟南芥189拟南芥76油菜33梧桐科Sterculiaceae可可99亚麻科Linaceae亚麻77杨柳科Salicaceae杨树1010芸香科Rutaceae甜橙55
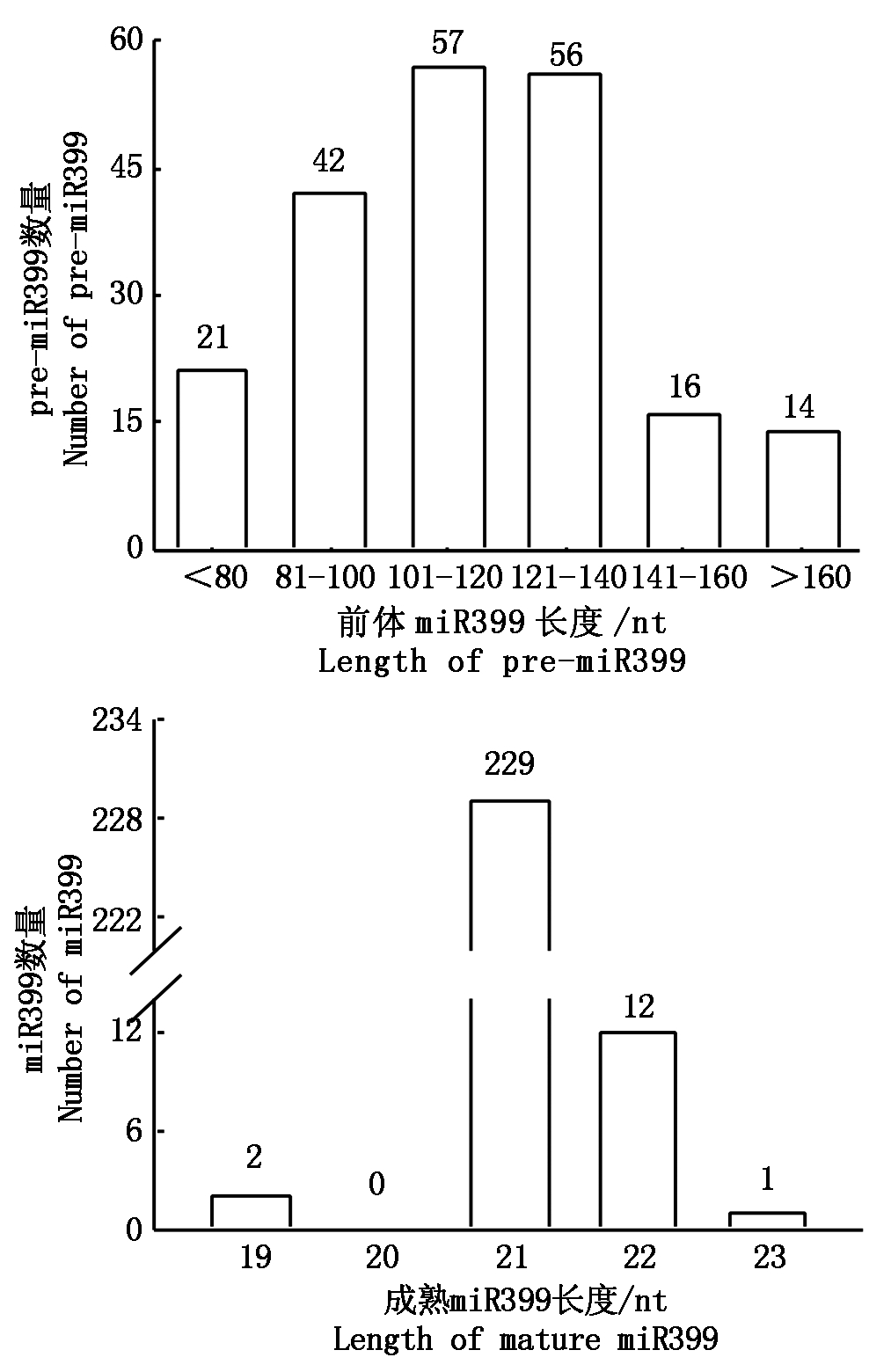
图1 植物前体miR399和成熟miR399核苷酸长度分布
Fig.1 Size distribution of plant pre-miR399 and mature miR399
为了解植物miR399家族进化特征,以双子叶植物中豆科的大豆、茄科的烟草、十字花科的拟南芥、杨柳科的杨树、蔷薇科的苹果和单子叶植物中禾本科的水稻、玉米为代表,构建系统发育树,并对成熟体序列进行比对分析(图3)。除玉米外,其他6个物种miR399序列一致性较高,水稻既有部分miR399与大豆的聚在一组,也有部分miR399与玉米、杨树和苹果聚在一组,说明miR399在植物种间的保守性较强,该家族在植物的生命调控中发挥重要且稳定的作用。
2.3 植物miR399家族二级结构预测

图2 前体miR399保守核苷酸序列标签
Fig.2 Sequence logo analysis of plant pre-miR399

图中miRNA物种缩写分别为:gma.大豆;osa.水稻;ptc.杨树;mdm.苹果;ath.拟南芥;zma.玉米;nta.烟草。
The miRNA species abbreviations:gma.Glycine max;osa.Oryza sativa;ptc.Populus trichocarpa;mdm.Malus domestica;ath.Arabidopsis thaliana;zma.Zea mays;nta.Nicotiana tabacum, respectively.
图3 不同科植物miR399家族聚类及成熟体序列比对
Fig.3 Phylogenetic tree of different family plants miR399
family and multiple alignment of mature sequences
植物pre-miR399经RNase Ⅲ-Dicer切割产生成熟miRNA[8]。pre-miRNA二级结构及成熟miRNA序列位置的研究,对序列切割和折叠预测具有重要的参考价值。本研究利用Mfold在线软件对部分pre-miR399二级结构进行预测,发现植物pre-miR399主要分为3种类型:①产生1条成熟miR399,且位于pre-miR399的3′端,大部分植物pre-miR399均属此类;②产生2条成熟miR399,分别位于pre-miR399的3′端和5′端,产生2条成熟miR399的前体均属此类;③产生1条成熟miR399,且位于pre-miR399的5′端,仅有gra-miR399和stu-miR399h属此类(图4)。这些不同类型的miR399可能在植物中发挥了不同的功能。
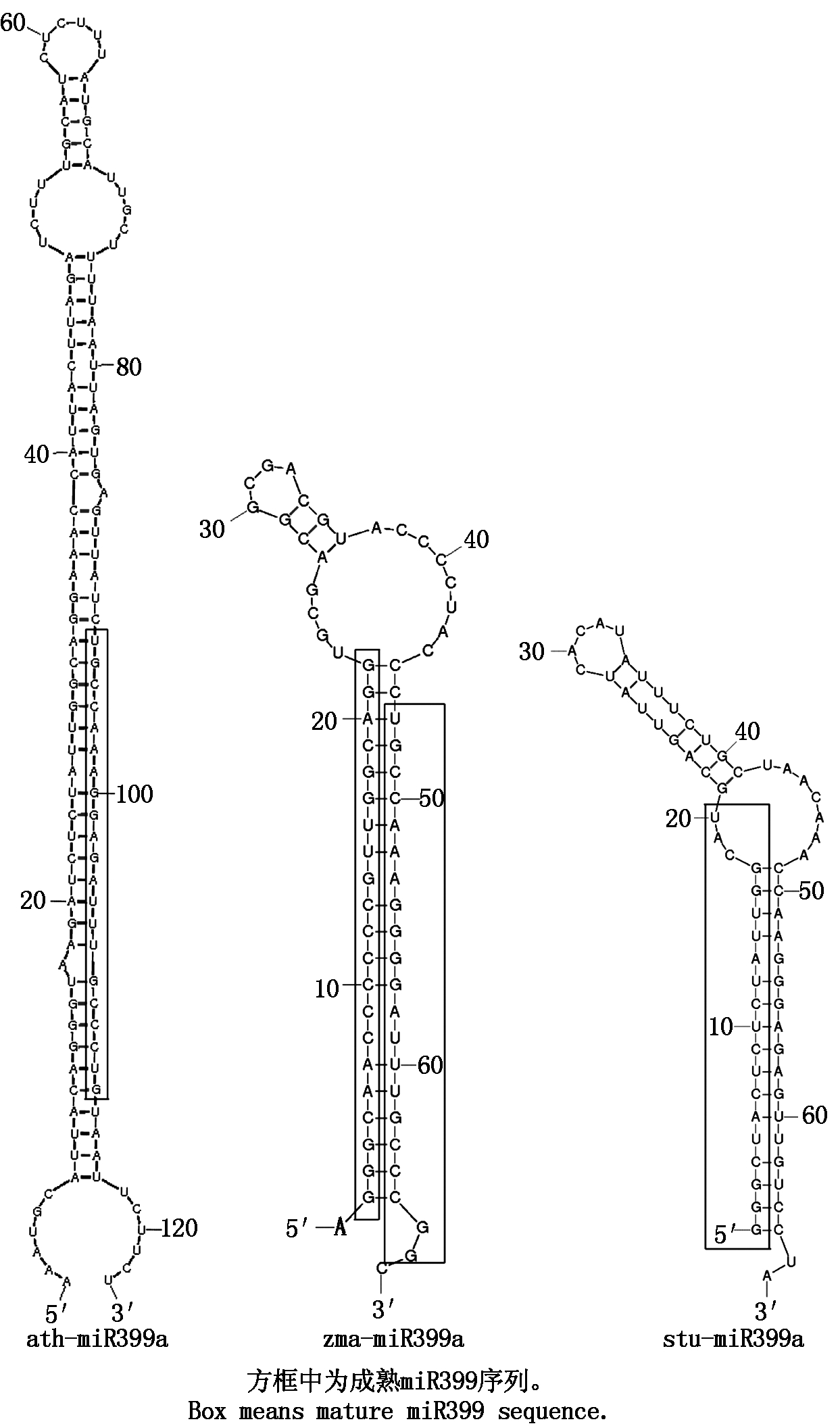
图4 植物pre-miR399家族发夹结构
Fig.4 Predicted hairpin secondary structures of selected plant pre-miR399
2.4 启动子特征分析
通过PlantCARE数据库对拟南芥和水稻miR399家族pre-miRNA上游1.5 kb启动子区顺式作用元件进行了分析(表2),发现所有基因启动子区均含有多个TATA-box和CAAT-box,部分基因含有Py-rich;所有基因启动子区均含有1个到多个激素响应元件,包括脱落酸响应元件ABRE、茉莉酸响应元件CGTCA-motif、赤霉素响应元件GARE-motif等;多数基因启动子区均含有多个胁迫响应元件,包括热激响应元件HSE、低温反应顺式作用元件LTR、干旱胁迫响应MYB结合位点MBS、防御和胁迫相关响应元件TC-rich。除此之外,大部分基因含有厌氧响应元件ARE和蛋白代谢调控顺式元件O2-site。基因含有的激素或胁迫响应元件数量和类型不同,可能是不同的基因在不同的信号通路中发挥作用,也说明该家族基因功能的多样性和复杂性。
表2 拟南芥和水稻miR399基因上游1.5 kb启动子区顺式作用元件信息
Tab.2 Information about putative cis-acting elements in the 1.5 kb upstream promoter region of miR399 genes in Arabidopsis and rice
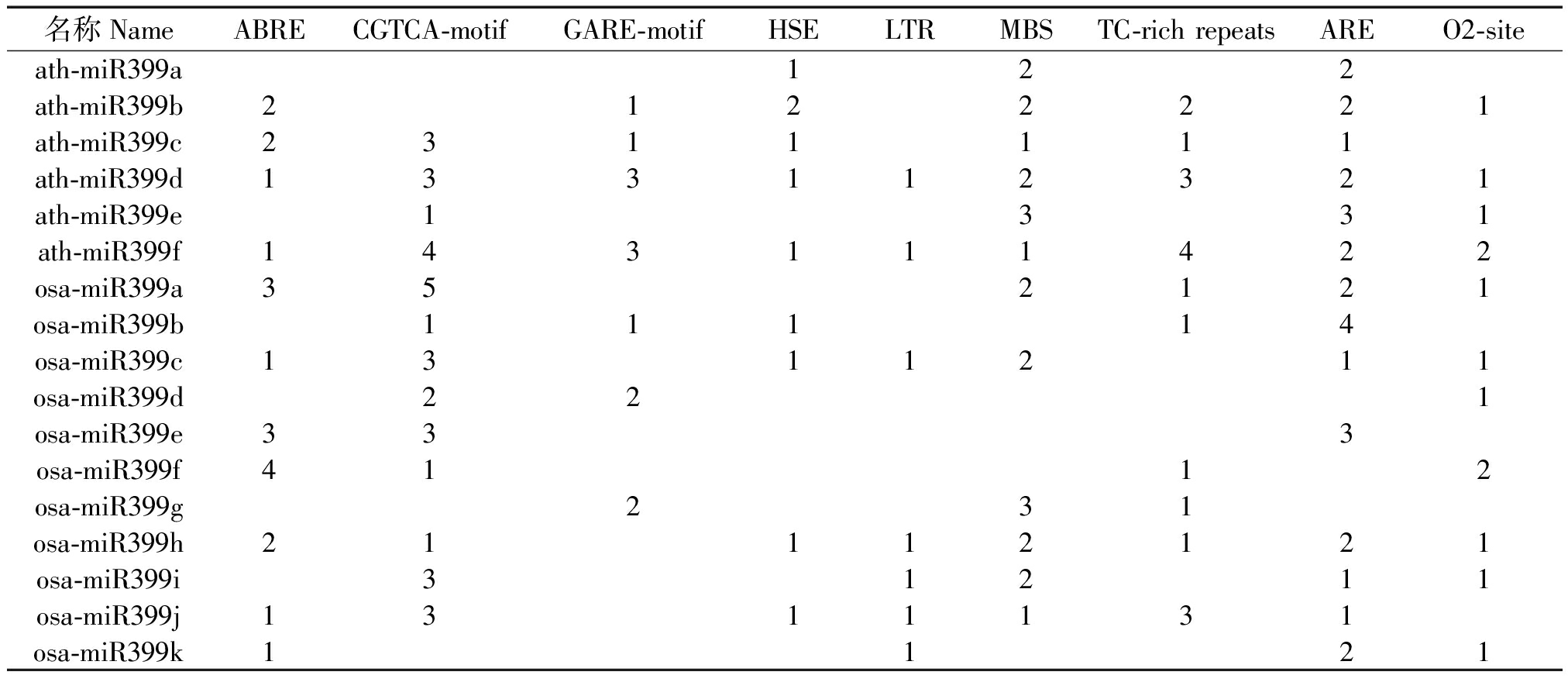
名称NameABRECGTCA-motifGARE-motifHSELTRMBSTC-rich repeatsAREO2-siteath-miR399a122ath-miR399b2122221ath-miR399c2311111ath-miR399d133112321ath-miR399e1331ath-miR399f143111422osa-miR399a352121osa-miR399b11114osa-miR399c1311211osa-miR399d221osa-miR399e333osa-miR399f4112osa-miR399g231osa-miR399h21112121osa-miR399i31211osa-miR399j1311131osa-miR399k1121
注:ABRE.脱落酸响应元件;CGTCA-motif.茉莉酸响应元件;GARE-motif.赤霉素响应元件;HSE.参与热激反应的顺式作用元件;LTR.参与低温反应的顺式作用元件;MBS.干旱响应MYB结合位点;TC-rich repeats.防御和胁迫相关响应元件;ARE.厌氧诱导的顺式作用元件;O2-site.参与玉米醇溶蛋白代谢调控的顺式作用元件。
Note: ABRE.Cis-acting element involved in the abscisic acid responsiveness; CGTCA-motif.Cis-acting regulatory element involved in the MeJA-responsiveness;GARE-motif.Gibberellin-responsive element; HSE.Cis-acting element involved in heat stress responsiveness;LTR.Cis-acting element involved in low-temperature responsiveness; MBS.MYB binding site involved in drought-inducibility;TC-rich repeats.Cis-acting element involved in defense and stress responsiveness;ARE.Cis-acting regulatory element essential for the anaerobic induction; O2-site.Cis-acting regulatory element involved in zein metabolism regulation.
2.5 植物miR399家族靶基因分析
miRNA通过特异性剪切靶标mRNA、阻止靶标mRNA翻译等方式调控基因表达[9]。预测并分析靶基因,对于深入了解miR399家族功能特征及其进化规律具有重要意义。本研究利用PMRD和psRNATarget数据库对拟南芥和水稻miR399家族靶基因进行预测,分析发现拟南芥和水稻miR399分别调控了11,22个靶基因。利用AGRiGO数据库对水稻miR399家族靶基因的基因本体进行分析(图5),发现其功能涉及生物学过程、细胞组分及分子功能3大类共7个小类。生物学过程分类中,胞内过程类型基因占比最大,比水稻该类基因本底提高了15.1%,其次为代谢过程;细胞组分分类中,细胞和细胞部分类别占比均为59.1%,其次为细胞器类型,其中,细胞类型比水稻该类基因本底降低了5.2%;分子功能分类中,催化活性和结合类型占比分别为40.9%,45.5%,分别比水稻该类基因本底提高了1.5%,1.4%。
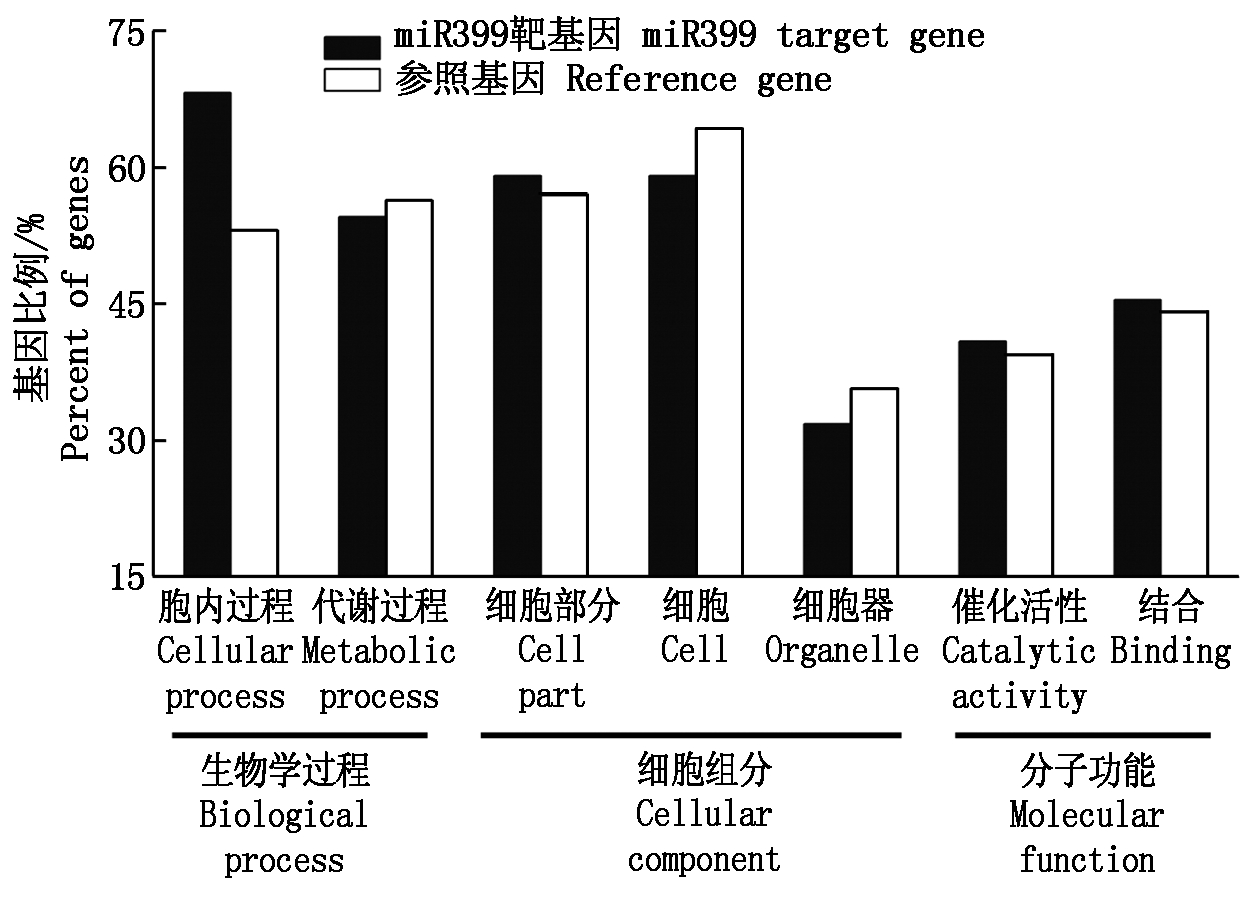
图5 水稻miR399家族靶基因基因本体分析
Fig.5 Gene ontology analysis of rice miR399 target genes
拟南芥miR399家族调控靶基因主要包括泛素联结酶、磷酸盐转运蛋白、丝氨酸/苏氨酸蛋白激酶、钠胆汁酸转运蛋白等基因(表3),这些基因主要参与了RNA合成和代谢调控、转录调控等过程。水稻miR399家族调控靶基因主要为磷酸盐转运蛋白、丝氨酸/苏氨酸蛋白激酶、类受体激酶2、DnaJ结构域蛋白、泛素联结酶、抗病蛋白、反转录转座子蛋白、WW结构域蛋白等基因(表3),这些基因主要参与了植物磷代谢、应激响应、基因表达调控等多项进程。
3 结论与讨论
miRNAs是一类参与植物生长、发育和应激反应的非编码小分子调节因子[10],在植物抵御环境胁迫过程中发挥作用,因此,miRNAs的鉴定和功能分析对植物抗逆新品种选育具有重要意义。传统的miRNAs研究需要构建小RNA文库、Northern Blot、qRT-PCR等一系列复杂的试验步骤,在实际研究中存在诸多困难。随着基因组学和测序技术的发展,计算方法被广泛应用于生物学研究中,利用多种计算软件和方法,对不同物种miRNA及其靶基因进行预测和分析[11],这些研究大大缩减了科研工作量,使得科研工作更加快速、高效地开展。本研究利用miRBase数据库对植物miR399家族成员进行系统分析,搜索到32个植物物种的244个成熟miR399,说明该家族miRNA在大部分植物物种中普遍存在,但在各物种中的分布数量很不均衡。一半以上的pre-miR399长度为101~140 nt,极大部分成熟miR399为21 nt,说明该家族成员进化上具有较高的保守性。
表3 植物miR399家族预测靶基因信息
Tab.3 Information of predicted target genes of plant miR399 family
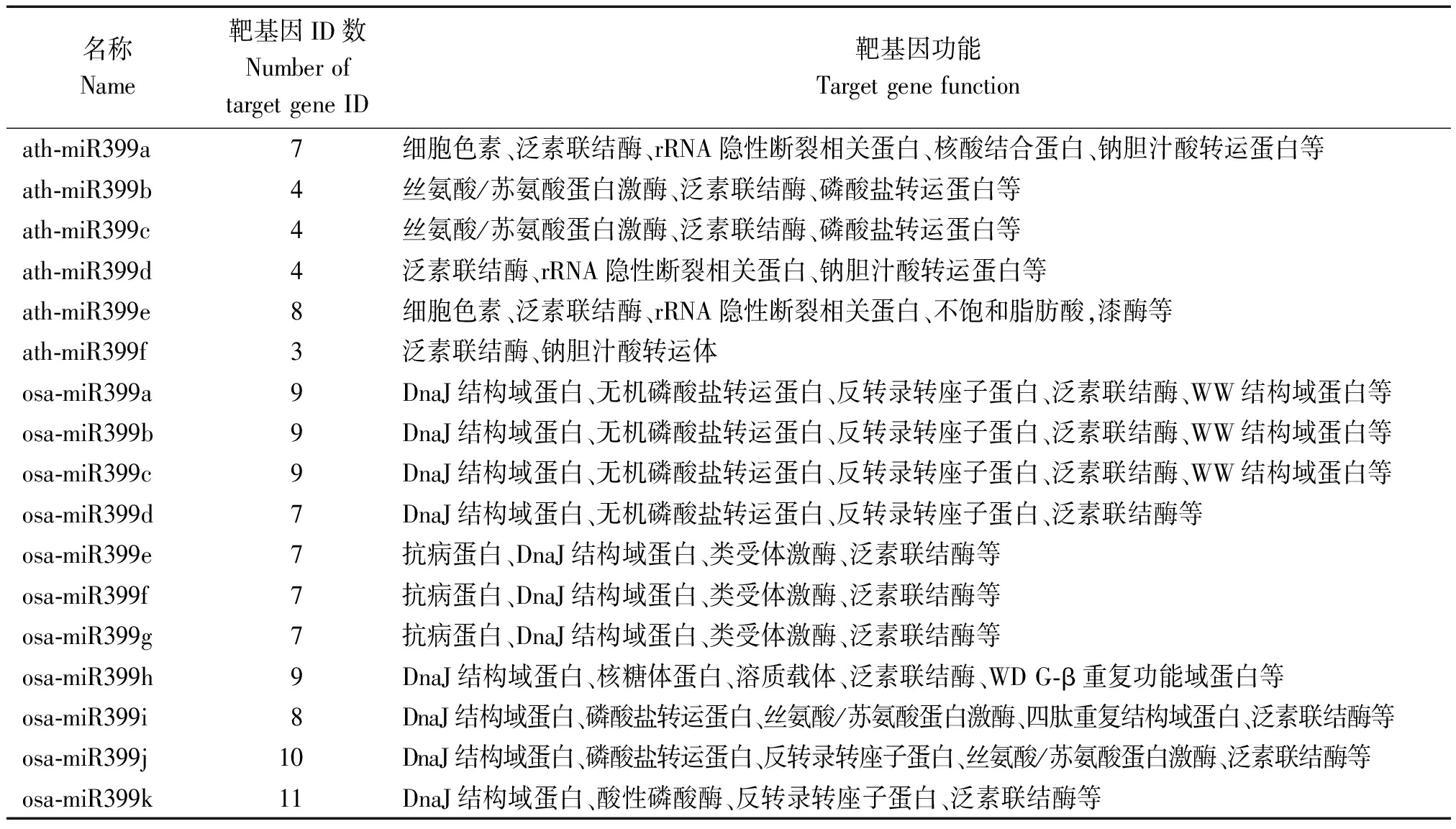
名称Name靶基因ID数Number of target gene ID靶基因功能Target gene functionath-miR399a7细胞色素、泛素联结酶、rRNA隐性断裂相关蛋白、核酸结合蛋白、钠胆汁酸转运蛋白等ath-miR399b4丝氨酸/苏氨酸蛋白激酶、泛素联结酶、磷酸盐转运蛋白等ath-miR399c4丝氨酸/苏氨酸蛋白激酶、泛素联结酶、磷酸盐转运蛋白等ath-miR399d4泛素联结酶、rRNA隐性断裂相关蛋白、钠胆汁酸转运蛋白等ath-miR399e8细胞色素、泛素联结酶、rRNA隐性断裂相关蛋白、不饱和脂肪酸,漆酶等ath-miR399f3泛素联结酶、钠胆汁酸转运体osa-miR399a9DnaJ结构域蛋白、无机磷酸盐转运蛋白、反转录转座子蛋白、泛素联结酶、WW结构域蛋白等osa-miR399b9DnaJ结构域蛋白、无机磷酸盐转运蛋白、反转录转座子蛋白、泛素联结酶、WW结构域蛋白等osa-miR399c9DnaJ结构域蛋白、无机磷酸盐转运蛋白、反转录转座子蛋白、泛素联结酶、WW结构域蛋白等osa-miR399d7DnaJ结构域蛋白、无机磷酸盐转运蛋白、反转录转座子蛋白、泛素联结酶等osa-miR399e7抗病蛋白、DnaJ结构域蛋白、类受体激酶、泛素联结酶等osa-miR399f7抗病蛋白、DnaJ结构域蛋白、类受体激酶、泛素联结酶等osa-miR399g7抗病蛋白、DnaJ结构域蛋白、类受体激酶、泛素联结酶等osa-miR399h9DnaJ结构域蛋白、核糖体蛋白、溶质载体、泛素联结酶、WD G-β重复功能域蛋白等osa-miR399i8DnaJ结构域蛋白、磷酸盐转运蛋白、丝氨酸/苏氨酸蛋白激酶、四肽重复结构域蛋白、泛素联结酶等osa-miR399j10DnaJ结构域蛋白、磷酸盐转运蛋白、反转录转座子蛋白、丝氨酸/苏氨酸蛋白激酶、泛素联结酶等osa-miR399k11DnaJ结构域蛋白、酸性磷酸酶、反转录转座子蛋白、泛素联结酶等
分析pre-miRNA二级结构,对了解成熟miRNA的剪切和折叠具有重要意义。pre-miR399在5′和3′端均有较保守序列,其中3′端序列保守性较强。大部分植物miR399位于pre-miRNA的3′端,除部分pre-miR399能产生5′和3′端2条成熟miR399外,极少miR399位于5′端。进化分析发现,除玉米外,该研究的6个物种miR399序列一致性较高,在植物种间的保守性较强,且不同种属间进化分组存在交叉。
植物miRNA通过序列互补的方式与靶基因结合,从而导致靶基因的降解或功能失活。基于该原则,对miRNA靶基因进行预测分析,对深入研究miRNA的功能具有重要指导意义。研究表明,miR399参与了植物对干旱、高盐和低磷胁迫的响应[10,12-13]。AtMYB2通过调控miR399表达,提高了拟南芥对磷元素的吸收[14]。Hvu-miR399受低磷诱导上调表达,调控其靶基因HvPHO2下调表达,并释放出磷转运蛋白PHO1和PHT1s,促进磷酸盐的吸收和转运[15]。Zma-miR399通过调控其靶基因ZmPHT1;3、ZmPHT1;8和ZmPHO2的表达,影响了玉米对低磷胁迫的耐受性[16]。Osa-miR399参与了水稻对多种营养饥饿胁迫的调控[17]。上述研究表明,miR399在植物生长发育和抵御环境胁迫过程中发挥了重要作用。该研究中,miR399家族靶基因功能分析显示,ath-miR399a-f均调控了泛素联结酶E2基因AT2G33770,泛素链接酶E2基因表达受植物中磷的调控,参与了磷酸盐饥饿响应,介导了PHO1和PHT1s在内膜中的降解[18-20]。ath-miR399b和ath-miR399c均调控了PHT1蛋白编码基因AT3G54700,PHT1为磷酸盐转运蛋白,在植物中发挥磷素转运功能;ath-miR399a、ath-miR399d和ath-miR399e均调控了RH39蛋白编码基因AT4G09730,RH39为DEAD盒蛋白,参与叶绿体23S rRNA的隐性断裂,突变体大大降低了核糖1,5-二磷酸羧化酶/加氧酶的水平[21]。osa-miR399a~osa-miR399k均调控了DnaJ功能域包含蛋白和泛素连接酶,DnaJ功能域包含蛋白属于热激蛋白家族,在多种生物进程中发挥作用[22],OsPHO2编码E2泛素联结酶,并且作为磷饥饿响应信号途径中的重要成员,间接负调控下游的多个无机磷转运体[23-24]。osa-miR399a-d、osa-miR399a-i、osa-miR399a-j调控了无机磷酸盐转运蛋白OsPT6[25]。多个miR399成员调控了同一类靶基因,说明该家族存在冗余现象,对细胞响应多种信号和细胞功能的稳定具有重要作用。
Pre-miR399启动子区含有多个激素和胁迫响应元件,miR399家族多个靶基因在磷饥饿响应、磷转运和信号转导等过程中起作用,表明植物miR399家族在植物响应环境胁迫中发挥作用,单个miR399成员对胁迫环境的响应模式及对靶基因的具体调控机制值得进一步深入研究。
[1] Ha M, Kim V N.Regulation of microRNA biogenesis[J].Nature Reviews Molecular Cell Biology,2014,15(8):509-524.doi:10.1038/nrm3838.
[2] Jones-Rhoades M W, Bartel D P, Bartel B. MicroRNAs and their regulatory roles in plants[J]. Annual Review of Plant Biology, 2006, 57: 19-53. doi: org/10.1146/annurev.arplant.57.032905.105218.
[3] Lauressergues D, Couzigou J M, Clemente H S, Martinez Y, Dunand C, Bécard G, Combier J P.Primary transcripts of microRNAs encode regulatory peptides[J].Nature,2015,520(7545):90-93.doi:10.1038/nature14346.
[4] Chien P S, Chiang C B, Wang Z R, Chiou T J.MicroRNA-mediated signaling and regulation of nutrient transport and utilization[J].Current Opinion in Plant Biology,2017,39:73-79.doi:10.1016/j.pbi.2017.06.007.
[5] Kuo H F, Chiou T J.The role of microRNAs in phosphorus deficiency signaling[J].Plant Physiology,2011,156(3):1016-1024.doi:10.1104/pp.111.175265.
[6] Liu T Y, Lin W Y, Huang T K, Chiou T J.MicroRNA-mediated surveillance of phosphate transporters on the move[J].Trends in Plant Science,2014,19(10):647-655.doi:10.1016/j.tplants.2014.06.004.
[7] Bologna N G, Schapire A L, Zhai J, Chorostecki U, Boisbouvier J, Meyers B C, Palatnik J F.Multiple RNA recognition patterns during microRNA biogenesis in plants[J].Genome Research,2013,23(10):1675-1689.doi:10.1101/gr.153387.112.
[8] Park M Y, Wu G, Gonzalez-Sulser A, Vaucheret H, Poethig R S.Nuclear processing and export of microRNAs in Arabidopsis[J].Proceedings of the National Academy of Sciences of the United States of America,2005,102(10):3691-3696.doi:10.1073/pnas.0405570102.
[9] Krol J, Loedige I, Filipowicz W.The widespread regulation of microRNA biogenesis, function and decay[J].Nature Reviews Genetics,2010,11(9):597-610.doi:10.1038/nrg2843.
[10] Baek D, Chun H J, Kang S, Shin G, Park S J, Hong H, Kim C, Kim D H, Lee S Y, Kim M C, Yun D J.A role for Arabidopsis miR399f in salt, drought, and ABA signaling[J].Molecules and Cells,2016,39(2):111-118.doi:10.14348/molcells.2016.2188.
[11] Lu Y, Yang X.Computational identification of novel MicroRNAs and their targets in Vigna unguiculata[J].Comparative and Functional Genomics,2010.doi:10.1155/2010/128297.
[12] 陶平. miR399调控大豆磷平衡及开花的初步研究[D]. 广州: 华南农业大学, 2016.
Tao P. Studies on the roles of miR399 in Phosphorus homeostasis and flowering time in soybean[D].Guangzhou:South China Agricultural University, 2016.
[13] Huen A K,Rodriguez-Medina C,Ho A Y Y,Atkins C A,Smith P M C.Long-distance movement of phosphate starvation-responsive microRNAs in Arabidopsis[J].Plant Biology,2017,19(4):643-649. doi: org/10.1111/plb.12568.
[14] Baek D, Kim M C, Chun H J, Kang S, Park H C, Shin G, Park J, Shen M, Hong H, Kim W Y, Kim D H, Lee S Y, Bressan R A, Bohnert H J, Yun D J.Regulation of miR399f transcription by AtMYB2 affects phosphate starvation responses in Arabidopsis[J].Plant Physiology,2013,161(1):362-373.doi:10.1104/pp.112.205922.
[15] Hackenberg M, Shi B J, Gustafson P, Langridge P.Characterization of phosphorus-regulated miR399 and miR827 and their isomirs in barley under phosphorus-sufficient and phosphorus-deficient conditions[J].BMC Plant Biology,2013,13(1):214.doi:10.1186/1471-2229-13-214.
[16] 王开. ZmmiR399在玉米适应低磷胁迫中的分子机制研究[D]. 北京: 中国农业科学院, 2017.
Wang K. Molecular mechanism of ZmmiR399 to low phosphate stress in maize[D]. Beijing: Chinese Academy of Agricultural Sciences, 2017.
[17] Hu B, Wang W, Deng K, Li H, Zhang Z, Zhang L, Chu C.MicroRNA399 is involved in multiple nutrient starvation responses in rice[J].Frontiers in Plant Science,2015,6:188.doi:10.3389/fpls.2015.00188.
[18] 陈晓婷, 刘军, 孙翠霞, 林桂芳. 草酸胁迫下拟南芥三个差异表达microRNA的分析[J]. 农业生物技术学报, 2014, 22(4): 432-439. doi: 10.3969/j.issn.1674-7968.2014.04.005.
Chen X T, Liu J, Sun C X, Lin G F. Three differentially expressed microRNAs in Arabidopsis thaliana under the stress of oxalic acid[J]. Journal of Agricultural Biotechnology, 2014,22(4):432-439.
[19] Balyan S C, Mutum R D, Kansal S, Kumar S, Mathur S, Raghuvanshi S. Insights into the small RNA-mediated networks in response to abiotic stress in plants[M].New York: Springer, 2015: 45-91.
[20] Yuan J, Zhang Y, Dong J, Sun Y, Lim B L, Liu D, Lu Z J.Systematic characterization of novel lncRNAs responding to phosphate starvation in Arabidopsis thaliana[J].BMC Genomics,2016,17(1):655.doi:10.1186/s12864-016-2929-2.
[21] Nishimura K, Ashida H, Ogawa T, Yokota A.A DEAD box protein is required for formation of a hidden break in Arabidopsis chloroplast 23S rRNA[J].The Plant Journal: for Cell and Molecular Biology,2010,63(5):766-777.doi:10.1111/j.1365-313X.2010.04276.x.
[22] Liberek K, Georgopoulos C.Autoregulation of the Escherichia coli heat shock response by the DnaK and DnaJ heat shock proteins[J].Proceedings of the National Academy of Sciences of the United States of America,1993,90(23):11019-11023. doi: org/10.1073/pnas.90.23.11019.
[23] Cai H M, Xie W B, Zhu T, Lian X M.Transcriptome response to phosphorus starvation in rice[J].Acta Physiologiae Plantarum,2012,34(1):327-341.doi:10.1007/s11738-011-0832-7.
[24] 应颖慧. 水稻磷信号重要因子OsPHO2的互作蛋白的鉴定和功能分析[D]. 杭州: 浙江大学, 2013.
Ying Y H. Identification and characterization of proteins interacting with OsPHO2,a major component of phosphate signaling pathway in rice[D].Hangzhou:Zhejiang University, 2013.
[25] Wu P, Xu G, Lian X. Nitrogen and phosphorus uptake and utilization[M]. New York: Springer,2013: 217-226.