藏绵羊是中国最古老的绵羊品种和三大粗毛绵羊品种之一,繁育在3 000~6 000 m气候寒冷、氧分压低的青藏高原地区。在漫长的适应性进化过程中,藏绵羊在生理、生化和形态学上具有了稳定适应高原低氧环境的遗传学特性,逐渐在形态、细胞和分子水平上形成了适应和调节机制,是适应青藏高原低氧环境的优势物种[1]。
脯氨酸羟化酶(Proline hydroxylase, PHDs)是一类双加氧酶,主要参与调控低氧诱导因子及低氧反应[2]。有PHD1、PHD2、PHD3和PHD4 4种亚型[3],PHD2(Proline hydroxylase domain protein 2,PHD2)即脯氨酸羟化酶2,又名EGLN1(Egl nine homolog 1)基因。绵羊EGLN1基因位于其25号染色体上,有7个外显子,长约5 kb,编码661个氨基酸[4]。EGLN1基因是一重要的氧气传感基因[5],具有负调控低氧诱导因子1a(Hypoxia-inducible factor-1a, HIF-1a)的作用,对低氧环境较为敏感,能够调控低氧环境条件下HIF-1a的表达,是其蛋白降解的主要因子[6]。EGLN1基因表达的降低可减少HIF-1a的降解,可形成HIF-1a的蓄积,使HIF-1a诱导的低氧反应基因的转录对低氧环境适应反应的细胞进行调控,如血管内皮生长因子(Vascular endothelial growth factor,VEGF)、血红素氧化酶-1(Heme oxygenase-1, HO-1)、诱导型一氧化氮合酶(Induced nitric oxide synthase, iNOS)、促红细胞生成素(Erythropoietin, EPO)等,从而提高细胞和组织对低氧的耐受力,使细胞较好的适应低氧环境,从而达到低氧适应形成对细胞的保护。
EGLN1基因的变异分析可有效研究动物的高原低氧适应性进化。利用全基因组关联分析(Genome wide association study, GWAS)方法对藏族人进行检测分析,结果发现,EGLN1基因是藏族与汉族人基因频率差异最大的基因,他们与藏族血红蛋白浓度以及红细胞计数呈显著相关,并直接参与了藏族的低氧适应进化过程[7]。EGLN1 基因在人的跨种族基因检测中表现出了很强的自然选择性[8],与蒙古人血红蛋白含量具有显著的相关性[9],以及慢性高原病[10]、高原水肿[11]和红细胞增多症[12]等均具有相关性。
截至目前, 对EGLN1基因的研究对象主要以世居高原上的土著居民为主,如EGLN1基因与藏族人群的高原性疾病、癌症、高原环境的适应等方面研究。以海拔高度不同进行的遗传特征差异以及基因表达差异研究鲜有报道。因此,本试验以藏绵羊为研究对象,低海拔湖羊为对照,采用qRT-PCR技术和PCR-SSCP方法分析EGLN1基因在高低海拔绵羊品种间、不同组织间的表达差异以及遗传变异特征与低氧适应的相关性。进而为藏绵羊高原低氧适应性机制以及耐低氧分子标记筛选提供基础。
1 材料和方法
1.1 试验样品
样品包括340只藏绵羊和249只湖羊血样(表1),用于基因组DNA提取。部分血液滴至FTA卡(Whatman Bioscience, Middlesex, UK)上,自然晾干后保存备用。组织样分别采集3只藏绵羊和3只湖羊的心、肝、脾、肺、肾和骨骼肌组织,立即置于液氮中,带回实验室-70 ℃冻存备用。
表1 绵羊基因组样品
Tab.1 The sheep′s genome sample

群体Group地点Location形式Sample form数量/只No.藏绵羊Tibetan sheep甘肃省玛曲县FTA340湖羊Hu sheep浙江省临安县FTA249
1.2 试验方法
1.2.1 FTA卡基因组DNA提取 FTA卡基因组提取采用两步法[13]。
1.2.2 总RNA提取 采用RNA simple Total RNA Kit试剂盒(北京天根)提取各组织的总RNA。利用Fast King RT Kit(With gDNase)试剂盒将总RNA反转录成cDNA,-20 ℃保存备用。
1.3 荧光定量引物设计与扩增
根据GenBank中绵羊EGLN1基因CDS序列(No:XM_012156646.2),利用在线Primer 6.0软件设计特异引物,内参基因18S参考文献[14]引物(P1和P2),引物序列见表2,引物合成由华大基因科技有限公司完成。利用Applied Biosystems Q6实时荧光定量PCR仪进行基因表达量测定,总反应体系为20 μL,包括荧光定量PCR预混液(AceQ® qPCR SYBR® Green Master Mix)10.0 μL、ROX Reference Dye1 0.4 μL、上下游引物各0.4 μL、模板cDNA 2.0 μL、灭菌蒸馏水6.8 μL,每个组织重复3个样品检测。机设条件为:预变性,95 ℃,5 min;变性,95 ℃,10 s,退火,60 ℃,30 s,延伸,72 ℃,30 s,40个循环;熔解反应为,变性95 ℃,15 s;然后60 ℃,60 s。
1.4 绵羊EGLN1基因PCR-SSCP检测
依据NCBI数据库中已有的绵羊EGLN1基因序列(NC_019482.2),利用Primer 6.0设计特异引物P3(表2),扩增intron6-intron7部分区域。
反应体系为20 μL:包括Taq预混酶10 μL(Vazyme),ddH2O 7.6 μL,引物0.8 μL和DNA模板0.8 μL。
PCR反应条件为:预变性,95 ℃,5 min;变性,95 ℃,30 s,退火,62 ℃,30 s,延伸,72 ℃,30 s,35个循环;延伸,72 ℃,7 min;4 ℃保存。利用1.5%琼脂糖凝胶对PCR产物进行电泳检测。
SSCP检测:取PCR产物2.5 μL,加入变性缓冲液7.5 μL混匀,在105 ℃进行加热变性5 min,然后置冰水混合物中。在14%的非变性聚丙烯酰胺凝胶(Acr∶Bis=37.5∶1)和0.5×的TBE液中电泳,外循环温度为15 ℃(恒温)的条件下,220 V电泳20 h。电泳结束后,将非变性聚丙烯酰胺凝胶进行银染显色,判型。
表2 引物序列
Tab.2 Primers sequence
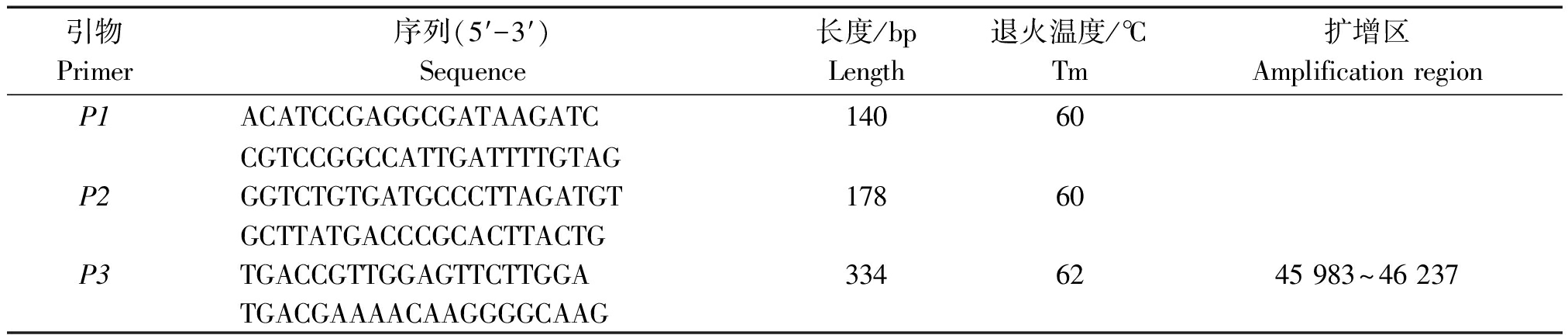
引物Primer序列(5′-3′)Sequence长度/bpLength退火温度/℃Tm扩增区Amplification regionP1ACATCCGAGGCGATAAGATC14060CGTCCGGCCATTGATTTTGTAGP2GGTCTGTGATGCCCTTAGATGT17860GCTTATGACCCGCACTTACTGP3TGACCGTTGGAGTTCTTGGA3346245 983~46 237TGACGAAAACAAGGGGCAAG
1.5 测序与数据分析
根据银染结果,确定SSCP带型,将纯合型样品直接测序,杂合型则进行切胶测序[15]。采用MEGA 5.0软件对核苷酸序列进行比较,利用Popgene 32.0软件对基因型频率和等位基因频率进行统计,然后对序列纯合度(Homozygosity, Ho)、杂合度(Heterozygosity, He)、有效等位基因数(Effective number of alleles, Ne)进行计算,并进行χ2检验分析,采用PIC 6.0软件对样品多态性信息含量(Polymorphism information content, PIC)进行计算。
采用2-ΔΔCT法[16]计算EGLN1基因mRNA的表达情况,ΔCT和ΔΔCT计算采用以下公式计算;ΔCT=CTEGLN1-CT18S,ΔΔCT=ΔCT试验组-ΔCT对照组,肾的表达数据为对照。应用SPSS 16.0软件对试验数据进行方差分析。
2 结果与分析
2.1 绵羊EGLN1基因组织差异表达分析
2.1.1 荧光定量PCR熔解曲线 绵羊EGLN1基因和内参基因18S的熔解曲线如图1所示,两者均为单一波峰,可知荧光定量PCR扩增产物特异,可进行定量表达测定分析。
2.1.2 绵羊EGLN1基因表达量测定 藏绵羊和湖羊2个品种的EGLN1基因组织表达量测定结果如图2所示。结果表明,在2个绵羊品种中,心、肝、肺、脾、肾和骨骼肌中均有表达。其中,肝在湖羊各组织中表达量最高,分别是心、脾、肺和肾和骨骼肌表达量的9.7,15.4,2.8,29.7和7.5倍;在藏绵羊中表达量最高的为脾,是心、肝、肺、肾和骨骼肌的1.7,3.4,2.1,1.3和1.1倍。2个品种相同组织间,湖羊各组织表达量均高于藏绵羊。尤其在心、肝和肺组织中最为明显。
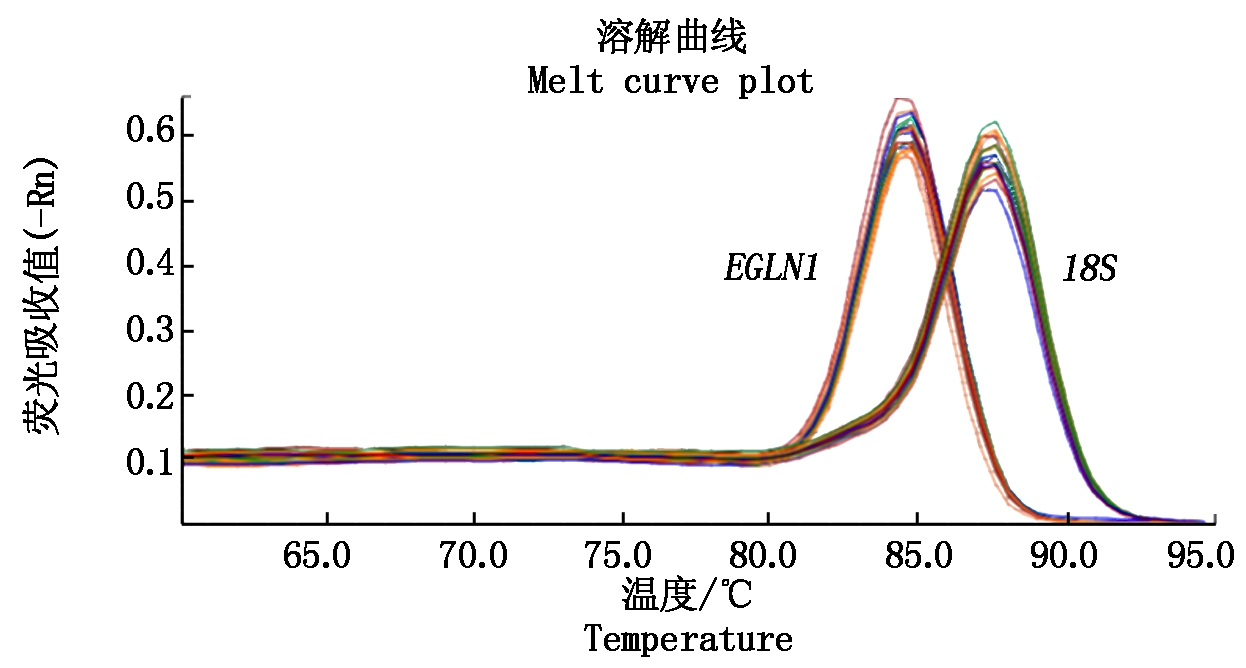
图1 EGLN1基因和18S rRNA熔解曲线
Fig.1 EGLN1 gene and 18S rRNA fluorescence melt curve plot
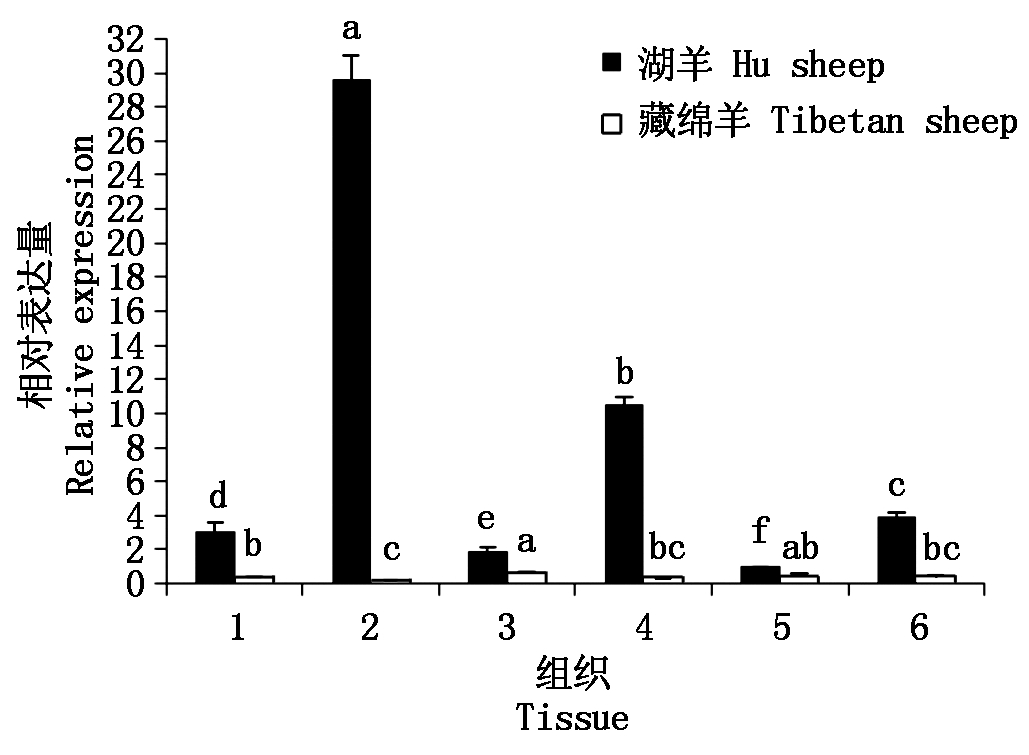
1.心脏;2.肝脏;3.脾脏;4.肺脏;5.肾脏;6.骨骼肌;湖羊肾为对照。
1. Heart;2. Liver;3. Spleen;4. Lung; 5. Kidney;6. Skeletal muscle; Kidney of Hu sheep represents control group.
图2 绵羊不同组织EGLN1基因相对表达量
Fig.2 Expression level histogram of EGLN1 gene in different tissues
2.2 EGLN1基因变异特征分析
2.2.1 SSCP检测结果 经SSCP检测,在藏绵羊和湖羊EGLN1基因intron6-intron7部分区域共发现4个(A~D)等位基因,形成AA、BB、CC、AB、AC、BC、AD、BD、CD共9种基因型(图3)。其中在湖羊群体中未发现BD基因型。序列分析表明,扩增区域共发现6个SNPs(c.1921-125A>G、c.1921-122A>G、c.1921-73C>A、c.1895G>A、c.1921+33G>A、c.1921+85T>G)以及1个碱基插入(c.1921-72插入G)和1个碱基缺失位点(c.1921-44缺失T)。变异位点c.1895G>A位于第6外显子区,未引起氨基酸的改变,属于同义突变。
2.2.2 群体遗传特征 藏绵羊和湖羊的遗传变异特征分析见表3,4。结果显示,藏绵羊和湖羊的优势等位基因分别为等位基因B和A,藏绵羊优势基因型为AB,湖羊优势基因型为AA,其中,在湖羊中未检测到基因型BD。经χ2适应性检验,2个绵羊群体均未达到显著水平(P>0.05)(表4),表明2个绵羊群体EGLN1基因处于Hardy-Weinberg平衡。
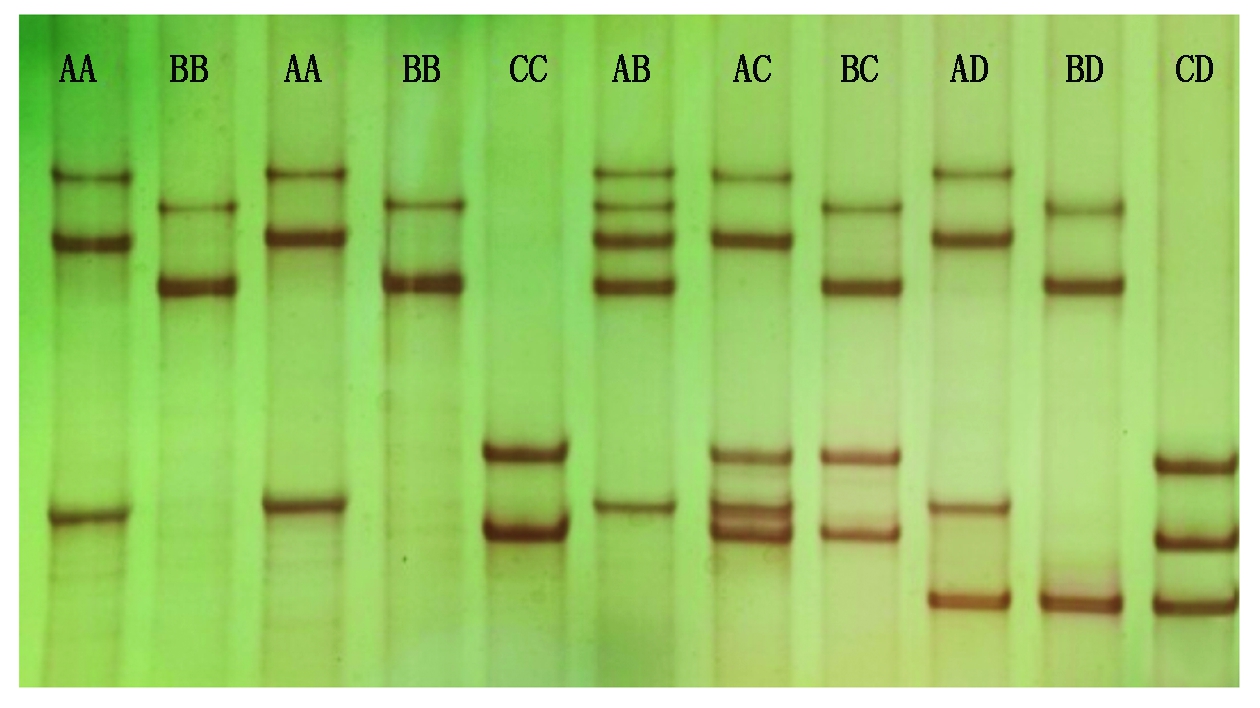
图3 绵羊EGLN1基因intron6-intron7区PCR-SSCP检测结果
Fig.3 Detection the region of intron6-intron7 of
EGLN1 gene in sheep by PCR-SSCP
表3 绵羊EGLN1基因的基因型频率和基因频率
Tab.3 Genotype frequencies and gene frequencies of EGLN1 gene in sheep
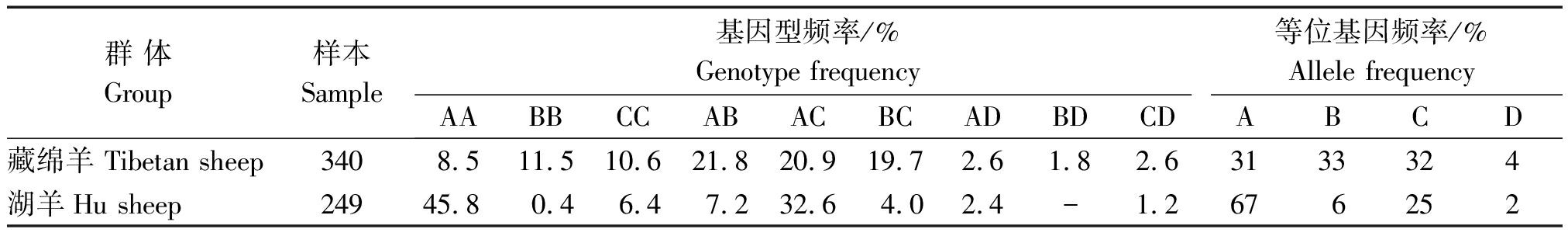
群 体Group样本Sample基因型频率/%Genotype frequency等位基因频率/%Allele frequencyAABBCCABACBCADBDCDABCD藏绵羊 Tibetan sheep3408.511.510.621.820.919.72.61.82.63133324湖羊Hu sheep24945.80.46.47.232.64.02.4-1.2676252
2个绵羊群体的遗传变异参数见表4。藏绵羊的多态信息含量为0.63(PIC>0.5),属于高度多态;湖羊的多态信息含量为0.42,属于中度多态。
表4 绵羊EGLN1基因的遗传变异参数
Tab.4 Genetic variation parameters of EGLN1 gene in sheep
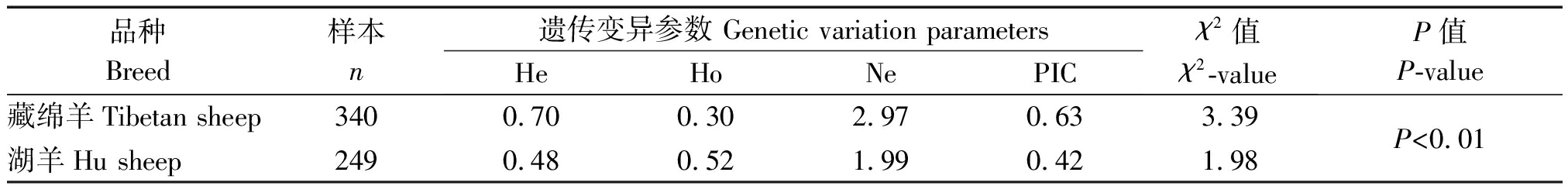
品种Breed样本n遗传变异参数 Genetic variation parametersHeHoNePICχ2值χ2-valueP值P-value藏绵羊Tibetan sheep3400.700.302.970.633.39湖羊Hu sheep2490.480.521.990.421.98P<0.01
注:PIC>0.5高度多态;PIC<0.25低度多态;P.表示2个群体间基因型频率差异。
Note: PIC>0.5 means high diversity; PIC<0.25 means low diversity; P.Indicating the difference in genotype frequencies between the two populations.
3 结论与讨论
3.1 绵羊EGLN1基因组织表达分析
高原低氧适应是指生活在高原的人及土著动物,为生存和适应高原低氧环境,所形成的可遗传的表型特征,以及生理生化方面的变化。EGLN1基因是动物低氧诱导通路上的关键调控基因,在其适应高原低氧环境上具有重要作用。在低氧条件下,EGLN1基因的羟化活性明显降低,从而使HIF-1α和HIF-2α累积,进而特异的增强其下游靶基因VEGF、EPO及VEGFR、乳酸脱氢酶A(Lactate dehydrogenase A,LDHA)、甘油醛-3-磷酸脱氢酶(Glyceral dehyde-3-Phosphate dehydrogenase,GAPDH)、内皮素-1(Endothelin-1,ET-1)、血红素氧化酶(Hemeoxygenase,HO)等基因的表达,进而参与机体低氧适应的代谢过程,增强生物体对高寒低氧的适应性[17-19]。
本研究qRT-PCR检测结果显示,EGLN1基因在藏绵羊和湖羊2个品种的不同组织器官中均有表达,但表达具有组织特异性,且与其他动物的相应研究结果相似[20-21]。同时,EGLN1基因的表达具有品种特异性,藏绵羊和湖羊分别为繁育在高海拔与低海拔的品种。2个品种间,藏绵羊各组织表达量均低于湖羊,但在心、肝、肾组织中差异更为显著,表明EGLN1基因的表达与海拔高度明显相关。研究表明,心、肺、肾是动物机体呼吸、内分泌调节以及信号传递等过程的主要调节器官。肝是机体新陈代谢的主要器官,在维持三大营养物质平衡中发挥着重要作用,如肝糖原调节血糖浓度稳定[22]。这些组织器官在动物的适应性方面所发挥的作用进一步说明,EGLN1基因与动物的高原低氧适应性具有相关性。叶银子[23]研究发现,EGLN1基因在不同海拔高度的沙蜥蜴心、肝中的表达量存在显著差异(P<0.01),如低海拔沙蜥蜴肝中表达量极显著高于(P<0.01)高海拔沙蜥蜴。但在心脏中差异不显著,与上述研究不一致,可能与研究物种不同有关。EGLN1基因对低氧诱导因子具有负调控作用,而藏绵羊EGLN1基因表达量下降,则可说明降低了对低氧诱导因子的抑制,增强其基因的表达量,发挥其在动物低氧适应过程中的重要作用,使动物机体能够更好地适应高原低氧环境。
3.2 绵羊EGLN1基因群体遗传及适应性特征
本研究在藏绵羊EGLN1基因intron6-intron7区发现了6个SNPs、1个插入和1个缺失位点,以及相应的4个等位基因。其中,1个SNPs位于外显子区,属于同义突变,其余均位于内含子上。内含子是基因上的一个重要原件,对基因表达进行双向调控,其发生突变可能会改变基因的表达效率[24-25]。结果显示:EGLN1基因在2个品种间的基因型和等位基因频率具有统计学意义(P<0.05),推测这种差异可能与其生活环境有关。由于藏绵羊繁育在青藏高原低氧环境,EGLN1基因可能在进化过程中受到了自然选择作用,使有利于高寒低氧适应的基因型(AB、BB、BC)受到选择并富集。李骞等[26]研究表明,在EGLN1基因5′UTR区和内含子区有6个SNPs在高原藏族群体和低海拔汉族群体之间存在显著差异,说明6个SNPs位点可能与藏族高原低氧适应性有关。对藏绵羊和湖羊EGLN1基因的研究也得出相似的结果,发现第4内含子中的2个SNPs的纯合型个体在2个绵羊间差异极显著(P<0.01),推测与藏绵羊的高原低氧环境适应相关[27],与本研究结果一致。根据PIC相关标准[28],藏绵羊EGLN1基因的PIC值是0.63,大于0.5,呈高度多态,表明藏绵羊该基因遗传变异程度较高,多态性也较为丰富,则选择潜力较大。因此,藏绵羊EGLN1基因遗传多样性丰富,具有作为高原低氧分子标记的潜力和优势。
EGLN1基因在藏绵羊和湖羊的6个不同组织均存在不同程度的表达。藏绵羊各组织表达量低于湖羊,尤其在心、肝、肺组织中。表现出明显的品种特异性以及组织特异性,这可能与2个品种的生活环境相关。在EGLN1基因intron6-intron7区检测到6个SNPs、1个碱基插入和1个碱基缺失,说明藏绵羊EGLN1基因遗传多样性丰富,且其优势等位基因B和优势基因型AB的频率极显著高于湖羊(P<0.01),具有作为高原低氧分子标记的优势和潜力。
[1] 唐晓惠, 强巴央宗. 藏绵羊对高原低氧环境的适应性及抗病性相关基因研究进展[J]. 畜牧与兽医, 2017, 49(2): 115-118.
Tang X H, Qiang B Z. Research progress in Tibetan sheep adaptability to hypoxia and disease-related genes[J]. Animal Husbandry & Veterinary Medicine, 2017,49(2):115-118.
[2] Nunomiya A, Shin J, Kitajima Y, Dan T, Miyata T, Nagatomi R.Activation of the hypoxia-inducible factor pathway induced by prolyl hydroxylase domain 2 deficiency enhances the effect of running training in mice[J].Acta Physiologica,2017,220(1):99-112.doi:10.1111/apha.12751.
[3] Appelhoff R J, Tian Y M, Raval R R, Turley H, Harris A L, Pugh C W, Ratcliffe P J, Gleadle J M.Differential function of the prolyl hydroxylases PHD1, PHD2, and PHD3 in the regulation of hypoxia-inducible factor[J].Journal of Biological Chemistry,2004,279(37):38458-38465.doi:10.1074/jbc.M406026200.
[4] Dupuy D, Aubert I, Duperat V G, Petit J, Taine L, Stef M, Bloch B, Arveiler B M.Characterization,and expression analysis of theSM-20 human homologue,c1orf12,and identification of a novel related gene,SCAND2[J]. Genomics, 2000, 69:348-354. doi: 10.1006/geno.2000.6343.
[5] Kiss J, Kirchberg J, Schneider M.Molecular Oxygen sensing: implications for visceral surgery[J].Langenbeck′s Archives of Surgery,2012,397(4):603-610.doi:10.1007/s00423-012-0930-z.
[6] Semenza G L.Hypoxia-inducible factor 1 (HIF-1) pathway[J].Science′s STKE,2007(407):cm8.doi:10.1126/stke.4072007cm8.
[7] Hu H, Petousi N, Glusman G, Yu Y, Bohlender R, Tashi T, Downie J M, Roach J C, Cole A M, Lorenzo F R, Rogers A R, Brunkow M E, Cavalleri G, Hood L, Alpatty S M, Prchal J T, Jorde L B, Robbins P A, Simonson T S, Huff C D.Evolutionary history of Tibetans inferred from whole-genome sequencing[J].PLoS Genetics,2017,13(4):e1006675.doi:10.1371/journal.pgen.1006675.
[8] Bigham A, Bauchet M, Pinto D, Mao X, Akey J M, Mei R, Scherer S W, Julian C G, Wilson M J, L pez Herr ez D, Brutsaert T, Parra E J, Moore L G, Shriver M D.Identifying signatures of natural selection in Tibetan and Andean populations using dense genome scan data[J].PLoS Genetics,2010,6(9):e1001116.doi:10.1371/journal.pgen.1001116.
[9] Xing J C, Wuren T, Simonson T S, Watkins W, Witherspoon D J, Wu W, Qin G, Huff C D, Jorde L B, Ge R L.Genomic analysis of natural selection and phenotypic variation in High-Altitude mongolians[J].PLoS Genetics,2013,9(7):1003634.doi:10.1371/journal.pgen.1003634.
[10] Tashi T, Scott Reading N, Wuren T, Zhang X, Moore L G, Hu H, Tang F, Shestakova A, Lorenzo F, Burjanivova T, Koul P, Guchhait P, Wittwer C T, Julian C G, Shah B, Huff C D, Gordeuk V R, Prchal J T, Ge R.Gain-of-function EGLN1 prolyl hydroxylase (PHD2 D4E:C127S) in combination with EPAS1 (HIF-2α) polymorphism lowers hemoglobin concentration in Tibetan highlanders[J].Journal of Molecular Medicine (Berlin, Germany),2017,95(6):665-670.doi:10.1007/s00109-017-1519-3.
[11] Mishra A, Mohammad G, Thinlas T, Pasha M A.EGLN1 variants influence expression and SaO2 levels to associate with high-altitude pulmonary oedema and adaptation[J].Clinical Science,2013,124(7):479-489.doi:10.1042/CS20120371.
[12] Xu J, Yang Y Z, Tang F, Ga Q, Tana W, Ge R L.EPAS1 gene polymorphisms are associated with high altitude polycythemia in tibetans at the Qinghai-Tibetan plateau[J].Wilderness & Environmental Medicine,2015,26(3):288-294.doi:10.1016/j.wem.2015.01.002.
[13] Zhou H, Hickford J G, Fang Q.A two-step procedure for extracting genomic DNA from dried blood spots on filter paper for polymerase chain reaction amplification[J].Analytical Biochemistry,2006,354(1):159-161.doi:10.1016/j.ab.2006.03.042.
[14] 刘芳. 藏羚羊STAT3,HIF-1α 和HIF-2α基因表达的生物学意义[D]. 西宁: 青海大学医学院, 2012.
Liu F. Biological significance of signal transducer and activator of transcription 3, hypoxia inducible factor 1 alpha and hypoxia inducible factor 2 alpha in High Altitude Hypoxic Adaptation Species-Tibetan antelope[D]. Xining: Qinghai University Medical College,2012.
[15] Gong H, Zhou H, Hickford J G.Diversity of the glycine/tyrosine-rich keratin-associated protein 6 gene (KAP6) family in sheep[J].Molecular Biology Reports,2011,38(1):31-35.doi:10.1007/s11033-010-0074-6.
[16] Livak K J, Schmittgen T D.Analysis of relative gene expression data using Real-time quantitative PCR and the 2-ΔΔCT method[J].Methods,2001,25(4):402-408. doi: 10.1006/meth.2001.1262.
[17] Rodriguez J, Herrero A, Li S, Rauch N, Quintanilla A, Wynne K, Krstic A, Acosta J C, Taylor C, Schlisio S, Von Kriegsheim A.PHD3 regulates p53 protein stability by hydroxylating proline 359[J].Cell Reports,2018,24(5):1316-1329.doi:10.1016/j.celrep.2018.06.108.
[18] Aggarwal S, Gheware A, Agrawal A, Ghosh S, Prasher B, Mukerji M, Indian Genome Variation Consortium.Combined genetic effects of EGLN1 and VWF modulate thrombotic outcome in hypoxia revealed by Ayurgenomics approach[J].Journal of Translational Medicine,2015,13:184.doi:10.1186/s12967-015-0542-9.
[19] Hu K, Babapoor-Farrokhran S, Rodrigues M, Deshpande M, Puchner B, Kashiwabuchi F, Hassan S J, Asnaghi L, Handa J T, Merbs S, Eberhart C G, Semenza G L, Montaner S, Sodhi A.Hypoxia-inducible factor 1 upregulation of both VEGF and ANGPTL4 is required to promote the angiogenic phenotype in uveal melanoma[J].Oncotarget,2016,7(7):7816-7828.doi:10.18632/oncotarget.6868.
[20] Han D, Wen L, Chen Y.Molecular cloning of phd1 and comparative analysis of phd1, 2, and 3 expression in Xenopus laevis[J].The Scientific World Journal,2012:689287.doi:10.1100/2012/689287.
[21] Huang M, Paglialunga S, Wong J M, Hoang M, Pillai R, Joseph J W.Role of prolyl hydroxylase domain proteins in the regulation of insulin secretion[J].Physiological Reports,2016,4(5):12722.doi:10.14814/phy2.12722.
[22] 陈杰. 家畜生理学[M]. 北京: 中国农业出版社, 2012:55-98.
Chen J. Livestock physiology[M]. Beijing: China Agriculture Press, 2012: 55-98.
[23] 叶银子. 蜥蜴低氧适应的分子机制:西藏沙蜥和草原沙蜥的比较研究[D]. 杭州: 杭州师范大学, 2016.
Ye Y Z. The molecular mechanisms of hypoxia adaption in lizards: a comparsion between the Bald′s Toad-headed agama (Phrynocephalus theobaldi) and the steppe toad-headed agama (Phrynocephalus frontalis)[D]. Hangzhou: Hangzhou Normal University,2016.
[24] 张开慧. 内含子的功能及应用[J]. 中国畜牧兽医, 2012, 39(7): 80-83. doi:10.3969/j.issn.1671-7236.2012.07.019.
Zhang K H. Intron function and application[J]. China Animal Husbandry & Veterinary Medicine, 2012,39(7):80-83. doi:10.3969/j.issn.1671-7236.2012.07.019.
[25] 张恩宇, 罗玉柱, 李少斌, 王继卿, 刘秀, 闫伟, 管生栋, 徐洪福. 甘肃高山细毛羊及其杂种羊GH基因第3内含子多态性与生长性状的相关性[J]. 华北农学报, 2015, 30(5): 71-76. doi:10.7668/hbnxb.2015.05.012.
Zhang E Y, Luo Y Z, Li S B, Wang J Q, Liu X, Yan W, Guan S D, Xu H F. Correlation ship investigation of GH gene intron 3 polymorphism with growth traits in Gansu alpine merino and crossbred sheep[J]. Acta Agriculturae Boreali-Sinica, 2015,30(5):71-76. doi:10.7668/hbnxb.2015.05.012.
[26] 李骞, 刘舒媛, 林克勤, 孙浩, 于亮, 黄小琴, 褚嘉祐, 杨昭庆. EGLN1基因6个单核苷酸多态性与高海拔低氧适应的相关性[J]. 遗传, 2013, 35(8): 992-998.
Li Q, Liu S Y, Lin K Q, Sun H, Yu L, Huang X Q, Chu J Y, Yang Z Q.Association between six single nucleotide polymorphisms of EGLN1 gene and adaptation to high-altitude hypoxia[J]. Hereditas, 2013,35(8):992-998.
[27] Geng R Q, Wang L P.The PCR-SSCP and DNA sequencing methods detecting genetic mutations of EGLN1 gene in different sheep breeds[J].Indian Journal of Animal Research,2015,49(1):44-47.doi:10.5958/0976-0555.2015.00009.6.
[28] Botstein D, White R L, Skolnick M, Davis R W.Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J].American Journal of Human Genetics,1980,32(3):314-331.