光周期是影响植物生长发育及生态适应性的重要因素[1]。在Garner等[2]发现光周期反应现象之后,有关植物光周期敏感性研究的报道不断增加。如唐志明等[3]认为,株高不同的水稻与光温敏感性的大小有关,研究结果发现株高越高的水稻光温敏感性也越强。王翠玲等[4]对热带、亚热带玉米光周期敏感性进行了研究,为克服我国玉米种质资源的局限性提供了方案。杨玉杰等[5]研究了植物种子的萌芽、幼苗的生长、开花以及根系生长与光周期间的关系,表明光周期对植物生长发育存在全方位的影响。此外,对小麦[6]、芝麻[7]等作物的生长、结实与光周期之间的联系也进行了研究。
谷子是常见的食用杂粮,最早发现于黄河流域,在我国已有悠久的种植历史。由于谷子属于短日照喜温作物,对光温比较敏感,导致其生态适应性狭窄,生产上缺少跨区的主栽品种。目前对谷子光周期敏感性的研究较少,赵治海等[8]利用遗传学的方法在292杂交后代中选育出了光(温)敏不育系821,发现该不育材料在长日照下是低敏不育的,在育性转换方面适合种植在北方春谷区常年气候条件,不育性稳定,利用面广。河南科技大学农学院与河北省农林科学院谷子研究所合作开展研究,前期已经对谷子主要农艺性状的光温敏感性进行了系统评价,发现抽穗期、穗长、穗码数对光周期较敏感,其次是叶片数,这4个性状适合作为谷子光周期敏感性评价的指标[9-10]。国内外学者利用谷子开花期作为指标性状,利用经典的杂交群体作图法将光周期敏感性位定位于3,4,9号染色体上[11-12]。为检测到新的光周期敏感性位点,进一步揭示谷子光周期敏感性的遗传基础,本研究在海南、洛阳2个不同光周期条件下调查了45份来自不同生态区谷子品种的抽穗期,用两地抽穗日数相对差值计算光周期敏感性,用40个SSR标记对45份谷子材料进行多态性扫描,并进行标记与谷子光周期敏感性的关联分析,发掘与光周期敏感性关联的SSR标记,并在关联标记区域进行候选基因分析。
1 材料和方法
1.1 试验材料
45份谷子材料见表1,分别于2015年5月中旬、10月底种植在河南省洛阳市河南科技大学开元校区试验田、海南省乐东县河北省农林科学院谷子研究所试验田,2015年6月-2016年1月在两地进行抽穗期调查。
表1 试验所用谷子品种材料
Tab.1 The foxtail millet cultivars used in this study
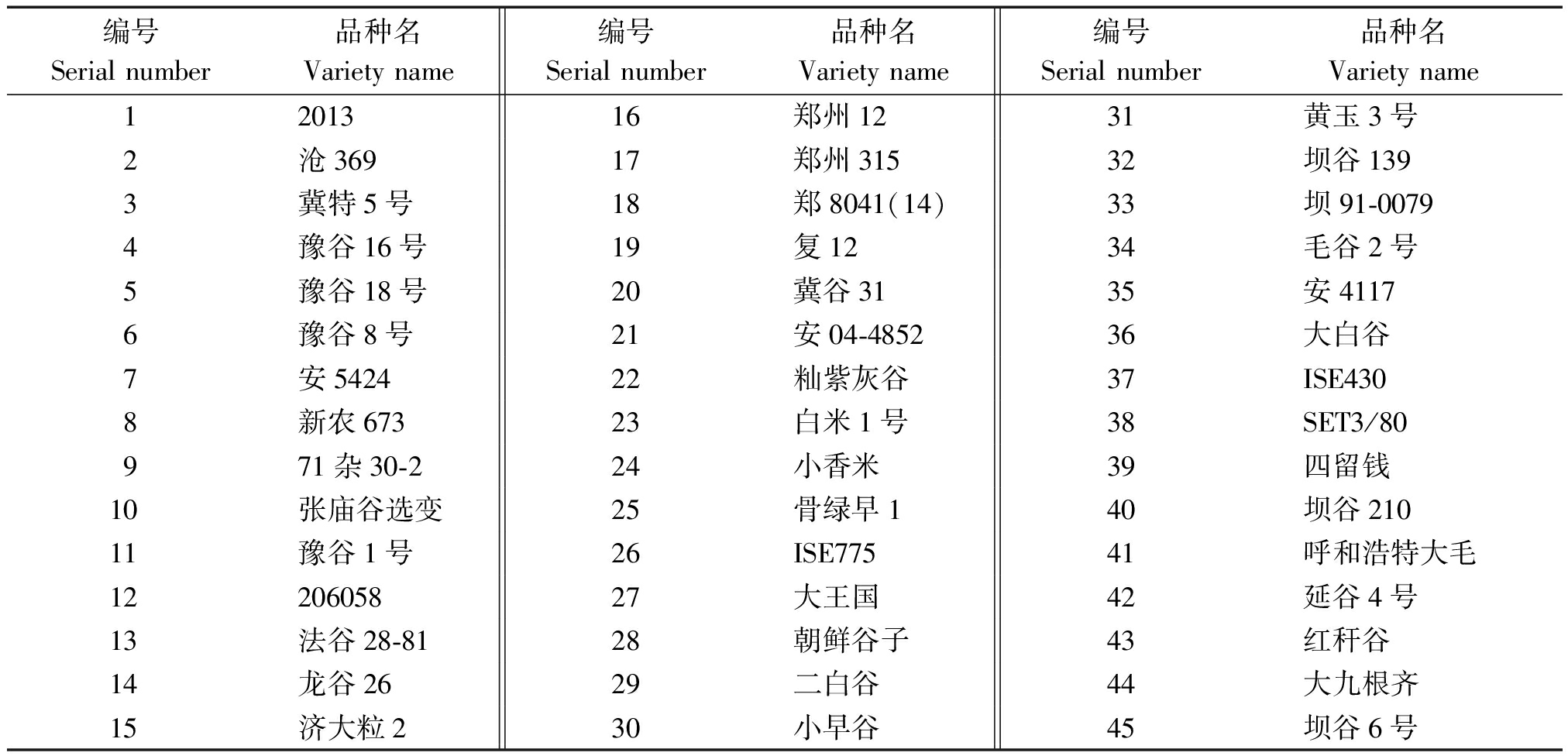
编号Serial number品种名Variety name编号Serial number品种名Variety name编号Serial number品种名Variety name1201316郑州1231黄玉3号2沧36917郑州31532坝谷1393冀特5号18郑8041(14)33坝91-00794豫谷16号19复1234毛谷2号5豫谷18号20冀谷3135安41176豫谷8号21安04-485236大白谷7安542422籼紫灰谷37ISE4308新农67323白米1号38SET3/80971杂30-224小香米39四留钱10张庙谷选变25骨绿早140坝谷21011豫谷1号26ISE77541呼和浩特大毛1220605827大王国42延谷4号13法谷28-8128朝鲜谷子43红秆谷14龙谷2629二白谷44大九根齐15济大粒230小早谷45坝谷6号
1.2 试验设计与农艺指标调查
45份谷子材料按照行距40 cm、株距3~5 cm、保护行35 cm进行种植,管理方法按当地常规管理。记录出苗期、抽穗期的方法见文献[13-14]。穗日数=抽穗期-出苗期,每个品种的光周期敏感性=(洛阳长日照抽穗日数-海南短日照抽穗日数)/短日照抽穗日数×100%。
1.3 谷子DNA的提取及PCR检测
采用2×CTAB法提取谷子DNA。40个SSR标记信息来自文献[15](表2)。PCR扩增体系(10 μL):基因组DNA为1 μL,上下游引物各0.5 μL,2×Power Taq PCR Master Mix 为5 μL,ddH2O(灭菌) 为3 μL。PCR扩增程序:94 ℃预变性4 min;94 ℃变性35 s,55~59 ℃退火30 s,72 ℃延伸1.5 min,34个循环;72 ℃延伸5 min。
表2 本研究所用的40个SSR标记信息
Tab.2 The information of 40 SSR markers used in this study

引物名称Primer name重复域Repeat motif引物序列Primer sequence退火温度/℃Annealing temperature预期片段大小/bpExpected sizep3(GT)34F:GCAGAAAGCATGCCGTAGTC57198R:GCTTGGAGTCCACATGGATAGp5(CA)27F:CTTCCCTCCCTCCCTGAC55182R:CTGAGCTGAGCTGCCTTTGp8(AC)26F:CGATCGAATGATCGATGAAC55203R:CCCTTTGTCCGATCACGTCp10(GT)21F:CAATCACATCCGAGCATTTC55198R:CACCCACCGTGTTGATCTGp12X(AC)29F:ACGAGTCACAAATCACAGCAC59224R:ATGCCTGAGCGGAACGGAAp13(CA)6F:GGAGAGATTCCGGGCTCTAGT57166R:ACGGTTCCGACATTTTAACGp56(CA)24F:GATGTGTACGGGTTGCATTG55199R:TGGGTTTCAGGGCTCTCTCp59(AC)22F:TAATTTTGTGGCGTGGGATG57176R:GCACTGGTTTTGTTGAATGGp61(CA)17F:CATCCGCGTCATCTGAATC56200R:ACCTGCTGCTATCCATCACCp91(GT)23F:AGCTGTGCTCCTCTGATCTTG57181R:TAACGTGGGGATGCACTAGCp98(GT)13F:ATTCATCAGTAGCACAGC55155R:TGGAACTAAGAACAGGAAACp6(GT)14F:AAGGATGGAATTTGCCACTG58188R:TTTCGACGATTTGCTTCAACb227(GA)26F:TGATCTGGCAGAACGAACA55156R:CAATTCCTGGACCAATATGCb233(GA)41F:GCCACGCACACCAACTT55226R:CTCCCGCAGAACACGCAb159(CT)24F:GCCAGTCCGAGATGGTTAAG57185R:AGCTCTAGCAGTTGGGGACAb101(CT)33F:ATCTAGGTGCCGATGCGT57264R:TGTGGGAAGAAGCTAGGGAAb187(CT)36F:TTGGACAAATGACGCTATGC59195R:CTGCATCAAATCAGGACCACb190(GA)25F:GAAATTTCACAAGTGTTGGTG55302R:TGATCGGAGCAGAGTGTTGAb234(CT)26F:GCCGCAACGAACAACCG55290R:CCTGTCCCTATCCCTGTCGb107(CT)25F:AGAACGAGGTGGTGTGTGG57253R:GGGTCTCACGCTCTCATCAb123(AC)19F:GGTGTTCTCCTGTGTGC58130R:AGAGTTATTTCCAGCATTAGTGb185(CT)30F:GCACGTGTGACTTTCCACAT55167R:GTGAATGGCACACGAAACTGb225(GA)28F:ACCAAGAACTGCCTGCAC55131R:TGCTTAGAACCCACTTGATCG
表2(续)
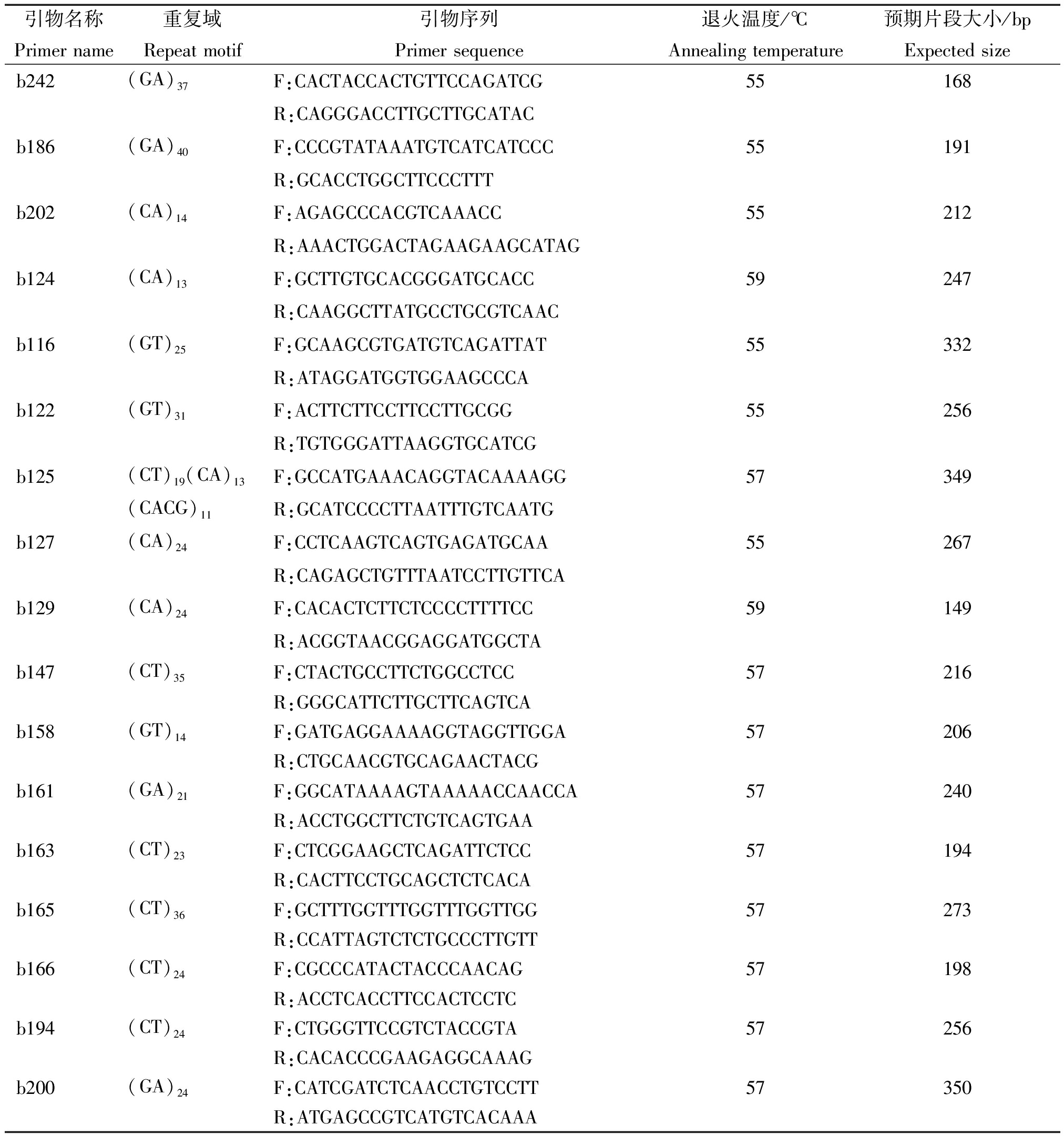
引物名称Primer name重复域Repeat motif引物序列Primer sequence退火温度/℃Annealing temperature预期片段大小/bpExpected sizeb242(GA)37F:CACTACCACTGTTCCAGATCG55168R:CAGGGACCTTGCTTGCATACb186(GA)40F:CCCGTATAAATGTCATCATCCC55191R:GCACCTGGCTTCCCTTTb202(CA)14F:AGAGCCCACGTCAAACC55212R:AAACTGGACTAGAAGAAGCATAGb124(CA)13F:GCTTGTGCACGGGATGCACC59247R:CAAGGCTTATGCCTGCGTCAACb116(GT)25F:GCAAGCGTGATGTCAGATTAT55332R:ATAGGATGGTGGAAGCCCAb122(GT)31F:ACTTCTTCCTTCCTTGCGG55256R:TGTGGGATTAAGGTGCATCGb125(CT)19(CA)13F:GCCATGAAACAGGTACAAAAGG57349(CACG)11R:GCATCCCCTTAATTTGTCAATGb127(CA)24F:CCTCAAGTCAGTGAGATGCAA55267R:CAGAGCTGTTTAATCCTTGTTCAb129(CA)24F:CACACTCTTCTCCCCTTTTCC59149R:ACGGTAACGGAGGATGGCTAb147(CT)35F:CTACTGCCTTCTGGCCTCC57216R:GGGCATTCTTGCTTCAGTCAb158(GT)14F:GATGAGGAAAAGGTAGGTTGGA57206R:CTGCAACGTGCAGAACTACGb161(GA)21F:GGCATAAAAGTAAAAACCAACCA57240R:ACCTGGCTTCTGTCAGTGAAb163(CT)23F:CTCGGAAGCTCAGATTCTCC57194R:CACTTCCTGCAGCTCTCACAb165(CT)36F:GCTTTGGTTTGGTTTGGTTGG57273R:CCATTAGTCTCTGCCCTTGTTb166(CT)24F:CGCCCATACTACCCAACAG57198R:ACCTCACCTTCCACTCCTCb194(CT)24F:CTGGGTTCCGTCTACCGTA57256R:CACACCCGAAGAGGCAAAGb200(GA)24F:CATCGATCTCAACCTGTCCTT57350R:ATGAGCCGTCATGTCACAAA
1.4 数据分析
1.4.1 谷子材料群体结构分析 用STRUCTURE 1.8软件对45份谷子材料进行群体遗传结构分析,确定最佳群体数K值,用Evanno等[16]的方法计算出ΔK,并选择适宜的K值。
1.4.2 SSR标记间连锁不平衡分析 用TASSEL 2.1软件估计SSR标记间的连锁不平衡情况,计算SSR成对位点间R2值和成对位点间LD的概率P值。
1.4.3 SSR标记与光周期敏感性的关联分析 用TASSEL 2.1软件中的GLM程序进行光周期敏感值与SSR标记的关联分析,并估计出统计检验的P值。
2 结果与分析
2.1 45份谷子材料光周期敏感性评价
45份谷子材料的光周期敏感值为0~1.917,其中郑8041(14)的光周期敏感值最大,为1.917,最敏感,其次为呼和浩特大毛谷,光周期敏感度值为1.667。光周期敏感值最小的为SET3/80,敏感度值为0。龙谷26、小早谷光周期敏感值也较小,均为0.417(表3)。
表3 谷子品种光周期敏感值
Tab.3 Photoperiod sensitivity values of foxtail millet cultivars
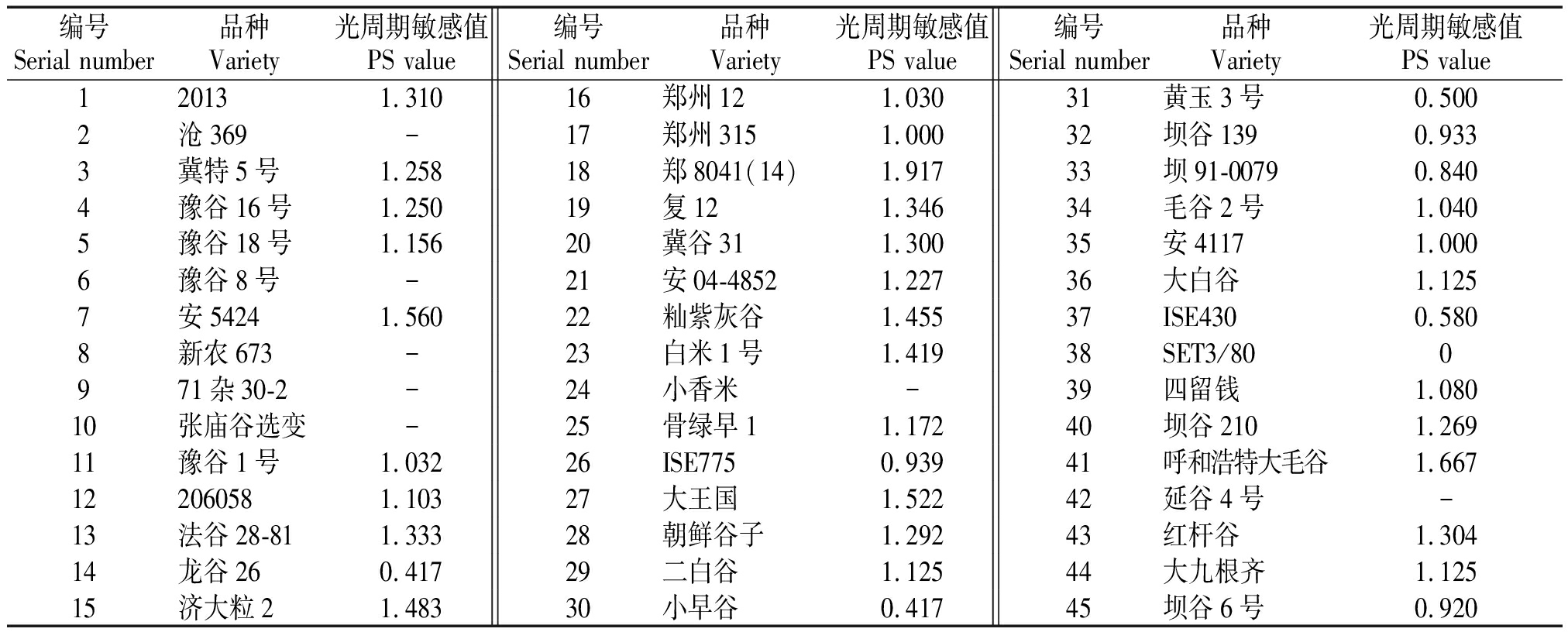
编号Serial number品种Variety 光周期敏感值PS value编号Serial number品种Variety光周期敏感值PS value编号Serial number品种Variety光周期敏感值PS value120131.31016郑州121.03031黄玉3号0.5002沧369-17郑州3151.00032坝谷1390.9333冀特5号1.25818郑8041(14)1.91733坝91-00790.8404豫谷16号1.25019复121.34634毛谷2号1.0405豫谷18号1.15620冀谷311.30035安41171.0006豫谷8号-21安04-48521.22736大白谷1.1257安54241.56022籼紫灰谷1.45537ISE4300.5808新农673-23白米1号1.41938SET3/800971杂30-2-24小香米-39四留钱1.08010张庙谷选变-25骨绿早11.17240坝谷2101.26911豫谷1号1.03226ISE7750.93941呼和浩特大毛谷1.667122060581.10327大王国1.52242延谷4号-13法谷28-811.33328朝鲜谷子1.29243红杆谷1.30414龙谷260.41729二白谷1.12544大九根齐1.12515济大粒21.48330小早谷0.41745坝谷6号0.920
2.2 SSR标记多态性检测
用40个SSR引物标记在45份谷子材料进行扩增,进行PAGE电泳,检测引物多态性。40个引物中检测到的等位基因个数为2~6,共检测出150个等位基因,平均每个引物检测等位基因数为3.75个。其中,p91、b158和p61引物检测出等位基因数最少,为2个;b200和b124引物检测等位基因数最多,为6个(表4和图1)。
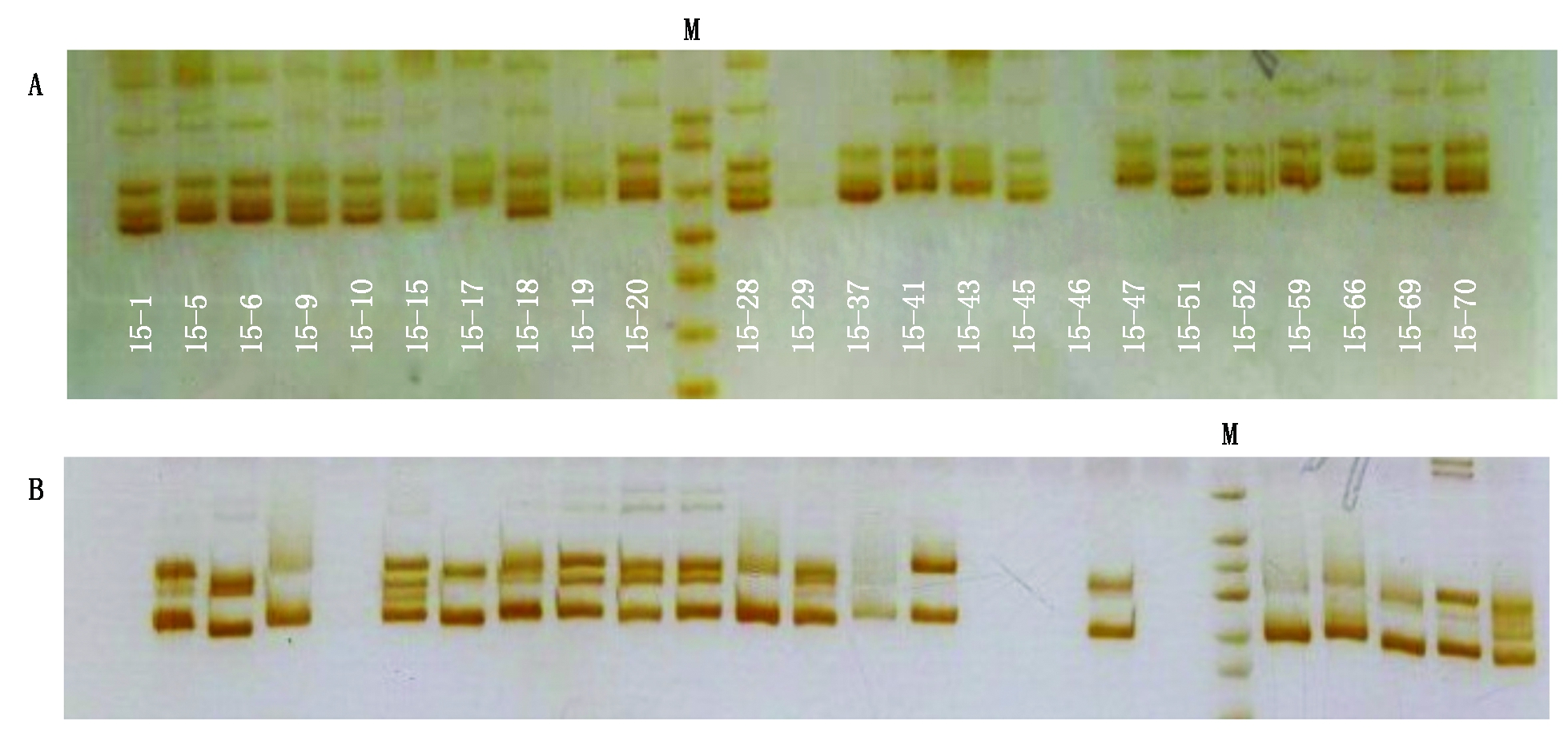
M. 50 bp lander。
图1 b200(A)、b124(B)在不同谷子品种中的扩增产物电泳
Fig.1 Electrophoresis results of amplification products of b200 (A) and b124 (B) among different foxtail millet cultivars
表4 40个SSR标记的等位基因数统计
Tab.4 Allele number statistics of 40 SSR markers
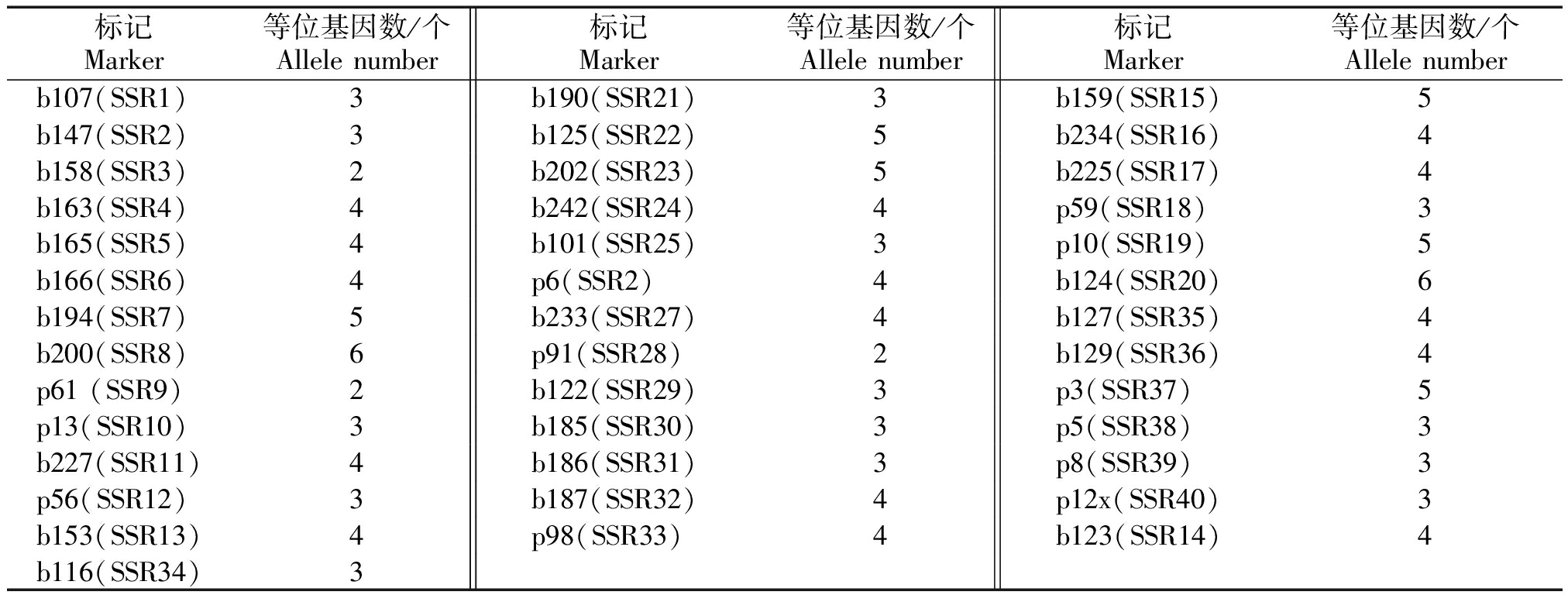
标记Marker等位基因数/个Allele number标记Marker等位基因数/个Allele number标记Marker等位基因数/个Allele numberb107(SSR1)3b190(SSR21)3b159(SSR15)5b147(SSR2)3b125(SSR22)5b234(SSR16)4b158(SSR3)2b202(SSR23)5b225(SSR17)4b163(SSR4)4b242(SSR24)4p59(SSR18)3b165(SSR5)4b101(SSR25)3p10(SSR19)5b166(SSR6)4p6(SSR2)4b124(SSR20)6b194(SSR7)5b233(SSR27)4b127(SSR35)4b200(SSR8)6p91(SSR28)2b129(SSR36)4p61 (SSR9)2b122(SSR29)3p3(SSR37)5p13(SSR10)3b185(SSR30)3p5(SSR38)3b227(SSR11)4b186(SSR31)3p8(SSR39)3p56(SSR12)3b187(SSR32)4p12x(SSR40)3b153(SSR13)4p98(SSR33)4b123(SSR14)4b116(SSR34)3
2.3 谷子群体遗传结构分析
群体结构分析确定谷子品种群体适宜的亚群数为3,结果见图2。从图中可以看出,有5个品种在第1亚群,分别来源于河北省、河南省。16个品种在第2亚群,分别来源于法国、河南省、河北省、山东省、黑龙江省、内蒙古自治区。21个品种在第3亚群,分别来源于朝鲜、德国、日本、美国、内蒙古自治区、黑龙江省、北京市、河北省、陕西省、河南省。剩余3个品种来自河南省、内蒙古自治区、德国,这3个品种分群模糊,组成混合组。
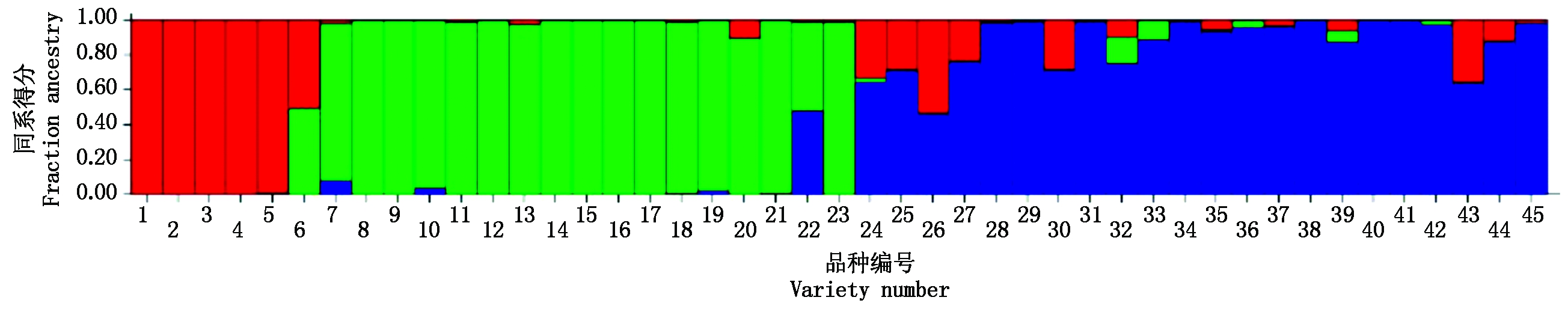
图2 每个谷子品种归属3个亚群的概率
Fig.2 Probability diagram of each cultivar belonging to three subgroups
2.4 SSR标记间的连锁不平衡分析
SSR标记间的连锁不平衡分析结果见图3。相关系数R2是位于对角线上方的三角形中,其相对应的P值位于对角线下方的三角形中。自然群体中用连锁不平衡系数R2越靠近1代表连锁关系越强,从图3可以看出,40个SSR标记间不存在明显的连锁不平衡现象。

黑色对角线上方的每一格表示SSR成对位点间R2值;对角线下方每一格表示成对位点LD的概率P值。
Each cell above the black diagonal represents the R2 value between the SSR paired sites; Cell below the diagonal represents probability P value of LD between the paired sites.
图3 40个SSR标记间LD分析结果
Fig.3 Linkage disequilibrium analysis result among 40 SSR markers
2.5 SSR标记与谷子光周期敏感性的关联分析
关联分析结果表明,在P<0.05的显著水平下共检测到2个与光周期敏感性相关联的SSR标记,即b200和b127,分别位于谷子的7号染色体和5号染色体(表5)。用这2个标记的引物序列搜索谷子基因组数据库(https://phytozome.jgi.doe.gov/pz/portal.html),发现这2个标记分别位于2个基因内部,其中b200位于登录号为Seita.7G284800的基因内部,该基因编码Myb类型的转录因子,根据基因组数据库提供的基因共表达信息,发现Myb转录因子与组蛋白及其修饰相关的基因存在共表达,如组蛋白H3基因(Seita.6G070000)、组蛋白-赖氨酸-氮-甲基转移酶基因EZH2(Seita.4G060900)、组蛋白-赖氨酸-氮-甲基转移酶基因SU(VAR)(Seita.3G095100)。此外Myb转录因子还与生长素应答蛋白14(Seita.9G162900)存在共表达,因此,推断所关联的基因是通过表观遗传学修饰的方式以及生长素信号传导的方式调控谷子光周期途径开花。而b127位于登录号为Seita.5G224300的基因内部,该基因编码一个功能未注释的蛋白DUF1644,根据数据库提供的基因表达信息,发现该基因在蓝光和远红光诱导条件下高度表达,与许多光周期响应蛋白存在相似性,推测该基因受光周期调控,与谷子光周期敏感性存在一定关系。
表5 与光周期敏感性关联的SSR位点及候选基因
Tab.5 The SSR loci and candidate genes associated with photoperiod sensitivity
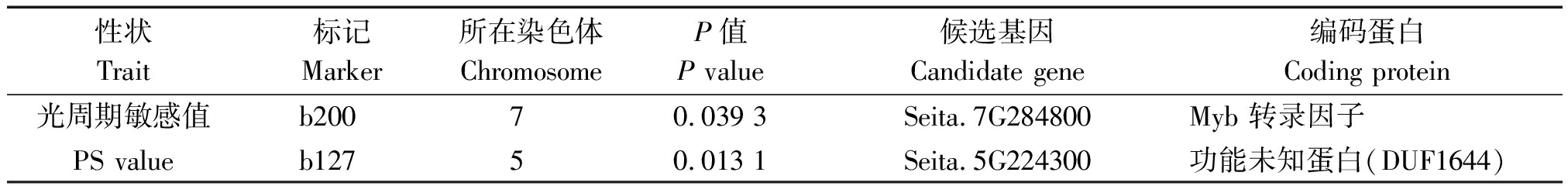
性状Trait标记Marker所在染色体ChromosomeP值P value候选基因Candidate gene编码蛋白Coding protein光周期敏感值b20070.039 3Seita.7G284800Myb 转录因子PS valueb12750.013 1Seita.5G224300功能未知蛋白(DUF1644)
3 结论与讨论
目前有关谷子光周期敏感性研究的报道较少,国内外学者利用杂交分离群体进行了谷子光周期敏感位点的QTL定位研究,发现在谷子4号染色体存在短日照条件表达的QTL位点,而在3,9号染色体检测到长日照条件下表达的QTL位点[11-12],进一步的研究发现,长日照(16 h)控制开花的机制与短日照(8~12 h)存在较大的差异[17]。这些研究都是基于双亲杂交分离群体进行的,能够检测到的变异位点有限,而基于自然群体的关联分析法能够克服杂交亲本变异位点有限的缺陷,检测到更多的基因位点。利用SSR标记和重测序开发的高密度SNP标记已经成功用于谷子主要农艺性状的全基因组关联分析研究[18-19],但是有关光周期敏感性的关联分析研究还未见报道。本研究利用SSR标记首次开展了谷子光周期敏感性的关联分析,在谷子5号染色体和7号染色体各检测到1个与光周期敏感性关联的SSR标记(b127与b200),并且这2个SSR位点均位于功能基因的内部,其中b127位于一个功能未注释的基因DUF1644内部,b200位于一个Myb转录因子基因内部,这2个基因在水稻、拟南芥光周期敏感性研究中还未报道过,因此,可能为新的光周期途径调控基因。
本研究在7号染色体关联到1个Myb转录因子基因,该基因与1个组蛋白基因H3、2个组蛋白修饰基因组蛋白-赖氨酸-氮-甲基转移酶基因EZH2、SU(VAR)存在共表达。在拟南芥开花调控研究中,发现组蛋白H3特定赖氨酸残基甲基化可以调节环境温度诱导的开花基因可变剪切体发生变化,从而影响开花期[20]。而在水稻的研究中,发现在长日照条件下抑制组蛋白赖氨酸甲基转移酶基因OsTrx1的表达可以延迟水稻开花[21]。因此,推测本研究所关联的Myb转录因子基因可能通过参与对组蛋白H3甲基化修饰来调控谷子开花。在5号染色体关联到的一个功能未注释基因DUF1644,该基因在蓝光和远红光诱导下高度表达。研究表明,拟南芥F-BOX基因FOA2受蓝光、远红光诱导表达,突变蓝光受体、红光受体及远红光受体则导致该基因光诱导表达效应减弱或消失,说明FOA2基因为光信号通路基因[22]。本研究获得的受蓝光、远红光诱导表达的DUF1644基因可能为光信号通路相关基因,参与光周期途径调控的谷子开花,但仍需进一步进行功能验证。
[1] 邓杰, 戴林建, 杨苏, 周田. 光温条件对烤烟生长发育和品质形成的影响综述[J]. 作物研究, 2015, 9(6): 678-681. doi: 10.3969/j.issn.1001-5280.2015.06.26.
Deng J, Dai L J, Yang S, Zhou T. Research progress on effect of light and temperature on the growth and quality of flue-cured tobacco[J]. Crop Research, 2015,9(6):678-681. doi: 10.3969/j.issn.1001-5280.2015.06.26.
[2] Garner W W, Allard H A. Effect of relative length of day and night and other factors of the environment on growth and reproduction in plants[J].J Agric Res,1920,18:553-606.
[3] 唐志明, 李晓方, 庄国富, 金晓春, 卢东柏. 水稻株高差异与光温生态敏感性的关系初探[J]. 广东农业科学, 2007 (4): 7-10. doi:10.3969/j.issn.1004-874X.2007.04.002.
Tang Z M, Li X F, Zhuang G F, Jin X C, Lu D B. Searching for the correlative factors of photosensitiveness and temperature sensibility in rice[J]. Guangdong Agricultural Sciences, 2007 (4):7-10. doi:10.3969/j.issn.1004-874X.2007.04.002.
[4] 王翠玲, 程芳芳, 孙朝晖, 库丽霞, 陈晓, 陈彦惠. 玉米光周期敏感性的遗传特性及相关基因的研究进展[J]. 玉米科学, 2008, 16(1): 11-14, 19.
Wang C L, Cheng F F, Sun C H, Ku L X, Chen X, Chen Y H. Advances in genetic research and related genes of photoperiod sensitivity in maize[J]. Journal of Maize Science, 2008,16(1):11-14, 19.
[5] 杨玉杰, 李海云. 光周期对植物生长发育的影响研究进展[J]. 安徽农业科学, 2010, 38(13): 6693-6694, 6740. doi:10.3969/j.issn.0517-6611.2010.13.026.
Yang Y J, Li H Y. Research progress on the effect of photoperiod on plant growth and development[J]. Journal of Anhui Agricultural Science, 2010,38(13):6693-6694, 6740. doi:10.3969/j.issn.0517-6611.2010.13.026.
[6] 沈韫赜, 郭双生. 受控环境下光周期对小麦生长发育、产量及营养品质的影响[J]. 麦类作物学报, 2015, 35(1): 64-70. doi:10.7606/j.issn.1009-1041.2015.01.10.
Shen Y Z, Guo S S. Effects of photoperiod on growth,development,yield and nutritional quality of wheat in controlled system[J]. Journal of Triticeae Crops, 2015,35(1):64-70. doi:10.7606/j.issn.1009-1041.2015.01.10.
[7] 李雪, 温昕, 邵铁梅, 孔卫娜, 刘培, 焦展, 安胜军. 芝麻遗传工程的研究进展[J]. 中国农学通报, 2012, 28(6): 153-158. doi:10.3969/j.issn.1000-6850.2012.06.029.
Li X, Wen X, Shao T M, Kong W N, Liu P, Jiao Z, An S J. Progress on genetic engineering of sesame[J]. Chinese Agricultural Science Bulletin, 2012,28(6):153-158. doi:10.3969/j.issn.1000-6850.2012.06.029.
[8] 赵治海, 崔文生, 杜贵, 杨少青. 谷子光(温)敏不育系821选育及其不育性与光温关系的研究[J]. 中国农业科学, 1996, 29(5): 23-31.
Zhao Z H, Cui W S, Du G, Yang S Q. The selection of millet photo(thermo)sensitive sterile line 821 and a study on the relation of sterility to illumination and temperature[J]. Scientia Agricultura Sinica, 1996,29(5):23-31.
[9] 贾小平, 袁玺垒, 李剑峰, 张博, 张小梅, 郭秀璞, 陈春燕. 不同光温条件谷子资源主要农艺性状的综合评价[J]. 中国农业科学, 2018, 51(13): 578-1752. doi: 10.3864/j.issn.0578-1752.2018.13.001.
Jia X P, Yuan X L, Li J F, Zhang B, Zhang X M, Guo X P, Chen C Y. Comprehensive evaluation of main agronomic traits of millet resources under different light and temperature conditions[J]. Scientia Agricultura Sinica, 2018,51(13):578-1752. doi: 10.3864/j.issn.0578-1752.2018.13.001.
[10] 贾小平, 李剑峰, 全建章, 王永芳, 董志平, 张博, 袁玺垒. 不同光周期条件下谷子农艺性状的光周期敏感性评价[J]. 植物遗传资源学报, 2018, 19(5): 919-924. doi: 10.13430/j.cnki.jpgr.20180207005.
Jia X P, Li J F, Quan J Z, Wang Y F, Dong Z P, Zhang B, Yuan X L. Evaluation of photoperiod sensitivity of agronomic traits of foxtail millet varieties(Setaria italica)under different photoperiod conditions[J]. Journal of Plant Genetic Resources, 2018,19(5):919-924. doi: 10.13430/j.cnki.jpgr.20180207005.
[11] 谢丽莉. 谷子光周期敏感相关性状的QTL定位与分析[D]. 郑州: 河南农业大学, 2012.
Xie L L. QTL mapping and analysis of photoperiod sensitivity of agronomic traits of foxtail millet varieties(Setaria italica)[D].Zhengzhou:Henan Agricultural University, 2012.
[12] Margarita Mauro-Herrera, Wang X W, Hugues Barbier, Thomas Brutnell, Katrien M Devos, Andrew N Doust. Genetic control and comparative genomic analysis of flowering time in setaria(poaceae)[J].G3,2013,3(2):283-295. doi: 10.1534/g3.112.005207.
[13] 贾小平, 董普辉, 张红晓, 孔祥生. 不同谷子品种(系)生长发育特性及抗倒性分析[J]. 河南农业科学, 2015, 44(8): 27-31.doi:10.15933/j.cnki.1004-3268.2015.08.006.
Jia X P, Dong P H, Zhang H X, Kong X S. Analysis of growth and development characteristics and lodging resistance of different foxtail millet cultivars(strains)[J]. Journal of Henan Agricultural Sciences, 2015,44(8):27-31.doi:10.15933/j.cnki.1004-3268.2015.08.006.
[14] 贾小平, 袁玺垒, 陆平, 范丙友, 黄华, 戴凌峰. 中国71个谷子种质资源的灰色关联度分析及综合评价[J]. 种子, 2017, 36(9): 1001-4705. doi:10.16590/j.cnki.1001-4705.2017.09.063.
Jia X P, Yuan X L, Lu P, Fan B Y, Huang H, Dai L F. Grey incidence analysis and comprehensive evaluation of 71 millet germplasm resources in China[J]. Seed, 2017,36(9):1001-4705. doi:10.16590/j.cnki.1001-4705.2017.09.063.
[15] 贾小平. 谷子SSR标记的开发及遗传图谱的构建[D]. 北京: 中国农业大学, 2008.
Jia X P. Development and genetic mapping of SSR markers in foxtail millet(Setaria italica)[D].Beijing:China Agricultural University, 2008.
[16] Evanno G, Regnaut S, Goudet J.Detecting the number of clusters of individuals using the software STRUCTURE: a simulation study[J].Molecular Ecology,2005,14(8):2611-2620. doi:10.1111/j.1365-294X.2005.02553.x.
[17] Doust A N, Mauro-Herrera M, Hodgeand J G, Jessica S.The C4 model grass Setaria is a short day plant with secondary long day genetic regulation[J].Frontiers in Plant Science,2017(8):1-10.doi:10.3389/fpls.
[18] Jia G, Huang X, Zhi H, Zhao Y, Zhao Q, Li W, Chai Y, Yang L, Liu K, Lu H, Zhu C, Lu Y, Zhou C, Fan D, Weng Q, Guo Y, Huang T, Zhang L, Lu T, Feng Q, Hao H, Liu H, Lu P, Zhang N, Li Y, Guo E, Wang S, Wang S, Liu J, Zhang W, Chen G, Zhang B, Li W, Wang Y, Li H, Zhao B, Li J, Diao X, Han B.A haplotype map of genomic variations and genome-wide association studies of agronomic traits in foxtail millet (Setaria italica)[J].Nature Genetics,2013,45(8):957-961.doi:10.1038/ng.2673.
[19] Gupta S, Kumari K, Muthamilarasan M, Parida S K, Prasad M.Population structure and association mapping of yield contributing agronomic traits in foxtail millet[J].Plant Cell Reports,2014,33(6):881-893. doi:10.1007/s00299-014-1564-0.
[20] Pajoro A, Severing E, Angenent G C, Immink R.Histone H3 lysine 36 methylation affects temperature-induced alternative splicing and flowering in plants[J].Genome Biology,2017,18(102):1-12. doi:10.1186/s13059-017-1235-x.
[21] Choi S C, Lee S, Kim S R,Lee Y S,Liu C Y,Cao X F,An G H.Trithorax group protein Oryza sativa Trithorax1 controls flowering time in rice via interaction with Early heading date 3[J]. Plant Physiol, 2014, 164: 1326-1337. doi:10.1104/pp.113.228049.
[22] 段桂芳. 拟南芥F-box家族基因FOA2的功能初步研究[D]. 长沙: 湖南大学, 2014.
Duan G F. Preliminary study on the function of F-box family gene FOA2 in Arabidopsis thaliana[D].Changsha:Hunan University, 2014.