小麦是我国三大粮食作物之一,在我国粮食生产中占重要地位。光周期是植物从营养生长转为生殖生长的重要影响因子,光敏色素互作因子(Phytochrome interacting factors,PIFs)在植物的光周期途径中起重要作用。bHLH(Basic helix-loop-helix)家族是植物中第二大类转录因子家族,参与植物不同组织众多代谢过程的调控, 在植物光信号传导、抗逆胁迫和次生代谢等过程中发挥重要作用[1]。PIFs是一类bHLH转录因子,能直接与光激活后的远红光吸收型光敏色素相互作用来控制光调节基因的表达[1-2]。目前,拟南芥中已经克隆的PIFs成员共有7个:PIF1(PIL5)、PIF3、PIF4、PIF5(PIL6)、PIF6(PIL2)、PIF7、PIF8(UNE10)。研究表明,PIFs是位于光受体下游的负调控因子,可以抑制光形态建成[3]。科学家用酵母双杂交法从拟南芥中筛选出第1个可与光敏色素直接相互作用的PIF3蛋白, PIF3蛋白具有E-box结合功能, 定位于细胞核内, 是HLH家族的一员[4]。之后通过反向遗传学的方法筛选得到PIFs家族的第2个成员PIF4,它可以和远红光吸收型光敏色素B相互作用[5]。在光信号传递到转录网络的过程中,远红光吸收型光敏色素由细胞质迁移到细胞核并与PIFs相互作用诱导靶细胞的转录应答[6]。PIFs中保守的bHLH结构域能特异结合到启动子中的G-box元件上,从而调控靶基因的表达[7]。研究发现,黑暗条件下拟南芥幼苗中PIF3基因表达量高,光照条件下其表达量迅速减少,推测光敏色素负调控PIF3基因的表达[8]。后来研究表明,PIF3含量的减少是由于光敏色素A或光敏色素B直接结合到PIF3的光敏色素结合区APA或APB位点上,诱导PIF3快速磷酸化并通过泛素-蛋白酶体系统(UPS)快速泛素化和降解造成的[9-10]。之后,关于PIF1、PIF4和PIF5的研究也得到与PIF3类似的结果[11-13]。PIFs参与植物温度信号转导途径,植物在低温条件下生长减缓,高温条件下表现出一系列形态变化, 如胚轴伸长、提早开花, 研究证明这些反应是由PIF4调控的[14]。此外,PIFs还参与激素信号转导途径[15]。已有研究证明,PIF4参与植物中赤霉素(GA)[16]、生长素(IAA)[17]、油菜素内酯(BR)[18]的信号传递,协同调控植物的生长发育。目前,关于PIF4基因的研究主要集中在拟南芥中[19-21],在小麦中并未见报道。为此,从光周期相关数字基因表达谱中筛选光周期调节基因TaPIF4,然后克隆该基因并对其序列进行生物信息学分析,同时利用实时荧光定量 PCR(qRT-PCR)检测该基因在长、短日照条件下的节律表达特性及非生物胁迫下的表达模式,为进一步研究该基因调控小麦光周期反应的机制及抗逆功能奠定理论基础。
1 材料和方法
1.1 试验材料
基因克隆供试材料为小麦品种辽春10号,qRT-PCR扩增材料为光敏小麦品种宁春36号。大肠杆菌 DH5α菌株(Escherichia coli)购自北京天根生化科技有限公司,DNA聚合酶(高保真酶)、普通琼脂糖胶回收试剂盒、质粒小量提取试剂盒、克隆载体pMD19-T、反转录试剂盒、荧光定量试剂盒SYBR Green Ⅰ均购自TaKaRa公司,TRIzol试剂盒购自Invitrogen公司,氨苄青霉素和卡纳青霉素购自Sigma公司。引物合成及测序均由北京六合华大基因科技有限公司完成,其余常规药品均为进口或国产分析纯。
1.2 试验处理
选取饱满小麦种子,灭菌后置于25 ℃条件下浸泡12~24 h,萌动后播种于装有营养土的种植盆中,放置于22 ℃培养箱中培养。辽春10号放置于16 h光照(LD)条件下培养。宁春36设置2个光照处理,分别放置在6 h光照(SD)和16 h光照(LD)2种条件下培养。每天常规水肥管理,21 d后剪取辽春10号的叶片用于目的基因克隆,同时剪取宁春36在2个光周期处理条件下的叶片用于目的基因的昼夜节律性表达分析。取样时间以光照开始时设为0 h,每隔3 h取样一次,连续取样48 h,样品用液氮速冻后置于-80 ℃冰箱备用。胁迫处理试验的小麦种子在16 h光照条件下培养,培养液为1/2 MS培养液,每隔2 d更换一次培养液。21 d后进行胁迫处理,分别用含有100 μmol/L脱落酸(ABA)、100 mmol/L NaCl、20% PEG6000的1/2MS培养液继续培养幼苗,温度为22 ℃;另取一部分幼苗置于4 ℃条件下进行冷处理;以不进行任何处理的材料为对照,分别剪取各处理0.5,6.0,12.0,24.0 h的叶片,用液氮速冻后置于-80 ℃冰箱备用。
1.3 TaPIF4基因的克隆
国家小麦工程技术研究中心小麦遗传育种研究室前期采用Illumina/Solexa基因测序技术对不同光周期处理下辽春10号小麦品种进行了光周期发育转录组文库构建和数字基因表达谱差异分析[22]。本研究从所得的光周期表达谱中进行筛选,得到光周期相关基因的EST序列,在 NCBI 网站的nr数据库中进行 Blast比对分析,因与粗山羊草的PIF4-like基因高度同源且有较高一致性,故命名该基因序列为TaPIF4。利用Primer Premier 5.0在该序列开放阅读框(ORF)的5′和3′非编码区设计2 条引物(表1)用于ORF片段的扩增。小麦叶片DNA的提取采用CTAB法,小麦总RNA的提取参照Invitrogen 的TRIzol试剂盒说明书,取1 μg RNA, 使用PrimeScriptTM RT reagent Kit with gDNA Eraser试剂盒(TaKaRa 公司)进行反转录合成第一链cDNA。PCR反应体系参照TaKaRa公司的PremiSTAR Max DNA Polymerase说明书配制,体积为20 μL:2× PCR PrimeSTAR Max Premix 10 μL、10 μmol/L上下游引物各0.5 μL、模板1 μL,用ddH2O补齐至20 μL。以辽春10号cDNA为模板时,PCR 反应条件为:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸2 min,35个循环;72 ℃最后延伸10 min。以DNA为模板时,PCR 反应条件为:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸3 min,35个循环;72 ℃最后延伸10 min。PCR 产物经1%琼脂糖凝胶电泳检测后,切下目的条带,经回收后与pMD19-T载体连接,然后转化大肠杆菌DH5α感受态细胞,筛选阳性克隆后送至公司进行测序分析。
表1 TaPIF4基因PCR及qRT-PCR扩增所用引物序列
Tab.1 Primers used for TaPIF4 gene PCR and qRT-PCR amplification
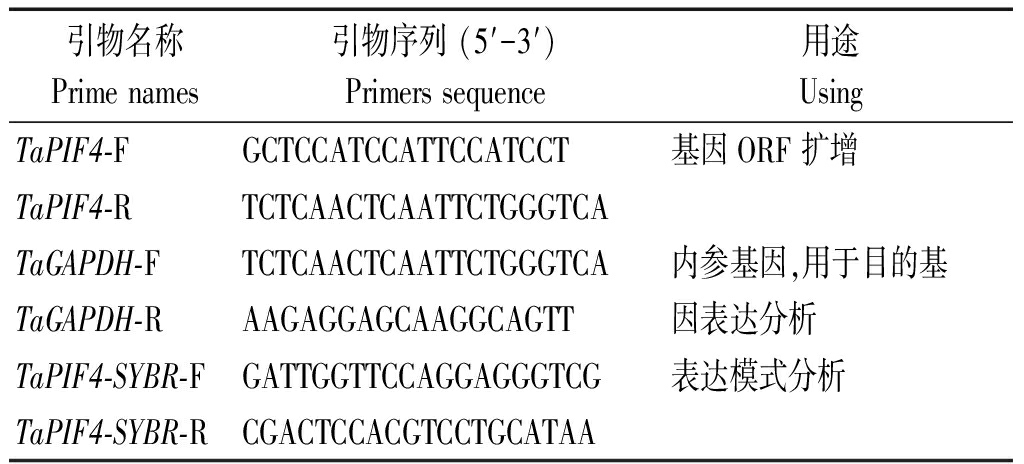
引物名称 Prime names引物序列 (5′-3′) Primers sequence用途 UsingTaPIF4-FGCTCCATCCATTCCATCCT基因ORF 扩增TaPIF4-RTCTCAACTCAATTCTGGGTCATaGAPDH-FTCTCAACTCAATTCTGGGTCA内参基因,用于目的基TaGAPDH-RAAGAGGAGCAAGGCAGTT因表达分析 TaPIF4-SYBR-FGATTGGTTCCAGGAGGGTCG表达模式分析 TaPIF4-SYBR-RCGACTCCACGTCCTGCATAA
1.4 TaPIF4基因的生物信息学分析
利用DNAMAN 软件对DNA、cDNA序列进行比对分析,确定外显子和内含子。通过NCBI(http:// www.ncbi. nlm.nih.gov/Structure/cdd/wrpsb.cgi)在线分析基因的保守结构域。采用在线分析工具ProtParam(http://web.expasy. org/protparam/)、Predictprotein(https://www.predictprotein.org/)、SOPMA(https:// npsa-prabi.ibcp.fr)预测目的基因编码蛋白质的结构和性质。利用Blast(https://blast.ncbi.nlm.nih.gov/Blast.cgi)在线分析工具对TaPIF4基因编码的氨基酸序列进行相似性分析,利用MEGA 6.0软件进行系统进化树构建。
1.5 TaPIF4基因的表达分析
参照qRT-PCR引物设计原则,根据GenBank登录的小麦内参基因GAPDH(gi:7579063) cDNA序列设计特异引物TaGAPDH-F、TaGAPDH-R,同时设计TaPIF4基因引物TaPIF4-SYBR-F、TaPIF4-SYBR-R(表1)。qRT-PCR反应体系参照TaKaRa公司的SYBR Premix Ex TaqTMⅡ(Tli RNaseH Plus) 试剂盒说明书,20 μL 体系含 SYBR Premix Ex TaqTMⅡ(2×) 10 μL、cDNA 模板 2.0 μL、10 μmol/L 的正反向引物各1.0 μL、ddH2O 6.0 μL。每个样品设置3个重复。反应条件为:95 ℃预变性30 s;95 ℃变性 15 s,60 ℃退火 30 s,72 ℃ 延伸30 s,40个循环。根据扩增曲线确定基因的Ct值,相对表达量采用2-ΔΔCt方法计算。
2 结果与分析
2.1 小麦TaPIF4基因的克隆及结构分析
以辽春10号叶片cDNA为模板,经PCR扩增得到1条长1 244 bp的片段(图1),与预期片段大小基本一致,其中ORF为1 053 bp。利用同一引物以DNA为模板进行PCR扩增,扩增产物长度为1 624 bp(图1)。用DNAMAN进行序列比对分析(图2)表明,TaPIF4基因编码区包含4个外显子(分别为60,702,66,225 bp)和3个内含子(分别为113,99,129 bp)。保守结构域分析表明(图3),TaPIF4蛋白在多肽链C端有1个bHLH功能域,该功能域含有碱性DNA结合域和HLH结构域,并含有典型的E-box/N-box识别位点。
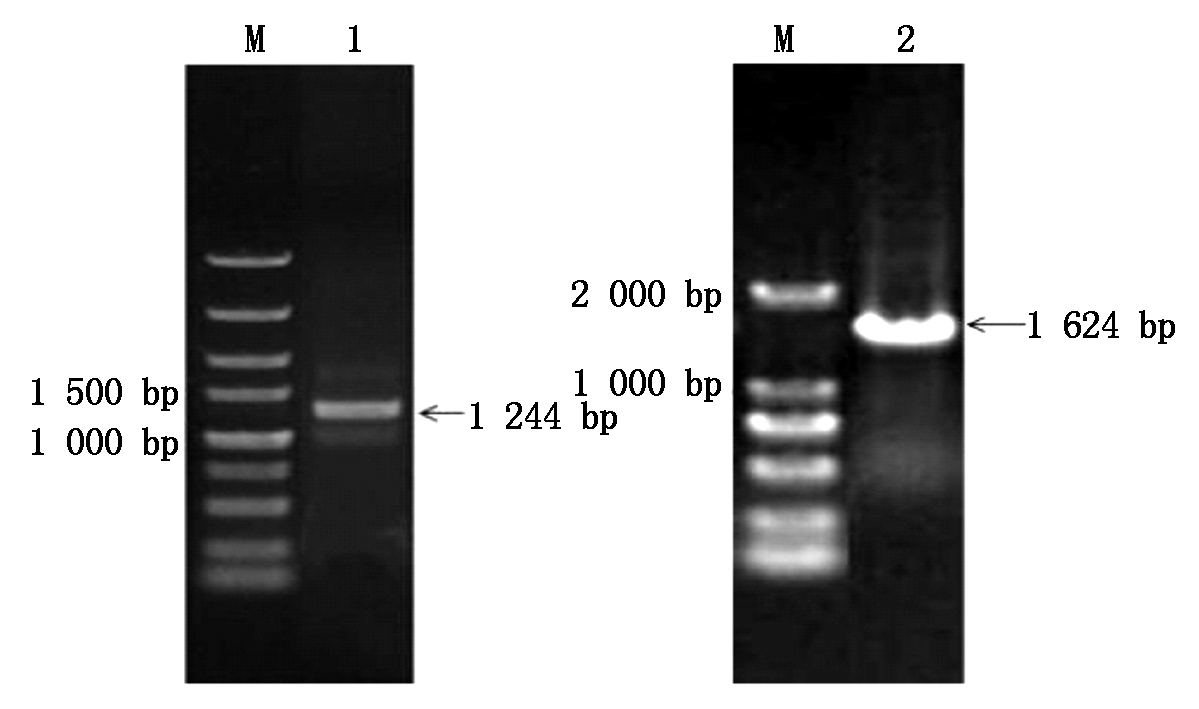
M.DNA分子质量标准(左.DL5000,右.DL2000);1.cDNA的PCR扩增产物;2.DNA的PCR扩增产物。
M. DNA molecular weight Marker(Left. DL5000, Right. DL2000); 1. PCR products of cDNA ; 2. PCR products of DNA.
图1 小麦TaPIF4基因的PCR扩增结果
Fig.1 PCR amplification product of TaPIF4 gene from wheat
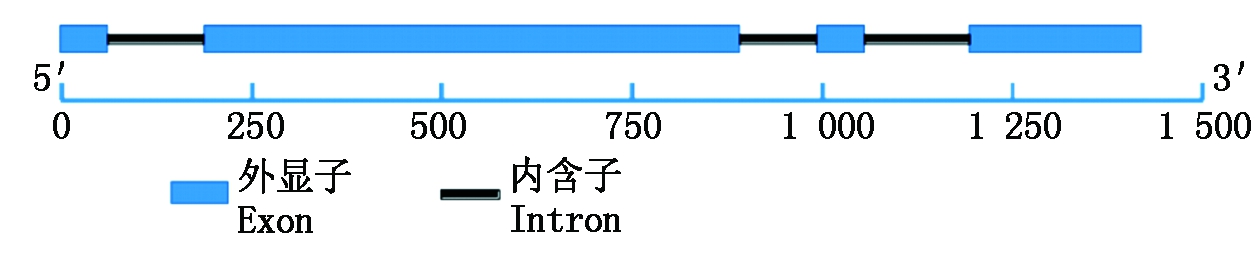
图2 TaPIF4基因编码区结构
Fig.2 Coding region structure of TaPIF4 gene
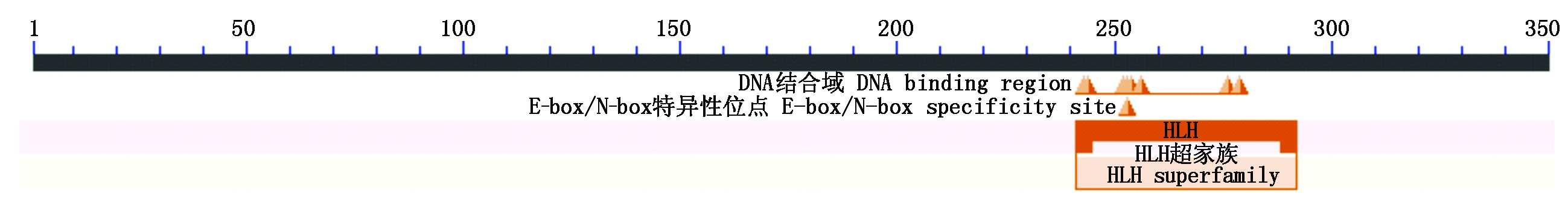
图3 TaPIF4蛋白的保守结构域分析
Fig.3 Analysis of conservative domains of TaPIF4 protein
2.2 TaPIF4基因的生物信息学分析
TaPIF4基因ORF编码350个氨基酸。经预测,TaPIF4蛋白分子质量为37.96 ku,分子式为C1618H2585N491O525S20,酸性氨基酸(Asp+Glu)残基数为45个,碱性氨基酸(Arg+Lys)残基数为40个,理论等电点为5.87。脂肪系数为63.91,平均亲水性(GRAVY)值为-0.579,预测为亲水性蛋白(图4)。不稳定指数为64.87,预测该蛋白为不稳定蛋白,易发生水解、氧化、消旋等反应。
经预测,TaPIF4蛋白无跨膜螺旋和双硫键,定位于细胞核中。TaPIF4蛋白的二级结构包括α-螺旋、β-转角、无规则卷曲、延伸链。其中,构成无规则卷曲的氨基酸数最多,为155个,占44.29%;构成螺旋和延伸链的氨基酸数分别为120,49个,占34.29%,14.00%;构成β-转角的氨基酸数最少,为26个,仅占7.43%(图5)。
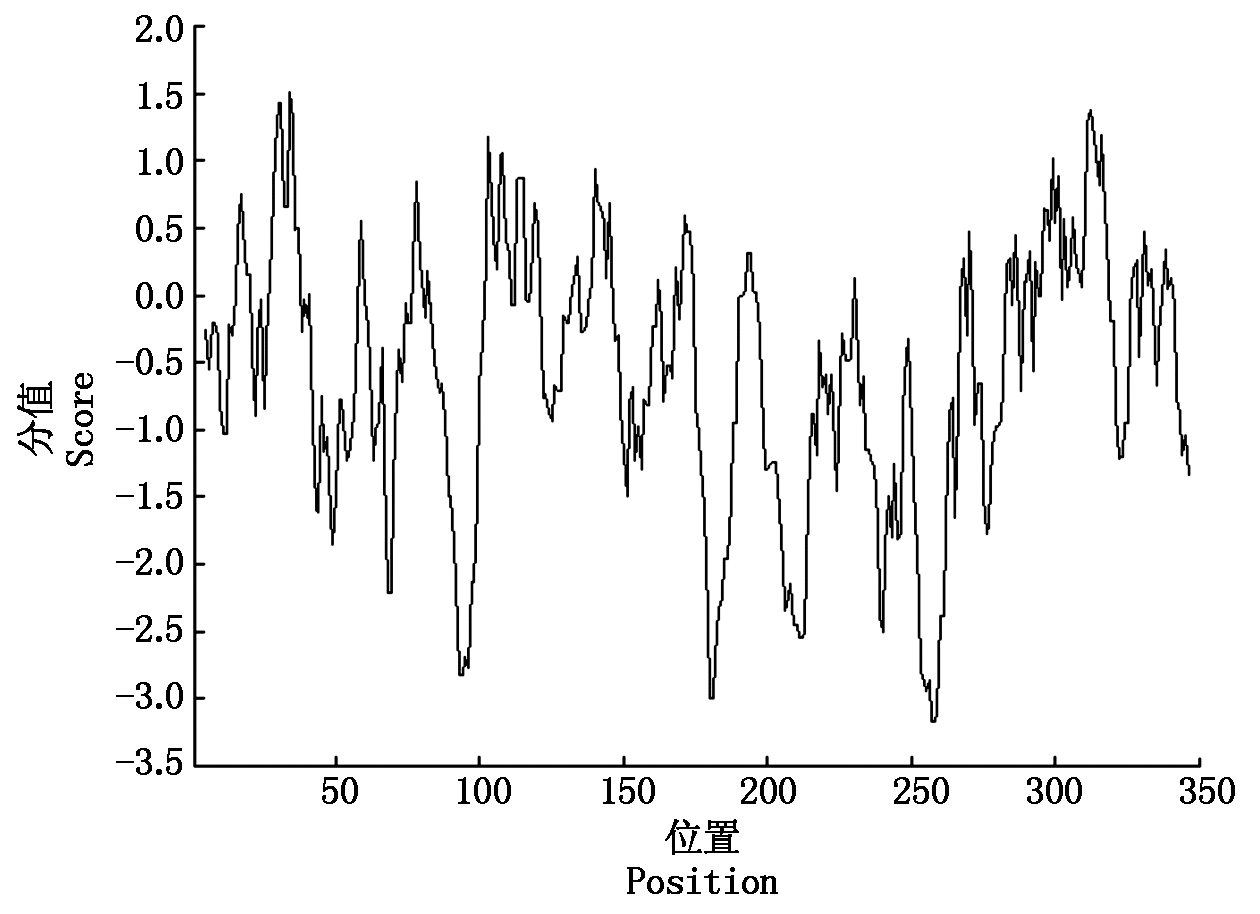
图4 TaPIF4蛋白疏水区预测
Fig.4 Prediction of hydrophobic region of TaPIF4 protein
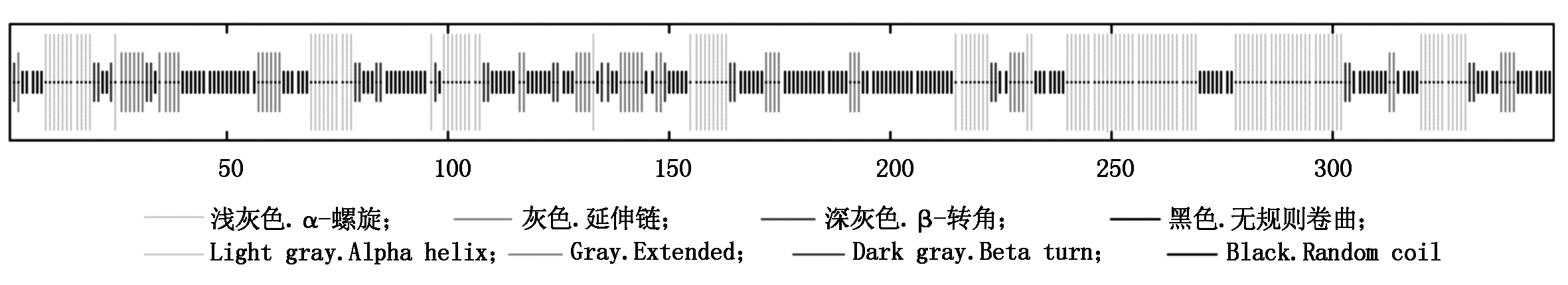
图5 TaPIF4蛋白的二级结构预测
Fig.5 Prediction of secondary structure of TaPIF4 protein
2.3 TaPIF4氨基酸序列比对及系统进化分析
将TaPIF4蛋白氨基酸序列与粗山羊草(Aegilops tauschii, XP_020189473.1)、大麦(Hordeum vulgare, BAJ92170.1)、谷子(Setaria italica, XP_012699734.1)、黍(Panicum hallii, PAN 2.1836.1)、水稻(Oryza sativa, XP_015618080.1)、高粱(Sorghum bicolor, KXG23955.1)、玉米(Zea mays, ONM07085.1)等植物的同源PIF蛋白氨基酸序列进行比对分析(图6)发现,TaPIF4蛋白氨基酸序列与其他物种同源PIF蛋白氨基酸序列长度相近,所有参比序列均含有1个bHLH结构域,该结构域在序列上非常保守,其中有47个氨基酸在不同PIF蛋白中是完全相同的,占bHLH结构域氨基酸总数的88.5%。TaPIF4蛋白序列与粗山羊草、大麦、谷子、黍、水稻、高粱、玉米的氨基酸序列同源性分别达100%,79%,56%,54%,51%,46%,40%。
将小麦TaPIF4蛋白氨基酸序列与粗山羊草、大麦、谷子、黍、水稻、高粱、玉米、短柄草(Brachypodium distachyon, XP_003559649.1)、菠萝(Ananas comosus, XP_020095267.1)、海枣(Phoenix dactylifera, XP_008776592.1)、油棕(Elaeis guineensis, XP_010906815.1)、小果野蕉(Musa acuminata, XP_009399053.1)、芦笋(Asparagus officinalis, ONK74413.1)、黄麻(Corchorus capsularis, OMO49600.1)、可可(Theobroma cacao, XP_017971849.1、柑橘(Citrus clementina, XP_006437071.1)、巨桉(Eucalyptus grandis, XP_010054152.1)、胡杨(Populus euphratica, XP_011032742.1)、枣(Ziziphus jujuba, XP_015880601.1)、木豆(Cajanus cajan, KYP73322.1)等20个物种中同源蛋白质氨基酸序列进行进化分析(图7)发现,所有参比的植物可以分为3类,分别是粗山羊草、大麦等亲缘关系较近的单子叶禾本科植物,菠萝、海枣等单子叶非禾本科植物,以及黄麻、可可等双子叶植物。其中,TaPIF4蛋白与粗山羊草同源蛋白质亲缘关系最近,其次为大麦、谷子、黍、水稻。

下划线.bHLH结构域;黑色阴影.相同氨基酸;灰色阴影.差异氨基酸。
The underlined part is the bHLH domain; The amino acids in black are the same amino acid; The amino acids in gray are the different amino acids.
图6 TaPIF4蛋白与其他植物同源PIF蛋白氨基酸序列比对分析
Fig.6 Deduced amino acid sequence aligment between TaPIF4 and homologous PIF proteins of other plants
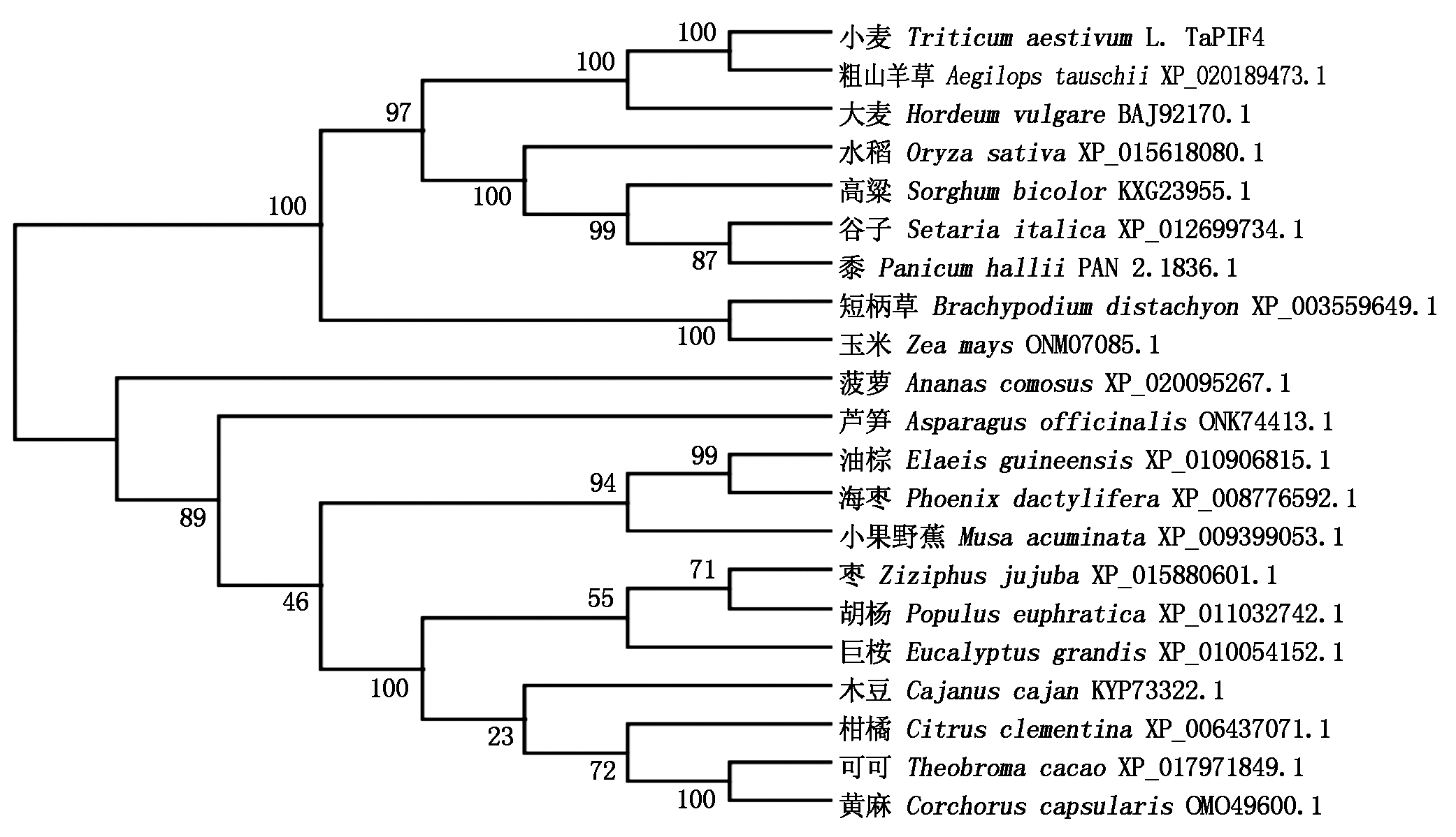
图7 TaPIF4蛋白的系统进化树
Fig.7 Phylogenetic tree of TaPIF4 protein
2.4 TaPIF4基因的表达特性分析
2.4.1 长短光处理下TaPIF4基因的表达分析 研究表明,拟南芥PIF4基因的表达具有昼夜节律性[23],但未见单子叶作物小麦TaPIF4基因表达的昼夜节律性研究。为此,分析了TaPIF4基因的昼夜节律性表达。由图8可知,在长日照和短日照条件下,TaPIF4基因表达总体上均表现出昼夜节律性。在长光照处理条件下,0~16 h光照期间TaPIF4基因的表达量总体呈先下降后趋于平稳之后再稍微升高的趋势,之后在黑暗条件下TaPRF4基因表达量在18~24 h中先显著提升后迅速下降;在第2个循环(24~48 h)中,TaPIF4基因表达量在光照条件下先迅速下降并基本趋于平稳,之后在黑暗期间TaPIF4基因表达量在42~48 h中显著提升后迅速下降(P<0.05)。在短光照处理条件下,TaPIF4基因表达量在光照期间(0~6 h)先持续下降后趋于平稳,在黑暗条件下其表达量在9~21 h中经历2次显著提升和下降;后下降,然后再上升后下降;在第2个循环(24~48 h)中,TaPIF4基因表达量的升降趋势同第1个循环(0~24 h),在33~39 h,42~45 h中分别经历2次显著提升后下降(P<0.05)。这表明该基因的表达具有昼夜节律性,可能在光周期信号转导途径中发挥作用。
2.4.2 非生物胁迫下TaPIF4基因的表达分析 TaPIF4基因含有bHLH结构域,暗示其可能参与植物的逆境胁迫信号通路。对TaPIF4基因在不同胁迫处理条件下的表达特性进行分析(图9)发现,在外源ABA处理条件下,TaPIF4基因的表达被明显抑制,在12,24 h该基因的表达量仅为CK的9.5%,7.5%。在20% PEG6000处理条件下,前期干旱诱导TaPIF4基因的表达,其表达量在0.5 h时远超过对照,在6 h时该基因的表达量是对照的1.44倍;但从干旱处理12 h开始,干旱开始显著抑制TaPIF4基因的表达。在100 mmol/L NaCl处理条件下,TaPIF4基因的表达无明显的规律。在4 ℃低温处理条件下,0.5 h时TaPIF4基因表达量与CK相比无明显差异,在6~12 h时该基因明显被抑制,随后在24 h时该基因被诱导表达。
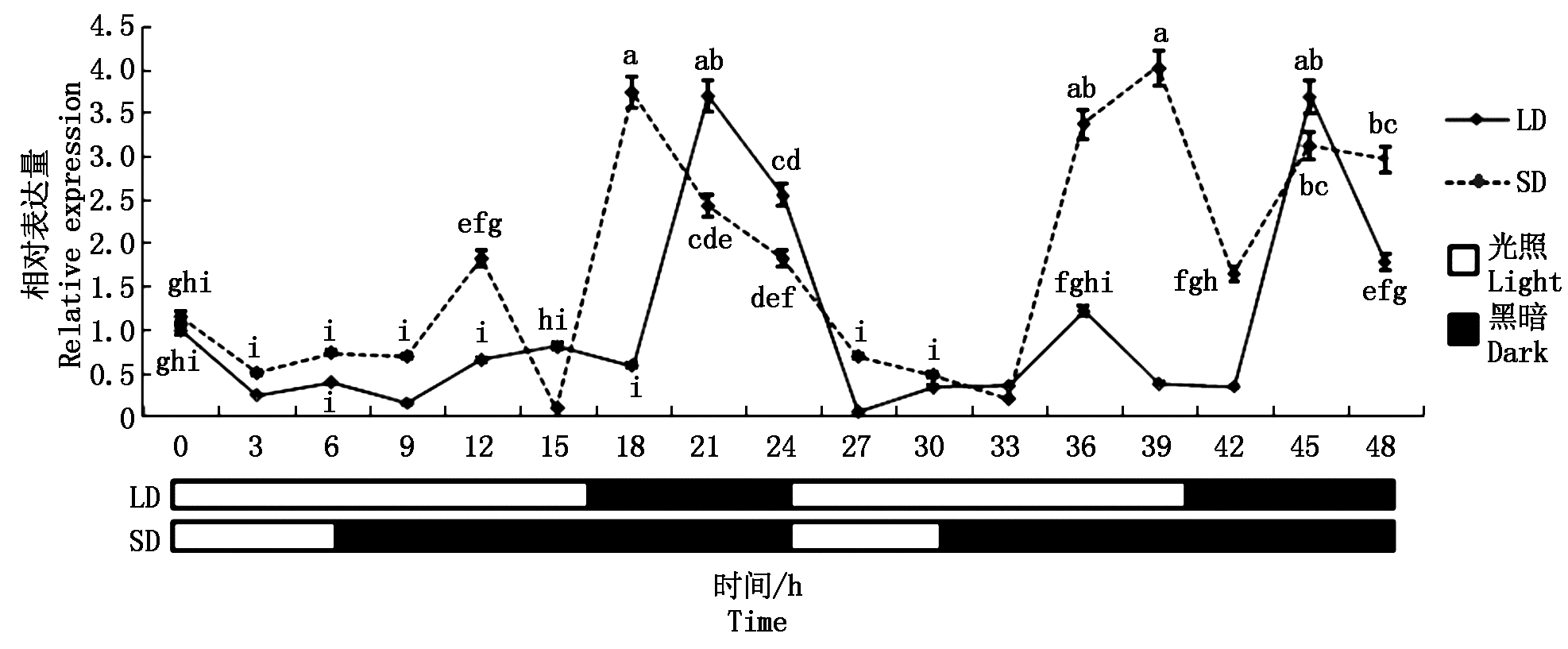
不同小写字母表示差异显著(P<0.05)。图9同。
Different letters indicate significant differences(P<0.05).The same as Fig.9.
图8 长、短日照处理下TaPIF4 的表达分析
Fig.8 Expression analysis of TaPIF4 under long and short days
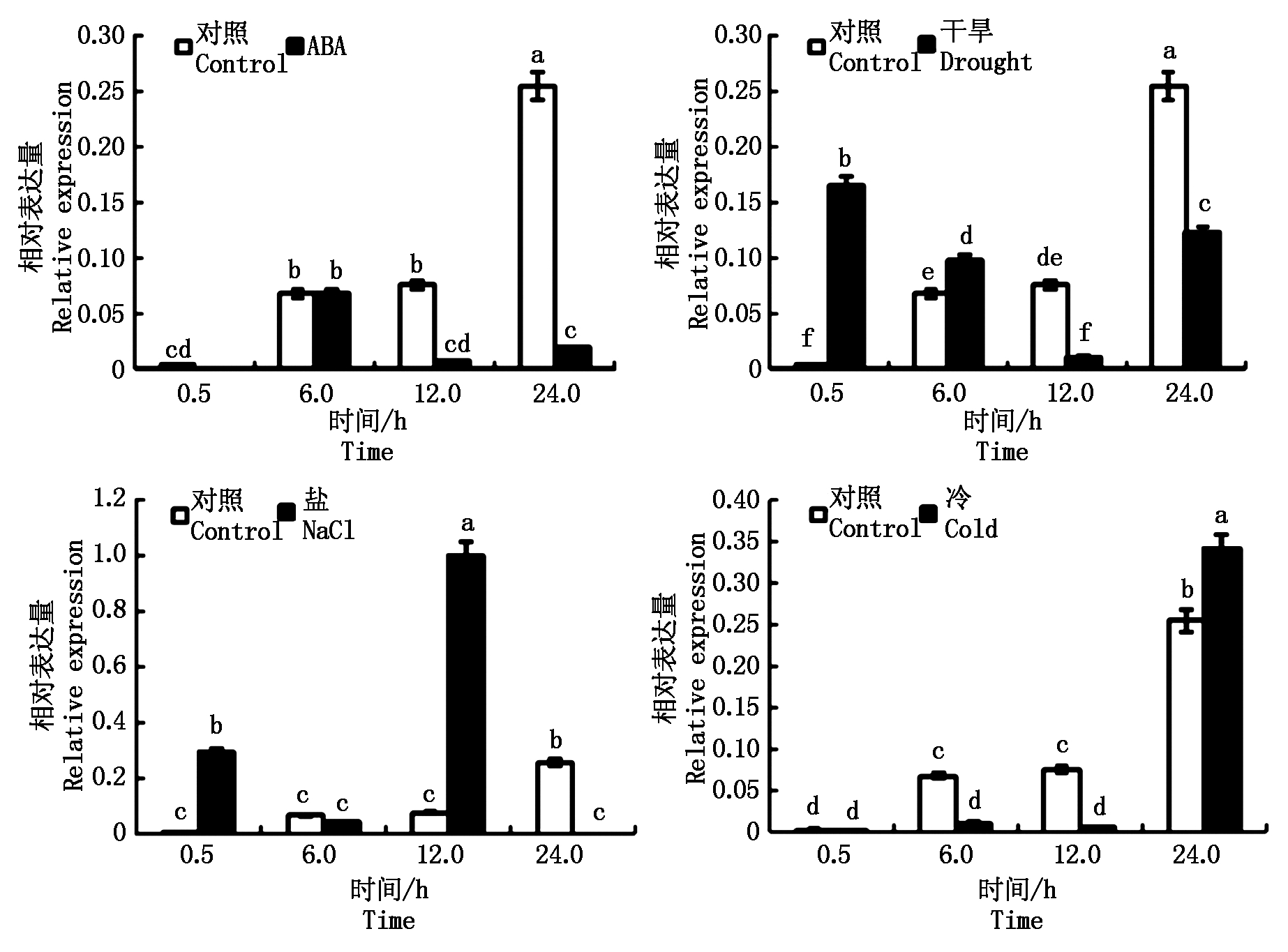
图9 TaPIF4在4种非生物胁迫条件下的表达分析
Fig.9 Expression analysis of TaPIF4 gene under four abiotic stresses
3 结论与讨论
本研究从小麦品种辽春10号中克隆到与光周期相关的基因TaPIF4,该基因ORF长1 053 bp,编码350个氨基酸,定位在细胞核中。小麦TaPIF4编码的氨基酸序列与粗山羊草(小麦的DD基因组供体)的一致性达到100%,推测TaPIF4基因可能完全来源于粗山羊草。TaPIF4蛋白序列相当保守,含有典型的bHLH结构域,具有E-box/N-box识别位点,含有与DNA片段结合的碱性区域,能够与其他PIFs或其他蛋白质形成同源或异源二聚体,拓展了TaPIF4作为转录因子进行植物生理系统调控的范围。经过近几十年的研究发现,PIFs不仅参与光信号调控活动,还参与植物的多种生物学过程,如种子萌发、植株形态建成、激素信号响应及昼夜节律等[18,24]。此外,bHLH转录因子还参与植物抗逆调控。虽然人们对PIFs参与多种信号转导途径、调控植物生长发育的机制已有了一定的认识, 然而研究主要集中在双子叶植物拟南芥上,对单子叶植物还需进一步研究。
研究发现,在拟南芥中许多bHLH转录因子的表达具有昼夜节律性,如bHLH69和bHLH92[25]。bHLH 转录因子家族的PIFs可以通过与远红光吸收型光敏色素互作来调控植物开花,其中,PIF4和PIF5的表达具有节律性,可能通过与节律钟基因的相互作用来调控植物的生长、开花[21]。研究发现,在光照条件下,受激活的光敏色素既可以通过光诱导的磷酸化作用与PIFs蛋白互作,也可以与PIFs蛋白直接互作,诱导PIFs蛋白迅速降解[26-28];同时光敏色素还可以通过调控COP1(Constitutively photomorphogenic 1)-SPA(Suppressor of phytochrome A)复合体的活性, 间接影响PIFs蛋白的稳定性[29],降低PIFs的表达水平。在黑暗条件下,光敏色素处于失活状态,PIFs不断累积,COP1-SPA复合物又通过抑制HY5(Hypocotyl 5)[29]、HFR1(Hypocotyl in far-red 1)[30]等光信号通路正调控因子的活性,间接提高PIFs蛋白的表达水平。本研究中,TaPIF4基因的表达总体表现出昼夜节律性,在光照条件下低水平表达,在黑暗条件下则高水平表达,但在持续的黑暗中表达量会出现升-降的波动现象。以上结果表明,TaPIF4在植物生命活动中参与光信号传导并可能影响细胞核中生物钟调节基因的表达,推测TaPIF4可以通过远红光吸收型光敏色素诱导降解,同时又能反馈调节光敏色素的表达水平来削弱这种抑制作用,使其不再继续降解而保持在一个较低的表达水平。初步推测TaPIF4基因可能在植物光周期途径中发挥作用。
PIFs不仅在光信号传递路径中起关键作用, 还参与GA、IAA、BR、ABA、乙烯等植物内源信号的传递,协同调控植物的生长、发育[16-18]。本研究发现,外源ABA抑制了TaPIF4基因的表达,推测TaPIF4基因可能参与ABA调控的反应。bHLH转录因子参与植物的抗逆调控网络,具有抗非生物胁迫的能力,如OsbHLH148[31]、OrbHLH001基因[32]。PIFs是一类bHLH转录因子,本研究发现,TaPIF4基因在外源ABA处理下表达被明显抑制;在干旱处理下,短时间内其表达量升高,之后表达被抑制,推测这种现象可能与TaPIF4基因参与光合作用有关,TaPIF4基因具有抵御干旱胁迫的能力;冷处理下,短时间内其表达被抑制,之后开始大量表达,推测TaPIF4基因具有抵御冷害的能力;盐处理下,TaPIF4基因的表达无明显规律。由以上结果推测,TaPIF4基因可能参与植物ABA、干旱、低温胁迫的信号通路。
[1] Jeong J, Choi G. Phytochrome-interacting factors have both shared and distinct biological roles[J]. Molecules and Cells, 2013, 35(5):371-380. doi:10.1007/s10059-013-0135-5.
[2] Toledo-Ortiz G, Huq E, Quail P H. The Arabidopsis basic/helix-loop-helix transcription factor family[J]. Plant Cell, 2003, 15(8): 1749-1770. doi: 10.1105/tpc.013 839.
[3] Leivar P, Quail P H. PIFs: Pivotal components in a cellular signaling hub[J]. Trends in Plant Science, 2011, 16(1): 19-28. doi:10.1016/j.tplants.2010.08.003.
[4] Ni M, Tepperman J M, Quail P H. PIF3, a phytochrome-interacting factor necessary for normal photoinduced signal transduction, is a novel basic helix- loop-helix protein[J]. Cell, 1998, 95(5): 657-667. doi: 10.1016/S0092-8674(00)81636-0.
[5] Huq E, Quail P H. PIF4, a phytochrome-interacting bHLH factor, functions as a negative regulator of phytochrome B signaling in Arabidopsis[J]. The EMBO Journal, 2002, 21 (10): 2441-2450. doi: 10.1093/ emboj/21.10.2441.
[6] Quail P H. Phytochrome-interacting factors[J]. Seminars in Cell and Developmental Biology, 2000, 11(6): 457-466. doi: 10.1006/scdb.2000.0199.
[7] Zhang Y, Mayba O, Pfeiffer A, Shi H, Tepperman J M, Speed T, Quail P H. A Quartet of PIF bHLH Factors provides a transcriptionally centered signaling hub that regulates seedling morphogenesis through differential expression-patterning of shared target genes in Arabidopsis[J].Plos Genetics, 2013,9(1): e1003244. doi:10.1371/journal.pgen.1003244.
[8] Liu X, Chen C Y, Wang K C,Luo M, Tai R, Yuan L, Zhao M, Yang S, Tian G, Cui Y, Hsieh H L, Wu K. PHYTOCHROME INTERACTING FACTOR3 associates with the histone deacetylase HDA15 in repression of chlorophyll biosynthesis and photosynthesis in etiolated Arabidopsis seedlings[J]. Plant Cell, 2013, 25(4): 1258-1273. doi:10.1105/tpc.113.109710.
[9] Park E, Kim J, Lee Y, Shin J, Oh E, Chung W, Liu J R, Choi G. Degradation of phytochrome interacting factor 3 in phytochrome-mediated light signaling[J]. Plant & Cell Physiology, 2004, 45(8): 968-975. doi: 10.1093/pcp/pch125.
[10] Al-Sady B, Ni W M, Kircher S, Kircher S, Sch?fer E, Quail P H. Photoactivated phytochrome induces rapid PIF3 phosphorylation prior to proteasome- mediated degradation[J]. Molecular Cell, 2006, 23(3): 439-446. doi: 10.1016/j.molcel.2006.06.011.
[11] Oh E, Yamaguchi S, Kamiya Y, Bae G, Chung W I, Choi G. Light activates the degradation of PIL5 protein to promote seed germination through gibberellin in Arabidopsis[J]. Plant Journal, 2006, 47(1): 124-139. doi: 10.1111/j.1365-313x.2006.02773.x.
[12] Shen Y, Khanna R, Carle C M, Quail P H. Phytochrome induces rapid PIF5 phosphorylation and degradation in response to red-light activation[J]. Plant Physiology, 2007, 145(3): 1043-1051. doi: 10.1104/pp.107.105601.
[13] Ni W, Xu S L, Chalkley R J, Pham T N D, Guan S H, Maltby D A, Burlingame A L, Wang Z Y, Quail P H.Multisite light-induced phosphorylation of the transcription factor PIF3 is necessary for both its rapid degradation and concomitant negative feedback modulation of photoreceptor phyB levels in Arabidopsis[J]. Plant Cell, 2013, 25(7):2679-2698. doi:10.1105/tpc.113.112342.
[14] Proveniers M C G, Zanten M V. High temperature acclimation through PIF4 signaling[J]. Trends in Plant Science, 2013, 18(2): 59-64. doi: 10.1016/j. tplants.2012.09.002.
[15] de Lucas M, Prat S. PIFs get BRright: PHYTOCHROME INTERACTING FACTORs as integrators of light and hormonal signals[J]. New Phytologist, 2014, 202(4):1126-1141. doi:10.1111/nph.12725.
[16] Lucas M D, Salomé P. PIFs get BRright: PHYTOCHROME INTERACTING FACTORs as integrators of light and hormonal signals[J]. New Phytologist, 2014, 202(4):1126-1141. doi: 10.1111/nph.12725.
[17] Sun J Q, Qi L L, Li Y N, Zhai Q Z, Li C Y. PIF4 and PIF5 transcription factors link blue light and auxin to regulate the phototropic response in Arabidopsis[J]. Plant Cell, 2013, 25(6):2102-2114. doi: 10.1105/tpc. 113.112417.
[18] Oh E, Zhu J Y, Wang Z Y. Interaction between BZR1 and PIF4 integrates brassinosteroid and environmental responses[J]. Nature Cell Biology, 2012, 14 (8): 802-809. doi: 10.1038/ncb2545.
[19] Nieto C, López-Salmerón V, Davière J M, Prat S. ELF3-PIF4 interaction regulates plant growth independently of the evening complex[J]. Current Biology, 2015, 25(1):187-193. doi: 10.1016/j.cub. 2014. 10.070.
[20] Huq E, Quail P H. PIF4, a phytochrome-interacting bHLH factor, functions as a negative regulator of phytochrome B signaling in Arabidopsis[J]. Embo Journal, 2014, 21(10):2441-2450. doi: 10.1093/emboj/21. 10.2441.
[21] Gangappa S N, Kumar S V. DET1 and HY5 control PIF4-mediated thermosensory elongation growth through distinct mechanisms[J]. Cell Reports, 2017, 18(2):344-351. doi: 10.1016/j.celrep.2016.12.046.
[22] Feng Y L, Zhao Y Y, Wang K T, Li Y C, Wang X, Yin J. Identification of vernalization responsive genes in the winter wheat cultivar Jing841 by transcriptome sequencing.[J]. Journal of Genetics, 2016, 95(4):957-964. doi: 10.1007/s12041-016-0724-0.
[23] Niwa Y,Yamashino T,Mizuno T. The circadian clock regulates the photoperiodic response of hypocotyl elongation through a coincidence mechanism in Arabidopsis thaliana[J]. Plant and Cell Physiology,2009, 50(4):838-854. doi: 10.1093/pcp/pcp028.
[24] Shin J, Anwer M U, Davis S J. Phytochrome-interacting factors (PIFs) as bridges between environmental signals and the circadian clock: diurnal regulation of growth and development[J]. Molecular Plant, 2013, 6(3):592-595. doi:10.1093/mp/sst060.
[25] Hanano S, Stracke R, Jakoby M, Merkle T, Domagalska M A, Weisshaar B, Davis S J. A systematic survey in Arabidopsis thaliana of transcription factors that modulate circadian parameters[J]. BMC Genomics, 2008, 9: 182. doi: 10.1186/1471-2164-9-182.
[26] Jeong J, Choi G. Phytochrome-interacting factors have both shared and distinct biological roles[J]. Molecules and Cells, 2013, 35(5): 371-380. doi: 10.100 7/s10059-013-0135-5.
[27] Bu Q Y, Zhu L, Huq E. Multiple kinases promote light-induced degradation of PIF1[J]. Plant Signaling and Behavior,2011,6(8) : 1119-1121. doi: 10.41 61/psb.6.8.16049.
[28] Ni W M, Xu S L, Chalkley R J, Pham T N D, Guan S H, Maltby D A, Burlingame A L, Wang Z Y, Quail P H. Multisite light-induced phosphorylation of the transcription factor PIF3 is necessary for both its rapid degradation and concomitant negative feedback modulation of photoreceptor phyB levels in Arabidopsis[J]. Plant Cell, 2013, 25(7):2679-2698. doi: 10.1105/tpc.113.112342.
[29] Leivar P, Monte E, Oka Y, Liu T, Carle C, Castillon A, Huq E, Quail P H. Multiple phytochrome- interacting bhlh transcription factors repress premature seedling photomorphogenesis in darkness [J]. Current Biology, 2008, 18(23):1815-1823. doi: 10.1016/j.cub.2008.10.058.
[30] Shi H, Zhong S W, Mo X R, Liu N, Nezames C D, Deng X W. Hfr1 sequesters pif1 to govern the transcriptional network underlying light-initiated seed germination in Arabidopsis[J]. Plant Cell, 2013, 25(10): 3770-3784. doi: 10.1105/tpc.113.117424.
[31] Seo J S, Joo J, Kim M J, Kim Y K, Nahm B H, Song S I, Cheong J J, Lee J S, Kim J K, Choi Y D. OsbHLH148, a basic helix-loop-helix protein, interacts with OsJAZ proteins in a jasmonate signaling pathway leading to drought tolerance in rice[J]. Plant Journal, 201l, 65(6): 907-921. doi: 10.1111/j.1365-313 X.2010.04477.x.
[32] Li F, Guo S Y, Zhao Y, Chen D Z, Chong K, Xu Y Y. Overexpression of a homopeptide repeat-containing bHLH protein gene (OrbHLH001) from Dongxiang wild rice confers freezing and salt tolerance in transgenic Arabidopsis[J]. Plant Cell Reports, 2010, 29(9): 977-986. doi: 10.1007/s00299-010-0 883-z.