大豆(Glycine max (L.) Merrill)起源于中国[1],大豆籽粒中蛋白质含量约占干质量的40%左右,是人类重要的植物蛋白来源。21世纪以来,品质性状改良成为大豆育种的主要目标之一,而提高大豆籽粒蛋白质含量也成为国内外品质育种的重要指标[2-3]。目前,通过常规杂交育种方法培育高蛋白大豆品种难度较大,而已发表的QTL定位结果有时也难以用于指导作物育种实践。因此,如何利用大豆资源中的优异基因,从分子水平揭示复杂的蛋白质合成代谢机制,是进行分子育种的重要前提。
国内外关于大豆蛋白质含量的研究有诸多报道。普遍认为大豆蛋白质合成受微效多基因调控,是数量性状遗传,加性效应明显[4]。试验材料的不同,环境条件的差异对蛋白质的含量也均有较大的影响。目前,在大豆所有的20个染色体上均发现有相关的QTL位点,共有298个QTL,详细信息可参考公共网站(http://soybase.org/)。连锁分析和关联分析均显示20号染色体检测到的大豆籽粒蛋白质合成相关QTL的频率最高[5-7],且在该染色体上有一个QTL区段(A688~Satt239)在多个环境和群体中均被检测到[8]。同时,大量研究表明,蛋白质含量与油分含量、单株产量、株高、主茎节数、主茎分枝数、开花期、成熟期均呈负相关[9-11],而蛋白质与脂肪两性状很难通过染色体片段重组的方法分开,因此,推测这2个性状紧密连锁或一因多效[12]。
对于蛋白质合成积累的研究,早期主要集中在阐述蛋白质构成因子、物质积累过程和环境条件几个方面。蛋白质的基本组成单位是氨基酸,氨基酸通过脱水缩合形成肽链。蛋白质是由1条或多条多肽链组成的生物大分子,每一条多肽链有20至数百个氨基酸残基不等。关于蛋白质合成的调控,前人研究发现,磷酸烯醇式丙酮酸羧化酶(PEPC)是控制种子中蛋白质和脂肪酸合成的重要调控基因,广泛分布于高等植物的细胞质中。Chen等[13]发现利用反义PEP基因转化油菜籽粒后,检测到蛋白质含量降低,同时含油量比对照增加了15%以上,该研究证明了底物竞争调控籽粒蛋白质、脂肪比率的理论。转录因子是一类蛋白质合成的重要调控因子,它们在蛋白质代谢中发挥着重要作用。大豆转录因子GmDof4和GmDof11转化拟南芥后,抑制了蛋白质合成相关基因的表达[14]。近些年来,研究人员通过比较不同物种间蛋白质合成相关的基因,从中发现一些共性的调控基因:LEC1、LEC2、ABI3、FUS3,这些关键的转录因子调控着胚胎发生和种子的成熟。LEC1是ABI3、FUS3上游的正向调控因子,而 ABI3、FUS3、LEC1 基因协同作用以控制种子发育过程中的多个基本过程,当其中某个基因发生突变后,不仅贮藏蛋白的含量降低,同时也降低了对ABA的敏感性[15]。通过对蛋白质合成基因启动子区域的顺式调控元件进行研究发现,贮藏蛋白基因的启动子区域具有2个保守的结构域 RY/G和B-box,其中RY/G结构域包括2个作用元件 RY(CATGCA)和G-box(CACGTG)分别与B3或bZIP、bHLH类转录因子结合,而B-box由DistB (GCCACTTGTC)和ProxB(CAAACACC)2个作用元件组成,分别与bZIP或MYB类转录因子结合,因此,推测这些转录因子对蛋白质的合成具有调控作用[16]。
转录组测序技术(RNA-Seq)的发展使得功能基因和蛋白的发现步伐大大加快。该技术将 RNA 进行反转录得到 cDNA,然后对 cDNA 文库进行测序,旨在快速全面的获取某物种在某状态的下的全部转录本[17]。其以速度快、准确性高、运行成本低等特性受到广泛关注。与基因芯片技术相比,RNA-Seq技术能够对特定细胞或组织在特定状态下的基因表达种类和丰度进行更精准的定性定量分析。近年来 RNA-Seq 技术在作物研究中被广泛应用于基因资源挖掘、揭示突变体变异机制、探究作物生理生化过程、研究抗病、抗逆的机制等方面,为不同生物的功能基因组学开展提供了新的思路与研究方法[18-19]。在基因资源的挖掘方面,Wang等[20]利用转录组测序技术对野生和突变型棉花的毛状体纤维细胞的分裂与延长过程进行探究;和小燕等[21]以含油量高及含油量低的2个花生品系为研究材料,构建了与油脂累积相关的2个发育时期的转录组测序文库,发现各类调控脂肪酸生物合成的基因在花生种子油脂合成初期十分活跃,并挖掘到油体固醇蛋白等基因可能参与了油脂的合成与代谢;除此之外,研究人员利用 454 平台从玉米[22]、羊草、油橄榄[23]等植物中发现了新基因。RNA-Seq技术对揭示突变体变异机制也具有一定的作用。栾海业等[24]以大麦白化颖壳突变体与野生型植株为研究材料进行转录组测序分析,结果表明,导致大麦白化颖壳突变体的出现,可能是因为叶绿体形成和发育的相关基因其表达受到了相关抑制,从而导致了光合作用的降低。在探究作物生理生化过程方面,陈静[25]利用 RNA-Seq 技术对花生种子休眠与解除休眠过程进行了研究,发现赤霉素 20 氧化酶、EREBP-like 因子热激蛋白等综合调控花生种子休眠解除以及萌发。在抗病虫害研究中,李海燕等[26]以抗病的五寨黑豆为材料,对大豆胞囊线虫侵染前与侵染后的转录组文库进行检测对比,发现苯丙烷类代谢途径及氧化磷酸化代谢通路对五寨黑豆的抗病有重要作用。黄启秀等[27]利用转录组测序技术对抗枯萎病抗性表现不同的7种材料进行研究,发现了可能与海岛棉枯萎病抗性相关的代谢通路-类黄酮代谢通路。除此之外,研究人员还应用转录组技术在水稻[28]、小麦[29-30]以及甘薯[31] 等作物中进行了相关的抗病试验,取得了一定的结果。在抗逆境领域,任梦露等[32]利用转录组测序技术研究大豆茎秆对荫蔽胁迫的响应,发现南豆 12 在荫蔽胁迫下其细胞壁多糖的含量和木质素含量有明显提升,并对生长素发生响应,以此方式来增加植株茎的强度,从而保持茎的某种形态优势,并提升其抗倒伏性,最终实现提高对荫蔽环境的适应程度。孙爱清等[33]以强抗旱花生品种为材料,对干旱处理过的花生叶片进行 RNA-Seq 分析,旨在探究花生干旱胁迫下的植株响应,结果发现类黄酮代谢途径可能在该过程中有重要作用。目前,作物中利用转录组测序技术的应用十分广泛,但在挖掘大豆发育时期籽粒中蛋白质合成相关基因的研究鲜有报道。
因此,本研究以遗传背景相近的大豆高蛋白品系冀HJ117及其回交亲本冀豆12为研究材料,利用测序技术检测两样本开花后14 d籽粒的表达谱。通过对差异表达基因进行功能注释、GO功能分类、富集代谢途径的分析以及荧光定量PCR分析,筛选出与蛋白质合成密切相关的基因,为揭示蛋白质合成代谢机制奠定理论基础。
1 材料和方法
1.1 试验材料
亲本组合冀豆12与茶秣食豆杂交后代与母本冀豆12(蛋白质含量46.48%)回交2次,得到高蛋白大豆冀HJ117(蛋白质含量52.99%)。冀HJ117和冀豆12由河北省农林科学院粮油作物研究所提供。将冀HJ117和冀豆12大豆籽粒种植于河北省农林科学院粮油作物研究所堤上试验站,当试验材料进入开花盛期时,分别对冀HJ117和冀豆12同一天开花的花朵进行标记,并对开花后14 d的籽粒进行取样,使用液氮速冻保存。
1.2 试验方法
1.2.1 RNA-Seq文库的构建及测序 RNA-Seq文库构建及测序工作由华大基因科技有限公司完成。
1.2.2 数据处理及差异表达基因的筛选 对测序得到的原始数据进行去杂处理得到有效序列(Clean reads),使用比对软件SOAPaligner/soap2将有效序列比对到参考基因组。利用唯一比对上基因的reads数目和比对上参考序列的总reads数来计算基因表达量,基因表达量的计算使用RPKM法[34],差异表达基因的筛选参照Audic等[35]发表在Genome Research上的基于测序的差异基因检测方法。基于差异基因的功能注释,对所有差异表达基因进行Gene Ontology(GO)功能显著性富集分析,分析将计算得到的P值(P-value)通过Bonferroni校正之后,以corrected P-value≤0.05为阈值,满足此条件的GO条目(GO term)定义为在差异表达基因中显著富集的GO term,以此确定差异表达基因行使的主要生物学功能。代谢通路(Pathway)显著性富集分析将Q值(Q value)≤0.05的Pathway定义为在差异表达基因中显著富集的Pathway,从而确定差异表达基因参与的最主要生化代谢途径和信号转导途径。
1.2.3 qRT-PCR检测候选基因的表达量 随机选取9个差异表达基因进行基因的表达量检测,使用北京天根公司的RNA prep pure Plant Kit试剂盒分别提取大豆冀HJ117及冀豆12 14 d籽粒的RNA,并使用TaKaRa RNA PCRTM Kit(AMV) Ver.3.0反转录得到cDNA,进行荧光定量PCR分析,选用CYP2基因为内参基因。
试验所选基因及引物序列如表1,合成于生工生物工程(上海)股份有限公司。qRT-PCR试验参照TaKaRa Premix Ex TaqTMⅡ试剂盒,反应体系含SYBR Premix Ex Taq Ⅱ 10 μL、模板0.5 ng、PCR正向引物(10 μmol/L) 1 μL、PCR反向引物(10 μmol/L) 1 μL, 补水至总体积20 μL。扩增反应程序为:95 ℃孵育30 s;40个循环的95 ℃ 5 s,60 ℃ 30 s,55 ℃ 30 s。该反应使用美国Bio-Rad Lcycleri Q5荧光定量PCR仪进行扩增。
试验数据分析采用 2-ΔΔCt 法进行相对定量分析,计算过程如下:先分别计算出每组的ΔΔCt=(Ct目的基因1-Ct内参基因)-(Ct目的基因2-Ct内参基因),再根据ΔΔCt 求出 2-ΔΔCt 以及标准误。使用Microsoft Excel 2007 软件处理数据并作图。
表1 基因名称及引物序列
Tab.1 Genes and primers for qRT-PCR
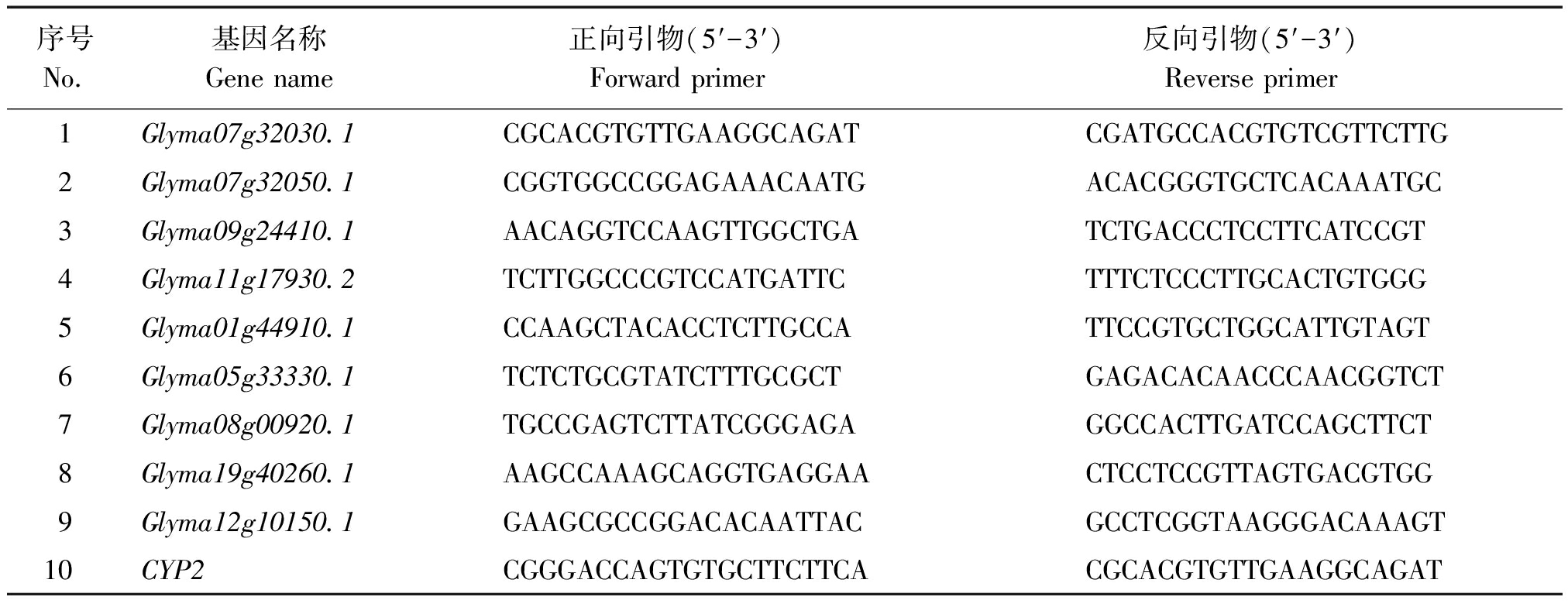
序号No.基因名称Gene name正向引物(5′-3′)Forward primer 反向引物(5′-3′)Reverse primer 1Glyma07g32030.1CGCACGTGTTGAAGGCAGATCGATGCCACGTGTCGTTCTTG2Glyma07g32050.1CGGTGGCCGGAGAAACAATGACACGGGTGCTCACAAATGC3Glyma09g24410.1AACAGGTCCAAGTTGGCTGATCTGACCCTCCTTCATCCGT4Glyma11g17930.2TCTTGGCCCGTCCATGATTCTTTCTCCCTTGCACTGTGGG5Glyma01g44910.1CCAAGCTACACCTCTTGCCATTCCGTGCTGGCATTGTAGT6Glyma05g33330.1TCTCTGCGTATCTTTGCGCTGAGACACAACCCAACGGTCT7Glyma08g00920.1TGCCGAGTCTTATCGGGAGAGGCCACTTGATCCAGCTTCT8Glyma19g40260.1AAGCCAAAGCAGGTGAGGAACTCCTCCGTTAGTGACGTGG9Glyma12g10150.1GAAGCGCCGGACACAATTACGCCTCGGTAAGGGACAAAGT10CYP2CGGGACCAGTGTGCTTCTTCACGCACGTGTTGAAGGCAGAT
2 结果与分析
2.1 测序质量评估与数据比对统计
通过高通量测序仪Illumina HiSeqTM 2000对冀豆12和冀HJ117 14 d籽粒的cDNA测序,分别得到原始序列8 747 900,8 486 919条。通过过滤,冀豆12得到有效序列8 684 049条,占原始序列的99.27%;冀HJ117得到有效序列8 410 499条,占原始序列的99.10%。表明测序质量较高,适合进行后续的生物信息分析。
将冀豆12与冀HJ117过滤得到的有效序列与大豆参考基因组进行比对,表2是各样品与参考基因组的数据比对统计。从比对结果看,两样本成功比对上的序列均在700万条以上,效率分别为85.75%,86.01%。
表2 有效序列与参考基因组比对结果统计
Tab.2 Comparison between clear reads and reference genomes
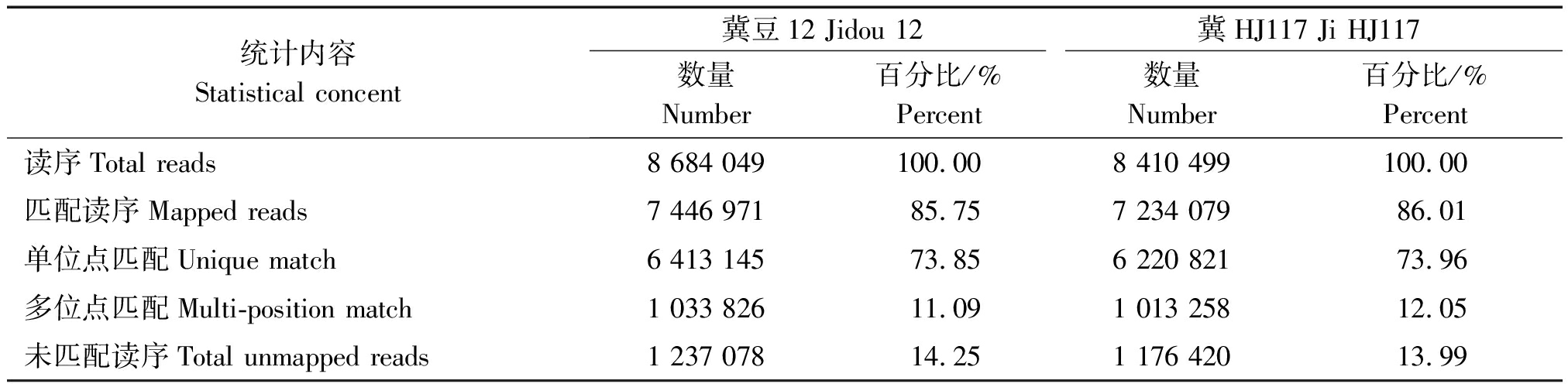
统计内容Statistical concent冀豆12 Jidou 12冀HJ117 Ji HJ117数量Number百分比/%Percent数量Number百分比/%Percent读序Total reads8 684 049100.008 410 499100.00匹配读序Mapped reads7 446 97185.757 234 07986.01单位点匹配Unique match6 413 14573.856 220 82173.96多位点匹配Multi-position match 1 033 82611.091 013 25812.05未匹配读序Total unmapped reads 1 237 07814.251 176 42013.99
2.2 cDNA片段随机性检验
由于不同参考基因有不同的长度,笔者把reads在参考基因上的位置标准化到相对位置(reads在基因上的位置与基因长度的比值),然后统计基因不同位置比对上的reads数量。从图1可以看出,2个样品的reads在参考基因上不同位置的分布相对比较均匀,表明cDNA片段化的随机性好,保证了测序质量。
2.3 差异表达基因的筛选
将两样本差异检验的P-value作多重假设检验(FDR)校正,并根据基因的表达量(RPKM值)计算该基因在不同样本间的差异表达倍数。将同时满足FDR≤0.001且倍数差异不低于2倍的基因定义为差异表达基因。经分析冀HJ117和冀豆12 2个转录组,共得到差异表达基因336个,其中冀HJ117较冀豆12上调表达基因195个,下调表达基因141个(图2-A)。
2.4 差异表达基因的功能注释
通过差异表达基因的筛选,获得差异表达基因的GO IDs。对冀豆12和冀HJ117的差异表达基因进行功能注释,在336个差异表达基因中,获得功能注释的基因为234个,未找到匹配信息的基因数为102个,2种情况占比分别为69.94%,30.36%(图2-B)。
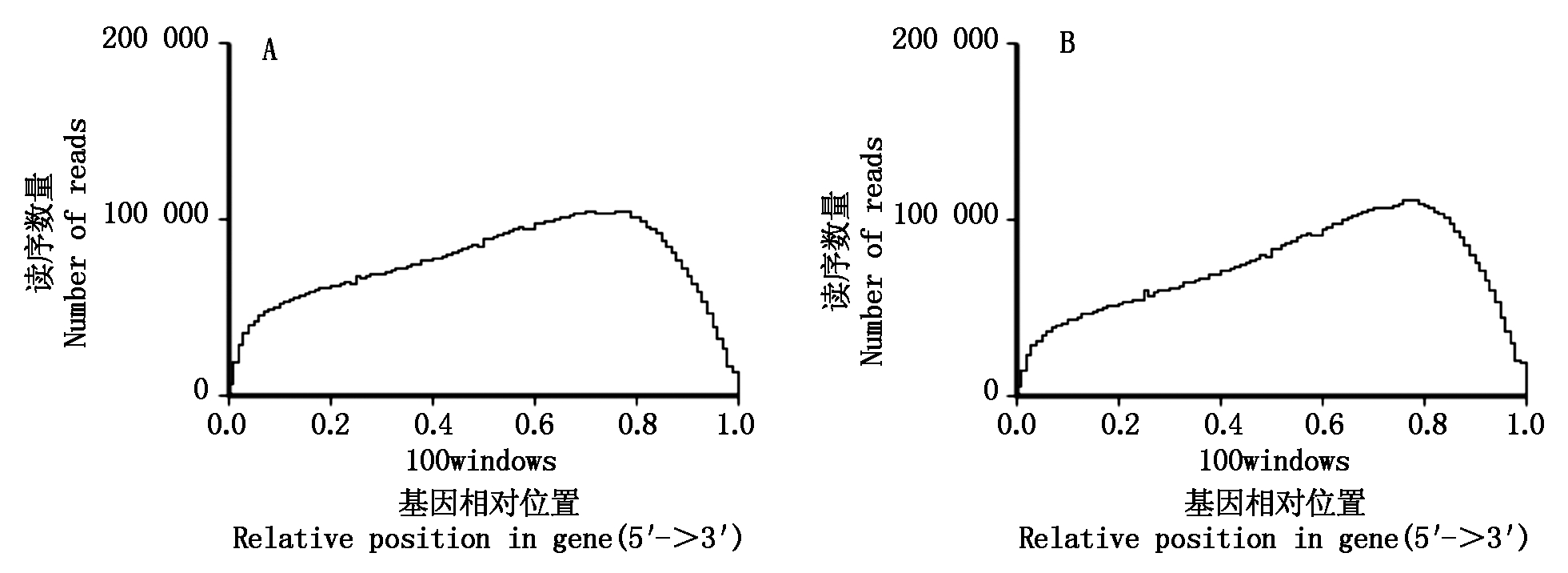
A.冀豆12的reads在参考基因上的分布均匀性统计;B.冀HJ117的reads在参考基因上的分布均匀性统计。
A.Distribution statistics of reads mapped to reference gene in Jidou 12; B. Distribution statistics of reads mapped to reference gene in Ji HJ117.
图1 cDNA片段随机性检验
Fig.1 Randomness test of cDNA fragment
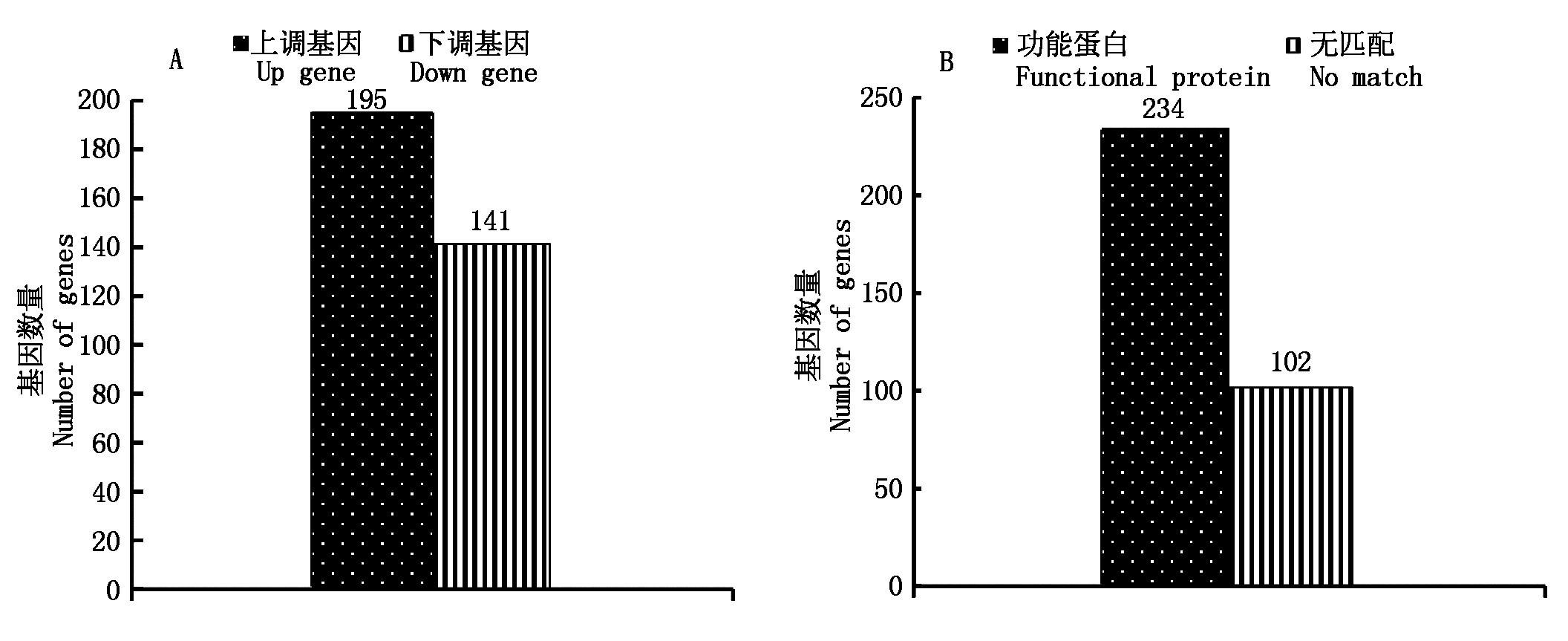
A.差异表达基因的筛选;B.差异表达基因的功能注释统计。
A.Screening of differential transcription genes; B. Functional annotation of differential transcription genes.
图2 差异转录基因的筛选及功能注释统计
Fig.2 Screening and functional annotation of differential transcription genes
2.5 差异表达基因的GO分类
对样本冀HJ117与冀豆12的336个差异表达基因的GO功能注释进行统计分析,GO总共有3个类目,分别描述基因参与的生物过程、所处的细胞位置以及基因的分子功能。
图3是冀HJ117与冀豆12样品差异表达基因GO注释聚类图,从图中可知,在生物过程分组的差异表达基因注释到了15个GO条目中,其中新陈代谢过程有95个差异表达基因,占生物过程分类的22.9%,其中61个基因上调表达,34个基因下调表达;细胞过程有76个差异表达基因,占生物过程的18.4%,47个基因上调表达,29个基因下调表达;应激反应有58个差异表达基因,占14.0%,上调表达基因41个,下调表达基因17个;而生物过程正向调控所占比例最低,仅有2个差异表达基因下调表达,占比例0.5%。
在所处的细胞位置类目下差异表达基因共注释到6个GO条目中,其中细胞、细胞组分、细胞器3个条目的差异表达基因较多,分别有160,144,98个差异表达基因,占细胞位置的36.2%,32.7%,22.2%;封闭性膜和大分子复合物的差异表达基因数目最少,均为4个,占0.9%比例。
然而在分子功能分组中差异表达基因注释到5个GO条目,其中具有催化活性的差异表达基因有117个,占49.5%;具有连接活性的差异表达基因有101个,占42.8%;而酶调节活性和分子传感器活性均仅有2个差异表达基因,占该分组比例0.8%。
2.6 GO功能的显著性富集分析
GO功能显著性富集分析可以筛选出在差异表达基因中显著富集的GO功能条目,并筛选出差异表达基因与哪些生物学功能显著相关。结果如表3所示,参考基因在生物过程分组中有注释的基因共25 633个,本研究有注释的差异表达的基因共171个。在该分组中有3个条目显著富集,主要涉及应激反应、化学稳态以及离子稳态;在细胞位置分组,有注释的参考基因共27 869个,本研究注释到162个基因差异表达,并在高尔基堆,高尔基池2个条目达到显著富集;在分子功能分组,有注释的参考基因共29 635个,本研究有179个注释的差异表达基因,这些差异表达基因在氧化还原酶类、氧化金属离子、受体氧,氧化还原酶类、氧化金属离子,氧化还原酶类3个条目显著富集。
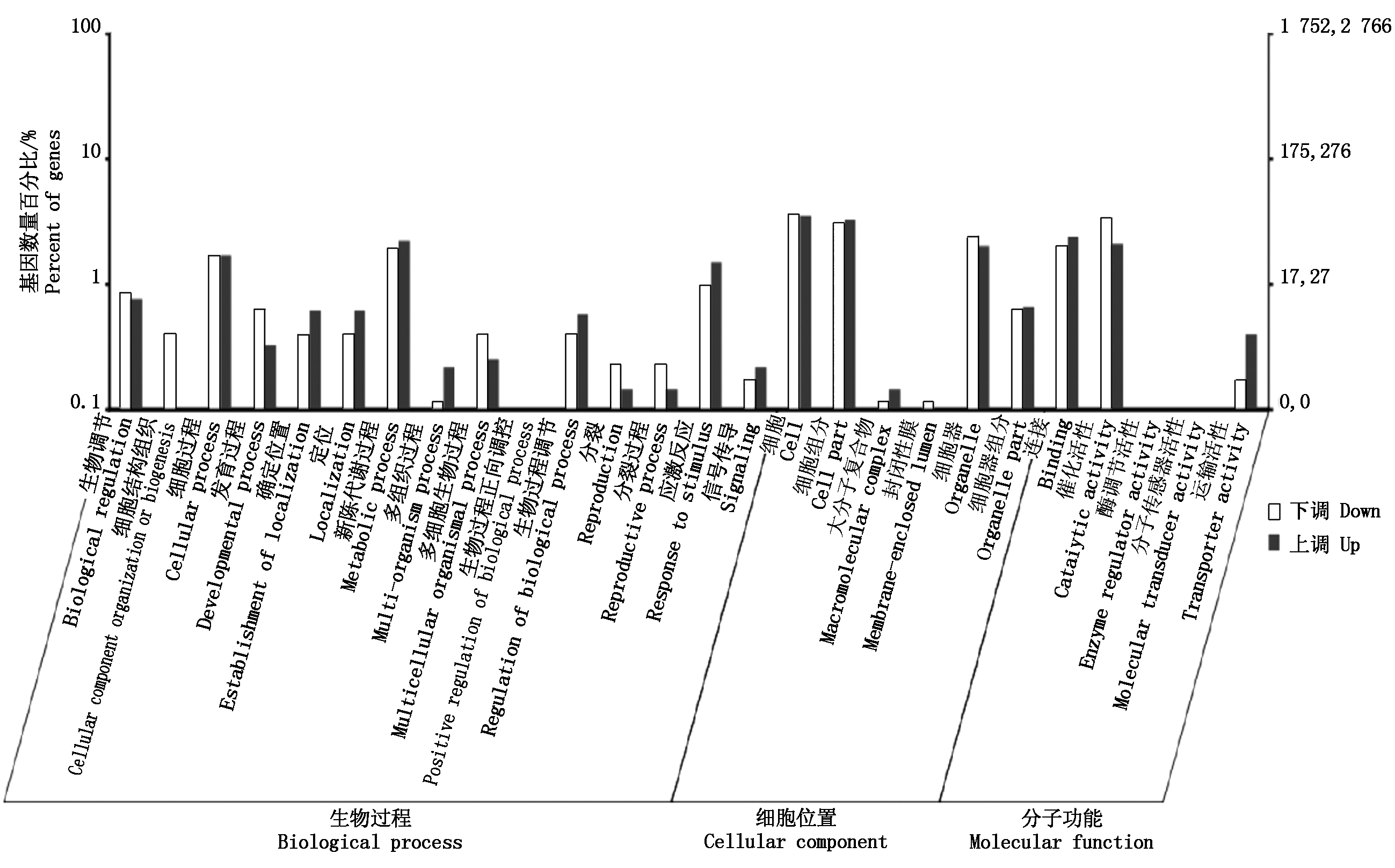
图3 差异表达基因GO注释聚类图
Fig.3 GO annotation of differentially expressed genes
表3 差异表达基因中显著富集的GO terms
Tab.3 Significantly enriched GO items in DEGs
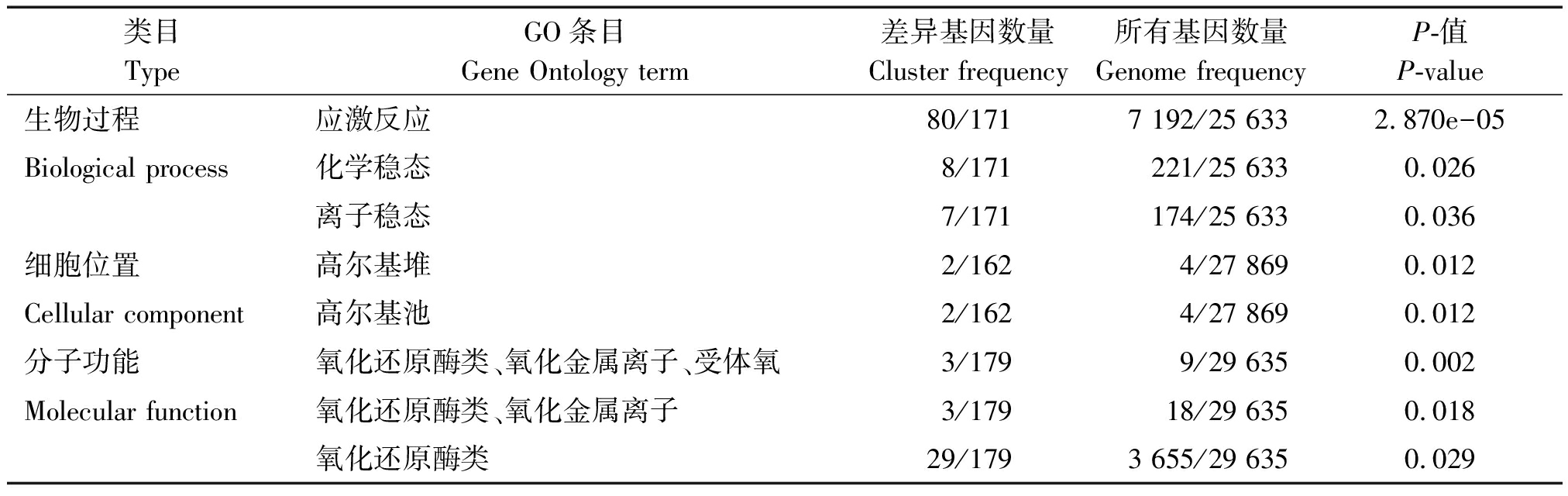
类目TypeGO条目Gene Ontology term差异基因数量Cluster frequency所有基因数量Genome frequencyP-值P-value生物过程应激反应80/1717 192/25 633 2.870e-05Biological process化学稳态8/171 221/25 633 0.026离子稳态7/171 174/25 633 0.036细胞位置高尔基堆2/162 4/27 869 0.012Cellular component高尔基池2/162 4/27 869 0.012分子功能氧化还原酶类、氧化金属离子、受体氧3/179 9/29 635 0.002Molecular function氧化还原酶类、氧化金属离子3/17918/29 635 0.018氧化还原酶类29/179 3 655/29 635 0.029
2.7 KEGG分析
将样本冀HJ117与冀豆12的表达基因与KEGG数据库比对,数据库中具有通路注释的基因共29 169个,本研究中注释到的差异表达的基因共有197个,参与了72个代谢通路。将Qvalue≤0.05的pathway定义为在差异表达基因中显著富集的Pathway。本试验的Pathway显著性富集分析结果发现,差异基因仅显著富集在蛋白质内质网合成途径中,该途径共有34个差异表达基因,占有注释的差异表达基因的17.26%,其中冀HJ117较冀豆12有33个基因上调表达,1个基因下调表达。
除此之外,差异表达基因还参与黄酮和黄酮醇的生物合成,丙氨酸、天门冬氨酸和谷氨酸代谢,脂肪酸生物合成,柠檬烯和蒎烯降解,类黄酮生物合成以及各类氨基酸的代谢和蛋白质输出途径等通路中,其详细信息统计于表4中。
图4为KEGG数据库中蛋白质内质网合成途径的详细信息,其中加粗方框为上调基因所在位置,虚线方框为同时存在上调基因和下调基因。差异表达基因推测的蛋白名称、所对应的KO号以及该基因的log2ratio值统计于表5中。从表中发现,差异表达基因所对应的表达蛋白主要有钙联蛋白(Calnexin,CNX)、结合免疫球蛋白(Binding immunoglobulin protein,Bip)、内质网膜结合连接酶(RING-finger protein with membrane anchor 1,RMA1)以及热激蛋白(Heat shoct proteins,Hsp20、Hsp40、Hsp70、Hsp90)。其中热激蛋白Hsp20的差异表达基因最多,共21个,其中20个基因上调表达,1个基因下调表达。
表4 Pathway显著性富集分析结果
Tab.4 Pathway enrichment analysis of DEGs
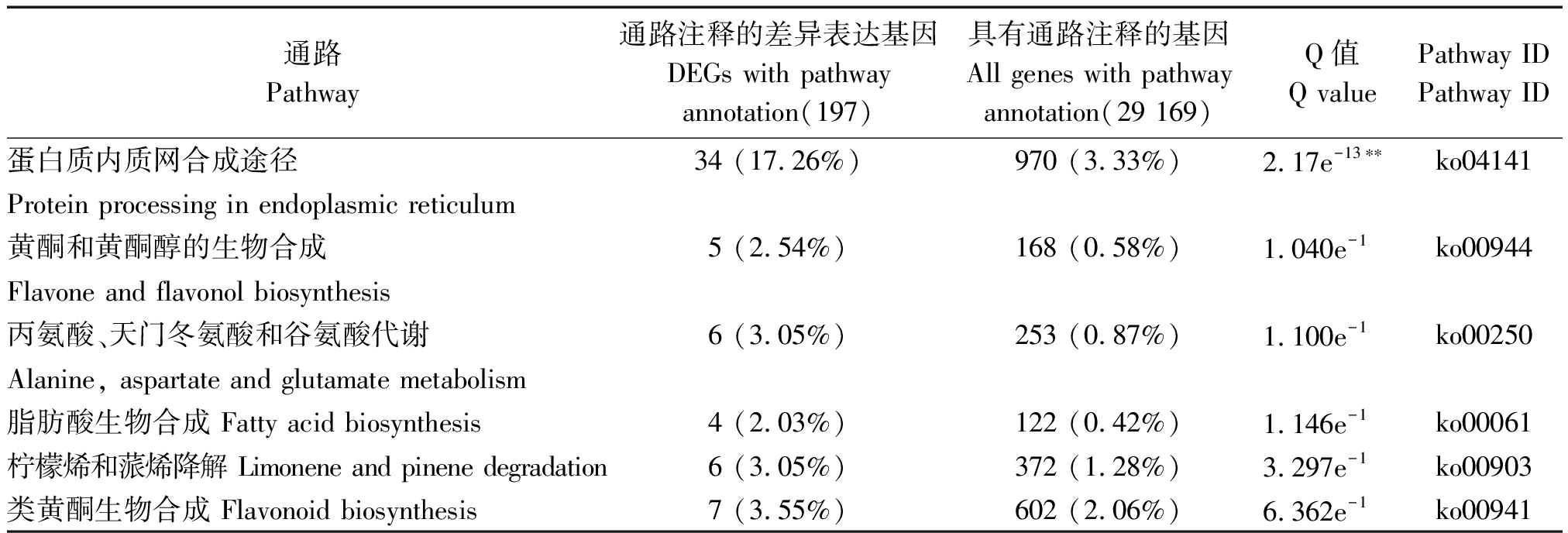
通路Pathway通路注释的差异表达基因DEGs with pathway annotation(197)具有通路注释的基因All genes with pathway annotation(29 169)Q值Q valuePathway IDPathway ID蛋白质内质网合成途径34 (17.26%)970 (3.33%)2.17e-13∗∗ko04141Protein processing in endoplasmic reticulum黄酮和黄酮醇的生物合成5 (2.54%)168 (0.58%)1.040e-1ko00944Flavone and flavonol biosynthesis丙氨酸、天门冬氨酸和谷氨酸代谢6 (3.05%)253 (0.87%)1.100e-1ko00250Alanine, aspartate and glutamate metabolism脂肪酸生物合成 Fatty acid biosynthesis4 (2.03%)122 (0.42%)1.146e-1ko00061柠檬烯和蒎烯降解 Limonene and pinene degradation6 (3.05%)372 (1.28%)3.297e-1ko00903类黄酮生物合成 Flavonoid biosynthesis7 (3.55%)602 (2.06%)6.362e-1ko00941
注:**.达到极显著差异。
Note:**.The difference is significant at the 0.01 level.

加粗标记为上调基因所在位置;虚线标记为下调基因所在位置。
The bold marker is the location of the up-regulated genes; The dotted marker is the location of the down-regulated gene.
图4 蛋白质内质网合成途径的详细信息
Fig.4 The detailed information of protein processing in endoplasmic reticulum
表5 蛋白质内质网合成途径差异表达基因详细信息
Tab.5 Details of genes in protein processing in endoplasmic reticulum

蛋白名称NameKO号KO number基因名称Gene name上/下调Up/downlog2ratioCNXK08054Glyma05g33330.1上调1.3Glyma08g00920.1上调1.0BiPK09490Glyma05g36620.1上调1.2RMA1K10666Glyma06g45850.1上调1.0Hsp40K09517Glyma19g40260.1上调1.0Hsp40K09503Glyma11g17930.2上调1.5Glyma12g10150.1上调1.1Hsp70K03283Glyma01g44910.1上调1.5Glyma02g36700.1上调1.3Glyma15g09430.1上调1.2Glyma18g52650.1上调1.1Hsp90K04079Glyma09g24410.1上调1.8Glyma16g29750.1上调1.6sHSF(Hsp20)K13993Glyma08g07330.1上调3.5Glyma19g01440.1上调3.5Glyma14g11430.1上调3.3Glyma08g07340.1上调3.0Glyma14g11420.1上调2.9Glyma16g33130.1上调2.8Glyma07g32030.1上调2.7Glyma17g34220.1上调2.6Glyma08g07350.1上调2.5Glyma13g24460.1上调2.1Glyma04g05720.1上调2.1Glyma06g05740.1上调2.0Glyma20g01930.1上调1.8Glyma02g41150.1上调1.6Glyma13g24480.1上调1.6Glyma13g24510.1上调1.6Glyma07g32050.1上调1.5Glyma11g14250.1上调1.4Glyma06g16490.1上调1.4Glyma13g24490.1上调1.3K13993Glyma07g32070.1下调-2.8
2.8 调控表达的转录因子分析
在冀HJ117与冀豆12差异表达的336个差异表达基因中,共有6个调控表达的转录因子,分属在5个转录因子家族中,分别是MYB、AP2、HSF、B3、以及Trihelix。其中有4个转录因子在冀HJ117中表达量较高,有2个转录因子在冀豆12中表达量较高,表6是差异表达基因中的转录因子的详细信息。
表6 差异表达基因中的转录因子
Tab.6 Transcription factors of differential transcription genes
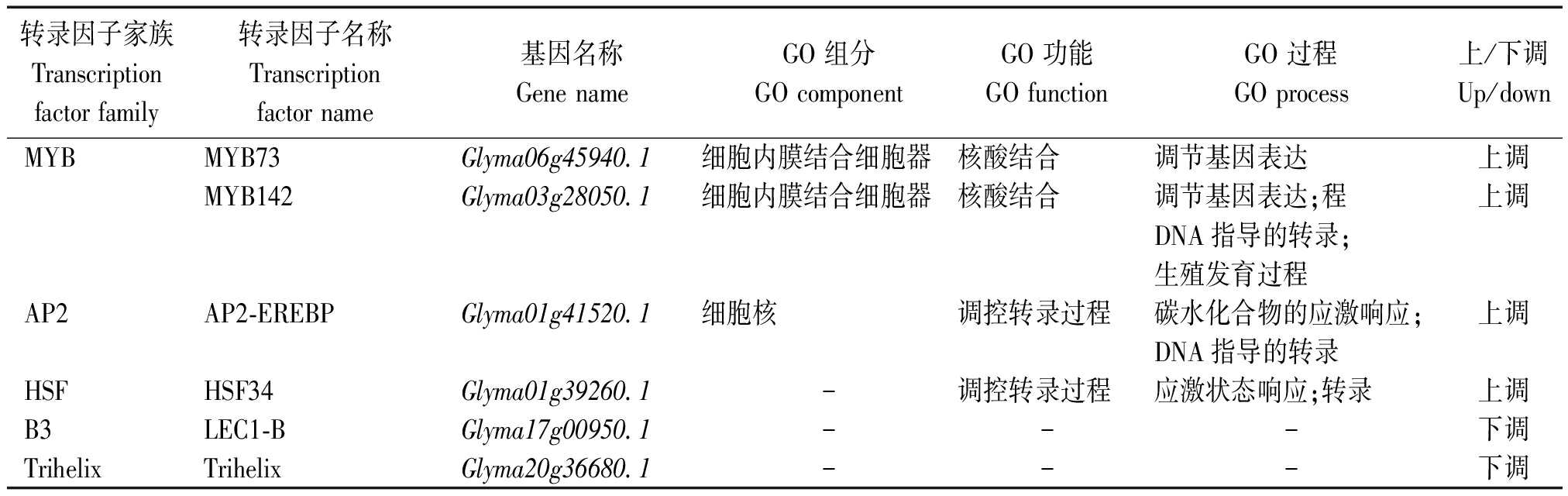
转录因子家族Transcription factor family转录因子名称Transcription factor name基因名称Gene nameGO 组分GO componentGO 功能GO functionGO 过程GO process上/下调Up/downMYB MYB73Glyma06g45940.1细胞内膜结合细胞器核酸结合调节基因表达上调MYB142Glyma03g28050.1细胞内膜结合细胞器核酸结合调节基因表达;程上调DNA指导的转录;生殖发育过程AP2AP2-EREBPGlyma01g41520.1细胞核调控转录过程碳水化合物的应激响应;上调DNA指导的转录HSFHSF34Glyma01g39260.1-调控转录过程应激状态响应;转录上调B3 LEC1-BGlyma17g00950.1---下调TrihelixTrihelixGlyma20g36680.1---下调
2.9 实时荧光定量PCR验证
为验证转录组测序数据的可靠性,从蛋白质内质网合成途径的34个差异表达基因中随机挑选9个基因(表1)进行qRT-PCR检测,经检测候选基因扩增的引物熔解曲线均为单峰,表明该引物对基因的扩增具有特异性。
通过对9个基因分别在冀HJ117与冀豆12 14 d籽粒中的表达量分析发现,候选基因在冀HJ117中的表达量均高于在冀豆12中的表达量(图5),该结果与RNA-Seq的分析结果趋势相一致。
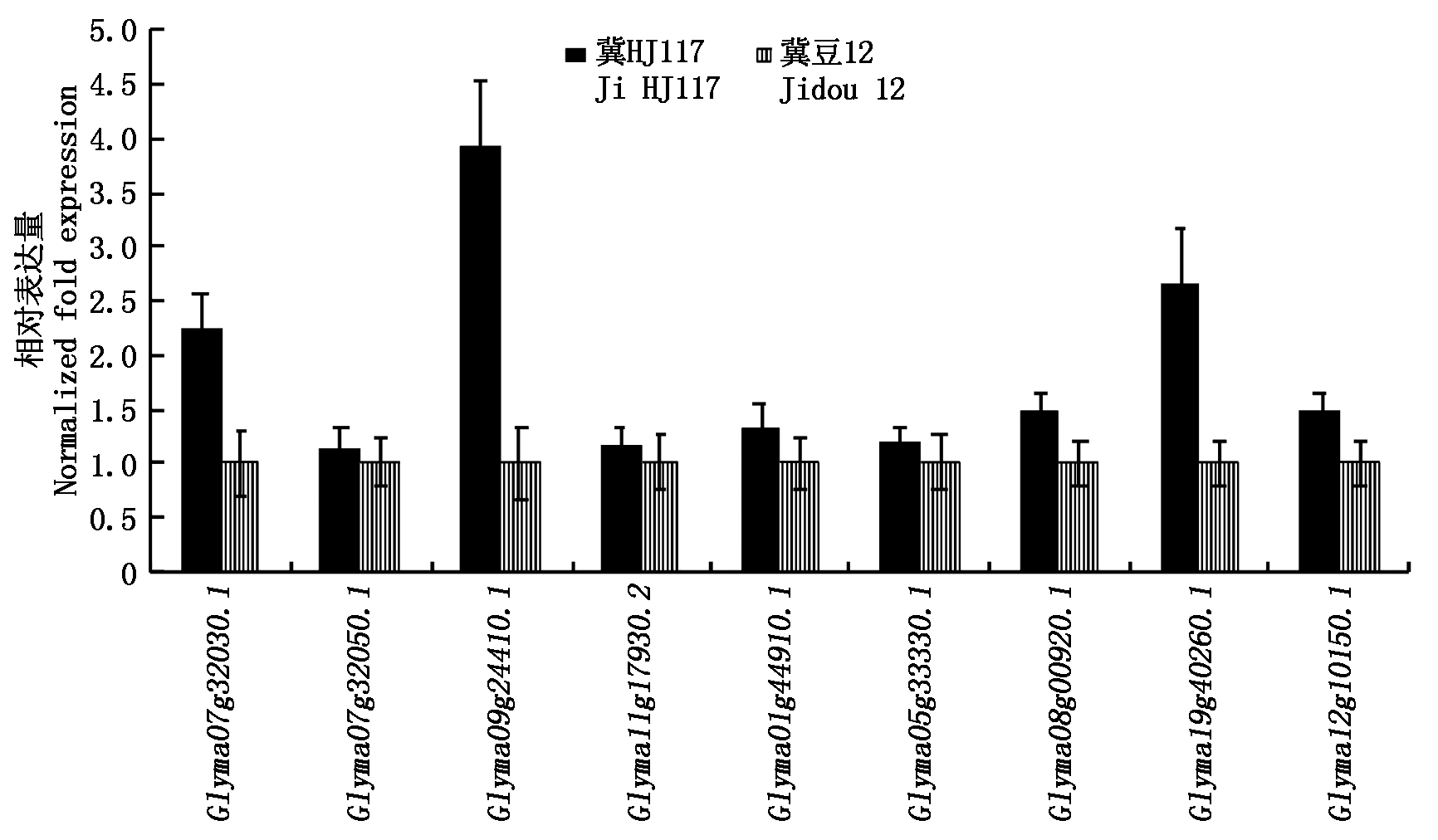
图5 候选基因在冀HJ117与冀豆12的表达量
Fig.5 Expression of candidate genes in Ji HJ117 and Jidou12
3 结论与讨论
蛋白质的合成代谢是个复杂的过程,主要涉及两方面:一是氨基酸的生理合成,即碳素和氮素的调运,二是贮藏蛋白基因的转录翻译及加工。大豆作为固氮植物将大气中的氮气转化为氨,氨通过转氨作用为其他氨基酸提供氨基,其中谷氨酰胺合成酶是氨代谢中的一个主要调控点。对于氨基酸的生物合成机理,多数氨基酸有其各自的合成通路,并与众多基因和酶的参与有关,其发生作用的位点、启动功能的时期以及如何使细胞内的各组分发生变化目前尚无定论[36]。杜若琛[37]研究发现,大豆种子的发育可分为 3 个时期:开花后 10~30 d为前期,该时期为蛋白质合成阶段;30~80 d为中期,该时期蛋白质发生快速积累,并合成贮藏蛋白;80~100 d为后期,蛋白质积累缓慢。其中贮藏蛋白占种子总蛋白含量的80%左右[38],可分为白蛋白、球蛋白、谷蛋白和醇溶蛋白4类。在种子的胚发育过程中,醇溶蛋白在粗面内质网合成,而后形成蛋白质聚集体,直接形成蛋白体并于其中贮存。白蛋白、球蛋白和谷蛋白首先在粗面内质网上形成分子量较大的前体,经过高尔基体的受体分选,进入特定的运输囊泡,经由受依赖型运输或聚集体形式运输至蛋白质贮存液泡中,最后经过液泡加工酶等的剪接转换为成熟型贮藏蛋白质[39]。
在本研究中,冀HJ117与冀豆12的差异表达基因显著富集在蛋白质内质网合成途径中,其差异表达的基因主要为钙联蛋白(CNX)、结合免疫球蛋白(Bip)、内质网膜结合连接酶(RMA1)以及热激蛋白(Hsp20、Hsp40、Hsp70、Hsp90)。该途径中,差异表达的Hsp基因所占比例最高,尤其以Hsp20的差异表达基因最多,共有21个。Hsp是一类从细菌到高等真核生物中普遍存在且高度保守的蛋白,根据分子量的大小可以分为5个家族,即Hsp100、Hsp90、Hsp70、Hsp60和小分子热激蛋白(sHSF),因为sHSF的分子量普遍在20 ku左右,故也称为Hsp20[40]。许多研究表明,热激蛋白在细胞中可以作为“分子伴侣”参与合成中的多肽反应,使其正确折叠;它还能够帮助新生肽穿过细胞膜结构,使蛋白质运转到细胞的不同部位发生作用;在高温等胁迫环境中,热激蛋白可以组织热变性蛋白的聚集,阻止蛋白的不可逆变性或有利于蛋白质变性后的复性[41]。
钙联蛋白是内质网中的一种磷酸化的钙结合蛋白,在哺乳细胞中它会与单葡糖基化修饰的糖蛋白相连接,帮助其成熟[42]。对于那些需要更多加工的非天然蛋白质,将会被UDP葡萄糖糖蛋白葡萄糖基转移酶(Glycoprotein glucosyltransferase,GT)识别,并使其聚糖分子A分支重新葡萄糖基化,这样就可以再次与钙联接蛋白或钙网织蛋白相连,进行重新折叠,这就是钙联接蛋白-钙网织蛋白循环(Calnexin-calreticulin cycle)[43]。Bip属于Hsp70家族蛋白在内质网体中的成员,参与进入内质网体的分泌型蛋白质和膜蛋白的折叠、贮存和转运过程[44]。
本研究中对差异表达基因进行GO功能的显著性富集分析,显示差异表达基因在氧化还原酶类条目中显著富集。而有研究表明,当蛋白质经过内质网的时候,会有一套“质控”系统将折叠正确的蛋白质释放,对于发生错误的蛋白将通过内质网相关的降解途径(ER-associated degradation,ERAD)运出内质网并降解[45]。在该降解途径中,Hsp70伴侣蛋白会与非天然蛋白结合,并使其与内质网膜结合酶Doa10、RMA1、HRD相结合,防止其转运至高尔基体。Hsp70分子蛋白还可以与氧化还原酶类蛋白一起帮助构象不正确的蛋白重新折叠,改变其构象[46]。携带有膜锚定结构的RMA1存在于内质网中,该蛋白是一种在拟南芥到人类中都广泛存在、非常保守的泛素连接酶[32]。RMA1蛋白能与UBE2J1蛋白相互作用,如果过表达RMA1蛋白会降低细胞内囊性纤维化病跨膜传导调节蛋白(Cystic fibrosis transmembrane conductance regulator,CFTR)的水平[47]。RMA1蛋白最主要的作用是在CFTR蛋白翻译过程中或翻译刚刚完成之后检测CFTR蛋白N末端结构域的折叠情况。
本研究中还筛选到一些与蛋白质合成相关的转录因子,主要包括5个家族的转录因子,分别是MYB、AP2、Hsf、B3、以及Trihelix家族的转录因子。前人研究表明,热休克转录因子是一组结构及功能具有广泛同源性,普遍存在于真核生物细胞中的蛋白质,其结构高度保守[48]。植物中Hsfs分为A、 B、 C 3个家族,又分为16个亚族。前人研究表明,植物中存在的Hsfs,能够与Hsps的启动子区域的热机元件(HSEs)结合,从而启动热激反应,激活下游基因的表达,对提高植物的耐热性有重要的调控作用。大豆中有38个Hsfs基因,划分到3个家族,12个亚族中,其中GmHsf-34基因在拟南芥中的超量表达,提高了植株对干旱和热胁迫的耐受性[49]。Chauhan等[50]从小麦种子中克隆了TaHsfA2d,转TaHsfA2d 拟南芥植株不仅表现耐高温,还表现出耐盐和抗旱性,同时转基因植株还表现出较高的产量和生物量积累。在黄淮海地区,大豆的生长季在6-10月之间,而大豆一般在7月下旬进入开花期,因此,大豆的开花期至鼓粒期是黄淮海地区最热的时期。本研究中检测到一个Hsf转录因子的高表达,推测该基因与提高大豆的耐热性有关,对蛋白质合成的影响还需进一步的研究。除此之外,在冀HJ117与冀豆12的差异表达基因中还检测到MYB和B3类转录因子,Ezcurra [18]和Reidt [15]等研究表明,该类转录因子对蛋白质的合成具有调控影响。因此,推测本研究筛选到的转录因子与蛋白质合成密切相关,但这些转录因子具体的作用机制还需深入的研究。
综合上述研究,本研究以遗传背景接近的冀HJ117与冀豆12为材料,对开花后14 d的籽粒进行了转录组测序,筛选到与蛋白质合成相关的基因。差异表达基因富集的GO分子功能条目与蛋白折叠有关,并且通过Pathway显著性富集分析发现差异表达基因富集在蛋白质内质网合成途径中,该途径中的钙联蛋白、结合免疫球蛋白、热激蛋白以及内质网膜结合连接酶均在蛋白质合成通路中起关键作用,因此,该途径是冀HJ117与冀豆12蛋白质含量产生差异的重要通路。
RNA-Seq分析获得了大豆中蛋白质合成相关的基因的基本信息及其可能的生物学功能。蛋白质内质网合成途径可能是冀豆12与冀HJ117蛋白含量产生差异的重要通路,因此,在该途径中富集的差异表达基因以及筛选到的差异表达的转录因子很可能是与蛋白合成密切相关的基因。
[1] 赵团结, 盖钧镒. 栽培大豆起源与演化研究进展[J]. 中国农业科学, 2004, 37(7): 954-962. doi: 10.3321/j.issn:0578-1752.2004.07.004.
Zhao T J, Gai J Y. The origin and evolution of cultivated soybean[J]. Scientia Agricultura Sinica, 2004, 37(7): 954-962. doi: 10.3321/j.issn:0578-1752.2004.07.004.
[2] 郑宇宏, 陈亮, 孟凡凡, 范旭红, 张云峰, 孙星邈, 王明亮, 王曙明. 吉林省不同年代大豆育成品种产量与品质性状变化趋势[J]. 东北农业科学, 2016, 41(6): 45-49. doi: 10.16423/j.cnki.1003-8701.2016.06.010.
Zheng Y H, Chen L, Meng F F, Fan X H, ZhangY F, Sun X M, Wang M L, Wang S M. Changes of yield and quality traits of soybean cultivars released during different stages in Jilin Province[J]. Journal of Northeast Agricultural Sciences, 2016, 41(6):45-49. doi: 10.16423/j.cnki.1003-8701.2016.06.010.
[3] 张伟, 王曙明, 邱强, 闫晓艳, 彭宝, 张晓霞, 姜海英. 从品种志分析吉林省八十五年来大豆育成品种产量和品质的演变[J]. 大豆科学, 2009, 28(6): 970-975. doi: 10.11861/j.issn.1000-9841.2009.06.970.
Zhang W, Wang S M, Qiu Q, Yan X Y, Peng B, Zhang X X, Jiang H Y. Changes of yield and quality traits of released soybean cultivars during Past 85 Years in Jilin Province[J]. Soybean Science, 2009, 28(6): 970-975. doi: 10.11861/j.issn.1000-9841.2009.06.970.
[4] Hwang E Y, Song Q J, Jia G F, Specht J E, Hyten D L, Costa1 J, Cregan P B. A genome-wide association study of seed protein and oil content in soybean[J]. BMC Genomics 2014, 15:1. doi: 10.1186/1471-2164-15-1.
[5] Bolon Y T, Joseph B, Cannon S B, Graham M A, Diers B W, Farmer A D, May G D, Muehlbauer G J, Specht J E, Tu Z J, Weeks N, Xu W W, Shoemaker R C, Vance C P. Complementary genetic and genomic approaches help characterize the linkage group I seed protein QTL in soybean[J]. BMC Plant Biology, 2010, 10: 41. doi: 10.1186/1471-2229-10-41.
[6] Kyujung Van, McHale L K. Meta-Analyses of QTLs associated with protein and oil contents and compositions in soybean9(Glycine max (L.) Merr.)seed[J]. International Journal of Molecular Sciences, 2017, 18(6): 1180. doi: 10.3390/ijms18061180.
[7] Patil G, Mian R, Vuong T, Pantalone V, Song Q, Chen P, Shannon G J, Carter T C, Nguyen H T. Molecular mapping and genomics of soybean seed protein: a review and perspective for the future[J]. Theoretical and Applied Genetics, 2017, 130(10):1975-1991. doi: 10.1007/s00122-017-2955-8.
[8] 魏荷, 王金社, 卢为国. 大豆籽粒蛋白质含量分子遗传研究进展[J]. 中国油料作物学报, 2015, 37(3): 394-410. doi: 1007-9084.2015.03.021.
Wei H, Wang J S, Lu W G. Molecular genetic advances in soybean seed protein[J]. Chinese Journal of Oil Crop Sciences, 2015, 37(3): 394-410. doi: 1007-9084.2015.03.021.
[9] Cober E R, Voldeng H D. Developing high-protein, high-yield soybean populations and lines[J]. Crop Science, 2000, 40(1): 39-42. doi: 10.2135/cropsci2000.40139x.
[10] Schmutz J, Cannon S B, Schlueter J, Ma J, Mitros T, Nelson W, Hyten D L, Song Q, Thelen J J, Cheng J, Xu D, Hellsten U, May G D, Yu Y, Sakurai T, Umezawa T, Bhattacharyya M K, Sandhu D, Valliyodan B, Lindquist E, Peto M, Grant D, Shu S, Goodstein D, Barry K, Futrell-Griggs M, Abernathy B, Du J, Tian Z, Zhu L, Gill N, Joshi T, Libault M, Sethuraman A, Zhang X C, Shinozaki K, Nguyen H T, Wing R A, Cregan P, Specht J, Grimwood J, Rokhsar D, Stacey G, Shoemaker R C, Jackson S A. Genome sequence of the palaeopolyploid soybean[J]. Nature, 2010, 463(7278): 178-183. doi: 10.1038/nature08670.
[11] Phansak P, Soonsuwon W, Hyten D L, Song Q J, Cregan P B, Graef G L, Specht J E.Multi-Population selective genotyping to identify soybean [Glycine max (L.) Merr.] seed protein and oil QTLs[J]. G3 Genesgenetics, 2016, 6: 1635-1648. doi: 10.1534/g3.116.027656.
[12] Nichols D M, Glover K D, Carlson S R, Specht J E, Diers B W. Fine mapping of a seed protein QTL on soybean linkage group I and its correlated effects on agronomic traits[J]. Crop Science, 2006, 46(2): 834-839. doi: 10.2135/cropsci2005.05-0168.
[13] Chen J Q, Lang C X, Hu Z H. Antisense PEP gene to ratio of protein and lipid content in Brassica napus seeds[J]. Journal of Agricultural Biotechnology, 1999, 7(4): 316-320. doi: 10.1016/S0140-6736(69)91396-8.
[14] Wang H W, Zhang B, Hao Y J, Tian A G, Liao Y, Zhang J S, Chen S Y. The soybean Dof-type transcription factor genes, GmDof4 and GmDof11, enhance lipid content in the seeds of transgenic Arabidopsis plants[J]. The Plant Journal, 2007, 52(4): 716-729. doi: 10.1111/j.1365-313X.2007.03268.x.
[15] Verdier J, Thompson R D. Transcriptional regulation of storage protein synthesis during dicotyledon seed filling[J]. Plant Cell Physiol, 2008, 49(9): 1263-1271. doi:10.1093/pcp/pcn116.
[16] Ezcurra I, Wycliffe P, Nehlin L, Ellerstrom M, Rask L. Transactivation of the Brassica napus napin promoter by ABI3 requires interaction of the conserved B2 and B3 domains of ABI3 with different cis-elements: B2 mediates activation through an ABRE, whereas B3 interacts with an RY/G-box[J]. Plant Journal, 2000, 24(1): 57-66. doi: 10.1046/j.1365-313x.2000.00857.x.
[17] 王楠. 谷子与锈菌互作的转录组和表达谱研究及相关基因表达分析[D].石家庄:河北师范大学, 2015.
Wang N. The bac library construction and transcriptome analysis of wheat material which carry YR26 gene[D].Shijiazhuang: Hebei Normal University, 2015.
[18] Luan H Y, Shen H Q, Zhang Y H, Zang H, Qiao H L, Hong T, Chen J, Chen H. Comparative transcriptome analysis of barley(Hordeum vulgare L.)glossy mutant using RNA-Seq[J]. Brazilian Journal of Botany, 2016, 27(11): 1-10. doi: 10.1007/s40415-016-0328-1.
[19] Wang L, Liu L, Ma Y, Li S, Dong S, Zu W. Transcriptome profilling analysis characterized the gene expression patterns responded to combined drought and heat stresses in soybean[J]. Computation Biology Chemistry, 2018, 77: 413-429. doi: 10.1016/j.compbiolchem.2018.09.012.
[20] Wang Q Q, Liu F, Chen X S, Ma X J, Zeng H Q, Yang Z M. Transcriptome profiling of early developing cotton fiber by deep-sequencing reveals significantly differential expression of genes in a fuzzless/lintless mutant[J]. Genomics, 2010, 96(6): 369-376. doi:10.1016/j.ygeno.2010.08.009.
[21] 和小燕, 胡晓锋, 王允, 张幸果, 李贺敏, 崔党群, 殷冬梅. 花生籽仁不同发育时期的转录组测序分析[J]. 分子植物育种, 2016, 14(11): 2930-2943.
He X Y, Hu X F, Wang Y, Zhang X G, Li H M, Cui D Q, Yin D M. Sequencing analysis of transcriptome during the different developmental stages in peanut seed[J]. Molecular Plant Breeding, 2016, 14(11): 2930-2943.
[22] Julio V, Enrique I, Beatriz J,Martínez O, Vielle-Calzada J P, Herrera-Estrella L, Herrera-Estrella A. Deep sampling of the palomero maize transcriptome by a high throughput strategy of pyrosequencing[J]. BMC Genomics, 2009, 10(1): 1-10. doi: 10.1186/1471-2164-10-299.
[23] Alagna F, D′Agostino N, Torchia L, Servili M, Rao R, Pietrella M, Giuliano G, Chiusano M L, Baldoni L, Perrotta G. Comparative 454 pyrosequencing of transcriptsfrom two olive genotypes during fruit development[J]. BMC Genomics, 2009, 10(1): 399-414. doi: 10.1186/1471-2164-10-399.
[24] 栾海业, 臧慧, 沈会权,张英虎,乔海龙,陶红,申玉香. 大麦白化颖壳突变体的转录组学分析[J]. 核农学报, 2017, 31(12): 2332-2339.
Luan H Y, Zang H, Shen H Q, Zhang Y H, Qiao H L, Tao H, Shen Y X. Transcriptome analysis of albino lemma mutant in barley[J]. Journal of Nuclear Agricultural Sciences, 2017, 31(12): 2332-2339.
[25] 陈静. 花生种子休眠解除过程中相关基因转录组学研究[D].南京:南京农业大学,2004.
Chen J. Analysis on transcriptome of genes involved in peanut seed dormancy release[D].Nanjing: Nanjing Agricultural University, 2004.
[26] 李海燕, 王芳, 段玉玺, 陈立杰. 大豆胞囊线虫侵染诱导五寨黑豆早期的转录组分析[J]. 中国油料作物学报, 2015, 37(2): 194-200. doi: 10.7505/j.issn.1007-9084.2015.02.011.
Li H Y, Wang F, Duan Y X, Chen L J. Transcriptome analysis of Wuzhai heidou infected by Heterodera glycine[J]. Chinese Journal of Oil Crop Sciences, 2015, 37(2): 194-200. doi: 10.7505/j.issn.1007-9084.2015.02.011.
[27] 黄启秀, 曲延英, 姚正培,李梦雨,陈全家. 海岛棉枯萎病抗性与类黄酮代谢途径基因表达量的相关性[J]. 作物学报, 2017, 43(12): 1791-1801. doi: 10.3724/SP.J.1006.2017.01791.
Huang Q X,Qu Y Y, Yao Z P, Li M Y, Chen Q J. Correlation between resistance to Fusarium wilt and expression of flavonoid metabolism related genes in Gossypium barbadense L.[J].Acta Agronimica Sinica,2017, 43(12):1791-1801. doi: 10.3724/SP.J.1006.2017.01791.
[28] Cohen S P, Liu H, Argueso C T, Pereira A, Vera Cruz C, Verdier V, Leach J E. RNA-Seq analysis reveals insight into enhanced rice Xa7-mediated bacterial blight resistance at high temperature[J]. PLoS One, 2017,12(11):e0187625. doi: 10.1371/journal.pone.0187625.
[29] 李永辉, 陈琳琳, 孙炳剑, 王利民,邢小萍,袁虹霞,丁胜利,李洪连. 假禾谷镰孢侵染小麦后 3 种植物激素相关基因的差异表达分析[J]. 作物学报, 2017, 43(11): 1632-1642. doi: 10.3724/SP.J.1006.2017.01632.
Li Y H, Chen L L, Sun B J, Wang L M, Xing X P, Yuan H X, Ding S L, Li H L. Differential expression of three plant hormone related genes in wheat in-fected by Fusarium pseudograminearum[J]. Acta Agronmica Sinica,2017, 43(11):1632-1642. doi: 10.3724/SP.J.1006.2017.01632.
[30] 曾庆东. 小麦抗条锈病基因Yr26载体材料BAC文库构建及转录组测序[D].杨凌:西北农林科技大学,2016.
Zeng Q D. The BAC library construction and transcriptome analysis of wheat material which carry Yr26 gene[J].Yangling: North West Agriculture and Forestry University,2016.
[31] 许家磊. 基于甘薯徐781和徐薯18转录组测序的SNP标记开发[D]. 北京: 中国农业科学院, 2015. doi: 10.7666/d.Y2787474.
Xu J L. SNP marker development based on sequencing of sweet potato Xu 781 and Xushu 18 transcriptome[D]. Beijing:China Academic Journal Electronic Publishing House, 2015.doi: 10.7666/d.Y2787474.
[32] 任梦露, 刘卫国, 刘婷, 杜勇利, 邓榆川, 邹俊林, 袁晋, 杨文钰. 荫蔽胁迫下大豆茎秆形态建成的转录组分析[J]. 作物学报, 2016, 42(9): 1319-1331. doi: 10.3724/SP.J.1006.2016.01319.
Ren M L, Liu W G, Liu T, Du Y L, Deng Y C, Zou J L, Yuan J, Yang W Y. Transcriptome analysis of stem morphogenesis under shade stress in soybean[J]. Acta Agtonomica Sinica, 2016, 42(9): 1319-1331.doi: 10.3724/SP.J.1006.2016.01319.
[33] 孙爱清, 张杰道, 万勇善, 刘风珍, 张昆, 孙利. 花生干旱胁迫响应基因的数字表达谱分析[J]. 作物学报, 2013, 39(6): 1045-1053. doi:10.3724/SP.J.1006.2013.01045.
Sun A Q, Sun J D, Wan Y S, Liu F Z, Zhang K, Sun L. In silico expression profile of genes in response todrought in peanut[J]. Acta Agronomica Sinica, 2016, 39(6): 1045-1053. doi:10.3724/SP.J.1006.2013.01045.
[34] Mortazavi A, Williams B A, Mccue K, Schaeffer L, Wold W. Mapping and quantifying mammalian transcriptomes by RNA-Seq[J]. Nature Methods, 2008, 5(7): 621-628. doi: 10.1038/nmeth.1226.
[35] Audic S, Claverie J M. The significance of digital gene expression profiles[J]. Genome Research, 1997, 7(10): 986-995. doi: 10.1101/gr.7.10.986.
[36] 陈欢. 大豆籽粒不同发育时期基因表达谱的研究[D]. 长春:吉林农业大学, 2012.
Chen H. Gene expression profile of developing soybean seed[D]. Changchun:Jilin Agricultural University, 2012.
[37] 杜若琛. 大豆籽粒发育过程中 ABI3-like 与贮藏蛋白基因的关系及调控机制的研究[D].晋中:山西农业大学,2016.
Du R C. Study on the relationship between ABI3-like and storage protein genes during the development of soybean seeds and its regulation mechanism[D]. Jinzhong:Shanxi Agricultural University, 2016.
[38] Fuji K, Shimada T, Takahashi H, Tamura K, Koumoto Y, Utsumi S, Nishizawa K, Maruyama N, Hara-Nishimura I. Arabidopsis vacuolar sorting mutants(green fluorescent seed)can be identified efficiently by secretion of vacuole-targeted green fluorescent protein in their seeds[J]. Plant Cell, 2007, 19(2): 597-609. doi: 10.1105/tpc.106.045997.
[39] 韩宝达, 李立新. 植物种子贮藏蛋白质及其细胞内转运与加工[J]. 植物学报, 2010, 45(4): 492-505. doi:10.3969/j.issn.1674-3466.2010.04.013.
Han B D, Li L X. Seed storage proteins and their intracellular transport and processing[J]. Chinese Bulletin of Botany, 2010, 45(4): 492-505.doi:10.3969/j.issn.1674-3466.2010.04.013.
[40] 张宁, 姜晶. 植物中小分子热激蛋白基因家族(sHSPs)研究进展[J]. 植物生理学报, 2017(6): 943-948. doi: CNKI:SUN:ZWSL.0.2017-06-006.
Zhang N, Jiang J. Research advances of small heat shock protein gene family (sHSPs) in plants[J]. Plant Physiology Journal, 2017, 53(6): 943-948. doi: CNKI:SUN:ZWSL.0.2017-06-006.
[41] 王义菊. 过量表达小分子热激蛋白对番茄耐热性的影响[D]. 济南: 山东师范大学, 2005.
Wang Y J. The effects of overexpression of small shock proteins on the thermotolerence in tomato[D]. Jinan: Shandong Normal University, 2005.
[42] Molinari M. N-glycan structure dictates extension of protein folding or onset of disposal[J]. Nature Chemical Biology, 2007, 3(6): 313-320. doi: 10.1038/nchembio880.
[43] Soldà T, Galli C, Kaufman R J, Molinari M. Substrate-specific requirements for UGT1-dependent release from calnexin[J]. Molecular Cell, 2007, 27(2): 238-249. doi: 10.1016/j.molcel.2007.05.032.
[44] Hebert D N, Foellmer B, Helenius A. Glucose trimming and reglucosylation determine glycoprotein association with calnexin in the endoplasmic reticulum[J]. Cell, 1995, 81(3): 425-433. doi: 10.1016/0092-8674(95)90395-x.
[45] Hirsch C, Gauss R, Horn S C, Neuber O, Sommer T. The ubiquitylation machinery of the endoplasmic reticulum[J]. Nature, 2009, 458(7237): 453-460. doi: 10.1038/nature07962.
[46] Gurkan C, Stagg S M, Lapointe P, Balch W E. The COPII cage: unifying principles of vesicle coat assembly[J]. Nature Reviews Molecular Cell Biology, 2006, 7(10): 727-738. doi: 10.1038/nrm2025.
[47] Younger J M, Chen L, Ren H Y, Rosser M F, Turnbull E L, Fan C Y, Patterson C, Cyr D M. Sequential quality-control checkpoints triage misfolded cystic fibrosis transmembrane conductance regulator[J]. Cell, 2006, 126(3): 571-582. doi: 10.1016/j.cell.2006.06.041.
[48] 吴迪. 内质网中FKBP23与BiP结合后对BiP的ATPase酶活性的影响[D]. 天津: 南开大学, 2005. doi: 10.7666/d.y804786.
Wu D. Effect of binding of FKBP23 to BiP on ATPase activity of BiP in endoplasmic reticulum[D].Tianjin: Nankai University, 2005. doi: 10.7666/d.y804786.
[49] Li P S, Yu T F, He G H, Chen M, Zhou Y B, Chai S H, Xu Z S, Ma Y Z. Genome-wide analysis of the Hsf family in soybean and functional identification of GmHsf34 involvement in drought and heat stresses[D].BMC Genomics, 2014,15(1):1-16. doi: 10.1186/1471-2164-15-1009.
[50] Chauhan H, Khurana N, Agarwal P, Khurana J P, Khurana P. A seeds preferential heat shock transcription factor from wheat provides abiotic stress tolerance and yield enhancement in transgenic Arabidopsis under heat stress environment[J]. PloS One, 2013, 8 (11):e79577. doi: 10.1371/journal.pone.0079577.