薏苡属(Coix)植物属于禾本科玉蜀黍族的一年生或多年生草本植物,世界上大约有10个种(变种),根、茎、叶、籽粒均可入药,其籽粒脱壳之后为薏(苡)仁米,具有营养、抗肿瘤、降血糖、降血脂、美白养颜、镇痛等多种滋养和药理功效,为“药(医)食同源”植物之典型代表。薏苡属资源分布广泛,主要分布于东南亚、印度、中国、日本、韩国等国家和地区,中国除了青、甘、宁未见报道外,其余各省(区)均有分布。因此,如何有效的评价薏苡的遗传多样性,对于加快优异资源、基因的保护和利用具有重要意义。薏苡种质资源的收集、保存等工作起步于1985-1995年期间的资源战略普查,黄亨履等[1]首次将中国薏苡资源划分为南方晚熟生态型、长江中下游中熟生态型和北方早熟生态型3个生态型,也有学者针对薏苡资源的农艺、种子性状的多样性等进行了比较详实的描述和分类[2-5]。贵州省薏苡工程技术研究中心为了有效规范薏苡种质资源的收集、整理和保存工作,在总结前人研究和多年薏苡资源种植观察的基础上制定了描述规范和数据采集标准[6],有效地规范了薏苡种质资源的收集、保存和整理工作,也对部分资源的表型多样性特征进行了分类和描述[7],为薏苡的资源的高效利用提供了重要参考。SSR分子标记具有技术简单、稳定性好以及共显性等特点,在遗传多样性分析[8-9]、DNA指纹图谱鉴定[10-11]、基因定位和分子育种[12]等方面应用广泛。目前,已有学者开发和利用SSR标记来探索薏苡资源的多样性,Ma等[13-14]开发了17对SSR引物并用以分析了来自中国和韩国的79个薏苡种质资源,聚类分析发现,大部分中国薏苡聚为一类,而所有的韩国薏苡聚为一类,揭示出中国薏苡和韩国薏苡有着比较远的亲缘关系;郭银萍等[15]从85对玉米SSR引物和72对水稻SSR引物中筛选出11对通用性较好引物将供试薏苡自交系划分为4类。直至2019年,中国农业大学和四川农业大学首次联合破译了栽培种(cultivar Beijing coix)和水生薏苡(Coix aquatica)的基因组序列[16-17];但是基于核基因组开发的SSR标记引物数量却还相当有限,标记覆盖度低,因此在多样性分析、指纹鉴定中的鉴别力也极为受限。而首个薏苡叶绿体基因组已经测序组装完成[18],本研究中心前期也基于叶绿体序列信息进行了SSR标记开发[19],为研究薏苡属遗传多样性及其演化提供了重要手段。
本研究利用叶绿体基因组SSR标记对薏苡种质资源进行遗传多样性和亲缘关系分析,并揭示不同地域、生态类型及野生、栽培类群间的遗传多样性和演化关系,为薏苡属资源的保护和种质创新提供理论支撑。
1 材料和方法
1.1 试验材料
供试材料来自中国农业科学院作物研究所的中期库薏苡种质资源,共93份,主要来源于中国16个省份(北京1份、山东1份、河北5份、山西3份、辽宁3份、吉林1份、湖南3份、江西1份、江苏2份、四川3份、贵州26份、云南29份、广西1份,福建7份、浙江2份、台湾1份)和5份老挝种质(表1),包含了薏苡属的6个种(变种)。根据不同材料的地理分布,将所有材料划分为3个生态型(区):北方早熟生态型(北京、山东、河北、山西、辽宁、吉林)、长江中下游中熟生态型(湖南、江西、江苏、四川)、南方晚熟生态型(贵州、云南、广西,福建、浙江、台湾);老挝种质划分为国外类型。
1.2 试验方法
1.2.1 DNA的提取 取饱满的籽粒温室育苗,待生长至3~4叶,采用天根植物新型基因组DNA提取试剂盒进行总DNA提取,1.0%琼脂糖凝胶电泳检测DNA的完整性,紫外分光光度法检测DNA浓度,稀释至30~50 ng/μL使用。
1.2.2 SSR标记检测 扩增引物选取贵州省薏苡工程技术研究中心基于叶绿体基因组开发的38对SSR引物[19]。PCR反应总体系10 μL, 包括2×Taq PCR MasterMix(Sangon Biotech)5 μL,正向引物(10 μmol/L)0.25 μL,反向引物(10 μmol/L)0.25 μL,DNA模板1.0 μL,ddH2O 3.5 μL。反应程序:94 ℃预变性5 min;94 ℃变性30 s,50~60 ℃退火30 s,72 ℃延伸1 min,32个循环;最后72 ℃延伸5 min,12 ℃保存。Fragment Analyzer毛细管电泳进行SSR分型检测。
1.3 数据统计分析
每对SSR引物检测一个位点,每个多态性条带视为一个等位基因,在相同迁移率的位置上,以1(有条带)、0(无条带)记录每个样品的扩增带。Popgene软件[20]计算不同位点和群体的观测等位基因数(Na)、有效等位基因数(Ne)、Nei′s多样性指数(H)和Shannon′s信息指数(I)。NTSYS.2.10e软件[21]计算群体Nei′s遗传距离和相似系数(一致度)并进行亲缘关系聚类。
表1 薏苡属种质资源信息
Tab.1 Details of Coix L. resources

编号Number种质库编号Accession number名称Name来源Origin拉丁名Latin name类型Type生态型 Ecotype1ZI000285嘎洒野生薏苡中国云南C. puellarum Balansa野生南方晚熟生态型2ZI000286嘎洒野生薏苡中国云南C. puellarum Balansa野生南方晚熟生态型3ZI000287巴达野生薏苡中国云南C. puellarum Balansa野生南方晚熟生态型4ZI000288野生薏苡中国云南C. lacryma-jobi var. lacryma-jobi野生南方晚熟生态型5ZI000289象明薏苡中国云南C. lacryma-jobi var. lacryma-jobi野生南方晚熟生态型6ZI000290嘎洒野生薏苡2号中国云南C. lacryma-jobi var. lacryma-jobi野生南方晚熟生态型7ZI000291嘎洒野生薏苡3号中国云南C. lacryma-jobi var. lacryma-jobi野生南方晚熟生态型8ZI000292嘎洒野生薏苡4号中国云南C. lacryma-jobi var. lacryma-jobi野生南方晚熟生态型9ZI000293兰壳薏苡中国云南C. chinensis var. formosana(Ohwi)L.栽培南方晚熟生态型10ZI000294佛鑫3号中国云南C. chinensis var. chinesis Tod.栽培南方晚熟生态型11ZI000295巴达栽培薏苡中国云南C. chinensis var. chinesis Tod.栽培南方晚熟生态型12ZI000296浙江小粒中国浙江C. chinensis var. chinesis Tod.栽培南方晚熟生态型13ZI000297浙江大粒中国浙江C. chinensis var. chinesis Tod.栽培南方晚熟生态型14ZI000298太谷2-4中国山西C. chinensis var. chinesis Tod.栽培北方早熟生态型15ZI000299太谷2-14中国山西C. chinensis var. chinesis Tod.栽培北方早熟生态型16ZI000300安国薏苡中国河北C. chinensis var. chinesis Tod.栽培北方早熟生态型17ZI000301承德薏苡中国河北C. chinensis var. chinesis Tod.栽培北方早熟生态型18ZI000302川紫薏苡中国四川C. chinensis var. chinesis Tod.栽培长江中下游中熟生态型19ZI000303澧县圆粒薏苡中国湖南C. lacryma-jobi var. lacryma-jobi野生长江中下游中熟生态型20ZI000304澧县长粒薏苡中国湖南C. lacryma-jobi var. lacryma-jobi野生长江中下游中熟生态型21ZI000305苡仁中国贵州C. chinensis var. chinesis Tod.栽培南方晚熟生态型22ZI000306六合薏苡中国贵州C. lacryma-jobi var. maxima Makino野生南方晚熟生态型23ZI000307本地六谷中国贵州C. chinensis var. chinesis Tod.栽培南方晚熟生态型24ZI000308本地六谷中国贵州C. lacryma-jobi var. Lacryma-jobi野生南方晚熟生态型25ZI000309本地六谷中国贵州C. lacryma-jobi var. maxima Makino栽培南方晚熟生态型26ZI000310六谷(薏9)中国贵州C. chinensis var. chinesis Tod.栽培南方晚熟生态型27ZI000311薏苡中国贵州C. chinensis var. chinesis Tod.栽培南方晚熟生态型28ZI000312野生薏苡中国贵州C. chinensis var. formosana(Ohwi)L.野生南方晚熟生态型29ZI000313野生薏苡中国贵州C. lacryma-jobi var. lacryma-jobi野生南方晚熟生态型30ZI000314野生薏苡中国贵州C. lacryma-jobi var. maxima Makino野生南方晚熟生态型31ZI000315野生薏苡中国贵州C. lacryma-jobi var. maxima Makino野生南方晚熟生态型32ZI000316薏苡中国贵州C. chinensis var. chinesis Tod.栽培南方晚熟生态型33ZI000317薏苡中国贵州C. chinensis var. chinesis Tod.栽培南方晚熟生态型34ZI000318陆谷中国贵州C. chinensis var. chinesis Tod.栽培南方晚熟生态型35ZI000319细长陆谷中国贵州C. stenocarpa Balansa野生南方晚熟生态型36ZI000320薏仁中国贵州C. lacryma-jobi var. lacryma-jobi野生南方晚熟生态型37ZI000322罗平薏苡中国贵州C. chinensis var. chinesis Tod.栽培南方晚熟生态型38ZI000323六谷中国云南C. chinensis var. chinesis Tod.栽培南方晚熟生态型39ZI000324糯六谷中国云南C. chinensis var. chinesis Tod.栽培南方晚熟生态型40ZI000325六谷中国云南C. chinensis var. chinesis Tod.栽培南方晚熟生态型41ZI000326桥头六谷中国云南C. chinensis var. chinesis Tod.栽培南方晚熟生态型42ZI000327糯六谷中国云南C. chinensis var. chinesis Tod.栽培南方晚熟生态型43ZI000328六谷中国云南C. chinensis var. chinesis Tod.栽培南方晚熟生态型44ZI000329铁六谷中国云南C. lacryma-jobi var. lacryma-jobi野生南方晚熟生态型45ZI000330数珠谷中国云南C. lacryma-jobi var. lacryma-jobi野生南方晚熟生态型46ZI000331六谷中国云南C. lacryma-jobi var. lacryma-jobi野生南方晚熟生态型47ZI000332六谷中国云南C. chinensis var. chinesis Tod.栽培南方晚熟生态型48ZI000333饭六谷中国云南C. chinensis var. chinesis Tod.栽培南方晚熟生态型49ZI000334糯六谷中国云南C. chinensis var. chinesis Tod.栽培南方晚熟生态型50ZI000335糯六谷中国云南C. chinensis var. chinesis Tod.栽培南方晚熟生态型51ZI000336旧腮六谷中国云南C. chinensis var. chinesis Tod.栽培南方晚熟生态型52ZI000337花甲六谷中国云南C. chinensis var. chinesis Tod.栽培南方晚熟生态型53ZI000338花甲六谷中国云南C. chinensis var. chinesis Tod.栽培南方晚熟生态型54ZI000340黑糯六谷中国云南C. chinensis var. chinesis Tod.栽培南方晚熟生态型55ZI000343富薏1号中国云南C. chinensis var. chinesis Tod.栽培南方晚熟生态型
表1(续)
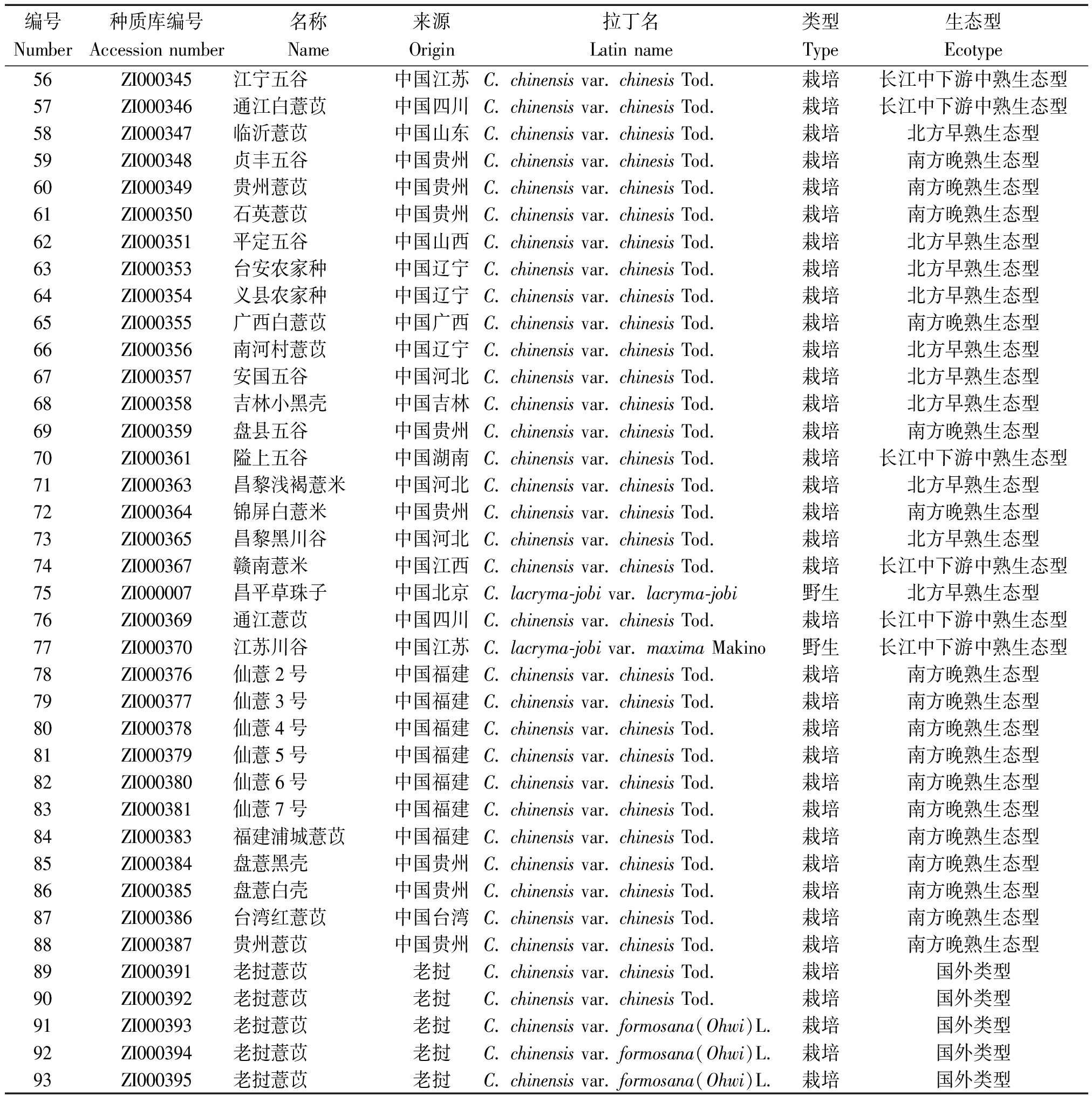
编号Number种质库编号Accession number名称Name来源Origin拉丁名Latin name类型Type生态型 Ecotype56ZI000345江宁五谷中国江苏C. chinensis var. chinesis Tod.栽培长江中下游中熟生态型57ZI000346通江白薏苡中国四川C. chinensis var. chinesis Tod.栽培长江中下游中熟生态型58ZI000347临沂薏苡中国山东C. chinensis var. chinesis Tod.栽培北方早熟生态型59ZI000348贞丰五谷中国贵州C. chinensis var. chinesis Tod.栽培南方晚熟生态型60ZI000349贵州薏苡中国贵州C. chinensis var. chinesis Tod.栽培南方晚熟生态型61ZI000350石英薏苡中国贵州C. chinensis var. chinesis Tod.栽培南方晚熟生态型62ZI000351平定五谷中国山西C. chinensis var. chinesis Tod.栽培北方早熟生态型63ZI000353台安农家种中国辽宁C. chinensis var. chinesis Tod.栽培北方早熟生态型64ZI000354义县农家种中国辽宁C. chinensis var. chinesis Tod.栽培北方早熟生态型65ZI000355广西白薏苡中国广西C. chinensis var. chinesis Tod.栽培南方晚熟生态型66ZI000356南河村薏苡中国辽宁C. chinensis var. chinesis Tod.栽培北方早熟生态型67ZI000357安国五谷中国河北C. chinensis var. chinesis Tod.栽培北方早熟生态型68ZI000358吉林小黑壳中国吉林C. chinensis var. chinesis Tod.栽培北方早熟生态型69ZI000359盘县五谷中国贵州C. chinensis var. chinesis Tod.栽培南方晚熟生态型70ZI000361隘上五谷中国湖南C. chinensis var. chinesis Tod.栽培长江中下游中熟生态型71ZI000363昌黎浅褐薏米中国河北C. chinensis var. chinesis Tod.栽培北方早熟生态型72ZI000364锦屏白薏米中国贵州C. chinensis var. chinesis Tod.栽培南方晚熟生态型73ZI000365昌黎黑川谷中国河北C. chinensis var. chinesis Tod.栽培北方早熟生态型74ZI000367赣南薏米中国江西C. chinensis var. chinesis Tod.栽培长江中下游中熟生态型75ZI000007昌平草珠子中国北京C. lacryma-jobi var. lacryma-jobi野生北方早熟生态型76ZI000369通江薏苡中国四川C. chinensis var. chinesis Tod.栽培长江中下游中熟生态型77ZI000370江苏川谷中国江苏C. lacryma-jobi var. maxima Makino野生长江中下游中熟生态型78ZI000376仙薏2号中国福建C. chinensis var. chinesis Tod.栽培南方晚熟生态型79ZI000377仙薏3号中国福建C. chinensis var. chinesis Tod.栽培南方晚熟生态型80ZI000378仙薏4号中国福建C. chinensis var. chinesis Tod.栽培南方晚熟生态型81ZI000379仙薏5号中国福建C. chinensis var. chinesis Tod.栽培南方晚熟生态型82ZI000380仙薏6号中国福建C. chinensis var. chinesis Tod.栽培南方晚熟生态型83ZI000381仙薏7号中国福建C. chinensis var. chinesis Tod.栽培南方晚熟生态型84ZI000383福建浦城薏苡中国福建C. chinensis var. chinesis Tod.栽培南方晚熟生态型85ZI000384盘薏黑壳中国贵州C. chinensis var. chinesis Tod.栽培南方晚熟生态型86ZI000385盘薏白壳中国贵州C. chinensis var. chinesis Tod.栽培南方晚熟生态型87ZI000386台湾红薏苡中国台湾C. chinensis var. chinesis Tod.栽培南方晚熟生态型88ZI000387贵州薏苡中国贵州C. chinensis var. chinesis Tod.栽培南方晚熟生态型89ZI000391老挝薏苡老挝C. chinensis var. chinesis Tod.栽培国外类型90ZI000392老挝薏苡老挝C. chinensis var. chinesis Tod.栽培国外类型91ZI000393老挝薏苡老挝C. chinensis var. formosana(Ohwi)L.栽培国外类型92ZI000394老挝薏苡老挝C. chinensis var. formosana(Ohwi)L.栽培国外类型93ZI000395老挝薏苡老挝C. chinensis var. formosana(Ohwi)L.栽培国外类型
群体Nei′s遗传距离计算公式:DS=-LN[Jxy/(JxJy)1/2],其中,![]() 表示从X群体中随机选出2个基因相同的平均数;
表示从X群体中随机选出2个基因相同的平均数;![]() 表示从Y群体中随机选出2个基因相同的平均数;
表示从Y群体中随机选出2个基因相同的平均数;![]() 表示从X、Y 2个群体中随机选出2个基因相同的平均数。
表示从X、Y 2个群体中随机选出2个基因相同的平均数。
群体遗传相似系数计算公式:![]() 其中Nij为单倍型i、j共有的等位基因数量,Ni、Nj分别是单倍型i、j的等位基因数。
其中Nij为单倍型i、j共有的等位基因数量,Ni、Nj分别是单倍型i、j的等位基因数。
2 结果与分析
2.1 叶绿体基因组SSR标记位点多样性
利用38对SSR引物对93份薏苡材料进行扩增,有33对引物在供试材料中表现出多态性,占所有扩增引物的86.84%(表2、图1)。33对SSR引物在所有的材料中共检测出153个等位变异,每个位点的等位基因数目为3~8个,平均4.64个;有效等位基因数为1.073 7~4.819 7个,平均1.777 7个;Nei′s多样性指数(H)和Shannon′s信息指数(I)的变异范围分别为0.068 7~0.792 6和0.175 2~1.816 3,平均为0.368 8和0.720 2,两者参数值变化趋于一致,也说明供试材料之间存在丰富的多样性。
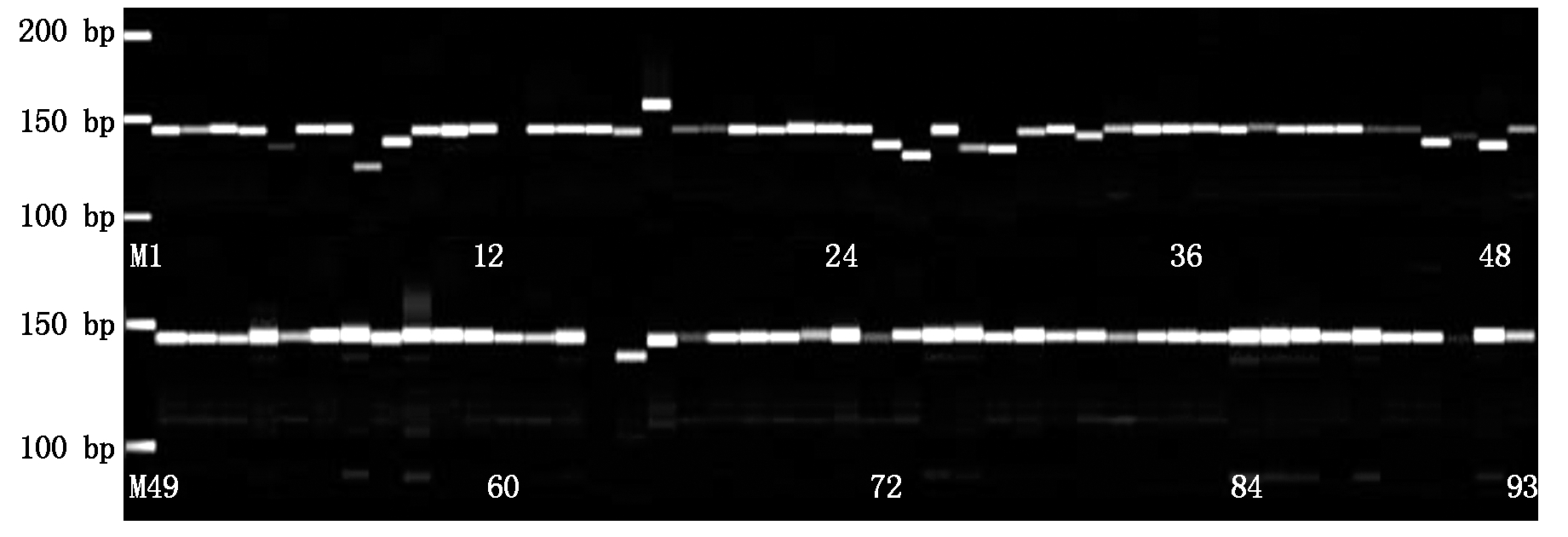
图1 引物cp1的扩增电泳图
Fig.1 Image of electrophoresis amplified by cp1 primer
表2 33对SSR引物的遗传参数
Tab.2 Genetic parameters from the 33 polymorphic SSR markers
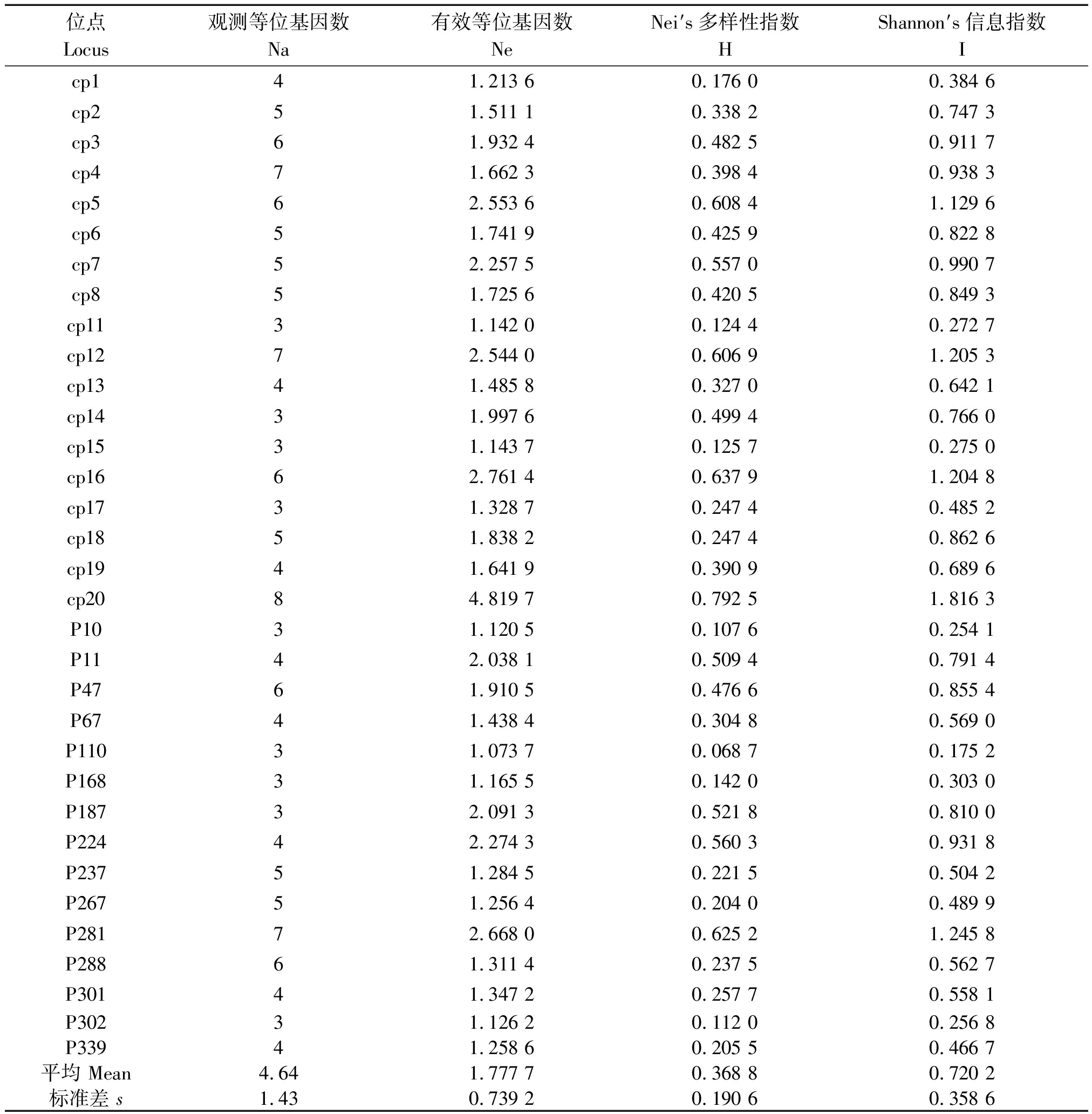
位点Locus观测等位基因数Na有效等位基因数NeNei′s多样性指数HShannon′s信息指数Icp141.213 60.176 00.384 6cp251.511 10.338 20.747 3cp361.932 40.482 50.911 7cp471.662 30.398 40.938 3cp562.553 60.608 41.129 6cp651.741 90.425 90.822 8cp752.257 50.557 00.990 7cp851.725 60.420 50.849 3cp1131.142 00.124 40.272 7cp1272.544 00.606 91.205 3cp1341.485 80.327 00.642 1cp1431.997 60.499 40.766 0cp1531.143 70.125 70.275 0cp1662.761 40.637 91.204 8cp1731.328 70.247 40.485 2cp1851.838 20.247 40.862 6cp1941.641 90.390 90.689 6cp2084.819 70.792 51.816 3P1031.120 50.107 60.254 1P1142.038 10.509 40.791 4P4761.910 50.476 60.855 4P6741.438 40.304 80.569 0P11031.073 70.068 70.175 2P16831.165 50.142 00.303 0P18732.091 30.521 80.810 0P22442.274 30.560 30.931 8P23751.284 50.221 50.504 2P26751.256 40.204 00.489 9P28172.668 00.625 21.245 8P28861.311 40.237 50.562 7P30141.347 20.257 70.558 1P30231.126 20.112 00.256 8P33941.258 60.205 50.466 7平均 Mean4.641.777 70.368 80.720 2标准差 s1.430.739 20.190 60.358 6
注:Na.观测等位基因数;Ne.有效等位基因数;H. Nei′s多样性指数;I. Shannon′s信息指数。表2同。
Note:Na.Observed number of alleles; Ne.Effective number of alleles; H.Nei′s gene diversity index; I.Shannon′s information index. The same as Tab.2.
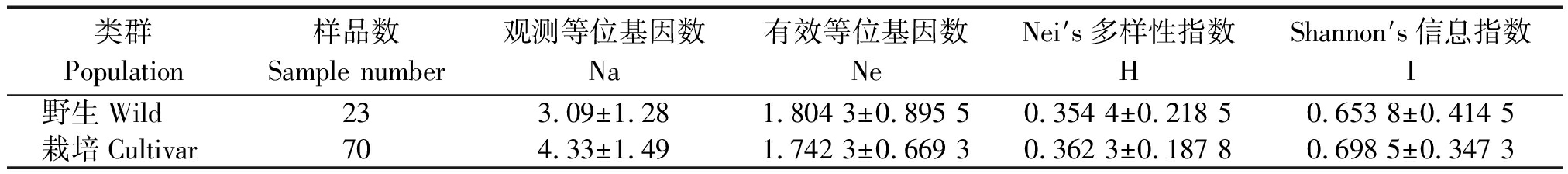
2.2 薏苡野生、栽培类群间的遗传多样性
根据野生、栽培薏苡群体的划分,计算不同类群的遗传多样性参数。结果显示,除了野生类群有效等位基因数高于栽培类群,野生类群的观测等位基因数、Nei′s多样性指数和Shannon′s信息指数均略低于栽培类群(表3);2个类群的遗传距离和一致度(相似系数)分别为0.036 2和0.964 4,说明野生和栽培类群的变异主要在类群内部,而2个类群之间的变异较小,野生和栽培类群间没有表现出明显的遗传分化。
表3 薏苡属野生和栽培类群的遗传多样性
Tab.3 Genetic diversity of wild and cultivar populations in Coix L.
类群Population样品数Sample number观测等位基因数Na有效等位基因数NeNei′s多样性指数HShannon′s信息指数I野生Wild233.09±1.281.804 3±0.895 50.354 4±0.218 50.653 8±0.414 5栽培Cultivar704.33±1.491.742 3±0.669 30.362 3±0.187 80.698 5±0.347 3
2.3 薏苡不同生态类型种质间的遗传多样性
根据薏苡种质资源的地理来源,将全部材料划分为4个生态型类群。由表4可知,4个生态型类群的平均观测等位基因数为1.45~4.33个,有效等位基因数为1.325 1~1.861 1。Nei′s多样性指数和Shannon′s信息指数显示,北方早熟生态型和南方晚熟生态型2个类群内遗传多样性较高,长江中下游中熟生态型类群次之,国外类型最低。遗传距离和一致度(表5)分析表明,4个生态型类群的遗传距离变化为0.028 1~0.224 6,一致度变化为0.798 8~0.972 2,北方早熟生态型、长江中下游晚熟和南方晚熟生态型类群之间的遗传距离相对较近、一致度高,但是与国外类型的遗传差异明显。
表4 薏苡属不同生态型类群的遗传多样性
Tab.4 Genetic diversity of ecotype populations in Coix L.
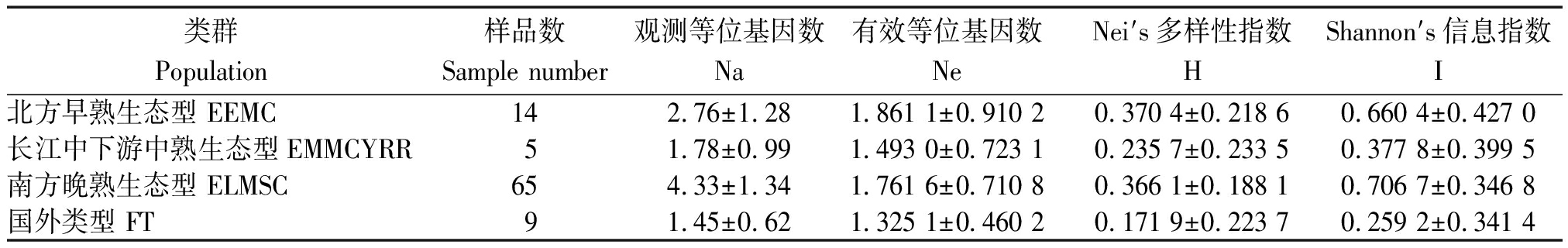
类群Population样品数Sample number观测等位基因数Na有效等位基因数NeNei′s多样性指数HShannon′s信息指数I北方早熟生态型 EEMC142.76±1.281.861 1±0.910 20.370 4±0.218 60.660 4±0.427 0长江中下游中熟生态型EMMCYRR 51.78±0.991.493 0±0.723 10.235 7±0.233 50.377 8±0.399 5南方晚熟生态型 ELMSC654.33±1.341.761 6±0.710 80.366 1±0.188 10.706 7±0.346 8国外类型 FT 91.45±0.621.325 1±0.460 20.171 9±0.223 70.259 2±0.341 4
表5 不同生态型类群的遗传距离和一致度
Tab.5 Genetic distance and Nei′s genetic identity in different ecotype populations of Coix L.

类群Population北方早熟生态型EEMC长江中下游中熟生态型EMMCYRR南方晚熟生态型ELMSC国外类型FT北方早熟生态型 EEMC10.958 50.951 00.798 8长江中下游中熟生态型EMMCYRR0.042 410.972 20.877 8南方晚熟生态型 ELMSC0.050 20.028 110.887 4国外类型 FT0.224 60.130 30.119 51
注:上三角为遗传一致度矩阵,下三角为遗传距离矩阵。
Note: Nei′s genetic identity(above diagonal) and genetic distance(below diagonal).
2.4 薏苡属不同种(变种)的遗传分化
从植物学分类角度将全部资源划分为6个种或变种,计算不同种(变种)间的相似系数和聚类分析。在相似系数0.85时,小珠薏苡种(C. puellarum Balansa)和窄果薏苡种(C. stenocarpa Balansa)分别为单独的一分支,在进化上也更为原始;台湾薏苡变种(C. chinensis var. formosana(Ohwi)L. Liu)、念珠薏苡变种(C. lacryma-jobi var. maxima Makino)、薏苡变种(C. lacryma-jobi var. lacryma-jobi)和薏米变种(C. chinensis var. chinesis Tod.)类群则聚在一起(图2)。聚类分析表明,台湾薏苡变种、念珠薏苡变种、薏苡变种和薏米变种4个类群之间的遗传差异相对较小;小珠薏苡种和窄果薏苡种则遗传分化明显,暗示了这2个薏苡种可能具有更早的物种起源,也为薏苡的植物学分类提供了重要分子证据。
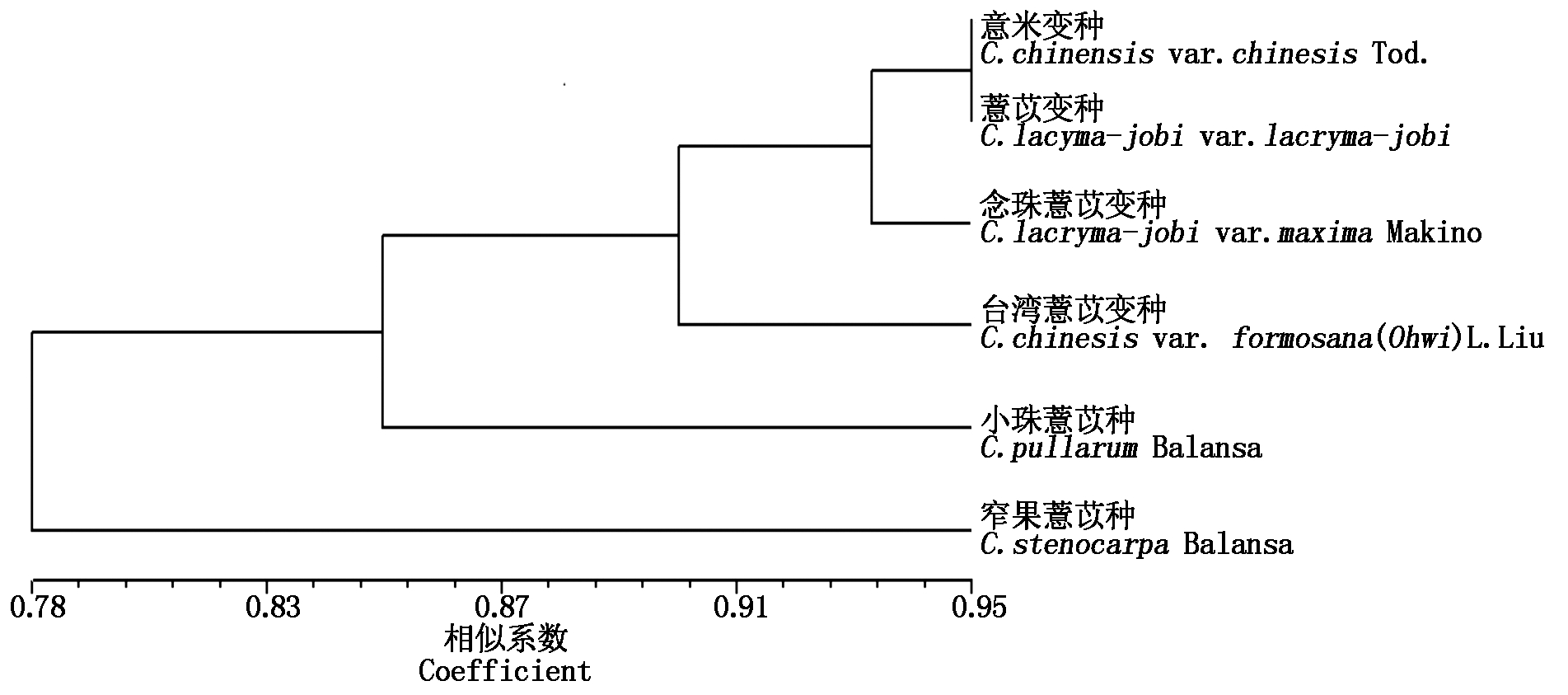
图2 薏苡属内不同种(变种)的聚类
Fig.2 Clustering of different species(varieties) in Coix L.
2.5 薏苡属种质资源的亲缘关系
将每个省份或地区的资源作为一个群体,并基于UPGMA法绘制聚类图(图3)。在遗传距离为0.74处,来自中国浙江、云南、贵州、福建、河北、江苏、湖南、四川、辽宁、江西及老挝种质聚为一类,山西、台湾、吉林、北京、山东和广西的种质则分别为单独的分支,揭示出不同薏苡属种质间的亲缘关系与地理分布距离不存在绝对的相关性,可能人类活动和比较频繁的基因渐渗有关。
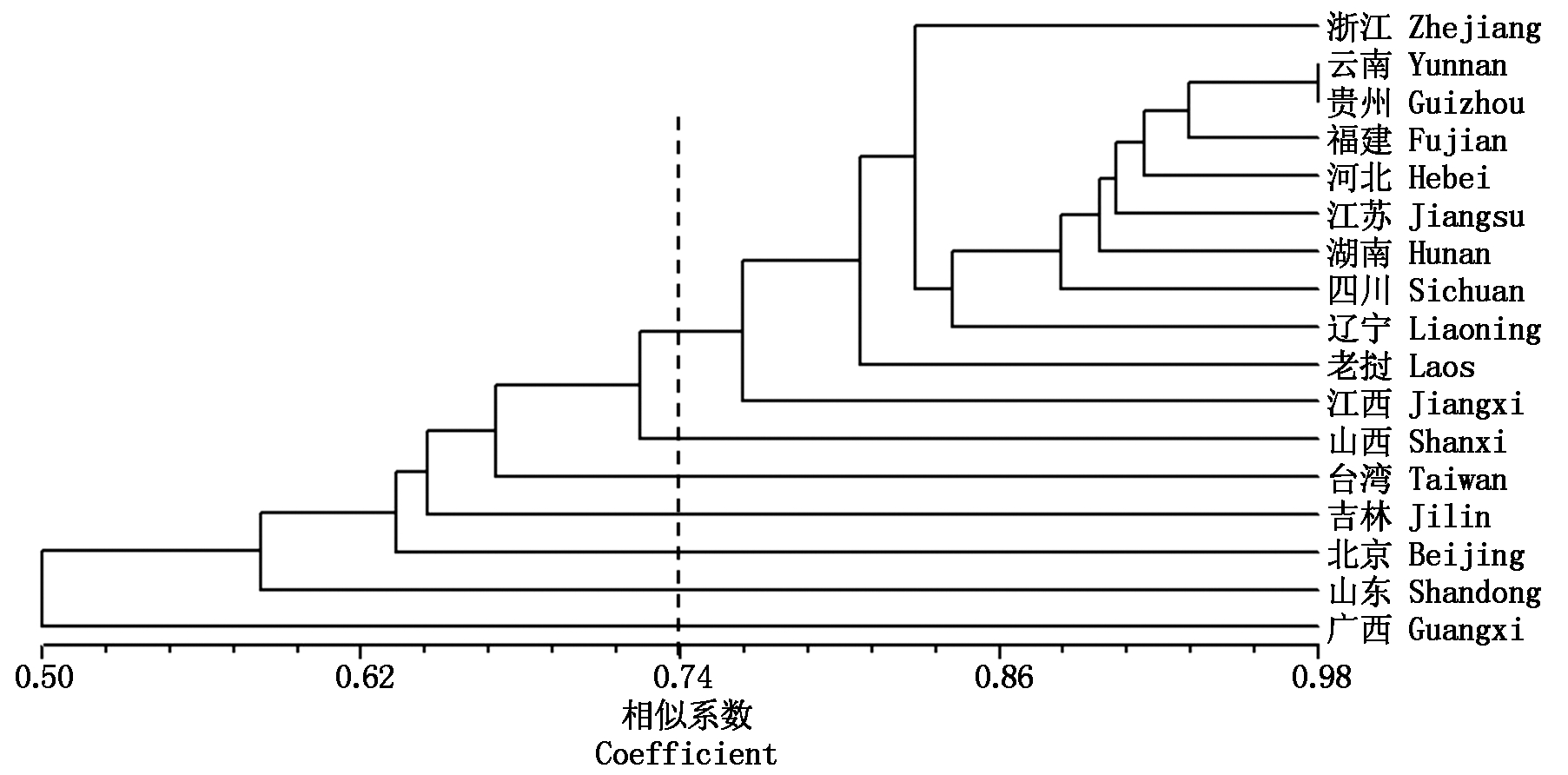
图3 薏苡属不同地域类群的UPGMA聚类
Fig.3 Clustering of regional populations in Coix L. by UPGMA method
3 讨论
3.1 叶绿体SSR标记在遗传多样性研究中的应用
叶绿体DNA单性遗传,有独立的进化路线,不依赖于其他任何数据即可构建分子系统树,可从历史和系统发育的角度解释生物多样性,并提供可靠和准确的信息,被认为是研究系统发生和群体遗传学的理想方法。因此,以叶绿体基因组信息开发的SSR标记不仅具备共显性和单倍型的特点,还继承了叶绿体基因组的遗传进化特征。迄今为止,基于叶绿体基因组的SSR标记开发[22-24]、遗传多样性分析[25-27]、物种分类和演化[28-29]等方面基础和应用研究也是方兴未艾。Chen等[30]利用22对cpSSR引物分析了苎麻属下7个种的亲缘关系。Wang等[31]则成功筛选出11对cpSSR和4对mtSSR标记对异源胞质的甘蓝不育系进行区分,并作为鉴定异源胞质材料的有效工具。本研究从38对薏苡cpSSR引物中筛选出33对有效引物,在93份薏苡属种质资源中扩增出153个等位变异,每个位点的平均等位基因数目4.64个,Nei′s多样性指数(H)和Shannon′s多样性指数(I)平均为0.368 8和0.720 2,在供试材料中具有较高多态性,也从另一层面揭示出了薏苡属种质资源丰富的遗传变异。虽然与核基因组SSR标记相比,cpSSR在进化上更为保守、多态性低,但是通过不同标记组合的单倍型在群体遗传分化、物种鉴定中却独具优势。本研究通过cpSSR标记对薏苡属的6个种(变种)类群的遗传分化分析,初步确立了不同种(变种)的亲缘关系,也为薏苡属的植物学分类提供了重要证据。此外,cpSSR标记进行商用品种的指纹鉴定时,由于标记多态性偏低,品种鉴别力有限,但在标记数量丰富、品种数量有限的前提下,依然是简单而有效的选择。
3.2 薏苡属种质资源的群体结构和地域分布关系探讨
由于叶绿体DNA具有单亲遗传的特点,与核基因组标记相比,鲜有基因重组发生,因此广泛用于群体结构和地理系谱学[32-33]分析。Xu等[34]用cpSSR标记分析了326份野生和栽培类型大豆资源遗传结构,并为栽培大豆的多起源学说提供了分子证据。魏潇等[35]通过对58份普通杏资源进行cpSSR的单倍型分析阐明了不同区域普通杏的演化关系。本研究利用cpSSR标记对薏苡属的野生、栽培及不同生态型类群进行遗传分化分析,揭示出野生和栽培类群的遗传分化主要在种群内部,种群间遗传分化不明显;国外类群(FT)与国内类群遗传距离相对较远,中国的3个生态型之间也没有明显的遗传分化。聚类分析也表明,薏苡属种质间的亲缘关系与地理分布距离不存在绝对的相关性,这与前人的研究结果基本一致[16]。令人出乎意料的是,野生类群与栽培类群的分化并不明显,可能与人类活动和基因渐渗有关,导致较多的过渡型种质产生。已有报道表明,薏苡属作物除了水生薏苡(种)以根茎为载体进行营养繁殖外,其余的种或变种之间不存在生殖隔离[36]。值得一提的是,如何有效开发分子标记并建立相应的高效检测体系将是今后薏苡属分子育种、物种鉴定、品种指纹鉴定和系统进化研究的关键。截至目前,本课题组已完成薏苡属多个种(变种)的叶绿体和线粒体基因组测序和组装工作,今后将通过叶绿体基因组的比较基因组学分析,重构薏苡属的系统进化关系,开发、整合和建立不同分子标记(SSR、SNP、Indel)检测体系,这对于理解薏苡属在禾本科植物中的系统发育地位和物种鉴定、分类等方面均具有重要指导意义。
[1] 黄亨履, 陆平, 朱玉兴, 李英材. 中国薏苡的生态型、多样性及利用价值[J]. 作物品种资源, 1995(4): 4-8. doi:10.19462/j.cnki.1671-895x.1995.04.002.
Huang H L, Lu P, Zhu Y X, Li Y C. Ecotype diversity and utility value in Chinese Job′s Tears[J].Journal of Crop Variety Resources, 1995(4): 4-8.
[2] 梁云涛, 陈成斌, 梁世春, 赖群珍, 黄娟, 徐志健. 中日韩三国薏苡种质资源遗传多样性研究[J]. 广西农业科学, 2006, 37(4): 341-344. doi:10.3969/j.issn.2095-1191.2006.04.001.
Liang Y T, Chen C B, Liang S C, Lai Q Z, Huang J, Xu Z J. Research on genetic diversity of Job′s Tears(Coix lacryma-jobi L.) germplasm from China Japan and Korea[J]. Guangxi Agricultural Sciences, 2006, 37(4): 341-344.
[3] 王硕, 张世鲍, 何金宝, 陆光著, 杨志清. 薏苡资源性状的主成分和聚类分析[J]. 云南农业大学学报, 2013, 28(2): 157-162. doi:10.3969/j.issn.1004-390X(n).2013.02.002.
Wang S, Zhang S B, He J B, Lu G Z, Yang Z Q. The principal component analysis and cluster analysis of Coix resource characteristics[J]. Journal of Yunnan Agricultural University, 2013, 28(2): 157-162.
[4] 李春花, 王艳青, 卢文洁, 王莉花. 云南薏苡种质资源农艺性状的主成分和聚类分析[J]. 植物遗传资源学报, 2015, 16(2): 277-281. doi:10.13430/j.cnki.jpgr.2015.02.010.
Li C H, Wang Y Q, Lu W J, Wang L H. The principal component and cluster analysis of agronomic traits of Coix germplasm resources in Yunnan[J]. Journal of Plant Genetic Resources, 2015, 16(2): 277-281.
[5] 金关荣, 奚秀洁, 程舟, 陈常理, 洛霞虹, 李珊. 薏苡种子形态性状多样性评价[J]. 植物遗传资源学报, 2017, 18(3): 421-428. doi:10.13430/j.cnki.jpgr.2017.03.006.
Jin G R, Xi X J, Cheng Z, Chen C L, Luo X H, Li S. Evaluation of seed morphological characteristics diversity of Job′s Tears( Coix lacryma-jobi) germplasm[J]. Journal of Plant Genetic Resources, 2017, 18(3): 421-428.
[6] 石明, 李祥栋, 秦礼康. 薏苡种质资源描述规范和数据标准[M]. 北京: 中国农业出版社, 2017.
Shi M, Li X D, Qin L K. Descriptors and data standard for Job′s Tears(Coix lacryma-jobi L.) [M]. Beijing: China Agriculture Press, 2017.
[7] 李祥栋, 潘虹, 陆秀娟, 魏心元, 陆平, 石明, 练启仙. 薏苡属种质资源的主要表型性状多样性研究[J]. 植物遗传资源学报, 2019, 20(1): 229-238. doi:10.13430/j.cnki.jpgr.20180604003.
Li X D, Pan H, Lu X J, Wei X Y, Lu P, Shi M, Lian Q X. Analysis of main phenotypic characteristics in Coix L. germplasm resources[J]. Journal of Plant Genetic Resources, 2019, 20(1): 229-238.
[8] 张立娜, 曹桂兰, 韩龙植. 利用SSR标记揭示中国粳稻地方品种遗传多样性[J]. 中国农业科学, 2012, 45(3): 405-413. doi:10.3864/j.issn.0578-1752.2012.03.001.
Zhang L N, Cao G L, Han L Z. Analysis of genetic diversity of Japonica rice landrace in China with microsatellite marker[J]. Scientia Agricultura Sinica, 2012, 45(3):405-413.
[9] 连帅, 陆平, 乔治军, 张琦, 张茜, 刘敏轩, 王瑞云. 利用SSR分子标记研究国内外黍稷地方品种和野生资源的遗传多样性[J].中国农业科学, 2016, 49(17): 3264-3275. doi:10.3864/j.issn.0578-1752.2016.17.002.
Lian S, Lu P, Qiao Z J, Zhang Q, Zhang Q, Liu M X, Wang R Y. Genetic diversity in Broomcorn Millet(Panicum miliaceum L.)from China and abroad by using SSR markers[J]. Scientia Agricultura Sinica, 2016, 49(17): 3264-3275.
[10] 陆徐忠, 倪金龙, 李莉, 汪秀峰, 马卉, 张小娟, 杨剑波. 利用SSR 分子指纹和商品信息构建水稻品种身份证[J]. 作物学报, 2014, 40(5): 823-829. doi:10.3724/SP.J.1006.2014.00823.
Lu X Z, Ni J L, Li L, Wang X F, Ma H, Zhang X J, Yang J B. Construction of rice variety indentity using SSR fingerprint and commodity information[J]. ACTA Agronomica Sinica, 2014, 40(5): 823-829.
[11] 王瑞, 张福耀, 程庆军, 田承华, 凌亮. 利用SSR荧光标记构建20个高粱品种指纹图谱[J]. 作物学报, 2015, 41(4): 658-665. doi:10.3724/SP.J.1006.2015.00658.
Wang R, Zhang F Y, Cheng Q J, Tian C H, Ling L. Establishment of 20 sorghum hybrids fingerprints using SSR fluorescent marker[J]. ACTA Agronomica Sinica, 2015, 41(4): 658-665.
[12] 郭建斌, 黄莉, 成良强, 陈伟刚, 任小平, 陈玉宁, 周小静, 沈金雄, 姜慧芳. 利用3个F2群体整合高密度栽培种花生遗传连锁图[J]. 作物学报, 2016, 42(2): 159-169. doi:10.3724/SP.J.1006.2016.00159.
Guo J B, Huang L, Cheng L Q, Chen W G, Ren X P, Chen Y N, Zhou X J, Shen J X, Jiang H F. An integrated genetic linkage map from three F2 populations of cultivated peanut(Arachis hypogaea L.)[J]. ACTA Agronomica Sinica, 2016, 42(2): 159-169.
[13] Ma K H, Kim K H, Dixit A, Yu J W, Chung J W, Lee J H, Cho E G, Kim T S, Park Y G. Newly developed polymorphic microsatellite markers in Job′s tears(Coix lacryma-jobi L.)[J]. Molecular Ecology Notes, 2006, 6(3): 689-691. doi:10.1111/j.1471-8286.2006.01311.x.
[14] Ma K H, Kim K H, Dixit A, Chung I M, Gwag J G, Kim T S, Park Y G. Assessment of genetic diversity and relationships among Coix lacryma-jobi accessions using microsatellite markers[J]. Biologia Plantarum, 2006, 54(2): 272-278. doi:10.1007/s10535-010-0047-6.
[15] 郭银萍, 彭忠华, 赵致, 陈志兴, 高翔, 刘胜传, 谢云. 基于SSR标记的贵州薏苡种质资源遗传多样性分析[J].植物遗传资源学报, 2012, 13(2): 317-320. doi:10.3969/j.issn.1672-1810.2012.02.027.
Guo Y P, Peng Z H, Zhao Z, Chen Z X, Gao X, Liu S C, Xie Y. Genetic diversity analysis of Jobs Tears germplasm resources by SSR markers in Guizhou Province[J]. Journal of Plant Genetic Resources, 2012, 13(2): 317-320.
[16] Liu H B, Shi J P, Cai Z X, Huang Y M, Lü M L, Du H L, Gao Q, Zuo Y, Dong Z B, Huang W, Qin R, Liang C Z, Lai J S, Jin W W. Evolution and domestication footprints uncovered from the genomes of coix[J]. Molecular Plant,2019.doi: 10.1016/j.molp.2019.11.009.
[17] Guo C, Wang Y N, Yang A G, He J, Xiao C W, Lü S H, Han F M, Yuan Y B, Yuan Y, Dong X L, Guo J, Yang Y W, Liu H L, Zuo N Z, Hu Y X, Zhao K X, Jiang Z B, Wang X, Jiang T T, Shen Y O, Cao M J, Wang Y, Long Z B, Rong T Z, Huang L Q, Zhou S F. The Coix genome provides insights into Panicoideae evolution and papery hull domestication[J]. Molecular Plant,2019.doi: 10.1016/j.molp.2019.11.008.
[18] Leseberg C H, Duvall M R. The complete chloroplast genome of Coix lacryma-jobi and a comparative molecular evolutionary analysis of plastomes in cereals[J]. Journal of Molecular Evolution, 2009, 69(4): 311-318. doi:10.1007/s00239-009-9275-9.
[19] 李祥栋, 潘虹, 陆秀娟, 魏心元, 陆平, 石明, 秦礼康. 薏苡叶绿体基因组的SSR位点分析及种质亲缘关系鉴定 [J]. 贵州农业科学, 2018, 46(8): 1-5. doi:10.3969/j.issn.1001-3601.2018.08.002.
Li X D, Pan H, Lu X J, Wei X Y, Lu P, Shi M, Qin L K. SSR locus analysis of chloroplast genome and identification of genetic relationships in 14 semen adlay germplasm resoures[J]. Guizhou Agricultural Sciences,2018, 46(8): 1-5.
[20] Yeh F, Yang R C, Boyle T. Popgene: Microsoft window-based freeware for population genetic analysis, version1.31[M]. Edmonton: University of Alberta, 1999.
[21] Rohlf F J. NTSYS-pc Numerical taxonomy and multivariate analysis system(Version 2.1)[M]. New York: User Guide. Exeter Software Setauket, 2000.
[22] 杨丽, 赵韩生, 彭镇华, 董丽莉, 高志民. 竹子叶绿体基因组SSR分子标记的开发及其应用[J]. 热带亚热带植物学报, 2014, 22(3): 263-269. doi:10.3969/j.issn.1005-3395.2014.03.008.
Yang L, Zhao H S, Peng Z H, Dong L L, Gao Z M. Development and application of SSR molecular markers from the chloroplast genome of bamboo[J]. Journal of Tropical and Subtropical Botany, 2014, 22(3): 263-269.
[23] Beyer M, Nazareno A G, Lohmann L G. Using genomic data to develop chloroplast DNA SSRs for the Neotropical liana Stizophyllum riparium(Bignonieae, Bignoniaceae)[J]. Applications in Plant Sciences, 2017, 5(10): 1700061. doi:10.3732/apps.1700061.
[24] 朱国忠, 戴伟民, 陈晓锋, 张晶旭, 强盛. 基于基因组数据库的杂草稻叶绿体分子标记开发[J]. 南京农业大学学报, 2015, 38(2): 240-247. doi:10.7685/j.issn.1000-2030.2015.02.010.
Zhu G Z, Dai W M, Chen X F, Zhang J X, Qiang S. Development of molecular markers in weedy rice chloroplast based on genomic database[J]. Journal of Nanjing Agricultural University, 2015, 38(2): 240-247.
[25] 程丽莉, 胡广隆, 苏淑钗, 黄武刚. 板栗及其近缘种叶绿SSR遗传多样性分析[J].华北农学报, 2015, 30(2): 145-149. doi:10.7668 /hbnxb.2015.02.026.
Cheng L L, Hu G L, Su S C, Huang W G. Diversity of cultivated chestnuts and it′s close relative species using chloroplast SSRs[J]. ACTA Agriculturae Boreali-Sinaca, 2015, 30(2): 145-149.
[26] 李航, 杨晓明, 丁德宽, 王小丽, 谢宗周, 叶俊丽, 彭抒昂, 邓秀新, 柴利军. 基于cpSSR和nSSR标记的地方柑橘资源‘枳雀’亲本分析[J]. 果树学报, 2018, 359(10): 1161-1169. doi:10.13925/j.cnki.gsxb.20180001.
Li H, Yang X M, Ding D K, Wang X L, Xie Z Z, Ye J L, Peng S A, Deng X X, Chai L J. Parentage analysis of indigenous citrus ′ZhiQue′ based on nuclear and chloroplast SSR markers[J]. Journal of Fruit Science, 2018, 35(10): 1161-1169.
[27] 曾建敏, 陈学军, 吴兴富, 焦芳婵, 肖炳光, 李永平, 童治军. 基于烟草叶绿体基因组和线粒体基因组SSR标记的烟属植物遗传多样性分析[J]. 中国烟草学报, 2016, 22(4): 89-97. doi:10.16472/j.chinatobacco.2016.130.
Zeng J M, Chen X J, Wu X F, Jiao F C, Xiao B G, Li Y P. Genetic diversity analysis of genus Nicotiana based on SSR markers in chloroplast genome and mitochondria genome[J]. ACTA Tabacaria Sinica, 2016, 22(4): 89-97.
[28] 杜久军, 左力辉, 刘易超, 于晓跃, 董研, 王进茂, 杨敏生. 裂叶榆叶绿体基因组及CP-SSR位点分析[J]. 植物遗传资源学报, 2018, 19(6): 1187-1196. doi:10.13430/j.cnki.jpgr.20180425001.
Du J J, Zuo L H, Liu Y C, Yu X Y, Dong Y, Wang J M, Yang M S. Chloroplast genome and CP-SSR Site analyses of Ulmus laciniata[J]. Journal of Plant Genetic Resources, 2018, 19(6): 1187-1196.
[29] Li J, Ge X J, Cao H L, Ye W H. Chloroplast DNA diversity in Castanopsis hystrix populations in South China[J]. Forest Ecology and Management, 2017, 243(1): 94-101. doi:10.1016/j.foreco.2007.02.012.
[30] Chen P,Yu C M,Wang Y Z, Chen J K,Lu L X,Xiong H P. Analysis of genetic relationship among Boehmeria varieties using cpSSR markers[J]. Agricultural Biotechnology 2015, 4(3): 24-26. doi:10.19759/j.cnki.2164-4993.2015.03.006.
[31] Wang Q B, Zhang Y Y, Fang Z Y, Liu Y M, Yang L M, Zhuang M. Chloroplast and mitochondrial SSR help to distinguish allo-cytoplasmic male sterile types in cabbage(Brassica oleracea L.var.capitata)[J]. Molocular Breeding, 2012, 30(2):709-716. doi:10.1007/s11032-011-9656-9.
[32] Butorac L, Hancˇevic'K, LuksÏic'K,  kvorc Ž, Leko M, Maul E, Goran Zdunic'. Assessment of wild grapevine(Vitis vinifera ssp. sylvestris) chlorotypes and accompanying woody species in the Eastern Adriatic region[J]. PLoS One, 2018, 13(6): e0199495. doi:10.1371/journal.pone.0199495.
kvorc Ž, Leko M, Maul E, Goran Zdunic'. Assessment of wild grapevine(Vitis vinifera ssp. sylvestris) chlorotypes and accompanying woody species in the Eastern Adriatic region[J]. PLoS One, 2018, 13(6): e0199495. doi:10.1371/journal.pone.0199495.
[33] 张靖国, 曹玉芬, 陈启亮, 杨晓平, 范净, 田瑞, 胡红菊. 基于叶绿体DNA变异的湖北梨属种质系统进化及遗传多样性分析[J]. 植物遗传资源学报, 2016, 17(4): 766-772. doi:10.13430/j.cnki.jpgr.2016.04.023.
Zhang J G, Cao Y F, Chen Q L, Yang X P, Fan J, Tian R, Hu H J. Genetic diversty and phylogenetics of pear(Pyrus L.) germplasm resources from Hubei province revealed by chloroplast DNA variation[J]. Journal of Plant Genetic Resources, 2016, 17(4): 766-772.
[34] Xu D, Abe J, Gai J, Shimamoto Y. Diversity of chloroplast DNA SSRs in wild and cultivated soybeans: evidence for multiple origins of cultivated soybean[J]. Theoretical and Applied Genetics, 2002, 105(5): 645-653. doi:10.1007/s00122-002-0972-7.
[35] 魏潇, 章秋平, 刘威生, 刘宁, 张玉萍, 徐铭, 刘硕, 张玉君, 马小雪. 基于叶绿体SSR 单倍型分析普通杏演化关系[J].植物遗传资源学报, 2018, 19(4): 705-712. doi:10.13430/j.cnki.jpgr.20171103001.
Wei X, Zhang Q P, Liu W S, Liu N, Zhang Y P, Xu M. Liu S, Zhang Y J, Ma X X. Phylogenetic relationship analysis of common apricot(Prunus armeniaca L. ) revealed by chloroplast SSR haplotypes[J]. Journal of Plant Genetic Resources, 2018, 19(4): 705-712.
[36] 陆平, 左志明. 广西水生薏苡种的发现与鉴定[J].广西农业科学,1996(1): 18-20.
Lu P, Zuo Z M. Discovery and identification of C. aduatica Roxb. in Guangxi[J]. Journal of Guangxi Agricultural Sciences, 1996(1): 18-20.