羊口疮(Orf)又称羊传染性脓疱,是由羊口疮病毒(Orf virus, ORFV)引发的接触性人畜共患传染病, 目前常以区域性流行危害当地养羊业发展。临床上主要感染山羊和绵羊,表现为口唇、舌、鼻、乳房等部位形成丘疹、水疱、脓疱和疣状结痂[1-3]。本病传染性强、流行迅速呈群发性,主要侵害3~6月龄的羔羊,发病率可高达100%,造成羔羊生长发育迟缓[4-5]。该病随着养羊业的发展广泛分布于世界各地,在我国以西部地区流行较广,是家畜疫病防控中的重要疫病之一,对全球养羊业造成重大经济损失[6-7]。
ORFV属于痘病毒科(Poxviridae)脊索动物痘病毒亚科(Chordopoxvirinae)副痘病毒属(Parapoxvirus),为有囊膜的线性双股DNA病毒,基因组大小将近150 kp, 编码130多个基因。中间的核心区域(ORF009~ORF111),该区域基因在病毒的复制、组装和释放过程中起着重要作用;两端为含有闭合发卡结构的反向末端重复序列(ORF001~ORF008、ORF112~ORF134),多与病毒的致病机理有关[8-11]。121基因位于ORFV基因组的3′反向末端重复序列区,相关研究表明该基因编码一种NF-κB信号通路抑制剂,在胞质中可直接与NF-κB-p65结合并抑制其磷酸化和核转运进程,此外该基因的缺失致使ORFV的毒力显著降低[12]。目前,国内对ORFV 121蛋白的研究不多,也未见对其进行表达的详细报道。本试验为进一步研究该蛋白的生物学功能提供了依据。
1 材料和方法
1.1 主要材料与试剂
ORFV AH-F10毒株由兽医病理生物学与疫病防控安徽省重点实验室保存[13]。pMD-19T载体、Solution Ⅰ、rTaq DNA聚合酶及限制性内切酶BamH Ⅰ和EcoR Ⅰ购自大连宝生物(TaKaRa)有限公司;大肠埃希菌DH5α和Rosetta感受态细胞购自天根生化科技(北京)有限公司;琼脂糖凝胶回收试剂盒、小量提取质粒试剂盒购自AXYGEN公司;SDS-PAGE凝胶快速配制试剂盒、DAB试剂盒购自碧云天生物技术公司;10×PBS缓冲液购自北京索莱宝科技有限公司;抗GST标签鼠源单抗、羊抗鼠IgG/HRP购自北京中杉金桥生物技术有限公司;6周龄雌性BALB/c小鼠购自安徽医科大学实验动物中心。ECL化学发光试剂盒购自美国ThermoFisher公司;其他试剂均为国产或进口分析纯。
1.2 试验方法
1.2.1 引物设计与合成 根据NCBI中GenBank数据库已发表的ORFV GO株(登录号:KP010354.1)全基因序列,利用Primer Premier 5.0软件设计一对121基因的特异性引物。上游引物F1:5′-CGGG ATCCATGGCTGGCTTCCTAGGTG-3′ (下划线处为BamH Ⅰ酶切位点),下游引物F2:5′-GGAATTCTC ACAGAACTTCCTCCACTTT-3′ (下划线处为EcoR Ⅰ酶切位点),引物由通用生物系统(安徽)有限公司合成。
1.2.2 121基因的克隆 按病毒基因组提取试剂盒使用说明书提取ORFV AH-F10株DNA,以提取的DNA为模板利用PCR技术扩增目的基因。PCR扩增体系(20 μL):rTaq Mix 10 μL,ddH2O 7 μL,上、下游引物及模板各1 μL。PCR扩增条件:94 ℃预变性5 min;94 ℃变性50 s,53 ℃退火1 min,72 ℃延伸1 min,共35个循环;72 ℃再延伸10 min。PCR产物回收后克隆至pMD-19T载体,转化至DH5α感受态细胞中,涂布于Amp (100 μg/mL)平板,37 ℃过夜培养。次日挑取单菌落提质粒,命名为pMD-19T-121,交由南京擎科生物科技有限公司测序。
1.2.3 重组质粒pGEX-6P-1-121的构建 将pMD-19T-121与pGEX-6P-1空载体质粒用限制性内切酶BamH Ⅰ和EcoR Ⅰ进行双酶切,分别回收目的片段,16 ℃连接3 h后同上述相同方法构建原核表达重组质粒pGEX-6P-1-121,经PCR、双酶切鉴定后交由南京擎科生物科技有限公司测序。
1.2.4 重组蛋白的诱导表达与鉴定 将pGEX-6P-1-121及pGEX-6P-1空载体质粒分别转化至E.coli Rosetta感受态细胞中,接种于Amp (100 μg/mL)平板,37 ℃过夜培养。按照1∶100比例接种Amp/LB液体培养基,37 ℃ 200 r/min培养,待OD600nm值在0.6~0.8时加入1 mmol/L IPTG,于35 ℃、150 r/min条件下诱导表达,将诱导出的蛋白进行SDS-PAGE电泳验证。少量诱导表达成功后进行大量诱导,超声裂解并离心后收集上清和沉淀进行SDS-PAGE电泳验证。
1.2.5 蛋白纯化 用Tris缓冲液洗涤沉淀,4 ℃条件下6 000 r/min离心10 min,弃上清,反复洗涤3次。依次用含有2,4,6 mol/L尿素的Tris缓冲液重悬沉淀,4 ℃ 6 000 r/min离心10 min弃上清,收集的沉淀用含8 mol/L尿素的Tris缓冲液重悬,4 ℃过夜。室温下12 000 r/min离心15 min,收集上清至透析袋中,用含6,5,4,2,0 mol/L尿素的Tris缓冲液各透析12 h,对重组蛋白进行梯度透析复性,蔗糖浓缩后收集目的蛋白。
1.2.6 Western-blot检测 将目的蛋白SDS-PAGE电泳,凝胶半干转硝酸纤维素膜后,5%脱脂奶粉4 ℃封闭过夜,TBST洗涤3次,每次10 min;加入1∶1 500稀释的GST标签鼠源单抗作为一抗,放入37 ℃温箱孵育1 h后TBST漂洗3次;加入1∶2 000稀释的辣根过氧化物酶(HRP)标记羊抗鼠IgG 为二抗,37 ℃孵育1 h后TBST漂洗3次每次10 min。DAB试剂盒显色观察。
1.2.7 多克隆抗体的制备 将纯化的121蛋白作为抗原50 μg/只免疫6周龄BALB/c雌鼠。首次免疫时,将蛋白与等体积的弗氏完全佐剂混匀,皮下注射。14 d后进行2次免疫,将蛋白与等体积的弗氏不完全佐剂混匀,皮下注射。按相同方法每隔14 d进行1次加强免疫。4次免疫后对小鼠进行眼眶采血并分离血清。
1.2.8 生物信息学分析 使用ProtParam在线程序分析AH-F10-121蛋白的理化性质;ProtScale软件分析其疏水性;通过在线服务器SignalP 4.1 Server、NetPhos 3.1 Server和TMHMM Server v. 2.0预测该蛋白的信号肽、磷酸化位点与跨膜结构域;SOPMA在线程序预测121蛋白的二级结构;使用DNAStar软件的Protean与ABCpred 在线程序对其B细胞表位进行综合分析。
2 结果与分析
2.1 基因的克隆与分析
以ORFV AH-F10株基因组DNA为模板进行PCR扩增,经琼脂糖凝胶电泳后显示出一条特异性条带,大小约为909 bp(图1),与预期结果符合。
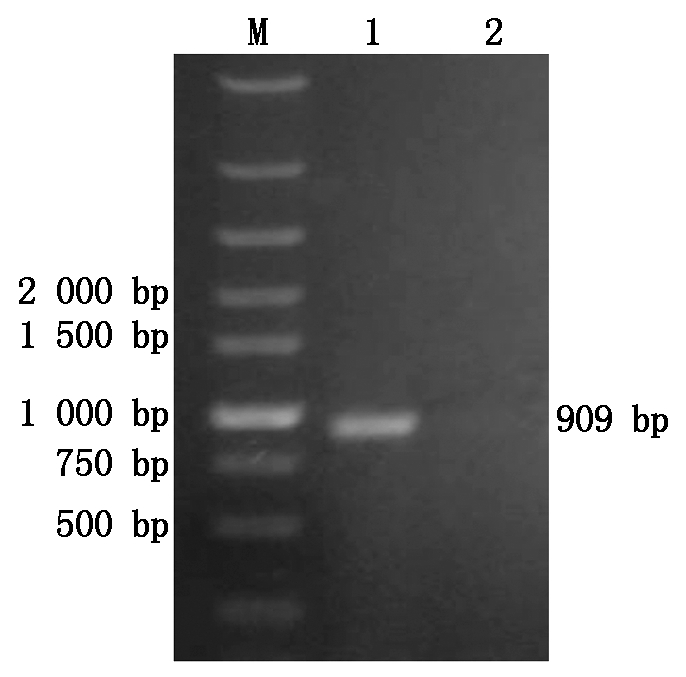
M.DNA Marker;1.121基因的PCR扩增产物;2.阴性对照。
M.DNA Marker;1.121 gene PCR product;2.Negative control.
图1 121基因PCR扩增产物
Fig.1 PCR amplification product of 121 gene
2.2 重组质粒pGEX-6P-1-121的双酶切鉴定
重组质粒pGEX-6P-1-121用限制性内切酶BamH Ⅰ和EcoR Ⅰ双酶切后,经1%琼脂糖凝胶电泳验证,结果显示目的基因条带大小与预期结果相符(图2),测序结果表明重组质粒构建成功。
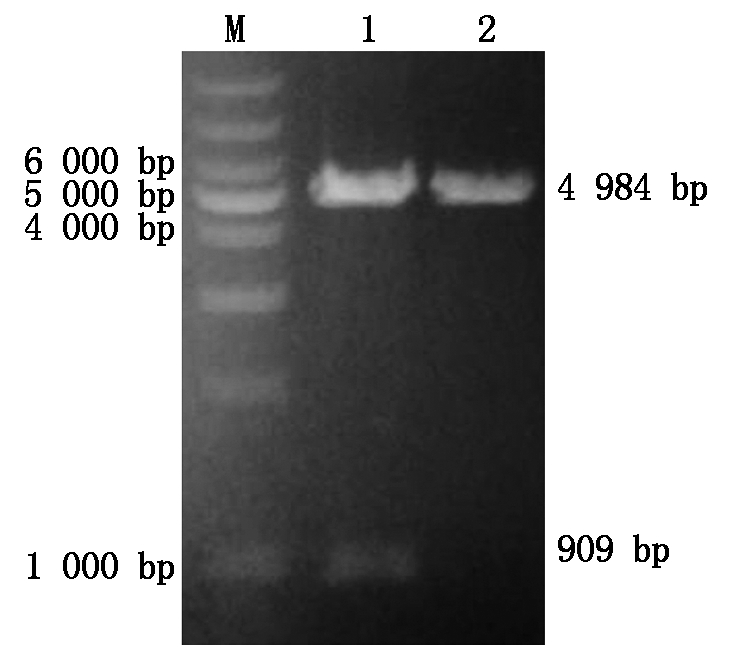
M.DNA Marker;1.重组质粒pGEX-6P-1-121双酶切产物;2.阴性对照。
M.DNA Marker; 1.Product of recombinant plasmid pGEX-6P-1-121
digested by double enzyme; 2.Negative control.
图2 重组质粒pGEX-6P-1-121 的
BamH Ⅰ和EcoR Ⅰ酶切鉴定
Fig.2 Product of recombinant plasmid pGEX-6P-1-121
digested with restriction enzymes BamH Ⅰ and EcoR Ⅰ
2.3 SDS-PAGE分析
将pGEX-6P-1-121质粒转化至Rosetta感受态细胞,成功诱导出大小约为60 ku的特异性蛋白条带(图3),与预期结果相符。
2.4 重组蛋白表达产物的SDS-PAGE电泳裂解上清及沉淀分析
将诱导表达产物超声裂解收集上清和沉淀进行SDS-PAGE电泳验证,结果表明,ORFV 121蛋白在大肠杆菌中主要以包涵体形式表达(图4)。

M.蛋白质分子质量标准;1.pGEX-6P-1空载体未经IPTG诱导;2.pGEX-6P-1空载体经IPTG诱导6 h;3.pGEX-6P-1-121未经IPTG诱导;4.pGEX-6P-1-121经IPTG诱导3 h;5.pGEX-6P-1-121经IPTG诱导4 h;6.pGEX-6P-1-121经IPTG诱导5 h;7.pGEX-6P-1-121经IPTG诱导6 h。
M.Protein Marker;1.pGEX-6P-1 empty vector was not induced by IPTG;2.pGEX-6P-1 empty vector was induced by IPTG for 6 h;3.pGEX-6P-1-121 was not induced by IPTG; 4.pGEX-6P-1-121 was induced by IPTG for 3 h; 5.pGEX-6P-1-121 was induced by IPTG for 4 h;6.pGEX-6P-1-121 was induced by IPTG for 5 h; 7.pGEX-6P-1-121 was induced by IPTG for 6 h.
图3 pGEX-6P-1-121蛋白产物SDS-PAGE电泳
Fig.3 SDS-PAGE electrophoresis analysis of
the pGEX-6P-1-121 protein product
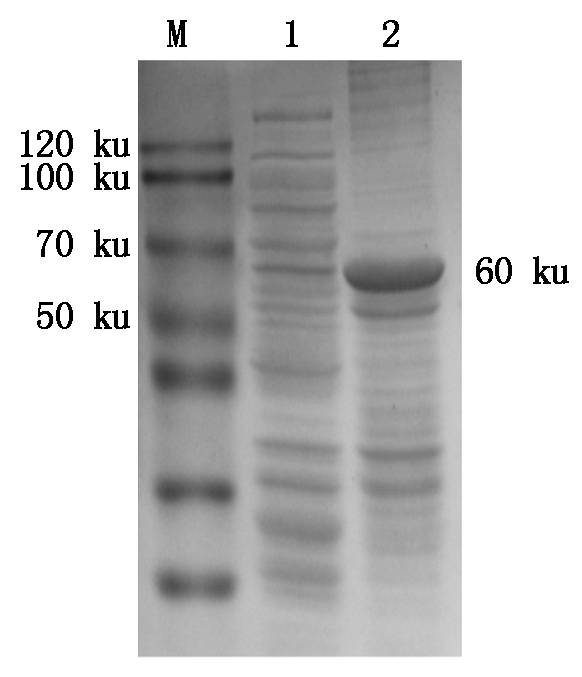
M.蛋白质分子质量标准;1.诱导菌体裂解上清;2.诱导菌体裂解沉淀。
M. Protein Marker; 1.Supernatant of lysate; 2.Precipitation of lysate.
图4 ORFV 121蛋白表达产物SDS-PAGE电泳
Fig.4 SDS-PAGE electrophoresis analysis of
ORFV 121 protein expression products
2.5 Western-blot鉴定
硝酸纤维素膜上显示出一条大小约为60 ku的特异性蛋白条带(图5),与预期结果相符。121蛋白与抗体特异性结合,表明重组蛋白具有良好的反应原性。
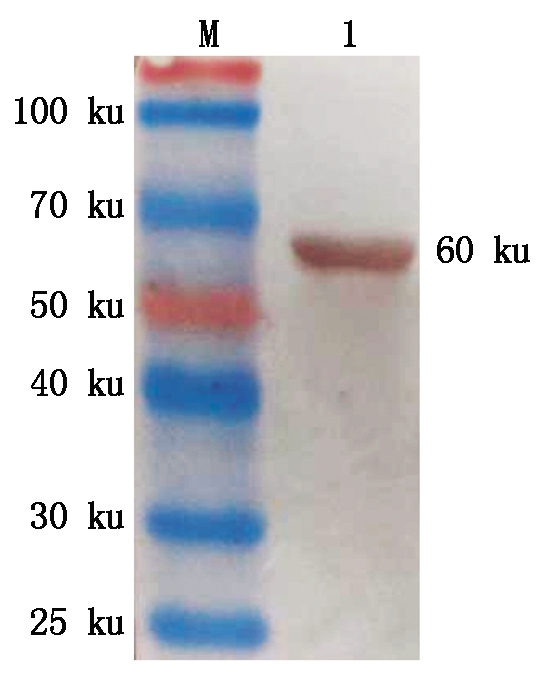
M.蛋白质分子质量标准; 1.pGEX-6P-1-121 重组质粒经IPTG诱导表达。
M.Protein Marker; 1.pGEX-6P-1-121 recombinant
plasmid was induced by IPTG.
图5 ORFV 121蛋白的Western-blot鉴定
Fig.5 Western-blot identification of ORFV 121 protein
2.6 鼠源多抗的Western-blot鉴定
以鼠源血清为一抗对纯化后的121蛋白进行Western-blot鉴定,结果表明,目的蛋白能与血清中的抗体特异性结合(图6),成功制备鼠源多抗。
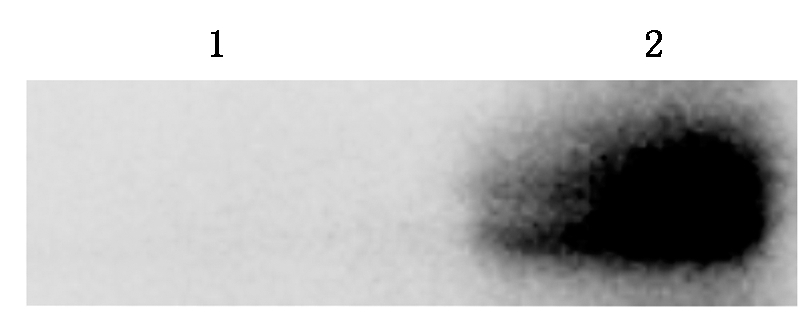
1.pGEX-6P-1空载体经IPTG诱导表达;2.pGEX-6P-1-121
重组质粒经IPTG诱导表达。
1.pGEX-6P-1 empty vector was induced by IPTG;
2.pGEX-6P-1-121 recombinant plasmid was induced by IPTG.
图6 纯化后121蛋白的Western-blot分析
Fig.6 Western-blot analysis of purified 121 protein
2.7 理化性质分析
利用ProtParam在线程序分析该毒株121蛋白的理化性质,结果显示该蛋白由302个氨基酸组成,分子式为C1433H2278N482O464S17,分子量为34 ku,理论pI (等电点)值为8.86,带负电荷的氨基酸数(Asp+Glu)为39,带正电荷的氨基酸数(Arg+Lys)为46。不稳定系数为67.37 (>40),属于不稳定蛋白。平均亲水值(GRAV)为-1.081,脂肪族系数为53.38。利用ProtScale在线服务器(窗口大小设定为11,算法为默认算法)对121蛋白的亲疏水性进行分析,显示标度值<0的区域比>0的较为密集(图7),结合GRAV值为-1.081,预测121蛋白为亲水性蛋白。
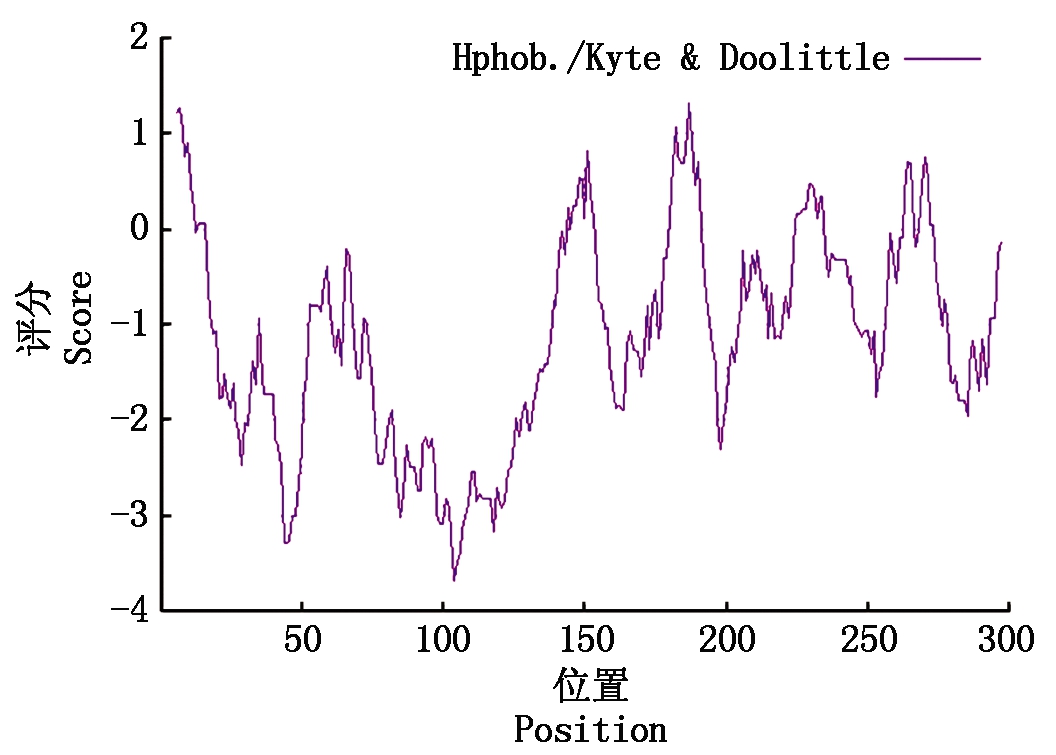
图7 AH-F10-121蛋白的亲疏水性分析
Fig.7 Hydrophilic and hydrophobic
analysis of AH-F10-121 protein
2.8 信号肽、磷酸化位点及跨膜结构域的预测分析
利用SignalP 4.1 Server在线程序对121进行信号肽分析(图8),结果显示该蛋白无信号肽;运用在线软件NetPhos 3.1 Server对121蛋白进行磷酸化位点的预测(图9),当潜在磷酸化位点的阈值为0.5时,121蛋白存在50个磷酸化位点,其中有31个丝氨酸位点,12个苏氨酸位点,7个酪氨酸位点;利用TMHMM Server v.2.0 在线程序预测121蛋白的跨膜结构域(图10),结果表明该蛋白无跨膜结构域。
图8 AH-F10-121蛋白的信号肽
Fig.8 Signal peptide of AH-F10-121 protein
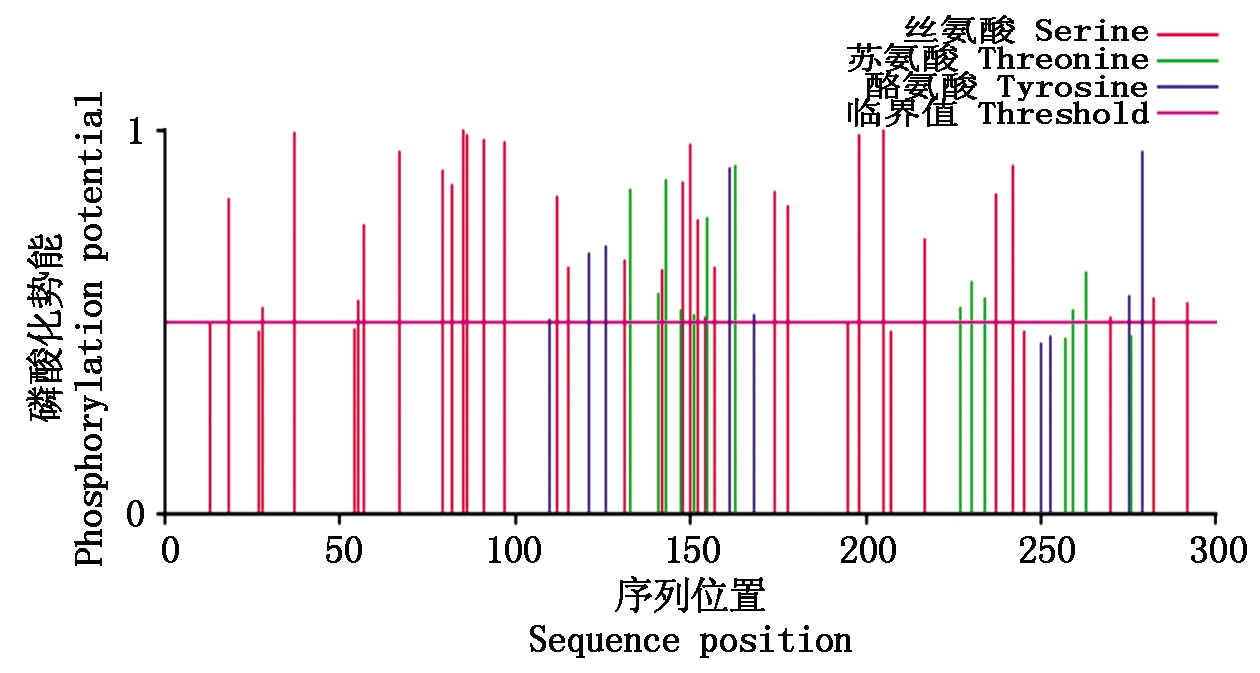
图9 ORFV 121蛋白的磷酸化位点
Fig.9 Phosphorylation site of ORFV 121 protein
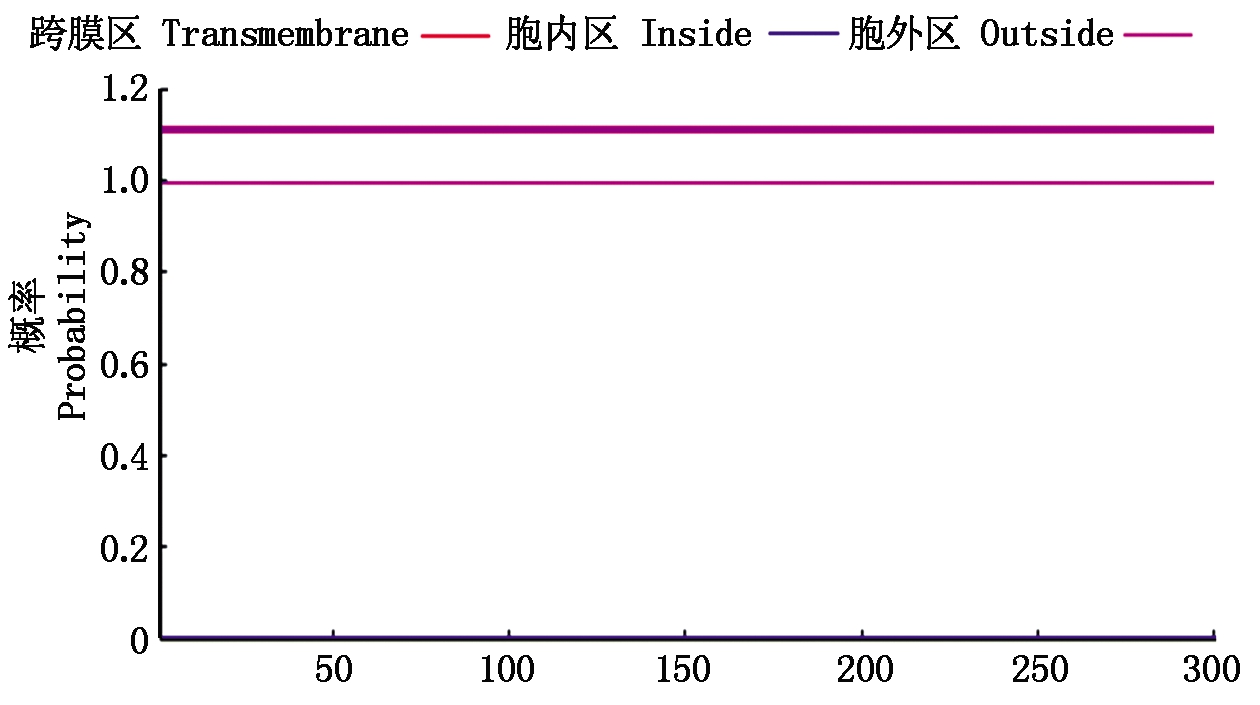
图10 ORFV 121蛋白的跨膜结构域
Fig.10 Transmembrane domain of ORFV 121 protein
2.9 二级结构预测
以SOPMA预测(图11),121蛋白的二级结构由α-螺旋、β-转角、无规则卷曲以及延伸链4种结构组成,其中α-螺旋(h)占27.81%、β-转角(t)占2.98%、无规则卷曲(c)占62.25%、延伸链(e)占6.95%。β-转角为凸出结构,与无规则卷曲多出现在蛋白表面且结构松散,较结构稳定的α-螺旋相比更易与抗体结合,可作为抗原表位,优势区段为20-37,42-55,61-70,73-121,124-142,150-154,159-166,170-222,267-271,277-292。

图11 121蛋白的二级结构
Fig.11 Secondary structure of 121 protein
2.10 B细胞表位的分析
运用DNAStar软件中的Protean程序预测B细胞抗原表位(图12),Kyte-Doolittle法预测AH-F10-121氨基酸亲水性,在16-144,154-178,192-224,241-257,273-297区段的亲水性较强。Karplus-Schulz法预测柔韧性,结果显示有19个柔韧性区段,其中27-37,49-57,75-124,127-157,171-180,193-205,276-289区段柔韧性较强,易发生扭曲折叠,形成B细胞抗原表位的几率较高。Jameson-Wolf法预测抗原性,18-70,72-140,159-178,193-226,253-261,276-302区段抗原指数较高。Emini法预测表面可及性,其中40-52,101-120,193-202区段具有较高的表面可及性。对该蛋白的二级结构、亲水性、柔韧性、抗原性和表面可及性进行综合分析(表1),在20-37,40-57,61-70,73-142,159-166,170-178,193-222,276-292处可能存在B细胞表位。将单参数综合分析结果结合ABCpred[14-15](设置阈值为0.51)初步预测筛选肽段的结果(表2),综合分析预测在22-37,40-52,64-70,95-110,159-166,172-178,201-216,285-292区段可能存在B细胞优势表位。
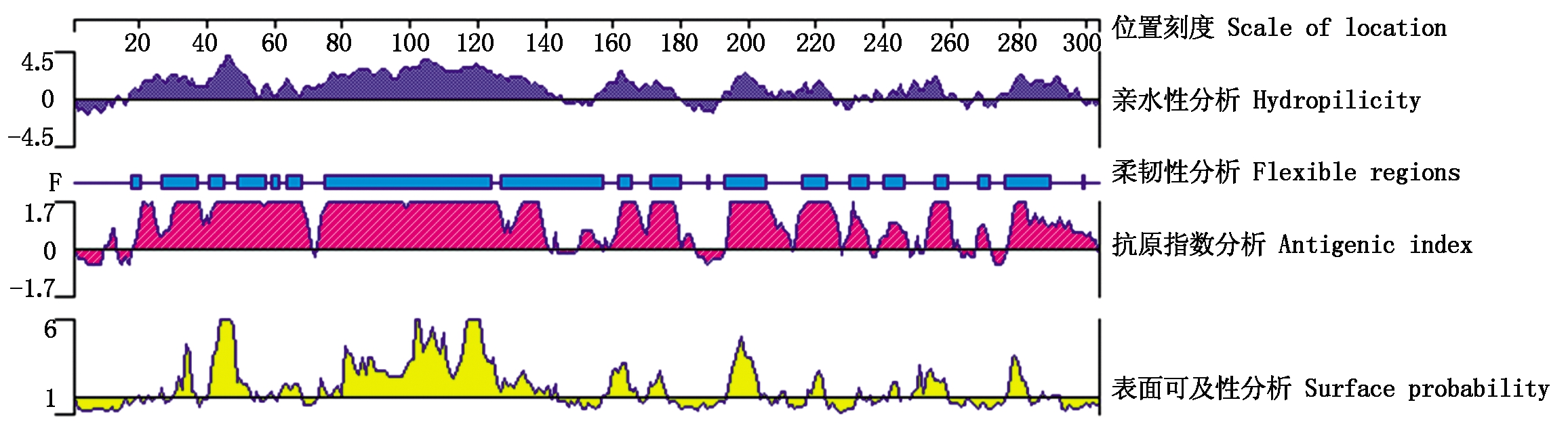
图12 121蛋白的B细胞表位预测
Fig.12 B cell epitope prediction of 121 protein
表1 综合不同数据分析ORFV 121蛋白B细胞抗原表位的位置
Tab.1 Analysis of the location of the ORFV 121 protein B cell epitopes in combination with different data

方法Method位置Position二级结构 Secondary structure20-37,42-55,61-70,73-121,124-142,150-154,159-166,170-222,267-271,277-292亲水性 Hydrophilicity16-144,154-178,192-224,241-257,273-297柔韧性 Flexibility27-37,49-57,75-124,127-157,171-180,193-205,276-289抗原性 Antigenicity18-70,72-140,159-178,193-226,253-261,276-302表面可及性 Surface probality40-52,101-120,193-202综合分析 Parameter analysis20-37,40-57,61-70,73-142,159-166,170-178,193-222,276-292
表2 ABCpred程序初步预测筛选肽段的分析
Tab.2 The analysis of the peptides obtained by preliminary prediction with ABCpred program

肽位22-3737-5264-7995-110155-170172-187201-216285-300Position of peptides分值 Score0.950.930.890.930.880.840.880.78
3 讨论
羊口疮是由ORFV引起的高接触性嗜上皮性人畜共患传染病。ORFV感染宿主后,口唇周围的病变影响采食,继发的病菌感染提高了患畜的死亡率。近几年的相关报道中显示该病的宿主范围呈现扩大趋势,人感染羊口疮的病例也逐年增多[16-17]。此外葛士坤等[18]的研究发现中国ORFV毒株进化特殊,突变率高,加大了我国针对羊口疮制定统一防控措施的难度。ORFV 121位于基因组的末端变异区,在全基因组中缺失时可有效降低ORFV的毒力使患畜出现弱毒症表型,对ORFV的毒力起决定性作用[19-20]。因此,对121基因及其所编码蛋白的深入研究对羊口疮的诊断防控具有重要意义。
本研究以ORFV AH-F10为模板扩增121基因,选用具有与重组蛋白融合表达率高及确保纯化蛋白纯度等优势的pGEX-6P-1大肠杆菌表达载体[21],成功构建重组质粒pGEX-6P-1-121。利用原核表达系统实现ORFV 121基因在大肠杆菌体系中的外源表达,并对IPTG浓度,诱导温度、时间、转速等因素进行优化,结果显示,在35 ℃、150 r/min、IPTG终浓度为1 mmol/L条件下诱导6 h,蛋白表达量最大。对目的蛋白进行SDS-PAGE分析显示重组蛋白主要以不溶性的包涵体形式存在。为提高纯化过程中变性蛋白的复性率,试验采用透析复性,通过逐渐降低复性液中的尿素浓度进行梯度复性,有效减少了复性过程中无活性蛋白聚体的出现。Western-blot结果显示ORFV 121蛋白能与鼠源血清发生特异性免疫反应,表明血清中含抗ORFV的多克隆抗体,多抗血清制备成功。利用生物信息学相关软件对ORFV 121蛋白进一步分析,结果表明其为亲水性不稳定蛋白,分子量为34 ku,与制备的重组蛋白大小相符。无信号肽与跨膜结构域,有文献报道信号肽对分泌蛋白的分泌效果起主导作用,利用信号肽可使外源基因在原核表达系统中实现可溶性表达[22],121蛋白无信号肽可能为重组蛋白主要以包涵体表达的原因之一,为下一步获得可溶性蛋白提供思路。磷酸化是蛋白质最重要的翻译后修复过程之一,是调控蛋白活性功能的通用机制,在细胞信号传导过程中起重要作用,NetPhos 3.1 Server预测121蛋白共有50个磷酸化位点,推测该蛋白可能通过潜在位点识别蛋白激酶进行磷酸化反应参与干扰宿主细胞的信号传导。二级结构分析结果表明121蛋白属混合型蛋白,以无规则卷曲、α-螺旋结构居多,间杂少量的β-转角结构。其中无规则卷曲和β-转角结构突出、稳定性差,易于盘旋和扭曲,容易与抗体分子结合,因此该区域可能存在优势抗原表位[23]。对线性B细胞表位进行预测,显示该蛋白含有8个亲水性、柔韧性、抗原性和表面可及性指数高的区段,并结合ABCpred预测结果进行综合分析,结果显示,在第22-37,40-52,64-70,95-110,159-166,172-178,201-216,285-292位可能存在优势抗原表位。进而推断该蛋白具有一定的免疫原性,与制备的鼠源多抗血清的Western-blot鉴定结果相印证。
综上,本试验通过原核表达系统成功制备了具有良好免疫原性的ORFV 121蛋白,并对其基本理化性质、结构与功能特性、潜在B细胞抗原表位进行生物信息学分析。为后续对该蛋白的深入研究以及抗体诊断试剂盒的研发奠定基础。
[1] Yu Y Z, Tan Q, Zhao W B, Zhang X, Ma J Z, Wu Z J, Zhu Z B, Cui Y D. Characterization of an Orf virus isolate from an outbreak in Heilongjiang Province, China[J]. Archives of Virology, 2017, 162(10): 3143-3149. doi: 10.1007/s00705-017-3426-x.
[2] Peralta A, Robles C, Martínez A, Alvarez L, Valera A, Calamante G, König G A. Identification and molecular characterization of Orf virus in Argentina[J]. Virus Genes, 2015, 50(3): 381-388. doi: 10.1007/s11262-015-1189-6.
[3] Yang Y, Qin X D, Wang G X, Jin J X, Shang Y J, Zhang Z D. Development of an isothermoal amplification-based assay for rapid visual detection of an Orf virus[J]. Virology Journal, 2016, 13(1): 46. doi: 10.1186/s12985-016-0502-x.
[4] Chen H Q, Li W, Kuang Z Z, Chen D X, Liao X Q, Li M, Luo S H, Hao W B. The whole genomic analysis of Orf virus strain HN3/12 isolated from Henan Province, central China[J]. Bmc Veterinary Research, 2017, 13(1): 260. doi: 10.1186/s12917-017-1178-1.
[5] Chan K W, Lin J W, Lee S H, Liao C J, Tsai M C, Hsu W L, Wong M L, Shih H C. Identification and phylogenetic analysis of Orf virus from goats in Taiwan[J]. Virus Genes, 2007, 35(3): 705-712. doi: 10.1007/s11262-007-0144-6.
[6] 成伟伟, 张克山, 刘永杰, 孔汉金, 尚佑军, 刘湘涛, 刘磊. 羊传染性脓疱病毒基因组结构和主要基因功能研究进展[J]. 动物医学进展, 2014, 35(7): 82-85. doi: 10.16437/j.cnki.1007-5038.2014.07.011.
Cheng W W, Zhang K S, Liu Y J, Kong H J, Shang Y J, Liu X T, Liu L. Advance in genome structure and major gene function of Orf virus[J]. Progress in Veterinary Medicine, 2014, 35(7): 82-85.
[7] 冯倩, 吴锦艳, 陈妍, 刘永杰, 张克山, 尚佑军, 刘湘涛. 羊口疮病毒疫苗株与野毒株VIL-10基因的比较分析[J]. 中国畜牧兽医, 2018, 45(9): 2368-2376. doi: 10.16431/j.cnki.1671-7236.2018.09.003.
Feng Q, Wu J Y, Chen Y, Liu Y J, Zhang K S, Shang Y J, Liu X T, Comparative analysis of VIL-10 gene between the vaccine and wild strains of Orf virus[J]. China Animal Husbandry & Veterinary Medicine, 2018, 45(9): 2368-2376.
[8] 孙正楠, 毛从剑, 张焕容. 山羊口疮病毒四川分离株F1L基因分子特征分析及抗原表位预测[J]. 中国畜牧兽医, 2018, 45(7): 1942-1948. doi: 10.16431/j.cnki.1671-7236.2018.07.027.
Sun Z N, Mao C J, Zhang H R. Molecular characteristics analysis and antigen epitope prediction of F1L gene of Orf virus isolate from Sichuan[J]. China Animal Husbandry & Veterinary Medicine, 2018, 45(7): 1942-1948.
[9] Olivero N, Reolon E, Arbiza J, Berois M. Genetic diversity of Orf virus isolated from sheep in Uruguay[J]. Archives of Virology, 2018, 163(5): 1285-1291. doi: 10.1007/s00705-018-3717-x.
[10] 何亚鹏, 张琪, 庞文静, 徐丽美, 付明哲, 许信刚. 羊口疮病毒陕西分离株B2L基因的克隆、序列分析及原核表达[J]. 动物医学进展, 2016, 37(12): 19-23. doi: 10.16437/j.cnki.1007-5038.2016.12.004.
He Y P, Zhang Q, Pang W J, Xu L M, Fu M Z, Xu X G. Cloning, sequence analysis and prokaryotic expression of B2L gene of Orf virus Shaanxi strain[J]. Progress in Veterinary Medicine, 2016, 37(12): 19-23.
[11] 闫丰超, 邵佳, 窦永喜. 羊口疮病毒分子生物学的研究进展[J]. 中国兽医科学, 2013,43(1): 103-109. doi: 10.16656/j.issn.1673-4696.2013.01.009.
Yan F C, Shao J, Dou Y X, Progress in molecular biology of Orf virus[J]. Chinese Veterinary Science, 2013,43(1): 103-109.
[12] Diel D G, Luo S, Delhon G, Peng Y, Flores E F, Rock D L. Orf virus ORFV121 encodes a novel inhibitor of NF-κB that contributes to virus virulence[J]. Journal of Virology, 2011, 85(5): 2037-2049. doi: 10.1128/JVI.02236-10.
[13] Wang Y, Yang K K, Bai C X, Yin D D, Li G, Qi K Z, Wang G J, Li Y D. Development of a SYBR Green I Real-time PCR for the detection of the Orf virus[J]. Amb Express, 2017, 7(1): 21. doi: 10.1186/s13568-016-0322-9.
[14] Saha S, Raghava G P S. Prediction of continuous B-cell epitopes in an antigen using recurrent neural network[J]. Proteins-structure Function & Bioinformatics, 2010, 65(1):40-48. doi: 10.1002/prot.21078.
[15] 王光祥, 尚佑军, 吕占禄, 张克山, 田宏, 刘湘涛. 羊口疮病毒F1L蛋白二级结构分析与表位预测[J]. 中国人兽共患病学报, 2012, 28(12): 1185-1190. doi: 10.3969/cjz.j.issn.1002-2694.2012.12.006.
Wang G X, Shang Y J, Lü Z L, Zhang K S, Tian H, Liu X T. Prediction of secondary structures and cell epitopes of F1L protein of ORFV[J]. Chinese Journal of Zoonoses, 2012, 28(12): 1185-1190.
[16] Tryland M, Beckmen K B, Burek-Huntington K A, Breines E M, Klein J. Orf virus infection in Alaskan mountain goats, Dall′s sheep, muskoxen, caribou and Sitka black-tailed deer[J]. Acta Veterinaria Scandinavica, 2018, 60(1): 12. doi: 10.1186/s13028-018-0366-8.
[17] Hasheminasab S S, Mahmoodi A, Mahmoodi P, Maghsood H. Orf virus infection in human Ecthyma contagiosum: a report of two cases in the West of Iran[J]. Virusdisease, 2016, 27(2): 209-210. doi: 10.1007/s13337-016-0304-1.
[18] 葛士坤, 张凯照, 鲁荣, 刘健新, 宁章勇. 2006-2016年中国羊口疮病毒的遗传演化分析[J]. 中国畜牧兽医, 2018, 45(2): 439-447. doi: 10.16431/j.cnki.1671-7236.2018.02.019.
Ge S K, Zhang K Z, Lu R, Liu J X, Ning Z Y. Genetic and phylogenetic analysis of Orf virus isolated in China from 2006 to 2016[J]. China Animal Husbandry & Veterinary Medicine, 2018, 45(2): 439-447.
[19] 刘方, 赵玄多, 安贝, 刘慧, 高洋, 张立强, 贾芸晓, 陈德坤. 羊口疮病毒凤翔株ORFV121和vIL-10基因的生物信息学分析[J]. 动物医学进展, 2014, 35(7): 52-58. doi: 10.16437/j.cnki.1007-5038.2014.07.009.
Liu F, Zhao X D, An B, Liu H, Gao Y, Zhang L Q, Jia Y X, Chen D K. Bioinformatics analyses ORFV121 gene and vIL-10 gene of ORFV-FX0910 strain[J]. Progress in Veterinary Medicine, 2014, 35(7): 52-58.
[20] Khatiwada S, Delhon G, Nagendraprabhu P, Chaulagain S, Luo S, Diel D G, Flores E F, Rock D L. A parapoxviral virion protein inhibits NF-κB signaling early in infection[J]. PLoS Pathogens, 2017, 13(8):e1006561. doi: 10.1371/journal.ppat.1006561.
[21] 赵巍, 苏川, 吴海玮, 胡雪梅, 沈蕾, 王荣芝, 马磊, 陈淑贞, 张兆松. 重组质粒pGEX-6P-1/Sj-FABPc的构建及在大肠杆菌中的表达[J]. 中国血吸虫病防治杂志, 2000(5): 261-264. doi: 10.16250/j.32.1374.2000.05.003.
Zhao W, Su C, Wu H W, Hu X M, Shen L, Wang R Z, Ma L, Chen S Z, Zhang Z S. Construction of recombinant pGEX-6P-1/Sj-FABPc and expression in E.coli[J]. Chinese Journal of Schistosomiasis Contral, 2000(5): 261-264.
[22] 黄宇良, 汪立平, 陆克文, 邵会娟, 王正全. 片球菌素pedA基因的原核表达、纯化及生物信息学分析[J]. 生物技术通报, 2017, 33(12): 144-150. doi: 10.13560/j.cnki.biotech.bull.1985.2017-0373.
Huang Y L, Wang L P, Lu K W, Shao H J, Wang Z Q. Prokaryotic expression, purification and bioinformatics analysis of pediocin pedA gene[J]. Biotechnology Bulletin, 2017, 33(12): 144-150.
[23] 李林杰, 常秋燕, 马鹏, 王悦萦, 马晓霞, 柏家林. PPRV Nigeria75/1 H蛋白原核表达及抗原表位预测[J]. 华北农学报, 2018, 33(5): 111-116. doi: 10.7668/hbnxb.2018.05.016.
Li L J, Chang Q Y, Ma P, Wang Y Y, Ma X X, Bai J L. Prokaryotic expression of PPRV Nigeria75/1 H protein and prediction of antigen epitope[J]. Acta Agriculturae Boreali-Sinica, 2018, 33(5): 111-116.