从遗传上提高猪抗病力及胴体瘦肉率对降低防御成本和获得最佳经济效益具有重要的现实意义,同时,也能更好地满足消费者对猪肉品质越来越高的要求。STAB2基因又称Stabilin-2,是清道夫受体家族成员之一,可通过其内皮生长因子样结构域识别乙酰化的低密度脂蛋白、透明质酸,硫酸软骨素、肝素、晚期糖基化终末产物等有害物质、体内原发性坏死细胞以及体外病原体膜表面的磷脂酰丝氨酸继而实现对这些物质的清除[1-4]。同时,Stabilin-2也参与淋巴细胞对肝窦内皮细胞的黏附及凋亡细胞清除等[5-6]。此外,研究还证实,STAB2基因可促进肠癌淋巴结的转移,而抑制STAB2可促进体内循环透明质酸的水平,并防止肿瘤的转移[7],同时,肝脏肿瘤周围STAB2的表达与患者的存活率之间呈负相关关系[8]。最新的研究还发现,Stabilin-2蛋白也可参与调节肌源性分化和肌肉再生过程中肌细胞融合的效率[9-10]。由此可见,Stabilin-2蛋白STAB2基因在机体的免疫和肌肉分化和肌细胞融合中均扮演者重要角色。但是,有关STAB2基因的转录调控机制目前仍然还不清楚。
本研究以荣昌猪基因组为模板,以人293细胞为细胞模型,通过克隆和构建荣昌猪转录调控区不同缺失长度的报告基因系统,初步分析了猪STAB2基因的启动子活性以及转录调控元件,为进一步研究猪STAB2基因的结构、功能及表达机制等奠定了基础。
1 材料和方法
1.1 试验材料
供体猪耳组织样由重庆市畜牧科学院荣昌猪国家级保种场提供;DNA提取试剂盒购自QIAGEN公司;限制性内切酶Xho Ⅰ和Hind Ⅲ、Q5超保真DNA聚合酶和T4 DNA连接酶购自NEB公司;人293细胞购自重庆源于启生物技术有限公司;胎牛血清和DMEM培养基购自Gbico公司;脂质体LipofectamineTM 2000转染试剂购自Invitrogen公司;pGL4.17骨架载体、pRL-TK内参载体、蛋白酶K和双荧光素酶报告基因系统试剂盒购自Promega公司;去内毒素质粒提取试剂盒购自OMEGA公司。
1.2 STAB2转录调控区生物信息学分析
STAB2基因转录调控区内核心启动子分布用Neural Network Promoter Prediction(http://www.fruitfly.org/seq_tools/promoter.html)软件预测;STAB2基因转录调控区内CpG岛用MethPrimer软件预测;STAB2基因核心启动子区转录因子结合位点用JASPAR(http://jaspar.genereg.net/)软件预测,阈值设置为0.9。
1.3 STAB2基因转录调控区各5′端缺失片段的扩增
参考NCBI数据库中的猪STAB2基因组序列(GenBank: NC_010447.5),利用Primer 3.0软件设计STAB2基因候选转录调控区各5′端缺失片段的扩增引物,同时利用DNAman软件分析候选转录调控序列所包含的酶切位点,再结合pGL4.17载体上包含的多克隆位点,进而确定本试验的最适酶切位点,引物信息详见表1。
试验用DNA模板的提取参照试剂盒说明书。DNA浓度及OD值用Nanodrop 2000测定,完整性通过1%琼脂糖凝胶电泳鉴定。随后以提取的DNA为模板,利用PCR的方法,扩增STAB2基因转录调控区各5′端缺失片段。扩增所使用的反应体系及反应程序均参照参考文献[11]。
表1 STAB2重组载体构建引物
Tab.1 Primers for the reporter construction of STAB2
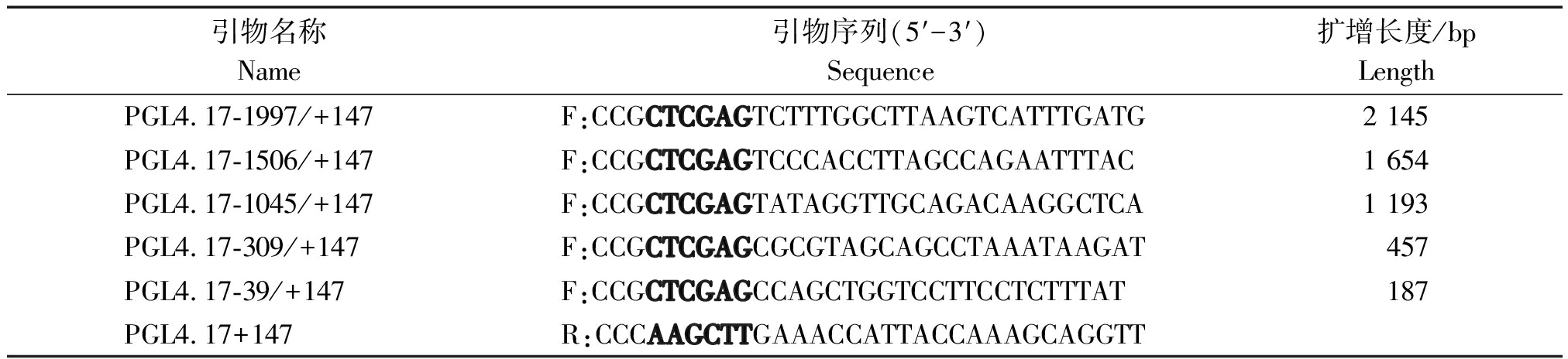
引物名称Name引物序列(5′-3′)Sequence 扩增长度/bpLengthPGL4.17-1997/+147F:CCGCTCGAGTCTTTGGCTTAAGTCATTTGATG2 145PGL4.17-1506/+147F:CCGCTCGAGTCCCACCTTAGCCAGAATTTAC1 654PGL4.17-1045/+147F:CCGCTCGAGTATAGGTTGCAGACAAGGCTCA1 193PGL4.17-309/+147F:CCGCTCGAGCGCGTAGCAGCCTAAATAAGAT457PGL4.17-39/+147F:CCGCTCGAGCCAGCTGGTCCTTCCTCTTTAT187PGL4.17+147R:CCCAAGCTTGAAACCATTACCAAAGCAGGTT
注:黑色加粗的大写字母代表酶切位点;黑色加粗大写字母左侧为酶切位点保护碱基。
Note:Bold capital letters represent cleavage sites and its left letters are protection base of cleavage sites.
1.4 STAB2基因转录调控区各5′端缺失重组载体的构建
将PCR扩增得到的各5′端缺失片段产物进行纯化,随后将纯化产物用Xho Ⅰ和Hind Ⅲ内切酶进行双酶切,具体反映体系参照试剂说明书。酶切产物经2%琼脂糖凝胶后进行胶回收以获得目的片段。最后,将目的片段与骨架载体pGL4.17进行4 ℃过夜连接,将连接产物转化进大肠杆菌后,利用菌液PCR来鉴定阳性克隆。同时利用双酶切和测序来进行重组载体的验证。
1.5 人293细胞培养及细胞转染
人293细胞的培养及细胞转染方法参照参考文献[11]。每个重组载体设置3个生物学重复。
1.6 STAB2基因转录调控区活性分析
转染细胞后48 h收集细胞。利用双荧光素酶活性检测试剂盒分析各重组载体的荧光素酶活性,具体操作见试剂盒使用说明。
1.7 统计分析
用Excel对获得的试验数据进行分析,同时利用T检验分析各重组载体之间荧光素酶活性的显著性差异,以P<0.05为差异显著。
2 结果与分析
2.1 STAB2 候选启动子区生物信息学分析
本试验首先利用巢式PCR成功扩增到STAB2基因转录起始位点上游1 997 bp的区域。核心启动子分布分析发现这1 997 bp序列中包含4个核心启动子,分别位于转录起始位点上游1 982-1 933 bp,1 372-1 323 bp,927-878 bp和228-179 bp(表2)。CpG岛分析结果揭示,这段序列中存在1个CpG岛,位置为-331--14 bp(318 bp)(图1)。
表2 STAB2调控区域内核心启动子分布
Tab.2 The distribution of core promoters in STAB2 regulation area
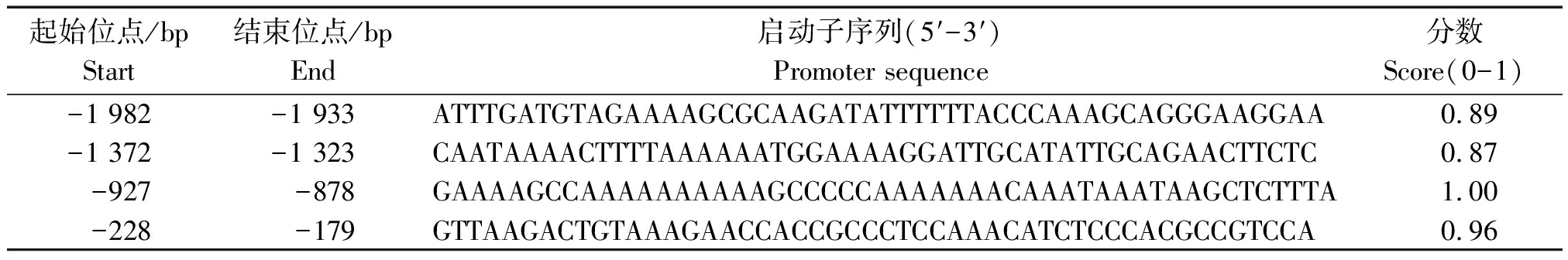
起始位点/bpStart结束位点/bpEnd启动子序列(5′-3′)Promoter sequence 分数Score(0-1)-1 982-1 933ATTTGATGTAGAAAAGCGCAAGATATTTTTTACCCAAAGCAGGGAAGGAA0.89-1 372-1 323CAATAAAACTTTTAAAAAATGGAAAAGGATTGCATATTGCAGAACTTCTC0.87-927-878GAAAAGCCAAAAAAAAAAGCCCCCAAAAAAACAAATAAATAAGCTCTTTA1.00-228-179GTTAAGACTGTAAAGAACCACCGCCCTCCAAACATCTCCCACGCCGTCCA0.96
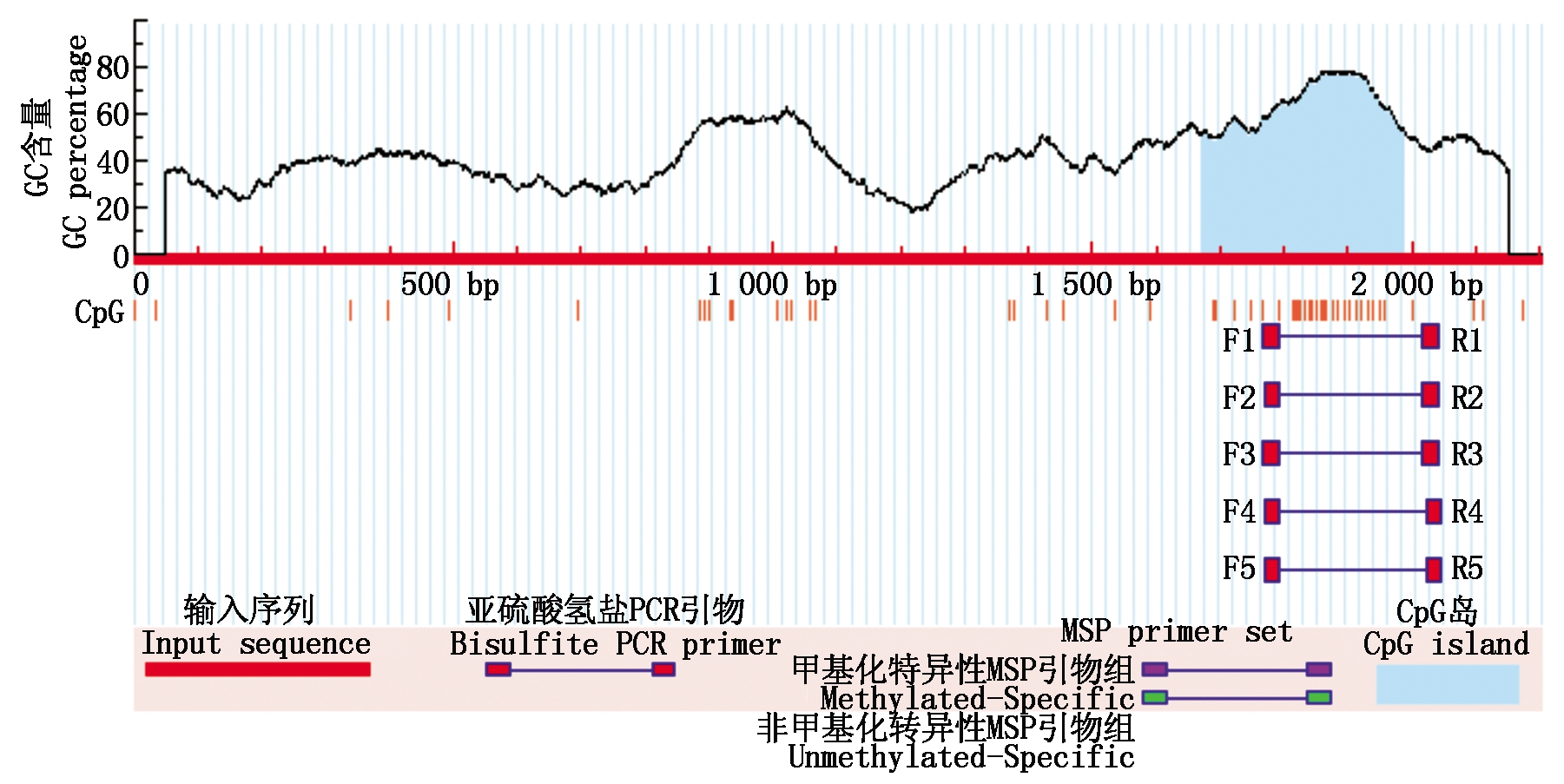
图1 STAB2基因启动子区CpG岛的预测
Fig.1 CpG island prediction of STAB2 gene promoter regions
2.2 STAB2基因转录调控区各5′端缺失片段的获取
利用PCR扩增和1%琼脂糖凝胶电泳检测发现,STAB2基因转录调控区各5′端缺失片段大小与预期的大小一致(图2)。
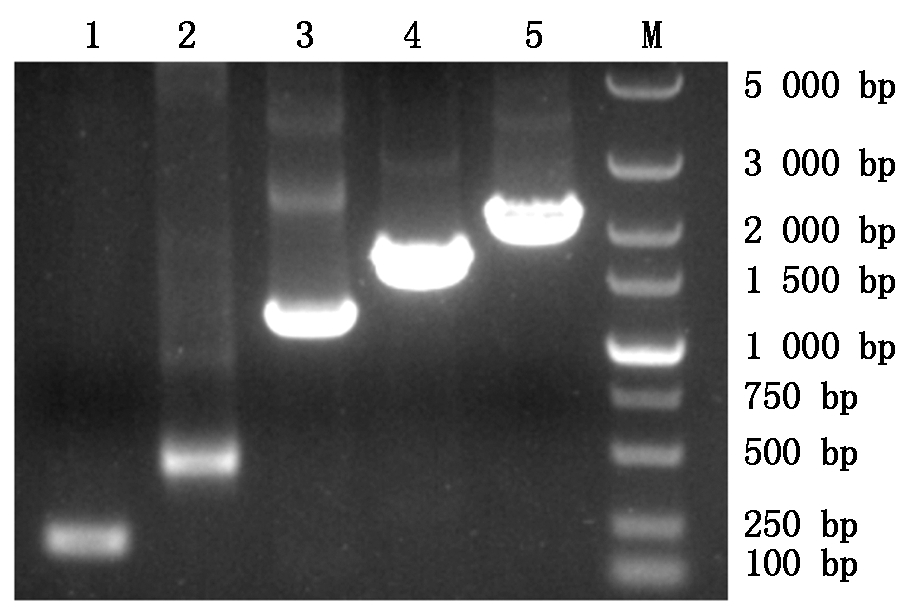
1.187 bp;2.457 bp;3.1 193 bp;4.1 654 bp;
5.2 145 bp;M.DL5000 Marker。
图2 STAB2基因转录调控区扩增的琼脂糖凝胶电泳图
Fig.2 Agarose gel electrophoresis of amplified fragments
of the STAB2 gene transcriptional regulatory region
2.3 STAB2基因转录调控区各5′端缺失重组载体的构建
本试验共构建了5个不同长度的5′端缺失重组载体:pGL4.17-1997、pGL4.17-1506、pGL4.17-1045、pGL4.17-309、pGL4.17-39。利用菌液PCR和双酶切试验进行鉴定,结果证实所构建的重组载体中插入片段的长度大小与预期一致(图3)。测序和序列比对的方法则进一步确认重组载体中的插入片段为目的片段。
2.4 STAB2基因启动子活性分析
利用双荧光素酶报告系统分析各重组质粒的活性,结果发现,除pGL4.17-39与对照载体pGL4.17之间差异不显著外,所构建的剩余4个不同长度的重组载体的启动子活性均显著高于对照载体pGL4.17。其中,pGL4.17-1045的活性又显著性高于pGL4.17-1997、pGL4.17-1506和pGL4.17-309,而pGL4.17-1997、pGL4.17-1506和pGL4.17-309三者之间的活性差异不显著(图4)。由此可知,STAB2基因的核心启动子区位于转录起始位点上游-309--39 bp区域,在-1 045--309 bp可能存在一个正向调控元件,而在-1 506--1 045 bp可能存在一个负向调控元件。
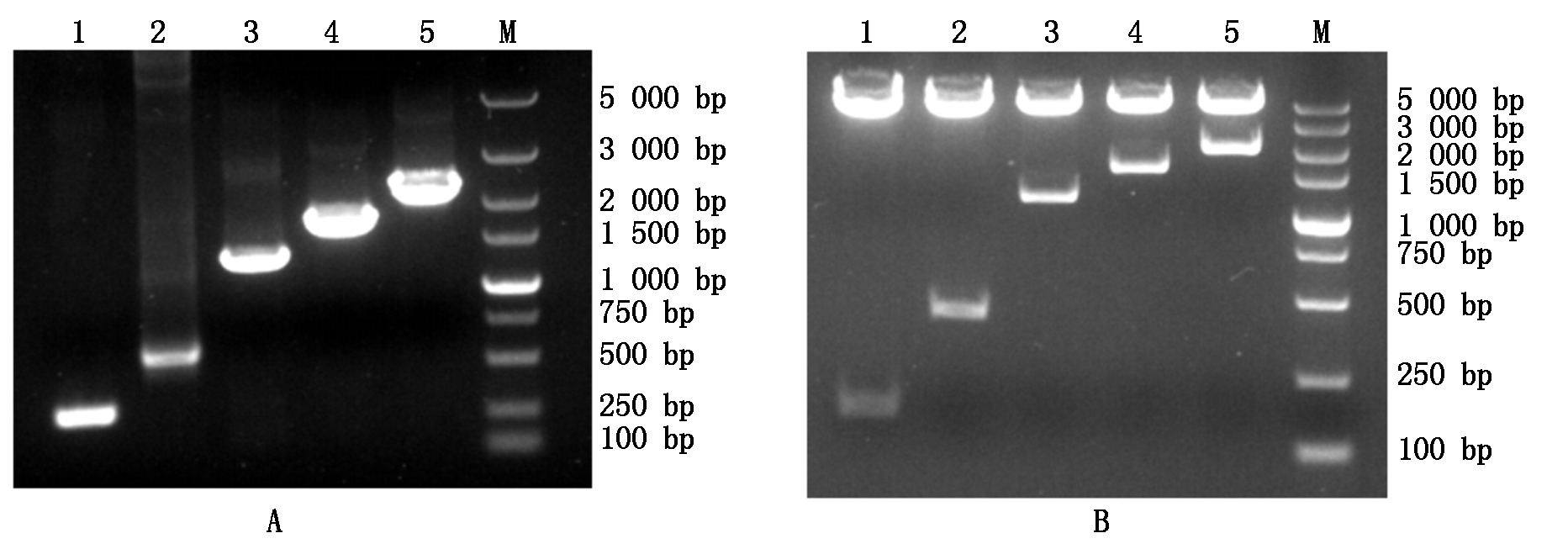
图3 重组载体PCR及双酶切鉴定
Fig.3 PCR and double enzyme identification of recombinant vector
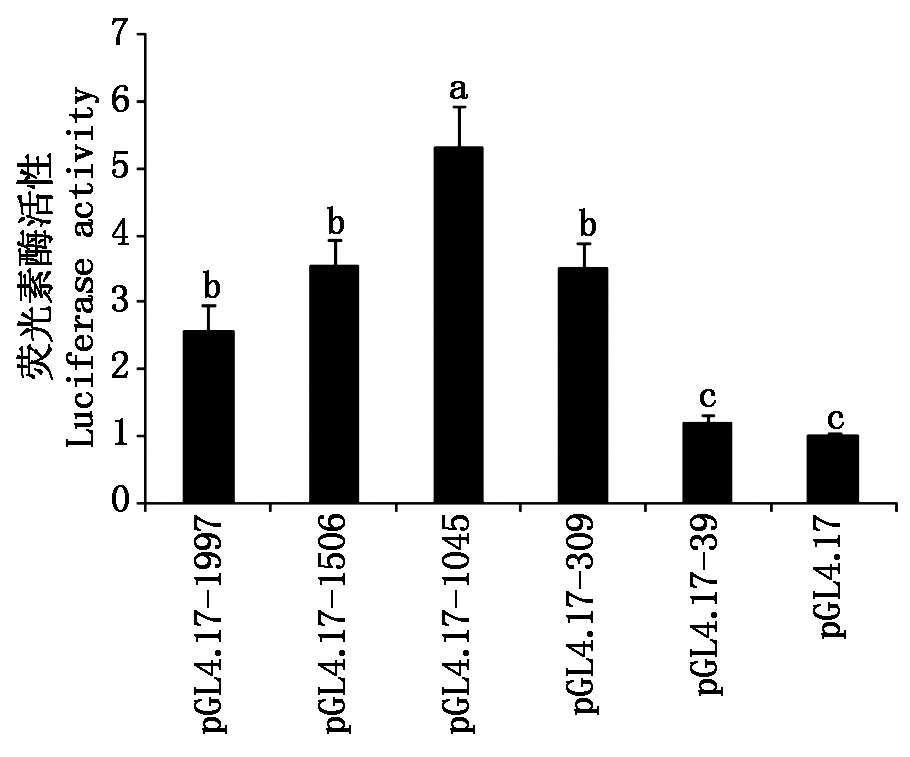
图4 不同长度片段荧光素酶活性分析
Fig.4 Luciferase activity analysis of
different length fragments
表3 STAB2基因的核心启动子区转
录因子及其结合位点预测
Tab.3 Prediction of transcription factors and its putative
binding sites of core promoter regions of STAB2 gene
转录因子Transcriptionfactor预测位点序列Predicted site sequence结合位点个数Number of binding sitesArnt∶∶AhrGGCGTG17BRCA1ACAACGC1FOXC1AGACTGTA1FOXO3GGAAAACA1HIF1A∶∶ARNTGGGCGTGG10HltfGCACATATCT1Klf4AGGGCGTGGA10KLF5TCCACGCCCT10MafbGCTGACTC1NFATC2TTTTCCA1NFICCTGGCA1SOX10CGTTGT1ZNF354CAACCAC17
2.5 STAB2基因核心启动子区序列分析
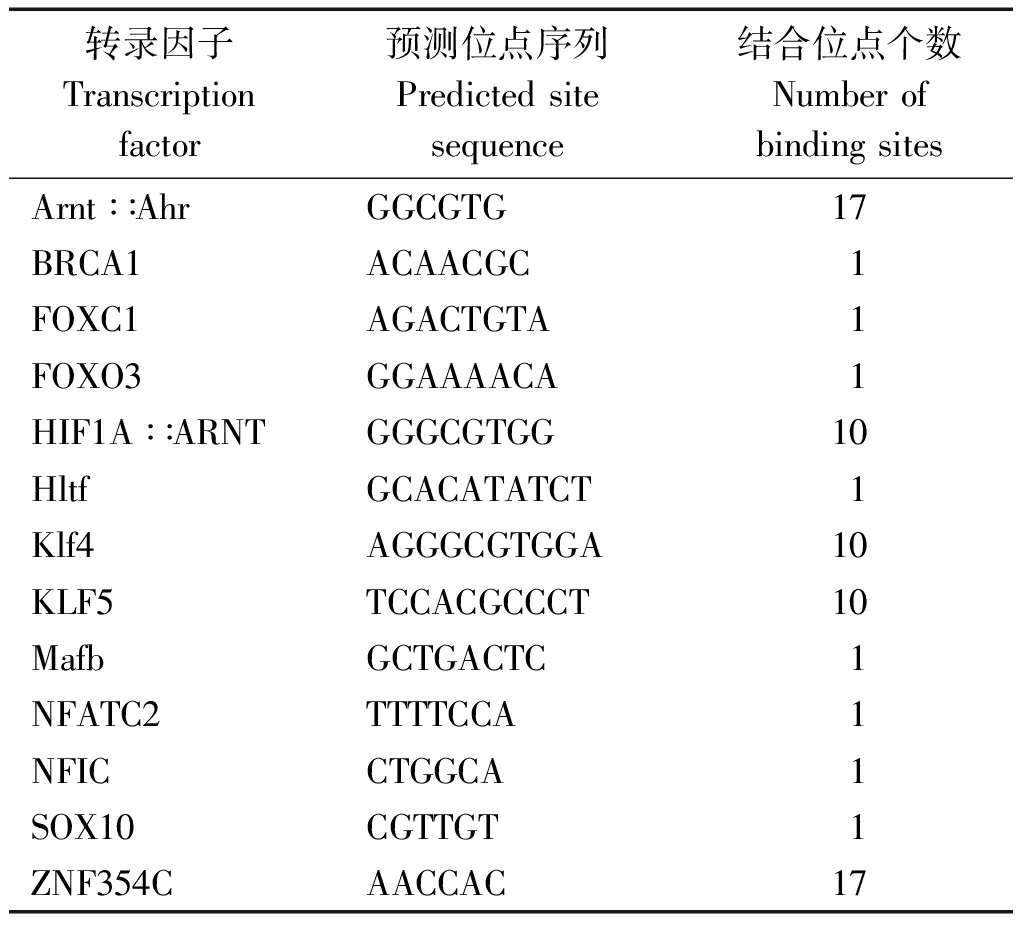
STAB2核心启动子区长度为271 bp(-309--39 bp)区域,位于所预测的甲基化岛区域内(-331--14 bp)。对这一区域中的潜在转录因子及其结合位点进行分析发现,这一区域包含72个转录因子结合位点,部分转录因子在这一区域具有多个结合位点,如转录因子Arnt∷Ahr和ZNF354C的结合位点各有17个,Klf4和KLF5的结合位点各有10个(表3);并且,转录因子结合位点之间也存在重合区。
3 讨论与结论
STAB2基因具有清除体内有害物质、体内原发性坏死细胞、体外病原体以及调节肌细胞肌源性分化和肌肉再生的功能,因此在机体的免疫和肌肉分化和肌细胞融合中均扮演者重要角色。但是,STAB2基因在猪上的转录调控机制目前仍不清楚。本研究首次对荣昌猪STAB2基因的启动子区域进行了克隆及生物信息学分析,同时利用双荧光素酶报告基因系统验证了核心启动子区、关键调控区以及位于启动子区域内的正向、负向调控元件,最后,对关键调控区域内的转录因子及其结合位点进行了分析,试验结果为进一步研究猪STAB2基因的表达调控机制以及其在调控猪抗病和肌肉品质中的功能提供了理论依据。
本试验首先对克隆得到的STAB2基因转录起始位点上游长1 997 bp的候选启动子区序列中核心启动子的分布情况进行分析发现,该区域包含4个核心启动子,分别位于转录起始位点上游-1 982--1 933 bp、-1 372--1 323 bp、-927--878 bp和-228--179 bp随后,对这一区域内的CpG岛进行分析,发现STAB2基因转录起始位点上游-331--14 bp处存在于一个CpG岛。最后,利用双荧光素酶报告基因载体试验证实,STAB2基因的核心启动子区位于转录起始位点上游-309--39 bp区域。因此,本研究从生物信息学和试验验证两方面均证实了STAB2基因的核心启动子区的位置,为进一步研究猪STAB2基因的结构、功能及表达机制等奠定了基础。
众所周知,基因转录的调节不仅依赖于与DNA结合的转录因子,还涉及特定转录因子结合其靶DNA的能力[12]。而启动子区的CpG位点在这一调节过程中扮演着重要角色,其极易发生甲基化,而CpG位点的甲基化可通过影响转录因子与其靶DNA的结合能力[13]及染色质的凝聚[12]等来调控转录因子与其靶DNA的结合能力,进而调控基因的表达。本研究发现STAB2基因的核心启动子区存在1个CpG岛,位于-331--14 bp(318 bp),这为研究STAB2基因核心启动子区甲基化程度与STAB2基因表达之间的关系奠定了基础。
本研究所构建的5个5′端缺失的重组载体中,pGL4.17-1045的荧光素酶活性显著高于其他载体,而pGL4.17-1997、pGL4.17-1506和pGL4.17-309之间的荧光素酶活性差异不显著,这说明在转录起始位点上游-1 506--1 045 bp可能存在一个抑制子从而使得pGL4.17-1506的荧光素酶活性与pGL4.17-1045相比显著降低,而在-1 045--309 bp则存在一个增强子使得pGL4.17-1045的荧光素酶活性与pGL4.17-309相比显著升高。这说明,STAB2基因的核心启动子区位于转录起始位点上游-309--39 bp区域,正向调控元件位于-1 045--309 bp区域,而负向调控元件位于-1 506--1 045 bp区域。
对STAB2基因的核心启动子区域进行转录因子及其结合位点的预测,总共发现了72个转录因子结合位点,如BRCA1、Hltf、Klf4、KLF5、NFATC2、ZNF354C等。这些转录因子或发挥转录激活功能,或发挥转录抑制功能,共同调控了STAB2基因的转录。BRCA1是一种肿瘤抑制子,其主要通过在DNA修复、细胞周期检查点控制、转录调控、细胞凋亡以及mRNA剪切等过程发挥作用来实现其维持基因组稳定性的功能[14]。转录因子Hltf具有解旋酶和ATP酶活性,可通过改变靶基因周围的染色质结构来调节靶基因的转录[15]。Klf4是一种广泛表达的转录因子,参与调控细胞增殖和分化等重要生理进程,且是皮肤屏障功能正常发育所必需的,缺失Klf4的小鼠在出生后不久就会因皮肤屏障功能丧失而死亡[16]。KLF5主要发挥转录活化的作用,既可以通过介导细胞周期蛋白D1的表达来促进细胞的增殖[17],也可以通过阻断ERK/p38MAPK信号通路来抑制细胞的凋亡[18]。NFATC2在免疫细胞的活化中具有重要作用[19],NFATC2缺陷型小鼠体内的炎性反应以及肽段特异性CTL应答均会严重受损,具体表现为早期炎性因子的生成降低、淋巴结肿大、朗格汉斯细胞迁移受损以及肥大细胞功能受损等[20]。ZNF354C主要作为转录抑制子在成骨细胞的分化中发挥重要作用[21],同时研究也发现,ZNF354C可转录出2种转录本,其中一种在骨骼肌中高表达,推测其可能在机体出生后的肌肉生成以及卫星细胞的自我更新中发挥重要功能[22]。综上,笔者发现,这些转录因子的功能主要集中在调控细胞增殖、细胞免疫及肌细胞生成等相关基因的转录上,这与前人报道的STAB2基因的功能相一致。
本试验利用基因克隆、双荧光素酶报告基因系统以及生物信息学分析等方法,克隆得到了STAB2基因的候选启动子序列并分析了其序列特征,确定了STAB2基因的核心启动子区域及关键调控区域,分析了关键调控区域内的相关转录因子及其结合位点,为进一步研究猪STAB2基因的表达调控机制以及其在调控猪抗病和肌肉品质中的功能提供了理论依据。
[1] Harris E N, Weigel P H. The ligand-binding profile of HARE: hyaluronan and chondroitin sulfates A, C, and D bind to overlapping sites distinct from the sites for heparin, acetylated low-density lipoprotein, dermatan sulfate, and CS-E[J]. Glycobiology, 2008, 18(8): 638-648.doi: 10.1093/glycob/cwn045.
[2] Tamura Y, Adachi H, Osuga J, Ohashi K, Yahagi N, Sekiya M, Okazaki H, Tomita S, Iizuka Y, Shimano H, Nagai R, Kimura S, Tsujimoto M, Ishibashi S. FEEL-1 and FEEL-2 are endocytic receptors for advanced glycation end products[J]. Journal of Biological Chemistry, 2003, 278(15): 12613-12617. doi: 10.1074/jbc.M210211200.
[3] D′souza S,Park S Y,Kim I S.Stabilin-2 acts as an engulfment receptor for the phosphatidylserine-dependent clearance of primary necrotic cells[J].Biochemical and Biophysical Research Communications, 2013, 432(3): 412-417. doi: 10.1016/j.bbrc.2013.01.133.
[4] Adachi H, Tsujimoto M. FEEL-1, a novel scavenger receptor with in vitro bacteria-binding and angiogenesis-modulating activities[J]. Journal of Biological Chemistry, 2002, 277(37): 34264-34270. doi: 10.1074/jbc.M204277200.
[5] Jung M Y, Park S Y, Kim I S.Stabilin-2 is involved in lymphocyte adhesion to the hepatic sinusoidal endothelium via the interaction with alphaMbeta2 integrin[J]. Journal of Leukocyte Biology, 2007, 82(5):1156-1165. doi: 10.1189/jlb.0107052.
[6] Park S Y, Jung M Y, Kim H J, Lee S J, Kim S Y, Lee B H, Kwon T H, Park R W, Kim I S. Rapid cell corpse clearance by stabilin-2, a membrane phosphatidylserine receptor[J]. Cell Death and Differentiation, 2008, 15(1): 192-201. doi: 10.1038/sj.cdd.4402242.
[7] Han M W, Lee J C, Park S Y, Kim Y M, Cho K J, Kim S W, Lee M, Nam S Y, Kim I S, Kim S Y. Homotypic interaction of Stabilin-2 plays a critical role in lymph node metastasis of tongue cancer[J]. Anticancer Research,2016, 36(12): 6611-6618. doi: 10.21873/anticanres.11267.
[8] Géraud C, Mogler C, Runge A, Evdokimov K, Lu S, Schledzewski K, Arnold B, Hämmerling G, Koch P S, Breuhahn K, Longerich T, Marx A, Weiss C, Damm F, Schmieder A, Schirmacher P, Augustin H G, Goerdt S. Endothelial transdifferentiation in hepatocellular carcinoma: loss of Stabilin-2 expression in peri-tumourous liver correlates with increased survival[J]. Liver International, 2013, 33(9): 1428-1440. doi: 10.1111/liv.12262.
[9] Park S Y, Yun Y, Lim J S, Kim M J, Kim S Y, Kim J E, Kim I S. Stabilin-2 modulates the efficiencyof myoblast fusion during myogenic differentiation and muscle regeneration[J]. Nature Communications,2016, 7: 10871. doi: 10.1038/ncomms10871.
[10] Hamoud N, Tran V, Aimi T, Kakegawa W, Lahaie S, Thibault M P, Pelletier A, Wong G W, Kim I S, Kania A, Yuzaki M, Bouvier M, C té J F. Spatiotemporal regulation of the GPCR activity of BAI3 by C1qL4 and Stabilin-2 controls myoblast fusion[J]. Nature Communication, 2018, 9(1): 4470. doi: 10.1038/s41467-018-06897-5.
té J F. Spatiotemporal regulation of the GPCR activity of BAI3 by C1qL4 and Stabilin-2 controls myoblast fusion[J]. Nature Communication, 2018, 9(1): 4470. doi: 10.1038/s41467-018-06897-5.
[11] 龙熙, 柴捷, 赵久刚, 蓝静, 郭宗义, 张廷焕. 猪EIF2S3基因启动子的克隆、活性及转录调控元件分析[J]. 华北农学报, 2018, 33(S1):38-44. doi: 10.7668/hbnxb.2018.S1.007.
Long X,Chai J,Zhao J G,Lan J,Guo Z Y,Zhang T H.Cloning,transcriptional activity and regulatory elements analysis of porcine EIF2S3 gene promoter[J]. Acta Agriculturae Boreali-sinica, 2018, 33(S1):38-44.
[12] Attwood J T, Yung R L, Richardson B C. DNA methylation and the regulation of genetranscription[J]. Cellular and Molecular Life Sciences, 2002, 59(2): 241-257. doi: 10.1007/s00018-002-8420-z.
[13] Banovich N E, Lan X, McVicker G, van de Geijn B, Degner J F, Blischak J D, Roux J, Pritchard J K, Gilad Y. Methylation QTLs are associated with coordinated changes in transcription factor binding, histone modifications, and gene expression levels[J]. Plos Genetics, 2014,10(9): e1004663. doi: 10.1371/journal.pgen.1004663.
[14] Savage K I, Harkin D P.BRCA,a ′complex′ protein involved in the maintenance of genomic stability[J]. Febs Journal, 2015, 282(4): 630-646. doi: 10.1111/febs.13150.
[15] Poole L A, Cortez D. Functions of SMARCAL1, ZRANB3, and HLTF in maintaining genome stability[J]. Critical Reviews in Biochemistry and Molecular Biology, 2017, 52(6): 696-714. doi: 10.1080/10409238.2017.1380597.
[16] Segre, J A, Bauer C, Fuchs E. Klf4 is a transcription factor required for establishing the barrier function of the skin[J]. Nature Genetics, 1999, 22(4): 356-360. doi: 10.1038/11926.
[17] Liu Y, Wen J K, Dong L H, Zheng B, Han M. Krüppel-like factor (KLF) 5 mediates cyclin D1expression and cell proliferation via interaction with c-Jun in Ang II-induced VSMCs[J]. Acta Pharmacologica Sinica, 2010, 31(1): 10-18. doi: 10.1038/aps.2009.185.
[18] Rane M J, Zhao Y G, Cai L. Krupsilonppel-like factors (KLFs) in renal physiology and disease[J]. EbioMedicine, 2019, 40: 743-750. doi: 10.1016/j.ebiom.2019.01.021.
[19] Walters R D, Drullinger L F, Kugel J F, Goodrich J A. NFATc2 recruits cJun homodimers to an NFAT site to synergistically activate interleukin-2 transcription[J]. Molecular Immunology, 2013,56(1-2): 48-56. doi: 10.1016/j.molimm.2013.03.022.
[20] Becker M, Heib V, Klein M, Doener F, Bopp T, Taube C, Radsak M, Schild H, Schmitt E, Stassen M. Impaired mast cell-driven immune responses in mice lacking the transcription factor NFATc2[J].The Journal of Immunology, 2009, 182(10): 6136-6142. doi: 10.4049/jimmunol.0802878.
[21] Fei Q, Lin J S, Meng H, Wang B Q, Yang Y, Wang Q, Su N, Li J J, Li D. Identification of upstream regulators for synovial expression signature genes in osteoarthritis[J]. Joint Bone Spine, 2016,83(5): 545-551. doi: 10.1016/j.jbspin.2015.09.001.
[22] Gao L, Sun C, Qiu H L, Liu H, Shao H J, Wang J, Li W X. Cloning and characterization of a novel human zinc finger gene, hKid3, from a C2H2-ZNF enriched human embryonic cDNA library[J]. Biochemical and Biophysical Research Communications, 2004, 325(4): 1145-1152. doi: 10.1016/j.bbrc.2004.10.158.